Method Article
Análise por microscopia confocal da classificação de proteínas para plasmodesmos em Nicotiana benthamiana
Neste Artigo
Resumo
Este protocolo descreve a seleção de marcadores plasmodésmicos ideais para análises baseadas em microscopia confocal de direcionamento de proteínas para plasmodesmos durante interações vírus-plasmodesmos ou transporte plasmodesmal.
Resumo
Plasmodesmos são nanoporos membranosos que conectam o citoplasma de células vegetais adjacentes e permitem o tráfego célula a célula de nutrientes, macromoléculas, bem como vírus invasores. Os plasmodesmos desempenham papéis fundamentais na regulação da comunicação intercelular, contribuindo para o desenvolvimento das plantas, respostas ambientais e interações com patógenos virais. A descoberta da localização plasmodésmica de proteínas vegetais ou virais pode fornecer informações funcionais úteis sobre a proteína e é importante para a compreensão dos mecanismos das interações planta-vírus. Para facilitar esses estudos, descrevemos um protocolo para análise baseada em microscopia confocal de diferentes proteínas de direcionamento plasmodesmal para selecionar o melhor marcador plasmodesmal para estudar as interações vírus-plasmodesmos ou transporte plasmodesmal. Especificamente, as análises desses eventos são ilustradas usando a proteína de movimento célula a célula (MP) do vírus de limpeza da veia do nabo (TVCV), a proteína localizada em plasmodesmos de Arabidopsis 5 (PDLP5) e a proteína de ligação à calose de plasmodesmos 1 (PDCB1). Os dados de localização plasmodésica da proteína são analisados em paralelo com a visualização global dos plasmodesmos usando coloração com azul de anilina dos tecidos amostrados. Essas abordagens podem ser facilmente adaptadas para analisar a localização plasmodésmica de qualquer proteína celular ou patogênica na planta.
Introdução
Os plasmodesmos (PD) desempenham um papel fundamental no controle do desenvolvimento vegetal, respostas ambientais e interações com patógenos virais por meio da regulação da comunicação intercelular 1,2. A DP se forma inicialmente durante a citocinese, com centenas de PD inseridas na nova célula entre as duas células-filhas, fornecendo assim os canais para a comunicação célula a célula 3,4. A DP é uma estrutura rica em membrana, contendo a membrana derivada do retículo endoplasmático (RE), um desmotúbulo trans-PD, na parte central dos poros que são revestidos pela membrana plasmática 3,4. Abordagens proteômicas comparativas identificaram inúmeras proteínas funcionais da DP, incluindo β-1,3-glucanases (BGs), calose sintases (CALSs), proteínas localizadas em plasmodesmos (PDLPs), proteínas de ligação à calose (PDCBs), proteínas da região transmembrana de múltiplos domínios C2 (MCTPs)3 e quinases semelhantes a receptores repetidos ricos em leucina (RLK)5. Recentemente, Kirk et al. desenvolveram uma ferramenta denominada plasmodesmos in silico proteome 1 (PIP1), que possibilitou prever novas proteínas PD em 22 espécies de plantas6. A DP varia em permeabilidade e estrutura durante o desenvolvimento da planta e a resposta a vários estresses. A deposição e hidrólise da calose (β-1,3-glucana) na região do pescoço ao redor da DP é um dos mecanismos amplamente conhecidos da regulação da DP7.
Muitos micróbios patogênicos, incluindo fungos, bactérias e vírus, podem manipular a dilatação ou estrutura da DP durante sua infecção 2,8,9. Magnaporthe oryzae, o agente causador da brusone do arroz, implanta hifas invasivas intracelulares para se mover de célula para célula através da DP8. Um patógeno bacteriano Pseudomonas syringae pv. o tomate requer uma proteína efetora HopO1-1 para movimento intercelular e disseminação na planta hospedeira por meio da interação e desestabilização do PDLP7, aumentando assim o fluxo molecular nas células vizinhas em Arabidopsis9. No entanto, os vírus de plantas são mais versáteis na regulação da DP durante sua transmissão intercelular, com a proteína de movimento viral (MP) promovendo o movimento célula a célula2. Devido à sua importante função na regulação do desenvolvimento e crescimento das plantas, bem como sua interação com micróbios patogênicos de plantas, a DP tem ganhado cada vez mais atenção nos últimos anos. Em Arabidopsis thaliana, existem dois tipos principais de proteínas funcionais de PD, PDLPs (1-8) e PDCBs (1-5), e muitas delas, por exemplo, PDLP5 1,10,11, PDLP112, PDLP613, PDLP714 e PDCB115, foram encontradas para desempenhar um papel na manipulação da permeabilidade da PD através da regulação da deposição de calose. No entanto, alguns PDLPs apresentaram redundância funcional, por exemplo, mutantes knockout de pdlp1 e pdlp1,2 não afetaram o tráfego molecular, embora mutantes knockout duplos de pdlp1,3 e pdlp2,3 tenham mostrado aumento da permeabilidade plasmodésica16. Curiosamente, a regulação negativa / nocaute ou superexpressão de PDLP5 isoladamente resulta em um aumento ou diminuição na permeabilidade plasmodésmica, respectivamente 1,17. Recentemente, Li et al. descobriram que PDLP5 e PDLP6 funcionam em diferentes interfaces celulares13. Esses resultados indicam que o PDLP5 pode ter funções não redundantes com outros PDLPs.
Devido à função crítica da DP na comunicação intercelular, desenvolvemos um protocolo para implantar as proteínas PD de plantas PDLP5 e PDCB1 e a proteína de movimento célula a célula viral (MP) do vírus de limpeza de veias de nabo (TVCV) como marcadores de DP simples, convenientes e confiáveis para experimentação de biologia celular. Para verificação posterior, a visualização da DP usando coloração com azul de anilina dos tecidos amostrados ocorreu em paralelo. Os protocolos descritos para localização de PD de PDLP5, PDCB1 e TVCV MP podem ser facilmente adaptados para analisar a localização potencial de PD de qualquer proteína celular ou derivada de patógenos em plantas vivas.
Protocolo
Os detalhes dos reagentes e dos equipamentos utilizados neste estudo estão listados na Tabela de Materiais.
1. Crescimento das plantas
- Cultive sementes de Nicotiana benthamiana em solo úmido em uma câmara de ambiente controlado a 23 ° C sob 16 h de luz e 8 h de escuridão.
- Após cerca de 2 semanas, transfira cuidadosamente as mudas com os pellets de turfa ao redor de suas raízes para vasos maiores e continue o crescimento nas mesmas condições por 4-5 semanas para os experimentos de agroinfiltração.
NOTA: Não use as plantas quando elas começarem a florescer, pois o acúmulo de GFP diminuirá no estágio18 da inflorescência.
2. Construção vetorial
- Use PCR para amplificar as sequências de codificação de interesse com a DNA polimerase de alta fidelidade Q5 e cloná-las no vetor de entrada pDONR207 pela reação BP19 usando um kit de clonase BP disponível comercialmente.
- Transfira os genes-alvo nos plasmídeos de entrada resultantes para o vetor de destino pPZP-RCS2A-nptII-DEST-EGFP-N1 pela reação LR19 usando um kit de clonase LR disponível comercialmente para produzir plasmídeos com a etiqueta EGFP fundida ao terminal C das proteínas-alvo.
- Verificar todas as construções por PCR e sequenciamento19.
3. Agroinfiltração
- Estrias Agrobacterium tumefaciens EHA105 contendo diferentes vetores em placas de ágar LB e incubadas por 2 dias sob 28 °C.
- Transfira uma única colônia para 2 mL de caldo LB e incube durante a noite a 28 ° C com agitação (250 rpm).
- Remover 1 mL da cultura durante a noite, seguido pela adição de 4 mL de caldo fresco LB e recultivar por 1 h nas mesmas condições.
- Ajustar a suspensão bacteriana para OD600 = 0,1 (OD600 = 0,2 para a co-expressão de duas proteínas) com tampão de infiltração (MgCl2, 10 mM; MES, 10 mM, pH 5,6).
- Misture diferentes suspensões bacterianas na proporção de 1:1 (v/v) para obter uma concentração final de OD600 = 0,1. (opcional).
- Incubar a suspensão bacteriana à temperatura ambiente durante 3 h com agitação suave.
- Infiltre a superfície abaxial das folhas totalmente expandidas de diferentes plantas com uma seringa descartável sem agulha de 1 mL e marque a área infiltrada (uma cor mais escura do que o tecido circundante não infiltrado) com uma caneta à prova d'água.
- Prossiga para a seção Microscopia confocal deste protocolo (etapa 5).
NOTA: Outras cepas de Agrobacterium tumefaciens adequadas para a transformação de espécies vegetais de interesse também podem ser utilizadas.
4. Mancha azul de anilina
- Adicione uma alíquota de 200 μL de azul de anilina a 1% (em tampão fosfato de potássio 50 mM, pH 8,0) a uma lâmina de microscópio20.
- Extirpar a área infiltrada de aproximadamente 0,5 cm × 0,5 cm, longe da nervura, com uma lâmina, e transferir as amostras de tecido foliar para a solução de azul de anilina (lado abaxial para cima) na lâmina do microscópio, e certifique-se de que as amostras de tecido foliar estejam submersas na solução de azul de anilina e, em seguida, cubra-a com uma lamínula (22 mm x 50 mm).
- Coloque as lâminas do microscópio com as amostras em um dessecador conectado a uma bomba de vácuo e evacue por 2 min (<0,8 Pa), seguido de uma liberação lenta da pressão e incubação no escuro por 30 min em temperatura ambiente.
- Visualize o sinal fluorescente do azul de anilina sob um microscópio confocal de varredura a laser com software compatível.
NOTA: Aqui, foi utilizado o método de coloração com azul de anilina de Huang et al.20 . Em uma modificação desta técnica, o mesmo experimento foi realizado sem a etapa de vácuo de 2 min, produzindo resultados semelhantes e sugerindo que a secagem a vácuo não é essencial para a coloração com anilina. Além disso, a concentração da mancha azul de anilina e o tempo de coloração podem variar de acordo com a fonte do tecido foliar, etc.
5. Microscopia confocal
- Colha duas folhas de duas plantas em momentos diferentes após a infiltração. Corte a zona de infiltração em fatias de aproximadamente 0,5 cm × 0,5 cm de distância da veia com uma lâmina.
- Coloque as amostras de tecido em uma gota de água estéril (lado abaxial para cima) na parte central de uma lâmina de microscópio e cubra-as com uma lamínula (22 mm x 50 mm), tomando cuidado para evitar bolhas.
- Certifique-se de que os comprimentos de onda de excitação para a detecção de sinais CFP, GFP e RFP sejam 405 nm, 488 nm e 561 nm, respectivamente, e os filtros de emissão para detecção sejam 410-602 nm para CFP e 400-602 nm para EGFP e RFP, com o orifício 1 UA e o ganho mestre definido como 769 V.
- Visualize o sinal fluorescente de etiquetas autofluorescentes na área infiltrada usando um microscópio confocal de varredura a laser com software compatível em 1 dia, 2 dias, 3 dias, 5 dias, 7 dias e 10 dias após a infiltração.
- Use uma lente objetiva de 10x e filtros GFP para localizar células com o sinal fluorescente e, em seguida, mude para uma lente objetiva de 40x para visualização da localização subcelular e gravação de imagens.
- Colete 20 imagens para cada condição, usando pelo menos duas réplicas biológicas independentes.
- Pontue a localização da PD da proteína testada com base na aparência puntiforme diagnóstica do sinal na periferia da célula21,22.
NOTA: Ao detectar a localização subcelular de uma proteína desconhecida, recomenda-se o uso de pelo menos três plantas.
6. Análise dos dados
- Use o software Fiji para dividir canais e adicionar a barra de escala às imagens para visualização.
- Use o software Fiji para dividir os canais antes de medir o valor médio da escala de cinza de cada imagem para avaliar a intensidade de fluorescência GFP (para MP, PDCB1 e PDLP5) ou CFP (para coloração com azul de anilina) em diferentes pontos de tempo.
- Use o software Fiji para dividir os canais antes da normalização das imagens de GFP (para MP, PDCB1 e PDLP5) ou CFP (para coloração com azul de anilina). A área da imagem foi medida e os pontos de PD foram contados manualmente. O número de pontos de DP por 100 μm2 foi calculado.
- Use ANOVA de duas vias com o teste de comparações múltiplas de Tukey23 para determinar os valores de P entre as diferentes amostras e diferentes pontos de tempo com software estatístico e gráfico.
NOTA: Para uma imagem de fluorescência de canal único, o valor da escala de cinza de cada pixel representa a intensidade de fluorescência desse ponto24. Aqui, o valor médio da escala de cinza foi usado para avaliar a intensidade de fluorescência de cada amostra em diferentes momentos.
Resultados
Para facilitar os estudos da função da DP na fisiologia vegetal e nas interações com patógenos, três proteínas de referência simples e confiáveis foram desenvolvidas para a localização da DP. Duas proteínas PD celulares e uma proteína MP derivada do patógeno codificada pelo tobamovírus vegetal TVCV foram selecionadas. A localização subcelular dessas proteínas foi visualizada usando um repórter autofluorescente EGFP fundido ao terminal C de cada proteína. Em uma abordagem alternativa, os DP foram visualizados usando coloração com azul de anilina dos depósitos de calose associados à DP. Os experimentos mostraram que tanto PDLP5 quanto MP exibiram localização PD pontilhada, com MP também localizada no núcleo da célula (Figura 1A), como observado anteriormente25. Em contraste, PDCB1 pareceu particionar entre PD e ER (Figura 1A). Para pesquisadores menos experientes na interpretação da localização subcelular de proteínas marcadas com GFP, a Figura 1A também ilustra o padrão nucleocitoplasmático característico do EGFP expresso em células vegetais. Para confirmar o sinal ER do PDCB1, co-expressamos PDCB1 com um marcador ER e descobrimos que o sinal PDCB1 se sobrepunha ao sinal do marcador ER (Figura 1B). Para avaliar a eficiência da localização de PD das proteínas testadas, foi calculada a porcentagem de células que expressam o sinal fluorescente que exibiram localização de PD desse sinal (Tabela 1).
Para a identificação da localização subcelular empregando proteínas de referência, é útil entender a escala de tempo da expressão da proteína de referência e as possíveis mudanças de sua localização. A Figura 2 mostra essa cinética para MP, PDLP5 e PDCB1 quando os próprios PD foram visualizados pela coloração com azul de anilina. Tanto o MP quanto o PDLP5 começaram a se acumular no PD já no pós-infiltração de um dia (dpi), e esse acúmulo atingiu um máximo aparente em 2 dpi, permanecendo estável por mais 5 dias, após os quais a intensidade do sinal diminuiu, provavelmente devido à degradação da proteína. O curso de tempo da classificação subcelular do PDCB1 foi mais complexo: nos primeiros 3 dpi, o PDCB1 exibiu um sinal ER comparativamente forte, que obscureceu quase completamente a localização do sinal específico do PD, enquanto que em 5 dpi, este último sinal tornou-se mais pronunciado (Figura 2), embora em menos células. Curiosamente, o surgimento do padrão de localização PD claro de PDCB1 após 5 dpi coincidiu com substancialmente menos células mostrando o sinal EGFP (Figura Suplementar 1). Assim, PDLP5 e MP podem funcionar como proteínas de referência de PD com pouco background na maioria dos outros compartimentos celulares, com uma janela de tempo de visualização ideal de 2-5 dpi. PDCB1, por outro lado, não é tão adequado quanto um marcador de DP.
Verificou-se, então, que os marcadores de DP mantêm sua especificidade de localização subcelular na presença de outras proteínas de DP. Para este fim, MP-EGFP ou PDLP5-EGFP foi co-expresso com um PD-localizando26 MP de outro vírus de planta, o vírus do mosaico do tabaco (TMV) marcado com CFP. A Figura 3 mostra que, em ambos os casos, as proteínas co-expressas exibiram um padrão claro de localização da DP. Da mesma forma, a coloração histológica de azul de anilina específica para PD dos tecidos que expressam MP-EGFP ou PDLP5-EGFP não detectou interferência na localização de PD dos sinais de EGFP e azul de anilina (Figura 3).
Os estudos de localização de DP geralmente se beneficiam de uma estimativa mais quantitativa da expressão geral da proteína de referência e do sinal de PD. Essa abordagem de quantificação é ilustrada na Figura 4 e na Figura 5. Para a quantificação da expressão geral, foi medida a intensidade do sinal de PDLP5 e MP. Ambas as proteínas apresentaram sinais fortes em todos os momentos de visualização durante o período de 10 dpi, com intensidade máxima de sinal em 3 dpi para PDLP5 e em 2 dpi para MP (Figura 4). Não houve diferenças estatisticamente significativas na intensidade do sinal entre PDLP5 e MP em nenhum dos momentos de visualização. A intensidade do sinal PDCB1 exibiu maior flutuação entre os momentos, com o sinal mais forte observado em 7 dpi (Figura 4), embora tenha sido difícil coletar dados estatisticamente significativos neste momento devido à escassez de células de expressão. Assim, os primeiros 3 dpi representam o tempo ideal para detectar a expressão de PDCB1. Como esperado, as amostras de coloração com azul de anilina exibiram um sinal mais estável do que as proteínas expressas transitoriamente (Figura 4).
Os pontos de PD formados por PDLP5 e MP foram então quantificados. O PDLP5 mostrou um número estável de pontos de DP durante todo o período de observação, sem diferenças estatisticamente significativas entre os dois pontos de tempo testados (Figura 5). O maior número de pontos de PD formados por MP foi detectado em 2 dpi, e eles permaneceram relativamente estáveis até o final do período de observação. A comparação dos números de pontos de DP entre PDLP5 e MP não revelou diferenças estatisticamente significativas na maioria dos momentos de amostragem (Figura 5). O número de pontos de PD corados com azul de anilina também permaneceu estável, embora tenha diminuído ligeiramente a 10 dpi. Os números de pontos de PD decorados por azul de anilina, PDLP5 ou MP foram semelhantes entre si com significância estatística (Figura 5), consistente com a especificidade de PD desses marcadores. Os números de pontos de PD para PDCB1 não foram analisados porque, em pontos de tempo anteriores, o sinal de PD foi obscurecido pelo sinal de ER e, em pontos de tempo posteriores, foi observado em apenas algumas células relativamente poucas (ver Figura 2).

Figura 1: Localização de plasmodesmos (PD) de TVCV MP, PDLP5 e PDCB1 expressos transitoriamente em Nicotiana benthamiana e coloração de azul de anilina de PD. (A) Localização de PD de MP-EGFP, PDLP5-EGFP e PDCB1-EGFP e coloração de anilina. O sinal EGFP está em verde, o sinal azul de anilina está em azul e a autofluorescência de plastídio está em vermelho. (B) Co-expressão de MP-EGFP, PDLP5-EGFP e PDCB1-EGFP com o marcador de retículo endoplasmático (ER) marcado com mRFP. As imagens foram registradas 2 dias após a infiltração (dpi) e são seções confocais únicas representativas de dois experimentos independentes (N = 20 imagens de 2 plantas). O sinal EGFP está em verde, o sinal mRFP está em roxo e a autofluorescência de plastídio está em vermelho. As setas amarelas indicam PDCB1 localizado no ER. Barras de escala = 5 μm. Clique aqui para ver uma versão maior desta figura.

Figura 2: Curso temporal da localização da DP de TVCV MP, PDLP5 e PDCB1 expressos transitoriamente em Nicotiana benthamiana e da coloração com azul de anilina da DP. As imagens foram gravadas a 2 dpi e são seções confocais únicas representativas de dois experimentos independentes (N = 20 imagens de 2 plantas para todos os sistemas, exceto 7 dpi e 10 dpi para PDCB1-EGFP, em que menos células exibiram o sinal). O sinal EGFP está em verde e o sinal azul de anilina está em azul. Barras de escala = 5 μm. Clique aqui para ver uma versão ampliada desta figura.
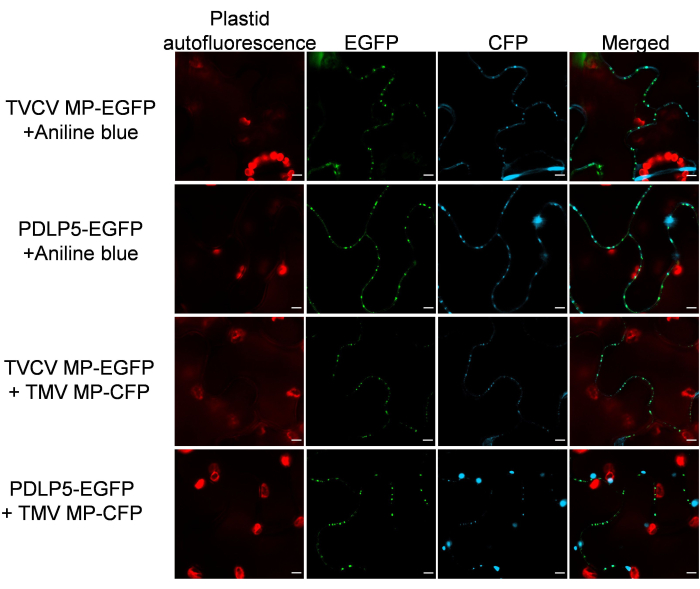
Figura 3: TVCV MP e PDLP5 mantêm sua localização PD após a co-expressão com um MP TMV localizador de PD e cocoloração com azul de anilina. As imagens foram gravadas a 2 dpi e são seções confocais únicas representativas de N ≥ 10 imagens de 4-6 plantas. O sinal EGFP está em verde, o sinal CFP e o sinal azul de anilina estão em azul e a autofluorescência do plastídio está em vermelho. Barras de escala = 5 μm. Clique aqui para ver uma versão ampliada desta figura.
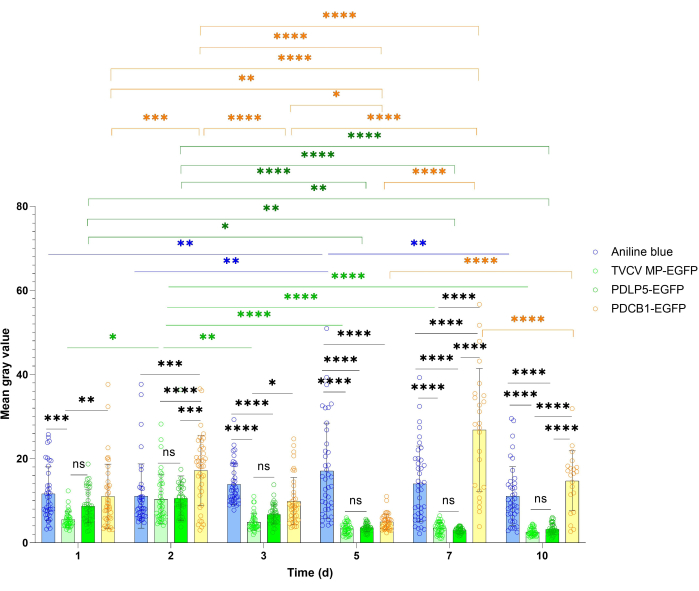
Figura 4: Curso temporal da intensidade de fluorescência de TVCV MP, PDLP5 e PDCB1 expressos transitoriamente na coloração Nicotiana benthamiana e azul de anilina PD. Os dados quantitativos foram coletados dos experimentos descritos na Figura 2. As barras de erro representam o desvio padrão da média de vários experimentos. Os pontos de dados individuais são indicados como a média para cada medição. As diferenças entre os valores médios avaliados pela ANOVA de duas vias com o teste de comparações múltiplas de Tukey são estatisticamente significativas para os valores de P. *P < 0,05, **P < 0,01 e ***P < 0,001, ****P < 0,0001; ns, não estatisticamente significativo. Barras azuis, coloração de anilina; barras verdes claras, MP-EGFP, barras verdes escuras, PDLP5-EGFP; barras amarelas, PDCB1-EGFP. Clique aqui para ver uma versão maior desta figura.

Figura 5: Curso temporal da formação de pontos de PD por TVCV MP e PDLP5 expressos transitoriamente em Nicotiana benthamiana e por coloração com azul de anilina de PD. Os dados quantitativos foram coletados dos experimentos descritos na Figura 2. As barras de erro representam o desvio padrão da média de vários experimentos. Os pontos de dados individuais são indicados como a média para cada medição. As diferenças entre os valores médios avaliados pela ANOVA de duas vias com o teste de comparações múltiplas de Tukey são estatisticamente significativas para os valores de P. *P < 0,05, **P < 0,01, ***P < 0,001, ****P < 0,0001. Barras azuis, coloração de anilina; barras verdes claras, MP-EGFP, barras verdes escuras, PDLP5-EGFP. Clique aqui para ver uma versão maior desta figura.
| Experiência I | ||||
| Total de células | Células com localização PD | Células com sinal misto | Células com localização de DP (%) | |
| Azul de anilina | 40 | 40 | 4 | 100 |
| MP-EGFP | 40 | 40 | 4 | 100 |
| PDCB1-EGFP | 40 | 3 | 38 | 7.5 |
| PDLP5-EGFP | 40 | 40 | 0 | 100 |
| Experiência II | ||||
| Total de células | Células com localização PD | Células com sinal misto | Células com localização de DP (%) | |
| Azul de anilina | 40 | 40 | 8 | 100 |
| MP-EGFP | 40 | 39 | 8 | 97.5 |
| PDCB1-EGFP | 40 | 12 | 31 | 30 |
| PDLP5-EGFP | 40 | 39 | 2 | 97.5 |
Tabela 1: Eficiência da localização de DP.
Figura suplementar 1: Localização PD de TVCV MP, PDLP5 e PDCB1 expressos transitoriamente em Nicotiana benthamiana. As imagens foram gravadas a 5 dpi (A), 7 dpi (B) e 10 dpi (C). O sinal EGFP é verde e a autofluorescência plastidial é vermelha. Barras de escala = 20 μm. Clique aqui para baixar este arquivo.
Discussão
Quaisquer estudos biológicos celulares de comunicação intercelular de plantas e transporte célula a célula durante o desenvolvimento e morfogênese normais da planta, bem como durante as interações planta-patógeno, requerem a detecção e monitoramento da classificação de proteínas - endógenas e codificadas por patógenos - para conexões intercelulares de plantas, os plasmodesmos (PD). Esses experimentos seriam substancialmente facilitados pelo uso de proteínas de referência, endógenas ou derivadas de patógenos, que se localizam fiel e consistentemente na DP, decorando e visualizando essas estruturas. Essa necessidade é atendida aqui selecionando três proteínas localizadoras de PD conhecidas: Arabidopsis PDLP5 e PDCB1 e MP do patógeno tobamoviral TVCV. Essas proteínas foram marcadas em seus terminais C com o repórter autofluorescente EGFP, e a capacidade das proteínas de fusão resultantes de classificar para PD foi determinada após sua expressão transitória em folhas de Nicotiana benthamiana e análises de microscopia confocal dos tecidos expressantes. Para visualização geral da DP nesses tecidos, foi utilizado o método padrão de coloração com azul de anilina.
Acredita-se que os MPs virais de plantas se enquadrem em dois grupos principais em relação aos seus efeitos na estrutura e integridade do PD: MPs que aumentam a permeabilidade do PD de forma reversível e sem quaisquer alterações estruturais no PD e MPs que bloqueiam o PD modificando sua estrutura. Os MPs de tobamovírus, como o TVCV, pertencem ao grupo PM do primeiro grupo, posicionando-os como marcadores atraentes de DP para estudos de localização subcelular27,28.
Esses experimentos posicionaram PDLP5-EGFP e MP-EGFP como marcadores biológicos confiáveis e reprodutíveis de DP, com a eficiência de detecção de DP comparável à do corante azul de anilina. A localização de PD de PDLP5-EGFP e MP-EGFP foi detectada durante todo o período de observação de 10 dpi, com o tempo ideal para observar o padrão de localização característico de PD entre 2 dpi e 3 dpi. Embora a expressão do promotor 35S possa resultar em silenciamento pós-transcricional do gene expresso, isso geralmente ocorre após expressão prolongada em plantas transgênicas transformadas de forma estável. Por outro lado, o uso desse forte promotor constitutivo é importante para a fácil detecção da proteína marcadora. Se a expressão da proteína de referência diminuir após 7-10 dpi, períodos mais curtos, ou seja, 2-3 dpi, devem ser usados. Diferenças significativas no padrão geral de localização subcelular entre PDLP5 e MP não foram detectadas aqui, exceto que o MP se acumulou no núcleo da célula além do PD. Por outro lado, o PDCB1 não é recomendado para uso como marcador de DP em si devido à complexidade de seu padrão de localização subcelular, que inclui localização substancial no RE.
A facilidade de detecção do sinal de fluorescência EGFP também dependia do estágio de crescimento da planta; especificamente, em N. benthamiana, o menor acúmulo de sinal de EGFP foi observado no estágio de inflorescência18.
Este estudo teve como objetivo desenvolver os marcadores de localização da DP ao máximo exclusivos para outros compartimentos subcelulares. Assim, seu uso como referência para a localização de proteínas de interesse com capacidades adicionais de localização subcelular deve ser combinado com a expressão de outros marcadores subcelulares, como aqueles específicos para o RE, mitocôndrias, membrana plasmática, etc. Nesse caso, a inclusão de células adicionais de Agrobacterium para a expressão desses marcadores deve ser levada em consideração no cálculo da concentração final das culturas de agroinfiltração, ou seja, OD600 das culturas líquidas. Obviamente, a concentração celular e os tempos de crescimento das plantas devem ser adaptados para expressão máxima e detecção de padrões de localização de DP em outras espécies de plantas.
Divulgações
Os autores declaram não haver interesses conflitantes.
Agradecimentos
O trabalho no laboratório VC foi apoiado por doações do NIH (R35GM144059), NSF (MCB 1913165 e IOS 1758046) e BARD (IS-5276-20) para VC. Os financiadores não tiveram nenhum papel no desenho do estudo, coleta e interpretação de dados, ou na decisão de publicar.
Materiais
| Name | Company | Catalog Number | Comments |
| ABT AC 1 phase motor | BRANDTECH | ABF63/4C-7RQ | |
| Agrobacterium tumefaciens EHA105 | |||
| Contamination control | CCI | ||
| Gateway BP Clonase II Enzyme mix | Invitrogen | #11789020 | |
| Gateway LR Clonase II Enzyme mix | Invitrogen | #11791020 | |
| GraphPad Prism 8.0.1. | GraphPad Software Inc. | ||
| Image J | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation | ||
| Laser scanning confocal microscope | Zeiss | LSM 900 | |
| Nicotiana benthamiana | Plant species | ||
| pDONR207 | Invitrogen | #12213013 | |
| Q5 High-Fidelity DNA Polymerase | NEB | #M0491S |
Referências
- Lee, J., et al. A plasmodesmata-localized protein mediates crosstalk between cell-to-cell communication and innate immunity in Arabidopsis. The Plant Cell. 23 (9), 3353-3373 (2011).
- Benitez-Alfonso, Y., Faulkner, C., Ritzenthaler, C., Maule, A. J. Plasmodesmata gateways to local and systemic virus infection. Mol Plant-Microbe Interact. 23 (11), 1403-1412 (2010).
- Bayer, E. M., Benitez-Alfonso, Y. Plasmodesmata: Channels under pressure. Annu Rev Plant Biol. 75, 21(2024).
- Maule, A. J. Plasmodesmata: Structure, function and biogenesis. Curr Opin Plant Biol. 11, 680-686 (2008).
- Fernandez-Calvino, L., et al. Arabidopsis plasmodesmal proteome. PLoS One. 6 (4), e18880(2011).
- Kirk, P., Amsbury, S., German, L., Gaudioso-Pedraza, R., Benitez-Alfonso, Y. A comparative meta-proteomic pipeline for the identification of plasmodesmata proteins and regulatory conditions in diverse plant species. BMC Biol. 20, 128(2022).
- Levy, A., Erlanger, M., Rosenthal, M., Epel, B. L. A plasmodesmata-associated β-1,3-glucanase in Arabidopsis. The Plant J. 49 (4), 669-682 (2007).
- Kankanala, P., Czymmek, K., Valent, B. Roles for rice membrane dynamics and plasmodesmata during biotrophic invasion by the blast fungus. The Plant Cell. 19 (2), 706-724 (2007).
- Aung, K., et al. Pathogenic bacteria target plant plasmodesmata to colonize and invade surrounding tissues. The Plant Cell. 32 (3), 595-611 (2020).
- Cui, W., Lee, J. Arabidopsis callose synthases CalS1/8 regulate plasmodesmal permeability during stress. Nat Plants. 2 (5), 160(2016).
- Liu, N. J., et al. Phytosphinganine affects plasmodesmata permeability via facilitating PDLP5-stimulated callose accumulation in Arabidopsis. Mol Plant. 13 (1), 128-143 (2020).
- Caillaud, M. C., et al. The plasmodesmal protein PDLP1 localizes to haustoria-associated membranes during downy mildew infection and regulates callose deposition. PLoS Pathog. 10 (11), e1004496(2014).
- Li, Z., Su-Ling, L., Christian, M., Walley, J. W., Aung, K. Plasmodesmata-located protein 6 regulates plasmodesmal function in Arabidopsis vasculature. The Plant Cell. , (2024).
- Chen, X., et al. Arabidopsis PDLP7 modulated plasmodesmata function is related to BG10-dependent glucosidase activity required for callose degradation. Sci Bull. , (2024).
- Simpson, C., Thomas, C., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. The Plant Cell. 21 (2), 581-594 (2009).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLoS Biol. 6 (1), e7(2008).
- Lim, G., et al. Plasmodesmata localizing proteins regulate transport and signaling during systemic acquired immunity in plants. Cell Host Microbe. 19 (4), 541-549 (2016).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnol Bioeng. 96 (3), 608-614 (2007).
- Walhout, A. J. M., et al. GATEWAY recombinational cloning: Application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Huang, C., et al. dsRNA-induced immunity targets plasmodesmata and is suppressed by viral movement proteins. The Plant Cell. 35 (10), 3845-3869 (2023).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. The plasmodesmal localization signal of TMV MP is recognized by plant synaptotagmin SYTA. mBio. 9 (4), e01314-e01318 (2018).
- Ward, S. J., Ramirez, M. D., Neelakantan, H., Walker, E. A. Cannabidiol prevents the development of cold and mechanical allodynia in paclitaxel-treated female C57Bl6 mice. Anesth Analg. 113 (4), 947-950 (2011).
- Erkkilä, M. T., et al. Widefield fluorescence lifetime imaging of protoporphyrin IX for fluorescence-guided neurosurgery: An ex vivo feasibility study. J Biophotonics. 12 (6), e201800378(2019).
- Levy, A., Zheng, J. Y., Lazarowitz, S. G. The tobamovirus Turnip vein clearing virus 30-kilodalton movement protein localizes to novel nuclear filaments to enhance virus infection. J Virol. 87 (11), 6428-6440 (2013).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell-movement protein. mBio. 7 (1), e2052-e2015 (2016).
- Kumar, G., Dasgupta, I. Variability, functions and interactions of plant virus movement proteins: what do we know so far. Microorganisms. 9 (4), 695(2021).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of nondestructive cell-to-cell and systemic movement of plant viruses. Crit Rev Plant Sci. 23 (3), 195-250 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados