Method Article
Konfokalmikroskopische Analyse der Proteinsortierung zu Plasmodesmata bei Nicotiana benthamiana
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Auswahl optimaler plasmodesmaler Marker für konfokalmikroskopische Analysen von Proteinen, die während Virus-Plasmodesmata-Interaktionen oder des plasmosmodesmalen Transports auf Plasmodesmen abzielen.
Zusammenfassung
Plasmodesmen sind membranöse Nanoporen, die das Zytoplasma benachbarter Pflanzenzellen verbinden und den Zell-zu-Zell-Transport von Nährstoffen, Makromolekülen sowie eindringenden Viren ermöglichen. Plasmodesmata spielen eine grundlegende Rolle bei der Regulation der interzellulären Kommunikation und tragen zur Pflanzenentwicklung, zu Umweltreaktionen und Interaktionen mit viralen Krankheitserregern bei. Die Entdeckung der plasmosmodesmalen Lokalisation von pflanzlichen oder viralen Proteinen könnte nützliche funktionelle Informationen über das Protein liefern und ist wichtig für das Verständnis der Mechanismen von Pflanzen-Virus-Interaktionen. Um diese Studien zu erleichtern, beschreiben wir ein Protokoll für die konfokale mikroskopische Analyse verschiedener plasmodesmaler Zielproteine, um den besten plasmodesmalen Marker für die Untersuchung der Virus-Plasmodesmata-Wechselwirkungen oder des plasmodesmalen Transports auszuwählen. Konkret werden die Analysen dieser Ereignisse anhand des Zell-zu-Zell-Bewegungsproteins (MP) des Turnip-Venen-Clearing-Virus (TVCV), des Arabidopsis Plasmodesmata-Localized Protein 5 (PDLP5) und des Plasmodesmata Callose-Binding Protein 1 (PDCB1) veranschaulicht. Die plasmosmodesmalen Lokalisationsdaten des Proteins werden parallel zur globalen Visualisierung der Plasmodesmen mittels Anilinblau-Färbung der beprobten Gewebe analysiert. Diese Ansätze können leicht angepasst werden, um die plasmmodesmale Lokalisation von zellulären oder pathogenen Proteinen in planta zu analysieren.
Einleitung
Plasmodesmata (PD) spielen eine grundlegende Rolle bei der Kontrolle der Pflanzenentwicklung, der Umweltreaktionen und der Wechselwirkungen mit viralen Krankheitserregern durch die Regulation der interzellulären Kommunikation 1,2. Parkinson bildet sich zunächst während der Zytokinese, wobei Hunderte von Parkinson zwischen den beiden Tochterzellen in die neue Zelle eingefügt werden und so die Kanäle für die Zell-zu-Zell-Kommunikation liefern 3,4. PD ist eine membranreiche Struktur, die die vom endoplasmatischen Retikulum (ER) abgeleitete Membran, ein trans-PD-Desmotubulum, im zentralen Teil der Poren enthält, die von der Plasmamembranausgekleidet sind 3,4. Vergleichende proteomische Ansätze identifizierten zahlreiche funktionelle Parkinson-Proteine, darunter β-1,3-Glucanasen (BGs), Callose-Synthasen (CALSs), Plasmodesmata-lokalisierte Proteine (PDLPs), Callose-bindende Proteine (PDCBs), Transmembranregionenproteine mit mehreren C2-Domänen (MCTPs)3 und leucinreiche Repeatrezeptor-ähnliche Kinasen (RLK)5. Vor kurzem haben Kirk et al. ein Werkzeug namens Plasmodesmata in silico proteome 1 (PIP1) entwickelt, das es ermöglichte, neue Parkinson-Proteine in 22 Pflanzenartenvorherzusagen 6. PD variiert in Durchlässigkeit und Struktur während der Pflanzenentwicklung und der Reaktion auf verschiedene Stressfaktoren. Die Ablagerung und Hydrolyse von Callose (β-1,3-Glucan) in der Halsregion, die die Parkinson-Krankheit umgibt, ist einer der weithin bekannten Mechanismen der Parkinson-Regulation7.
Viele pathogene Mikroben, darunter Pilze, Bakterien und Viren, können die Erweiterung oder Struktur der Parkinson-Krankheit während ihrer Infektion manipulieren 2,8,9. Magnaporthe oryzae, der Erreger der Reisexplosion, setzt intrazelluläre invasive Hyphen ein, um durch PD8 von Zelle zu Zelle zu gelangen. Ein bakterieller Erreger Pseudomonas syringae pv. Die Tomate benötigt ein Effektorprotein HopO1-1 für die interzelluläre Bewegung und Ausbreitung in der Wirtspflanze durch Interaktion mit und Destabilisierung von PDLP7, wodurch der molekulare Fluss in benachbarten Zellen in Arabidopsis9 erhöht wird. Pflanzenviren sind jedoch vielseitiger bei der Regulierung von Parkinson während ihrer interzellulären Übertragung, wobei das virale Bewegungsprotein (MP) die Bewegung von Zelle zu Zelle fördert2. Aufgrund ihrer wichtigen Funktion bei der Regulierung der Pflanzenentwicklung und des Pflanzenwachstums sowie ihrer Interaktion mit pflanzenpathogenen Mikroben hat die Parkinson-Krankheit in den letzten Jahren zunehmend an Bedeutung gewonnen. In Arabidopsis thaliana gibt es zwei Haupttypen von PD-funktionellen Proteinen, PDLPs (1-8) und PDCBs (1-5), und viele von ihnen, z. B. PDLP5 1,10,11, PDLP112, PDLP613, PDLP714 und PDCB115, spielten eine Rolle bei der Manipulation der PD-Permeabilität durch Regulation der Calloseablagerung. Es wurde jedoch festgestellt, dass einige PDLPs eine funktionelle Redundanz aufwiesen, z.B. beeinflussten Knockout-Mutanten von pdlp1 und pdlp1,2 den molekularen Transport nicht, obwohl Doppel-Knockout-Mutanten von pdlp1,3 und pdlp2,3 eine erhöhte plasmosmodesmale Permeabilität zeigten16. Interessanterweise führt eine Herunterregulation/Knockout oder Überexpression von PDLP5 allein zu einer Erhöhung bzw. Abnahme der plasmosmodesmalen Permeabilität 1,17. Kürzlich haben Li et al. herausgefunden, dass PDLP5 und PDLP6 an unterschiedlichen Zellgrenzflächen funktionieren13. Diese Ergebnisse deuten darauf hin, dass PDLP5 möglicherweise nicht redundante Funktionen mit anderen PDLPs hat.
Aufgrund der kritischen Funktion von Parkinson in der interzellulären Kommunikation haben wir ein Protokoll entwickelt, um die pflanzlichen Parkinson-Proteine PDLP5 und PDCB1 sowie das virale Zell-zu-Zell-Bewegungsprotein (MP) des Turnip-Venen-Clearing-Virus (TVCV) als einfache, bequeme und zuverlässige Parkinson-Marker für zellbiologische Experimente einzusetzen. Zur weiteren Verifizierung wurde parallel die Visualisierung der Parkinson-Krankheit mittels Anilinblau-Färbung der entnommenen Gewebe durchgeführt. Die beschriebenen Protokolle für die PD-Lokalisierung von PDLP5, PDCB1 und TVCV MP können leicht angepasst werden, um eine potenzielle PD-Lokalisierung von zellulären oder pathogenen Proteinen in lebenden Pflanzen zu analysieren.
Protokoll
Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Pflanzenwachstum
- Nicotiana benthamiana Samen in feuchter Erde in einer kontrollierten Umgebung bei 23 °C unter 16 h Licht und 8 h Dunkelheit anbauen.
- Nach ca. 2 Wochen setzen Sie die Sämlinge mit den Torfpellets um die Wurzeln vorsichtig in größere Töpfe um und setzen Sie das Wachstum unter den gleichen Bedingungen für die Agroinfiltrationsversuche 4-5 Wochen lang fort.
HINWEIS: Verwenden Sie die Pflanzen nicht, wenn sie zu blühen beginnen, da die GFP-Akkumulation im Stadium des Blütenstandes18 abnimmt.
2. Vektor-Konstruktion
- Verwenden Sie PCR, um die kodierenden Sequenzen von Interesse mit Q5 High-Fidelity DNA-Polymerase zu amplifizieren und sie durch die BP-Reaktion19 unter Verwendung eines kommerziell erhältlichen BP-Clonase-Kits in den Eintrittsvektor pDONR207 zu klonieren.
- Übertragen Sie die Zielgene in den resultierenden Eintrittsplasmiden in den Zielvektor pPZP-RCS2A-nptII-DEST-EGFP-N1 durch die LR-Reaktion19 unter Verwendung eines kommerziell erhältlichen LR-Clonase-Kits, um Plasmide herzustellen, bei denen der EGFP-Tag an den C-Terminus der Zielproteine fusioniert ist.
- Verifizieren Sie alle Konstrukte durch PCR und Sequenzierung19.
3. Agroinfiltration
- Streak Agrobacterium tumefaciens EHA105 Zellen, die verschiedene Vektoren auf LB-Agarplatten enthalten und 2 Tage lang unter 28 °C inkubiert wurden.
- Eine einzelne Kolonie in 2 mL LB-Bouillon überführen und über Nacht bei 28 °C unter Rühren (250 U/min) inkubieren.
- 1 ml aus der Übernachtkultur nehmen, anschließend 4 ml frische LB-Brühe hinzufügen und 1 h unter den gleichen Bedingungen rekulturieren.
- Die bakterielle Suspension wird mit Infiltrationspuffer (MgCl2, 10 mM; MES, 10 mM, pH 5,6).
- Mischen Sie verschiedene bakterielle Suspensionen im Verhältnis 1:1 (v/v), um eine Endkonzentration von OD600 = 0,1 zu erreichen. (fakultativ).
- Inkubieren Sie die bakterielle Suspension 3 h lang bei Raumtemperatur unter leichtem Rühren.
- Infiltrieren Sie die abaxiale Oberfläche der vollständig expandierten Blätter verschiedener Pflanzen mit einer nadellosen 1-ml-Einwegspritze und markieren Sie den infiltrierten Bereich (eine dunklere Farbe als das umgebende, nicht infiltrierte Gewebe) mit einem wasserfesten Stift.
- Fahren Sie mit dem Abschnitt Konfokale Mikroskopie dieses Protokolls fort (Schritt 5).
HINWEIS: Andere Stämme von Agrobacterium tumefaciens, die für die Umwandlung von Pflanzenarten von Interesse geeignet sind, können ebenfalls verwendet werden.
4. Anilinblauer Fleck
- Ein Aliquot von 200 μl 1 % Anilinblau (in 50 mM Kaliumphosphatpuffer, pH 8,0) wird auf einen Objektträger20 gegeben.
- Schneiden Sie den infiltrierten Bereich von ca. 0,5 cm × 0,5 cm von der Vene weg mit einer Klinge heraus und überführen Sie die Blattgewebeproben in die Anilinblaulösung (abaxiale Seite nach oben) auf dem Objektträger, stellen Sie sicher, dass die Blattgewebeproben in die Anilinblaulösung getaucht sind, und decken Sie sie dann mit einem Deckglas (22 mm x 50 mm) ab.
- Legen Sie die Objektträger mit den Proben in einen Exsikkator, der an einer Vakuumpumpe befestigt ist, und evakuieren Sie sie für 2 Minuten (<0,8 Pa), gefolgt von einem langsamen Ablassen des Drucks und einer 30-minütigen Inkubation im Dunkeln bei Raumtemperatur.
- Visualisieren Sie das Fluoreszenzsignal von Anilinblau unter einem konfokalen Laser-Scanning-Mikroskop mit kompatibler Software.
HINWEIS: Hier wurde die Anilinblau-Färbemethode von Huang et al.20 verwendet. In einer Modifikation dieser Technik wurde das gleiche Experiment ohne den 2-minütigen Vakuumschritt durchgeführt, was zu ähnlichen Ergebnissen führte und darauf hindeutet, dass die Vakuumtrocknung für die Anilinfärbung nicht unbedingt erforderlich ist. Außerdem können die Konzentration des Anilinblau-Flecks und die Färbezeit je nach Quelle des Blattgewebes usw. variieren.
5. Konfokale Mikroskopie
- Ernte zwei Blätter von zwei Pflanzen zu unterschiedlichen Zeitpunkten nach der Infiltration. Schneiden Sie die Infiltrationszone mit einer Klinge in ca. 0,5 cm × 0,5 cm große Scheiben von der Vene entfernt.
- Legen Sie die Gewebeproben in einen Tropfen sterilen Wassers (abaxiale Seite nach oben) in den mittleren Teil eines Objektträgers und decken Sie sie mit einem Deckglas (22 mm x 50 mm) ab, wobei Sie darauf achten, dass keine Blasen entstehen.
- Stellen Sie sicher, dass die Anregungswellenlängen für die Detektion von CFP-, GFP- und RFP-Signalen 405 nm, 488 nm bzw. 561 nm betragen und die Emissionsfilter für die Detektion 410-602 nm für CFP und 400-602 nm für EGFP und RFP betragen, wobei die Lochblende 1 AU und die Master-Verstärkung auf 769 V eingestellt sind.
- Visualisieren Sie das Fluoreszenzsignal von autofluoreszierenden Tags im infiltrierten Bereich mit einem konfokalen Laserscanning-Mikroskop mit kompatibler Software 1 Tag, 2 Tage, 3 Tage, 5 Tage, 7 Tage und 10 Tage nach der Infiltration.
- Verwenden Sie eine 10-fach-Objektivlinse und GFP-Filter, um Zellen mit dem Fluoreszenzsignal zu lokalisieren, und wechseln Sie dann zu einer 40-fach-Objektivlinse, um die subzelluläre Lokalisierung und Bildaufzeichnung zu visualisieren.
- Sammeln Sie 20 Bilder für jede Bedingung mit mindestens zwei unabhängigen biologischen Replikaten.
- Score Die PD-Lokalisation des getesteten Proteins basiert auf dem diagnostischen punktuellen Auftreten des Signals an der Peripherie der Zelle21,22.
HINWEIS: Beim Nachweis der subzellulären Lokalisation eines unbekannten Proteins wird die Verwendung von mindestens drei Pflanzen empfohlen.
6. Datenanalyse
- Verwenden Sie die Fidschi-Software, um Kanäle zu teilen und die Maßstabsleiste zur Visualisierung zu den Bildern hinzuzufügen.
- Verwenden Sie die Fiji-Software, um Kanäle zu teilen, bevor Sie den mittleren Graustufenwert für jedes Bild messen, um die Fluoreszenzintensität von GFP (für MP, PDCB1 und PDLP5) oder CFP (für Anilinblau-Färbung) zu verschiedenen Zeitpunkten zu beurteilen.
- Verwenden Sie die Fiji-Software, um Kanäle vor der Normalisierung der Bilder von GFP (für MP, PDCB1 und PDLP5) oder CFP (für Anilinblau-Färbung) zu teilen. Der Bereich des Bildes wurde vermessen, und die PD-Punkte wurden manuell gezählt. Es wurde die Anzahl der PD-Punkte pro 100μm 2 berechnet.
- Verwenden Sie die bidirektionale ANOVA mit dem Tukey-Mehrfachvergleichstest23 , um die P-Werte zwischen den verschiedenen Stichproben und verschiedenen Zeitpunkten mit Statistik- und Grafiksoftware zu bestimmen.
HINWEIS: Bei einem Einkanal-Fluoreszenzbild stellt der Graustufenwert jedes Pixels die Fluoreszenzintensität dieses Punktes24 dar. Hier wurde der mittlere Graustufenwert verwendet, um die Fluoreszenzintensität jeder Probe zu verschiedenen Zeitpunkten zu beurteilen.
Ergebnisse
Um die Untersuchung der Funktion von Parkinson in der Pflanzenphysiologie und der Wechselwirkungen mit Krankheitserregern zu erleichtern, wurden drei einfache und zuverlässige Referenzproteine für die Parkinson-Lokalisierung entwickelt. Es wurden zwei zelluläre PD-Proteine und ein von Krankheitserregern abgeleitetes MP-Protein ausgewählt, das vom pflanzlichen Tobamovirus TVCV kodiert wird. Die subzelluläre Lokalisation dieser Proteine wurde mit Hilfe eines autofluoreszierenden Reporters EGFP visualisiert, der mit dem C-Terminus jedes Proteins fusioniert war. In einem alternativen Ansatz wurden die PD durch Anilinblau-Färbung der PD-assoziierten Callose-Ablagerungen sichtbar gemacht. Die Experimente zeigten, dass sowohl PDLP5 als auch MP eine punktuelle PD-Lokalisation aufwiesen, wobei MP auch im Zellkern lokalisiert war (Abbildung 1A), wie bereits erwähnt25. Im Gegensatz dazu schien sich PDCB1 zwischen PD und ER aufzuteilen (Abbildung 1A). Für Forscher, die weniger Erfahrung in der Interpretation der subzellulären Lokalisation von GFP-markierten Proteinen haben, veranschaulicht Abbildung 1A auch das charakteristische nukleozytoplasmatische Muster von EGFP, das in Pflanzenzellen exprimiert wird. Um das ER-Signal von PDCB1 zu bestätigen, exprimierten wir PDCB1 mit einem ER-Marker und fanden heraus, dass das PDCB1-Signal das Signal des ER-Markers überlappte (Abbildung 1B). Um die Effizienz der Parkinson-Lokalisation der getesteten Proteine zu bewerten, wurde der Prozentsatz der Zellen, die das Fluoreszenzsignal exprimieren und eine Parkinson-Lokalisation dieses Signals aufwiesen, berechnet (Tabelle 1).
Für die Identifizierung der subzellulären Lokalisation durch die Verwendung von Referenzproteinen ist es nützlich, die Zeitskala der Expression des Referenzproteins und mögliche Änderungen seiner Lokalisation zu verstehen. Abbildung 2 zeigt solche Kinetiken für MP, PDLP5 und PDCB1, wenn die PD selbst durch Anilinblau-Färbung sichtbar gemacht wurde. Sowohl MP als auch PDLP5 begannen sich bereits einen Tag nach der Infiltration (dpi) bei PD zu akkumulieren, und diese Akkumulation erreichte ein scheinbares Maximum bei 2 dpi und blieb für weitere 5 Tage stabil, danach nahm die Signalintensität ab, höchstwahrscheinlich aufgrund des Proteinabbaus. Der zeitliche Verlauf der subzellulären Sortierung von PDCB1 war komplexer: Für die ersten 3 dpi zeigte PDCB1 ein vergleichbar starkes ER-Signal, das die PD-spezifische Signallokalisation fast vollständig verdeckte, während bei 5 dpi dieses letztere Signal stärker ausgeprägt war (Abbildung 2), wenn auch in weniger Zellen. Interessanterweise fiel das Auftreten des klaren PD-Lokalisationsmusters von PDCB1 nach 5 dpi mit wesentlich weniger Zellen zusammen, die das EGFP-Signal zeigten (Ergänzende Abbildung 1). Somit können PDLP5 und MP als PD-Referenzproteine mit wenig Hintergrund in den meisten anderen zellulären Kompartimenten fungieren, mit einem optimalen Visualisierungszeitfenster von 2-5 dpi. PDCB1 hingegen ist nicht so gut geeignet wie ein PD-Marker.
Anschließend konnte nachgewiesen werden, dass die PD-Marker ihre subzelluläre Lokalisierungsspezifität in Gegenwart anderer PD-Proteine beibehalten. Zu diesem Zweck wurde MP-EGFP oder PDLP5-EGFP mit einem PD-lokalisierenden26 MP eines anderen Pflanzenvirus, dem Tabakmosaikvirus (TMV), koexprimiert, das mit CFP markiert ist. Abbildung 3 zeigt, dass die co-exprimierten Proteine in beiden Fällen ein klares PD-Lokalisationsmuster aufwiesen. In ähnlicher Weise zeigte die PD-spezifische histologische Anilinblau-Färbung des MP-EGFP- oder PDLP5-EGFP-exprimierenden Gewebes keine Interferenz mit der PD-Lokalisation der EGFP- und Anilinblau-Signale (Abbildung 3).
Parkinson-Lokalisationsstudien profitieren oft von einer quantitativeren Abschätzung der Gesamtexpression und des Parkinson-Signals des Referenzproteins. Dieser Quantifizierungsansatz ist in Abbildung 4 und Abbildung 5 dargestellt. Zur Quantifizierung der allgemeinen Expression wurde die Signalintensität von PDLP5 und MP gemessen. Beide Proteine zeigten während des 10-dpi-Zeitraums zu allen Visualisierungszeitpunkten starke Signale, mit maximaler Signalintensität bei 3 dpi für PDLP5 und bei 2 dpi für MP (Abbildung 4). Es gab zu keinem der Visualisierungszeitpunkte statistisch signifikante Unterschiede in der Signalintensität zwischen PDLP5 und MP. Die Intensität des PDCB1-Signals wies eine größere Fluktuation zwischen den Zeitpunkten auf, wobei das stärkste Signal bei 7 dpi beobachtet wurde (Abbildung 4), obwohl es zu diesem Zeitpunkt aufgrund des Mangels an exprimierenden Zellen schwierig war, statistisch signifikante Daten zu sammeln. Somit stellen die ersten 3 dpi das optimale Timing für den Nachweis der PDCB1-Expression dar. Wie erwartet zeigten Anilinblau-Färbeproben ein stabileres Signal als die transient exprimierten Proteine (Abbildung 4).
Die aus PDLP5 und MP gebildeten PD-Punkte wurden dann quantifiziert. PDLP5 zeigte eine stabile Anzahl von PD-Puncta während des gesamten Beobachtungszeitraums, ohne statistisch signifikante Unterschiede zwischen zwei der getesteten Zeitpunkte (Abbildung 5). Die höchste Anzahl von PD-Punkten, die von MP gebildet wurden, wurde bei 2 dpi nachgewiesen und blieb bis zum Ende des Beobachtungszeitraums relativ stabil. Der Vergleich der Anzahl der PD puncta zwischen PDLP5 und MP ergab bei den meisten Probenahmezeitpunkten keine statistisch signifikanten Unterschiede (Abbildung 5). Die Anzahl der mit Anilinblau gefärbten PD-Puncta blieb ebenfalls stabil, wenn auch leicht abnehmend bei 10 dpi. Die Anzahl der mit Anilinblau, PDLP5 oder MP dekorierten PD-Puncta war mit statistischer Signifikanz ähnlich (Abbildung 5), was mit der PD-Spezifität dieser Marker übereinstimmt. Die Anzahl der PD-Punkte für PDCB1 wurde nicht analysiert, da das PD-Signal zu früheren Zeitpunkten durch das ER-Signal verdeckt wurde und zu späteren Zeitpunkten nur in relativ wenigen Zellen beobachtet wurde (siehe Abbildung 2).

Abbildung 1: Plasmodesmata (PD)-Lokalisation von TVCV MP, PDLP5 und PDCB1 transient exprimiert in Nicotiana benthamiana und Anilinblau-Färbung von PD. (A) PD-Lokalisation von MP-EGFP, PDLP5-EGFP und PDCB1-EGFP sowie Anilin-Färbung. Das EGFP-Signal ist grün, das Anilinblau-Signal ist blau und die Plastiden-Autofluoreszenz ist rot. (B) Koexpression von MP-EGFP, PDLP5-EGFP und PDCB1-EGFP mit dem Marker für das endoplasmatische Retikulum (ER), der mit mRFP markiert ist. Die Bilder wurden 2 Tage nach der Infiltration (dpi) aufgenommen und sind einzelne konfokale Schnitte, die repräsentativ für zwei unabhängige Experimente sind (N = 20 Bilder von 2 Pflanzen). Das EGFP-Signal ist grün, das mRFP-Signal ist violett und die Plastiden-Autofluoreszenz ist rot. Gelbe Pfeile zeigen PDCB1 an, das an der Notaufnahme lokalisiert ist. Maßstabsbalken = 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Zeitverlauf der PD-Lokalisation von TVCV MP, PDLP5 und PDCB1 transient exprimiert in Nicotiana benthamiana und der Anilinblau-Färbung von PD. Die Bilder wurden bei 2 dpi aufgenommen und sind einzelne konfokale Schnitte, die repräsentativ für zwei unabhängige Experimente sind (N = 20 Bilder von 2 Pflanzen für alle Systeme, außer 7 dpi und 10 dpi für PDCB1-EGFP, in denen weniger Zellen das Signal zeigten). Das EGFP-Signal ist grün und das Anilinblau-Signal blau. Maßstabsbalken = 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
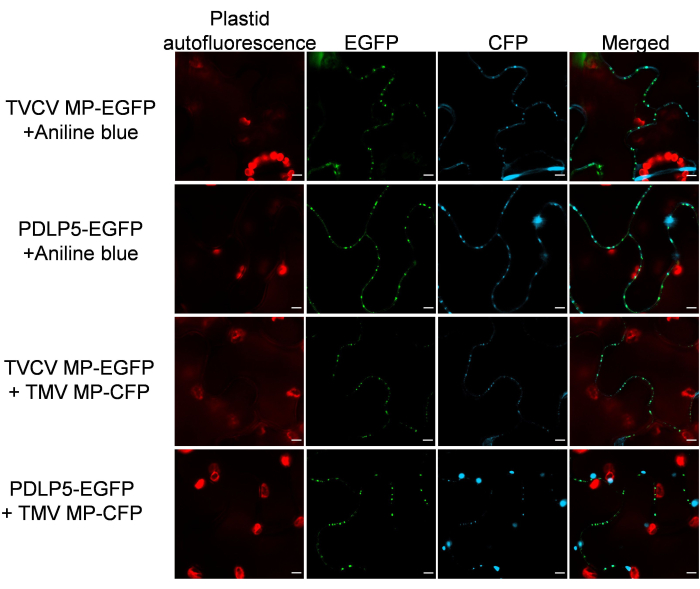
Abbildung 3: TVCV MP und PDLP5 behalten ihre PD-Lokalisation nach der Co-Expression mit einem PD-lokalisierenden TMV MP und der Cofärbung mit Anilinblau bei. Die Bilder wurden bei 2 dpi aufgenommen und sind einzelne konfokale Schnitte, die für N ≥ 10 Bilder von 4-6 Pflanzen repräsentativ sind. Das EGFP-Signal ist grün, das CFP-Signal und das Anilinblau-Signal sind blau und die Plastiden-Autofluoreszenz ist rot. Maßstabsbalken = 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
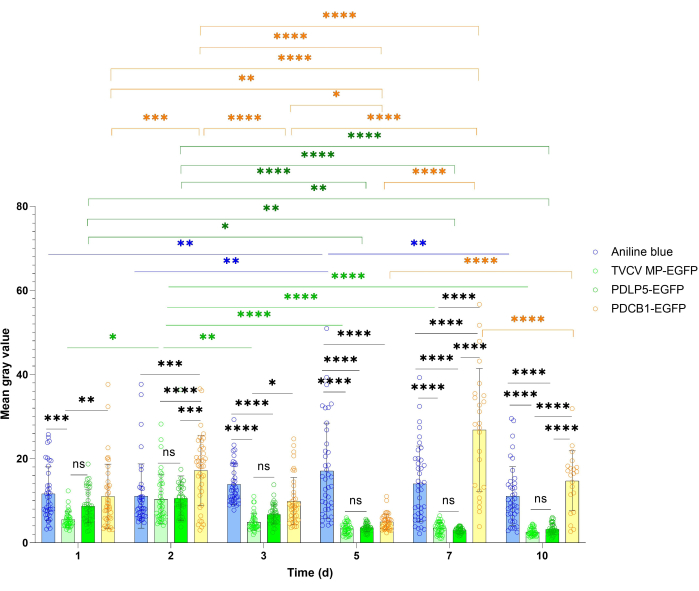
Abbildung 4: Zeitverlauf der Fluoreszenzintensität von TVCV MP, PDLP5 und PDCB1 transient exprimiert in der PD-Färbung von Nicotiana benthamiana und Anilinblau. Quantitative Daten wurden aus den in Abbildung 2 beschriebenen Experimenten gesammelt. Fehlerbalken stellen die Standardabweichung des Mittelwerts aus mehreren Experimenten dar. Die einzelnen Datenpunkte werden als Mittelwert für jede Messung angegeben. Die Unterschiede zwischen den Mittelwerten, die durch die bidirektionale ANOVA mit dem Mehrfachvergleichstest nach Tukey ermittelt werden, sind für die P-Werte statistisch signifikant. *P < 0,05, **P < 0,01 und ***P < 0,001, ****P < 0,0001; ns, nicht statistisch signifikant. Blaue Balken, Anilinfärbung; hellgrüne Balken, MP-EGFP, dunkelgrüne Balken, PDLP5-EGFP; gelbe Balken, PDCB1-EGFP. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Zeitverlauf der Bildung von PD-Puncta durch TVCV MP und PDLP5 transient exprimiert in Nicotiana benthamiana und durch Anilinblau-Färbung von PD. Quantitative Daten wurden aus den in Abbildung 2 beschriebenen Experimenten gesammelt. Fehlerbalken stellen die Standardabweichung des Mittelwerts aus mehreren Experimenten dar. Die einzelnen Datenpunkte werden als Mittelwert für jede Messung angegeben. Die Unterschiede zwischen den Mittelwerten, die durch die bidirektionale ANOVA mit dem Mehrfachvergleichstest nach Tukey ermittelt werden, sind für die P-Werte statistisch signifikant. *P < 0,05, **P < 0,01, ***P < 0,001, ****P < 0,0001. Blaue Balken, Anilinfärbung; hellgrüne Balken, MP-EGFP, dunkelgrüne Balken, PDLP5-EGFP. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Versuch I | ||||
| Zellen gesamt | Zellen mit Parkinson-Lokalisation | Zellen mit gemischtem Signal | Zellen mit Parkinson-Lokalisation (%) | |
| Anilinblau | 40 | 40 | 4 | 100 |
| Abgeordneter-EGFP | 40 | 40 | 4 | 100 |
| PDCB1-EGFP | 40 | 3 | 38 | 7.5 |
| PDLP5-EGFP | 40 | 40 | 0 | 100 |
| Versuch II | ||||
| Zellen gesamt | Zellen mit Parkinson-Lokalisation | Zellen mit gemischtem Signal | Zellen mit Parkinson-Lokalisation (%) | |
| Anilinblau | 40 | 40 | 8 | 100 |
| Abgeordneter-EGFP | 40 | 39 | 8 | 97.5 |
| PDCB1-EGFP | 40 | 12 | 31 | 30 |
| PDLP5-EGFP | 40 | 39 | 2 | 97.5 |
Tabelle 1: Effizienz der PD-Lokalisierung.
Ergänzende Abbildung 1: PD-Lokalisation von TVCV MP, PDLP5 und PDCB1 transient exprimiert in Nicotiana benthamiana. Die Bilder wurden mit 5 dpi (A), 7 dpi (B) und 10 dpi (C) aufgenommen. Das EGFP-Signal ist grün und die Autofluoreszenz der Plastiden rot. Maßstabsstäbe = 20 μm. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Alle zellbiologischen Untersuchungen der interzellulären Kommunikation und des Zell-zu-Zell-Transports von Pflanzen während der normalen Pflanzenentwicklung und Morphogenese sowie während der Interaktionen zwischen Pflanzen und Krankheitserregern erfordern den Nachweis und die Überwachung der Sortierung von Proteinen - sowohl endogenen als auch pathogenen, die für pflanzliche interzelluläre Verbindungen kodiert sind, die Plasmodesmata (PD). Diese Experimente würden wesentlich erleichtert durch die Verwendung von Referenzproteinen, ob endogen oder von Krankheitserregern abgeleitet, die treu und konsistent an Parkinson lokalisieren und diese Strukturen dekorieren und visualisieren. Dieser Bedarf wird hier durch die Selektion von drei bekannten PD-lokalisierenden Proteinen adressiert: Arabidopsis PDLP5 und PDCB1 sowie MP aus dem tobamoviralen Erreger TVCV. Diese Proteine wurden an ihren C-Termini mit dem autofluoreszierenden Reporter EGFP markiert, und die Fähigkeit der resultierenden Fusionsproteine, sich nach PD zu sortieren, wurde nach ihrer transienten Expression in Blättern von Nicotiana benthamiana und konfokalmikroskopischen Analysen der exprimierenden Gewebe bestimmt. Für die allgemeine Visualisierung der Parkinson-Krankheit in diesen Geweben wurde die Standard-Anilinblau-Färbemethode verwendet.
Es wird angenommen, dass pflanzliche virale MP in Bezug auf ihre Auswirkungen auf die PD-Struktur und -Integrität in zwei Hauptgruppen fallen: MP, die die PD-Permeabilität reversibel und ohne strukturelle Veränderungen bei PD erhöhen, und MP, die PD durch Modifikation ihrer Struktur hemmen. MP von Tobamoviren, wie z.B. TVCV, gehören zur ersten Gruppe der MP-Gruppe, was sie als attraktive Marker für Parkinson für subzelluläre Lokalisationsstudien positioniert27,28.
Diese Experimente positionierten PDLP5-EGFP und MP-EGFP als zuverlässige und reproduzierbare biologische Marker für Parkinson, wobei die Effizienz des PD-Nachweises mit der des Farbstoffs Anilinblau vergleichbar ist. Die PD-Lokalisation von PDLP5-EGFP und MP-EGFP wurde über den gesamten Beobachtungszeitraum von 10 dpi detektiert, wobei der optimale Zeitpunkt für die Beobachtung des charakteristischen PD-Lokalisationsmusters zwischen 2 dpi und 3 dpi lag. Obwohl die Expression des 35S-Promotors zu einem posttranskriptionellen Silencing des exprimierten Gens führen kann, tritt dies häufig nach längerer Expression in stabil transformierten transgenen Pflanzen auf. Zum anderen ist die Verwendung dieses starken konstitutiven Promotors wichtig für den einfachen Nachweis des Markerproteins. Wenn die Expression des Referenzproteins nach 7-10 dpi abnimmt, sollten kürzere Zeiträume, d. h. 2-3 dpi, verwendet werden. Signifikante Unterschiede im gesamten subzellulären Lokalisationsmuster zwischen PDLP5 und MP wurden hier nicht festgestellt, mit der Ausnahme, dass sich MP zusätzlich zur Parkinson im Zellkern akkumulierte. Umgekehrt wird PDCB1 aufgrund der Komplexität seines subzellulären Lokalisationsmusters, das eine erhebliche Lokalisation zum ER beinhaltet, nicht für die Verwendung als Parkinson-Marker per se empfohlen.
Wie leicht das EGFP-Fluoreszenzsignal detektiert werden konnte, hing auch vom Wachstumsstadium der Pflanze ab. insbesondere bei N. benthamiana wurde die geringste EGFP-Signalakkumulation im Blütenstandsstadium18 festgestellt.
Ziel dieser Studie war es, die PD-Lokalisationsmarker so zu entwickeln, dass sie maximal exklusiv für andere subzelluläre Kompartimente sind. Daher sollte ihre Verwendung als Referenz für die Lokalisierung von Proteinen von Interesse mit zusätzlichen subzellulären Lokalisierungsfähigkeiten mit der Expression anderer subzellulärer Marker, wie z.B. solcher, die spezifisch für das ER, Mitochondrien, Plasmamembran usw. sind, kombiniert werden. In diesem Fall sollte die Einbeziehung zusätzlicher Agrobacterium-Zellen für die Expression dieser Marker bei der Berechnung der Endkonzentration von Agroinfiltrationskulturen, d. h. OD600 der Flüssigkulturen, berücksichtigt werden. Offensichtlich sollten die Zellkonzentration und die Wachstumszeiten der Pflanzen angepasst werden, um die maximale Expression und die Erkennung von PD-Lokalisierungsmustern in anderen Pflanzenarten zu ermöglichen.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Die Arbeit im VC-Labor wurde durch Zuschüsse von NIH (R35GM144059), NSF (MCB 1913165 und IOS 1758046) und BARD (IS-5276-20) an VC unterstützt. Die Geldgeber spielten keine Rolle beim Studiendesign, bei der Datenerhebung und -interpretation oder bei der Entscheidung über die Veröffentlichung.
Materialien
| Name | Company | Catalog Number | Comments |
| ABT AC 1 phase motor | BRANDTECH | ABF63/4C-7RQ | |
| Agrobacterium tumefaciens EHA105 | |||
| Contamination control | CCI | ||
| Gateway BP Clonase II Enzyme mix | Invitrogen | #11789020 | |
| Gateway LR Clonase II Enzyme mix | Invitrogen | #11791020 | |
| GraphPad Prism 8.0.1. | GraphPad Software Inc. | ||
| Image J | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation | ||
| Laser scanning confocal microscope | Zeiss | LSM 900 | |
| Nicotiana benthamiana | Plant species | ||
| pDONR207 | Invitrogen | #12213013 | |
| Q5 High-Fidelity DNA Polymerase | NEB | #M0491S |
Referenzen
- Lee, J., et al. A plasmodesmata-localized protein mediates crosstalk between cell-to-cell communication and innate immunity in Arabidopsis. The Plant Cell. 23 (9), 3353-3373 (2011).
- Benitez-Alfonso, Y., Faulkner, C., Ritzenthaler, C., Maule, A. J. Plasmodesmata gateways to local and systemic virus infection. Mol Plant-Microbe Interact. 23 (11), 1403-1412 (2010).
- Bayer, E. M., Benitez-Alfonso, Y. Plasmodesmata: Channels under pressure. Annu Rev Plant Biol. 75, 21 (2024).
- Maule, A. J. Plasmodesmata: Structure, function and biogenesis. Curr Opin Plant Biol. 11, 680-686 (2008).
- Fernandez-Calvino, L., et al. Arabidopsis plasmodesmal proteome. PLoS One. 6 (4), e18880 (2011).
- Kirk, P., Amsbury, S., German, L., Gaudioso-Pedraza, R., Benitez-Alfonso, Y. A comparative meta-proteomic pipeline for the identification of plasmodesmata proteins and regulatory conditions in diverse plant species. BMC Biol. 20, 128 (2022).
- Levy, A., Erlanger, M., Rosenthal, M., Epel, B. L. A plasmodesmata-associated β-1,3-glucanase in Arabidopsis. The Plant J. 49 (4), 669-682 (2007).
- Kankanala, P., Czymmek, K., Valent, B. Roles for rice membrane dynamics and plasmodesmata during biotrophic invasion by the blast fungus. The Plant Cell. 19 (2), 706-724 (2007).
- Aung, K., et al. Pathogenic bacteria target plant plasmodesmata to colonize and invade surrounding tissues. The Plant Cell. 32 (3), 595-611 (2020).
- Cui, W., Lee, J. Arabidopsis callose synthases CalS1/8 regulate plasmodesmal permeability during stress. Nat Plants. 2 (5), 160 (2016).
- Liu, N. J., et al. Phytosphinganine affects plasmodesmata permeability via facilitating PDLP5-stimulated callose accumulation in Arabidopsis. Mol Plant. 13 (1), 128-143 (2020).
- Caillaud, M. C., et al. The plasmodesmal protein PDLP1 localizes to haustoria-associated membranes during downy mildew infection and regulates callose deposition. PLoS Pathog. 10 (11), e1004496 (2014).
- Li, Z., Su-Ling, L., Christian, M., Walley, J. W., Aung, K. Plasmodesmata-located protein 6 regulates plasmodesmal function in Arabidopsis vasculature. The Plant Cell. , (2024).
- Chen, X., et al. Arabidopsis PDLP7 modulated plasmodesmata function is related to BG10-dependent glucosidase activity required for callose degradation. Sci Bull. , (2024).
- Simpson, C., Thomas, C., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. The Plant Cell. 21 (2), 581-594 (2009).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLoS Biol. 6 (1), e7 (2008).
- Lim, G., et al. Plasmodesmata localizing proteins regulate transport and signaling during systemic acquired immunity in plants. Cell Host Microbe. 19 (4), 541-549 (2016).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnol Bioeng. 96 (3), 608-614 (2007).
- Walhout, A. J. M., et al. GATEWAY recombinational cloning: Application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Huang, C., et al. dsRNA-induced immunity targets plasmodesmata and is suppressed by viral movement proteins. The Plant Cell. 35 (10), 3845-3869 (2023).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. The plasmodesmal localization signal of TMV MP is recognized by plant synaptotagmin SYTA. mBio. 9 (4), e01314-e01318 (2018).
- Ward, S. J., Ramirez, M. D., Neelakantan, H., Walker, E. A. Cannabidiol prevents the development of cold and mechanical allodynia in paclitaxel-treated female C57Bl6 mice. Anesth Analg. 113 (4), 947-950 (2011).
- Erkkilä, M. T., et al. Widefield fluorescence lifetime imaging of protoporphyrin IX for fluorescence-guided neurosurgery: An ex vivo feasibility study. J Biophotonics. 12 (6), e201800378 (2019).
- Levy, A., Zheng, J. Y., Lazarowitz, S. G. The tobamovirus Turnip vein clearing virus 30-kilodalton movement protein localizes to novel nuclear filaments to enhance virus infection. J Virol. 87 (11), 6428-6440 (2013).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell-movement protein. mBio. 7 (1), e2052-e2015 (2016).
- Kumar, G., Dasgupta, I. Variability, functions and interactions of plant virus movement proteins: what do we know so far. Microorganisms. 9 (4), 695 (2021).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of nondestructive cell-to-cell and systemic movement of plant viruses. Crit Rev Plant Sci. 23 (3), 195-250 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten