Method Article
Analyse par microscopie confocale du tri des protéines vers les plasmodesmes chez Nicotiana benthamiana
Dans cet article
Résumé
Ce protocole décrit la sélection de marqueurs plasmodesmaux optimaux pour les analyses basées sur la microscopie confocale du ciblage des protéines sur les plasmodesmes lors d’interactions virus-plasmodesmes ou de transport plasmodesmal.
Résumé
Les plasmodesmes sont des nanopores membraneux qui relient le cytoplasme des cellules végétales adjacentes et permettent le trafic de cellules à cellules de nutriments, de macromolécules, ainsi que de virus envahisseurs. Les plasmodesmes jouent un rôle fondamental dans la régulation de la communication intercellulaire, contribuant au développement des plantes, aux réponses environnementales et aux interactions avec les agents pathogènes viraux. La découverte de la localisation plasmodesmale des protéines végétales ou virales pourrait fournir des informations fonctionnelles utiles sur la protéine et est importante pour comprendre les mécanismes des interactions plantes-virus. Pour faciliter ces études, nous décrivons un protocole d’analyse par microscopie confocale de différentes protéines de ciblage plasmodesmal afin de sélectionner le meilleur marqueur plasmodesmal pour l’étude des interactions virus-plasmodesmes ou du transport plasmodesmal. Plus précisément, les analyses de ces événements sont illustrées à l’aide de la protéine de mouvement de cellule à cellule (MP) du virus de clarification des veines de navet (TVCV), de la protéine 5 localisée d’Arabidopsis Plasmodesmata (PDLP5) et de la protéine de liaison 1 de Plasmodesmata Callose (PDCB1). Les données de localisation plasmodesmale des protéines sont analysées en parallèle avec la visualisation globale des plasmodesmes à l’aide de la coloration au bleu d’aniline des tissus échantillonnés. Ces approches peuvent être facilement adaptées pour analyser la localisation plasmodesmale de toute protéine cellulaire ou pathogène dans le planta.
Introduction
Les plasmodesmes jouent un rôle fondamental dans le contrôle du développement des plantes, des réponses environnementales et des interactions avec les agents pathogènes viraux grâce à la régulation de la communication intercellulaire 1,2. La MP se forme initialement au cours de la cytokinèse, avec des centaines de DP insérées dans la nouvelle cellule entre les deux cellules filles, fournissant ainsi les canaux de communication de cellule à cellule 3,4. La MP est une structure riche en membrane, contenant la membrane dérivée du réticulum endoplasmique (RE), un desmotubule trans-, dans la partie centrale des pores qui sont tapissés par la membrane plasmique 3,4. Des approches protéomiques comparatives ont permis d’identifier de nombreuses protéines fonctionnelles de la MP, notamment les β-1,3-glucanases (BG), les callose synthases (CALS), les protéines situées dans les plasmodesmes (PDLP), les protéines de liaison aux callosités (PDCB), les protéines de la région transmembranaire à plusieurs domaines C2 (MCTP)3 et les kinases de type récepteur répété riches en leucine (RLK)5. Récemment, Kirk et al. ont développé un outil appelé plasmodesmata in silico proteome 1 (PIP1), qui a permis de prédire de nouvelles protéines chez 22 espèces végétales6. La perméabilité et la structure de la MP varient au cours du développement de la plante et de la réponse à divers stress. Le dépôt et l’hydrolyse de callose (β-1,3-glucane) dans la région du cou entourant la DP sont l’un des mécanismes largement connus de la régulation de la DP7.
De nombreux microbes pathogènes, notamment les champignons, les bactéries et les virus, peuvent manipuler la dilatation ou la structure de la MP au cours de leurinfection2,8,9. Magnaporthe oryzae, l’agent causal de la pyriculariose du riz, déploie des hyphes envahissants intracellulaires pour se déplacer de cellule en cellule jusqu’à8. Un pathogène bactérien Pseudomonas syringae pv. la tomate a besoin d’une protéine effectrice HopO1-1 pour se déplacer intercellulaire et se propager dans la plante hôte en interagissant avec PDLP7 et en la déstabilisant, augmentant ainsi le flux moléculaire dans les cellules voisines chez Arabidopsis9. Cependant, les virus végétaux sont plus polyvalents dans la régulation de la MP au cours de leur transmission intercellulaire, la protéine de mouvement viral (MP) favorisant le mouvement de cellule à cellule2. En raison de leur rôle important dans la régulation du développement et de la croissance des plantes, ainsi que de leur interaction avec les microbes pathogènes des plantes, les MP ont fait l’objet d’une attention croissante ces dernières années. Chez Arabidopsis thaliana, il existe deux principaux types de protéines fonctionnelles de la, les PDLP (1-8) et les PDCB (1-5), et beaucoup d’entre elles, par exemple, PDLP5 1,10,11, PDLP112, PDLP613, PDLP714 et PDCB115, jouent un rôle dans la manipulation de la perméabilité de la par la régulation du dépôt de callose. Cependant, certains PDLP se sont avérés avoir une redondance fonctionnelle, par exemple, les mutants knock-out de pdlp1 et pdlp1,2 n’ont pas affecté le trafic moléculaire, bien que les mutants doubles knock-out de pdlp1,3 et pdlp2,3 aient montré une perméabilité plasmodesmale accrue16. Il est intéressant de noter que la régulation négative/l’inactivation ou la surexpression de PDLP5 seul entraîne une augmentation ou une diminution de la perméabilité plasmodesmale, respectivement 1,17. Récemment, Li et al. ont découvert que PDLP5 et PDLP6 fonctionnent à différentes interfaces cellulaires13. Ces résultats indiquent que PDLP5 pourrait avoir des fonctions non redondantes avec d’autres PDLP.
En raison de la fonction critique de la MP dans la communication intercellulaire, nous avons développé un protocole pour déployer les protéines PDP PDLP5 et PDCB1 de la plante et la protéine virale de mouvement de cellule à cellule (MP) du virus du nettoyage des veines du navet (TVCV) en tant que marqueurs de DP simples, pratiques et fiables pour l’expérimentation en biologie cellulaire. Pour une vérification plus poussée, la visualisation de la DP à l’aide de la coloration au bleu d’aniline des tissus échantillonnés s’est déroulée en parallèle. Les protocoles décrits pour la localisation de PDLP5, PDCB1 et TVCV MP peuvent être facilement adaptés pour analyser la localisation potentielle de toute protéine cellulaire ou dérivée d’un agent pathogène dans les plantes vivantes.
Protocole
Les détails des réactifs et de l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Croissance des plantes
- Cultivez les graines de Nicotiana benthamiana dans un sol humide dans une chambre à environnement contrôlé à 23 °C sous 16 h de lumière et 8 h d’obscurité.
- Après environ 2 semaines, transférez soigneusement les plants avec les granulés de tourbe autour de leurs racines dans des pots plus grands et continuez la croissance dans les mêmes conditions pendant 4-5 semaines pour les expériences d’agro-infiltration.
REMARQUE : N’utilisez pas les plantes lorsqu’elles commencent à fleurir, car l’accumulation de GFP diminuera au stade d’inflorescence18.
2. Construction vectorielle
- Utilisez la PCR pour amplifier les séquences codantes d’intérêt avec l’ADN polymérase haute fidélité Q5 et clonez-les dans le vecteur d’entrée pDONR207 par la réaction BP19 à l’aide d’un kit BP Clonase disponible dans le commerce.
- Transférer les gènes cibles dans les plasmides d’entrée résultants dans le vecteur de destination pPZP-RCS2A-nptII-DEST-EGFP-N1 par la réaction LR19 à l’aide d’un kit de clonase LR disponible dans le commerce pour produire des plasmides avec l’étiquette EGFP fusionnée à l’extrémité C-terminale des protéines cibles.
- Vérifier toutes les constructions par PCR et séquençage19.
3. L’agro-infiltration
- Cellules striées d’Agrobacterium tumefaciens EHA105 contenant différents vecteurs sur plaques de gélose LB et incubées pendant 2 jours à 28 °C.
- Transvaser une seule colonie dans 2 mL de bouillon LB et incuber toute la nuit à 28 °C en agitant (250 tr/min).
- Retirer 1 mL de la culture pendant la nuit, puis ajouter 4 mL de bouillon LB frais, et recultiver pendant 1 h dans les mêmes conditions.
- Ajuster la suspension bactérienne àOD 600 = 0,1 (OD600 = 0,2 pour la co-expression de deux protéines) avec tampon d’infiltration (MgCl2, 10 mM ; MES, 10 mM, pH 5,6).
- Mélanger différentes suspensions bactériennes dans un rapport de 1:1 (v/v) pour obtenir une concentration finale deDO 600 = 0,1. (facultatif).
- Incuber la suspension bactérienne à température ambiante pendant 3 h avec une agitation douce.
- Infiltrez la surface abaxiale des feuilles complètement déployées de différentes plantes à l’aide d’une seringue jetable sans aiguille de 1 ml et marquez la zone infiltrée (d’une couleur plus foncée que le tissu environnant non infiltré) à l’aide d’un stylo étanche.
- Passez à la section Microscopie confocale de ce protocole (étape 5).
REMARQUE : D’autres souches d’Agrobacterium tumefaciens adaptées à la transformation des espèces végétales d’intérêt peuvent également être utilisées.
4. Teinture bleu aniline
- Ajouter une aliquote de 200 μL de bleu d’aniline à 1 % (dans un tampon de phosphate de potassium de 50 mM, pH 8,0) à une lame de microscope20.
- Exciser la zone infiltrée d’environ 0,5 cm × 0,5 cm, loin de la veine, à l’aide d’une lame, et transférer les échantillons de tissu foliaire dans la solution de bleu d’aniline (côté abaxial vers le haut) sur la lame de microscope, et s’assurer que les échantillons de tissu foliaire sont immergés dans la solution de bleu d’aniline, puis recouvrir le tout d’un verre de protection (22 mm x 50 mm).
- Placez les lames de microscope avec les échantillons dans un dessiccateur fixé à une pompe à vide et évacuez pendant 2 min (<0,8 Pa), puis relâchez lentement la pression et laissez incuber dans l’obscurité pendant 30 min à température ambiante.
- Visualisez le signal fluorescent du bleu d’aniline sous un microscope confocal à balayage laser avec un logiciel compatible.
REMARQUE : Ici, la méthode de coloration au bleu d’aniline de Huang et al.20 a été utilisée. Dans une modification de cette technique, la même expérience a été réalisée sans l’étape de vide de 2 minutes, produisant des résultats similaires et suggérant que le séchage sous vide n’est pas essentiel pour la coloration à l’aniline. De plus, la concentration de la coloration au bleu d’aniline et le temps de coloration peuvent varier en fonction de la source du tissu foliaire, etc.
5. Microscopie confocale
- Récoltez deux feuilles de deux plantes à des moments différents après l’infiltration. Coupez la zone d’infiltration en tranches d’environ 0,5 cm × 0,5 cm de la veine à l’aide d’une lame.
- Placez les échantillons de tissus dans une goutte d’eau stérile (côté abaxial vers le haut) dans la partie centrale d’une lame de microscope, et couvrez-les d’un verre de protection (22 mm x 50 mm), en prenant soin d’éviter les bulles.
- Assurez-vous que les longueurs d’onde d’excitation pour la détection des signaux CFP, GFP et RFP sont respectivement de 405 nm, 488 nm et 561 nm, et que les filtres d’émission pour la détection sont de 410 à 602 nm pour le CFP et de 400 à 602 nm pour l’EGFP et le RFP, avec le sténopé 1 AU et le gain principal réglé sur 769 V.
- Visualisez le signal fluorescent des balises autofluorescentes dans la zone infiltrée à l’aide d’un microscope confocal à balayage laser avec un logiciel compatible à 1 jour, 2 jours, 3 jours, 5 jours, 7 jours et 10 jours après l’infiltration.
- Utilisez une lentille d’objectif 10x et des filtres GFP pour localiser les cellules avec le signal fluorescent, puis passez à une lentille d’objectif 40x pour la visualisation de la localisation subcellulaire et l’enregistrement d’images.
- Collectez 20 images pour chaque condition, en utilisant au moins deux réplicats biologiques indépendants.
- Score localisation de la protéine testée basée sur l’apparition diagnostique ponctuée du signal à la périphérie de la cellule21,22.
REMARQUE : Lors de la détection de la localisation subcellulaire d’une protéine inconnue, il est recommandé d’utiliser au moins trois plantes.
6. Analyse des données
- Utilisez le logiciel Fiji pour diviser les canaux et ajouter la barre d’échelle aux images pour la visualisation.
- Utilisez le logiciel Fiji pour diviser les canaux avant de mesurer la valeur moyenne en niveaux de gris de chaque image afin d’évaluer l’intensité de fluorescence GFP (pour MP, PDCB1 et PDLP5) ou CFP (pour la coloration au bleu d’aniline) à différents points temporels.
- Utilisez le logiciel Fiji pour diviser les canaux avant la normalisation des images de GFP (pour MP, PDCB1 et PDLP5) ou CFP (pour la coloration au bleu d’aniline). La surface de l’image a été mesurée et les points ont été comptés manuellement. Le nombre de points de par 100 μm2 a été calculé.
- Utilisez l’ANOVA à deux facteurs avec le test de comparaisons multiples23 de Tukey pour déterminer les valeurs P entre les différents échantillons et les différents points temporels à l’aide d’un logiciel statistique et graphique.
REMARQUE : Pour une image de fluorescence monocanal, la valeur en niveaux de gris de chaque pixel représente l’intensité de fluorescence de ce point24. Ici, la valeur moyenne en niveaux de gris a été utilisée pour évaluer l’intensité de fluorescence de chaque échantillon à différents points temporels.
Résultats
Pour faciliter l’étude de la fonction de la DP dans la physiologie végétale et des interactions avec les agents pathogènes, trois protéines de référence simples et fiables ont été développées pour la localisation de la DP. Deux protéines cellulaires et une protéine MP dérivée d’un agent pathogène codées par le TVCV du tobamovirus végétal ont été sélectionnées. La localisation subcellulaire de ces protéines a été visualisée à l’aide d’un rapporteur autofluorescent EGFP fusionné à l’extrémité C-terminale de chaque protéine. Dans une approche alternative, les DP ont été visualisées à l’aide de la coloration au bleu d’aniline des dépôts de callose associés à la DP. Les expériences ont montré que PDLP5 et MP présentaient tous deux une localisation ponctuée de la, la MP étant également localisée dans le noyau cellulaire (Figure 1A), comme indiqué précédemment25. En revanche, PDCB1 semblait se répartir entre et RE (Figure 1A). Pour les chercheurs moins expérimentés dans l’interprétation de la localisation subcellulaire des protéines marquées à la GFP, la figure 1A illustre également le profil nucléocytoplasmique caractéristique de l’EGFP exprimé dans les cellules végétales. Pour confirmer le signal RE de PDCB1, nous avons co-exprimé PDCB1 avec un marqueur RE et avons constaté que le signal PDCB1 chevauchait le signal du marqueur RE (Figure 1B). Pour évaluer l’efficacité de la localisation des protéines testées, le pourcentage de cellules exprimant le signal fluorescent qui présentaient une localisation de ce signal a été calculé (Tableau 1).
Pour l’identification de la localisation subcellulaire à l’aide de protéines de référence, il est utile de comprendre l’échelle de temps de l’expression de la protéine de référence et les changements potentiels de sa localisation. La figure 2 montre une telle cinétique pour MP, PDLP5 et PDCB1 lorsque les eux-mêmes ont été visualisés par coloration au bleu d’aniline. MP et PDLP5 ont commencé à s’accumuler au niveau de la déjà un jour après l’infiltration (dpi), et cette accumulation a atteint un maximum apparent à 2 dpi, restant stable pendant 5 jours supplémentaires, après quoi l’intensité du signal a diminué, très probablement en raison de la dégradation des protéines. L’évolution temporelle du tri subcellulaire de PDCB1 était plus complexe : pour les premiers 3 dpi, PDCB1 présentait un signal RE comparativement fort, qui obscurcissait presque complètement la localisation du signal spécifique de, tandis qu’à 5 dpi, ce dernier signal devenait plus prononcé (Figure 2), bien que dans moins de cellules. Il est intéressant de noter que l’émergence du modèle de localisation clair de PDCB1 après 5 dpi a coïncidé avec beaucoup moins de cellules montrant le signal EGFP (Figure supplémentaire 1). Ainsi, PDLP5 et MP peuvent fonctionner comme des protéines de référence avec peu de bruit de fond dans la plupart des autres compartiments cellulaires, avec une fenêtre de temps de visualisation optimale de 2 à 5 dpi. PDCB1, en revanche, n’est pas aussi approprié qu’un marqueur.
Il a ensuite été vérifié que les marqueurs de la conservent leur spécificité de localisation subcellulaire en présence d’autres protéines de la. À cette fin, MP-EGFP ou PDLP5-EGFP a été co-exprimé avec un-localisant 26 MP d’un autre virus végétal, le virus de la mosaïque du tabac (TMV) marqué avec CFP. La figure 3 montre que, dans les deux cas, les protéines co-exprimées présentaient un schéma clair de localisation de la. De même, la coloration histologique au bleu d’aniline spécifique à la des tissus exprimant MP-EGFP ou PDLP5-EGFP n’a détecté aucune interférence avec la localisation des signaux EGFP et bleu d’aniline (Figure 3).
Les études de localisation de la bénéficient souvent d’une estimation plus quantitative de l’expression globale de la protéine de référence et du signal de la. Cette approche de quantification est illustrée à la figure 4 et à la figure 5. Pour la quantification de l’expression générale, l’intensité du signal de PDLP5 et MP a été mesurée. Les deux protéines ont montré des signaux forts à tous les points temporels de visualisation pendant la période de 10 dpi, avec une intensité de signal maximale à 3 dpi pour PDLP5 et à 2 dpi pour MP (Figure 4). Il n’y avait aucune différence statistiquement significative dans l’intensité du signal entre PDLP5 et MP à aucun des points temporels de visualisation. L’intensité du signal PDCB1 a montré une plus grande fluctuation entre les points temporels, le signal le plus fort étant observé à 7 ppp (figure 4), bien qu’il ait été difficile de recueillir des données statistiquement significatives à ce moment-là en raison de la rareté des cellules exprimées. Ainsi, les 3 premiers dpi représentent le moment optimal pour détecter l’expression de PDCB1. Comme prévu, les échantillons de coloration au bleu d’aniline ont montré un signal plus stable que les protéines exprimées transitoirement (Figure 4).
Les puncta formés par PDLP5 et MP ont ensuite été quantifiés. PDLP5 a montré un nombre stable de points tout au long de la période d’observation, sans différences statistiquement significatives entre deux des points temporels testés (figure 5). Le plus grand nombre de puncta formés par MP a été détecté à 2 dpi, et ils sont restés relativement stables jusqu’à la fin de la période d’observation. La comparaison du nombre de points entre PDLP5 et MP n’a révélé aucune différence statistiquement significative à la plupart des moments d’échantillonnage (figure 5). Le nombre de puncta teintés au bleu d’aniline est également resté stable, bien qu’il ait légèrement diminué à 10 dpi. Les nombres de puncta décorés de bleu d’aniline, de PDLP5 ou de MP étaient similaires les uns aux autres avec une signification statistique (figure 5), ce qui correspond à la spécificité DP de ces marqueurs. Le nombre de points pour PDCB1 n’a pas été analysé parce que, à des moments antérieurs, le signal était obscurci par le signal RE, et à des moments ultérieurs, il n’a été observé que dans un nombre relativement restreint de cellules (voir la figure 2).

Figure 1 : Localisation en plasmodesmes () de TVCV MP, PDLP5 et PDCB1 exprimée transitoirement dans Nicotiana benthamiana et coloration au bleu d’aniline de. (A) Localisation de MP-EGFP, PDLP5-EGFP et PDCB1-EGFP et coloration à l’aniline. Le signal EGFP est en vert, le signal bleu aniline est en bleu et l’autofluorescence plastide est en rouge. (B) Co-expression de MP-EGFP, PDLP5-EGFP et PDCB1-EGFP avec le marqueur du réticulum endoplasmique (RE) marqué par mRFP. Les images ont été enregistrées 2 jours après l’infiltration (dpi) et sont des coupes confocales simples représentatives de deux expériences indépendantes (N = 20 images de 2 plantes). Le signal EGFP est en vert, le signal mRFP est en violet et l’autofluorescence des plastes est en rouge. Les flèches jaunes indiquent que PDCB1 est localisé au RE. Barres d’échelle = 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 2 : Évolution temporelle de la localisation de la MP du TVCV MP, du PDLP5 et du PDCB1 exprimés transitoirement chez Nicotiana benthamiana et de la coloration au bleu d’aniline de la MP. Les images ont été enregistrées à 2 dpi et sont des coupes confocales uniques représentatives de deux expériences indépendantes (N = 20 images de 2 plantes pour tous les systèmes, sauf 7 dpi et 10 dpi pour PDCB1-EGFP, dans lesquels moins de cellules présentaient le signal). Le signal EGFP est en vert et le signal bleu aniline est en bleu. Barres d’échelle = 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
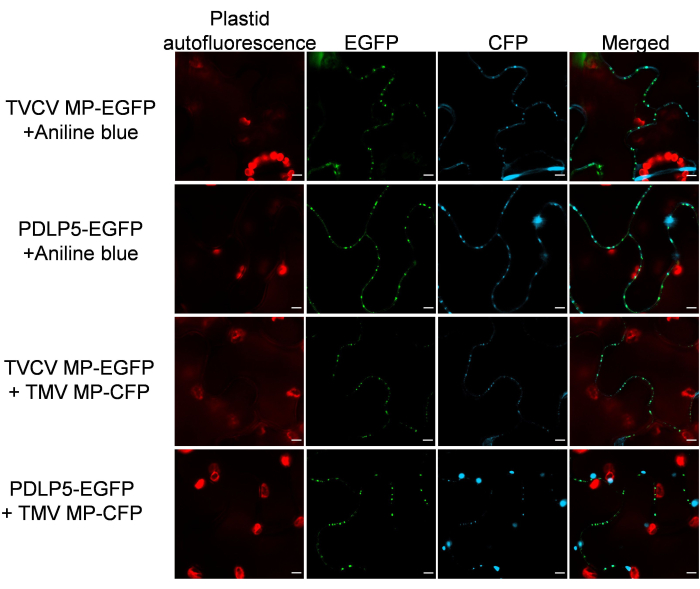
Figure 3 : TVCV MP et PDLP5 conservent leur localisation après co-expression avec un MP TMV localisant et co-coloration avec le bleu d’aniline. Les images ont été enregistrées à 2 dpi et sont des coupes confocales simples représentatives de N ≥ 10 images de 4 à 6 plantes. Le signal EGFP est en vert, le signal CFP et le signal bleu aniline sont en bleu et l’autofluorescence plastide est en rouge. Barres d’échelle = 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
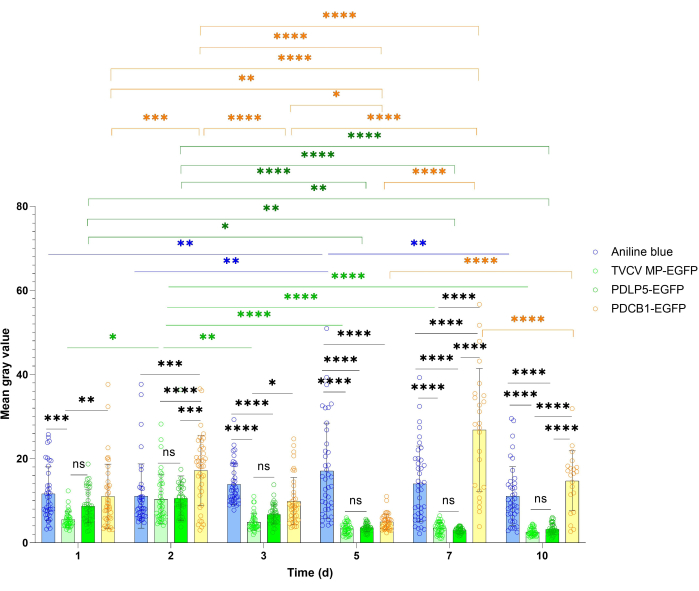
Figure 4 : Évolution temporelle de l’intensité de fluorescence du TVCV MP, PDLP5 et PDCB1 exprimée transitoirement dans la coloration à Nicotiana benthamiana et au bleu d’aniline. Des données quantitatives ont été recueillies à partir des expériences décrites à la figure 2. Les barres d’erreur représentent l’écart-type de la moyenne de plusieurs expériences. Les points de données individuels sont indiqués comme moyenne pour chaque mesure. Les différences entre les valeurs moyennes évaluées par l’ANOVA à deux facteurs avec le test de comparaisons multiples de Tukey sont statistiquement significatives pour les valeurs P. *P < 0,05, **P < 0,01 et ***P < 0,001, ****P < 0,0001 ; ns, non statistiquement significatif. Barres bleues, coloration à l’aniline ; barres vert clair, MP-EGFP, barres vert foncé, PDLP5-EGFP ; barres jaunes, PDCB1-EGFP. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Évolution temporelle de la formation de puncta par TVCV MP et PDLP5 exprimée transitoirement chez Nicotiana benthamiana et par coloration au bleu d’aniline de. Des données quantitatives ont été recueillies à partir des expériences décrites à la figure 2. Les barres d’erreur représentent l’écart-type de la moyenne de plusieurs expériences. Les points de données individuels sont indiqués comme moyenne pour chaque mesure. Les différences entre les valeurs moyennes évaluées par l’ANOVA à deux facteurs avec le test de comparaisons multiples de Tukey sont statistiquement significatives pour les valeurs P. *P < 0,05, **P < 0,01, ***P < 0,001, ****P < 0,0001. Barres bleues, coloration à l’aniline ; barres vert clair, MP-EGFP, barres vert foncé, PDLP5-EGFP. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Expérience I | ||||
| Nombre total de cellules | Cellules avec localisation de la MP | Cellules à signal mixte | Cellules avec localisation de la MP (%) | |
| Bleu aniline | 40 | 40 | 4 | 100 |
| MP-EGFP | 40 | 40 | 4 | 100 |
| PDCB1-EGFP | 40 | 3 | 38 | 7.5 |
| PDLP5-EGFP | 40 | 40 | 0 | 100 |
| Expérience II | ||||
| Nombre total de cellules | Cellules avec localisation de la MP | Cellules à signal mixte | Cellules avec localisation de la MP (%) | |
| Bleu aniline | 40 | 40 | 8 | 100 |
| MP-EGFP | 40 | 39 | 8 | 97.5 |
| PDCB1-EGFP | 40 | 12 | 31 | 30 |
| PDLP5-EGFP | 40 | 39 | 2 | 97.5 |
Tableau 1 : Efficacité de la localisation de la DP.
Figure supplémentaire 1 : Localisation de TVCV MP, PDLP5 et PDCB1 exprimée transitoirement chez Nicotiana benthamiana. Les images ont été enregistrées à 5 dpi (A), 7 dpi (B) et 10 dpi (C). Le signal EGFP est vert et l’autofluorescence du plaste est rouge. Barres d’échelle = 20 μm. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Toute étude de biologie cellulaire de la communication intercellulaire végétale et du transport de cellule à cellule au cours du développement et de la morphogenèse normaux des plantes, ainsi que lors des interactions plantes-pathogènes, nécessite la détection et la surveillance du tri des protéines, à la fois endogènes et codées par des agents pathogènes, aux connexions intercellulaires des plantes, les plasmodesmes (). Ces expériences seraient considérablement facilitées par l’utilisation de protéines de référence, qu’elles soient endogènes ou dérivées d’agents pathogènes, qui se localisent fidèlement et systématiquement dans la, décorant et visualisant ces structures. Ce besoin est résolu ici par la sélection de trois protéines connues localisantes de : Arabidopsis PDLP5 et PDCB1, et MP de l’agent pathogène tobamoviral TVCV. Ces protéines ont été marquées à leur extrémité C-terminale avec le rapporteur autofluorescent EGFP, et la capacité des protéines de fusion résultantes à trier en MP a été déterminée à la suite de leur expression transitoire dans les feuilles de Nicotiana benthamiana et d’analyses en microscopie confocale des tissus exprimants. Pour la visualisation générale de la MP dans ces tissus, la méthode standard de coloration au bleu d’aniline a été utilisée.
On pense que les MP virales végétales se divisent en deux grands groupes en ce qui concerne leurs effets sur la structure et l’intégrité de la MP : les MP qui augmentent la perméabilité de la MP de manière réversible et sans aucune altération structurelle de la MP et les MP qui contrôlent la MP en modifiant leur structure. Les MP des tobamovirus, tels que le TVCV, appartiennent au premier groupe MP, ce qui les positionne comme des marqueurs attrayants de la MP pour les études de localisation subcellulaire27,28.
Ces expériences ont positionné PDLP5-EGFP et MP-EGFP comme des marqueurs biologiques fiables et reproductibles de la MP, avec une efficacité de détection de la DP comparable à celle du colorant bleu d’aniline. La localisation DP de PDLP5-EGFP et MP-EGFP a été détectée tout au long de la période d’observation de 10 dpi, le moment optimal pour l’observation du modèle de localisation caractéristique de la DP se situant entre 2 dpi et 3 dpi. Bien que l’expression du promoteur 35S puisse entraîner un silençage post-transcriptionnel du gène exprimé, cela se produit souvent après une expression prolongée chez des plantes transgéniques transformées de manière stable. D’autre part, l’utilisation de ce puissant promoteur constitutif est importante pour la détection facile de la protéine marqueur. Si l’expression de la protéine de référence diminue après 7-10 dpi, des périodes plus courtes, c’est-à-dire 2-3 dpi, doivent être utilisées. Des différences significatives dans le profil global de localisation subcellulaire entre PDLP5 et MP n’ont pas été détectées ici, sauf que MP s’est accumulé dans le noyau cellulaire en plus de la MP. Inversement, il n’est pas recommandé d’utiliser PDCB1 comme marqueur de la MP en soi en raison de la complexité de son modèle de localisation subcellulaire, qui comprend une localisation substantielle dans le RE.
La facilité de détection du signal de fluorescence EGFP dépendait également du stade de croissance de la plante ; plus précisément, chez N. benthamiana, l’accumulation de signal EGFP la plus faible a été observée au stade18 de l’inflorescence.
Cette étude visait à développer les marqueurs de localisation de la MP de manière à ce qu’ils soient exclusivement associés à d’autres compartiments subcellulaires. Ainsi, leur utilisation comme références pour la localisation de protéines d’intérêt avec des capacités de localisation subcellulaire supplémentaires devrait être combinée avec l’expression d’autres marqueurs subcellulaires, tels que ceux spécifiques du RE, des mitochondries, de la membrane plasmique, etc. Dans ce cas, l’inclusion de cellules Agrobacterium supplémentaires pour l’expression de ces marqueurs doit être prise en compte lors du calcul de la concentration finale des cultures d’agro-infiltration, c’est-à-dire DO600 des cultures liquides. De toute évidence, la concentration cellulaire et les temps de croissance des plantes doivent être adaptés pour une expression maximale et la détection du modèle de localisation chez d’autres espèces végétales.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Les travaux dans le laboratoire VC ont été soutenus par des subventions des NIH (R35GM144059), de la NSF (MCB 1913165 et IOS 1758046) et de BARD (IS-5276-20) à VC. Les bailleurs de fonds n’ont joué aucun rôle dans la conception de l’étude, la collecte et l’interprétation des données, ni dans la décision de publication.
matériels
| Name | Company | Catalog Number | Comments |
| ABT AC 1 phase motor | BRANDTECH | ABF63/4C-7RQ | |
| Agrobacterium tumefaciens EHA105 | |||
| Contamination control | CCI | ||
| Gateway BP Clonase II Enzyme mix | Invitrogen | #11789020 | |
| Gateway LR Clonase II Enzyme mix | Invitrogen | #11791020 | |
| GraphPad Prism 8.0.1. | GraphPad Software Inc. | ||
| Image J | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation | ||
| Laser scanning confocal microscope | Zeiss | LSM 900 | |
| Nicotiana benthamiana | Plant species | ||
| pDONR207 | Invitrogen | #12213013 | |
| Q5 High-Fidelity DNA Polymerase | NEB | #M0491S |
Références
- Lee, J., et al. A plasmodesmata-localized protein mediates crosstalk between cell-to-cell communication and innate immunity in Arabidopsis. The Plant Cell. 23 (9), 3353-3373 (2011).
- Benitez-Alfonso, Y., Faulkner, C., Ritzenthaler, C., Maule, A. J. Plasmodesmata gateways to local and systemic virus infection. Mol Plant-Microbe Interact. 23 (11), 1403-1412 (2010).
- Bayer, E. M., Benitez-Alfonso, Y. Plasmodesmata: Channels under pressure. Annu Rev Plant Biol. 75, 21(2024).
- Maule, A. J. Plasmodesmata: Structure, function and biogenesis. Curr Opin Plant Biol. 11, 680-686 (2008).
- Fernandez-Calvino, L., et al. Arabidopsis plasmodesmal proteome. PLoS One. 6 (4), e18880(2011).
- Kirk, P., Amsbury, S., German, L., Gaudioso-Pedraza, R., Benitez-Alfonso, Y. A comparative meta-proteomic pipeline for the identification of plasmodesmata proteins and regulatory conditions in diverse plant species. BMC Biol. 20, 128(2022).
- Levy, A., Erlanger, M., Rosenthal, M., Epel, B. L. A plasmodesmata-associated β-1,3-glucanase in Arabidopsis. The Plant J. 49 (4), 669-682 (2007).
- Kankanala, P., Czymmek, K., Valent, B. Roles for rice membrane dynamics and plasmodesmata during biotrophic invasion by the blast fungus. The Plant Cell. 19 (2), 706-724 (2007).
- Aung, K., et al. Pathogenic bacteria target plant plasmodesmata to colonize and invade surrounding tissues. The Plant Cell. 32 (3), 595-611 (2020).
- Cui, W., Lee, J. Arabidopsis callose synthases CalS1/8 regulate plasmodesmal permeability during stress. Nat Plants. 2 (5), 160(2016).
- Liu, N. J., et al. Phytosphinganine affects plasmodesmata permeability via facilitating PDLP5-stimulated callose accumulation in Arabidopsis. Mol Plant. 13 (1), 128-143 (2020).
- Caillaud, M. C., et al. The plasmodesmal protein PDLP1 localizes to haustoria-associated membranes during downy mildew infection and regulates callose deposition. PLoS Pathog. 10 (11), e1004496(2014).
- Li, Z., Su-Ling, L., Christian, M., Walley, J. W., Aung, K. Plasmodesmata-located protein 6 regulates plasmodesmal function in Arabidopsis vasculature. The Plant Cell. , (2024).
- Chen, X., et al. Arabidopsis PDLP7 modulated plasmodesmata function is related to BG10-dependent glucosidase activity required for callose degradation. Sci Bull. , (2024).
- Simpson, C., Thomas, C., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. The Plant Cell. 21 (2), 581-594 (2009).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLoS Biol. 6 (1), e7(2008).
- Lim, G., et al. Plasmodesmata localizing proteins regulate transport and signaling during systemic acquired immunity in plants. Cell Host Microbe. 19 (4), 541-549 (2016).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnol Bioeng. 96 (3), 608-614 (2007).
- Walhout, A. J. M., et al. GATEWAY recombinational cloning: Application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Huang, C., et al. dsRNA-induced immunity targets plasmodesmata and is suppressed by viral movement proteins. The Plant Cell. 35 (10), 3845-3869 (2023).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. The plasmodesmal localization signal of TMV MP is recognized by plant synaptotagmin SYTA. mBio. 9 (4), e01314-e01318 (2018).
- Ward, S. J., Ramirez, M. D., Neelakantan, H., Walker, E. A. Cannabidiol prevents the development of cold and mechanical allodynia in paclitaxel-treated female C57Bl6 mice. Anesth Analg. 113 (4), 947-950 (2011).
- Erkkilä, M. T., et al. Widefield fluorescence lifetime imaging of protoporphyrin IX for fluorescence-guided neurosurgery: An ex vivo feasibility study. J Biophotonics. 12 (6), e201800378(2019).
- Levy, A., Zheng, J. Y., Lazarowitz, S. G. The tobamovirus Turnip vein clearing virus 30-kilodalton movement protein localizes to novel nuclear filaments to enhance virus infection. J Virol. 87 (11), 6428-6440 (2013).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell-movement protein. mBio. 7 (1), e2052-e2015 (2016).
- Kumar, G., Dasgupta, I. Variability, functions and interactions of plant virus movement proteins: what do we know so far. Microorganisms. 9 (4), 695(2021).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of nondestructive cell-to-cell and systemic movement of plant viruses. Crit Rev Plant Sci. 23 (3), 195-250 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon