Method Article
Análisis microscópico confocal de la clasificación de proteínas a plasmodesmos en Nicotiana benthamiana
En este artículo
Resumen
Este protocolo describe la selección de marcadores plasmodesmales óptimos para análisis basados en microscopía confocal de proteínas dirigidas a plasmodesmos durante las interacciones virus-plasmodesmos o el transporte plasmodesmal.
Resumen
Los plasmodesmos son nanoporos membranosos que conectan el citoplasma de las células vegetales adyacentes y permiten el tráfico de nutrientes, macromoléculas y virus invasores de célula a célula. Los plasmodesmos desempeñan un papel fundamental en la regulación de la comunicación intercelular, contribuyendo al desarrollo de las plantas, las respuestas ambientales y las interacciones con los patógenos virales. El descubrimiento de la localización plasmodésmica de proteínas vegetales o virales podría proporcionar información funcional útil sobre la proteína y es importante para comprender los mecanismos de las interacciones planta-virus. Para facilitar estos estudios, describimos un protocolo para el análisis basado en microscopía confocal de diferentes proteínas diana plasmodesmales para seleccionar el mejor marcador plasmodesmal para estudiar las interacciones virus-plasmodesmos o el transporte plasmodesmal. Específicamente, los análisis de estos eventos se ilustran utilizando la proteína de movimiento de célula a célula (MP) del virus de limpieza de venas de nabo (TVCV), la proteína 5 localizada por plasmodesmos de Arabidopsis (PDLP5) y la proteína de unión a callosa 1 de Plasmodesmata (PDCB1). Los datos de localización del plasmodesma de proteínas se analizan en paralelo con la visualización global de plasmodesmos mediante tinción con azul de anilina de los tejidos muestreados. Estos enfoques se pueden adaptar fácilmente para analizar la localización plasmodésmica de cualquier proteína celular o patógena en la planta.
Introducción
Los plasmodesmos (PD) desempeñan un papel fundamental en el control del desarrollo de las plantas, las respuestas ambientales y las interacciones con patógenos virales a través de la regulación de la comunicación intercelular 1,2. La EP se forma inicialmente durante la citocinesis, con cientos de PD insertados en la nueva célula entre las dos células hijas, suministrando así los canales para la comunicación de célula a célula 3,4. La EP es una estructura rica en membranas, que contiene la membrana derivada del retículo endoplásmico (RE), un desmotúbulo trans-PD, en la parte central de los poros revestidos por la membrana plasmática 3,4. Los enfoques proteómicos comparativos identificaron numerosas proteínas funcionales de la PD, incluidas las β-1,3-glucanasas (BG), las calosa sintasas (CALS), las proteínas localizadas en plasmodesmos (PDLP), las proteínas de unión a callosa (PDCB), las proteínas de la región transmembrana de múltiples dominios C2 (MCTP)3 y las quinasas similares a receptores repetidos (RLK) ricas en leucina5. Recientemente, Kirk et al. desarrollaron una herramienta denominada plasmodesmos en el proteoma in silico 1 (PIP1), que permitió predecir nuevas proteínas PD en 22 especies de plantas6. La DP varía en permeabilidad y estructura durante el desarrollo de la planta y la respuesta a diversos estreses. La deposición e hidrólisis de calosa (β-1,3-glucano) en la región del cuello que rodea la EP es uno de los mecanismos ampliamente conocidos de la regulación de la EP7.
Muchos microbios patógenos, incluidos hongos, bacterias y virus, pueden manipular la dilatación o la estructura de la EP durante su infección 2,8,9. Magnaporthe oryzae, el agente causante del brusone del arroz, despliega hifas invasivas intracelulares para pasar de una célula a otra a través de PD8. Un patógeno bacteriano Pseudomonas syringae pv. El tomate requiere una proteína efectora HopO1-1 para el movimiento intercelular y la propagación en la planta huésped a través de la interacción y la desestabilización de PDLP7, aumentando así el flujo molecular en las células vecinas en Arabidopsis9. Sin embargo, los virus de plantas son más versátiles en la regulación de la EP durante su transmisión intercelular, con la proteína de movimiento viral (MP) promoviendo el movimiento de célula a célula2. Debido a su importante función en la regulación del desarrollo y crecimiento de las plantas, así como a su interacción con los microbios fitopatógenos, la EP ha ganado cada vez más atención en los últimos años. En Arabidopsis thaliana, hay dos tipos principales de proteínas funcionales de la EP, PDLP (1-8) y PDCB (1-5), y se encontró que muchas de ellas, por ejemplo, PDLP5 1,10,11, PDLP112, PDLP613, PDLP714 y PDCB115, desempeñan un papel en la manipulación de la permeabilidad de la PD a través de la regulación de la deposición de calosa. Sin embargo, se encontró que algunos PDLPs tenían una redundancia funcional, por ejemplo, los mutantes knockout de pdlp1 y pdlp1,2 no afectaron el tráfico molecular, aunque los mutantes knockout dobles de pdlp1,3 y pdlp2,3 mostraron una mayor permeabilidad plasmodésmica16. Curiosamente, la regulación a la baja/knockout o la sobreexpresión de PDLP5 por sí sola da lugar a un aumento o disminución de la permeabilidad plasmodésica, respectivamente 1,17. Recientemente, Li et al. han encontrado que PDLP5 y PDLP6 funcionan en diferentes interfaces celulares13. Estos resultados indican que PDLP5 podría tener funciones no redundantes con otros PDLP.
Debido a la función crítica de la EP en la comunicación intercelular, desarrollamos un protocolo para desplegar las proteínas PDLP5 y PDCB1 de las plantas y la proteína de movimiento viral de célula a célula (MP) del virus de limpieza de venas de nabo (TVCV) como marcadores de EP simples, convenientes y confiables para la experimentación en biología celular. Para una mayor verificación, se procedió en paralelo a la visualización de la DP mediante la tinción con azul de anilina de los tejidos muestreados. Los protocolos descritos para la localización de PD de PDLP5, PDCB1 y TVCV MP se pueden adaptar fácilmente para analizar la posible localización de PD de cualquier proteína celular o derivada de patógenos en plantas vivas.
Protocolo
Los detalles de los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Crecimiento de las plantas
- Cultive semillas de Nicotiana benthamiana en suelo húmedo en una cámara de ambiente controlado a 23 °C bajo 16 h de luz y 8 h de oscuridad.
- Después de aproximadamente 2 semanas, transfiera con cuidado las plántulas con los gránulos de turba alrededor de sus raíces a macetas más grandes y continúe el crecimiento en las mismas condiciones durante 4-5 semanas para los experimentos de agroinfiltración.
NOTA: No utilice las plantas cuando comiencen a florecer, ya que la acumulación de GFP disminuirá en la etapa de inflorescencia18.
2. Construcción vectorial
- Utilice la PCR para amplificar las secuencias codificantes de interés con la ADN polimerasa de alta fidelidad Q5 y clonarlas en el vector de entrada pDONR207 mediante la reacción BP19 utilizando un kit de clonasa BP disponible en el mercado.
- Transfiera los genes diana de los plásmidos de entrada resultantes al vector de destino pPZP-RCS2A-nptII-DEST-EGFP-N1 mediante la reacciónLR 19 utilizando un kit de clonasa LR disponible en el mercado para producir plásmidos con la etiqueta EGFP fusionada con el extremo C-terminal de las proteínas diana.
- Verificar todos los constructos por PCR y secuenciación19.
3. Agroinfiltración
- Estrías de células de Agrobacterium tumefaciens EHA105 que contienen diferentes vectores en placas de agar LB e incubadas durante 2 días a 28 °C.
- Transfiera una sola colonia a 2 mL de caldo LB e incube durante la noche a 28 °C con agitación (250 rpm).
- Retirar 1 mL del cultivo durante la noche, seguido de la adición de 4 mL de caldo LB fresco, y recultivar durante 1 h en las mismas condiciones.
- Ajuste la suspensión bacteriana a DE600 = 0,1 (DE600 = 0,2 para la coexpresión de dos proteínas) con tampón de infiltración (MgCl2, 10 mM; MES, 10 mM, pH 5,6).
- Mezclar diferentes suspensiones bacterianas en una proporción de 1:1 (v/v) para obtener una concentración final de OD600 = 0,1. (Opcional).
- Incubar la suspensión bacteriana a temperatura ambiente durante 3 h con agitación suave.
- Infiltre la superficie abaxial de las hojas completamente expandidas de diferentes plantas con una jeringa desechable sin aguja de 1 ml y marque el área infiltrada (un color más oscuro que el tejido circundante no infiltrado) con un bolígrafo a prueba de agua.
- Continúe con la sección Microscopía confocal de este protocolo (paso 5).
NOTA: También se pueden utilizar otras cepas de Agrobacterium tumefaciens adecuadas para la transformación de especies vegetales de interés.
4. Mancha azul de anilina
- Añadir una alícuota de 200 μL de azul de anilina al 1% (en tampón de fosfato potásico de 50 mM, pH 8,0) a un portaobjetos de microscopio20.
- Extirpar el área infiltrada de aproximadamente 0,5 cm × 0,5 cm, lejos de la vena, con una cuchilla, y transferir las muestras de tejido foliar a la solución de azul de anilina (lado abaxial hacia arriba) en el portaobjetos del microscopio, y asegúrese de que las muestras de tejido foliar estén sumergidas en la solución de azul de anilina, y luego cúbrala con un cubreobjetos (22 mm x 50 mm).
- Coloque los portaobjetos del microscopio con las muestras en un desecador conectado a una bomba de vacío y evacúe durante 2 min (<0,8 Pa), seguido de una liberación lenta de la presión e incubación en la oscuridad durante 30 min a temperatura ambiente.
- Visualice la señal fluorescente del azul de anilina bajo un microscopio confocal de barrido láser con software compatible.
NOTA: En este caso se utilizó el método de tinción con azul de anilina de Huang et al.20 . En una modificación de esta técnica, se realizó el mismo experimento sin el paso de vacío de 2 minutos, produciendo resultados similares y sugiriendo que el secado al vacío no es esencial para la tinción con anilina. Además, la concentración de la tinción de azul de anilina y el tiempo de tinción pueden variar según la fuente del tejido de la hoja, etc.
5. Microscopía confocal
- Coseche dos hojas de dos plantas en diferentes momentos después de la infiltración. Corta la zona de infiltración en rodajas de aproximadamente 0,5 cm × 0,5 cm de la vena con una cuchilla.
- Coloque las muestras de tejido en una gota de agua estéril (con el lado abaxial hacia arriba) en la parte central de un portaobjetos de microscopio y cúbralas con un cubreobjetos (22 mm x 50 mm), teniendo cuidado de evitar burbujas.
- Asegúrese de que las longitudes de onda de excitación para la detección de señales CFP, GFP y RFP sean de 405 nm, 488 nm y 561 nm, respectivamente, y que los filtros de emisión para la detección sean de 410-602 nm para CFP y de 400-602 nm para EGFP y RFP, con el agujero de alfiler 1 UA y la ganancia maestra establecida en 769 V.
- Visualice la señal fluorescente de las etiquetas autofluorescentes en el área infiltrada utilizando un microscopio confocal de barrido láser con software compatible 1 día, 2 días, 3 días, 5 días, 7 días y 10 días después de la infiltración.
- Utilice una lente de objetivo de 10x y filtros GFP para localizar las células con la señal fluorescente y, a continuación, cambie a una lente de objetivo de 40x para la visualización de la localización subcelular y la grabación de imágenes.
- Recoja 20 imágenes para cada afección, utilizando al menos dos réplicas biológicas independientes.
- Puntuación de la localización de la PD de la proteína ensayada en función del aspecto punteado diagnóstico de la señal en la periferia de la célula21,22.
NOTA: Al detectar la localización subcelular de una proteína desconocida, se recomienda utilizar al menos tres plantas.
6. Análisis de datos
- Utilice el software Fiji para dividir los canales y añadir la barra de escala a las imágenes para su visualización.
- Utilice el software Fiji para dividir los canales antes de medir el valor medio de la escala de grises de cada imagen para evaluar la intensidad de fluorescencia GFP (para MP, PDCB1 y PDLP5) o CFP (para tinción con azul de anilina) en diferentes puntos temporales.
- Utilice el software Fiji para dividir los canales antes de la normalización de las imágenes de GFP (para MP, PDCB1 y PDLP5) o CFP (para la tinción con azul de anilina). Se midió el área de la imagen y se contaron manualmente los puntos de DP. Se calculó el número de puntos de PD por 100 μm2 .
- Utilice el ANOVA de dos factores con la prueba de comparaciones múltiples23 de Tukey para determinar los valores P entre las diferentes muestras y los diferentes puntos de tiempo con software estadístico y gráfico.
NOTA: Para una imagen de fluorescencia de un solo canal, el valor de la escala de grises de cada píxel representa la intensidad de fluorescencia de ese punto24. Aquí, se utilizó el valor medio de la escala de grises para evaluar la intensidad de fluorescencia de cada muestra en diferentes puntos temporales.
Resultados
Para facilitar los estudios de la función de la EP en la fisiología de las plantas y las interacciones con los patógenos, se desarrollaron tres proteínas de referencia sencillas y fiables para la localización de la EP. Se seleccionaron dos proteínas PD celulares y una proteína MP derivada del patógeno codificada por el tobamovirus vegetal TVCV. La localización subcelular de estas proteínas se visualizó utilizando un EGFP reportero autofluorescente fusionado con el extremo C-terminal de cada proteína. En un enfoque alternativo, la DP se visualizó mediante la tinción con azul de anilina de los depósitos de callosa asociados a la EP. Los experimentos mostraron que tanto PDLP5 como MP exhibían una localización punteada de la PD, con MP también localizada en el núcleo de la célula (Figura 1A), como se señaló anteriormente25. Por el contrario, PDCB1 parecía particionarse entre PD y ER (Figura 1A). Para los investigadores con menos experiencia en la interpretación de la localización subcelular de las proteínas marcadas con GFP, la Figura 1A también ilustra el patrón nucleocitoplasmático característico de EGFP expresado en células vegetales. Para confirmar la señal ER de PDCB1, coexpresamos PDCB1 con un marcador ER y descubrimos que la señal PDCB1 se superponía a la señal del marcador ER (Figura 1B). Para evaluar la eficiencia de la localización de la PD de las proteínas ensayadas, se calculó el porcentaje de células que expresan la señal fluorescente que exhiben la localización de la PD de esta señal (Tabla 1).
Para la identificación de la localización subcelular mediante el empleo de proteínas de referencia, es útil comprender la escala temporal de la expresión de la proteína de referencia y los posibles cambios de su localización. La Figura 2 muestra dicha cinética para MP, PDLP5 y PDCB1 cuando la DP se visualizó mediante tinción con azul de anilina. Tanto MP como PDLP5 comenzaron a acumularse en PD ya al día post-infiltración (dpi), y esta acumulación alcanzó un máximo aparente a los 2 dpi, permaneciendo estable durante 5 días más, después de los cuales la intensidad de la señal disminuyó, muy probablemente debido a la degradación de la proteína. El curso temporal de la clasificación subcelular de PDCB1 fue más complejo: durante los primeros 3 dpi, PDCB1 exhibió una señal ER comparativamente fuerte, que oscureció casi por completo la localización de la señal específica de PD, mientras que a los 5 dpi, esta última señal se volvió más pronunciada (Figura 2), aunque en menos células. Curiosamente, la aparición del claro patrón de localización de PD de PDCB1 después de 5 dpi coincidió con un número sustancialmente menor de células que mostraban la señal de EGFP (Figura suplementaria 1). Por lo tanto, PDLP5 y MP pueden funcionar como proteínas de referencia de PD con poco fondo en la mayoría de los otros compartimentos celulares, con una ventana de tiempo de visualización óptima de 2-5 dpi. PDCB1, por otro lado, no es tan adecuado como un marcador de DP.
A continuación, se comprobó que los marcadores de EP conservan su especificidad de localización subcelular en presencia de otras proteínas de la EP. Con este fin, MP-EGFP o PDLP5-EGFP se coexpresó con26 MP localizadores de PD de otro virus de la planta, el virus del mosaico del tabaco (TMV) marcado con CFP. La Figura 3 muestra que, en ambos casos, las proteínas coexpresadas mostraron un claro patrón de localización de la EP. De manera similar, la tinción histológica con azul de anilina específica de PD de los tejidos que expresan MP-EGFP o PDLP5-EGFP no detectó interferencia con la localización de PD de las señales de EGFP y azul de anilina (Figura 3).
Los estudios de localización de la EP a menudo se benefician de una estimación más cuantitativa de la expresión general de la proteína de referencia y la señal de la EP. Este enfoque de cuantificación se ilustra en la Figura 4 y la Figura 5. Para la cuantificación de la expresión general, se midió la intensidad de la señal de PDLP5 y MP. Ambas proteínas mostraron señales fuertes en todos los puntos de tiempo de visualización durante el período de 10 dpi, con una intensidad de señal máxima de 3 dpi para PDLP5 y de 2 dpi para MP (Figura 4). No hubo diferencias estadísticamente significativas en la intensidad de la señal entre PDLP5 y MP en ninguno de los puntos temporales de visualización. La intensidad de la señal PDCB1 mostró una mayor fluctuación entre puntos de tiempo, con la señal más fuerte observada a 7 ppp (Figura 4), aunque fue difícil recopilar datos estadísticamente significativos en este punto de tiempo debido a la escasez de células expresivas. Por lo tanto, los primeros 3 ppp representan el momento óptimo para detectar la expresión de PDCB1. Como se esperaba, las muestras de tinción con azul de anilina mostraron una señal más estable que las proteínas expresadas transitoriamente (Figura 4).
A continuación, se cuantificaron los puntos de PD formados por PDLP5 y MP. PDLP5 mostró un número estable de puntos de PD durante todo el período de observación, sin diferencias estadísticamente significativas entre dos de los puntos de tiempo probados (Figura 5). El mayor número de puntos de PD formados por MP se detectó a los 2 dpi, y se mantuvieron relativamente estables hasta el final del período de observación. La comparación del número de puntos de PD entre PDLP5 y MP no reveló diferencias estadísticamente significativas en la mayoría de los momentos de muestreo (Figura 5). El número de puntos de PD teñidos con azul de anilina también se mantuvo estable, aunque disminuyó ligeramente a los 10 dpi. El número de puntos de DP decorados con azul de anilina, PDLP5 o MP fue similar entre sí con significación estadística (Figura 5), lo que concuerda con la especificidad de estos marcadores para la EP. No se analizó el número de puntos de PD para PDCB1 porque, en puntos de tiempo anteriores, la señal de PD estaba oscurecida por la señal de ER, y en puntos de tiempo posteriores, se observó solo en un número relativamente pequeño de células (ver Figura 2).

Figura 1: Localización de plasmodesmos (PD) de TVCV MP, PDLP5 y PDCB1 expresada transitoriamente en Nicotiana benthamiana y tinción con azul de anilina de PD. (A) Localización de PD de MP-EGFP, PDLP5-EGFP y PDCB1-EGFP y tinción con anilina. La señal EGFP está en verde, la señal azul de anilina está en azul y la autofluorescencia de plástidos está en rojo. (B) Coexpresión de MP-EGFP, PDLP5-EGFP y PDCB1-EGFP con el marcador de retículo endoplásmico (RE) marcado con mRFP. Las imágenes se registraron a los 2 días después de la infiltración (dpi) y son secciones confocales individuales representativas de dos experimentos independientes (N = 20 imágenes de 2 plantas). La señal EGFP está en verde, la señal mRFP está en púrpura y la autofluorescencia de plástidos está en rojo. Las flechas amarillas indican PDCB1 localizado en el ER. Barras de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Evolución temporal de la localización de la EP de TVCV MP, PDLP5 y PDCB1 expresada transitoriamente en Nicotiana benthamiana y de la tinción con azul de anilina de la EP. Las imágenes se registraron a 2 ppp y son secciones confocales individuales representativas de dos experimentos independientes (N = 20 imágenes de 2 plantas para todos los sistemas, excepto 7 ppp y 10 ppp para PDCB1-EGFP, en los que menos células exhibieron la señal). La señal EGFP está en verde y la señal azul de anilina está en azul. Barras de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
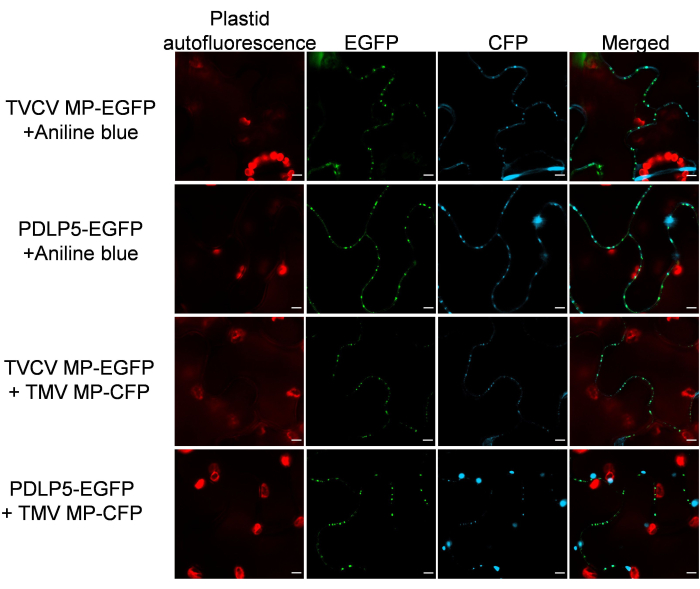
Figura 3: TVCV MP y PDLP5 conservan su localización de PD después de la coexpresión con un TMV MP que localiza PD y se cueca con azul de anilina. Las imágenes se registraron a 2 dpi y son secciones confocales individuales representativas de N ≥ 10 imágenes de 4-6 plantas. La señal EGFP está en verde, la señal CFP y la señal azul anilina están en azul y la autofluorescencia del plástido está en rojo. Barras de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
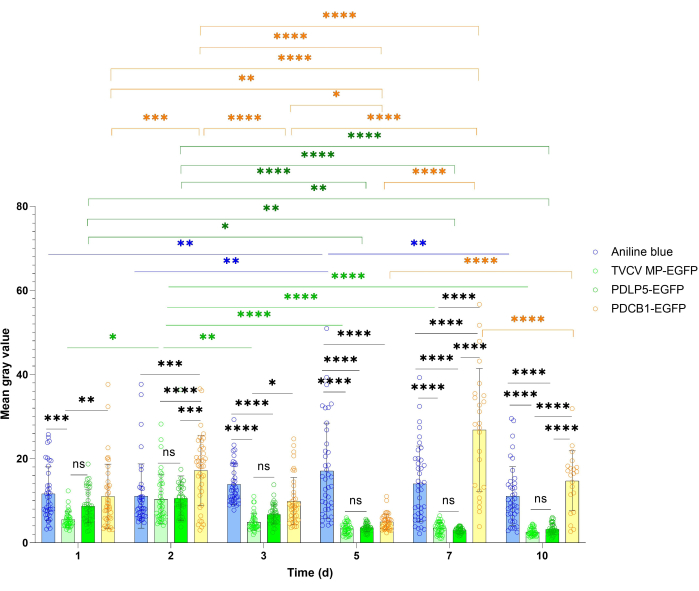
Figura 4: Curso temporal de la intensidad de fluorescencia de TVCV MP, PDLP5 y PDCB1 expresada transitoriamente en tinción de Nicotiana benthamiana y PD con azul de anilina. Los datos cuantitativos se recogieron de los experimentos descritos en la Figura 2. Las barras de error representan la desviación estándar de la media de varios experimentos. Los puntos de datos individuales se indican como la media de cada medición. Las diferencias entre los valores medios evaluados por ANOVA de dos factores con la prueba de comparaciones múltiples de Tukey son estadísticamente significativas para los valores P. *P < 0,05, **P < 0,01 y ***P < 0,001, ****P < 0,0001; ns, no estadísticamente significativo. Barras azules, tinción de anilina; barras de color verde claro, MP-EGFP, barras de color verde oscuro, PDLP5-EGFP; barras amarillas, PDCB1-EGFP. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Curso temporal de la formación de PD puncta por TVCV MP y PDLP5 expresados transitoriamente en Nicotiana benthamiana y por tinción con azul de anilina de PD. Los datos cuantitativos se recogieron de los experimentos descritos en la Figura 2. Las barras de error representan la desviación estándar de la media de varios experimentos. Los puntos de datos individuales se indican como la media de cada medición. Las diferencias entre los valores medios evaluados por ANOVA de dos factores con la prueba de comparaciones múltiples de Tukey son estadísticamente significativas para los valores P. *P < 0,05, **P < 0,01, ***P < 0,001, ****P < 0,0001. Barras azules, tinción de anilina; barras de color verde claro, MP-EGFP, barras de color verde oscuro, PDLP5-EGFP. Haga clic aquí para ver una versión más grande de esta figura.
| Experimento I | ||||
| Total de celdas | Células con localización de PD | Células con señal mixta | Células con localización de PD (%) | |
| Azul de anilina | 40 | 40 | 4 | 100 |
| MP-EGFP | 40 | 40 | 4 | 100 |
| PDCB1-EGFP | 40 | 3 | 38 | 7.5 |
| PDLP5-EGFP | 40 | 40 | 0 | 100 |
| Experimento II | ||||
| Total de celdas | Células con localización de PD | Células con señal mixta | Células con localización de PD (%) | |
| Azul de anilina | 40 | 40 | 8 | 100 |
| MP-EGFP | 40 | 39 | 8 | 97.5 |
| PDCB1-EGFP | 40 | 12 | 31 | 30 |
| PDLP5-EGFP | 40 | 39 | 2 | 97.5 |
Tabla 1: Eficiencia de la localización de la DP.
Figura suplementaria 1: Localización de PD de TVCV MP, PDLP5 y PDCB1 expresados transitoriamente en Nicotiana benthamiana. Las imágenes se grabaron a 5 ppp (A), 7 ppp (B) y 10 ppp (C). La señal EGFP es verde y la autofluorescencia del plástido es roja. Barras de escala = 20 μm. Haga clic aquí para descargar este archivo.
Discusión
Cualquier estudio biológico celular de la comunicación intercelular de las plantas y el transporte de célula a célula durante el desarrollo normal y la morfogénesis de las plantas, así como durante las interacciones entre plantas y patógenos, requiere la detección y el seguimiento de la clasificación de las proteínas, tanto endógenas como codificadas por patógenos, en las conexiones intercelulares de las plantas, los plasmodesmos (PD). Estos experimentos se verían facilitados sustancialmente por el uso de proteínas de referencia, ya sean endógenas o derivadas de patógenos, que se localizan fiel y consistentemente en la EP, decorando y visualizando estas estructuras. Esta necesidad se aborda aquí mediante la selección de tres proteínas localizadoras de PD conocidas: Arabidopsis PDLP5 y PDCB1, y MP del patógeno tobamoviral TVCV. Estas proteínas se marcaron en su extremo C con el indicador autofluorescente EGFP, y la capacidad de las proteínas de fusión resultantes para clasificarse en PD se determinó siguiendo su expresión transitoria en hojas de Nicotiana benthamiana y análisis de microscopía confocal de los tejidos expresores. Para la visualización general de la EP en estos tejidos, se utilizó el método estándar de tinción con azul de anilina.
Se cree que los MP virales de plantas se dividen en dos grupos principales en cuanto a sus efectos sobre la estructura e integridad de la EP: los MP que aumentan la permeabilidad a la PD de forma reversible y sin alteraciones estructurales en la DP y los MP que controlan la DP modificando su estructura. Los MP de tobamovirus, como el TVCV, pertenecen al primer grupo de los MP, posicionándolos como marcadores atractivos de EP para estudios de localización subcelular27,28.
Estos experimentos posicionaron a PDLP5-EGFP y MP-EGFP como marcadores biológicos fiables y reproducibles de la EP, con una eficiencia de detección de la EP comparable a la del colorante azul de anilina. La localización de PD de PDLP5-EGFP y MP-EGFP se detectó durante todo el período de observación de 10 dpi, con el momento óptimo para observar el patrón de localización característico de PD entre 2 dpi y 3 dpi. Aunque la expresión del promotor 35S puede resultar en el silenciamiento postranscripcional del gen expresado, esto ocurre a menudo después de una expresión prolongada en plantas transgénicas transformadas de manera estable. Por otro lado, el uso de este fuerte promotor constitutivo es importante para la fácil detección de la proteína marcadora. Si la expresión de la proteína de referencia disminuye después de 7-10 dpi, se deben usar períodos más cortos, es decir, 2-3 dpi. No se detectaron diferencias significativas en el patrón general de localización subcelular entre PDLP5 y MP, excepto que la MP se acumuló en el núcleo celular además de la PD. Por el contrario, no se recomienda el uso de PDCB1 como marcador de EP per se debido a la complejidad de su patrón de localización subcelular, que incluye una localización sustancial en el RE.
La facilidad de detección de la señal de fluorescencia del EGFP también dependía de la etapa de crecimiento de la planta; específicamente, en N. benthamiana, la menor acumulación de señal de EGFP se observó en la etapa de inflorescencia18.
Este estudio tuvo como objetivo desarrollar los marcadores de localización de la EP de forma máxima exclusiva para otros compartimentos subcelulares. Así, su uso como referencias para la localización de proteínas de interés con capacidades adicionales de localización subcelular debe combinarse con la expresión de otros marcadores subcelulares, como los específicos del RE, las mitocondrias, la membrana plasmática, etc. En este caso, la inclusión de células adicionales de Agrobacterium para la expresión de estos marcadores debe tenerse en cuenta al calcular la concentración final de los cultivos de agroinfiltración, es decir, OD600 de los cultivos líquidos. Obviamente, la concentración celular y los tiempos de crecimiento de la planta deben adaptarse para la máxima expresión y la detección de patrones de localización de PD en otras especies de plantas.
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
El trabajo en el laboratorio de VC fue apoyado por subvenciones de NIH (R35GM144059), NSF (MCB 1913165 e IOS 1758046) y BARD (IS-5276-20) a VC. Los financiadores no tuvieron ningún papel en el diseño del estudio, la recopilación e interpretación de los datos, ni en la decisión de publicarlo.
Materiales
| Name | Company | Catalog Number | Comments |
| ABT AC 1 phase motor | BRANDTECH | ABF63/4C-7RQ | |
| Agrobacterium tumefaciens EHA105 | |||
| Contamination control | CCI | ||
| Gateway BP Clonase II Enzyme mix | Invitrogen | #11789020 | |
| Gateway LR Clonase II Enzyme mix | Invitrogen | #11791020 | |
| GraphPad Prism 8.0.1. | GraphPad Software Inc. | ||
| Image J | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation | ||
| Laser scanning confocal microscope | Zeiss | LSM 900 | |
| Nicotiana benthamiana | Plant species | ||
| pDONR207 | Invitrogen | #12213013 | |
| Q5 High-Fidelity DNA Polymerase | NEB | #M0491S |
Referencias
- Lee, J., et al. A plasmodesmata-localized protein mediates crosstalk between cell-to-cell communication and innate immunity in Arabidopsis. The Plant Cell. 23 (9), 3353-3373 (2011).
- Benitez-Alfonso, Y., Faulkner, C., Ritzenthaler, C., Maule, A. J. Plasmodesmata gateways to local and systemic virus infection. Mol Plant-Microbe Interact. 23 (11), 1403-1412 (2010).
- Bayer, E. M., Benitez-Alfonso, Y. Plasmodesmata: Channels under pressure. Annu Rev Plant Biol. 75, 21(2024).
- Maule, A. J. Plasmodesmata: Structure, function and biogenesis. Curr Opin Plant Biol. 11, 680-686 (2008).
- Fernandez-Calvino, L., et al. Arabidopsis plasmodesmal proteome. PLoS One. 6 (4), e18880(2011).
- Kirk, P., Amsbury, S., German, L., Gaudioso-Pedraza, R., Benitez-Alfonso, Y. A comparative meta-proteomic pipeline for the identification of plasmodesmata proteins and regulatory conditions in diverse plant species. BMC Biol. 20, 128(2022).
- Levy, A., Erlanger, M., Rosenthal, M., Epel, B. L. A plasmodesmata-associated β-1,3-glucanase in Arabidopsis. The Plant J. 49 (4), 669-682 (2007).
- Kankanala, P., Czymmek, K., Valent, B. Roles for rice membrane dynamics and plasmodesmata during biotrophic invasion by the blast fungus. The Plant Cell. 19 (2), 706-724 (2007).
- Aung, K., et al. Pathogenic bacteria target plant plasmodesmata to colonize and invade surrounding tissues. The Plant Cell. 32 (3), 595-611 (2020).
- Cui, W., Lee, J. Arabidopsis callose synthases CalS1/8 regulate plasmodesmal permeability during stress. Nat Plants. 2 (5), 160(2016).
- Liu, N. J., et al. Phytosphinganine affects plasmodesmata permeability via facilitating PDLP5-stimulated callose accumulation in Arabidopsis. Mol Plant. 13 (1), 128-143 (2020).
- Caillaud, M. C., et al. The plasmodesmal protein PDLP1 localizes to haustoria-associated membranes during downy mildew infection and regulates callose deposition. PLoS Pathog. 10 (11), e1004496(2014).
- Li, Z., Su-Ling, L., Christian, M., Walley, J. W., Aung, K. Plasmodesmata-located protein 6 regulates plasmodesmal function in Arabidopsis vasculature. The Plant Cell. , (2024).
- Chen, X., et al. Arabidopsis PDLP7 modulated plasmodesmata function is related to BG10-dependent glucosidase activity required for callose degradation. Sci Bull. , (2024).
- Simpson, C., Thomas, C., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. The Plant Cell. 21 (2), 581-594 (2009).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLoS Biol. 6 (1), e7(2008).
- Lim, G., et al. Plasmodesmata localizing proteins regulate transport and signaling during systemic acquired immunity in plants. Cell Host Microbe. 19 (4), 541-549 (2016).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnol Bioeng. 96 (3), 608-614 (2007).
- Walhout, A. J. M., et al. GATEWAY recombinational cloning: Application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Huang, C., et al. dsRNA-induced immunity targets plasmodesmata and is suppressed by viral movement proteins. The Plant Cell. 35 (10), 3845-3869 (2023).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. The plasmodesmal localization signal of TMV MP is recognized by plant synaptotagmin SYTA. mBio. 9 (4), e01314-e01318 (2018).
- Ward, S. J., Ramirez, M. D., Neelakantan, H., Walker, E. A. Cannabidiol prevents the development of cold and mechanical allodynia in paclitaxel-treated female C57Bl6 mice. Anesth Analg. 113 (4), 947-950 (2011).
- Erkkilä, M. T., et al. Widefield fluorescence lifetime imaging of protoporphyrin IX for fluorescence-guided neurosurgery: An ex vivo feasibility study. J Biophotonics. 12 (6), e201800378(2019).
- Levy, A., Zheng, J. Y., Lazarowitz, S. G. The tobamovirus Turnip vein clearing virus 30-kilodalton movement protein localizes to novel nuclear filaments to enhance virus infection. J Virol. 87 (11), 6428-6440 (2013).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell-movement protein. mBio. 7 (1), e2052-e2015 (2016).
- Kumar, G., Dasgupta, I. Variability, functions and interactions of plant virus movement proteins: what do we know so far. Microorganisms. 9 (4), 695(2021).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of nondestructive cell-to-cell and systemic movement of plant viruses. Crit Rev Plant Sci. 23 (3), 195-250 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados