Method Article
Isolamento, caracterização e extração de DNA total para identificação de fungos endofíticos em plantas micoheterotróficas
Neste Artigo
Erratum Notice
Resumo
O presente artigo tem como objetivo fornecer protocolos detalhados e adequados para o isolamento de fungos endofíticos associados a plantas, preservação a longo prazo de isolados, caracterização morfológica e extração de DNA total para posterior identificação molecular e análises metagenômicas.
Resumo
As plantas micoheterotróficas apresentam uma das formas mais extremas de dependência micorrízica, tendo perdido totalmente sua capacidade autotrófica. Tão essenciais quanto qualquer outro recurso vital, os fungos com os quais essas plantas se associam intimamente são essenciais para elas. Assim, algumas das técnicas mais relevantes no estudo de espécies micoheterotróficas são as que possibilitam a investigação de fungos associados, especialmente aqueles que habitam raízes e órgãos subterrâneos. Nesse contexto, técnicas de identificação de fungos endofíticos dependentes e independentes de cultura são comumente aplicadas. O isolamento de endófitos fúngicos fornece um meio para identificá-los morfologicamente, analisar sua diversidade e manter inóculos para aplicações na germinação simbiótica de sementes de orquídeas. No entanto, sabe-se que existe uma grande variedade de fungos não cultiváveis habitando tecidos vegetais. Assim, técnicas de identificação molecular independentes de cultura oferecem uma cobertura mais ampla da diversidade e abundância de espécies. Este artigo tem como objetivo fornecer o suporte metodológico necessário para o início de dois procedimentos de investigação: um dependente de cultura e outro independente. Em relação ao protocolo cultura-dependente, são detalhados os processos de coleta e manutenção de amostras de plantas desde os locais de coleta até as instalações laboratoriais, além do isolamento de fungos filamentosos de órgãos subterrâneos e aéreos de plantas micoheterotróficas, manutenção de uma coleção de isolados, caracterização morfológica de hifas pela metodologia de cultura em lâminas e identificação molecular de fungos por extração de DNA total. Englobando metodologias independentes de cultura, os procedimentos detalhados incluem a coleta de amostras de plantas para análises metagenômicas e extração de DNA total de órgãos de plantas aclorofilas usando um kit comercial. Finalmente, protocolos de continuidade (por exemplo, reação em cadeia da polimerase [PCR], sequenciamento) também são sugeridos para análises, e técnicas são apresentadas aqui.
Introdução
Os fungos endofíticos são, por definição, aqueles que habitam o interior dos órgãos e tecidos vegetais em infecções imperceptíveis (isto é, sem causar danos ao seu hospedeiro)1,2. Esses fungos podem interagir de forma neutra ou benéfica com as plantas hospedeiras, podem conferir resistência a patógenos e condições ambientais desfavoráveis e podem contribuir para a síntese de compostos benéficos para a planta (por exemplo, fatores de crescimento e outros fitohormônios)1,3. Os endofíticos micorrízicos são fungos que estabelecem associações micorrízicas com a planta, participando da transferência denutrientes4. Em Orchidaceae, a interação com endófitos micorrízicos é fundamental para a germinação de sementes na grande maioria das espécies e estabelecimento de plântulas em todas as plantas da família5. Nesses contextos, as orquídeas micoheterotróficas representam um caso de total dependência em relação aos seus parceiros micorrízicos, uma vez que dependem da transferência de nutrientes minerais e compostos de carbono por esses fungos durante todo o seu ciclo devida6. Portanto, o isolamento e a identificação de fungos associados são uma base fundamental na investigação de estratégias de vida micoheterotrófica. Além disso, pouco se sabe sobre o papel dos fungos endófitos em plantas micoheterotróficas ou mesmo sobre a real diversidade desses fungos 7,8.
A investigação de fungos endofíticos pode ser realizada por meio de diferentes técnicas, tradicionalmente descritas como independentes ou dependentes de cultura, como, por exemplo: (a) observação direta, (b) isolamento fúngico e identificação morfológica e/ou molecular, e (c) extração total de DNA de tecidos vegetais e identificaçãomolecular9. Na observação direta (a), fungos endofíticos podem ser investigados ainda no interior de células e tecidos vegetais por microscopia de luz oueletrônica9, uma vez que diferentes protocolos de microscopia são detalhados por Pena-Passos et al.10. Pelos métodos de isolamento (b), os endófitos fúngicos podem ser caracterizados de acordo com suas colônias, hifas e morfologia da estrutura reprodutiva ou de resistência. Além disso, por meio de técnicas de isolamento, é possível realizar a identificação molecular de isolados por meio de extração de DNA, amplificação de sequências de identificação molecular (códigos de barras ou impressões digitais) e sequenciamento11. Esta última técnica (c) permite a identificação molecular de fungos endofíticos por extração de DNA no interior dos tecidos vegetais (metacódigo de barras), seguida de preparo e sequenciamento de bibliotecas12.
Além disso, isolados fúngicos podem ser aplicados em ensaios de germinação simbiótica, utilizando sementes de orquídeas autotróficas ou micoheterotróficas. Um exemplo dessa aplicação é a investigação conduzida por Sisti et al.13, descrevendo a germinação e os estágios iniciais de desenvolvimento do protocormo em Pogoniopsis schenckii, uma orquídea micoheterotrófica, em associação com alguns de seus isolados, compostos por fungos endofíticos não micorrízicos. O protocolo de germinação simbiótico aplicado é detalhado e apresentado em vídeo por Pena-Passos et al.10. O isolamento de fungos em associação com diferentes órgãos vegetais permite diversos enfoques de investigação sobre a natureza das interações planta-fungos (por exemplo, compreender aspectos ecológicos ou fisiológicos da associação, bem como investigações sobre a transferência de nutrientes dos fungos para a planta)9.
As metodologias apresentadas na seção 1 baseiam-se em uma coleção de amostras de órgãos subterrâneos, uma vez que esses órgãos apresentam maiores dificuldades de coleta, e são de grande interesse, uma vez que os endófitos micorrízicos os colonizam. No entanto, ambos os protocolos incluídos (etapas 1.1 e 1.2) podem ser aplicados a outros órgãos de plantas micoheterotróficas (por exemplo, rizomas, caules florais e frutos). A metodologia de coleta descrita na etapa 1.1 é designada para o isolamento de fungos endofíticos (seção 2) para caracterização morfológica (seções 4 e 5) e/ou extração total de DNA para identificação de isolados (seção 6). Por outro lado, a metodologia de coleta descrita na etapa 1.2 é atribuída exclusivamente à extração total de DNA de tecidos vegetais para técnicas de metabarcoding (seção 7). Na seção 3, são apresentados quatro métodos para armazenamento e preservação de fungos filamentosos, dois para armazenamento de curta duração (3-6 meses) e os outros dois adequados para armazenamento de longo prazo (>1 ano). A caracterização morfológica (seções 4 e 5) pode ser associada à identificação molecular para reforçá-la e fornecer informações importantes sobre macro e micromorfologia fúngica. A Figura 1 resume as metodologias coletivas descritas a seguir.

Figura 1: Sumarização esquemática dos métodos apresentados. Coleta de plantas e isolamento, preservação e identificação molecular de fungos por metodologias dependentes e independentes de cultura. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Coleta de amostras de plantas
- Coleta de amostras para métodos dependentes de cultura
- Cavar cuidadosamente os órgãos subterrâneos; Estes podem ser raízes, caules, rizomas ou órgãos de armazenamento da planta a ser coletada. Além de solos altamente compactos, colete essas amostras manualmente.
OBS: O uso de ferramentas como espátulas ou colheres nesta etapa é desaconselhável, pois pode danificar as estruturas frágeis das plantas micoheterotróficas e causar contaminação dos tecidos por fungos não endofíticos. - Colete o maior número possível de órgãos subterrâneos. Mantenha as amostras em sacos de papel dentro de um recipiente refrigerado (por exemplo, caixa de espuma de poliestireno ou saco térmico com gelo). Se os órgãos aéreos também forem coletados, transporte-os separadamente dos subterrâneos.
- Cavar cuidadosamente os órgãos subterrâneos; Estes podem ser raízes, caules, rizomas ou órgãos de armazenamento da planta a ser coletada. Além de solos altamente compactos, colete essas amostras manualmente.
- Coleta de amostras para métodos independentes de cultura
- Escavar cuidadosamente as raízes da planta a ser coletada sob as mesmas recomendações apontadas na NOTA da etapa 1.1.1.
- Colete o maior número possível de órgãos subterrâneos. Manter as amostras coletadas em criotubos dentro de nitrogênio líquido (opção desejável) ou utilizar tubos centrífugos envoltos por gelo seco (opção alternativa). Manter os órgãos aéreos separados, se coletados.
2. Isolamento de fungos endofíticos associados a órgãos vegetais14
NOTA: Todos os materiais, soluções e reagentes utilizados nesta secção devem ser estéreis. Aqueles que não puderem ser comprados já esterilizados devem ser autoclavados a 121 °C por 20 min.
- Desinfestação superficial de órgãos vegetais
- Lavar as amostras recolhidas (passo 1.1.2) em água corrente e remover o máximo possível de substrato e outros detritos que as amostras possam ter.
- Dentro de uma capela de fluxo laminar, mantenha as amostras lavadas submersas em etanol 70% por 1 min (um copo ou um frasco de vidro podem ser usados).
- Transfira as amostras para outro recipiente com hipoclorito de sódio com cloro ativo a 2% por 3 min.
- Mude as amostras para um recipiente com etanol 70% e mantenha submersas por 1 min. Em seguida, lave as amostras sequencialmente em dois recipientes com água destilada.
- Instalação de fragmentos vegetais em meio de cultura
NOTA: Cada etapa detalhada na etapa 2.2 deve ser conduzida dentro de uma capela de fluxo laminar.- Antes da instalação, prepare placas de Petri (8-10 cm de diâmetro) com 19,5 g/L de meio de cultura de batata-dextrose ágar (PDA) + 7 g/L de ágar bacteriológico + 3 mL/L de antibióticos (por exemplo, estreptomicina, penicilina, tetraciclina, ampicilina).
- Manter as placas de Petri com o meio de cultura a 36 °C por 24 h antes de usá-las para garantir que não haja contaminação. Descarte pratos contaminados com colônias de bactérias ou fungos.
OBS: A adição de antibióticos no meio é essencial nessa etapa, pois os órgãos são altamente infectados por bactérias que podem inibir o crescimento fúngico. Verificar se os antibióticos podem ser aquecidos durante a autoclavagem; alguns antibióticos devem ser adicionados após o resfriamento do meio a cerca de 36 °C (antes de colocá-lo nos pratos). - Imediatamente após a desinfestação superficial das amostras e ainda dentro de uma capela de fluxo laminar, em PDA, inocular algumas gotas da água destilada do último recipiente utilizado para lavar as amostras. Esta etapa é importante para avaliar a eficácia da desinfestação superficial das amostras.
- Em uma placa autoclavada vazia, coloque as amostras e use um bisturi flamejado e pinça para seccionar as amostras até uma espessura de cerca de 0,2 cm.
- Corte amostras cilíndricas longitudinalmente em duas metades, se desejado para amplificar a superfície em contato com o meio. Para expor melhor as frutas e outros órgãos mais esféricos, pique ou corte bem as estruturas. As sementes também podem ser importantes fontes fúngicas, portanto, certifique-se de que o fruto seccionado as exponha ao meio.
NOTA: Somente órgãos saudáveis, sem danos teciduais ou sinais de possíveis doenças ou infecções patogênicas, devem ser usados no isolamento de fungos endofíticos. - Distribuir cinco fragmentos dos órgãos subterrâneos nas placas de Petri com BDA + antibiótico. Certifique-se de que os fragmentos estejam o mais longe possível um do outro e também não estejam tocando as bordas do prato. Não organize nenhum fragmento na mídia. Preparar réplicas de cada órgão instalado, como precaução em caso de contaminação.
- Sele as placas de Petri com película aderente e guarde-as no escuro a 25-27 °C (de preferência em uma incubadora) por 5 dias.
- Cálculo da frequência de isolamento (FI)11
- Após 5 dias de incubação dos fragmentos de órgãos, calcular o FI, de acordo com o número de fragmentos incubados que apresentam colônias fúngicas crescendo dividido pelo total de fragmentos incubados, conforme representado na seguinte equação:

- Após 5 dias de incubação dos fragmentos de órgãos, calcular o FI, de acordo com o número de fragmentos incubados que apresentam colônias fúngicas crescendo dividido pelo total de fragmentos incubados, conforme representado na seguinte equação:
- Purificação de isolados fúngicos por estriação e subcultivo
NOTA: Cada passo detalhado na etapa 2.4 deve ser conduzido dentro de uma capela de fluxo laminar.- Preparar placas de Petri (5 cm de diâmetro) com 5-7 g/L de ágar-ágar (AA; somente ágar bacteriológico). Manter os pratos por 24 h a 36 °C para eliminar os possivelmente contaminados.
- Identifique cada colônia fúngica cultivada com um código e delimite suas margens no lado inferior do prato (pode ser feito com um marcador permanente). Diferencie as colônias por cor, padrão de crescimento, textura e formato de margem.
- Com um palito de madeira autoclavado, usando a ponta fina, recuperar uma pequena quantidade de micélio de uma colônia de fungos. Concentrar-se preferencialmente nas margens da colônia e escolher uma área o mais distante possível de outra colônia, evitando recuperar mais de um tipo de colônia ao mesmo tempo.
- Usando o mesmo palito com micélio na ponta, estriar o AA produzindo três estrias (sulcos). Certifique-se de que cada estria esteja a uma distância de 1 cm da outra e das bordas do prato. Escreva o código apropriado (usando papel adesivo e lápis), lacre os pratos e mantenha-os incubados no escuro a 25-27 °C por 3 dias.
- Prepare placas de Petri (8 cm de diâmetro) com 39 g/L de PDA. Não é necessário adicionar antibióticos nesta fase.
- Após incubar as placas AA, observe-as meticulosamente contra a luz (de uma lâmpada ou de uma janela), com o objetivo de identificar hifas finas formando colônias individuais. Delimitar a área de uma colônia individual por prato, usando um marcador permanente no lado inferior da placa de Petri.
- Em uma capela de fluxo laminar, use um palito autoclavado para cortar uma porção do meio que contém a colônia e transferir o volume de corte para o centro de uma nova placa de PDA.
- Identifique as placas de Petri com os códigos dos isolados, sele as placas com película aderente e mantenha-as no escuro a 25-27 °C por 7-14 dias.
3. Preservação de isolados fúngicos purificados
NOTA: Todos os materiais, soluções e reagentes utilizados nesta secção devem ser estéreis. Aqueles que não puderem ser comprados já esterilizados devem ser autoclavados a 121 °C por 20 min.
- Preservação pelo método de Castellani ou em óleo mineral (3-6 meses)11,15
- Preparar tubos de microcentrífuga de 2 mL com 0,5 mL de água destilada ou contendo 0,5 mL de óleo mineral (dependendo do método escolhido). Certifique-se de que os tubos estejam vazios autoclavados e que a água e o óleo autoclavados sejam adicionados aos tubos dentro de uma capela de fluxo laminar.
- Em uma capela de fluxo laminar, colocar as placas de Petri com isolados purificados já cultivados em BDA (39 g/L) por 7-14 dias. Usando um palito autoclavado, corte cuboides pequenos (0,5 cm x 0,5 cm na área superior) de meio contendo as margens do micélio.
- Coloque de quatro a seis cuboides nos tubos de microcentrífuga com água destilada (método de Castellani) ou óleo mineral. Conservar os tubos no escuro, a 25 °C pelo tempo que for necessário, observando as limitações de tempo do método.
NOTA: Evite adicionar muitos cuboides aos tubos e deixá-los cheios, o que pode aumentar as chances de contaminação. É possível manter os tubos refrigerados, o que pode favorecer a preservação de alguns isolados por mais tempo. Currah et al.16 recomendam o armazenamento de réplicas de isolados de fungos micorrízicos de orquídeas tropicais refrigerados e a 25 °C, pois podem ser perdidos quando armazenados em câmara fria. - Recupere um cuboide e coloque-o no centro de uma nova placa PDA para cultivar um isolado armazenado.
- Criopreservação em grãos de arroz descascados (>1 ano)17
- Lave os grãos de arroz descascados em água corrente e cozinhe-os até que a casca de arroz comece a abrir. Distribuir os grãos cozidos em tubos de ensaio de vidro com tampa de rosca e autoclave duas vezes com intervalo de 24 h entre elas.
- Em uma capela de fluxo laminar, colocar as placas de Petri com isolados purificados já cultivados em BDA (39 g/L) por 7-14 dias. Com palito autoclavado, recuperar cinco pequenos fragmentos de hifas das margens do micélio e inoculá-los no tubo contendo grãos de arroz autoclavados.
OBS: A inoculação de hifas em diferentes pontos e profundidades do tubo garante que o isolado colonize os grãos de arroz descascados em menos tempo. - Incubar os tubos no escuro a 25-27 °C durante 14 dias. Agite os tubos, agitando-os a cada 3 dias para manter os grãos individualizados.
- Depois de observar o crescimento de fungos nos grãos de arroz, distribua os grãos em uma placa de Petri autoclavada forrada com papel filtro para absorver a umidade, sobre o papel para que os grãos possam secar. Conservar no escuro a 25-27 °C durante 2-3 dias.
- Prepare criotubos com 1/3 de sílica gel na parte inferior e 1/3 de lã de vidro acima da sílica. Finalmente, distribua 1/3 dos grãos de arroz com micélio fúngico cultivado. Conservar a -20 °C durante 24 h.
- Após 24 h, armazenar os criotubos a -80 °C pelo tempo necessário, observando as limitações de tempo do método.
- Criopreservação em vermiculita com uso de crioprotetor (>1 ano)18
- Preparar um meio de cultura líquido composto de 0,2% de extrato de levedura e 2% de glicose em água destilada. Ajuste o pH para 5 e autoclave-o.
- Distribuir 0,2 g de vermiculita (utilizar granulometria fina) em criotubos com tampa de rosca e autoclavá-los. Adicionar 0,8 mL de meio líquido autoclavado aos criotubos contendo vermiculita.
- Em uma capela de fluxo laminar, colocar as placas de Petri com isolados purificados já cultivados em BDA (39 g/L) por 7-14 dias. Com palito autoclavado, recuperar de três a cinco fragmentos de hifas das margens do micélio e inocular ao longo do tubo contendo vermiculita + meio de cultura líquido. Lembre-se de identificar os criotubos com o respectivo código isolado.
OBS: A inoculação de hifas em diferentes pontos e profundidades do criotubo garante que o isolado colonize a vermiculita em menor tempo. - Armazenar os criotubos no escuro a 25-27 °C até que a colonização da maioria dos grãos de vermiculita possa ser observada, o que geralmente leva em torno de 14 dias.
- Preparar uma solução crioprotetora composta de glicerol a 5% e trealose a 5% em água destilada e autoclavá-la. Deixe a solução esfriar antes de usá-la.
- Após a vermiculita em criotubos ser colonizada pelo respectivo isolado fúngico, distribuir 0,4 mL de crioprotetor em cada tubo e manter os criotubos refrigerados a 4 °C por 48 h. Em seguida, manter os criotubos a -80 °C pelo tempo necessário, observando as limitações de tempo do método.
4. Caracterização macromorfológica de fungos filamentosos (morfologia de colônias)
- Manter um registro fotográfico do micélio cultivado em cada placa de Petri com 39 g/L de PCA por 7-14 dias. Lembre-se de registrar os dois lados da colônia, superior e inferior (reverso). Se os pratos vão ser prontamente usados na seção 6 ou não são mantidos, abra os pratos ao fotografá-los para obter fotos melhores.
- Se interessado em dados quantitativos sobre a morfologia da colônia, a subcultura replica os isolados e os mantém nas mesmas condições de crescimento e por um período constante para registrar o diâmetro da colônia e calcular a taxa de crescimento (geralmente em mm/h).
NOTA: Resultados quantitativos mais sofisticados podem ser obtidos usando diferentes tipos de meios de cultura para comparação19 e considerando as ferramentas estatísticas para tratar dados. - Observar as colônias sob estereomicroscópio para identificar características morfológicas e fotografar em magnificação. Avaliar as colônias de acordo com suas características macromorfológicas, conforme detalhado na seção de resultados. Consultar diversas fontes bibliográficas para ajudar a categorizar e relacionar a macromorfologia com a identificação molecular e/ou morfológica.
5. Caracterização micromorfológica de fungos filamentosos (morfologia das hifas)
NOTA: As técnicas micromorfológicas são comparadas na seção de discussão, considerando seus possíveis usos e desvantagens.
- Cultivar os isolados em placas de Petri com 39 g/L de PDA por 7-14 dias. Para avaliar possíveis fungos micorrízicos de orquídeas, cultivar os isolados em ágar 17 g/L de fubá (CMA; ou outro meio limitante de nutrientes) por 3-7 dias19.
- Montagem Tease
- Trabalhe dentro de uma capela de fluxo laminar. Coloque uma gota de uma mancha escolhida (passo 5.5) sobre uma lâmina de vidro limpa.
- Usando um palito autoclavado ou outro material estéril, remova cuidadosamente algumas hifas do isolado crescido e coloque-as na gota de mancha. Coloque uma lamínula (de preferência em um ângulo inicial de 45° para evitar bolhas de ar) e analise sob um microscópio de luz.
- Suporte para fita adesiva
NOTA: Esta técnica é geralmente aplicada em culturas fúngicas para ser prontamente utilizada na seção 6 ou não mantida, uma vez que a fita adesiva não pode ser autoclavada, e contaminações podem ocorrer após a execução do método.- Coloque uma gota de uma mancha escolhida (passo 5.5) sobre uma lâmina de vidro limpa. Corte uma tira de fita adesiva transparente em um tamanho que se encaixe bem na lâmina de vidro e a mancha central caia bem.
- Direcione a superfície pegajosa da tira para a superfície do micélio. Não pressione e tente coletar algumas hifas sem furar muitas delas.
- Cole a fita na lâmina de vidro, garantindo que a mancha esteja em contato com as hifas coletadas. Coloque uma gota de água acima da fita adesiva e coloque uma tampa. Analise a lâmina sob um microscópio de luz.
- Cultura em lâminas de fungos filamentosos20
- Dentro de uma placa de Petri grande (>9 cm de diâmetro, de preferência), coloque: papel filtro na parte inferior, uma haste de vidro em forma de U ou uma adaptação (o objetivo é fornecer uma elevação para a lâmina de vidro), uma lâmina de vidro e duas lamínulas. Placas de Petri mais altas facilitam a manipulação. Autoclave esses kits de placas de Petri, uma para cada isolado.
- Preparar um pequeno volume de 39 g/L PDA ou 17 g/L CMA, adicionar 10 g/L de ágar bacteriológico e autoclave (o volume para uma placa de Petri é suficiente para mais de 30 isolados). Os meios de cultura usados na cultura de slides devem ser mais difíceis do que os meios usuais. Autoclave 100 mL de água destilada.
- Trabalhe dentro de uma capela de fluxo laminar. Coloque o meio líquido em uma placa de Petri, produzindo uma camada em torno de 0,5 cm de altura. Deixe o meio se solidificar. Uma vez sólido, use um bisturi estéril para cortar quadrados do meio que tenham 1 cm x 1 cm de dimensão.
- Abra o kit de placa de Petri dentro de uma capela de fluxo laminar e use pinças flamejadas para organizar o material autoclavado dentro da placa. Disponha o material na ordem numerada, como pode ser visto na Figura 2A, colocando um quadrado do meio de cultura sobre a lâmina de vidro. Antes de colocar a tampa sobre o meio, avance para o passo 5.4.5.
- Use um palito autoclavado para recuperar algumas hifas de um isolado e esfregue cuidadosamente as quatro faces laterais do meio colocadas na lâmina de vidro. Coloque uma lamínula autoclavada sobre o quadrado médio usando pinça flamejada.
- Use uma ponta de pipeta estéril para colocar água autoclavada no papel de filtro, para criar uma câmara úmida. Use um volume para saturar o papel sem colocar água em excesso. Selar a placa de Petri com película aderente e identificá-la com o respectivo código isolado. Mantenha os pratos no escuro a 25-27 °C por 3-7 dias.
- Avaliar o crescimento de hifas na lâmina de vidro e na lamínula. A partir de cada kit de cultura de lâminas são produzidas duas lâminas, uma utilizando a lâmina de vidro com hifas e a lamínula autoclavada, a segunda utilizando a lamínula com hifas e outra com lâmina de vidro.
- Após a observação do crescimento das hifas, desprender cuidadosamente o quadrado do meio da lâmina de vidro (Figura 2B), montar as duas lâminas com a coloração escolhida (passo 5.5) e analisar ao microscópio de luz. Lembre-se de identificar o código de isolamento no slide. Para produzir lâminas semipermanentes, sele a lamínula com esmalte transparente.
- Para produzir lâminas permanentes, lave cuidadosamente o corante das hifas aderidas após a coloração e deixe a lâmina secar. Montagem com meio de montagem rápida (para detalhes, leia Pena-Passos et al.10).
NOTA: A desvantagem de produzir uma lâmina permanente com esta técnica é que esporos e estruturas não aderidas ao vidro podem ser lavados.
- Métodos de coloração e observação de hifas
- Lactofenol azul de algodão (LPCB)21: Adicionar 20 mL de ácido lático, 40 mL de glicerol e 20 mL de água destilada. Dissolva 20 g de cristais de fenol nesta solução aquecendo suavemente. Dissolver 0,05 g de azul de metilo (azul de algodão, ou 2 mL de solução aquosa a 1%). O LPCB também pode ser facilmente adquirido já preparado.
CUIDADO: O fenol é altamente tóxico e volátil; manipulá-lo exclusivamente dentro de um exaustor e usar luvas. - Azul de toluidina O10(TBO): Preparar 0,05% TBO em tampão fosfato 0,1 M (pH 6,8).
- Vermelho Congo22,23,24: Preparar uma solução de 1% de vermelho Congo em água destilada e filtrá-la. Incubar por 5-10 min. Este corante também pode ser aplicado para microscopia de fluorescência25.
- Ao observar e fotografar hifas fúngicas ao microscópio de luz, consulte diversas fontes bibliográficas para ajudar a identificar estruturas (confira a discussão).
- Lactofenol azul de algodão (LPCB)21: Adicionar 20 mL de ácido lático, 40 mL de glicerol e 20 mL de água destilada. Dissolva 20 g de cristais de fenol nesta solução aquecendo suavemente. Dissolver 0,05 g de azul de metilo (azul de algodão, ou 2 mL de solução aquosa a 1%). O LPCB também pode ser facilmente adquirido já preparado.

Figura 2: Procedimentos para cultivo em lâminas de fungos filamentosos . (A) Configuração esquemática de um kit de cultura de lâminas, onde os números indicam a ordem de disposição dos elementos. (B) Descolamento do quadrado do meio de cultura após o crescimento das hifas é observado na lâmina de vidro e na lamínula. Clique aqui para ver uma versão maior desta figura.
6. Extração total de DNA de isolados fúngicos (protocolo caseiro26 com modificações 27)
NOTA: Todos os materiais, soluções e reagentes utilizados nesta secção devem ser estéreis. Aqueles que não puderem ser comprados já esterilizados devem ser autoclavados a 121 °C por 20 min. Use luvas durante todo o protocolo e realize algumas etapas dentro de uma coifa.
- Preparar o tampão de extração: dodecil sulfato de sódio (SDS) a 1%, NaCl 250 mM, Tris-HCl 200 mM (pH 8,0) e ácido etilenodiaminotetracético (EDTA) 25 mM. Cultivar os isolados purificados em 39 g/L de PCA por 7-14 dias.
- Raspar cuidadosamente o micélio de um isolado usando uma espátula ou uma colher e transferir os fragmentos para uma argamassa autoclavada, evitando transferir o meio de cultura com o agregado de hifas. Usando um pilão de porcelana, triture o micélio com nitrogênio líquido para um pó fino. Não deixe o micélio ser moído derreter, adicionando nitrogênio para evitar isso.
- Adicionar 1 ml de tampão de extracção num tubo de microcentrífuga de 2 ml e colocar a amostra moída até à marca de 1,5 ml. Agite suavemente o conteúdo dos tubos para homogeneizar.
- Agitar os tubos em um vórtice por 5 s e colocá-los em um termobloco a 65 °C por 20 min. Homogeneize cuidadosamente o conteúdo por inversão a cada 7-10 min.
- Centrifugar os tubos a 10.000 x g por 10 min a 4 °C. Transferir 800 μL da fase superior para um novo tubo de microcentrífuga de 2 mL, adicionar 800 μL de fenol aos tubos e misturar o conteúdo por inversão.
CUIDADO: O fenol é um reagente altamente tóxico e volátil; Pode causar eritema, gangrena e necrose tecidual. Quando inalada, pode causar dispneia e tosse. A absorção sistêmica pode danificar o fígado, os rins e o sistema nervoso central. Manipule o fenol exclusivamente dentro de uma capa de fumaça e use luvas.
OBS: A partir do próximo passo, use luvas e conduza o protocolo dentro de um exaustor. - Centrifugar os tubos a 10.000 x g por 10 min a 4 °C e transferir 800 μL da fase superior para um novo tubo de 2 mL, evitando cuidadosamente a transferência do conteúdo da fase inferior.
- Adicionar 400 μL de fenol e 400 μL de clorofórmio aos tubos e misturar o conteúdo por inversão. Centrifugar-os a 10.000 x g por 10 min a 4 °C.
CUIDADO: O clorofórmio é um reagente altamente tóxico e volátil; Pode causar irritação e lesões quando em contato com a pele e, se inalada, afeta o sistema nervoso central e cardiorrespiratório, fígado e rins. Manipule o clorofórmio exclusivamente dentro de um exaustor e use luvas. - Recuperar 800 μL ou menos da fase superior para um novo tubo de 2 mL. Adicionar 800 μL de clorofórmio, misturar o conteúdo por inversão e centrifugar a 10.000 x g durante 10 min a 4 °C.
OBS: Nesta etapa, nenhum resíduo da fase inferior deve ser transferido para tubos novos. Assim, tenha cuidado redobrado ao recuperar a fase superior. - Transferir 600-800 μL da fase superior para um novo tubo de 1,5 mL e adicionar 450 μL de isopropanol. Misture o conteúdo por inversão e incube a 25 °C durante 5 min.
- Centrifugar os tubos a 10.000 x g durante 5 min a 4 °C e eliminar o sobrenadante utilizando uma micropipeta. Tenha cuidado para não descartar o pellet depositado no fundo do tubo.
- Adicionar 500 μL de etanol 80% e centrifugar por 5 min. Repita duas vezes caso o pellet não esteja clarificado.
- Retire o etanol usando uma micropipeta e seque o pellet a 37 °C por 30-60 min. Adicionar 30-50 μL de água deionizada, eluíndo o pellet usando uma micropipeta.
- Manter os tubos a 4 °C durante a noite para a eluição completa do ADN e congelar o conteúdo a -20 °C.
7. Extração total de DNA de órgãos vegetais para metodologia de metabarcoding (kit comercial)
OBS: Para a metodologia a seguir, é necessário adquirir o kit comercial indicado na Tabela de Materiais como kit de extração de DNA do solo. Todos os materiais, soluções e reagentes utilizados nesta secção devem ser estéreis. Aqueles que não puderem ser comprados já esterilizados devem ser autoclavados a 121 °C por 20 min. É altamente recomendável o uso de luvas durante todo o protocolo, e as etapas podem ser conduzidas dentro de uma capela de fluxo laminar. O protocolo descrito é modificado de De Souza et al.12, a partir do protocolo detalhado pelo fabricante.
- Com pilões e argamassas de porcelana, triture as raízes colhidas seguindo a etapa 1.2 em nitrogênio líquido, reduzindo as amostras a um pó fino. Adicionar 0,3 g das amostras moídas aos tubos PowerBead e agitar suavemente para homogeneizar.
- Adicione 60 μL de solução C1 (incluída no kit) aos tubos PowerBead e misture o conteúdo por inversão. Usando um homogeneizador de tecido e lisador celular (Tabela de Materiais), amarre os tubos firmemente a um suporte adequado, acople o suporte ao vórtice e ligue o equipamento na velocidade máxima por 10-20 min.
NOTA: Se a solução de C1 for precipitada, aqueça-a a 60 °C até à dissolução completa. - Centrifugar os tubos a 10.000 x g por 30 s a 25 °C. Transferir 500 μL do sobrenadante para tubos de microcentrífuga de 2 mL. O conteúdo transferido pode ser particulado.
- Adicionar 250 μL de solução C2 aos tubos e agitar num vórtice durante 5 s. Incubar a 4 °C durante 5 min e, em seguida, centrifugar a 10.000 x g durante 1-2 min a 25 °C.
- Transferir mais de 600 μL do sobrenadante para novos tubos de 2 mL, evitando o pellet. Adicionar 200 μL da solução C3 e agitar num vórtice durante 5 s.
- Incubar a 4 °C durante 5 min e, em seguida, centrifugar a 10.000 x g durante 1 min a 25 °C. Nesta etapa, certifique-se de que o sobrenadante não seja particulado.
- Transfira mais de 750 μL do sobrenadante para novos tubos de 2 mL, evitando o pellet. Homogeneizar bem a solução C4, adicionar 1.100 μL da solução C4 ao sobrenadante e agitar em vórtice por 5 s.
- Carregar 675 μL do conteúdo dos tubos nas colunas de spin MB, sobre o filtro, e centrifugar a 10.000 x g por 1 min a 25 °C. Descarte o conteúdo líquido.
- Repita a etapa anterior duas vezes até que todo o conteúdo de cada tubo seja processado. Em seguida, adicionar 500 μL da solução C5 no centro do filtro presente na coluna superior do tubo e centrifugar a 10.000 x g por 30 s a 25 °C.
- Descarte o conteúdo líquido e centrifugue novamente nas mesmas condições da etapa anterior. Transfira cuidadosamente a coluna superior da coluna de spin MB para novos tubos de microcentrífuga de 2 mL, evitando gotejar qualquer conteúdo líquido na coluna.
- Adicionar 85-100 μL da solução C6 ao centro do filtro e aguardar 1 min. Centrifugar a 10.000 x g por 30 s a 25 °C e descartar a coluna de rotação MB. Conservar os tubos a -80 °C.
8. Quantificação de DNA em espectrofotômetro (consulte a Tabela de Materiais)
- Usando o espectrofotômetro indicado, abra o software. Selecione a opção Ácido nucleico, escolha a opção DNA e defina a concentração como ng/μL.
- Adicionar 1 μL de água deionizada ao detector do espectrofotômetro e calibrar selecionando a opção Em branco. Após a leitura, limpe suavemente o detector com papel de seda macio.
- Nomeie a amostra de campo com o código isolado da amostra a ser lida e coloque 1 μL dela no detector do equipamento. Selecione a opção Medir; Um gráfico e uma tabela com os resultados são gerados.
- Repetir o passo 8.3 até que todas as amostras tenham sido lidas. Recomenda-se salvar a tabela com os resultados gerados para registro e análise: selecione a opção Relatórios e o local onde o arquivo .xml será salvo.
- Adicione 5 μL de água deionizada ao detector, aguarde alguns minutos e limpe-o suavemente com papel de seda macio.
Resultados
No protocolo de isolamento, considerando que há contaminação da água utilizada na última lavagem e a contaminação também é detectada nas placas de Petri com fragmentos inoculados, diferentes ações podem ser tomadas, dependendo do tipo de contaminante (Tabela 1). Esse procedimento deve ser repetido desde o início no caso de contaminantes fúngicos altamente esporulantes, que também apresentam crescimento acelerado, e bactérias multiplicadoras intensas, resistentes aos antibióticos escolhidos. Em vez disso, se o contaminante fúngico apresentar crescimento lento ou não esporular, é possível simplesmente evitar isolá-lo nas etapas subsequentes do protocolo. Finalmente, contaminações com cepas bacterianas que se multiplicam lentamente também podem ser facilmente evitadas nas etapas posteriores, especialmente na purificação, ao recuperar hifas.
Em todos os casos apresentados anteriormente, o cenário ideal é refazer o procedimento de desinfestação utilizando outras amostras e instalar os fragmentos da planta em meio de cultura que combine outros antibióticos, com um espectro de ação mais amplo, nos casos de bactérias resistentes contaminando as primeiras placas. Entretanto, considerando as dificuldades envolvidas na coleta de novas amostras e obtenção de órgãos de plantas micoheterotróficas, alguns casos podem ser resolvidos com sucesso nas etapas posteriores do protocolo, como apresentado na Tabela 1.
| Contaminante | Característica do contaminante | Possível consequência da contaminação | Ação necessária | |||
| Bactéria | Multiplicação intensa | Inibição total ou parcial do crescimento dos endófitos de interesse | Reiniciar o isolamento desde o início | |||
| Multiplicação lenta | Isolamento da bactéria junto com o endófito de interesse | Evitar colônias de bactérias durante a purificação | ||||
| Parede celular | Crescimento vigoroso e/ou altamente esporulante | Impossibilita isolar e/ou purificar os endófitos de interesse | Reiniciar o isolamento desde o início | |||
| Crescimento lento e/ou não esporulante | Isolamento do contaminante por engano | Evitar o isolamento do contaminante | ||||
Tabela 1: Descrição dos possíveis contaminantes no processo de isolamento de fungos endofíticos, consequências da contaminação e procedimentos de mitigação.
Após 5 dias da instalação dos fragmentos de órgãos no meio BDA, é possível visualizar o crescimento de pequenos grupos de micélios de fungos filamentosos, emergindo do interior dos fragmentos (Figura 3). Cada morfotipo micelial deve resultar em um isolado fúngico ao final do protocolo de isolamento, considerando que cada um deles deve ser estriado em uma nova placa de Petri com meio AA para purificação. Recomenda-se que as placas originais da instalação do fragmento não sejam descartadas antes de completar todo o protocolo de isolamento. Podem ser mantidos refrigerados a 4 °C até à obtenção das colónias purificadas. Além disso, em relação a essa parte do protocolo, a análise de FI pode ser muito útil na avaliação da porcentagem de amostras de tecido que resultam em fungos isolados entre o total de amostras instaladas.
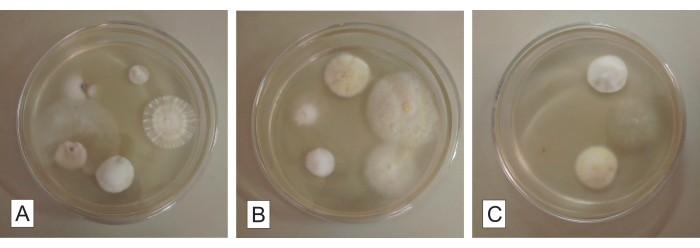
Figura 3: Fragmentos radiculares para isolamento fúngico em ágar batata-dextrose. Fungos endofíticos cultiváveis crescendo a partir de fragmentos radiculares de uma orquídea micoheterotrófica após cerca de 5 dias de incubação. Cada placa de Petri (A, B e C) contém cinco fragmentos de raízes. Clique aqui para ver uma versão maior desta figura.
Após 3 dias após a incubação das placas de AA para purificação do isolado, minúsculas colônias hifais, quase imperceptíveis, deveriam ter crescido a partir das estrias no meio AA. Nesta fase, caso houvesse contaminação por bactérias de crescimento lento nas placas de isolamento, é possível que essas bactérias também tenham sido transferidas para as placas AA com as hifas endofíticas fúngicas selecionadas. Se assim for, as bactérias ficarão restritas às regiões da estria, com pouco crescimento quando comparadas ao fungo, permitindo a recuperação apenas do fungo de interesse para novas placas de PDA.
Durante 7-14 dias da inoculação dos isolados fúngicos purificados em placas de PDA, a colônia pura deve crescer centrifugamente (do centro da placa até sua periferia), formando um único micélio circular (Figura 4). Possíveis contaminações são facilmente identificadas nessa etapa, pois comprometem consideravelmente a homogeneidade da colônia quanto ao seu crescimento, forma, aspecto, cor, produção de pigmentos no meio, etc. (Figura 5). Supondo que as colônias finais obtidas não sejam puras, os fungos inicialmente cultivados a partir dos fragmentos de órgãos devem ser submetidos novamente aos procedimentos de purificação, por estriação e subcultura (etapa 2.4). É possível recuperar os isolados dos órgãos inicialmente instalados ou das placas de isolamento finais contendo culturas heterogêneas.
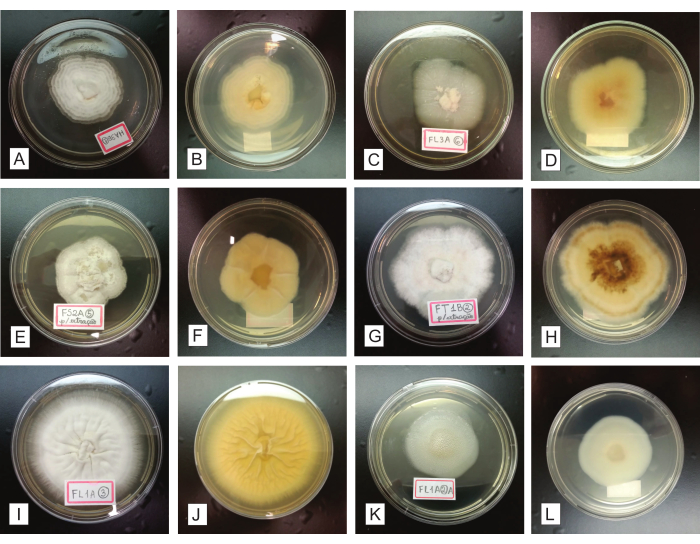
Figura 4: Representação de isolados fúngicos purificados, cultivados por 7-14 dias em meio de cultura BDA. Na primeira e terceira colunas (A, C, E, G, I e K), as colônias registradas são vistas do lado superior; a segunda e a quarta colunas (B, D, F, H, J e L) apresentam as mesmas colônias, respectivamente vistas de baixo. Clique aqui para ver uma versão maior desta figura.

Figura 5: Representação de isolados fúngicos impuros, cultivados por 7-14 dias em meio de cultura BDA. Na primeira coluna (A, D e G), uma visão geral das colônias do lado superior é vista; a segunda coluna (B, E e F) mostra as colônias em detalhes; a terceira coluna (C, F e I) mostra as colônias da parte inferior. Os números representam diferentes morfotipos fúngicos presentes em cada prato, e as linhas representam uma delimitação sutil entre os diferentes isolados fúngicos. Clique aqui para ver uma versão maior desta figura.
A preservação dos isolados não deve ser feita com um único método de armazenamento, pois cada fungo pode apresentar maior ou menor sensibilidade a cada método descrito. É altamente aconselhável escolher pelo menos dois tipos de armazenamento para cada isolado fúngico, garantindo mais chances de sucesso em preservá-lo. Ressaltamos que a inviabilidade de fungos preservados em Castellani ou óleo mineral é esperada após 6 meses ou menos. Os pesquisadores devem considerar se esses são os únicos métodos de preservação escolhidos, para evitar possíveis perdas inesperadas de isolados. Para estender esse período limitado, é possível reativar os isolados fúngicos cultivando-os no meio BDA (39 g/L) por 2-3 semanas e, em seguida, armazenando-os novamente. Após um período de armazenamento, espera-se que os isolados apresentem um crescimento mais lento, comparado à taxa de crescimento observada antes de sua preservação, e colônias com aspectos menos vigorosos (isto é, menos densas, de coloração diferente). Subcultivar algumas vezes em meio de cultura rico em nutrientes é suficiente para restabelecer tais características.
Ao avaliar a macromorfologia das colônias obtidas no procedimento de isolamento, dados qualitativos devem ser coletados, considerando o maior número possível de características: (a) cor da colônia (parte superior e inferior), que podem ser analisadas por meio de guias de cores impressos (por exemplo, Rayner28, Kornerup & Wanscher29, Ridgway30); (b) opacidade das colônias: transparente, opaca, translúcida; (c) pigmentos difusíveis no meio31 (presença/ausência e cor); (d)exsudato 31 (presença/ausência, cor, aspecto geral); (e) estruturas macroscópicas31 (presença/ausência, tipo e aparência; por exemplo, escleródios, picnidia); (f) micélio aéreo e submerso 19,32,21 (presença/ausência, aparência escassa ou abundante); (g) aparência da margem19 (cor, forma, uniformidade - submersa ou aérea); e (h) topografia da colônia (amontoada, enrugada, crateriforme, plana, etc.), a aparência geral e textura - cotonosa, aveludada, pulverulenta, lanosa, sebácea (cerosa), glabra, calcária, esbelta, coriácea, espinhosa, etc.19,21.
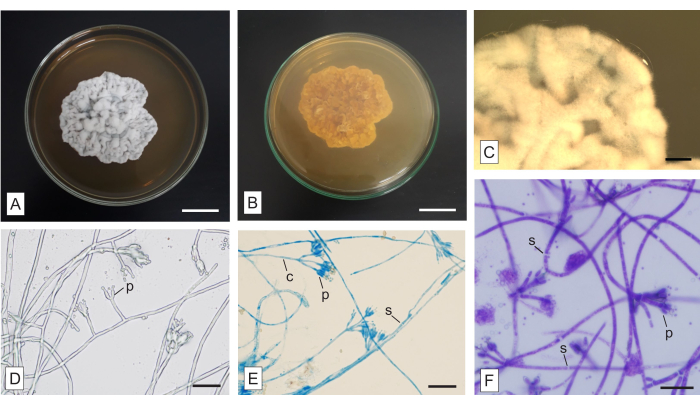
Figura 6: Macro e micromorfologia de um fungo não identificado isolado da orquídea micoheterotrófica Wullschlaegelia aphylla. (A) Aspectos superiores e (B) inferiores da colônia, (C) visão amplificada das hifas aéreas (como visto em um estereomicroscópio). Micromorfologia das hifas (D) sem coloração e coradas com (E) LPCB e (F) TBO. Abreviações: c = conidióforo, p = fialide, s = septo. Barras de escala: A,B = 2 cm; C = 2 mm; D-F = 20 μm. Clique aqui para ver uma versão maior desta figura.
Na Figura 6, é mostrada uma colônia isolada das raízes fusiformes da orquídea micoheterotrófica Wullschlaegelia aphylla, aplicando-se a metodologia descrita. A colônia é esbranquiçada a cinzenta na face superior (Figura 6A) e acastanhada na face inferior (Figura 6B). É opaco, sem pigmentos difusíveis no meio nem exsudatos. Os micélios são aéreos e abundantes, as margens são irregulares e aéreas, e a colônia tem textura aveludada e topografia enrugada (Figura 6A). Estruturas macroscópicas estão ausentes.
O método de cultura de lâminas é tradicional e vantajoso e, embora demorado, pode ser aplicado para produzir lâminas permanentes que podem ser examinadas posteriormente. Os diferentes corantes aqui apresentados podem ser utilizados para evidenciar estruturas importantes, e requerem testes nas amostras, para determinar o tempo de incubação mais adequado. Vermelho Congo e TBO são manchas de uso geral. O vermelho Congo é adequado para demonstrar algumas estruturas delicadas (leitura adicional: Malloch22). A TBO cora o conteúdo celular mais intensamente do que a parede celular do fungo (Figura 6F). Apesar de ser um corante usual para estruturas vegetais e não tão comum em microscopia fúngica, a TBO tem uma importante propriedade metacromática33, o que favorece outras aplicações, como a análise de fungos em tecidosvegetais10. LPCB é um corante amplamente aplicado na análise fúngica, embora exija cautela por causa do fenol. O azul de algodão (sinônimo: azul de metila) é a mancha, o ácido lático é um agente de limpeza e o fenol é um agente de morte21. O LPCB tem afinidade por quitina e evidencia paredes de esporos e ornamentações23,24. Os septos fúngicos não são corados por LPCB (Figura 6E) nem TBO (Figura 6F), facilitando a identificação destas estruturas. A aplicação de colorações é vantajosa para evidenciar estruturas que não são facilmente visualizadas quando as hifas não são coradas (Figura 6D).
Para a extracção de ADN de fungos e amostras de raízes, deve ser respeitada a quantidade de amostra triturada em azoto líquido a adicionar ao tampão de extracção, não excedendo a quantidade indicada no protocolo. Devemos ressaltar que uma grande quantidade de amostra processada não representa simplesmente a obtenção de uma alta concentração de DNA sem grandes inconvenientes, pois pode comprometer consideravelmente a qualidade final do DNA. Isso ocorre ao saturar os seguintes estágios de purificação do DNA. Além disso, a concentração de compostos putativos produzidos por plantas ou fungos (por exemplo, pigmentos, metabólitos secundários), presentes na solução final com DNA, pode atuar na redução da qualidade do DNA e/ou inibir a reação em cadeia da polimerase (PCR). Outra questão que pode reduzir significativamente a qualidade do DNA é sucatear completamente o meio de cultura com micélio fúngico e moê-lo, o que deve ser fortemente evitado. Alguns reagentes do procedimento de extração (por exemplo, fenol, álcool etílico, SDS) e outras substâncias (por exemplo, pigmentos fúngicos) podem ser inibidores interferindo na PCR.
Nas etapas de limpeza do DNA, ao utilizar fenol e clorofórmio, o sobrenadante deve sempre representar a fase com menos impurezas, compreendendo normalmente a fase mais clara. Durante tal estágio, a fase inferior não deve ser recuperada quando aspirar o sobrenadante que será transferido para novos tubos. Ao final do protocolo, o pellet (constituído por DNA) deve apresentar uma coloração translúcida (altamente desejável) a esbranquiçada. Na etapa de secagem, proceder à secagem do DNA em termobloco é fundamental para evaporar completamente o etanol. Após 12 h a 4 °C, o pellet deve ter eluído completamente na solução aquosa adicionada aos tubos, não sendo visível depois disso. Caso a eluição não esteja completa e a concentração de DNA esteja muito abaixo da concentração ideal, é possível manter a solução refrigerada por mais 12 h, sem perdas significativas em sua qualidade.
As análises de DNA no software geram uma tabela, representada na Tabela 2, onde são indicados os dados referentes à quantidade e qualidade do DNA. A quantificação do DNA é dada pela concentração de ácidos nucléicos em nanogramas por microlitro de uma amostra. Considerando a identificação molecular dos fungos isolados, as amostras de DNA devem ter concentração de aproximadamente 200 ng/μL, ideal para as etapas de PCR. Nos casos de amostras com concentrações mais elevadas de ácidos nucleicos, deve ser diluída uma alíquota da amostra, aproximando a concentração do valor anteriormente mencionado. Amostras com concentrações excessivamente altas, como as amostras 1 e 2 da Tabela 2, podem sugerir uma falsa detecção devido a contaminantes na solução. Enquanto isso, amostras com menores concentrações de ácidos nucleicos, como a de número 7 (Tabela 2), ainda podem ser utilizadas para PCR, sendo importante aplicar um volume maior de amostra nas reações. Em relação aos parâmetros de qualidade, é satisfatório que os quocientes 260/280 e 260/230 estejam entre 1,8 e 2,2, conforme indicado nas amostras 3, 5 e 8 (Tabela 2). As amostras que apresentarem tais valores (estando muito abaixo ou acima) devem ser submetidas a novas extrações de DNA, conforme indicado nas amostras 9 e 10 da Tabela 2. Nos casos de amostras com quantidade adequada de DNA e minimamente superior à faixa ideal de qualidade, como sugerem os valores 260/280 e 260/230 dos números 4 e 6 (Tabela 2), a diluição é o procedimento mais benéfico para tornar a concentração de ácido nucleico adequada para a PCR e diluir possíveis inibidores, como reagentes vestigiais do procedimento de extração, nas amostras.
| Amostra | Ácido Nucleico Conc. | Unidade | A260 | A280 | 260/280 | 260/230 | Tipo de amostra |
| 1 | 14491.7 | ng/μL | 289.833 | 141.175 | 2.05 | 1.8 | ADN |
| 2 | 13359.3 | ng/μL | 267.187 | 124.607 | 2.14 | 2.15 | ADN |
| 3 | 1137.6 | ng/μL | 22.751 | 10.574 | 2.15 | 2.13 | ADN |
| 4 | 1472.6 | ng/μL | 29.452 | 13.287 | 2.22 | 2.16 | ADN |
| 5 | 3464.8 | ng/μL | 69.295 | 33.329 | 2.08 | 1.88 | ADN |
| 6 | 1884.2 | ng/μL | 37.684 | 17.912 | 2.1 | 1.78 | ADN |
| 7 | 187.6 | ng/μL | 3.751 | 1.834 | 2.05 | 2.06 | ADN |
| 8 | 1580.3 | ng/μL | 31.607 | 15.281 | 2.07 | 1.98 | ADN |
| 9 | 923.3 | ng/μL | 18.466 | 9.196 | 2.01 | 1.37 | ADN |
| 10 | 2414.4 | ng/μL | 48.287 | 21.008 | 2.3 | 3.45 | ADN |
Tabela 2: Resultados gerados no software a partir de amostras de DNA fúngico analisadas em espectrofotômetro.
Diferente das amostras de DNA de fungos endofíticos isolados, a concentração de DNA nas amostras para análise de metabarcoding deve apresentar grandes quantidades de moléculas de DNA extraídas, com alta qualidade e peso molecular. As amostras devem ser avaliadas por eletroforese em gel de agarose, obtendo-se a máxima integridade possível, com pouca ou nenhuma mancha no gel.
Discussão
A desinfestação superficial de amostras de plantas é uma das etapas mais críticas do protocolo apresentado. Nenhuma contaminação nas placas de PDA com gotas da última lavagem são altamente desejáveis. Bactérias são frequentemente observadas como contaminantes nas placas de isolamento, geralmente mais do que fungos esporulantes transportados pelo ar, considerando que bactérias endofíticas também são comuns nos tecidos vegetais 3,11. Assim, a adição de antibióticos no meio de cultura na instalação dos fragmentos de órgãos é essencial. Melhores resultados são obtidos quando se combinam diferentes tipos de antibióticos, resultando em um espectro de ação mais amplo. Outra consideração importante é o viés intrínseco no isolamento dos fungos, pois o uso de PDA e AA inevitavelmente selecionará certas espécies e desfavorecerá outras. A combinação de outros meios de cultura pode minimizar o viés, mas não eliminar tais limitações9.
Geralmente, fungos que produzem esporos durante o crescimento em placas de Petri apresentam maiores taxas de sobrevivência em protocolos de preservação, em temperaturas mais baixas ou não, e isolados não esporulantes podem não tolerar a preservação de Castellani ou óleo mineral34,35,36, de modo que um método de criopreservação pode ser crucial nesses casos. Além disso, alguns isolados podem ser sensíveis ao congelamento, e a adição de crioprotetores auxilia na preservação, como no método apresentado comvermiculita18. As características da colônia e das hifas podem ser úteis para agrupar os isolados com características semelhantes, facilitando a seleção para uso em tratamentos simbióticos de germinação (conforme detalhado por Pena-Passos et al.10). Essas características também contribuem para a escolha dos métodos de preservação a serem aplicados, principalmente considerando a esporulação ou sua ausência.
A caracterização morfológica dos isolados também melhora e complementa as descrições morfológicas disponíveis na literatura especializada, especialmente quando associada à caracterização molecular. Apesar de ser um desafio com numerosos isolados, a caracterização morfológica dos fungos é demorada e depende de dados publicados. Mesmo que apenas a caracterização molecular esteja planejada para ser realizada, é aconselhável manter um registro fotográfico da aparência da colônia (macromorfologia) e publicá-lo sempre que possível, para contribuir com futuros procedimentos de identificação. A manutenção de fotos das placas onde os fragmentos estão instalados também é importante, para permitir a comparação entre os isolados finais obtidos e os morfotipos observados crescendo nas placas de instalação. Nesse caso, os fungos comparados devem ser cultivados no mesmo tipo de meio. Metodologias adicionais são encontradas em Currah et al.16 para avaliar fungos micorrízicos comuns de orquídeas, como cultivo em meio de ácido tânico (MAT) para diferenciar os gêneros Epulorhiza (teleomorfo: Tulasnella) e Ceratorhiza (teleomorfo: Ceratobasidium), e cultivo em CMA para estimular células monilioides para a caracterização de fungos do complexo Rhizoctonia .
Considerando os métodos detalhados para observar hifas fúngicas sob microscópio de luz, a montagem em tease e a fita adesiva são mais rápidas do que a cultura em lâminas, embora o método de tease mount não seja adequado para analisar esporos e medir ângulos de ramos. A montagem com fita adesiva preserva melhor a organização micelial do que a técnica de tease mount, embora o uso de fita adesiva não seja tão confiável quanto a cultura de lâminas para medir ângulos de ramos (observação dos autores). A cultura de lâminas, apesar de demorada, é a técnica mais adequada para a produção de lâminas semipermanentes e permanentes. A macro e a micromorfologia são analisadas considerando a literatura. Webster e Weber32 é uma extensa fonte geral de informações e referências adicionais. Currah et al.16 e Zettler eCorey37 fornecem informações úteis sobre fungos micorrízicos de orquídeas. Apesar de descreverem fungos de importância médica, Walsh et al.21 e McGinnis31 também são úteis sobre características gerais de colônias de fungos filamentosos, informações visuais/descritivas e referências adicionais para micromorfologia fúngica.
Segundo Yu et al.38, as análises de quantificação de DNA em espectrofotômetro apresentam grande precisão e acurácia, sendo consistentes com análises quantitativas de PCR em tempo real. Uma observação importante é que alguns isolados podem não fornecer amostras de DNA adequadamente puras para as etapas subsequentes de identificação molecular, uma vez que os endófitos fúngicos dos micoheterotróficos são extremamente diversos, especialmente aqueles associados a plantas tropicais39, assim como os metabólitos produzidos. Análises em espectrofotômetros podem ser usadas para avaliar esses casos e diferentes protocolos, ou kits comerciais aplicados para purificação do DNA das amostras. As etapas subsequentes sugeridas na identificação molecular dos isolados são a amplificação da região do espaçador interno transcrito (ITS) e o sequenciamento. A região ITS é um DNA expansor de RNA ribossomal extensivamente empregado na literatura especializada e formalmente aceito como o principal código de barras para identificação defungos9,40. Pode ser amplificada por PCR com os primers ITS1 e ITS441, depois sequenciada na plataforma de Sanger42.
Análises de metabarcode, além de permitirem investigar a riqueza, abundância e composição taxonômica de microrganismos em amostras ambientais e vegetais, podem fornecer resultados de interações positivas ou negativas entre esses microrganismos12. A etapa de maceração com nitrogênio líquido adicionado ao protocolo padrão DNeasy PowerSoil visa quebrar o maior número possível de células fúngicas em relação aos tecidos vegetais, possibilitando melhor amostragem da comunidade fúngica no tecido macerado11. A escolha de primers para sequências fúngicas (códigos de barras) na identificação molecular deve considerar a interferência com o DNA vegetal. Aconselhamos o uso de primers que amplificam regiões variáveis do gene 18S do fungo, por exemplo, ITS1-5,8S-ITS243. Após a obtenção das amostras de DNA, as etapas subsequentes são a preparação das bibliotecas metagenômicas, que dependem da plataforma de sequenciamento escolhida, o sequenciamento posterior das bibliotecas e a análise dos dados obtidos por meio de ferramentas de bioinformática. Recomendamos o uso da plataforma Illumina MiSeq, que representa uma opção mais barata e de alta saída, gerando sequências de 250 pb em curtos períodos de tempo11,44.
Divulgações
Os autores não têm nada a revelar e nenhum conflito de interesses.
Agradecimentos
Agradecemos o financiamento da FAPESP (2015/26479-6) e do CNPq (447453/2014-9). JLSM agradece ao CNPq pelas bolsas de produtividade (303664/2020-7). O MPP agradece à Capes (bolsa de mestrado, processo 88887.600591/2021-00) e ao CNPq.
Materiais
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Referências
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , Springer-Verlag. Berlin. 1-13 (2006).
- Smith, S. E., Read, D. J. Mycorrhizal Symbiosis. , Elsevier Science Academic Press. London. (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777(2022).
- Araújo, W. L., et al. Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, Santarém: UFOPA. 257(2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774(2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589(2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069(2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. Larone's Medically Important Fungi: A Guide to Identification. , ASM Press. (2018).
- Malloch, D. Moulds: Isolation, Cultivation, Identification. , Available from: http://website.nbm-mnb.ca/mycologywebpages/Moulds/Moulds.html (1997).
- Smith, P. Microscopy: Chemical Reagents. British Mycological Society. , Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022).
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , Santarém: UFOPA. 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. Methuen Handbook of Colour. Methuen handbook of colour. , Methuen & Co. Ltd. London. (1967).
- Ridgway, R. Color Standards and Color Nomenclature. , Washington, DC. Published by the author (1912).
- McGinnis, M. R. Laboratory Handbook of Medical Mycology. , Elsevier Science. (2012).
- Webster, J., Weber, R. Introduction to Fungi. , Cambridge University Press. Cambridge, UK. (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados