Method Article
Mycoheterotrophic 식물에서 내생균 균류를 식별하기 위한 분리, 특성화 및 전체 DNA 추출
Erratum Notice
요약
현재 논문은 식물 관련 내생균의 분리, 분리물의 장기 보존, 형태학적 특성 분석 및 후속 분자 식별 및 균유전체 분석을 위한 전체 DNA 추출을 위한 상세하고 적절한 프로토콜을 제공하는 것을 목표로 합니다.
초록
Mycoheterotrophic 식물은 가장 극단적 인 형태의 균근 의존성 중 하나를 나타내며 독립 영양 능력을 완전히 잃었습니다. 다른 중요한 자원과 마찬가지로 이 식물과 밀접하게 연관되어 있는 균류는 식물에게 필수적입니다. 따라서 mycoheterotrophic 종을 연구하는 데 가장 관련성이 높은 기술 중 일부는 관련 균류, 특히 뿌리 및 지하 기관에 서식하는 균류를 조사할 수 있는 기술입니다. 이러한 맥락에서, 배양 의존적 및 배양 비의존성 내생균을 식별하기 위한 기술이 일반적으로 적용된다. 곰팡이 내생식물을 분리하는 것은 형태학적으로 식별하고, 다양성을 분석하고, 난초 종자의 공생 발아에 적용하기 위한 접종물을 유지하는 수단을 제공합니다. 그러나 식물 조직에 서식하는 다양한 비배양 균류가 있는 것으로 알려져 있습니다. 따라서 배양에 독립적인 분자 식별 기술은 종의 다양성과 풍부도에 대한 더 넓은 범위를 제공합니다. 이 기사는 두 가지 조사 절차, 즉 문화 종속 및 독립 조사 절차를 시작하는 데 필요한 방법론적 지원을 제공하는 것을 목표로 합니다. 배양 의존적 프로토콜과 관련하여, 균종속영양 식물의 지하 및 공중 기관에서 사상균을 분리하고, 분리물을 수집하고, 슬라이드 배양 방법론에 의한 균사를 형태학적으로 특성화하고, 전체 DNA 추출을 통해 균류의 분자 식별과 함께 채취 장소에서 실험실 시설까지 식물 샘플을 수집하고 유지하는 과정이 자세히 설명되어 있습니다. 배양에 독립적인 방법론을 포괄하는 세부 절차에는 메타게놈 분석을 위한 식물 샘플 수집과 상용 키트를 사용하여 아클로로필 식물 장기에서 전체 DNA 추출이 포함됩니다. 마지막으로, 연속성 프로토콜(예: 중합효소연쇄반응[PCR], 염기서열분석)도 분석을 위해 제안되며, 여기에 기술이 제시됩니다.
서문
내생균(endophytic fungi)은 식물 장기와 조직의 내부에 눈에 띄지 않는 감염(즉, 숙주에게 해를 끼치지 않고)에 서식하는 균류입니다.1,2. 이러한 균류는 숙주 식물과 중성 또는 유익하게 상호 작용할 수 있고, 병원균 및 불리한 환경 조건에 대한 저항성을 부여할 수 있으며, 식물에 유익한 화합물(예: 성장 인자 및 기타 식물 호르몬)의 합성에 기여할 수 있습니다1,3. 균근 내생식물(Mycorrhizal endophytes)은 식물과 균근 결합을 이루는 균류로, 영양소 전달에 참여한다4. Orchidaceae에서 균근 내생식물과의 상호 작용은 대다수의 종에서 종자 발아와 가족의 모든 식물에서 묘목 설립에 기본입니다5. 이러한 맥락에서, 진균 종속영양 난초는 전체 생애 주기 동안 이러한 균류에 의한 미네랄 영양소와 탄소 화합물 전달에 의존하기 때문에 균근 파트너에 대한 완전한 의존의 경우를 나타낸다6. 따라서 연관 균류의 분리 및 식별은 mycoheterotrophic life 전략을 조사할 때 기본 기반입니다. 더욱이, 균 종속 영양 식물에서 곰팡이 내생식물의 역할이나 이러한 곰팡이 7,8의 실제 다양성에 대해서는 거의 알려져 있지 않습니다.
내생균의 조사는 전통적으로 배양 독립적 또는 의존적이라고 설명되는 다양한 기술을 통해 수행될 수 있는데, 예를 들어 (a) 직접 관찰, (b) 곰팡이 분리 및 형태학적 및/또는 분자 식별, (c) 식물 조직의 전체 DNA 추출 및 분자 식별9. 직접 관찰 (a)에서, 내생균류는 식물 세포 및 조직의 내부에 있는 동안 광 또는 전자 현미경(light or electron microscopy)9에 의해 조사될 수 있으며, 이는 Pena-Passos et al.10에 의해 상이한 현미경 프로토콜이 상세히 기술되어 있기 때문이다. 분리 방법 (b)에 의해, 곰팡이 내생식물은 그들의 군집, 균사 및 생식 또는 저항 구조 형태에 따라 특성화될 수 있다. 또한 분리 기술을 통해 DNA 추출, 분자 식별 서열(바코드 또는 지문)의 증폭 및 염기서열분석을 통해 분리물의 분자 식별을 수행할 수 있습니다11. 후자의 기술(c)은 식물 조직의 내부(메타바코드화)에 있는 동안 DNA 추출에 따라 내생균의 분자 식별을 가능하게 하고, 라이브러리 준비 및 염기서열분석12을 수행한다.
또한, 곰팡이 분리물은 독립 영양 또는 mycoheterotrophic 난초의 종자를 사용하여 공생 발아 시험에 적용될 수 있습니다. 이러한 응용의 예는 Sisti et al.13에 의해 수행된 조사로, 비균근 내생균을 포함하는 일부 분리물과 관련하여 진균종속영양 난초인 Pogoniopsis schenckii에서 원시관 발달의 발아 및 초기 단계를 설명합니다. 적용된 공생 발아 프로토콜은 Pena-Passos et al.10의 비디오에 자세히 설명되어 있습니다. 균류를 다른 식물 기관과 연관시켜 분리하면 식물-균류 상호 작용의 특성에 대한 다양한 조사에 초점을 맞출 수 있습니다(예: 연관성의 생태학적 또는 생리학적 측면을 이해하고 균류에서 식물로의 영양분 전달에 대한 조사)9.
섹션 1에 제시된 방법론은 지하 장기 샘플 수집을 기반으로 하는데, 이러한 장기는 수집에 가장 어려움을 나타내며 균근 내생식물이 군집을 이루기 때문에 주요 관심사입니다. 그러나, 포함된 프로토콜(단계 1.1 및 1.2)은 다른 진균종속영양 식물 기관(예를 들어, 뿌리줄기, 꽃줄기 및 과일)에 적용될 수 있다. 1.1단계에서 설명된 수집 방법은 형태학적 특성화(섹션 4 및 5)를 위한 내생균(섹션 2) 및/또는 분리 식별을 위한 전체 DNA 추출(섹션 6)을 위해 지정됩니다. 반면에, 1.2단계에서 설명한 수집 방법론은 메타바코딩 기법을 위한 식물 조직의 전체 DNA 추출에만 할당됩니다(섹션 7). 섹션 3에서는 사상균 저장 및 보존을 위한 4가지 방법을 제시하는데, 2개는 단기 보관(3-6개월)이고 다른 2개는 장기 보관(>1년)에 적합합니다. 형태학적 특성화(섹션 4 및 5)는 이를 강화하고 곰팡이 거대 및 미세 형태에 대한 중요한 정보를 제공하기 위해 분자 식별과 관련될 수 있습니다. 그림 1 은 그 이후에 설명된 집합적 방법론을 요약한 것입니다.

그림 1: 제시된 방법의 개략적 요약. 식물 수집 및 곰팡이 분리, 보존 및 배양 의존적 및 독립적 방법론에 의한 분자 식별. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 식물 시료 채취
- culture-dependent method에 대한 샘플 수집
- 지하 기관을 조심스럽게 파십시오. 이들은 수집할 식물의 뿌리, 줄기, 뿌리줄기 또는 저장 기관일 수 있습니다. 매우 조밀한 토양 외에도 이러한 샘플을 손으로 수집하십시오.
알림: 이 단계에서 흙손이나 스쿱과 같은 도구를 사용하는 것은 진균종속영양 식물의 취약한 구조를 손상시킬 수 있고 비내생균에 의한 조직 오염을 유발할 수 있으므로 권장되지 않습니다. - 가능한 한 많은 지하 장기를 수집하십시오. 샘플을 냉각된 용기(예: 폴리스티렌 폼 상자 또는 얼음이 든 보온 백) 안에 종이 봉지에 보관합니다. 공중 기관도 수집되는 경우 지하 기관과 별도로 운송하십시오.
- 지하 기관을 조심스럽게 파십시오. 이들은 수집할 식물의 뿌리, 줄기, 뿌리줄기 또는 저장 기관일 수 있습니다. 매우 조밀한 토양 외에도 이러한 샘플을 손으로 수집하십시오.
- culture-independent method에 대한 샘플 수집
- 1.1.1 단계의 NOTE에 지적된 것과 동일한 권장 사항에 따라 수집할 식물의 뿌리를 조심스럽게 파십시오.
- 가능한 한 많은 지하 장기를 수집하십시오. 수집된 시료를 액체 질소 내부의 극저온 튜브에 보관하거나(바람직한 옵션) 드라이아이스로 둘러싸인 원심분리기 튜브를 사용하십시오(대체 옵션). 공중 장기를 수집하는 경우 별도로 유지 관리하십시오.
2. 식물 기관과 관련된 내생균의 분리14
알림: 이 섹션에 사용된 모든 재료, 용액 및 시약은 멸균되어야 합니다. 이미 멸균된 상태로 구입할 수 없는 제품은 121°C에서 20분 동안 고압멸균해야 합니다.
- 식물 기관의 표재성 소독
- 수집된 샘플을 흐르는 물에 씻고(1.1.2단계) 샘플에 있을 수 있는 기질 및 기타 파편을 최대한 제거합니다.
- 층류 후드 내부에서 세척된 샘플을 70% 에탄올에 담근 상태로 1분 동안 유지합니다(비커 또는 유리병 사용 가능).
- 샘플을 2% 활성 염소가 포함된 차아염소산나트륨이 있는 다른 용기로 3분 동안 옮깁니다.
- 샘플을 70% 에탄올이 담긴 용기로 바꾸고 1분 동안 물에 담근 상태로 유지합니다. 그런 다음 증류수로 두 개의 용기에 샘플을 순차적으로 세척합니다.
- 배양 배지에 식물 조각 설치
알림: 2.2단계에 자세히 설명된 모든 단계는 층류 후드 내부에서 수행해야 합니다.- 설치하기 전에 8g/L 감자 포도당 한천 배양 배지(PDA) + 7g/L 세균 한천 + 3mL/L 항생제(예: 스트렙토마이신, 페니실린, 테트라사이클린, 암피실린)로 페트리 접시(직경 8-10cm)를 준비합니다.
- 페트리 접시를 사용하기 전에 배양 배지를 36°C에서 24시간 동안 유지하여 오염이 없는지 확인하십시오. 박테리아나 곰팡이 군집에 오염된 접시는 버리십시오.
알림: 장기가 곰팡이 성장을 억제할 수 있는 박테리아에 의해 심하게 감염되기 때문에 배지에 항생제를 추가하는 것이 이 단계에서 필수적입니다. 고압멸균 중에 항생제가 가열될 수 있는지 확인하십시오. 일부 항생제는 배지를 약 36°C로 식힌 후(접시에 넣기 전에) 추가해야 합니다. - 샘플의 표면 소독 직후 여전히 층류 후드 내부에 PDA에서 샘플을 세척하는 데 사용된 마지막 용기에서 증류수 몇 방울을 접종합니다. 이 단계는 샘플의 표면 소독의 효능을 평가하는 데 중요합니다.
- 빈 오토클레이브 접시에 샘플을 놓고 화염 메스와 집게를 사용하여 샘플을 약 0.2cm 두께로 절단합니다.
- 원통형 샘플을 세로로 두 부분으로 나누어 원하는 경우 매체와 접촉하는 표면을 증폭합니다. 과일과 다른 구형 기관을 더 잘 노출시키려면 구조를 완전히 자르거나 썰어주세요. 씨앗은 또한 중요한 곰팡이 공급원이 될 수 있으므로 절편된 과일이 배지에 노출되도록 하십시오.
알림: 조직 손상이나 가능한 질병 또는 병원성 감염의 신호가 없는 건강한 장기만 진균 내생식물 분리에 사용해야 합니다. - PDA + 항생제와 함께 페트리 접시에 지하 장기의 5 개 조각을 분배하십시오. 파편이 가능한 한 서로 멀리 떨어져 있고 접시 가장자리에 닿지 않도록 하십시오. 매체에 조각을 배열하지 마십시오. 오염에 대비하여 설치된 각 장기의 복제품을 준비합니다.
- 페트리 접시를 접착 필름으로 밀봉하고 25-27 ° C의 어두운 곳에서 5 일 동안 보관하십시오 (바람직하게는 인큐베이터에서).
- 절연 주파수(IF) 미적분11
- 장기 절편 배양 5일 후, 다음 방정식과 같이 배양된 단편의 총계로 나눈 값에 따라 성장하는 곰팡이 군집을 나타내는 배양된 단편의 수에 따라 IF를 계산합니다.

- 장기 절편 배양 5일 후, 다음 방정식과 같이 배양된 단편의 총계로 나눈 값에 따라 성장하는 곰팡이 군집을 나타내는 배양된 단편의 수에 따라 IF를 계산합니다.
- 줄무늬 및 subculturing에 의한 곰팡이 분리물의 정제
알림: 2.4단계에 설명된 모든 단계는 층류 후드 내부에서 수행해야 합니다.- 5-7g/L 한천(AA, 세균학적 한천만 해당)으로 페트리 접시(직경 5cm)를 준비합니다. 오염 가능성이 있는 접시를 제거하기 위해 24°C에서 36시간 동안 접시를 유지하십시오.
- 성장한 각 곰팡이 군체를 코드로 식별하고 접시 바닥면의 여백을 구분합니다(영구 마커로 수행할 수 있음). 색상, 성장 패턴, 질감 및 여백 형식으로 군체를 구별합니다.
- 고압 멸균 된 나무 이쑤시개로 미세한 팁을 사용하여 곰팡이 식민지에서 소량의 균사체를 회수합니다. 가급적이면 군체 가장자리에 집중하고 다른 군체에서 가능한 한 멀리 떨어진 지역을 선택하여 한 번에 두 가지 이상의 군체를 회복하지 마십시오.
- 끝에 균사체가 있는 동일한 이쑤시개를 사용하여 AA를 줄무늬로 만들어 3개의 줄무늬(홈)를 만듭니다. 각 줄무늬가 다른 줄무늬와 접시 가장자리에서 1cm 떨어져 있는지 확인하십시오. 적절한 코드(스티커 용지와 연필 사용)를 작성하고 접시를 밀봉한 다음 25-27°C에서 3일 동안 어두운 곳에서 배양된 상태로 유지합니다.
- 8g/L PDA로 페트리 접시(직경 39cm)를 준비합니다. 이 단계에서 항생제를 추가할 필요는 없습니다.
- AA 접시를 배양한 후 빛(램프 또는 창문)에 대해 꼼꼼하게 관찰하여 개별 군체를 형성하는 미세한 균사를 식별하는 것을 목표로 합니다. 페트리 접시의 바닥면에 있는 영구 마커를 사용하여 접시당 개별 식민지의 면적을 구분합니다.
- 층류 후드에서 오토클레이브 이쑤시개를 사용하여 콜로니가 포함된 매체의 일부를 절단하고 절단 부피를 새 PDA 접시의 중앙으로 옮깁니다.
- 분리 코드로 페트리 접시를 식별하고 접시를 접착 필름으로 밀봉하고 25-27 ° C의 어두운 곳에서 7-14 일 동안 유지합니다.
3. 정제된 곰팡이 분리물의 보존
알림: 이 섹션에 사용된 모든 재료, 용액 및 시약은 멸균되어야 합니다. 이미 멸균된 상태로 구입할 수 없는 제품은 121°C에서 20분 동안 고압멸균해야 합니다.
- Castellani's 방법 또는 미네랄 오일 보존(3-6개월)11,15
- 2mL의 증류수 또는 0.5mL의 미네랄 오일이 들어 있는 0.5mL 미세 원심분리기 튜브를 준비합니다(선택한 방법에 따라 다름). 튜브가 비어 있고 고압멸균된 물과 오일이 층류 후드 내부의 튜브에 추가되었는지 확인합니다.
- 층류 후드에 PDA(39g/L)에서 이미 자란 정제된 분리물과 함께 페트리 접시를 7-14일 동안 놓습니다. 오토클레이브 이쑤시개를 사용하여 균사체 가장자리가 포함된 배지의 작은 직육면체(상단 영역에서 0.5cm x 0.5cm)를 자릅니다.
- 4-6개의 직육면체를 증류수(Castellani's method) 또는 미네랄 오일과 함께 마이크로 원심분리기 튜브에 넣습니다. 튜브를 어두운 곳에서 필요한 만큼 25°C에 보관하고 방법의 시간 제한을 준수하십시오.
알림: 튜브에 너무 많은 직육면체를 추가하여 오염 가능성을 높일 수 있는 가득 채우지 마십시오. 튜브를 냉장 상태로 유지할 수 있으며, 이는 일부 분리물을 더 오래 보존하는 데 유리할 수 있습니다. Currah et al.16 은 열대 난초에서 분리한 균근 균류의 복제물을 냉장 보관하고 25°C에서 보관할 것을 권장하는데, 이는 냉장 보관 시 손실될 수 있기 때문입니다. - 직육면체를 회수하여 새 PDA 접시의 중앙에 놓아 저장된 분리물을 성장시킵니다.
- 껍질을 벗기지 않은 쌀알의 냉동 보존(>1년)17
- 껍질을 벗기지 않은 쌀알은 흐르는 물에 씻고 왕겨가 열리기 시작할 때까지 요리합니다. 조리된 곡물을 스크류 캡이 있는 유리 시험관에 분배하고 그 사이에 24시간 간격으로 두 번 오토클레이브합니다.
- 층류 후드에 PDA(39g/L)에서 이미 자란 정제된 분리물과 함께 페트리 접시를 7-14일 동안 놓습니다. 오토클레이브 이쑤시개를 사용하여 균사체 가장자리에서 5개의 작은 균사 조각을 회수하고 오토클레이브 쌀알이 들어 있는 튜브에 접종합니다.
알림: 튜브의 다른 지점과 깊이에 균사를 접종하면 분리물이 껍질을 벗기지 않은 쌀알을 더 짧은 시간에 식민지화할 수 있습니다. - 25-27 °C의 어두운 곳에서 14 일 동안 튜브를 배양합니다. 곡물을 개성화하기 위해 3일마다 튜브를 소용돌이쳐 튜브를 교반합니다.
- 쌀알에서 곰팡이가 자라는 것을 관찰한 후 습기를 흡수하기 위해 여과지를 깐 오토클레이브 페트리 접시에 곡물을 종이 위에 분배하여 쌀알이 건조될 수 있도록 합니다. 25-27 °C의 어두운 곳에서 2-3일 동안 보관하십시오.
- 바닥에 1/3 실리카겔이 있고 실리카 위에 1/3 유리솜이 있는 극저온 튜브를 준비합니다. 마지막으로 쌀알의 1/3을 자란 곰팡이 균사체와 함께 분배합니다. -20 °C에서 24시간 동안 보관하십시오.
- 24시간 후 -80°C에서 필요한 기간 동안 냉동 튜브를 보관하고 분석법의 시간 제한을 준수합니다.
- 동결 보호제를 사용한 질석의 동결 보존(>1년)18
- 증류수에 0.2% 효모 추출물과 2% 포도당으로 구성된 액체 배양 배지를 준비합니다. pH를 5로 조정하고 오토클레이브합니다.
- 0.2g의 질석(미세 입도계 사용)을 스크류 캡이 있는 극저온 튜브에 분배하고 고압멸균합니다. 0.8mL의 오토클레이브 액체 배지를 질석이 들어 있는 극저온 튜브에 추가합니다.
- 층류 후드에 PDA(39g/L)에서 이미 자란 정제된 분리물과 함께 페트리 접시를 7-14일 동안 놓습니다. 오토클레이브 이쑤시개를 사용하여 균사체 가장자리에서 3-5개의 균사 조각을 회수하고 질석 + 액체 배양 배지가 들어 있는 튜브를 따라 접종합니다. 각각의 분리 코드로 cryotube를 식별하는 것을 잊지 마십시오.
알림: 극저온 튜브의 다양한 지점과 깊이에 균사를 접종하면 분리물이 더 짧은 시간에 질석을 식민지화할 수 있습니다. - 대부분의 질석 입자의 집락화가 관찰될 수 있을 때까지 25-27°C의 어두운 곳에 cryotube를 보관하며, 이는 일반적으로 약 14일이 소요됩니다.
- 증류수에 5% 글리세롤과 5% 트레할로스로 구성된 동결 보호제 용액을 준비하고 고압 멸균합니다. 용액을 사용하기 전에 식히십시오.
- cryotubes의 질석이 각각의 fungal 분리물에 의해 집락화된 후 각 튜브에 0.4mL의 cryoprotectant를 분배하고 cryotube를 4°C에서 48시간 동안 냉장 보관합니다. 그런 다음 필요한 만큼 -80°C에서 극저온 튜브를 유지하고 분석법의 시간 제한을 관찰합니다.
4. 사상균의 거대형태학적 특성화(콜로니 형태학)
- 7-14일 동안 39g/L PDA로 각 페트리 접시에서 자란 균사체의 사진 기록을 유지합니다. 식민지의 양쪽, 위쪽과 아래쪽(역방향)을 등록하는 것을 잊지 마십시오. 섹션 6에서 접시를 쉽게 사용하거나 유지 관리하지 않을 경우 더 나은 사진을 얻기 위해 사진을 찍을 때 접시를 엽니다.
- 콜로니 형태에 대한 정량적 데이터에 관심이 있는 경우, 하위 배양은 분리체를 복제하고 동일한 성장 조건에서 일정한 기간 동안 유지하여 콜로니 직경을 등록하고 성장률(보통 mm/h 단위)을 계산합니다.
참고: 비교를 위해 다양한 유형의 배양 배지를 사용하고19 데이터를 처리하기 위한 통계 도구를 고려함으로써 보다 정교한 정량적 결과를 얻을 수 있습니다. - 또한 실체현미경으로 군체를 관찰하여 형태학적 특성을 식별하고 확대하여 사진을 찍습니다. 결과 섹션에 자세히 설명된 대로 거대형태학적 특성에 따라 군체를 평가합니다. 다양한 서지 출처를 참조하여 거대 형태학을 분류하고 분자 및/또는 형태학적 식별과 관련시키는 데 도움을 줍니다.
5. 사상균의 미세형태학적 특성화(균사 형태학)
참고 : micromorphological 기술은 가능한 용도와 단점을 고려하여 토론 섹션에서 비교됩니다.
- 7-14일 동안 39g/L PDA로 페트리 접시에서 분리물을 성장시킵니다. 추정 난초 균근 균류를 평가하기 위해, 17 g/L 옥수수 가루 한천(CMA; 또는 다른 영양 제한 배지)에서 3-7일 동안 분리물을 성장시킨다19.
- 티즈 마운트
- 층류 후드 내부에서 작업하십시오. 깨끗한 유리 슬라이드에 선택한 얼룩(5.5단계) 한 방울을 놓습니다.
- 오토클레이브 이쑤시개 또는 다른 멸균 재료를 사용하여 성장한 분리물에서 일부 균사를 조심스럽게 제거하고 얼룩 방울에 넣습니다. 커버슬립을 놓고(기포를 피하기 위해 초기 각도 45°를 사용하는 것이 좋음) 광학 현미경으로 분석합니다.
- 접착 테이프 마운트
참고: 이 기술은 일반적으로 접착 테이프를 고압멸균할 수 없고 방법을 실행한 후 오염이 발생할 수 있기 때문에 섹션 6에서 쉽게 사용하거나 유지 관리하지 않는 곰팡이 배양에 적용됩니다.- 깨끗한 유리 슬라이드에 선택한 얼룩(5.5단계) 한 방울을 놓습니다. 투명 접착 테이프 스트립을 유리 슬라이드와 중앙 얼룩 방울에 잘 맞는 크기로 자릅니다.
- 스트립의 끈적끈적한 표면을 균사체 표면으로 향하게 합니다. 누르지 말고 너무 많이 붙이지 않고 균사를 모으십시오.
- 테이프를 유리 슬라이드에 붙여서 얼룩이 수집된 균사와 접촉하도록 합니다. 테이프 위에 물 한 방울을 놓고 커버 슬립을 놓습니다. 광학 현미경으로 슬라이드를 분석합니다.
- 사상균의 슬라이드 배양20
- 큰 페트리 접시 (직경 >9cm, 바람직하게)에는 바닥 부분의 여과지, U 자형 유리 막대 또는 적응 장치 (유리 슬라이드의 높이를 제공하는 것이 목표), 유리 슬라이드 1 개 및 커버 슬립 2 개를 놓습니다. 더 높은 페트리 접시는 조작을 용이하게 합니다. 각 분리물에 대해 하나씩 이러한 Petri 접시 키트를 고압 멸균하십시오.
- 39g/L PDA 또는 17g/L CMA의 소량을 준비하고 10g/L 세균 한천을 추가하고 오토클레이브를 추가합니다(페트리 접시 하나의 부피는 30개 이상의 분리물에 충분함). 슬라이드 배양에 사용되는 배양 배지는 일반 배지보다 단단해야 합니다. 증류수 100mL를 오토클레이브합니다.
- 층류 후드 내부에서 작업하십시오. 액체 매체를 페트리 접시에 넣고 약 0.5cm 높이의 층을 만듭니다. 매체를 응고시키십시오. 고체가 되면 멸균 메스를 사용하여 크기가 1cm x 1cm인 정사각형의 매체를 자릅니다.
- 층류 후드 내부의 페트리 접시 키트를 열고 화염 집게를 사용하여 접시 내부에 고압 멸균 된 재료를 구성합니다. 그림 2A와 같이 번호가 매겨진 순서대로 재료를 배열하고 유리 슬라이드에 정사각형의 배양 배지를 놓습니다. 커버슬립을 매체 위에 놓기 전에 5.4.5단계로 진행하십시오.
- 오토클레이브 이쑤시개를 사용하여 분리물에서 일부 균사를 회수하고 유리 슬라이드에 놓인 배지의 4면에 조심스럽게 문지릅니다. 화염 집게를 사용하여 중간 사각형 위에 오토클레이브 커버슬립을 놓습니다.
- 멸균 피펫 팁을 사용하여 여과지에 오토클레이브된 물을 넣어 습한 챔버를 만듭니다. 물을 과도하게 넣지 않고 용지를 적시도록 볼륨을 사용하십시오. 페트리 접시를 접착 필름으로 밀봉하고 각각의 분리 코드로 식별합니다. 25-27 ° C의 어두운 곳에서 3-7 일 동안 접시를 유지하십시오.
- 유리 슬라이드와 커버슬립에서 균사 성장을 평가합니다. 각 슬라이드 배양 키트에서 두 개의 슬라이드가 생성되는데, 하나는 균사와 오토클레이브 커버슬립이 있는 유리 슬라이드를 사용하고, 두 번째는 균사가 있는 커버슬립과 다른 유리 슬라이드를 사용합니다.
- 균사 성장을 관찰한 후 유리 슬라이드에서 정사각형 배지를 조심스럽게 분리하고(그림 2B) 선택한 염색을 사용하여 두 슬라이드를 장착하고(5.5단계) 광학 현미경으로 분석합니다. 슬라이드에서 격리 코드를 식별해야 합니다. 반영구적인 슬라이드를 만들려면 투명한 매니큐어로 커버슬립을 밀봉하십시오.
- 영구적인 슬라이드를 만들려면 염색 후 부착된 균사에서 염료를 조심스럽게 씻고 슬라이드를 건조시킵니다. 빠른 장착 매체를 사용하여 장착합니다(자세한 내용은 Pena-Passos et al.10 참조).
알림: 이 기술로 영구 슬라이드를 생산할 때의 단점은 유리에 부착되지 않은 포자와 구조물이 세척될 수 있다는 것입니다.
- 염색 방법 및 균사 관찰
- 락토페놀 코튼 블루(LPCB)21: 젖산 20mL, 글리세롤 40mL, 증류수 20mL를 추가합니다. 이 용액에 20g의 페놀 결정을 부드럽게 가열하여 녹입니다. 0.05g의 메틸 블루(면 청색 또는 2mL의 1% 수용액)를 녹입니다. LPCB는 이미 준비된 제품으로도 쉽게 구입할 수 있습니다.
주의: 페놀은 독성이 강하고 휘발성이 강합니다. 흄 후드 내부에서만 조작하고 장갑을 착용하십시오. - 톨루이딘 블루 O10(TBO): 0.1M 인산염 완충액(pH 6.8)에 0.05% TBO를 준비합니다.
- 콩고 레드22,23,24 : 증류수에 1 % 콩고 레드 용액을 준비하고 여과하십시오. 5-10분 동안 배양합니다. 이 염료는 형광 현미경 검사에도 적용될 수 있다(25).
- 광학 현미경으로 곰팡이 균사를 관찰하고 사진을 찍을 때 구조를 식별하는 데 도움이 되도록 다양한 서지 출처를 참조하십시오(토론 확인).
- 락토페놀 코튼 블루(LPCB)21: 젖산 20mL, 글리세롤 40mL, 증류수 20mL를 추가합니다. 이 용액에 20g의 페놀 결정을 부드럽게 가열하여 녹입니다. 0.05g의 메틸 블루(면 청색 또는 2mL의 1% 수용액)를 녹입니다. LPCB는 이미 준비된 제품으로도 쉽게 구입할 수 있습니다.

그림 2: 사상균 의 슬라이드 배양 절차. (A) 슬라이드 배양 키트의 개략도 구성으로, 여기서 숫자는 요소 배열 순서를 나타냅니다. (B) 균사 성장이 유리 슬라이드와 커버슬립에서 관찰된 후 배양 배지의 사각형을 분리하는 단계. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
6. 곰팡이 분리물에서 총 DNA 추출(수정 27이 있는 수제 프로토콜 26)
알림: 이 섹션에 사용된 모든 재료, 용액 및 시약은 멸균되어야 합니다. 이미 멸균된 상태로 구입할 수 없는 제품은 121°C에서 20분 동안 고압멸균해야 합니다. 전체 프로토콜 동안 장갑을 착용하고 흄 후드 내부에서 일부 단계를 수행합니다.
- 추출 완충액을 준비합니다 : 1 % 도데 실 황산 나트륨 (SDS), 250mM NaCl, 200mM Tris-HCl (pH 8.0) 및 25mM 에틸렌 디아민 테트라 아세트산 (EDTA). 정제된 분리물을 39g/L PDA에서 7-14일 동안 성장시킵니다.
- 주걱이나 숟가락을 사용하여 분리물에서 균사체를 조심스럽게 긁어내고 절편을 고압멸균 모르타르로 옮기고 배양 배지를 균사 응집체와 함께 옮기지 않도록 합니다. 도자기 유봉을 사용하여 균사체를 액체 질소로 갈아 고운 가루로 만듭니다. 분쇄되는 균사체가 녹지 않도록 질소를 첨가하십시오.
- 2mL 미세 원심분리 튜브에 1mL의 추출 완충액을 넣고 1.5mL 표시가 될 때까지 분쇄된 샘플을 넣습니다. 균질화하기 위해 튜브의 내용물을 부드럽게 저어줍니다.
- 소용돌이에서 튜브를 5초 동안 저어주고 65°C의 써모블록에 20분 동안 넣습니다. 7-10분마다 반전으로 콘텐츠를 조심스럽게 균질화합니다.
- 튜브를 10,000 x g 에서 4°C에서 10분 동안 원심분리합니다. 상상에서 800μL를 새로운 2mL 마이크로 원심분리 튜브로 옮기고 800μL의 페놀을 튜브에 첨가한 다음 내용물을 반전으로 혼합합니다.
주의 : 페놀은 독성이 강하고 휘발성 시약입니다. 홍반, 괴저, 조직 괴사를 유발할 수 있습니다. 흡입하면 호흡곤란과 기침을 유발할 수 있습니다. 전신 흡수는 간, 신장 및 중추 신경계를 손상시킬 수 있습니다. 흄 후드 내부에서만 페놀을 조작하고 장갑을 착용하십시오.
알림: 다음 단계부터 장갑을 사용하고 흄 후드 내부에서 프로토콜을 수행합니다. - 4°C에서 10분 동안 10,000 x g 의 튜브를 원심분리하고 상상에서 새로운 2mL 튜브로 800μL를 옮기면서 하상(inferior) 내용물의 전이를 주의 깊게 방지합니다.
- 400 μL의 페놀과 400 μL의 클로로포름을 튜브에 넣고 내용물을 반전으로 혼합합니다. 10,000 x g 에서 4 °C에서 10 분 동안 원심 분리합니다.
주의 : 클로로포름은 독성이 강하고 휘발성 시약입니다. 피부에 닿으면 자극과 부상을 유발할 수 있으며 흡입하면 중추 신경계 및 심폐 시스템, 간 및 신장에 영향을 미칩니다. 흄 후드 내부에서만 클로로포름을 조작하고 장갑을 착용하십시오. - 상상에서 800 μL 이하를 새로운 2 mL 튜브로 회수합니다. 클로로포름 800μL를 첨가하고 내용물을 반전으로 혼합한 후 10,000 x g 에서 4°C에서 10분 동안 원심분리합니다.
알림: 이 단계에서는 하상의 잔류물을 새 튜브로 옮기지 않아야 합니다. 따라서 상부 단계를 회복할 때 각별히 주의하십시오. - 상상 600-800 μL를 새로운 1.5 mL 튜브로 옮기고 450 μL의 이소프로판올을 추가합니다. 내용물을 반전하여 혼합하고 25°C에서 5분 동안 배양합니다.
- 튜브를 10,000 x g 에서 4°C에서 5분 동안 원심분리하고 마이크로피펫을 사용하여 상층액을 버립니다. 튜브 바닥에 쌓인 펠릿을 버리지 않도록 주의하십시오.
- 500μL의 80% 에탄올을 넣고 원심분리기를 5분 동안 추가합니다. 펠릿이 명확하지 않은 경우 두 번 반복합니다.
- 마이크로피펫을 사용하여 에탄올을 제거하고 펠릿을 37°C에서 30-60분 동안 건조합니다. 30-50 μL의 탈이온수를 추가하고 마이크로피펫을 사용하여 펠릿을 용출합니다.
- 완전한 DNA 용출을 위해 튜브를 4°C에서 밤새 유지하고 내용물을 -20°C에서 동결합니다.
7. 메타바코딩 방법론을 위한 식물 장기의 총 DNA 추출(상용 키트)
알림: 다음 방법론의 경우 재료 표 에 표시된 상용 키트를 토양 DNA 추출 키트로 구입해야 합니다. 이 섹션에 사용된 모든 재료, 용액 및 시약은 멸균 상태여야 합니다. 이미 멸균된 상태로 구입할 수 없는 제품은 121°C에서 20분 동안 고압멸균해야 합니다. 전체 프로토콜 동안 장갑을 착용하는 것이 좋으며 단계는 층류 후드 내부에서 수행할 수 있습니다. 설명된 프로토콜은 제조업체에서 자세히 설명한 프로토콜에서 De Souza et al.12에서 수정되었습니다.
- 도자기 유봉과 절구를 사용하여 1.2 단계 이후에 수집 한 뿌리를 액체 질소로 갈아서 샘플을 미세한 분말로 줄입니다. 분쇄된 샘플에서 0.3g을 PowerBead 튜브에 추가하고 부드럽게 교반하여 균질화합니다.
- 60μL의 용액 C1(키트에 포함)을 PowerBead 튜브에 추가하고 내용물을 반전으로 혼합합니다. 조직 균질화기 및 세포 용해기(재료 표)를 사용하여 튜브를 적절한 지지대에 단단히 묶고 지지대를 와류에 결합한 다음 10-20분 동안 최대 속도로 장비를 시작합니다.
알림: C1 용액이 침전된 경우 완전히 용해될 때까지 60°C에서 가열하십시오. - 튜브를 10,000 x g 에서 25°C에서 30초 동안 원심분리합니다. 상층액 500μL를 2mL 마이크로 원심분리 튜브로 옮깁니다. 이송된 내용물은 미립자일 수 있습니다.
- 250μL의 용액 C2를 튜브에 추가하고 5초 동안 소용돌이에서 교반합니다. 4°C에서 5분 동안 배양한 다음 10,000 x g 에서 25°C에서 1-2분 동안 원심분리합니다.
- 600μL 이상의 상층액을 펠릿을 피하고 새 2mL 튜브로 옮깁니다. 200μL의 용액 C3를 추가하고 소용돌이에서 5초 동안 교반합니다.
- 4°C에서 5분 동안 배양한 다음 10,000 x g 에서 25°C에서 1분 동안 원심분리합니다. 이 단계에서 상층액이 미립자가 아닌지 확인하십시오.
- 750μL 이상의 상층액을 펠릿을 피하고 새 2mL 튜브로 옮깁니다. 용액 C4를 잘 균질화하고 1,100μL의 용액 C4를 상층액에 첨가하고 5초 동안 소용돌이에서 교반합니다.
- MB 스핀 컬럼의 튜브 내용물에서 675μL를 필터 위에 로드하고 25°C에서 1분 동안 10,000 x g 으로 원심분리합니다. 액체 내용물을 버리십시오.
- 각 튜브의 모든 내용물이 처리될 때까지 이전 단계를 두 번 반복합니다. 그런 다음 튜브의 상부 컬럼에 있는 필터 중앙에 500μL의 용액 C5를 추가하고 25°C에서 30초 동안 10,000 x g 에서 원심분리합니다.
- 액체 내용물을 버리고 이전 단계와 동일한 조건에서 다시 원심분리기를 사용하십시오. MB 스핀 컬럼의 우수한 컬럼을 새로운 2mL 마이크로 원심분리기 튜브로 조심스럽게 옮겨 컬럼에 액체 함량이 떨어지지 않도록 합니다.
- 85-100μL의 용액 C6을 필터 중앙에 추가하고 1분 동안 기다립니다. 25°C에서 30초 동안 10,000 x g 에서 원심분리하고 MB 스핀 컬럼을 폐기합니다. 튜브를 -80 °C에서 보관하십시오.
8. 분광 광도계에서 DNA 정량 (재료 표 확인)
- 표시된 분광 광도계를 사용하여 소프트웨어를 엽니다. 핵산 옵션을 선택하고 DNA 옵션을 선택한 다음 농도를 ng/μL로 설정합니다.
- 분광광도계 검출기에 1μL의 탈이온수를 추가하고 Blank 옵션을 선택하여 보정합니다. 판독 후에는 부드러운 티슈 페이퍼로 감지기를 부드럽게 닦으십시오.
- 필드 이름을 sample의 이름을 s로 지정합니다.amp읽을 샘플의 분리 코드를 지정하고 1μL를 장비 검출기에 넣습니다. 옵션을 선택하십시오 측정 ; 결과가 포함된 그래프와 테이블이 생성됩니다.
- 모든 샘플을 읽을 때까지 8.3단계를 반복합니다. 기록 및 분석을 위해 생성 된 결과와 함께 테이블을 저장하는 것이 좋습니다 : 보고서 옵션을 선택하고 .xml 파일을 저장할 위치를 선택하십시오.
- 검출기에 5μL의 탈이온수를 넣고 몇 분 정도 기다린 다음 부드러운 티슈 페이퍼로 부드럽게 닦습니다.
결과
격리 프로토콜에서 마지막 세척에 사용된 물로 인한 오염이 있고 접종된 파편이 있는 페트리 접시에서도 오염이 검출되는 것을 고려하면 오염 물질의 유형에 따라 다른 조치를 취할 수 있습니다(표 1). 이 절차는 포자가 많은 곰팡이 오염 물질의 경우 처음부터 반복해야 하며, 이는 또한 성장을 가속화하고 선택한 항생제에 내성이 있는 강렬한 증식 박테리아를 나타냅니다. 대신, 곰팡이 오염 물질이 느린 성장을 나타내거나 포자가 형성되지 않는 경우, 프로토콜의 후속 단계에서 분리하는 것을 피할 수 있습니다. 마지막으로, 천천히 증식하는 박테리아 균주에 의한 오염은 균사를 회수할 때 후방 단계, 특히 정제에서 쉽게 피할 수 있습니다.
이전에 제시된 모든 사례에서 이상적인 시나리오는 내성 박테리아가 첫 번째 접시를 오염시키는 경우 다른 샘플을 사용하여 소독 절차를 다시 수행하고 다른 항생제를 결합한 배양 배지에 식물 조각을 더 넓은 범위의 작용과 함께 설치하는 것입니다. 그러나, 새로운 시료를 채취하고 진균종속영양 식물로부터 장기를 얻는 데 수반되는 어려움을 고려할 때, 일부 사례는 표 1에 제시된 바와 같이 프로토콜의 후방 단계에서 성공적으로 해결될 수 있습니다.
| 오염 물질 | 오염 물질 특성 | 오염의 가능한 결과 | 필요한 작업 | |||
| 박테리아 | 강렬한 곱셈 | 관심있는 내생식물의 성장에 대한 전체 또는 부분적 억제 | 처음부터 격리 다시 시작 | |||
| 느린 곱셈 | 관심 있는 내생식물과 함께 박테리아의 분리 | 정제 중 박테리아 식민지를 피하십시오 | ||||
| 균 류 | 활발한 성장 및/또는 높은 포자 형성 | 관심 내생식물을 분리 및/또는 정제하는 것을 불가능하게 만듭니다. | 처음부터 격리 다시 시작 | |||
| 느린 성장 및/또는 비포자 형성 | 실수로 오염 물질의 분리 | 오염 물질의 분리를 피하십시오 | ||||
표 1: 내생균 분리 과정에서 발생할 수 있는 오염 물질, 오염 결과 및 완화 절차에 대한 설명.
PDA 배지에 장기 단편을 설치한 후 5일이 지나면 단편 내부에서 나오는 사상균 균사체의 작은 그룹의 성장을 시각화할 수 있습니다(그림 3). 각 균사체 형태형은 분리 프로토콜이 끝날 때까지 균류를 분리해야 하며, 각 균류는 정제를 위해 AA 배지가 있는 새로운 페트리 접시에 줄무늬가 있어야 한다는 점을 고려합니다. 전체 격리 프로토콜을 완료하기 전에 조각 설치의 원래 접시를 버리지 않는 것이 좋습니다. 정제된 콜로니가 얻어질 때까지 4°C에서 냉장 보관할 수 있습니다. 또한 프로토콜의 이 부분과 관련하여 IF 분석은 설치된 전체 샘플 중에서 분리된 곰팡이를 초래하는 조직 샘플의 비율을 평가하는 데 큰 도움이 될 수 있습니다.
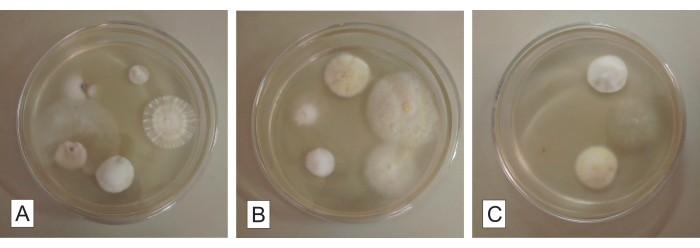
그림 3: 감자 포도당 한천에서 곰팡이 분리를 위한 뿌리 조각. 약 5일간의 배양 후 mycoheterotrophic 난초의 뿌리 조각에서 자라는 배양 가능한 내생균류. 각 페트리 접시(A, B, C)에는 5개의 뿌리 조각이 들어 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
분리 정제를 위해 AA 접시를 배양한 후 3일 후, 거의 감지할 수 없는 미세한 균사 콜로니가 AA 배지의 줄무늬에서 성장해야 합니다. 이 단계에서, 분리 접시에서 느리게 성장하는 박테리아로 인한 오염이 있는 경우, 이러한 박테리아는 선택된 곰팡이 내생식물 균사와 함께 AA 접시로 옮겨졌을 수도 있습니다. 만약 그렇다면, 박테리아는 균류와 비교할 때 거의 성장하지 않고 선조체 영역으로 제한될 것이며, 새로운 PDA 요리에 관심 있는 균류만 회수할 수 있습니다.
PDA 접시에서 정제된 분리 곰팡이를 접종하는 7-14일 동안 순수한 집락은 원심적으로(접시 중심에서 주변으로) 성장하여 유일한 원형 균사체를 형성해야 합니다(그림 4). 가능한 오염은 이 단계에서 쉽게 식별되는데, 이는 콜로니의 성장, 형태, 양상, 색상, 배지에서의 안료 생성 등과 관련하여 콜로니 균질성을 상당히 손상시키기 때문입니다(그림 5). 얻어진 최종 콜로니가 순수하지 않다고 가정하면, 장기 조각에서 처음 성장한 균류는 줄무늬 및 과배양에 의한 정제 절차에 다시 제출되어야 합니다(2.4단계). 초기에 설치된 장기 또는 이질적인 배양액을 포함하는 최종 분리 접시에서 분리물을 회수할 수 있습니다.
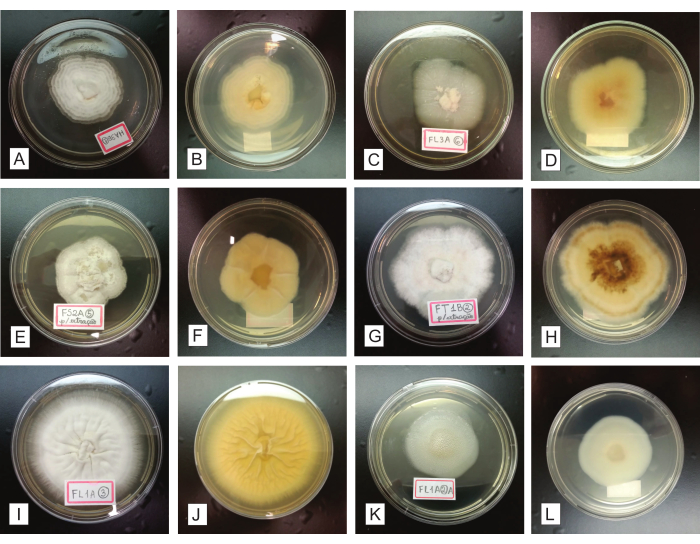
그림 4: PDA 배양 배지에서 7-14일 동안 성장한 정제된 진균 분리물의 표현. 첫 번째 및 세 번째 열(A, C, E, G, I 및 K)에서 등록된 콜로니는 위쪽에서 볼 수 있습니다. 두 번째 및 네 번째 열(B, D, F, H, J 및 L)은 각각 아래쪽에서 볼 때 동일한 콜로니를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: PDA 배양 배지에서 7-14일 동안 성장한 불순 진균 분리물의 표현. 첫 번째 열(A, D 및 G)에서는 위쪽에서 식민지의 일반적인 모습을 볼 수 있습니다. 두 번째 열(B, E 및 F)에는 식민지가 자세히 표시됩니다. 세 번째 열(C, F, I)은 아래쪽에서 콜로니를 보여줍니다. 숫자는 각 접시에 존재하는 서로 다른 곰팡이 형태형을 나타내며 선은 서로 다른 곰팡이 분리 사이의 미묘한 구분을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
분리물의 보존은 각 곰팡이가 설명된 각 방법에 대해 다소 민감성을 나타낼 수 있으므로 하나의 단독 보관 방법을 사용하여 이루어져서는 안 됩니다. 각 곰팡이 분리에 대해 최소 두 가지 유형의 보관을 선택하여 보존에 성공할 가능성을 높이는 것이 좋습니다. 우리는 Castellani 또는 미네랄 오일에 보존 된 곰팡이의 생존 불능이 6 개월 이하로 예상된다는 것을 강조합니다. 연구자들은 예상치 못한 분리물 손실을 피하기 위해 이러한 보존 방법이 선택되는 유일한 보존 방법인지 고려해야 합니다. 이 제한된 기간을 연장하기 위해 PDA 배지(39g/L)에서 2-3주 동안 성장시킨 다음 다시 저장하여 분리된 곰팡이를 재활성화할 수 있습니다. 보관 기간이 지나면 분리된 개체는 보존 전에 관찰된 성장률에 비해 성장이 느려지고 군체가 덜 활발한 측면(즉, 밀도가 낮고 색상이 다름)이 나타날 것으로 예상됩니다. 영양이 풍부한 배양 배지에서 몇 번 과배양하는 것만으로도 이러한 특성을 재확립하기에 충분합니다.
분리 절차에서 얻어진 콜로니의 거대형태를 평가할 때, 가능한 한 많은 특성을 고려하여 정성적 데이터를 수집해야 한다: (a) 인쇄된 컬러 가이드(예: Rayner28, Kornerup & Wanscher29, Ridgway30)를 사용하여 분석할 수 있는 콜로니의 색상(상단 및 하단); (b) 콜로니 불투명도 : 투명, 불투명, 반투명; (c) 매체(31) 내의 확산성 안료(존재/부재 및 색상); (d) 삼출물31 (존재/부재, 색상, 일반적인 외관); (e) 거시적 구조(macroscopic structures)31 (존재/부재, 유형 및 출현; 예를 들어, sclerotia, pycnidia); (f) 공중 및 수중 균사체 19,32,21 (존재/부재, 외관-부족 또는 풍부); (g) 여백 외관19 (색상, 형태, 균일 성 - 수중 또는 공중); (h) 군체 지형(쌓임, 주름, 분화구형, 평평함 등), 일반적인 모양과 질감 - 솜털, 벨벳, 가루, 털, 피지(왁스), 광택, 백악질, 끈적끈적한, 가죽질, 가시 등.19,21.
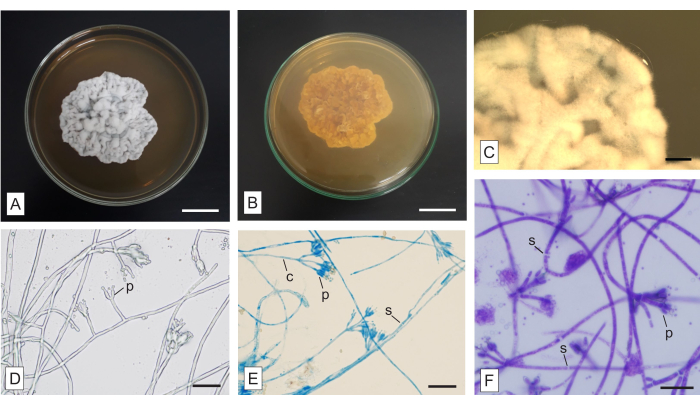
그림 6: mycoheterotrophic orchid Wullschlaegelia aphylla에서 분리한 미확인 균류의 거시형태 및 미시형태. (A) 군체의 상부 및 (B) 하부 측면, (C) 공중 균사의 증폭된 보기(실체현미경에서 볼 수 있음). 염색이 없고 (E) LPCB 및 (F) TBO로 염색된 균사 (D)의 미형태학. 약어: c = 분쇄포자, p = phialide, s = 격막. 눈금 막대: A,B = 2cm; 씨 = 2mm; D-F = 20μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 6에는 설명된 방법론을 적용하여 mycoheterotrophic orchid Wullschlaegelia aphylla의 방추형 뿌리에서 분리된 콜로니가 표시됩니다. 콜로니는 윗면이 희끄무레하거나 회색을 띠고(그림 6A), 아랫면이 갈색을 띤다(그림 6B). 불투명하며 매체에 확산성 안료도 없고 삼출물도 없습니다. 균사체는 공중에 풍부하고 가장자리는 불규칙하고 공중에 있으며 군체는 벨벳 같은 질감과 주름진 지형을 가지고 있습니다(그림 6A). 거시적 구조가 없습니다.
슬라이드 배양 방법은 전통적이고 유리하며, 시간이 많이 걸리지만 나중에 검사할 수 있는 영구 슬라이드를 만드는 데 적용할 수 있습니다. 여기에 제시된 다양한 염료는 중요한 구조를 나타내는 데 사용할 수 있으며 가장 적절한 배양 시간을 결정하기 위해 샘플에서 테스트가 필요합니다. 콩고 레드와 TBO는 범용 얼룩입니다. 콩고 레드는 섬세한 구조를 보여주기에 적절합니다 (추가 읽기 : Malloch22). TBO는 곰팡이 세포벽보다 세포 내용물을 더 강하게 염색합니다(그림 6F). 식물 구조에 대한 일반적인 염료이고 곰팡이 현미경에서는 그리 흔하지 않지만, TBO는 중요한 변색 특성(33)을 가지며, 이는 식물 조직에서의 곰팡이 분석(10)과 같은 다른 응용에 유리하다. LPCB는 곰팡이 분석에 광범위하게 적용되는 염료이지만 페놀 때문에 주의가 필요합니다. 코튼 블루(동의어: 메틸 블루)는 얼룩, 젖산은 투명제, 페놀은 살상제입니다21. LPCB는 키틴질에 대한 친화력을 가지고 있으며 포자 벽과 장식을 나타냅니다23,24. 곰팡이 격막은 LPCB(그림 6E) 또는 TBO(그림 6F)에 의해 염색되지 않으므로 이러한 구조를 쉽게 식별할 수 있습니다. 염색제의 적용은 균사가 염색되지 않았을 때 쉽게 볼 수 없는 구조를 입증하는 데 유리합니다(그림 6D).
곰팡이 및 뿌리 샘플에서 DNA를 추출하려면 추출 완충액에 추가할 액체 질소의 분쇄 샘플의 양을 준수해야 하며 프로토콜에 표시된 양을 초과하지 않아야 합니다. 우리는 많은 양의 처리된 샘플이 최종 DNA 품질을 상당히 손상시킬 수 있기 때문에 큰 단점 없이 단순히 고농도의 DNA를 나타내는 것이 아니라는 점을 강조해야 합니다. 이것은 DNA 정제의 다음 단계를 포화시킬 때 발생합니다. 또한, DNA와 함께 최종 용액에 존재하는 식물 또는 균류에 의해 생성된 추정 화합물(예: 색소, 2차 대사 산물)을 농축하면 DNA 품질을 저하시키거나 중합효소 연쇄 반응(PCR)을 억제하는 역할을 할 수 있습니다. DNA 품질을 크게 저하시킬 수 있는 또 다른 문제는 곰팡이 균사체로 배양 배지를 완전히 폐기하고 분쇄하는 것인데, 이는 강력히 피해야 합니다. 추출 절차로부터의 일부 시약(예를 들어, 페놀, 에틸 알코올, SDS) 및 다른 물질(예를 들어, 곰팡이 색소)은 PCR을 방해하는 억제제일 수 있다.
DNA 정화 단계에서 페놀과 클로로포름을 사용할 때 상층액은 항상 불순물이 적은 상을 나타내야 하며 일반적으로 가장 깨끗한 상을 이해해야 합니다. 이러한 단계에서 새 튜브로 옮겨질 상층액을 흡인할 때 하위가 회복되어서는 안 됩니다. 프로토콜이 끝날 때쯤, 펠릿(DNA에 의해 구성됨)은 반투명(매우 바람직한)에서 희끄무레한 색을 나타내야 합니다. 건조 단계에서 써모블록에서 DNA를 건조하는 것은 에탄올을 완전히 증발시키는 데 필수적입니다. 4°C에서 12시간 후, 펠릿은 튜브에 첨가된 수용액에서 완전히 용리되어야 하며, 그 후에는 보이지 않아야 합니다. 용출이 완료되지 않고 DNA 농도가 이상적인 농도보다 훨씬 낮은 경우 용액의 품질 저하 없이 용액을 12시간 이상 냉장 보관할 수 있습니다.
소프트웨어의 DNA 분석은 표 2에 표시된 대로 DNA의 양 및 품질과 관련된 데이터가 표시된 표를 생성합니다. DNA 정량화는 샘플의 마이크로리터당 나노그램 단위의 핵산 농도로 제공됩니다. 분리된 균류의 분자 식별을 고려할 때 DNA 샘플의 농도는 약 200ng/μL여야 하며, 이는 PCR 단계에 이상적입니다. 핵산 농도가 더 높은 샘플의 경우, 샘플의 부분 표본을 희석하여 농도를 이전에 언급한 값에 근접해야 합니다. 표 2의 샘플 1 및 2와 같이 농도가 과도하게 높은 샘플은 용액의 오염 물질로 인한 오검출을 암시할 수 있습니다. 한편, 번호 7 (표 2)과 같이 핵산 농도가 낮은 샘플은 여전히 PCR에 사용될 수 있으며, 이는 반응에서 더 많은 양의 샘플을 적용하는 것이 중요합니다. 품질 파라미터에 관해서는, 몫(260/280 및 260/230)이 샘플 3, 5 및 8에 나타난 바와 같이, 1.8 내지 2.2 사이인 것이 만족스럽다(표 2). 이러한 값(훨씬 낮거나 높음)을 나타내는 샘플은 표 2의 샘플 9 및 10에 표시된 대로 새로운 DNA 추출에 제출해야 합니다. 숫자 4 및 6의 260/280 및 260/230 값에 의해 제안된 바와 같이, 적절한 양의 DNA를 가지며 이상적인 품질 범위를 최소한으로 초과하는 샘플의 경우(표 2), 희석은 핵산 농도를 PCR에 적합하게 만들고 추출 절차로부터 흔적 시약과 같은 가능한 억제제를 희석하는 가장 유익한 절차이며, 샘플에서.
| 견본 | 핵산 Conc. | 단위 | 대답 260 | 대답 280 | 260/280 | 260/230 | 시료 유형 |
| 1 | 14491.7 | ng/μL | 289.833 | 141.175 | 2.05 | 1.8 | DNA (디엔제이 |
| 2 | 13359.3 | ng/μL | 267.187 | 124.607 | 2.14 | 2.15 | DNA (디엔제이 |
| 3 | 1137.6 | ng/μL | 22.751 | 10.574 | 2.15 | 2.13 | DNA (디엔제이 |
| 4 | 1472.6 | ng/μL | 29.452 | 13.287 | 2.22 | 2.16 | DNA (디엔제이 |
| 5 | 3464.8 | ng/μL | 69.295 | 33.329 | 2.08 | 1.88 | DNA (디엔제이 |
| 6 | 1884.2 | ng/μL | 37.684 | 17.912 | 2.1 | 1.78 | DNA (디엔제이 |
| 7 | 187.6 | ng/μL | 3.751 | 1.834 | 2.05 | 2.06 | DNA (디엔제이 |
| 8 | 1580.3 | ng/μL | 31.607 | 15.281 | 2.07 | 1.98 | DNA (디엔제이 |
| 9 | 923.3 | ng/μL | 18.466 | 9.196 | 2.01 | 1.37 | DNA (디엔제이 |
| 10 | 2414.4 | ng/μL | 48.287 | 21.008 | 2.3 | 3.45 | DNA (디엔제이 |
표 2: 분광광도계에서 분석한 진균 DNA 샘플에서 소프트웨어에서 생성된 결과.
분리된 내생균류의 DNA 샘플과 달리 메타바코딩 분석을 위한 샘플의 DNA 농도는 높은 품질과 분자량으로 추출된 DNA 분자의 양이 대량으로 나타나야 합니다. 샘플은 아가로스 겔에서 전기영동으로 평가해야 하며, 겔에 번짐이 거의 또는 전혀 없이 가능한 최대 무결성을 얻어야 합니다.
토론
식물 샘플의 표면적 소독은 제시된 프로토콜에서 가장 중요한 단계 중 하나입니다. PDA 접시에 마지막 세척 물방울이 있는 오염이 없는 것은 매우 바람직합니다. 박테리아는 격리 접시에서 오염 물질로 자주 관찰되며, 일반적으로 공기 중 포자 균류보다 더 많으며, 내생균은 식물 조직 내에서도 흔하다는 점을 고려하면 3,11. 따라서 장기 절편을 설치할 때 배양 배지에 항생제를 첨가하는 것이 필수적입니다. 다양한 유형의 항생제를 결합할 때 더 나은 결과를 얻을 수 있으며, 그 결과 작용의 스펙트럼이 더 넓어집니다. 또 다른 중요한 고려 사항은 PDA와 AA를 사용하면 필연적으로 특정 종을 선택하고 다른 종을 선호하지 않기 때문에 곰팡이를 분리하는 데 내재된 편향입니다. 다른 배양 배지의 조합은 편향을 최소화할 수 있지만, 그러한 한계를 없애지는 못한다9.
일반적으로, 페트리 접시에서 자라는 동안 포자를 생성하는 균류는 더 낮은 온도에서 또는 그렇지 않은 보존 프로토콜에서 더 높은 생존율을 나타내며, 비포자성 분리물은 Castellani's 또는 광유 보존을 용납하지 않을 수 있습니다34,35,36, 따라서 이러한 경우 냉동 보존 방법이 중요할 수 있습니다. 또한, 일부 분리물은 동결에 민감할 수 있으며, 동결 보호제의 첨가는 질석18을 사용하는 제시된 방법에서와 같이 보존을 돕는다. 콜로니 및 균사 특성은 유사한 형질을 가진 분리체를 그룹화하는 데 유용할 수 있으며, 공생 발아 처리에 사용하기 위한 선택을 용이하게 합니다(Pena-Passos et al.10에 자세히 설명되어 있음). 이러한 특성은 특히 포자 형성 또는 부재를 고려하여 적용할 보존 방법을 선택하는 데에도 기여합니다.
분리체를 형태학적으로 특성화하는 것은 특히 분자 특성화와 관련된 경우 전문 문헌에서 사용할 수 있는 형태학적 설명을 개선하고 보완합니다. 수많은 분리체가 있는 어려운 과제임에도 불구하고 곰팡이를 형태학적으로 특성화하는 것은 시간이 많이 걸리고 발표된 데이터에 의존합니다. 분자 특성화만 수행할 계획이더라도 콜로니 모양(거대형태학)의 사진 등록부를 유지하고 가능할 때마다 게시하여 향후 식별 절차에 기여하는 것이 좋습니다. 파편이 설치된 접시의 사진을 유지하는 것도 중요한데, 이는 최종적으로 얻어진 분리물과 설치 접시에서 자라는 것으로 관찰된 형태형을 비교할 수 있도록 하기 위함이다. 이러한 경우, 비교된 균류는 동일한 유형의 배지에서 배양되어야 합니다. Currah et al.16 에서는 Epulorhiza (teleomorph: Tulasnella) 및 Ceratorhiza (teleomorph: Ceratobasidium) 속을 구별하기 위한 탄닌산 배지(TAM)에서의 배양 및 Rhizoctonia 복합체의 곰팡이 특성 분석을 위해 모닐로이드 세포를 자극하기 위한 CMA 배양과 같은 일반적인 난초 균근 균류를 평가하기 위한 추가 방법론을 찾을 수 있습니다.
광학 현미경으로 곰팡이 균사를 관찰하는 세부적인 방법을 고려할 때, 티즈 마운트 및 접착 테이프 마운트는 슬라이드 배양보다 빠르지만 티즈 마운트 방법은 포자 분석 및 가지 각도 측정에 적합하지 않습니다. 점착 테이프 마운트는 티즈 마운트 기법보다 균사체 조직을 더 잘 보존하지만, 점착 테이프를 사용하는 것은 분기 각도를 측정하기 위한 슬라이드 배양만큼 신뢰할 수 없습니다(저자 관찰). 슬라이드 배양은 시간이 많이 걸리지만 반영구적 및 영구 슬라이드를 제작하는 데 가장 적합한 기술입니다. 거시형태학과 미시형태학은 문헌을 고려하여 분석됩니다. Webster and Weber32 는 광범위한 일반 정보 및 추가 참고 문헌입니다. Currah et al.16 및 Zettler and Corey37 은 난초 균근 균류에 대한 유용한 정보를 제공합니다. 의학적으로 중요한 균류를 설명하지만, Walsh et al.21 및 McGinnis31 은 사상균 군집의 일반적인 특성, 시각적/설명적 정보 및 진균 미세형태학에 대한 추가 참고 문헌에도 도움이 됩니다.
Yu et al.38에 따르면, 분광광도계의 DNA 정량 분석은 정량적 Real-Time PCR 분석과 일치하는 뛰어난 정밀도와 정확도를 제공합니다. 중요한 관찰은 일부 분리물이 후속 분자 식별 단계를 위해 적절하게 순수한 DNA 샘플을 제공하지 않을 수 있다는 것인데, 이는 마이코헤테로트로프의 곰팡이 내생식물이 극도로 다양하기 때문에, 특히 열대 식물과 관련된 내생식물(39)과 생성된 대사산물이 그러하기 때문이다. 분광 광도계 분석은 이러한 사례와 다양한 프로토콜 또는 샘플의 DNA 정제에 적용되는 상용 키트를 평가하는 데 사용할 수 있습니다. 분리체의 분자 식별에서 제안된 후속 단계는 내부 전사 스페이서(ITS) 영역의 증폭 및 염기서열분석입니다. ITS 영역은 전문 문헌에 광범위하게 사용되는 리보솜 RNA 스페이서 DNA이며 공식적으로 곰팡이 식별을 위한 주요 바코드로 승인되었습니다 9,40. 프라이머 ITS1 및 ITS4(41)를 이용한 PCR에 의해 증폭된 후, 생어 플랫폼(42)에서 서열분석될 수 있다.
메타바코드 분석은 환경 및 식물 샘플에서 미생물의 풍부함, 풍부도 및 분류학적 조성을 조사할 수 있을 뿐만 아니라 이들 미생물 간의 긍정적 또는 부정적 상호작용의 결과를 제공할 수 있다12. DNeasy PowerSoil 표준 프로토콜에 추가된 액체 질소를 사용하는 침용 단계는 식물 조직과 관련하여 가능한 한 많은 진균 세포를 파괴하여 침식된 조직11에서 진균 군집의 더 나은 샘플링을 가능하게 하는 것을 목표로 합니다. 분자 식별에서 진균 염기서열(바코드)에 대한 프라이머 선택은 식물 DNA와의 간섭을 고려해야 합니다. 곰팡이 18S 유전자의 가변 영역을 증폭하는 프라이머(예: ITS1-5,8S-ITS243)를 사용하는 것이 좋습니다. DNA 샘플을 얻은 후 후속 단계는 선택한 염기서열분석 플랫폼에 따라 달라지는 메타게놈 라이브러리를 준비하고, 라이브러리의 사후 염기서열을 분석하고, 생물정보학 도구를 사용하여 획득한 데이터를 분석하는 것입니다. 우리는 플랫폼 Illumina MiSeq를 사용하는 것이 좋은데, 이는 짧은 시간 동안 250 bp 서열을 생성하는 고출력의 저렴한 옵션을 나타냅니다11,44.
공개
저자는 공개할 것이 없으며 이해 상충도 없습니다.
감사의 말
FAPESP(2015/26479-6) 및 CNPq(447453/2014-9)의 자금 지원에 감사드립니다. JLSM은 생산성 보조금에 대해 CNPq에 감사드립니다(303664/2020-7). MPP는 Capes(석사 학위 장학금, 프로세스 88887.600591/2021-00)와 CNPq에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
참고문헌
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , Springer-Verlag. Berlin. 1-13 (2006).
- Smith, S. E., Read, D. J. Mycorrhizal Symbiosis. , Elsevier Science Academic Press. London. (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777(2022).
- Araújo, W. L., et al. Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, Santarém: UFOPA. 257(2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774(2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589(2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069(2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. Larone's Medically Important Fungi: A Guide to Identification. , ASM Press. (2018).
- Malloch, D. Moulds: Isolation, Cultivation, Identification. , Available from: http://website.nbm-mnb.ca/mycologywebpages/Moulds/Moulds.html (1997).
- Smith, P. Microscopy: Chemical Reagents. British Mycological Society. , Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022).
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , Santarém: UFOPA. 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. Methuen Handbook of Colour. Methuen handbook of colour. , Methuen & Co. Ltd. London. (1967).
- Ridgway, R. Color Standards and Color Nomenclature. , Washington, DC. Published by the author (1912).
- McGinnis, M. R. Laboratory Handbook of Medical Mycology. , Elsevier Science. (2012).
- Webster, J., Weber, R. Introduction to Fungi. , Cambridge University Press. Cambridge, UK. (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유