Method Article
Isolamento, caratterizzazione ed estrazione totale del DNA per l'identificazione di funghi endofiti in piante micoeterotrofe
In questo articolo
Erratum Notice
Riepilogo
Il presente articolo si propone di fornire protocolli dettagliati e adeguati per l'isolamento di funghi endofiti associati alle piante, la conservazione a lungo termine degli isolati, la caratterizzazione morfologica e l'estrazione del DNA totale per la successiva identificazione molecolare e analisi metagenomica.
Abstract
Le piante micoeterotrofe presentano una delle forme più estreme di dipendenza micorrizica, avendo perso totalmente la loro capacità autotrofica. Essenziali come qualsiasi altra risorsa vitale, i funghi a cui queste piante si associano intimamente sono essenziali per loro. Pertanto, alcune delle tecniche più rilevanti nello studio delle specie micoeterotrofe sono quelle che consentono di studiare i funghi associati, in particolare quelli che abitano le radici e gli organi sotterranei. In questo contesto, vengono comunemente applicate tecniche per l'identificazione di funghi endofiti coltura-dipendenti e coltura-indipendenti. L'isolamento degli endofiti fungini fornisce un mezzo per identificarli morfologicamente, analizzare la loro diversità e mantenere inoculi per applicazioni nella germinazione simbiotica dei semi di orchidea. Tuttavia, è noto che esiste una grande varietà di funghi non coltivabili che abitano i tessuti vegetali. Pertanto, le tecniche di identificazione molecolare indipendenti dalla coltura offrono una copertura più ampia della diversità e dell'abbondanza delle specie. Questo articolo si propone di fornire il supporto metodologico necessario per l'avvio di due procedure di indagine: una cultura-dipendente e una indipendente. Per quanto riguarda il protocollo coltura-dipendente, vengono descritti in dettaglio i processi di raccolta e mantenimento dei campioni vegetali dai siti di raccolta alle strutture di laboratorio, insieme all'isolamento dei funghi filamentosi dagli organi sotterranei e aerei delle piante micoeterotrofe, al mantenimento di una collezione di isolati, alla caratterizzazione morfologica delle ife mediante la metodologia di coltura a vetrino e all'identificazione molecolare dei funghi mediante estrazione totale del DNA. Comprendendo metodologie indipendenti dalla coltura, le procedure dettagliate includono la raccolta di campioni di piante per le analisi metagenomiche e l'estrazione del DNA totale dagli organi vegetali aclorofilliani utilizzando un kit commerciale. Infine, per le analisi vengono suggeriti anche protocolli di continuità (ad esempio, reazione a catena della polimerasi [PCR], sequenziamento) e vengono qui presentate tecniche.
Introduzione
I funghi endofiti sono, per definizione, quelli che abitano l'interno degli organi e dei tessuti vegetali in caso di infezioni poco appariscenti (cioè senza causare danni al loro ospite)1,2. Questi funghi possono interagire in modo neutro o benefico con le piante ospiti, possono conferire resistenza agli agenti patogeni e alle condizioni ambientali sfavorevoli e possono contribuire alla sintesi di composti benefici per la pianta (ad esempio, fattori di crescita e altri fitormoni)1,3. Gli endofiti micorrizici sono funghi che stabiliscono associazioni micorriziche con la pianta, prendendo parte al trasferimento dei nutrienti4. Nelle Orchidaceae, l'interazione con gli endofiti micorrizici è fondamentale per la germinazione dei semi nella stragrande maggioranza delle specie, e per l'insediamento delle piantine in tutte le piante della famiglia5. In tali contesti, le orchidee micoeterotrofe rappresentano un caso di totale dipendenza nei confronti dei loro partner micorrizici, in quanto dipendono dal trasferimento di nutrienti minerali e composti carboniosi da parte di questi funghi durante il loro intero ciclo vitale6. Pertanto, l'isolamento e l'identificazione dei funghi associati è una base fondamentale quando si studiano le strategie di vita micoeterotrofe. Inoltre, poco si sa sul ruolo degli endofiti fungini nelle piante micoeterotrofe o anche sulla reale diversità di questi funghi 7,8.
Lo studio dei funghi endofiti può essere condotto attraverso diverse tecniche, tradizionalmente descritte come coltura-indipendenti o -dipendenti, ad esempio: (a) osservazione diretta, (b) isolamento fungino e identificazione morfologica e/o molecolare, e (c) estrazione totale del DNA dei tessuti vegetali e identificazione molecolare9. Nell'osservazione diretta (a), i funghi endofiti possono essere studiati mentre si trovano ancora all'interno di cellule e tessuti vegetali mediante microscopia ottica o elettronica9, poiché diversi protocolli di microscopia sono dettagliati da Pena-Passos et al.10. Con i metodi di isolamento (b), gli endofiti fungini possono essere caratterizzati in base alle loro colonie, ife e morfologia della struttura riproduttiva o di resistenza. Inoltre, attraverso tecniche di isolamento, è possibile condurre l'identificazione molecolare degli isolati attraverso l'estrazione del DNA, l'amplificazione delle sequenze di identificazione molecolare (codici a barre o impronte digitali) e il sequenziamento11. Quest'ultima tecnica (c) consente l'identificazione molecolare di funghi endofiti mediante estrazione di DNA all'interno dei tessuti vegetali (metabarcoding), seguita dalla preparazione e dal sequenziamento della libreria12.
Inoltre, gli isolati fungini possono essere applicati in prove di germinazione simbiotica, utilizzando semi di orchidee autotrofe o micoeterotrofe. Un esempio di tale applicazione è l'indagine condotta da Sisti et al.13, che descrive la germinazione e le fasi iniziali dello sviluppo del protocormo in Pogoniopsis schenckii, un'orchidea micoeterotrofica, in associazione con alcuni dei suoi isolati, comprendenti funghi endofiti non micorrizici. Il protocollo di germinazione simbiotica applicato è dettagliato e presentato in un video di Pena-Passos et al.10. L'isolamento dei funghi in associazione con diversi organi della pianta consente di concentrarsi su diversi obiettivi di indagine riguardanti la natura delle interazioni pianta-fungo (ad esempio, per comprendere gli aspetti ecologici o fisiologici dell'associazione, nonché le indagini sul trasferimento di nutrienti dai funghi alla pianta)9.
Le metodologie presentate nella sezione 1 si basano su una raccolta di campioni di organi sotterranei, in quanto questi organi presentano le maggiori difficoltà di raccolta, e sono di grande interesse poiché gli endofiti micorrizici li colonizzano. Tuttavia, entrambi i protocolli inclusi (passaggi 1.1 e 1.2) possono essere applicati ad altri organi micoeterotrofi della pianta (ad esempio, rizomi, steli floreali e frutti). La metodologia di raccolta descritta nella fase 1.1 è designata per l'isolamento di funghi endofiti (sezione 2) per la caratterizzazione morfologica (sezioni 4 e 5) e/o l'estrazione di DNA totale per l'identificazione dell'isolato (sezione 6). D'altra parte, la metodologia di raccolta descritta nella fase 1.2 è assegnata esclusivamente all'estrazione totale del DNA di tessuti vegetali per tecniche di metabarcoding (sezione 7). Nella sezione 3 vengono presentati quattro metodi per la conservazione e la conservazione dei funghi filamentosi, due per la conservazione a breve termine (3-6 mesi) e gli altri due adeguati per la conservazione a lungo termine (>1 anno). La caratterizzazione morfologica (sezioni 4 e 5) può essere associata all'identificazione molecolare per rafforzarla e fornire importanti informazioni sulla macro- e micromorfologia fungina. La figura 1 riassume le metodologie collettive descritte in seguito.

Figura 1: Sintesi schematica dei metodi presentati. Raccolta delle piante e isolamento, conservazione e identificazione molecolare dei funghi mediante metodologie coltura-dipendenti e indipendenti dalla coltura. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Raccolta di campioni di piante
- Raccolta di campioni per metodi dipendenti dalle impostazioni cultura
- Scava con cura gli organi sotterranei; Questi possono essere radici, steli, rizomi o organi di deposito della pianta da raccogliere. Oltre ai terreni molto compatti, raccogli questi campioni a mano.
NOTA: L'uso di strumenti come cazzuole o palette in questa fase è sconsigliato, in quanto può danneggiare le fragili strutture delle piante micoeterotrofe e può causare la contaminazione dei tessuti da parte di funghi non endofiti. - Raccogli il maggior numero possibile di organi sotterranei. Conservare i campioni in sacchetti di carta all'interno di un contenitore raffreddato (ad es. scatola di polistirolo espanso o borsa termica con ghiaccio). Se vengono raccolti anche organi aerei, trasportarli separatamente da quelli sotterranei.
- Scava con cura gli organi sotterranei; Questi possono essere radici, steli, rizomi o organi di deposito della pianta da raccogliere. Oltre ai terreni molto compatti, raccogli questi campioni a mano.
- Raccolta di campioni per metodi indipendenti dalla coltura
- Scavare con cura le radici della pianta da raccogliere secondo le stesse raccomandazioni indicate nella NOTA dal punto 1.1.1.
- Raccogli il maggior numero possibile di organi sotterranei. Conservare i campioni raccolti in criotubi all'interno di azoto liquido (opzione desiderabile) o utilizzare provette da centrifuga circondate da ghiaccio secco (opzione alternativa). Mantenere separatamente gli organi aerei, se raccolti.
2. Isolamento di funghi endofiti associati ad organi vegetali14
NOTA: Ogni materiale, soluzione e reagente utilizzato in questa sezione deve essere sterile. Quelli che non possono essere acquistati già sterilizzati devono essere sterilizzati in autoclave a 121 °C per 20 min.
- Disinfestazione superficiale degli organi vegetali
- Lavare i campioni raccolti (passaggio 1.1.2) in acqua corrente e rimuovere il più possibile il substrato e altri detriti che i campioni potrebbero avere.
- All'interno di una cappa a flusso laminare, mantenere i campioni lavati immersi in etanolo al 70% per 1 minuto (è possibile utilizzare un becher o un barattolo di vetro).
- Trasferire i campioni in un altro contenitore con ipoclorito di sodio con cloro attivo al 2% per 3 minuti.
- Mettere i campioni in un contenitore con etanolo al 70% e tenerli immersi per 1 minuto. Successivamente, lavare i campioni in sequenza in due contenitori con acqua distillata.
- Installazione di frammenti vegetali nel terreno di coltura
NOTA: Ogni passaggio descritto nel passaggio 2.2 deve essere eseguito all'interno di una cappa a flusso laminare.- Prima dell'installazione, preparare piastre di Petri (8-10 cm di diametro) con 19,5 g/L di destrosio di patate agar terreno (PDA) + 7 g/L di agar batteriologico + 3 mL/L di antibiotici (ad es. streptomicina, penicillina, tetraciclina, ampicillina).
- Mantenere le piastre di Petri con il terreno di coltura a 36 °C per 24 ore prima di utilizzarle per garantire che non vi siano contaminazioni. Scartare le stoviglie contaminate da colonie di batteri o funghi.
NOTA: L'aggiunta di antibiotici nel terreno è essenziale in questa fase, poiché gli organi sono altamente infettati da batteri che possono inibire la crescita fungina. Verificare se gli antibiotici possono essere riscaldati durante la sterilizzazione in autoclave; alcuni antibiotici devono essere aggiunti dopo aver raffreddato il terreno a circa 36 °C (prima di metterlo nei piatti). - Subito dopo la disinfestazione superficiale dei campioni e sempre all'interno di una cappa a flusso laminare, in PDA, inoculare alcune gocce dell'acqua distillata dall'ultimo contenitore utilizzato per lavare i campioni. Questo passaggio è importante per valutare l'efficacia della disinfestazione superficiale dei campioni.
- In un piatto vuoto in autoclave, posizionare i campioni e utilizzare un bisturi fiammeggiato e una pinza per sezionare i campioni a uno spessore di circa 0,2 cm.
- Sezionare longitudinalmente i campioni cilindrici in due metà, se lo si desidera, per amplificare la superficie a contatto con il fluido. Per esporre meglio i frutti e altri organi più sferici, tritare o affettare accuratamente le strutture. I semi possono anche essere importanti fonti fungine, quindi assicurati che il frutto sezionato li esponga al substrato.
NOTA: Solo gli organi sani, senza danni ai tessuti o segnali di possibili malattie o infezioni patogene, dovrebbero essere utilizzati nell'isolamento degli endofiti fungini. - Distribuire cinque frammenti degli organi sotterranei nelle piastre di Petri con PDA + antibiotico. Assicurati che i frammenti siano il più lontano possibile l'uno dall'altro e che non tocchino i bordi del piatto. Non disporre alcun frammento sul supporto. Preparare le repliche di ogni organo installato, come precauzione in caso di contaminazione.
- Sigillare le piastre di Petri con pellicola trasparente e conservarle al buio a 25-27 °C (preferibilmente in un'incubatrice) per 5 giorni.
- Calcolo della frequenza di isolamento (IF)11
- Dopo 5 giorni di incubazione del frammento d'organo, calcolare l'IF, in base al numero di frammenti incubati che presentano colonie fungine in crescita diviso per il totale dei frammenti incubati, come rappresentato nella seguente equazione:

- Dopo 5 giorni di incubazione del frammento d'organo, calcolare l'IF, in base al numero di frammenti incubati che presentano colonie fungine in crescita diviso per il totale dei frammenti incubati, come rappresentato nella seguente equazione:
- Purificazione degli isolati fungini mediante striatura e subcoltura
NOTA: Ogni passaggio descritto nel passaggio 2.4 deve essere eseguito all'interno di una cappa a flusso laminare.- Preparare le piastre di Petri (5 cm di diametro) con 5-7 g/L di agar-agar (AA; solo agar batteriologico). Mantenere le stoviglie per 24 ore a 36 °C per eliminare quelle eventualmente contaminate.
- Identifica ogni colonia fungina coltivata con un codice e delimita i suoi margini sul lato inferiore del piatto (può essere fatto con un pennarello indelebile). Differenzia le colonie in base al colore, al modello di crescita, alla consistenza e al formato dei margini.
- Con uno stuzzicadenti in legno autoclavato, utilizzando la punta fine, recuperare una piccola quantità di micelio da una colonia fungina. Concentrarsi preferibilmente sui margini della colonia e scegliere un'area il più lontana possibile da un'altra colonia, evitando di recuperare più di un tipo di colonia contemporaneamente.
- Usando lo stesso stuzzicadenti con il micelio all'estremità, striare l'AA producendo tre strie (scanalature). Assicurati che ogni stria sia a una distanza di 1 cm dall'altra e dai bordi del piatto. Scrivere l'apposito codice (utilizzando carta adesiva e matita), sigillare le stoviglie e mantenerle incubate al buio a 25-27 °C per 3 giorni.
- Preparare le piastre di Petri (8 cm di diametro) con 39 g/L di PDA. Non è necessario aggiungere antibiotici in questa fase.
- Dopo aver incubato le piastre AA, osservarle meticolosamente in controluce (da una lampada o da una finestra), con l'obiettivo di identificare le sottili ife che formano le singole colonie. Delimitare l'area di una singola colonia per capanna, utilizzando un pennarello indelebile sul lato inferiore della capsula di Petri.
- In una cappa a flusso laminare, utilizzare uno stuzzicadenti autoclavato per tagliare una porzione del terreno contenente la colonia e trasferire il volume tagliato al centro di una nuova capsula PDA.
- Identificare le piastre di Petri con i codici degli isolati, sigillare le piastre con pellicola trasparente e mantenerle al buio a 25-27 °C per 7-14 giorni.
3. Conservazione degli isolati fungini purificati
NOTA: Ogni materiale, soluzione e reagente utilizzato in questa sezione deve essere sterile. Quelli che non possono essere acquistati già sterilizzati devono essere sterilizzati in autoclave a 121 °C per 20 min.
- Conservazione con metodo Castellani o in olio minerale (3-6 mesi)11,15
- Preparare provette per microcentrifuga da 2 mL con 0,5 mL di acqua distillata o contenenti 0,5 mL di olio minerale (a seconda del metodo scelto). Assicurarsi che le provette siano vuote in autoclave e che l'acqua e l'olio sterilizzati in autoclave vengano aggiunti alle provette all'interno di una cappa a flusso laminare.
- In una cappa a flusso laminare, posizionare le piastre di Petri con isolati purificati già coltivati in PDA (39 g/L) per 7-14 giorni. Usando uno stuzzicadenti autoclavato, tagliare dei piccoli cuboidi (0,5 cm x 0,5 cm nella zona superiore) di terreno contenente i margini del micelio.
- Mettere da quattro a sei cubi nelle provette della microcentrifuga con acqua distillata (metodo Castellani) o olio minerale. Conservare le provette al buio, a 25 °C per tutto il tempo necessario, rispettando i limiti di tempo del metodo.
NOTA: Evitare di aggiungere troppi parallelepipedi ai tubi e di riempirli, il che potrebbe aumentare le possibilità di contaminazione. E' possibile mantenere le provette refrigerate, il che può favorire la conservazione di alcuni isolati più a lungo. Currah et al.16 raccomandano di conservare le repliche degli isolati di funghi micorrizici delle orchidee tropicali sia refrigerati che a 25 °C, poiché possono essere persi durante la conservazione a freddo. - Recupera un parallelepipedo e posizionalo al centro di una nuova parabola PDA per far crescere un isolato immagazzinato.
- Crioconservazione in chicchi di riso non decorticati (>1 anno)17
- Lavare i chicchi di riso non decorticati in acqua corrente e cuocerli fino a quando il guscio di riso inizia ad aprirsi. Distribuire i chicchi cotti in provette di vetro con tappo a vite e sterilizzare in autoclave due volte con un intervallo di 24 ore tra una e l'altra.
- In una cappa a flusso laminare, posizionare le piastre di Petri con isolati purificati già coltivati in PDA (39 g/L) per 7-14 giorni. Utilizzando uno stuzzicadenti autoclavato, recuperare cinque piccoli frammenti di ife dai margini del micelio e inocularli nella provetta contenente i chicchi di riso autoclavati.
NOTA: L'inoculazione di ife in diversi punti e profondità della provetta garantisce che l'isolato colonizzi i chicchi di riso non decorticati in minor tempo. - Incubare le provette al buio a 25-27 °C per 14 giorni. Agitare i tubi facendoli vorticare ogni 3 giorni per mantenere i chicchi individualizzati.
- Dopo aver osservato la crescita fungina nei chicchi di riso, distribuire i chicchi in una capsula di Petri autoclavata rivestita con carta da filtro per assorbire l'umidità, sopra la carta in modo che i chicchi possano asciugarsi. Conservare al buio a 25-27 °C per 2-3 giorni.
- Preparare i criotubi con 1/3 di gel di silice sul fondo e 1/3 di lana di vetro sopra la silice. Infine, distribuire 1/3 dei chicchi di riso con micelio fungino cresciuto. Conservare a -20 °C per 24 h.
- Dopo 24 ore, conservare i criotubi a -80 °C per tutto il tempo necessario, rispettando i limiti di tempo del metodo.
- Crioconservazione in vermiculite con crioprotettore (>1 anno)18
- Preparare un terreno di coltura liquido composto dallo 0,2% di estratto di lievito e dal 2% di glucosio in acqua distillata. Regolare il pH a 5 e sterilizzarlo in autoclave.
- Distribuire 0,2 g di vermiculite (utilizzare granulometria fine) in criotubi con tappo a vite e sterilizzarli in autoclave. Aggiungere 0,8 mL di terreno liquido autoclavato ai criotubi contenenti vermiculite.
- In una cappa a flusso laminare, posizionare le piastre di Petri con isolati purificati già coltivati in PDA (39 g/L) per 7-14 giorni. Utilizzando uno stuzzicadenti autoclavato, recuperare da tre a cinque frammenti di ife dai margini del micelio e inoculare lungo la provetta contenente vermiculite + terreno di coltura liquido. Ricordarsi di identificare i criotubi con il rispettivo codice di isolamento.
NOTA: L'inoculazione di ife in diversi punti e profondità del criotubo garantisce che l'isolato colonizzi la vermiculite in meno tempo. - Conservare i criotubi al buio a 25-27 °C fino a quando non si osserva la colonizzazione della maggior parte dei grani di vermiculite, che di solito richiede circa 14 giorni.
- Preparare una soluzione crioprotettiva composta dal 5% di glicerolo e dal 5% di trealosio in acqua distillata e sterilizzarla in autoclave. Lasciare raffreddare la soluzione prima di utilizzarla.
- Dopo che la vermiculite nei criotubi è stata colonizzata dal rispettivo isolato fungino, distribuire 0,4 mL di crioprotettore in ciascuna provetta e mantenere i criotubi refrigerati a 4 °C per 48 ore. Successivamente, mantenere i criotubi a -80 °C per tutto il tempo necessario, rispettando i limiti di tempo del metodo.
4. Caratterizzazione macromorfologica di funghi filamentosi (morfologia delle colonie)
- Mantenere un registro fotografico del micelio coltivato in ciascuna capsula di Petri con 39 g/L PDA per 7-14 giorni. Ricordati di registrare entrambi i lati della colonia, la parte superiore e quella inferiore (rovescio). Se i piatti devono essere prontamente utilizzati nella sezione 6 o non vengono mantenuti, aprire i piatti quando li si fotografa per ottenere foto migliori.
- Se interessati a dati quantitativi sulla morfologia della colonia, la subcoltura replica gli isolati e li mantiene nelle stesse condizioni di crescita e per un periodo costante per registrare il diametro della colonia e calcolare il tasso di crescita (di solito in mm/h).
NOTA: Risultati quantitativi più sofisticati possono essere ottenuti utilizzando diversi tipi di terreni di coltura per il confronto19 e considerando gli strumenti statistici per il trattamento dei dati. - Osservare le colonie anche al microscopio stereoscopico per identificarne le caratteristiche morfologiche e fotografarle ingrandindole. Valutare le colonie in base alle loro caratteristiche macromorfologiche, come dettagliato nella sezione dei risultati. Consultare diverse fonti bibliografiche per aiutare a classificare e correlare la macromorfologia all'identificazione molecolare e/o morfologica.
5. Caratterizzazione micromorfologica di funghi filamentosi (morfologia ifale)
NOTA: Le tecniche micromorfologiche sono confrontate nella sezione di discussione, considerandone i possibili usi e svantaggi.
- Coltivare gli isolati su piastre di Petri con 39 g/L PDA per 7-14 giorni. Per valutare i funghi micorrizici putativi delle orchidee, coltivare gli isolati su 17 g/L di agar di farina di mais (CMA; o un altro mezzo limitante i nutrienti) per 3-7 giorni19.
- Stuzzicare il supporto
- Lavorare all'interno di una cappa a flusso laminare. Posizionare una goccia di una macchia scelta (passaggio 5.5) su un vetrino pulito.
- Utilizzando uno stuzzicadenti autoclavato o un altro materiale sterile, rimuovere con cura alcune ife dall'isolato coltivato e posizionarle nella goccia di colorante. Posizionare un vetrino coprioggetto (preferibilmente con un angolo iniziale di 45° per evitare bolle d'aria) e analizzarlo al microscopio ottico.
- Supporto per nastro adesivo
NOTA: Questa tecnica viene solitamente applicata nelle colture fungine per essere prontamente utilizzata nella sezione 6 o non mantenuta, poiché il nastro adesivo non può essere sterilizzato in autoclave e possono verificarsi contaminazioni dopo l'esecuzione del metodo.- Posizionare una goccia di una macchia scelta (passaggio 5.5) su un vetrino pulito. Tagliare una striscia di nastro adesivo trasparente di dimensioni che si adattino bene al vetrino e alla goccia di macchia centrale.
- Dirigere la superficie adesiva della striscia sulla superficie del micelio. Non premere e cerca di raccogliere alcune ife senza attaccarne troppe.
- Incolla il nastro adesivo sul vetrino, assicurandoti che la macchia sia a contatto con le ife raccolte. Metti una goccia d'acqua sopra il nastro e posiziona un vetrino coprioggetti. Analizza il vetrino al microscopio ottico.
- Coltura di vetrini di funghi filamentosi20
- All'interno di una capsula di Petri di grandi dimensioni (>9 cm di diametro, preferibilmente), posizionare: carta da filtro nella parte inferiore, un'asta di vetro a forma di U o un adattamento (lo scopo è quello di fornire un'elevazione per il vetrino), un vetrino e due vetrini coprioggetti. Le piastre di Petri più alte facilitano la manipolazione. Autoclavare questi kit di piastre di Petri, uno per ogni isolato.
- Preparare un piccolo volume di 39 g/L di PDA o 17 g/L di CMA, aggiungere 10 g/L di agar batteriologico e autoclave (il volume di una capsula di Petri è sufficiente per più di 30 isolati). I terreni di coltura utilizzati nella coltura dei vetrini devono essere più duri dei normali terreni. Autoclave 100 mL di acqua distillata.
- Lavorare all'interno di una cappa a flusso laminare. Mettere il terreno liquido in una capsula di Petri, producendo uno strato alto circa 0,5 cm. Lascia che il mezzo si solidifichi. Una volta solido, utilizzare un bisturi sterile per tagliare quadrati del terreno di dimensioni 1 cm x 1 cm.
- Aprire il kit della capsula di Petri all'interno di una cappa a flusso laminare e utilizzare una pinza fiammata per organizzare il materiale sterilizzato in autoclave all'interno della capsa. Disporre il materiale nell'ordine numerato, come si vede nella Figura 2A, posizionando un quadrato di terreno di coltura sul vetrino. Prima di posizionare il vetrino coprioggetto sul supporto, procedere al punto 5.4.5.
- Utilizzare uno stuzzicadenti autoclavato per recuperare alcune ife da un isolato e strofinare con cura le quattro facce laterali del terreno posto sul vetrino. Posizionare un vetrino coprioggetto autoclavato sul quadrato medio utilizzando una pinza fiammata.
- Utilizzare un puntale per pipetta sterile per inserire l'acqua sterilizzata in autoclave nella carta da filtro, per creare una camera umida. Utilizzare un volume per saturare la carta senza aggiungere acqua in eccesso. Sigillare la capsula di Petri con pellicola trasparente e identificarla con il rispettivo codice isolato. Mantenere le stoviglie al buio a 25-27 °C per 3-7 giorni.
- Valutare la crescita delle ife nel vetrino e nel vetrino. Da ogni kit di coltura vengono prodotti due vetrini, uno utilizzando il vetrino con ife e il vetrino coprioggetti autoclavato, il secondo utilizzando il vetrino coprioggetti con ife e un altro vetrino.
- Dopo aver osservato la crescita ifale, staccare con cautela il quadrato di terreno dal vetrino (Figura 2B), montare i due vetrini utilizzando un colorante scelto (passaggio 5.5) e analizzare al microscopio ottico. Ricordarsi di identificare il codice di isolamento sulla diapositiva. Per produrre vetrini semipermanenti, sigillare il vetrino coprioggetti con smalto trasparente.
- Per produrre vetrini permanenti, lavare accuratamente il colorante dalle ife aderenti dopo la colorazione e lasciare asciugare il vetrino. Montare con un mezzo di montaggio rapido (per i dettagli, leggere Pena-Passos et al.10).
NOTA: Lo svantaggio di produrre un vetrino permanente con questa tecnica è che le spore e le strutture non aderenti al vetro possono essere lavate.
- Metodi di colorazione e osservazione delle ife
- Lattofenolo blu di cotone (LPCB)21: aggiungere 20 ml di acido lattico, 40 ml di glicerolo e 20 ml di acqua distillata. Sciogliere 20 g di cristalli di fenolo in questa soluzione riscaldando delicatamente. Sciogliere 0,05 g di blu di metile (blu di cotone o 2 ml di soluzione acquosa all'1%). LPCB può anche essere facilmente acquistato già preparato.
ATTENZIONE: Il fenolo è altamente tossico e volatile; manipolarlo esclusivamente all'interno di una cappa aspirante e indossare i guanti. - Blu di toluidina O10 (TBO): Preparare TBO allo 0,05% in tampone fosfato 0,1 M (pH 6,8).
- Congo rosso22,23,24: Preparare una soluzione di rosso Congo all'1% in acqua distillata e filtrarla. Incubare per 5-10 min. Questo colorante può essere applicato anche per la microscopia a fluorescenza25.
- Quando si osservano e si fotografano le ife fungine al microscopio ottico, consultare diverse fonti bibliografiche per aiutare a identificare le strutture (controlla la discussione).
- Lattofenolo blu di cotone (LPCB)21: aggiungere 20 ml di acido lattico, 40 ml di glicerolo e 20 ml di acqua distillata. Sciogliere 20 g di cristalli di fenolo in questa soluzione riscaldando delicatamente. Sciogliere 0,05 g di blu di metile (blu di cotone o 2 ml di soluzione acquosa all'1%). LPCB può anche essere facilmente acquistato già preparato.

Figura 2: Procedure per la coltura di funghi filamentosi. (A) Configurazione schematica di un kit di coltura di vetrini, in cui i numeri indicano l'ordine di disposizione degli elementi. (B) Si osserva il distacco del quadrato del terreno di coltura dopo la crescita ifale nel vetrino e nel vetrino. Fare clic qui per visualizzare una versione più grande di questa figura.
6. Estrazione totale del DNA da isolati fungini (protocollo casalingo26 con modifiche 27)
NOTA: Ogni materiale, soluzione e reagente utilizzato in questa sezione deve essere sterile. Quelli che non possono essere acquistati già sterilizzati devono essere sterilizzati in autoclave a 121 °C per 20 min. Indossare i guanti durante tutto il protocollo ed eseguire alcune fasi all'interno di una cappa aspirante.
- Preparare il tampone di estrazione: 1% di sodio dodecil solfato (SDS), 250 mM NaCl, 200 mM Tris-HCl (pH 8,0) e 25 mM di acido etilendiamminotetraacetico (EDTA). Coltivare gli isolati purificati in 39 g/L PDA per 7-14 giorni.
- Raschiare con cura il micelio da un isolato utilizzando una spatola o un cucchiaio e trasferire i frammenti in una malta autoclavata, evitando di trasferire il terreno di coltura con l'aggregato di ife. Usando un pestello di porcellana, macinare il micelio con azoto liquido fino a ottenere una polvere fine. Non lasciare che il micelio macinato si sciolga, aggiungendo azoto per evitarlo.
- Aggiungere 1 mL di tampone di estrazione in una provetta per microcentrifuga da 2 mL e posizionare il campione macinato fino al segno di 1,5 mL. Agitare delicatamente il contenuto delle provette per omogeneizzare.
- Agitare i tubi in un vortice per 5 s e metterli in un termoblocco a 65 °C per 20 min. Omogeneizzare accuratamente il contenuto per inversione ogni 7-10 min.
- Centrifugare le provette a 10.000 x g per 10 minuti a 4 °C. Trasferire 800 μL dalla fase superiore a una nuova provetta per microcentrifuga da 2 mL, aggiungere 800 μL di fenolo alle provette e miscelare il contenuto per inversione.
ATTENZIONE: Il fenolo è un reagente altamente tossico e volatile; Può causare eritema, cancrena e necrosi tissutale. Se inalato, può causare dispnea e tosse. L'assorbimento sistemico può danneggiare il fegato, i reni e il sistema nervoso centrale. Manipolare il fenolo esclusivamente all'interno di una cappa aspirante e indossare guanti.
NOTA: Dalla fase successiva, utilizzare i guanti ed eseguire il protocollo all'interno di una cappa aspirante. - Centrifugare le provette a 10.000 x g per 10 minuti a 4 °C e trasferire 800 μL dalla fase superiore a una nuova provetta da 2 mL, evitando accuratamente il trasferimento del contenuto della fase inferiore.
- Aggiungere 400 μL di fenolo e 400 μL di cloroformio alle provette e mescolare il contenuto per inversione. Centrifugarli a 10.000 x g per 10 minuti a 4 °C.
ATTENZIONE: Il cloroformio è un reagente altamente tossico e volatile; Può causare irritazioni e lesioni a contatto con la pelle e, se inalato, colpisce il sistema nervoso centrale e cardiorespiratorio, il fegato e i reni. Manipolare il cloroformio esclusivamente all'interno di una cappa aspirante e indossare guanti. - Recuperare 800 μL o meno dalla fase superiore in una nuova provetta da 2 mL. Aggiungere 800 μL di cloroformio, mescolare il contenuto per inversione e centrifugare a 10.000 x g per 10 minuti a 4 °C.
NOTA: In questa fase, nessun residuo della fase inferiore deve essere trasferito ai nuovi tubi. Pertanto, fai molta attenzione quando recuperi la fase superiore. - Trasferire 600-800 μL dalla fase superiore a una nuova provetta da 1,5 mL e aggiungere 450 μL di isopropanolo. Miscelare il contenuto per inversione e incubare a 25 °C per 5 min.
- Centrifugare le provette a 10.000 x g per 5 minuti a 4 °C ed eliminare il surnatante utilizzando una micropipetta. Fare attenzione a non gettare il pellet depositato sul fondo del tubo.
- Aggiungere 500 μL di etanolo all'80% e centrifugare per 5 min. Ripetere due volte nel caso in cui il pellet non sia chiarificato.
- Rimuovere l'etanolo con una micropipetta e asciugare il pellet a 37 °C per 30-60 minuti. Aggiungere 30-50 μL di acqua deionizzata, eluendo il pellet utilizzando una micropipetta.
- Mantenere le provette a 4 °C per una notte per una completa eluizione del DNA e congelare il contenuto a -20 °C.
7. Estrazione totale del DNA da organi vegetali per metodologia di metabarcoding (kit commerciale)
NOTA: Per la seguente metodologia, è necessario acquistare il kit commerciale indicato nella Tabella dei Materiali come kit per l'estrazione del DNA del suolo. Ogni materiale, soluzione e reagente utilizzato in questa sezione deve essere sterile. Quelli che non possono essere acquistati già sterilizzati devono essere sterilizzati in autoclave a 121 °C per 20 min. Si consiglia vivamente di indossare guanti durante l'intero protocollo e i passaggi possono essere eseguiti all'interno di una cappa a flusso laminare. Il protocollo descritto è modificato da De Souza et al.12, dal protocollo dettagliato dal produttore.
- Utilizzando pestelli e mortai in porcellana, macinare le radici raccolte seguendo il passaggio 1.2 in azoto liquido, riducendo i campioni in polvere fine. Aggiungere 0,3 g dai campioni macinati alle provette PowerBead e agitare delicatamente per omogeneizzare.
- Aggiungere 60 μL di soluzione C1 (inclusa nel kit) alle provette PowerBead e miscelare il contenuto per inversione. Utilizzando un omogeneizzatore per tessuti e un lisatore cellulare (Tabella dei materiali), legare saldamente i tubi a un supporto adeguato, accoppiare il supporto al vortice e avviare l'apparecchiatura alla massima velocità per 10-20 minuti.
NOTA: Nel caso in cui la soluzione C1 sia precipitata, riscaldarla a 60 °C fino alla completa dissoluzione. - Centrifugare le provette a 10.000 x g per 30 s a 25 °C. Trasferire 500 μL del surnatante in provette da microcentrifuga da 2 mL. Il contenuto trasferito può essere particolato.
- Aggiungere 250 μL di soluzione C2 alle provette e agitare in un vortice per 5 s. Incubare a 4 °C per 5 minuti e poi centrifugare a 10.000 x g per 1-2 minuti a 25 °C.
- Trasferire più di 600 μL del surnatante in nuove provette da 2 mL, evitando il pellet. Aggiungere 200 μL di soluzione C3 e agitare a vortice per 5 s.
- Incubare a 4 °C per 5 minuti e poi centrifugare a 10.000 x g per 1 minuto a 25 °C. In questa fase, assicurarsi che il surnatante non sia particolato.
- Trasferire più di 750 μL del surnatante in nuove provette da 2 mL, evitando il pellet. Omogeneizzare bene la soluzione C4, aggiungere 1.100 μL di soluzione C4 al surnatante e agitare in un vortice per 5 s.
- Caricare 675 μL dal contenuto delle provette nelle colonne di rotazione MB, sopra il filtro, e centrifugare a 10.000 x g per 1 minuto a 25 °C. Scartare il contenuto liquido.
- Ripetere il passaggio precedente due volte fino a quando tutto il contenuto di ogni provetta non è stato elaborato. Quindi, aggiungere 500 μL di soluzione C5 al centro del filtro presente nella colonna superiore della provetta e centrifugare a 10.000 x g per 30 s a 25 °C.
- Scartare il contenuto liquido e centrifugare nuovamente nelle stesse condizioni del passaggio precedente. Trasferire con cautela la colonna superiore della colonna MB Spin su nuove provette per microcentrifuga da 2 mL, evitando di far gocciolare il contenuto di liquido nella colonna.
- Aggiungere 85-100 μL di soluzione C6 al centro del filtro e attendere 1 minuto. Centrifugare a 10.000 x g per 30 s a 25 °C ed eliminare la colonna di centrifuga MB. Conservare le provette a -80 °C.
8. Quantificazione del DNA in spettrofotometro (vedi Tabella dei Materiali)
- Utilizzando lo spettrofotometro indicato, aprire il software. Selezionare l'opzione Acido nucleico, scegliere l'opzione DNA e impostare la concentrazione su ng/μL.
- Aggiungere 1 μL di acqua deionizzata al rivelatore dello spettrofotometro e calibrare selezionando l'opzione Blank. Dopo la lettura, pulire delicatamente il rilevatore con carta velina morbida.
- Nominare il campione da campo con il codice di isolamento del campione da leggere e inserirne 1 μL nel rivelatore dell'apparecchiatura. Selezionare l'opzione Misura; Viene generato un grafico e una tabella con i risultati.
- Ripetere il passaggio 8.3 fino a quando tutti i campioni sono stati letti. Si consiglia di salvare la tabella con i risultati generati per la registrazione e l'analisi: selezionare l'opzione Report e la posizione in cui verrà salvato il file .xml.
- Aggiungere 5 μL di acqua deionizzata al rilevatore, attendere alcuni minuti e strofinare delicatamente con carta velina morbida.
Risultati
Nel protocollo di isolamento, considerando che c'è contaminazione dall'acqua utilizzata nell'ultimo lavaggio e che la contaminazione viene rilevata anche nelle piastre di Petri con frammenti inoculati, possono essere intraprese azioni diverse, a seconda del tipo di contaminante (Tabella 1). Questa procedura deve essere ripetuta fin dall'inizio in caso di contaminanti fungini altamente sporulanti, che presentano anche una crescita accelerata, e batteri a moltiplicazione intensa, resistenti agli antibiotici scelti. Se invece il contaminante fungino presenta una crescita lenta o non sporula, è possibile semplicemente evitare di isolarlo nelle fasi successive del protocollo. Infine, le contaminazioni con ceppi batterici che si moltiplicano lentamente possono essere facilmente evitate anche nelle fasi posteriori, soprattutto nella depurazione, durante il recupero delle ife.
In tutti i casi precedentemente presentati, lo scenario ideale è quello di rifare la procedura di disinfestazione utilizzando altri campioni e installando i frammenti vegetali in un terreno di coltura che combini altri antibiotici, con uno spettro d'azione più ampio, nei casi di batteri resistenti che contaminano le prime pietanze. Tuttavia, considerando le difficoltà che comporta la raccolta di nuovi campioni e l'ottenimento di organi da piante micoeterotrofe, alcuni casi possono essere risolti con successo nelle fasi posteriori del protocollo, come presentato nella Tabella 1.
| Contaminante | Caratteristica del contaminante | Possibili conseguenze della contaminazione | Azione richiesta | |||
| Batteri | Moltiplicazione intensa | Inibizione totale o parziale della crescita degli endofiti di interesse | Riavviare l'isolamento dall'inizio | |||
| Moltiplicazione lenta | Isolamento dei batteri insieme all'endofito di interesse | Evitare colonie di batteri durante la purificazione | ||||
| Funghi | Crescita vigorosa e/o altamente sporulante | Rende impossibile isolare e/o purificare gli endofiti di interesse | Riavviare l'isolamento dall'inizio | |||
| Crescita lenta e/o non sporulante | Isolamento del contaminante per errore | Evitare l'isolamento del contaminante | ||||
Tabella 1: Descrizione dei possibili contaminanti nel processo di isolamento dei funghi endofiti, conseguenze della contaminazione e procedure di mitigazione.
A distanza di 5 giorni dall'installazione dei frammenti d'organo nel terreno PDA, è possibile visualizzare la crescita di piccoli gruppi di miceli fungi filamentosi, che emergono dall'interno dei frammenti (Figura 3). Ogni morfotipo miceliare dovrebbe risultare in un isolato fungino entro la fine del protocollo di isolamento, considerando che ognuno di essi deve essere striato in una nuova capsula di Petri con terreno AA per la purificazione. Si consiglia di non scartare le piastre originali dell'installazione del frammento prima di aver completato l'intero protocollo di isolamento. Possono essere conservati in frigorifero a 4 °C fino ad ottenere le colonie purificate. Inoltre, per quanto riguarda questa parte del protocollo, l'analisi IF può essere molto utile per valutare la percentuale di campioni di tessuto che risultano in funghi isolati rispetto al totale dei campioni installati.
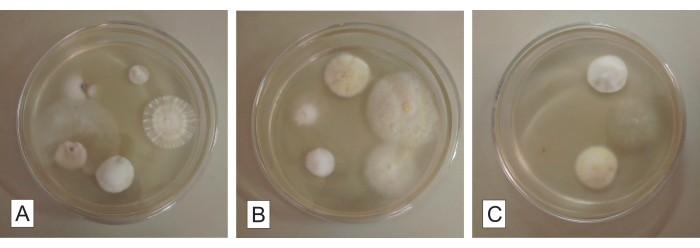
Figura 3: Frammenti di radici per l'isolamento fungino in agar destrosio di patata. Funghi endofiti coltivabili che crescono dai frammenti radicali di un'orchidea micoeterotrofia dopo circa 5 giorni di incubazione. Ogni capsula di Petri (A, B e C) contiene cinque frammenti di radici. Fare clic qui per visualizzare una versione più grande di questa figura.
Dopo 3 giorni dall'incubazione delle piastre AA per la purificazione dell'isolato, minuscole colonie ifali, quasi impercettibili, dovrebbero essere cresciute dalle strie nel terreno AA. In questa fase, nel caso in cui ci fosse una contaminazione da batteri a crescita lenta nelle piastre di isolamento, è possibile che questi batteri siano stati trasferiti anche nelle piastre AA con le ife endofite fungine selezionate. In tal caso, i batteri saranno limitati alle regioni della stria, con poca crescita rispetto al fungo, consentendo il recupero del solo fungo di interesse per i nuovi piatti PDA.
Durante 7-14 giorni dall'inoculazione degli isolati fungini purificati nelle piastre PDA, la colonia pura deve crescere centrifugamente (dal centro della capsula alla sua periferia), formando un unico micelio circolare (Figura 4). In questa fase sono facilmente identificabili possibili contaminazioni, in quanto compromettono considerevolmente l'omogeneità della colonia per quanto riguarda la crescita, la forma, l'aspetto, il colore, la produzione di pigmenti nel substrato, ecc. (Figura 5). Supponendo che le colonie finali ottenute non siano pure, i funghi inizialmente cresciuti dai frammenti d'organo devono essere sottoposti nuovamente a procedure di purificazione, mediante striatura e subcoltura (fase 2.4). È possibile recuperare gli isolati sia dagli organi installati inizialmente che dalle piastre di isolamento finali contenenti colture eterogenee.
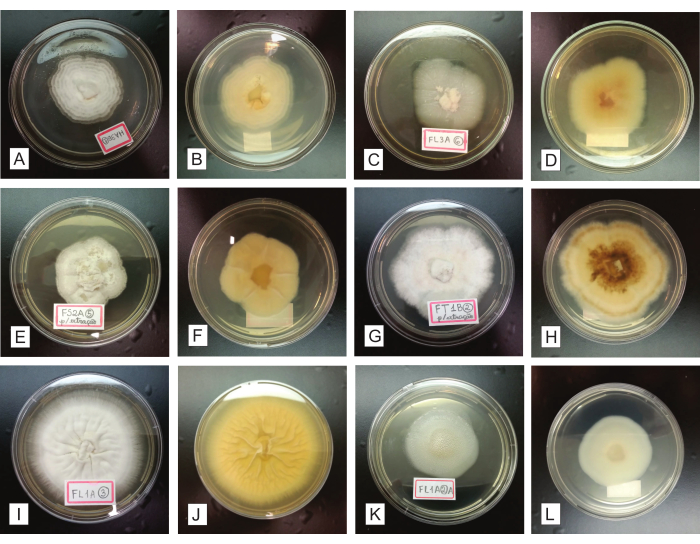
Figura 4: Rappresentazione di isolati fungini purificati, coltivati per 7-14 giorni in un terreno di coltura PDA. Nella prima e nella terza colonna (A, C, E, G, I e K), le colonie registrate sono viste dal lato superiore; la seconda e la quarta colonna (B, D, F, H, J e L) presentano le stesse colonie, viste rispettivamente dalla parte inferiore. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Rappresentazione di isolati fungini impuri, coltivati per 7-14 giorni in terreno di coltura PDA. Nella prima colonna (A, D e G) si vede una veduta generale delle colonie dal lato superiore; la seconda colonna (B, E e F) mostra le colonie in dettaglio; la terza colonna (C, F e I) mostra le colonie dalla parte inferiore. I numeri rappresentano i diversi morfotipi fungini presenti in ogni piatto e le linee rappresentano una sottile delimitazione tra i diversi isolati fungini. Fare clic qui per visualizzare una versione più grande di questa figura.
La conservazione degli isolati non deve essere effettuata utilizzando un unico metodo di conservazione, poiché ogni fungo può presentare una maggiore o minore sensibilità a ciascun metodo descritto. Si consiglia vivamente di scegliere almeno due tipi di conservazione per ogni isolato fungino, garantendo maggiori possibilità di successo nella conservazione. Evidenziamo che l'invitalità dei funghi conservati in Castellani o dell'olio minerale è prevista dopo 6 mesi o meno. I ricercatori dovrebbero considerare se questi sono gli unici metodi di conservazione scelti, per evitare possibili perdite impreviste di isolati. Per prolungare questo periodo limitato, è possibile riattivare gli isolati fungini coltivandoli nel terreno PDA (39 g/L) per 2-3 settimane e poi conservandoli nuovamente. Dopo un periodo di conservazione, ci si aspetta che gli isolati presentino una crescita più lenta, rispetto al tasso di crescita osservato prima della sua conservazione, e colonie con aspetti meno vigorosi (cioè meno densi, di colore diverso). È sufficiente subcoltivare alcune volte in un terreno di coltura ricco di sostanze nutritive per ristabilire tali caratteristiche.
Quando si valuta la macromorfologia delle colonie ottenute nella procedura di isolamento, è necessario raccogliere dati qualitativi, considerando il maggior numero possibile di caratteristiche: (a) colore della colonia (parte superiore e inferiore), che possono essere analizzati utilizzando guide di colore stampate (ad esempio, Rayner28, Kornerup & Wanscher29, Ridgway30); (b) opacità della colonia: trasparente, opaca, traslucida; (c) pigmenti diffusibili nel mezzo31 (presenza/assenza e colore); d) essudati31 (presenza/assenza, colore, aspetto generale); (e) strutture macroscopiche31 (presenza/assenza, tipo e aspetto; es., sclerozi, picnidi); f) miceli 19,32,21 aerei e sommersi (presenza/assenza, aspetto-scarso o abbondante); (g) aspetto del margine19 (colore, forma, uniformità - sommerso o aereo); e (h) topografia della colonia (ammucchiata, rugosa, crateriforme, piatta, ecc.), l'aspetto generale e la consistenza - cotonosa, vellutata, polverosa, lanosa, sebacea (cerosa), glabra, gessosa, viscida, coriacea, spinosa, ecc.19,21.
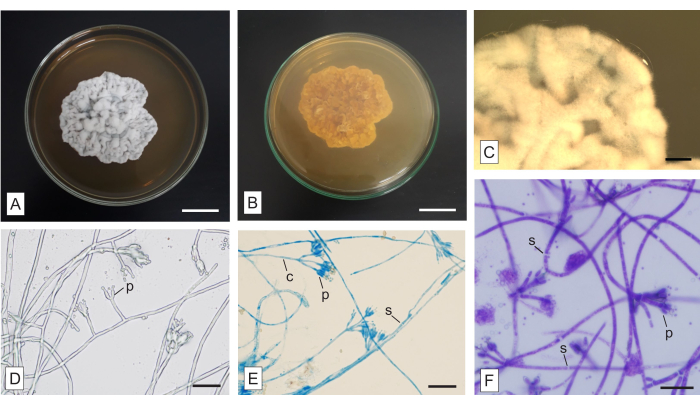
Figura 6: Macro- e micromorfologia di un fungo non identificato isolato dall'orchidea micoeterotrofica Wullschlaegelia aphylla. (A) Aspetti superiori e (B) inferiori della colonia, (C) visione amplificata delle ife aeree (come si vede in uno stereomicroscopio). Micromorfologia delle ife (D) senza colorazione e colorate con (E) LPCB e (F) TBO. Abbreviazioni: c = conidioforo, p = fialide, s = setto. Barre della scala: A,B = 2 cm; C = 2 mm; D-F = 20 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Nella Figura 6 è mostrata una colonia isolata dalle radici fusiformi dell'orchidea micoeterotrofica Wullschlaegelia aphylla, applicando la metodologia descritta. La colonia è da biancastra a grigia nella parte superiore (Figura 6A) e brunastra nella faccia inferiore (Figura 6B). È opaco, senza pigmenti diffusibili nel mezzo né essudati. I miceli sono aerei e abbondanti, i margini sono irregolari e aerei, e la colonia ha una tessitura vellutata e una topografia rugosa (Figura 6A). Le strutture macroscopiche sono assenti.
Il metodo di coltura dei vetrini è tradizionale e vantaggioso e, sebbene richieda molto tempo, può essere applicato per produrre vetrini permanenti che possono essere esaminati in seguito. I diversi coloranti qui presentati possono essere utilizzati per evidenziare strutture importanti e richiedono test nei campioni, per determinare il tempo di incubazione più adeguato. Il rosso Congo e il TBO sono colorazioni generiche. Il rosso Congo è adeguato a dimostrare alcune strutture delicate (approfondimenti: Malloch22). Il TBO colora il contenuto cellulare più intensamente della parete cellulare fungina (Figura 6F). Pur essendo un colorante usuale per le strutture vegetali e non così comune nella microscopia fungina, il TBO ha un'importante proprietà metacromatica33, che favorisce altre applicazioni, come l'analisi fungina nei tessuti vegetali10. L'LPCB è un colorante ampiamente applicato nell'analisi fungina, anche se richiede cautela a causa del fenolo. Il blu di cotone (sinonimo: blu metilico) è la macchia, l'acido lattico è un agente di pulizia e il fenolo è un agente di uccisione21. LPCB ha un'affinità per la chitina ed evidenzia pareti di spore e ornamenti23,24. I setti fungini non sono colorati né da LPCB (Figura 6E) né da TBO (Figura 6F), facilitando l'identificazione di tali strutture. L'applicazione di coloranti è vantaggiosa per evidenziare strutture che non sono facilmente visibili quando le ife non sono colorate (Figura 6D).
Per l'estrazione del DNA da funghi e campioni di radici, deve essere rispettata la quantità di campione macinato in azoto liquido da aggiungere al tampone di estrazione, non superiore alla quantità indicata nel protocollo. Va sottolineato che una grande quantità di campione processato non rappresenta semplicemente l'ottenimento di un'alta concentrazione di DNA senza grossi inconvenienti, in quanto può compromettere considerevolmente la qualità finale del DNA. Ciò si verifica quando si saturano le seguenti fasi di purificazione del DNA. Inoltre, la concentrazione di composti putativi prodotti da piante o funghi (ad esempio, pigmenti, metaboliti secondari), presenti nella soluzione finale con il DNA, può agire per ridurre la qualità del DNA e/o inibire la reazione a catena della polimerasi (PCR). Un altro problema che può ridurre significativamente la qualità del DNA è la rottamazione del terreno di coltura con micelio fungino e la macinazione, che deve essere fortemente evitata. Alcuni reagenti della procedura di estrazione (ad es. fenolo, alcol etilico, SDS) e altre sostanze (ad es. pigmenti fungini) possono essere inibitori che interferiscono con la PCR.
Nelle fasi di pulizia del DNA, quando si utilizzano fenolo e cloroformio, il surnatante deve sempre rappresentare la fase con meno impurità, comprendendo normalmente la fase più chiara. Durante tale fase, la fase inferiore non deve essere recuperata quando si aspira il surnatante che verrà trasferito in nuove provette. Entro la fine del protocollo, il pellet (costituito da DNA) dovrebbe presentare un colore da traslucido (altamente desiderabile) a biancastro. Nella fase di essiccazione, procedere con l'essiccazione del DNA in un termoblocco è fondamentale per la completa evaporazione dell'etanolo. Dopo 12 ore a 4 °C, il pellet dovrebbe essere completamente eluito nella soluzione acquosa aggiunta ai tubi, non essendo più visibile. Nel caso in cui l'eluizione non sia completa e la concentrazione di DNA sia molto al di sotto della concentrazione ideale, è possibile mantenere la soluzione refrigerata per altre 12 ore, senza perdite significative nella sua qualità.
Le analisi del DNA nel software generano una tabella, come rappresentato nella Tabella 2, in cui sono indicati i dati relativi alla quantità e alla qualità del DNA. La quantificazione del DNA è data dalla concentrazione di acidi nucleici in nanogrammi per microlitro di campione. Considerando l'identificazione molecolare di funghi isolati, i campioni di DNA dovrebbero avere una concentrazione di circa 200 ng/μL, ideale per le fasi di PCR. In caso di campioni con concentrazioni più elevate di acidi nucleici, è necessario diluire un'aliquota del campione, approssimando la concentrazione al valore precedentemente menzionato. I campioni con concentrazioni eccessivamente elevate, come i campioni 1 e 2 nella Tabella 2, possono suggerire un falso rilevamento a causa di contaminanti nella soluzione. Nel frattempo, i campioni con concentrazioni di acido nucleico più basse, come il numero 7 (Tabella 2), possono ancora essere utilizzati per la PCR, essendo importante applicare un volume maggiore di campione nelle reazioni. Per quanto riguarda i parametri di qualità, è soddisfacente che i quozienti 260/280 e 260/230 siano compresi tra 1,8 e 2,2, come indicato nei campioni 3, 5 e 8 (tabella 2). I campioni che presentano tali valori (molto al di sotto o al di sopra) devono essere sottoposti a nuove estrazioni di DNA, come indicato nei campioni 9 e 10 della tabella 2. Nel caso di campioni con una quantità adeguata di DNA e che supera minimamente l'intervallo di qualità ideale, come suggerito dai valori 260/280 e 260/230 nei numeri 4 e 6 (Tabella 2), la diluizione è la procedura più vantaggiosa per rendere la concentrazione di acido nucleico adeguata per la PCR e diluire eventuali inibitori, come i reagenti vestigiali della procedura di estrazione. nei campioni.
| Campione | Acido Nucleico Conc. | Unità | A260 | A280 | 260/280 | 260/230 | Tipo di campione |
| 1 | 14491.7 | ng/μL | 289.833 | 141.175 | 2.05 | 1.8 | DNA |
| 2 | 13359.3 | ng/μL | 267.187 | 124.607 | 2.14 | 2.15 | DNA |
| 3 | 1137.6 | ng/μL | 22.751 | 10.574 | 2.15 | 2.13 | DNA |
| 4 | 1472.6 | ng/μL | 29.452 | 13.287 | 2.22 | 2.16 | DNA |
| 5 | 3464.8 | ng/μL | 69.295 | 33.329 | 2.08 | 1.88 | DNA |
| 6 | 1884.2 | ng/μL | 37.684 | 17.912 | 2.1 | 1.78 | DNA |
| 7 | 187.6 | ng/μL | 3.751 | 1.834 | 2.05 | 2.06 | DNA |
| 8 | 1580.3 | ng/μL | 31.607 | 15.281 | 2.07 | 1.98 | DNA |
| 9 | 923.3 | ng/μL | 18.466 | 9.196 | 2.01 | 1.37 | DNA |
| 10 | 2414.4 | ng/μL | 48.287 | 21.008 | 2.3 | 3.45 | DNA |
Tabella 2: Risultati generati nel software da campioni di DNA fungino analizzati in uno spettrofotometro.
A differenza dei campioni di DNA provenienti da funghi endofiti isolati, la concentrazione di DNA nei campioni per le analisi di metabarcoding dovrebbe presentare enormi quantità di molecole di DNA estratto, con elevata qualità e peso molecolare. I campioni devono essere valutati mediante elettroforesi in gel di agarosio, ottenendo la massima integrità possibile, con poca o nessuna sbavatura nel gel.
Discussione
La disinfestazione superficiale dei campioni vegetali è una delle fasi più critiche del protocollo presentato. È altamente auspicabile che non si contaminino le stoviglie PDA con gocce dell'ultimo lavaggio. I batteri sono spesso osservati come contaminanti nelle piastre di isolamento, di solito più dei funghi sporulanti presenti nell'aria, considerando che i batteri endofiti sono comuni anche all'interno dei tessuti vegetali 3,11. Pertanto, l'aggiunta di antibiotici nel terreno di coltura durante l'installazione dei frammenti d'organo è essenziale. Si ottengono risultati migliori quando si combinano diversi tipi di antibiotici, con conseguente spettro d'azione più ampio. Un'altra considerazione importante è la distorsione intrinseca nell'isolare i funghi, poiché l'uso di PDA e AA selezionerà inevitabilmente alcune specie e ne sfavorirà altre. La combinazione di altri terreni di coltura può ridurre al minimo le distorsioni, anche se non eliminare tali limitazioni9.
Generalmente, i funghi che producono spore durante la crescita in piastre di Petri presentano tassi di sopravvivenza più elevati nei protocolli di conservazione, a temperature più basse o meno, e gli isolati non sporulanti potrebbero non tollerare la conservazione di Castellani o dell'olio minerale34,35,36, quindi un metodo di crioconservazione può essere cruciale in questi casi. Inoltre, alcuni isolati possono essere sensibili al congelamento e l'aggiunta di crioprotettori aiuta la conservazione, come nel metodo presentato utilizzando la vermiculite18. Le caratteristiche della colonia e dell'ifale possono essere utili per raggruppare gli isolati con caratteristiche simili, facilitando la selezione per l'uso in trattamenti di germinazione simbiotica (come dettagliato da Pena-Passos et al.10). Queste caratteristiche concorrono anche alla scelta dei metodi di conservazione da applicare, soprattutto considerando la sporulazione o la sua assenza.
La caratterizzazione morfologica degli isolati migliora e integra le descrizioni morfologiche disponibili nella letteratura specializzata, soprattutto se associata alla caratterizzazione molecolare. Nonostante sia una sfida con numerosi isolati, la caratterizzazione morfologica dei funghi richiede molto tempo e dipende dai dati pubblicati. Anche se si prevede di condurre solo la caratterizzazione molecolare, è consigliabile mantenere un registro fotografico dell'aspetto della colonia (macromorfologia) e pubblicarlo quando possibile, per contribuire alle future procedure di identificazione. È importante anche la conservazione delle foto delle parabole in cui sono installati i frammenti, per consentire il confronto tra gli isolati ottenuti finali e i morfotipi osservati crescere nelle piastre di installazione. In tal caso, i funghi confrontati devono essere coltivati nello stesso tipo di terreno. Ulteriori metodologie sono state trovate in Currah et al.16 per valutare i funghi micorrizici comuni delle orchidee, come la coltura su terreno acido tannico (TAM) per differenziare i generi Epulorhiza (teleomorfo: Tulasnella) e Ceratorhiza (teleomorfo: Ceratobasidium) e la coltura su CMA per stimolare cellule monilioidi per la caratterizzazione di funghi dal complesso Rhizoctonia .
Considerando i metodi dettagliati per osservare le ife fungine al microscopio ottico, il montaggio a strappo e il montaggio a nastro adesivo sono più veloci della coltura a vetrino, sebbene il metodo di montaggio a strappo non sia adeguato per analizzare le spore e misurare gli angoli dei rami. Il montaggio su nastro adesivo preserva l'organizzazione del micelio meglio della tecnica di montaggio a strappo, anche se l'uso del nastro adesivo non è affidabile come la coltura a vetrino per misurare gli angoli dei rami (osservazione degli autori). La coltura dei vetrini, nonostante richieda molto tempo, è la tecnica più adeguata per produrre vetrini semipermanenti e permanenti. La macro e la micromorfologia vengono analizzate considerando la letteratura. Webster e Weber32 è un'ampia fonte generale di informazioni e riferimenti aggiuntivi. Currah et al.16 e Zettler e Corey37 forniscono informazioni utili sui funghi micorrizici delle orchidee. Sebbene descrivano funghi di importanza medica, Walsh et al.21 e McGinnis31 sono utili anche sulle caratteristiche generali delle colonie di funghi filamentosi, informazioni visive/descrittive e riferimenti aggiuntivi per la micromorfologia fungina.
Secondo Yu et al.38, le analisi di quantificazione del DNA in uno spettrofotometro presentano una grande precisione e accuratezza, essendo coerenti con le analisi quantitative di PCR in tempo reale. Un'osservazione importante è che alcuni isolati potrebbero non fornire campioni di DNA sufficientemente puri per le successive fasi di identificazione molecolare, poiché gli endofiti fungini dei micoeterotrofi sono estremamente diversi, in particolare quelli associati alle piante tropicali39, così come i metaboliti prodotti. Le analisi spettrofotometriche possono essere utilizzate per valutare tali casi e diversi protocolli, o kit commerciali applicati per la purificazione del DNA dei campioni. Le fasi successive suggerite nell'identificazione molecolare degli isolati sono l'amplificazione della regione dello spaziatore trascritto interno (ITS) e il sequenziamento. La regione ITS è un DNA spaziatore di RNA ribosomiale ampiamente impiegato nella letteratura specializzata e formalmente accettato come codice a barre principale per l'identificazione fungina 9,40. Può essere amplificato mediante PCR con i primer ITS1 e ITS441, quindi sequenziato nella piattaforma Sanger42.
Le analisi di metabarcoding, oltre a consentire di studiare la ricchezza, l'abbondanza e la composizione tassonomica dei microrganismi in campioni ambientali e vegetali, possono fornire risultati di interazioni positive o negative tra questi microrganismi12. La fase di macerazione con azoto liquido aggiunto al protocollo standard DNeasy PowerSoil ha lo scopo di rompere il maggior numero possibile di cellule fungine in relazione ai tessuti vegetali, consentendo un migliore campionamento della comunità fungina nel tessuto macerato11. La scelta dei primer per le sequenze fungine (codici a barre) nell'identificazione molecolare deve considerare l'interferenza con il DNA delle piante. Si consiglia l'uso di primer che amplificano le regioni variabili del gene 18S del fungo, ad esempio ITS1-5,8S-ITS243. Dopo aver ottenuto i campioni di DNA, le fasi successive sono la preparazione delle librerie metagenomiche, che dipendono dalla piattaforma di sequenziamento scelta, il sequenziamento a posteriori delle librerie e l'analisi dei dati ottenuti utilizzando strumenti bioinformatici. Si consiglia di utilizzare la piattaforma Illumina MiSeq, che rappresenta un'opzione meno costosa con un'elevata potenza, generando sequenze da 250 bp in brevi periodi di tempo11,44.
Divulgazioni
Gli autori non hanno nulla da rivelare e nessun conflitto di interessi.
Riconoscimenti
Si ringraziano i finanziamenti di FAPESP (2015/26479-6) e CNPq (447453/2014-9). JLSM ringrazia CNPq per le sovvenzioni alla produttività (303664/2020-7). MPP ringrazia Capes (borsa di studio per laurea magistrale, processo 88887.600591/2021-00) e CNPq.
Materiali
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Riferimenti
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , Springer-Verlag. Berlin. 1-13 (2006).
- Smith, S. E., Read, D. J. Mycorrhizal Symbiosis. , Elsevier Science Academic Press. London. (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777(2022).
- Araújo, W. L., et al. Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, Santarém: UFOPA. 257(2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774(2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589(2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069(2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. Larone's Medically Important Fungi: A Guide to Identification. , ASM Press. (2018).
- Malloch, D. Moulds: Isolation, Cultivation, Identification. , Available from: http://website.nbm-mnb.ca/mycologywebpages/Moulds/Moulds.html (1997).
- Smith, P. Microscopy: Chemical Reagents. British Mycological Society. , Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022).
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , Santarém: UFOPA. 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. Methuen Handbook of Colour. Methuen handbook of colour. , Methuen & Co. Ltd. London. (1967).
- Ridgway, R. Color Standards and Color Nomenclature. , Washington, DC. Published by the author (1912).
- McGinnis, M. R. Laboratory Handbook of Medical Mycology. , Elsevier Science. (2012).
- Webster, J., Weber, R. Introduction to Fungi. , Cambridge University Press. Cambridge, UK. (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon