Method Article
Isolement, caractérisation et extraction totale de l’ADN pour identifier les champignons endophytes chez les plantes mycohétérotrophes
Dans cet article
Erratum Notice
Résumé
Le présent article vise à fournir des protocoles détaillés et adéquats pour l’isolement des champignons endophytes associés aux plantes, la conservation à long terme des isolats, la caractérisation morphologique et l’extraction totale de l’ADN pour l’identification moléculaire ultérieure et les analyses métagénomiques.
Résumé
Les plantes mycohétérotrophes présentent l’une des formes les plus extrêmes de dépendance mycorhizienne, ayant totalement perdu leur capacité autotrophe. Aussi essentiels que toute autre ressource vitale, les champignons auxquels ces plantes s’associent intimement leur sont essentiels. Par conséquent, certaines des techniques les plus pertinentes dans l’étude des espèces mycohétérotrophes sont celles qui permettent d’étudier les champignons associés, en particulier ceux qui habitent les racines et les organes souterrains. Dans ce contexte, des techniques d’identification des champignons endophytes dépendants et indépendants de la culture sont couramment appliquées. L’isolement des endophytes fongiques permet de les identifier morphologiquement, d’analyser leur diversité et de maintenir les inocules pour des applications dans la germination symbiotique des graines d’orchidées. Cependant, on sait qu’il existe une grande variété de champignons non cultivables qui habitent les tissus végétaux. Ainsi, les techniques d’identification moléculaire indépendantes de la culture offrent une couverture plus large de la diversité et de l’abondance des espèces. L’objectif de cet article est d’apporter le soutien méthodologique nécessaire à la mise en place de deux procédures d’investigation : l’une dépendante de la culture et l’autre indépendante. En ce qui concerne le protocole dépendant de la culture, les processus de collecte et de conservation des échantillons de plantes des sites de collecte aux installations de laboratoire sont détaillés, ainsi que l’isolement des champignons filamenteux des organes souterrains et aériens des plantes mycohétérotrophes, la conservation d’une collection d’isolats, la caractérisation morphologique des hyphes par la méthodologie de culture sur lame et l’identification moléculaire des champignons par extraction totale de l’ADN. Englobant des méthodologies indépendantes de la culture, les procédures détaillées comprennent la collecte d’échantillons de plantes pour des analyses métagénomiques et l’extraction totale de l’ADN à partir d’organes végétaux achlorophylliens à l’aide d’un kit commercial. Enfin, des protocoles de continuité (p. ex., réaction en chaîne par polymérase [PCR], séquençage) sont également suggérés pour les analyses, et les techniques sont présentées ici.
Introduction
Les champignons endophytes sont, par définition, ceux qui habitent l’intérieur des organes et des tissus végétaux lors d’infections discrètes (c’est-à-dire sans causer de dommages à leur hôte)1,2. Ces champignons peuvent interagir de manière neutre ou bénéfique avec les plantes hôtes, peuvent conférer une résistance aux agents pathogènes et aux conditions environnementales défavorables, et peuvent contribuer à la synthèse de composés bénéfiques pour la plante (par exemple, les facteurs de croissance et d’autres phytohormones)1,3. Les endophytes mycorhiziens sont des champignons qui établissent des associations mycorhiziennes avec la plante, participant au transfert des nutriments4. Chez les Orchidaceae, l’interaction avec les endophytes mycorhiziens est fondamentale pour la germination des graines chez la grande majorité des espèces, et l’établissement des plantules chez toutes les plantes de la famille5. Dans de tels contextes, les orchidées mycohétérotrophes représentent un cas de dépendance totale vis-à-vis de leurs partenaires mycorhiziens, car elles dépendent du transfert de nutriments minéraux et de composés carbonés par ces champignons pendant tout leur cycle de vie6. Par conséquent, l’isolement et l’identification des champignons associés est une base fondamentale lors de l’étude des stratégies de vie mycohétérotrophes. De plus, on sait peu de choses sur les rôles des endophytes fongiques chez les plantes mycohétérotrophes ou même sur la diversité réelle de ces champignons 7,8.
L’étude des champignons endophytes peut être menée à l’aide de différentes techniques, traditionnellement décrites comme indépendantes ou dépendantes de la culture, par exemple : (a) l’observation directe, (b) l’isolement fongique et l’identification morphologique et/ou moléculaire, et (c) l’extraction totale de l’ADN des tissus végétaux et l’identification moléculaire9. Dans l’observation directe (a), les champignons endophytes peuvent être étudiés alors qu’ils sont encore à l’intérieur des cellules et des tissus végétaux par microscopie optique ou électronique9, car différents protocoles de microscopie sont détaillés par Pena-Passos et al.10. Par des méthodes d’isolement (b), les endophytes fongiques peuvent être caractérisés en fonction de leurs colonies, de leurs hyphes et de la morphologie de leur structure de reproduction ou de résistance. De plus, grâce à des techniques d’isolement, il est possible d’effectuer l’identification moléculaire des isolats par extraction d’ADN, amplification de séquences d’identification moléculaire (codes-barres ou empreintes digitales) et séquençage11. Cette dernière technique (c) permet l’identification moléculaire de champignons endophytes par extraction d’ADN à l’intérieur des tissus végétaux (métabarcoding), suivie d’une préparation et d’un séquençage de la bibliothèque12.
De plus, des isolats fongiques peuvent être appliqués dans des essais de germination symbiotique, en utilisant des graines d’orchidées autotrophes ou mycohétérotrophes. Un exemple d’une telle application est l’étude menée par Sisti et al.13, décrivant la germination et les premiers stades de développement du protocorme chez Pogoniopsis schenckii, une orchidée mycohétérotrophe, en association avec certains de ses isolats, comprenant des champignons endophytes non mycorhiziens. Le protocole de germination symbiotique appliqué est détaillé et présenté dans une vidéo par Pena-Passos et al.10. L’isolement des champignons en association avec différents organes végétaux permet d’orienter diverses recherches sur la nature des interactions plantes-champignons (par exemple, pour comprendre les aspects écologiques ou physiologiques de l’association, ainsi que des enquêtes sur le transfert des nutriments des champignons à la plante)9.
Les méthodologies présentées dans la section 1 sont basées sur une collecte d’échantillons d’organes souterrains, car ces organes présentent le plus de difficultés à collecter, et ils présentent un intérêt majeur puisque les endophytes mycorhiziens les colonisent. Cependant, les deux protocoles inclus (étapes 1.1 et 1.2) peuvent être appliqués à d’autres organes végétaux mycohétérotrophes (p. ex., rhizomes, tiges florales et fruits). La méthode de collecte décrite à l’étape 1.1 est destinée à l’isolement des champignons endophytes (section 2) pour la caractérisation morphologique (sections 4 et 5) et/ou à l’extraction totale de l’ADN pour l’identification des isolats (section 6). D’autre part, la méthodologie de collecte décrite à l’étape 1.2 est exclusivement attribuée à l’extraction totale de l’ADN des tissus végétaux pour les techniques de métabarcoding (section 7). Dans la section 3, quatre méthodes de stockage et de conservation des champignons filamenteux sont présentées, deux pour le stockage à court terme (3 à 6 mois) et les deux autres adéquates pour le stockage à long terme (>1 an). La caractérisation morphologique (sections 4 et 5) peut être associée à l’identification moléculaire pour la renforcer et fournir des informations importantes sur la macro- et micromorphologie fongique. La figure 1 résume les méthodologies collectives décrites ci-dessous.

Figure 1 : Résumé schématique des méthodes présentées. Collecte de plantes et isolement fongique, préservation et identification moléculaire par des méthodologies dépendantes et indépendantes de la culture. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Prélèvement d’échantillons de plantes
- Prélèvement d’échantillons pour les méthodes dépendantes de la culture
- Creusez soigneusement les organes souterrains ; Il peut s’agir de racines, de tiges, de rhizomes ou d’organes de stockage de la plante à collecter. En dehors des sols très compacts, collectez ces échantillons à la main.
REMARQUE : L’utilisation d’outils tels que des truelles ou des pelles dans cette étape est déconseillée, car elle peut endommager les structures fragiles des plantes mycohétérotrophes et peut provoquer une contamination des tissus par des champignons non endophytes. - Collectez autant d’organes souterrains que possible. Conservez les échantillons dans des sacs en papier à l’intérieur d’un contenant réfrigéré (p. ex., une boîte en mousse de polystyrène ou un sac isotherme avec de la glace). Si des organes aériens sont également prélevés, transportez-les séparément des organes souterrains.
- Creusez soigneusement les organes souterrains ; Il peut s’agir de racines, de tiges, de rhizomes ou d’organes de stockage de la plante à collecter. En dehors des sols très compacts, collectez ces échantillons à la main.
- Prélèvement d’échantillons pour des méthodes indépendantes de la culture
- Creusez soigneusement les racines de la plante à collecter selon les mêmes recommandations que celles indiquées dans la NOTE de l’étape 1.1.1.
- Collectez autant d’organes souterrains que possible. Conservez les échantillons prélevés dans des cryotubes à l’intérieur d’azote liquide (option souhaitable) ou utilisez des tubes à centrifuger entourés de glace carbonique (option alternative). Conservez les organes aériens séparément, s’ils sont prélevés.
2. Isolement des champignons endophytes associés aux organes végétaux14
REMARQUE : Tous les matériaux, solutions et réactifs utilisés dans cette section doivent être stériles. Ceux qui ne peuvent pas être achetés déjà stérilisés doivent être autoclavés à 121 °C pendant 20 min.
- Désinfestation superficielle des organes végétaux
- Lavez les échantillons prélevés (étape 1.1.2) à l’eau courante et enlevez autant de substrat et d’autres débris que possible dans les échantillons.
- À l’intérieur d’une hotte à flux laminaire, maintenez les échantillons lavés immergés dans de l’éthanol à 70 % pendant 1 min (un bécher ou un bocal en verre peut être utilisé).
- Transférez les échantillons dans un autre récipient contenant de l’hypochlorite de sodium avec 2 % de chlore actif pendant 3 min.
- Changer les échantillons dans un récipient contenant 70% d’éthanol et les maintenir immergés pendant 1 min. Ensuite, lavez les échantillons séquentiellement dans deux récipients avec de l’eau distillée.
- Installation de fragments végétaux dans un milieu de culture
REMARQUE : Chaque étape détaillée à l’étape 2.2 doit être effectuée à l’intérieur d’une hotte à flux laminaire.- Avant l’installation, préparer des boîtes de Pétri (de 8 à 10 cm de diamètre) avec 19,5 g/L de milieu de culture de gélose au dextrose de pomme de terre (PDA) + 7 g/L de gélose bactériologique + 3 mL/L d’antibiotiques (p. ex., streptomycine, pénicilline, tétracycline, ampicilline).
- Maintenir les boîtes de Pétri avec le milieu de culture à 36 °C pendant 24 h avant de les utiliser pour s’assurer qu’il n’y a pas de contamination. Jetez les plats contaminés par des colonies de bactéries ou de champignons.
REMARQUE : L’ajout d’antibiotiques dans le milieu est essentiel dans cette étape, car les organes sont fortement infectés par des bactéries qui peuvent inhiber la croissance fongique. Vérifier si les antibiotiques peuvent être chauffés pendant l’autoclavage ; certains antibiotiques doivent être ajoutés après avoir refroidi le milieu à environ 36 °C (avant de le placer dans la vaisselle). - Immédiatement après la désinfestation superficielle des échantillons et toujours à l’intérieur d’une hotte à flux laminaire, en PDA, inoculer quelques gouttes de l’eau distillée du dernier récipient utilisé pour laver les échantillons. Cette étape est importante pour évaluer l’efficacité de la désinfestation superficielle des échantillons.
- Dans une boîte autoclavée vide, placez les échantillons et utilisez un scalpel flammé et une pince pour sectionner les échantillons sur une épaisseur d’environ 0,2 cm.
- Sectionnez les échantillons cylindriques longitudinalement en deux moitiés si vous le souhaitez pour amplifier la surface en contact avec le milieu. Pour mieux exposer les fruits et autres organes plus sphériques, hachez ou coupez soigneusement les structures. Les graines peuvent également être d’importantes sources fongiques, alors assurez-vous que les fruits sectionnés les exposent au milieu.
REMARQUE : Seuls les organes sains, sans lésions tissulaires ni signes de maladies possibles ou d’infections pathogènes, doivent être utilisés dans l’isolement des endophytes fongiques. - Répartissez cinq fragments d’organes souterrains dans les boîtes de Pétri avec PDA + antibiotique. Assurez-vous que les fragments sont aussi éloignés que possible les uns des autres et qu’ils ne touchent pas non plus les bords de la parabole. Ne disposez pas de fragments sur le support. Préparer des copies de chaque organe installé, par précaution en cas de contamination.
- Scellez les boîtes de Pétri avec du film alimentaire et conservez-les à l’abri de la lumière à 25-27 °C (de préférence dans un incubateur) pendant 5 jours.
- Calcul de la fréquence d’isolement (FI)11
- Après 5 jours d’incubation des fragments d’organes, calculer la FI, en fonction du nombre de fragments incubés qui présentent des colonies fongiques en croissance, divisé par le total des fragments incubés, comme le montre l’équation suivante :

- Après 5 jours d’incubation des fragments d’organes, calculer la FI, en fonction du nombre de fragments incubés qui présentent des colonies fongiques en croissance, divisé par le total des fragments incubés, comme le montre l’équation suivante :
- Purification d’isolats fongiques par striation et sous-culture
REMARQUE : Chaque étape détaillée à l’étape 2.4 doit être effectuée à l’intérieur d’une hotte à flux laminaire.- Préparer des boîtes de Pétri (5 cm de diamètre) avec 5 à 7 g/L d’agar-agar (AA ; gélose bactériologique uniquement). Maintenir la vaisselle pendant 24 h à 36 °C pour éliminer celles qui pourraient être contaminées.
- Identifiez chaque colonie fongique cultivée à l’aide d’un code et délimitez ses marges sur la face inférieure de la boîte (cela peut être fait avec un marqueur permanent). Différenciez les colonies par la couleur, le modèle de croissance, la texture et le format des marges.
- À l’aide d’un cure-dent en bois autoclave, à l’aide de la pointe fine, récupérez une infime quantité de mycélium d’une colonie fongique. Concentrez-vous de préférence sur les marges de la colonie et choisissez une zone aussi éloignée que possible d’une autre colonie, en évitant de récupérer plus d’un type de colonie à la fois.
- À l’aide du même cure-dent avec du mycélium à l’extrémité, strier l’AA en produisant trois stries (rainures). Assurez-vous que chaque strie est à une distance de 1 cm l’une de l’autre et des bords de la parabole. Écrivez le code approprié (à l’aide d’un papier autocollant et d’un crayon), scellez les plats et maintenez-les incubés dans l’obscurité à 25-27 °C pendant 3 jours.
- Préparez des boîtes de Pétri (8 cm de diamètre) avec 39 g/L de PDA. Il n’est pas nécessaire d’ajouter des antibiotiques à ce stade.
- Après avoir incubé les boîtes AA, observez-les méticuleusement à contre-jour (d’une lampe ou d’une fenêtre), dans le but d’identifier les hyphes fins formant des colonies individuelles. Délimitez la zone d’une colonie individuelle par boîte, à l’aide d’un marqueur permanent sur la face inférieure de la boîte de Pétri.
- Dans une hotte à flux laminaire, utilisez un cure-dent autoclavé pour couper une partie du milieu contenant la colonie et transférer le volume coupé au centre d’une nouvelle boîte PDA.
- Identifiez les boîtes de Pétri avec les codes des isolats, scellez les boîtes avec un film alimentaire et maintenez-les dans l’obscurité à 25-27 °C pendant 7 à 14 jours.
3. Conservation des isolats fongiques purifiés
REMARQUE : Tous les matériaux, solutions et réactifs utilisés dans cette section doivent être stériles. Ceux qui ne peuvent pas être achetés déjà stérilisés doivent être autoclavés à 121 °C pendant 20 min.
- Conservation avec la méthode de Castellani ou dans de l’huile minérale (3-6 mois)11,15
- Préparer des tubes de microcentrifugation de 2 mL avec 0,5 mL d’eau distillée ou contenant 0,5 mL d’huile minérale (selon la méthode choisie). Assurez-vous que les tubes sont vides à l’autoclave et que l’eau et l’huile autoclavées sont ajoutées aux tubes à l’intérieur d’une hotte à flux laminaire.
- Dans une hotte à flux laminaire, placez les boîtes de Pétri avec des isolats purifiés déjà cultivés en PDA (39 g/L) pendant 7 à 14 jours. À l’aide d’un cure-dent autoclave, découpez de petits cuboïdes (0,5 cm x 0,5 cm dans la partie supérieure) d’un milieu contenant les marges du mycélium.
- Placez quatre à six cuboïdes dans les tubes de la microcentrifugeuse avec de l’eau distillée (méthode de Castellani) ou de l’huile minérale. Conservez les tubes à l’obscurité, à 25 °C, aussi longtemps que nécessaire, en respectant les limites de temps de la méthode.
REMARQUE : Évitez d’ajouter trop de cuboïdes dans les tubes et de les remplir, ce qui pourrait augmenter les risques de contamination. Il est possible de maintenir les tubes au réfrigérateur, ce qui peut favoriser la conservation de certains isolats plus longtemps. Currah et al.16 recommandent de conserver les répétitions d’isolats de champignons mycorhiziens provenant d’orchidées tropicales à la fois au réfrigérateur et à 25 °C, car elles peuvent être perdues lorsqu’elles sont entreposées au froid. - Récupérez un parallélépipède et placez-le au centre d’une nouvelle parabole PDA pour faire pousser un isolat stocké.
- Cryoconservation dans les grains de riz non décortiqués (>1 an)17
- Lavez les grains de riz non décortiqués à l’eau courante et faites-les cuire jusqu’à ce que la coque de riz commence à s’ouvrir. Répartissez les grains cuits dans des tubes à essai en verre avec un bouchon à vis et autoclavez deux fois avec un intervalle de 24 h entre les deux.
- Dans une hotte à flux laminaire, placez les boîtes de Pétri avec des isolats purifiés déjà cultivés en PDA (39 g/L) pendant 7 à 14 jours. À l’aide d’un cure-dent autoclave, prélevez cinq petits fragments d’hyphes sur les marges du mycélium et inoculez-les dans le tube contenant des grains de riz autoclavés.
REMARQUE : L’inoculation d’hyphes à différents points et profondeurs du tube garantit que l’isolat colonise les grains de riz non décortiqués en moins de temps. - Incuber les tubes dans l’obscurité à 25-27 °C pendant 14 jours. Agitez les tubes en les vortex tous les 3 jours pour garder les grains individualisés.
- Après avoir observé la croissance fongique dans les grains de riz, répartissez les grains dans une boîte de Pétri autoclavée tapissée de papier filtre pour absorber l’humidité, sur le papier afin que les grains puissent sécher. Conserver à l’abri de la lumière à 25-27 °C pendant 2-3 jours.
- Préparez les cryotubes avec 1/3 de gel de silice au fond et 1/3 de laine de verre au-dessus de la silice. Enfin, répartissez 1/3 des grains de riz avec du mycélium fongique cultivé. Conserver à -20 °C pendant 24 h.
- Après 24 h, stocker les cryotubes à -80 °C aussi longtemps que nécessaire, en respectant les limites de temps de la méthode.
- Cryoconservation dans la vermiculite à l’aide d’un cryoprotecteur (>1 an)18
- Préparer un milieu de culture liquide composé de 0,2 % d’extrait de levure et de 2 % de glucose dans de l’eau distillée. Ajustez le pH à 5 et autoclavez-le.
- Répartissez 0,2 g de vermiculite (utilisez une granulométrie fine) dans des cryotubes à l’aide d’un bouchon à vis et autoclavez-les. Ajouter 0,8 mL de milieu liquide autoclavé dans les cryotubes contenant de la vermiculite.
- Dans une hotte à flux laminaire, placez les boîtes de Pétri avec des isolats purifiés déjà cultivés en PDA (39 g/L) pendant 7 à 14 jours. À l’aide d’un cure-dent autoclave, prélever trois à cinq fragments d’hyphes sur les marges du mycélium et inoculer le long du tube contenant vermiculite + milieu de culture liquide. N’oubliez pas d’identifier les cryotubes avec le code d’isolat correspondant.
REMARQUE : L’inoculation d’hyphes en différents points et profondeurs du cryotube garantit que l’isolat colonise la vermiculite en moins de temps. - Stockez les cryotubes dans l’obscurité à 25-27 °C jusqu’à ce que la colonisation de la plupart des grains de vermiculite puisse être observée, ce qui prend généralement environ 14 jours.
- Préparez une solution cryoprotectrice composée de 5 % de glycérol et de 5 % de tréhalose dans de l’eau distillée et autoclavez-la. Laissez refroidir la solution avant de l’utiliser.
- Une fois que la vermiculite dans les cryotubes est colonisée par l’isolat fongique respectif, répartissez 0,4 mL de cryoprotecteur dans chaque tube et conservez les cryotubes au réfrigérateur à 4 °C pendant 48 h. Ensuite, maintenez les cryotubes à -80 °C aussi longtemps que nécessaire, en respectant les limites de temps de la méthode.
4. Caractérisation macromorphologique des champignons filamenteux (morphologie des colonies)
- Maintenir un registre photographique du mycélium cultivé dans chaque boîte de Pétri avec 39 g/L de PDA pendant 7 à 14 jours. N’oubliez pas d’enregistrer les deux côtés de la colonie, le dessus et le dessous (à l’envers). Si la vaisselle doit être facilement utilisée dans la section 6 ou si elle n’est pas entretenue, ouvrez la vaisselle lorsque vous les photographiez pour obtenir de meilleures photos.
- Si vous êtes intéressé par des données quantitatives sur la morphologie des colonies, la sous-culture réplique les isolats et les maintient dans les mêmes conditions de croissance et pendant une période constante pour enregistrer le diamètre de la colonie et calculer le taux de croissance (généralement en mm/h).
NOTE : Des résultats quantitatifs plus sophistiqués peuvent être obtenus en utilisant différents types de milieux de culture à des fins de comparaison19 et en considérant les outils statistiques pour traiter les données. - Observez également les colonies au stéréomicroscope pour identifier les caractéristiques morphologiques et photographiez-les à la loupe. Évaluer les colonies en fonction de leurs caractéristiques macromorphologiques, telles que détaillées dans la section des résultats. Consulter diverses sources bibliographiques pour aider à catégoriser et à relier la macromorphologie à l’identification moléculaire et/ou morphologique.
5. Caractérisation micromorphologique des champignons filamenteux (morphologie des hyphes)
NOTE : Les techniques micromorphologiques sont comparées dans la section de discussion, en tenant compte de leurs utilisations possibles et de leurs inconvénients.
- Cultivez les isolats sur des boîtes de Pétri avec 39 g/L de PDA pendant 7 à 14 jours. Pour évaluer les champignons mycorhiziens putatifs des orchidées, cultiver les isolats sur de la gélose à la semoule de maïs à 17 g/L (CMA ; ou un autre milieu limitant les nutriments) pendant 3 à 7 jours19.
- Monture de taquinerie
- Travaillez à l’intérieur d’une hotte à flux laminaire. Placez une goutte d’une teinture choisie (étape 5.5) sur une lame de verre propre.
- À l’aide d’un cure-dent autoclavé ou d’un autre matériau stérile, retirez soigneusement certains hyphes de l’isolat cultivé et placez-les dans la goutte de tache. Placez une lamelle (de préférence à un angle initial de 45° pour éviter les bulles d’air) et analysez-la au microscope optique.
- Support de ruban adhésif
REMARQUE : Cette technique est généralement appliquée dans les cultures fongiques pour être facilement utilisée dans la section 6 ou non entretenue, car le ruban adhésif ne peut pas être autoclavé et des contaminations peuvent se produire après l’exécution de la méthode.- Placez une goutte d’une teinture choisie (étape 5.5) sur une lame de verre propre. Découpez une bande de ruban adhésif transparent d’une taille qui s’adapte bien à la lame de verre et à la goutte de tache centrale.
- Dirigez la surface collante de la bande vers la surface du mycélium. N’appuyez pas et essayez de recueillir quelques hyphes sans en coller trop.
- Collez le ruban adhésif sur la lame de verre, en vous assurant que la tache est en contact avec les hyphes collectés. Placez une goutte d’eau au-dessus du ruban adhésif et placez une lamelle. Analysez la lame au microscope optique.
- Culture de lames de champignons filamenteux20
- À l’intérieur d’une grande boîte de Pétri (>9 cm de diamètre, de préférence), placez : du papier filtre dans la partie inférieure, une tige de verre en U ou une adaptation (le but est de fournir une élévation pour la lame de verre), une lame de verre et deux lamelles. Des boîtes de Pétri plus hautes facilitent la manipulation. Autoclaver ces kits de boîtes de Pétri, un pour chaque isolat.
- Préparez un petit volume de 39 g/L de PDA ou 17 g/L de CMA, ajoutez 10 g/L de gélose bactériologique et autocchez (le volume d’une boîte de Pétri est suffisant pour plus de 30 isolats). Les milieux de culture utilisés dans la culture des lames doivent être plus durs que les milieux habituels. Autoclaver 100 mL d’eau distillée.
- Travaillez à l’intérieur d’une hotte à flux laminaire. Placez le milieu liquide dans une boîte de Pétri, en produisant une couche d’environ 0,5 cm de haut. Laissez le milieu se solidifier. Une fois solide, utilisez un scalpel stérile pour couper des carrés du milieu de 1 cm x 1 cm.
- Ouvrez la trousse de boîte de Pétri à l’intérieur d’une hotte à flux laminaire et utilisez une pince flammée pour organiser le matériau autoclavé à l’intérieur de la boîte. Disposez le matériel dans l’ordre numéroté, comme indiqué à la figure 2A, en plaçant un carré de milieu de culture sur la lame de verre. Avant de placer la lamelle sur le support, passez à l’étape 5.4.5.
- Utilisez un cure-dent autoclavé pour récupérer quelques hyphes d’un isolat et frottez soigneusement les quatre faces latérales du milieu placé sur la lame de verre. Placez une lamelle autoclavée sur le carré moyen à l’aide d’une pince flammée.
- Utilisez une pointe de pipette stérile pour placer de l’eau autoclavée dans le papier filtre, afin de créer une chambre humide. Utilisez un volume pour saturer le papier sans mettre d’eau en excès. Scellez la boîte de Pétri avec un film alimentaire et identifiez-la avec le code d’isolat correspondant. Conservez la vaisselle dans l’obscurité à 25-27 °C pendant 3 à 7 jours.
- Évaluez la croissance des hyphes dans la lame de verre et la lamelle. Deux lames sont produites à partir de chaque kit de culture de lames, l’une utilisant la lame de verre avec hyphes et la lamelle autoclave, la seconde utilisant la lamelle avec hyphes et une autre lame de verre.
- Une fois la croissance des hyphes observée, détachez soigneusement le carré de milieu de la lame de verre (Figure 2B), montez les deux lames à l’aide d’une coloration choisie (étape 5.5) et analysez au microscope optique. N’oubliez pas d’identifier le code d’isolement sur la lame. Pour produire des lames semi-permanentes, scellez la lamelle avec du vernis à ongles transparent.
- Pour produire des lames permanentes, lavez soigneusement le colorant des hyphes collés après la coloration et laissez sécher la lame. Montage à l’aide d’un support de montage rapide (pour plus de détails, lire Pena-Passos et al.10).
REMARQUE : L’inconvénient de la production d’une lame permanente avec cette technique est que les spores et les structures qui n’adhèrent pas au verre peuvent être lavées.
- Méthodes de coloration et observation des hyphes
- Lactophénol bleu de coton (LPCB)21 : Ajouter 20 mL d’acide lactique, 40 mL de glycérol et 20 mL d’eau distillée. Dissoudre 20 g de cristaux de phénol dans cette solution en chauffant doucement. Dissoudre 0,05 g de bleu de méthyle (bleu de coton, ou 2 mL de solution aqueuse à 1 %). Le LPCB peut également être facilement acheté déjà préparé.
ATTENTION : Le phénol est hautement toxique et volatil ; Manipulez-le exclusivement à l’intérieur d’une hotte et portez des gants. - Bleu de toluidine O10 (TBO) : Préparer 0,05 % de TBO dans un tampon phosphate de 0,1 M (pH 6,8).
- Rouge Congo22,23,24 : Préparez une solution de rouge Congo à 1% dans de l’eau distillée et filtrez-la. Incuber pendant 5 à 10 min. Ce colorant peut également être appliqué pour la microscopie à fluorescence25.
- Lorsque vous observez et photographiez des hyphes fongiques au microscope optique, consultez diverses sources bibliographiques pour vous aider à identifier les structures (consultez la discussion).
- Lactophénol bleu de coton (LPCB)21 : Ajouter 20 mL d’acide lactique, 40 mL de glycérol et 20 mL d’eau distillée. Dissoudre 20 g de cristaux de phénol dans cette solution en chauffant doucement. Dissoudre 0,05 g de bleu de méthyle (bleu de coton, ou 2 mL de solution aqueuse à 1 %). Le LPCB peut également être facilement acheté déjà préparé.

Figure 2 : Procédures de culture de lames de champignons filamenteux. (A) Configuration schématique d’un kit de culture de lames, où les numéros indiquent l’ordre de disposition des éléments. (B) Le détachement du carré du milieu de culture après la croissance des hyphes est observé dans la lame de verre et la lamelle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Extraction totale d’ADN à partir d’isolats fongiques (protocole maison26 avec modifications 27)
REMARQUE : Tous les matériaux, solutions et réactifs utilisés dans cette section doivent être stériles. Ceux qui ne peuvent pas être achetés déjà stérilisés doivent être autoclavés à 121 °C pendant 20 min. Portez des gants pendant tout le protocole et effectuez quelques étapes à l’intérieur d’une hotte.
- Préparez le tampon d’extraction : 1 % de dodécylsulfate de sodium (SDS), 250 mM de NaCl, 200 mM de Tris-HCl (pH 8,0) et 25 mM d’acide éthylènediaminetétraacétique (EDTA). Cultivez les isolats purifiés dans 39 g/L de PDA pendant 7 à 14 jours.
- Grattez soigneusement le mycélium d’un isolat à l’aide d’une spatule ou d’une cuillère et transférez les fragments dans un mortier autoclave, en évitant de transférer le milieu de culture avec l’agrégat d’hyphes. À l’aide d’un pilon en porcelaine, broyez le mycélium avec de l’azote liquide jusqu’à l’obtention d’une poudre fine. Ne laissez pas le mycélium moulu fondre, ajoutez de l’azote pour éviter cela.
- Ajouter 1 mL de tampon d’extraction dans un tube de microcentrifugation de 2 mL et placer l’échantillon moulu jusqu’à la marque de 1,5 mL. Agitez doucement le contenu des tubes pour l’homogénéiser.
- Agitez les tubes en tourbillon pendant 5 s et placez-les dans un thermobloc à 65 °C pendant 20 min. Homogénéisez soigneusement le contenu par inversion toutes les 7 à 10 minutes.
- Centrifuger les tubes à 10 000 x g pendant 10 min à 4 °C. Transférez 800 μL de la phase supérieure dans un nouveau tube de microcentrifugation de 2 mL, ajoutez 800 μL de phénol dans les tubes et mélangez le contenu par inversion.
ATTENTION : Le phénol est un réactif hautement toxique et volatil ; Il peut provoquer un érythème, une gangrène et une nécrose des tissus. Lorsqu’il est inhalé, il peut provoquer une dyspnée et une toux. L’absorption systémique peut endommager le foie, les reins et le système nerveux central. Manipulez le phénol exclusivement à l’intérieur d’une hotte et portez des gants.
REMARQUE : À partir de l’étape suivante, utilisez des gants et effectuez le protocole à l’intérieur d’une hotte. - Centrifuger les tubes à 10 000 x g pendant 10 min à 4 °C et transférer 800 μL de la phase supérieure vers un nouveau tube de 2 mL, en évitant soigneusement le transfert du contenu de la phase inférieure.
- Ajouter 400 μL de phénol et 400 μL de chloroforme dans les tubes et mélanger le contenu par inversion. Centrifugez-les à 10 000 x g pendant 10 min à 4 °C.
ATTENTION : Le chloroforme est un réactif hautement toxique et volatil ; Il peut provoquer des irritations et des blessures au contact de la peau et, s’il est inhalé, affecte les systèmes nerveux central et cardiorespiratoire, le foie et les reins. Manipulez le chloroforme exclusivement à l’intérieur d’une hotte et portez des gants. - Récupérez 800 μL ou moins de la phase supérieure dans un nouveau tube de 2 mL. Ajouter 800 μL de chloroforme, mélanger le contenu par inversion et centrifuger à 10 000 x g pendant 10 min à 4 °C.
REMARQUE : Dans cette étape, aucun résidu de la phase inférieure ne doit être transféré dans de nouveaux tubes. Ainsi, soyez très prudent lors de la récupération de la phase supérieure. - Transférez 600 à 800 μL de la phase supérieure dans un nouveau tube de 1,5 mL et ajoutez 450 μL d’isopropanol. Mélanger le contenu par inversion et incuber à 25 °C pendant 5 min.
- Centrifuger les tubes à 10 000 x g pendant 5 min à 4 °C et jeter le surnageant à l’aide d’une micropipette. Attention à ne pas jeter la pastille déposée au fond du tube.
- Ajouter 500 μL d’éthanol à 80 % et centrifuger pendant 5 min. Répétez deux fois au cas où la pastille ne serait pas clarifiée.
- Retirez l’éthanol à l’aide d’une micropipette et séchez le granulé à 37 °C pendant 30 à 60 min. Ajouter 30 à 50 μL d’eau déminéralisée en éluant la pastille à l’aide d’une micropipette.
- Maintenez les tubes à 4 °C pendant la nuit pour une élution complète de l’ADN et congelez le contenu à -20 °C.
7. Extraction totale d’ADN à partir d’organes végétaux pour la méthodologie de métabarcoding (kit commercial)
REMARQUE : Pour la méthodologie suivante, il est nécessaire d’acheter le kit commercial indiqué dans le tableau des matériaux en tant que kit d’extraction d’ADN du sol. Tous les matériaux, solutions et réactifs utilisés dans cette section doivent être stériles. Ceux qui ne peuvent pas être achetés déjà stérilisés doivent être autoclavés à 121 °C pendant 20 min. Il est fortement recommandé de porter des gants pendant tout le protocole, et les étapes peuvent être effectuées à l’intérieur d’une hotte à flux laminaire. Le protocole décrit est modifié à partir de De Souza et al.12, à partir du protocole détaillé par le fabricant.
- À l’aide de pilons et de mortiers en porcelaine, broyer les racines recueillies à l’étape 1.2 dans de l’azote liquide, en réduisant les échantillons en une fine poudre. Ajouter 0,3 g des échantillons moulus dans les tubes PowerBead et agiter doucement pour homogénéiser.
- Ajouter 60 μL de solution C1 (incluse dans le kit) dans les tubes PowerBead et mélanger le contenu par inversion. À l’aide d’un homogénéisateur de tissus et d’un lyseur cellulaire (Table des matériaux), attachez fermement les tubes à un support adéquat, couplez le support au vortex et démarrez l’équipement à la vitesse maximale pendant 10 à 20 minutes.
REMARQUE : Dans le cas où la solution C1 est précipitée, chauffez-la à 60 °C jusqu’à dissolution complète. - Centrifuger les tubes à 10 000 x g pendant 30 s à 25 °C. Transférer 500 μL du surnageant dans des tubes de microcentrifugation de 2 mL. Le contenu transféré peut être particulaire.
- Ajouter 250 μL de solution C2 dans les tubes et agiter en tourbillon pendant 5 s. Incuber à 4 °C pendant 5 min, puis centrifuger à 10 000 x g pendant 1 à 2 min à 25 °C.
- Transvaser plus de 600 μL de surnageant dans de nouveaux tubes de 2 mL, en évitant la pastille. Ajouter 200 μL de solution C3 et agiter sur un vortex pendant 5 s.
- Incuber à 4 °C pendant 5 min puis centrifuger à 10 000 x g pendant 1 min à 25 °C. À ce stade, assurez-vous que le surnageant n’est pas particulaire.
- Transvaser plus de 750 μL du surnageant dans de nouveaux tubes de 2 mL, en évitant la pastille. Bien homogénéiser la solution C4, ajouter 1 100 μL de solution C4 au surnageant et agiter en tourbillon pendant 5 s.
- Chargez 675 μL du contenu des tubes dans les colonnes d’essorage MB, sur le filtre, et centrifugez à 10 000 x g pendant 1 min à 25 °C. Jetez le contenu liquide.
- Répétez l’étape précédente deux fois jusqu’à ce que tout le contenu de chaque tube soit traité. Ensuite, ajouter 500 μL de solution C5 au centre du filtre présent au niveau de la colonne supérieure du tube et centrifuger à 10 000 x g pendant 30 s à 25 °C.
- Jetez le contenu liquide et centrifugez à nouveau dans les mêmes conditions que l’étape précédente. Transférez avec précaution la colonne supérieure de la colonne de centrifugation MB dans de nouveaux tubes de microcentrifugation de 2 mL, en évitant que le contenu liquide de la colonne ne s’égoutte.
- Ajouter 85 à 100 μL de solution C6 au centre du filtre et attendre 1 min. Centrifuger à 10 000 x g pendant 30 s à 25 °C et jeter la colonne d’essorage MB. Conservez les tubes à -80 °C.
8. Quantification de l’ADN dans un spectrophotomètre (voir le tableau des matériaux)
- À l’aide du spectrophotomètre indiqué, ouvrez le logiciel. Sélectionnez l’option Acide nucléique, choisissez l’option ADN et définissez la concentration sur ng/μL.
- Ajoutez 1 μL d’eau déminéralisée au détecteur du spectrophotomètre et étalonnez en sélectionnant l’option Blank. Après la lecture, essuyez délicatement le détecteur avec du papier de soie doux.
- Nommez l’échantillon de champ avec le code d’isolat de l’échantillon à lire et placez-en 1 μL dans le détecteur de l’équipement. Sélectionnez l’option Mesurer ; Un graphique et un tableau avec les résultats sont générés.
- Répétez l’étape 8.3 jusqu’à ce que tous les échantillons aient été lus. Il est recommandé d’enregistrer la table avec les résultats générés pour l’enregistrement et l’analyse : sélectionnez l’option Rapports et l’emplacement où le fichier .xml sera enregistré.
- Ajoutez 5 μL d’eau déminéralisée dans le détecteur, attendez quelques minutes et essuyez-le délicatement avec du papier de soie doux.
Résultats
Dans le protocole d’isolement, étant donné qu’il y a contamination par l’eau utilisée lors du dernier lavage et que la contamination est également détectée dans les boîtes de Pétri avec des fragments inoculés, différentes mesures peuvent être prises, selon le type de contaminant (tableau 1). Cette procédure doit être répétée dès le début en cas de contaminants fongiques très sporulants, qui présentent également une croissance accélérée, et de bactéries multiplicatrices intenses, résistantes aux antibiotiques choisis. Au lieu de cela, si le contaminant fongique présente une croissance lente ou ne sporule pas, il est possible d’éviter simplement de l’isoler dans les étapes ultérieures du protocole. Enfin, les contaminations par des souches bactériennes qui se multiplient lentement peuvent également être facilement évitées dans les étapes postérieures, en particulier lors de la purification, lors de la récupération des hyphes.
Dans tous les cas présentés précédemment, le scénario idéal est de refaire la procédure de désinfestation en utilisant d’autres échantillons et d’installer les fragments de plantes dans un milieu de culture qui combine d’autres antibiotiques, avec un spectre d’action plus large, en cas de contamination des premiers plats par des bactéries résistantes. Cependant, compte tenu des difficultés liées à la collecte de nouveaux échantillons et à l’obtention d’organes à partir de plantes mycohétérotrophes, certains cas peuvent être résolus avec succès dans les étapes ultérieures du protocole, comme le montre le tableau 1.
| Contaminant | Caractéristiques des contaminants | Conséquences possibles de la contamination | Action requise | |||
| Bactéries | Multiplication intense | Inhibition totale ou partielle de la croissance des endophytes d’intérêt | Redémarrer l’isolement depuis le début | |||
| Multiplication lente | Isolement de la bactérie avec l’endophyte d’intérêt | Éviter les colonies de bactéries pendant la purification | ||||
| Mycètes | Croissance vigoureuse et/ou fortement sporulante | Rend impossible l’isolement et/ou la purification des endophytes d’intérêt | Redémarrer l’isolement depuis le début | |||
| Croissance lente et/ou non sporulante | Isolement du contaminant par erreur | Éviter l’isolement du contaminant | ||||
Tableau 1 : Description des contaminants possibles dans le processus d’isolement des champignons endophytes, conséquences de la contamination et procédures d’atténuation.
Après 5 jours à partir de l’installation des fragments d’organes dans le milieu PDA, il est possible de visualiser la croissance de petits groupes de mycéliums de champignons filamenteux, émergeant de l’intérieur des fragments (Figure 3). Chaque morphotype mycélien devrait aboutir à un isolat fongique à la fin du protocole d’isolement, étant donné que chacun d’entre eux doit être strié dans une nouvelle boîte de Pétri avec un milieu AA pour la purification. Il est conseillé de ne pas jeter les plats d’origine de l’installation de fragments avant d’avoir terminé l’ensemble du protocole d’isolement. Ils peuvent être conservés au réfrigérateur à 4 °C jusqu’à l’obtention des colonies purifiées. De plus, en ce qui concerne cette partie du protocole, l’analyse IF peut être très utile pour évaluer le pourcentage d’échantillons de tissus qui entraînent des champignons isolés parmi le total des échantillons installés.
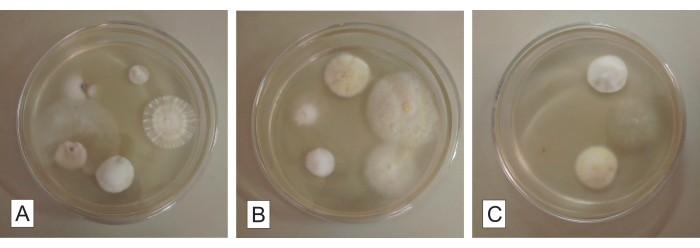
Figure 3 : Fragments de racines pour l’isolement fongique dans une gélose au dextrose de pomme de terre. Champignons endophytes cultivables poussant à partir des fragments de racines d’une orchidée mycohétérotrophe après environ 5 jours d’incubation. Chaque boîte de Pétri (A, B et C) contient cinq fragments de racines. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Après 3 jours suivant l’incubation des boîtes AA pour la purification des isolats, de minuscules colonies d’hyphes, presque imperceptibles, devraient s’être développées à partir des stries dans le milieu AA. À ce stade, en cas de contamination par des bactéries à croissance lente dans les boîtes d’isolement, il est possible que ces bactéries aient également été transférées dans les boîtes AA avec les hyphes endophytes fongiques sélectionnés. Si c’est le cas, les bactéries seront limitées aux régions striales, avec peu de croissance par rapport au champignon, ce qui permettra la récupération du seul champignon d’intérêt pour les nouveaux plats PDA.
Pendant 7 à 14 jours d’inoculation des isolats fongiques purifiés dans des boîtes PDA, la colonie pure doit croître par centrifugation (du centre de la boîte à sa périphérie), formant un seul mycélium circulaire (Figure 4). Les contaminations possibles sont facilement identifiées à ce stade, car elles compromettent considérablement l’homogénéité de la colonie en ce qui concerne sa croissance, sa forme, son aspect, sa couleur, sa production de pigments dans le milieu, etc. (Figure 5). En supposant que les colonies finales obtenues ne soient pas pures, les champignons initialement cultivés à partir des fragments d’organes doivent être soumis à nouveau à des procédures de purification, par striation et sous-culture (étape 2.4). Il est possible de récupérer les isolats soit dans les organes initialement installés, soit dans les boîtes d’isolement finales contenant des cultures hétérogènes.
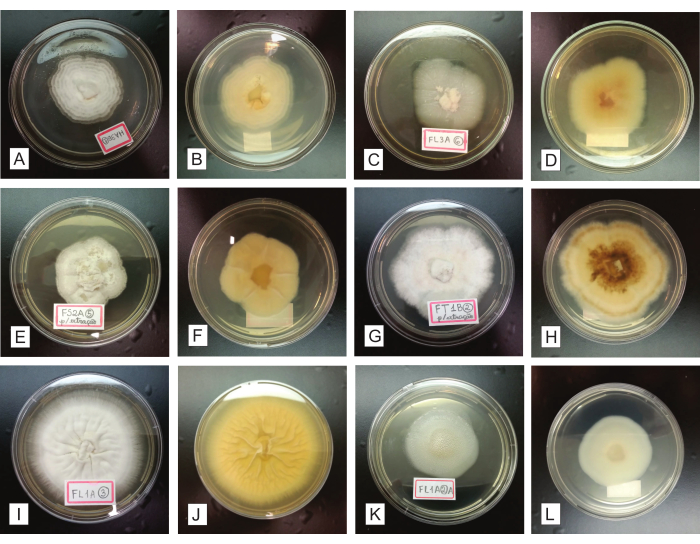
Figure 4 : Représentation d’isolats fongiques purifiés, cultivés pendant 7 à 14 jours dans un milieu de culture PDA. Dans les première et troisième colonnes (A, C, E, G, I et K), les colonies enregistrées sont vues du côté supérieur ; les deuxième et quatrième colonnes (B, D, F, H, J et L) présentent les mêmes colonies, vues respectivement du dessous. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Représentation d’isolats fongiques impurs, cultivés pendant 7 à 14 jours dans un milieu de culture PDA. Dans la première colonne (A, D et G), on voit une vue générale des colonies du côté supérieur ; la deuxième colonne (B, E et F) montre les colonies en détail ; la troisième colonne (C, F et I) montre les colonies par le dessous. Les chiffres représentent les différents morphotypes fongiques présents dans chaque boîte, et les lignes représentent une délimitation subtile entre les différents isolats fongiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La conservation des isolats ne doit pas se faire à l’aide d’une seule méthode de stockage, car chaque champignon peut présenter une sensibilité plus ou moins grande à chaque méthode décrite. Il est fortement conseillé de choisir au moins deux types de stockage pour chaque isolat fongique, garantissant plus de chances de succès dans sa conservation. Nous soulignons que l’inviabilité des champignons conservés dans Castellani ou l’huile minérale est attendue après 6 mois ou moins. Les chercheurs devraient se demander s’il s’agit des seules méthodes de conservation choisies, afin d’éviter d’éventuelles pertes inattendues d’isolats. Pour prolonger cette période limitée, il est possible de réactiver les isolats fongiques en les cultivant dans le milieu PDA (39 g/L) pendant 2-3 semaines, puis en les stockant à nouveau. Après une période de stockage, on s’attend à ce que les isolats présentent une croissance plus lente, par rapport au taux de croissance observé avant leur conservation, et des colonies avec des aspects moins vigoureux (c’est-à-dire moins denses, de couleur différente). La sous-culture quelques fois dans un milieu de culture riche en nutriments suffit à rétablir ces caractéristiques.
Lors de l’évaluation de la macromorphologie des colonies obtenues dans le cadre de la procédure d’isolement, des données qualitatives doivent être recueillies, en tenant compte du plus grand nombre possible de caractéristiques : (a) la couleur de la colonie (dessus et dessous), qui peut être analysée à l’aide de guides de couleurs imprimés (par exemple, Rayner28, Kornerup & Wanscher29, Ridgway30) ; b) opacité de la colonie : transparente, opaque, translucide ; c) pigments diffusibles dans le milieu31 (présence/absence et couleur) ; d) exsudats31 (présence/absence, couleur, aspect général) ; e) structures macroscopiques31 (présence/absence, type et aspect ; p. ex., sclérotes, pycnides) ; f) mycélium aérien et submergé 19,32,21 (présence/absence, apparence peu abondante ou abondante) ; g) l’aspect de la marge19 (couleur, forme, uniformité - submergée ou aérienne) ; et (h) la topographie de la colonie (entassée, ridée, cratériforme, plate, etc.), l’aspect général et la texture - cotonneux, velouté, poudré, laineux, sébacé (cireux), glabre, crayeux, visqueux, coriace, épineux, etc.19,21.
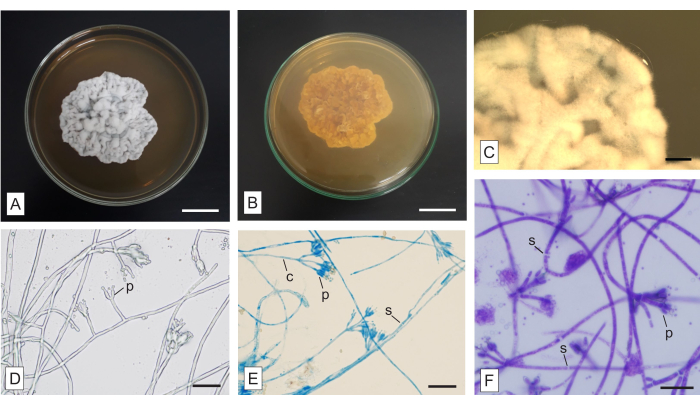
Figure 6 : Macro- et micromorphologie d’un champignon non identifié isolé de l’orchidée mycohétérotrophe Wullschlaegelia aphylla. (A) les aspects supérieurs et (B) inférieurs de la colonie, (C) vue amplifiée des hyphes aériens (tels qu’ils sont vus au stéréomicroscope). Micromorphologie des hyphes (D) sans coloration et colorés avec (E) LPCB et (F) TBO. Abréviations : c = conidiophore, p = phialide, s = septum. Barres d’échelle : A,B = 2 cm ; C = 2 mm ; D-F = 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La figure 6 montre une colonie isolée des racines fusiformes de l’orchidée mycohétérotrophe Wullschlaegelia aphylla, en appliquant la méthodologie décrite. La face supérieure de la colonie est blanchâtre à grise (figure 6A) et brunâtre sur la face inférieure (figure 6B). Il est opaque, sans pigments diffusibles dans le milieu ni exsudats. Les mycéliums sont aériens et abondants, les marges sont irrégulières et aériennes, et la colonie a une texture veloutée et une topographie ridée (figure 6A). Les structures macroscopiques sont absentes.
La méthode de culture des lames est traditionnelle et avantageuse et, bien qu’elle prenne du temps, elle peut être appliquée pour produire des lames permanentes qui peuvent être examinées par la suite. Les différents colorants présentés ici peuvent être utilisés pour mettre en évidence des structures importantes, et ils nécessitent des tests dans les échantillons, afin de déterminer le temps d’incubation le plus adéquat. Le rouge Congo et le TBO sont des teintures à usage général. Le rouge Congo est adéquat pour mettre en évidence certaines structures délicates (lecture complémentaire : Malloch22). Le TBO colore le contenu cellulaire plus intensément que la paroi cellulaire fongique (Figure 6F). Bien qu’il s’agisse d’un colorant habituel pour les structures végétales et qu’il ne soit pas si courant en microscopie fongique, le TBO possède une propriété métachromatique importante33, qui favorise d’autres applications, telles que l’analyse fongique dans les tissus végétaux10. Le LPCB est un colorant largement appliqué dans l’analyse fongique, bien qu’il exige de la prudence en raison du phénol. Le bleu de coton (synonyme : bleu de méthyle) est la tache, l’acide lactique est un agent de nettoyage et le phénol est un agent de destruction21. LPCB a une affinité pour la chitine et met en évidence des murs de spores et des ornementations23,24. Les cloisons fongiques ne sont colorées ni par le LPCB (Figure 6E) ni par le TBO (Figure 6F), ce qui facilite l’identification de ces structures. L’application de colorants est avantageuse pour mettre en évidence des structures qui ne sont pas facilement visibles lorsque les hyphes ne sont pas colorés (Figure 6D).
Pour l’extraction d’ADN à partir d’échantillons de champignons et de racines, la quantité d’échantillon moulu en azote liquide à ajouter au tampon d’extraction doit être respectée, sans dépasser la quantité indiquée dans le protocole. Il convient de souligner qu’une grande quantité d’échantillon traité ne représente pas simplement l’obtention d’une forte concentration d’ADN sans inconvénients majeurs, car elle peut compromettre considérablement la qualité finale de l’ADN. Cela se produit lors de la saturation des étapes suivantes de la purification de l’ADN. En outre, la concentration de composés putatifs produits par des plantes ou des champignons (par exemple, des pigments, des métabolites secondaires), présents dans la solution finale avec de l’ADN, peut agir pour réduire la qualité de l’ADN et/ou inhiber la réaction en chaîne par polymérase (PCR). Un autre problème qui peut réduire considérablement la qualité de l’ADN est la mise au rebut du milieu de culture avec du mycélium fongique et son broyage, ce qui doit être fortement évité. Certains réactifs de la procédure d’extraction (par exemple, le phénol, l’alcool éthylique, le SDS) et d’autres substances (par exemple, les pigments fongiques) peuvent être des inhibiteurs interférant avec la PCR.
Dans les étapes de nettoyage de l’ADN, lors de l’utilisation du phénol et du chloroforme, le surnageant doit toujours représenter la phase avec le moins d’impuretés, comprenant normalement la phase la plus claire. Au cours d’une telle étape, la phase inférieure ne doit pas être récupérée lors de l’aspiration du surnageant qui sera transféré dans de nouvelles trompes. À la fin du protocole, la pastille (constituée d’ADN) devrait présenter une couleur translucide (hautement souhaitable) à blanchâtre. Dans la phase de séchage, procéder au séchage de l’ADN dans un thermobloc est fondamental pour évaporer complètement l’éthanol. Après 12 h à 4 °C, la pastille doit avoir complètement élué dans la solution aqueuse ajoutée aux tubes, n’étant plus visible par la suite. Dans le cas où l’élution n’est pas complète et que la concentration d’ADN est bien inférieure à la concentration idéale, il est possible de conserver la solution réfrigérée pendant 12 h de plus, sans perte significative de sa qualité.
Les analyses d’ADN dans le logiciel génèrent un tableau, tel que représenté dans le tableau 2, où les données relatives à la quantité et à la qualité de l’ADN sont indiquées. La quantification de l’ADN est donnée par la concentration d’acides nucléiques en nanogrammes par microlitre d’un échantillon. Compte tenu de l’identification moléculaire des champignons isolés, les échantillons d’ADN devraient avoir une concentration d’environ 200 ng/μL, ce qui est idéal pour les étapes de PCR. Dans le cas d’échantillons avec des concentrations plus élevées d’acides nucléiques, une aliquote de l’échantillon doit être diluée, en rapprochant la concentration de la valeur mentionnée précédemment. Les échantillons présentant des concentrations excessivement élevées, tels que les échantillons 1 et 2 du tableau 2, peuvent suggérer une fausse détection en raison de contaminants dans la solution. Pendant ce temps, les échantillons avec des concentrations d’acides nucléiques plus faibles, comme le numéro 7 (tableau 2), peuvent toujours être utilisés pour la PCR, étant important pour appliquer un volume d’échantillon plus élevé dans les réactions. En ce qui concerne les paramètres de qualité, il est satisfaisant que les quotients 260/280 et 260/230 soient compris entre 1,8 et 2,2, comme l’indiquent les échantillons 3, 5 et 8 (tableau 2). Les échantillons qui présentent de telles valeurs (qui sont nettement inférieures ou supérieures) doivent être soumis à de nouvelles extractions d’ADN, comme l’indiquent les échantillons 9 et 10 du tableau 2. Dans le cas d’échantillons contenant une quantité suffisante d’ADN et dépassant au minimum la plage de qualité idéale, comme le suggèrent les valeurs de 260/280 et 260/230 des numéros 4 et 6 (tableau 2), la dilution est la procédure la plus avantageuse pour rendre la concentration d’acide nucléique adéquate pour la PCR et diluer les inhibiteurs éventuels, tels que les réactifs résiduels de la procédure d’extraction. dans les échantillons.
| Échantillon | Acide nucléique Conc. | Unité | Réf. A260 | Réf. A280 | 260/280 | 260/230 | Type d’échantillon |
| 1 | 14491.7 | ng/μL | 289.833 | 141.175 | 2.05 | 1.8 | ADN |
| 2 | 13359.3 | ng/μL | 267.187 | 124.607 | 2.14 | 2.15 | ADN |
| 3 | 1137.6 | ng/μL | 22.751 | 10.574 | 2.15 | 2.13 | ADN |
| 4 | 1472.6 | ng/μL | 29.452 | 13.287 | 2.22 | 2.16 | ADN |
| 5 | 3464.8 | ng/μL | 69.295 | 33.329 | 2.08 | 1.88 | ADN |
| 6 | 1884.2 | ng/μL | 37.684 | 17.912 | 2.1 | 1.78 | ADN |
| 7 | 187.6 | ng/μL | 3.751 | 1.834 | 2.05 | 2.06 | ADN |
| 8 | 1580.3 | ng/μL | 31.607 | 15.281 | 2.07 | 1.98 | ADN |
| 9 | 923.3 | ng/μL | 18.466 | 9.196 | 2.01 | 1.37 | ADN |
| 10 | 2414.4 | ng/μL | 48.287 | 21.008 | 2.3 | 3.45 | ADN |
Tableau 2 : Résultats générés dans le logiciel à partir d’échantillons d’ADN fongique analysés dans un spectrophotomètre.
Différente des échantillons d’ADN de champignons endophytes isolés, la concentration d’ADN dans les échantillons pour les analyses de métabarcoding doit présenter des quantités massives de molécules d’ADN extraites, de haute qualité et de poids moléculaire. Les échantillons doivent être évalués par électrophorèse dans du gel d’agarose, en obtenant la plus grande intégrité possible, avec peu ou pas de bavures dans le gel.
Discussion
La désinfestation superficielle des échantillons de plantes est l’une des étapes les plus critiques du protocole présenté. Il est fortement souhaitable qu’il n’y ait pas de contamination de la vaisselle PDA par des gouttes provenant du dernier lavage. Les bactéries sont fréquemment observées en tant que contaminants dans les boîtes d’isolement, généralement plus que les champignons sporulants en suspension dans l’air, étant donné que les bactéries endophytes sont également courantes dans les tissus végétaux 3,11. Ainsi, l’ajout d’antibiotiques dans le milieu de culture lors de l’installation des fragments d’organes est essentiel. De meilleurs résultats sont obtenus en combinant différents types d’antibiotiques, ce qui se traduit par un spectre d’action plus large. Une autre considération importante est le biais intrinsèque dans l’isolement des champignons, car l’utilisation de PDA et d’AA sélectionnera inévitablement certaines espèces et en défavorisera d’autres. La combinaison d’autres milieux de culture peut minimiser le biais, mais pas éliminer ces limitations9.
En général, les champignons qui produisent des spores en poussant dans des boîtes de Pétri présentent des taux de survie plus élevés dans les protocoles de conservation, à des températures plus basses ou non, et les isolats non sporulants peuvent ne pas tolérer la conservation de Castellani ou de l’huile minérale34,35,36, de sorte qu’une méthode de cryoconservation peut être cruciale dans de tels cas. De plus, certains isolats peuvent être sensibles à la congélation, et l’ajout de cryoprotecteurs facilite la conservation, comme dans la méthode présentée utilisant la vermiculite18. Les caractéristiques de la colonie et des hyphes peuvent être utiles pour regrouper les isolats ayant des caractères similaires, facilitant la sélection pour des utilisations dans des traitements de germination symbiotique (tels que détaillés par Pena-Passos et al.10). Ces caractéristiques contribuent également au choix des méthodes de conservation à appliquer, notamment en tenant compte de la sporulation ou de son absence.
La caractérisation morphologique des isolats permet également d’améliorer et de compléter les descriptions morphologiques disponibles dans la littérature spécialisée, en particulier lorsqu’elles sont associées à la caractérisation moléculaire. Bien qu’il s’agisse d’un défi avec de nombreux isolats, la caractérisation morphologique des champignons prend du temps et dépend des données publiées. Même si seule la caractérisation moléculaire est prévue, il est conseillé de tenir un registre photographique de l’aspect de la colonie (macromorphologie) et de le publier chaque fois que cela est possible, afin de contribuer aux futures procédures d’identification. Il est également important de conserver des photos des boîtes où les fragments sont installés, afin de permettre la comparaison entre les isolats finaux obtenus et les morphotypes observés dans les boîtes d’installation. Dans ce cas, les champignons comparés doivent être cultivés dans le même type de milieu. D’autres méthodologies sont trouvées dans Currah et al.16 pour évaluer les champignons mycorhiziens communs des orchidées, telles que la culture sur milieu d’acide tannique (TAM) pour différencier les genres Epulorhiza (téléomorphe : Tulasnella) et Ceratorhiza (téléomorphe : Ceratobasidium), et la culture sur CMA pour stimuler les cellules monilioïdes pour la caractérisation des champignons du complexe Rhizoctonia .
Compte tenu des méthodes détaillées d’observation des hyphes fongiques au microscope optique, le montage par taquine et le montage par ruban adhésif sont plus rapides que la culture sur lames, bien que la méthode de montage par taquin ne soit pas adéquate pour analyser les spores et mesurer les angles des branches. Le montage par ruban adhésif préserve mieux l’organisation du mycélium que la technique de montage par taquine, bien que l’utilisation du ruban adhésif ne soit pas aussi fiable que la culture de lames pour mesurer les angles des branches (observation des auteurs). La culture de lames, bien qu’elle prenne du temps, est la technique la plus adéquate pour produire des lames semi-permanentes et permanentes. La macro- et la micromorphologie sont analysées en tenant compte de la littérature. Webster and Weber32 est une source complète d’informations générales et de références supplémentaires. Currah et al.16 et Zettler et Corey37 fournissent des informations utiles sur les champignons mycorhiziens des orchidées. Bien qu’ils décrivent des champignons d’importance médicale, Walsh et al.21 et McGinnis31 sont également utiles pour les caractéristiques générales des colonies de champignons filamenteux, les informations visuelles/descriptives et les références supplémentaires pour la micromorphologie fongique.
Selon Yu et al.38, les analyses de quantification de l’ADN dans un spectrophotomètre présentent une grande précision et exactitude, étant cohérentes avec les analyses quantitatives de PCR en temps réel. Une observation importante est que certains isolats peuvent ne pas fournir des échantillons d’ADN suffisamment purs pour les étapes d’identification moléculaire ultérieures, car les endophytes fongiques des mycohétérotrophes sont extrêmement diversifiés, en particulier ceux associés aux plantes tropicales39, tout comme les métabolites produits. Des analyses spectrophotométriques peuvent être utilisées pour évaluer de tels cas et différents protocoles, ou des kits commerciaux appliqués pour la purification de l’ADN des échantillons. Les étapes subséquentes suggérées dans l’identification moléculaire des isolats sont l’amplification de la région de l’espaceur transcrit interne (ITS) et le séquençage. La région ITS est un ADN d’espacement de l’ARN ribosomique largement utilisé dans la littérature spécialisée et formellement accepté comme code-barres principal pour l’identification fongique 9,40. Il peut être amplifié par PCR avec les amorces ITS1 et ITS441, puis séquencé dans la plate-forme Sanger42.
Les analyses de métabarcoding, en plus de permettre d’étudier la richesse, l’abondance et la composition taxonomique des micro-organismes dans les échantillons environnementaux et végétaux, peuvent fournir des résultats d’interactions positives ou négatives entre ces micro-organismes12. L’étape de macération à l’azote liquide ajouté au protocole standard DNeasy PowerSoil vise à casser le plus grand nombre possible de cellules fongiques par rapport aux tissus végétaux, permettant ainsi un meilleur échantillonnage de la communauté fongique dans le tissu macéré11. Le choix des amorces pour les séquences fongiques (codes-barres) dans l’identification moléculaire doit tenir compte de l’interférence avec l’ADN de la plante. Nous conseillons l’utilisation d’amorces qui amplifient les régions variables du gène fongique 18S, par exemple, ITS1-5,8S-ITS243. Après l’obtention des échantillons d’ADN, les étapes suivantes consistent à préparer les banques métagénomiques, qui dépendent de la plate-forme de séquençage choisie, le séquençage a posteriori des banques et l’analyse des données obtenues à l’aide d’outils bioinformatiques. Nous recommandons d’utiliser la plate-forme Illumina MiSeq, qui représente une option moins coûteuse avec un rendement élevé, générant des séquences de 250 pb en peu de temps11,44.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer et n’ont aucun conflit d’intérêts.
Remerciements
Nous remercions les financements de la FAPESP (2015/26479-6) et du CNPq (447453/2014-9). JLSM remercie le CNPq pour les subventions à la productivité (303664/2020-7). Le MPP remercie Capes (bourse de maîtrise, processus 88887.600591/2021-00) et CNPq.
matériels
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Références
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , Springer-Verlag. Berlin. 1-13 (2006).
- Smith, S. E., Read, D. J. Mycorrhizal Symbiosis. , Elsevier Science Academic Press. London. (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777(2022).
- Araújo, W. L., et al. Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, Santarém: UFOPA. 257(2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774(2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589(2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069(2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. Larone's Medically Important Fungi: A Guide to Identification. , ASM Press. (2018).
- Malloch, D. Moulds: Isolation, Cultivation, Identification. , Available from: http://website.nbm-mnb.ca/mycologywebpages/Moulds/Moulds.html (1997).
- Smith, P. Microscopy: Chemical Reagents. British Mycological Society. , Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022).
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , Santarém: UFOPA. 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. Methuen Handbook of Colour. Methuen handbook of colour. , Methuen & Co. Ltd. London. (1967).
- Ridgway, R. Color Standards and Color Nomenclature. , Washington, DC. Published by the author (1912).
- McGinnis, M. R. Laboratory Handbook of Medical Mycology. , Elsevier Science. (2012).
- Webster, J., Weber, R. Introduction to Fungi. , Cambridge University Press. Cambridge, UK. (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon