Method Article
Aislamiento, caracterización y extracción de ADN total para identificar hongos endófitos en plantas micoheterótrofas
En este artículo
Erratum Notice
Resumen
El presente artículo tiene como objetivo proporcionar protocolos detallados y adecuados para el aislamiento de hongos endófitos asociados a plantas, la preservación a largo plazo de aislados, la caracterización morfológica y la extracción de ADN total para su posterior identificación molecular y análisis metagenómicos.
Resumen
Las plantas micoheterótrofas presentan una de las formas más extremas de dependencia micorrícica, habiendo perdido totalmente su capacidad autótrofa. Tan esencial como cualquier otro recurso vital, los hongos con los que estas plantas se asocian íntimamente son esenciales para ellas. De ahí que algunas de las técnicas más relevantes en el estudio de especies micoheterótrofas sean las que permiten la investigación de hongos asociados, especialmente aquellos que habitan en raíces y órganos subterráneos. En este contexto, se aplican comúnmente técnicas para identificar hongos endófitos dependientes e independientes del cultivo. El aislamiento de endófitos fúngicos proporciona un medio para identificarlos morfológicamente, analizar su diversidad y mantener los inóculos para aplicaciones en la germinación simbiótica de semillas de orquídeas. Sin embargo, se sabe que existe una gran variedad de hongos no cultivables que habitan en los tejidos vegetales. Por lo tanto, las técnicas de identificación molecular independientes del cultivo ofrecen una cobertura más amplia de la diversidad y abundancia de especies. El objetivo de este artículo es proporcionar el apoyo metodológico necesario para iniciar dos procedimientos de investigación: uno dependiente de la cultura y otro independiente. En cuanto al protocolo dependiente del cultivo, se detallan los procesos de recolección y mantenimiento de muestras de plantas desde los sitios de recolección hasta las instalaciones de laboratorio, junto con el aislamiento de hongos filamentosos de órganos subterráneos y aéreos de plantas micoheterótrofas, el mantenimiento de una colección de aislados, la caracterización morfológica de las hifas mediante la metodología de cultivo de portaobjetos y la identificación molecular de hongos mediante la extracción total de ADN. Abarcando metodologías independientes del cultivo, los procedimientos detallados incluyen la recolección de muestras de plantas para análisis metagenómicos y la extracción total de ADN de órganos de plantas aclorofílicas utilizando un kit comercial. Por último, también se sugieren protocolos de continuidad (p. ej., reacción en cadena de la polimerasa [PCR], secuenciación) para los análisis, y aquí se presentan técnicas.
Introducción
Los hongos endófitos son, por definición, aquellos que habitan en el interior de los órganos y tejidos de las plantas en infecciones discretas (es decir, sin causar daño a su huésped)1,2. Estos hongos pueden interactuar de forma neutra o beneficiosa con las plantas hospederas, pueden conferir resistencia a patógenos y condiciones ambientales desfavorables, y pueden contribuir a la síntesis de compuestos beneficiosos para la planta (por ejemplo, factores de crecimiento y otras fitohormonas)1,3. Los endófitos micorrícicos son hongos que establecen asociaciones micorrícicas con la planta, participando en la transferencia de nutrientes4. En Orchidaceae, la interacción con endófitos micorrícicos es fundamental para la germinación de semillas en la gran mayoría de las especies, y el establecimiento de plántulas en todas las plantas de la familia5. En tales contextos, las orquídeas micoheterótrofas representan un caso de dependencia total con respecto a sus socios micorrícicos, ya que dependen de la transferencia de nutrientes minerales y compuestos de carbono por parte de estos hongos durante todo su ciclo de vida6. Por lo tanto, el aislamiento e identificación de hongos asociados es una base fundamental a la hora de investigar estrategias de vida micoheterótrofas. Además, poco se sabe sobre el papel de los endófitos fúngicos en las plantas micoheterótrofas o incluso la diversidad real de estos hongos 7,8.
La investigación de hongos endófitos puede llevarse a cabo mediante diferentes técnicas, tradicionalmente descritas como independientes o dependientes del cultivo, por ejemplo: (a) observación directa, (b) aislamiento de hongos e identificación morfológica y/o molecular, y (c) extracción total de ADN de tejidos vegetales e identificación molecular9. En observación directa (a), los hongos endófitos pueden ser investigados mientras aún se encuentran en el interior de células y tejidos vegetales mediante microscopía óptica o electrónica9, ya que los diferentes protocolos de microscopía son detallados por Peña-Passos et al.10. Mediante métodos de aislamiento (b), los endófitos fúngicos pueden caracterizarse de acuerdo con sus colonias, hifas y morfología de la estructura reproductiva o de resistencia. Además, a través de técnicas de aislamiento, es posible llevar a cabo la identificación molecular de aislados a través de la extracción de ADN, la amplificación de secuencias de identificación molecular (códigos de barras o huellas dactilares) y la secuenciación11. Esta última técnica (c) permite la identificación molecular de hongos endófitos mediante la extracción de ADN en el interior de los tejidos vegetales (metacódigo de barras), seguida de la preparación y secuenciación de la biblioteca12.
Además, los aislados de hongos pueden aplicarse en ensayos de germinación simbiótica, utilizando semillas de orquídeas autótrofas o micoheterótrofas. Un ejemplo de esta aplicación es la investigación realizada por Sisti et al.13, describiendo la germinación y las etapas iniciales del desarrollo de protocormos en Pogoniopsis schenckii, una orquídea micoheterótrofa, en asociación con algunos de sus aislados, que comprenden hongos endófitos no micorrícicos. El protocolo de germinación simbiótica aplicado se detalla y presenta en un video de Pena-Passos et al.10. El aislamiento de hongos en asociación con diferentes órganos de la planta permite diversos enfoques de investigación con respecto a la naturaleza de las interacciones planta-hongo (por ejemplo, para comprender los aspectos ecológicos o fisiológicos de la asociación, así como indagaciones sobre la transferencia de nutrientes de los hongos a la planta)9.
Las metodologías presentadas en la sección 1 se basan en la recolección de muestras de órganos subterráneos, ya que estos órganos presentan más dificultades en la recolección, y son de gran interés ya que los endófitos micorrícicos los colonizan. Sin embargo, ambos protocolos incluidos (pasos 1.1 y 1.2) pueden aplicarse a otros órganos de plantas micoheterótrofas (p. ej., rizomas, tallos florales y frutos). La metodología de recolección descrita en el paso 1.1 está diseñada para aislar hongos endófitos (sección 2), para la caracterización morfológica (secciones 4 y 5) y/o la extracción total de ADN para la identificación aislada (sección 6). Por otro lado, la metodología de recolección descrita en el paso 1.2 se asigna exclusivamente a la extracción de ADN total de tejidos vegetales para técnicas de metabarcoding (sección 7). En la sección 3 se presentan cuatro métodos de almacenamiento y conservación de hongos filamentosos, dos para el almacenamiento a corto plazo (3-6 meses) y los otros dos adecuados para el almacenamiento a largo plazo (>1 año). La caracterización morfológica (secciones 4 y 5) puede asociarse con la identificación molecular para reforzarla y proporcionar información importante sobre la macro y micromorfología fúngica. En la Figura 1 se resumen las metodologías colectivas que se describen a continuación.

Figura 1: Resumen esquemático de los métodos presentados. Recolección de plantas y aislamiento, preservación e identificación molecular de hongos mediante metodologías dependientes e independientes del cultivo. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Recogida de muestras de plantas
- Recopilación de muestras para métodos dependientes de la referencia cultural
- Excava cuidadosamente los órganos subterráneos; Estos pueden ser raíces, tallos, rizomas u órganos de almacenamiento de la planta que se va a recolectar. Aparte de los suelos muy compactos, recoja estas muestras a mano.
NOTA: El uso de herramientas como llanas o cucharas en este paso es desaconsejable, ya que puede dañar las estructuras frágiles de las plantas micoheterótrofas y puede causar contaminación de los tejidos por hongos no endófitos. - Recoge tantos órganos subterráneos como sea posible. Mantenga las muestras en bolsas de papel dentro de un recipiente refrigerado (por ejemplo, una caja de espuma de poliestireno o una bolsa térmica con hielo). Si también se recogen órganos aéreos, transpórtelos por separado de los subterráneos.
- Excava cuidadosamente los órganos subterráneos; Estos pueden ser raíces, tallos, rizomas u órganos de almacenamiento de la planta que se va a recolectar. Aparte de los suelos muy compactos, recoja estas muestras a mano.
- Recogida de muestras para métodos independientes de la referencia cultural
- Excave cuidadosamente las raíces de la planta que se va a recolectar bajo las mismas recomendaciones señaladas en la NOTA del paso 1.1.1.
- Recoge tantos órganos subterráneos como sea posible. Mantenga las muestras recolectadas en criotubos dentro de nitrógeno líquido (opción deseable) o use tubos de centrífuga rodeados de hielo seco (opción alternativa). Mantenga los órganos aéreos por separado, si se recogen.
2. Aislamiento de hongos endófitos asociados a órganos vegetales14
NOTA: Todos los materiales, soluciones y reactivos utilizados en esta sección deben ser estériles. Los que no se pueden comprar ya esterilizados deben esterilizarse en autoclave a 121 °C durante 20 min.
- Desinfestación superficial de órganos vegetales
- Lave las muestras recogidas (paso 1.1.2) con agua corriente y elimine la mayor cantidad posible de sustrato y otros residuos que puedan tener las muestras.
- Dentro de una campana de flujo laminar, mantenga las muestras lavadas sumergidas en etanol al 70% durante 1 min (se puede usar un vaso de precipitados o un frasco de vidrio).
- Transfiera las muestras a otro recipiente con hipoclorito de sodio con cloro activo al 2% durante 3 min.
- Cambie las muestras a un recipiente con etanol al 70% y manténgalas sumergidas durante 1 min. Después, lave las muestras secuencialmente en dos recipientes con agua destilada.
- Instalación de fragmentos de plantas en medio de cultivo
NOTA: Cada paso detallado en el paso 2.2 debe realizarse dentro de una campana de flujo laminar.- Antes de la instalación, prepare placas de Petri (8-10 cm de diámetro) con 19,5 g/L de medio de cultivo en agar patata dextrosa (PDA) + 7 g/L de agar bacteriológico + 3 mL/L de antibióticos (por ejemplo, estreptomicina, penicilina, tetraciclina, ampicilina).
- Mantenga las placas de Petri con el medio de cultivo a 36 °C durante 24 h antes de usarlas para asegurarse de que no haya contaminación. Deseche los platos contaminados con colonias de bacterias u hongos.
NOTA: La adición de antibióticos en el medio es esencial en este paso, ya que los órganos están altamente infectados por bacterias que pueden inhibir el crecimiento de hongos. Verifique si los antibióticos se pueden calentar durante el autoclave; algunos antibióticos deben agregarse después de enfriar el medio a alrededor de 36 ° C (antes de colocarlo en los platos). - Inmediatamente después de la desinfestación superficial de las muestras y aún dentro de una campana de flujo laminar, en PDA, inocular unas gotas del agua destilada del último recipiente utilizado para lavar las muestras. Este paso es importante para evaluar la eficacia de la desinfestación superficial de las muestras.
- En un plato vacío esterilizado en autoclave, coloque las muestras y utilice un bisturí flameado y unas pinzas para seccionar las muestras hasta un grosor de unos 0,2 cm.
- Seccionar las muestras cilíndricas longitudinalmente en dos mitades si se desea amplificar la superficie en contacto con el medio. Para exponer mejor las frutas y otros órganos más esféricos, pique o corte bien las estructuras. Las semillas también pueden ser fuentes importantes de hongos, así que asegúrese de que la fruta seccionada las exponga al medio.
NOTA: Solo los órganos sanos, sin daño tisular o señales de posibles enfermedades o infecciones patógenas, deben usarse en el aislamiento de endófitos fúngicos. - Distribuya cinco fragmentos de los órganos subterráneos en las placas de Petri con PDA + antibiótico. Asegúrese de que los fragmentos estén lo más lejos posible entre sí y que no toquen los bordes del plato. No coloque ningún fragmento en el medio. Preparar réplicas de cada órgano instalado, como precaución en caso de contaminación.
- Selle las placas de Petri con film transparente y guárdelas en la oscuridad a 25-27 °C (preferiblemente en una incubadora) durante 5 días.
- Cálculo de frecuencia de aislamiento (IF)11
- Después de 5 días de incubación de los fragmentos de órganos, calcule el FI, de acuerdo con el número de fragmentos incubados que presentan colonias fúngicas en crecimiento dividido por el total de fragmentos incubados, como se representa en la siguiente ecuación:

- Después de 5 días de incubación de los fragmentos de órganos, calcule el FI, de acuerdo con el número de fragmentos incubados que presentan colonias fúngicas en crecimiento dividido por el total de fragmentos incubados, como se representa en la siguiente ecuación:
- Purificación de aislados fúngicos mediante estriado y subcultivo
NOTA: Cada paso detallado en el paso 2.4 debe realizarse dentro de una campana de flujo laminar.- Preparar placas de Petri (5 cm de diámetro) con 5-7 g/L de agar-agar (AA; solo agar bacteriológico). Mantener la vajilla durante 24 h a 36 °C para eliminar los posibles contaminantes.
- Identifique cada colonia de hongos cultivados con un código y delimite sus márgenes en la parte inferior del plato (se puede hacer con un marcador permanente). Diferencie las colonias por color, patrón de crecimiento, textura y formato de margen.
- Con un palillo de madera esterilizado en autoclave, con la punta fina, recupere una pequeña cantidad de micelio de una colonia de hongos. Concéntrese preferentemente en los márgenes de la colonia y elija un área lo más alejada posible de otra colonia, evitando recuperar más de un tipo de colonia a la vez.
- Usando el mismo palillo de dientes con micelio en la punta, estriar el AA produciendo tres estrías (surcos). Asegúrese de que cada estría esté a una distancia de 1 cm de la otra y de los bordes del plato. Escriba el código apropiado (con papel adhesivo y lápiz), selle los platos y manténgalos incubados en la oscuridad a 25-27 ° C durante 3 días.
- Preparar placas de Petri (8 cm de diámetro) con PDA de 39 g/L. No es necesario agregar antibióticos en esta etapa.
- Después de incubar las placas AA, obsérvelas meticulosamente a contraluz (de una lámpara o una ventana), con el objetivo de identificar hifas finas que forman colonias individuales. Delimite el área de una colonia individual por placa, usando un marcador permanente en la parte inferior de la placa de Petri.
- En una campana de flujo laminar, use un palillo de dientes esterilizado en autoclave para cortar una porción del medio que contiene la colonia y transfiera el volumen cortado al centro de una nueva placa PDA.
- Identifique las placas de Petri con los códigos de los aislados, selle las placas con film transparente y manténgalas en la oscuridad a 25-27 °C durante 7-14 días.
3. Conservación de aislados fúngicos purificados
NOTA: Todos los materiales, soluciones y reactivos utilizados en esta sección deben ser estériles. Los que no se pueden comprar ya esterilizados deben esterilizarse en autoclave a 121 °C durante 20 min.
- Conservación con el método de Castellani o en aceite mineral (3-6 meses)11,15
- Preparar tubos de microcentrífuga de 2 mL con 0,5 mL de agua destilada o que contengan 0,5 mL de aceite mineral (dependiendo del método elegido). Asegúrese de que los tubos estén vacíos en autoclave y que el agua y el aceite esterilizados en autoclave se agreguen a los tubos dentro de una campana de flujo laminar.
- En una campana de flujo laminar, colocar las placas de Petri con aislados purificados ya cultivados en PDA (39 g/L) durante 7-14 días. Con un palillo esterilizado en autoclave, corte cuboides pequeños (0,5 cm x 0,5 cm en la zona superior) de medio que contengan los márgenes del micelio.
- Coloque de cuatro a seis cuboides en los tubos de microcentrífuga con agua destilada (método de Castellani) o aceite mineral. Almacene los tubos en la oscuridad, a 25 °C durante el tiempo que sea necesario, respetando las limitaciones de tiempo del método.
NOTA: Evite agregar demasiados cuboides a los tubos y hacerlos llenos, lo que puede aumentar las posibilidades de contaminación. Es posible mantener los tubos refrigerados, lo que puede favorecer la conservación de algunos aislados durante más tiempo. Currah et al.16 recomiendan almacenar las réplicas de aislados de hongos micorrícicos de orquídeas tropicales tanto refrigeradas como a 25 °C, ya que pueden perderse cuando se almacenan en frío. - Recupere un cuboide y colóquelo en el centro de una nueva antena PDA para cultivar un aislado almacenado.
- Criopreservación en granos de arroz sin cáscara (>1 año)17
- Lave los granos de arroz sin cáscara en agua corriente y cocínelos hasta que la cáscara de arroz comience a abrirse. Distribuir los granos cocidos en tubos de ensayo de vidrio con tapón de rosca y esterilizar en autoclave dos veces con un intervalo de 24 h entre ellos.
- En una campana de flujo laminar, colocar las placas de Petri con aislados purificados ya cultivados en PDA (39 g/L) durante 7-14 días. Con un palillo esterilizado en autoclave, recupere cinco pequeños fragmentos de hifas de los márgenes del micelio e inocule en el tubo que contiene los granos de arroz esterilizados en autoclave.
NOTA: La inoculación de hifas en diferentes puntos y profundidades del tubo garantiza que el aislado colonice los granos de arroz sin cáscara en menos tiempo. - Incubar los tubos en la oscuridad a 25-27 °C durante 14 días. Agite los tubos en vórtice cada 3 días para mantener los granos individualizados.
- Después de observar el crecimiento de hongos en los granos de arroz, distribuya los granos en una placa de Petri esterilizada en autoclave forrada con papel de filtro para absorber la humedad, sobre el papel para que los granos se sequen. Conservar en la oscuridad a 25-27 °C durante 2-3 días.
- Prepare los criotubos con 1/3 de gel de sílice en la parte inferior y 1/3 de lana de vidrio por encima de la sílice. Finalmente, distribuya 1/3 de los granos de arroz con micelio fúngico cultivado. Conservar a -20 °C durante 24 h.
- Después de 24 h, almacenar los criotubos a -80 °C durante el tiempo que sea necesario, respetando las limitaciones de tiempo del método.
- Criopreservación en vermiculita mediante crioprotector (>1 año)18
- Preparar un medio de cultivo líquido compuesto por un 0,2% de extracto de levadura y un 2% de glucosa en agua destilada. Ajuste el pH a 5 y autoclave.
- Distribuir 0,2 g de vermiculita (utilizar granulometría fina) en criotubos con tapón de rosca y esterilizarlos en autoclave. Agregue 0,8 ml de medio líquido esterilizado en autoclave a los criotubos que contienen vermiculita.
- En una campana de flujo laminar, colocar las placas de Petri con aislados purificados ya cultivados en PDA (39 g/L) durante 7-14 días. Con un palillo esterilizado en autoclave, recupere de tres a cinco fragmentos de hifas de los márgenes del micelio e inocule a lo largo del tubo que contiene vermiculita + medio de cultivo líquido. Recuerde identificar los criotubos con el respectivo código de aislamiento.
NOTA: La inoculación de hifas en diferentes puntos y profundidades del criotubo garantiza que el aislado colonice la vermiculita en menos tiempo. - Almacenar los criotubos en la oscuridad a 25-27 °C hasta que se pueda observar la colonización de la mayoría de los granos de vermiculita, lo que suele tardar unos 14 días.
- Preparar una solución crioprotectora compuesta por un 5% de glicerol y un 5% de trehalosa en agua destilada y esterilizarla en autoclave. Deje que la solución se enfríe antes de usarla.
- Después de que la vermiculita en los criotubos sea colonizada por el respectivo aislado de hongo, distribuya 0,4 mL de crioprotector en cada tubo y mantenga los criotubos refrigerados a 4 °C durante 48 h. Posteriormente, mantenga los criotubos a -80 °C durante el tiempo que sea necesario, respetando las limitaciones de tiempo del método.
4. Caracterización macromorfológica de hongos filamentosos (morfología de colonias)
- Mantener un registro fotográfico del micelio cultivado en cada placa de Petri con 39 g/L PDA durante 7-14 días. Recuerde registrar ambos lados de la colonia, superior e inferior (reverso). Si los platos se van a usar fácilmente en la sección 6 o no se mantienen, abra los platos al fotografiarlos para obtener mejores fotos.
- Si está interesado en datos cuantitativos sobre la morfología de la colonia, el subcultivo replica los aislados y los mantiene en las mismas condiciones de crecimiento y durante un período constante para registrar el diámetro de la colonia y calcular la tasa de crecimiento (generalmente en mm/h).
NOTA: Se pueden lograr resultados cuantitativos más sofisticados utilizando diferentes tipos de medios de cultivo para la comparación19 y considerando las herramientas estadísticas para tratar los datos. - Observe las colonias bajo un microscopio estereoscópico para identificar las características morfológicas y fotografiarlas con aumento. Evaluar las colonias de acuerdo a sus características macromorfológicas, tal y como se detalla en el apartado de resultados. Consultar diversas fuentes bibliográficas que ayuden a categorizar y relacionar la macromorfología con la identificación molecular y/o morfológica.
5. Caracterización micromorfológica de hongos filamentosos (morfología de hifas)
NOTA: Las técnicas micromorfológicas se comparan en la sección de discusión, considerando sus posibles usos y desventajas.
- Cultive los aislados en placas de Petri con 39 g/L de PDA durante 7-14 días. Para evaluar los hongos micorrícicos putativos de las orquídeas, cultive los aislados en agar harina de maíz (CMA; u otro medio limitante de nutrientes) de 17 g/L durante 3-7 días19.
- Montura de burla
- Trabaje dentro de una campana de flujo laminar. Coloque una gota de un tinte elegido (paso 5.5) en un portaobjetos de vidrio limpio.
- Con un palillo de dientes esterilizado en autoclave u otro material estéril, retire con cuidado algunas hifas del aislado cultivado y colóquelas en la gota de mancha. Coloque un cubreobjetos (preferiblemente en un ángulo inicial de 45° para evitar burbujas de aire) y analice bajo un microscopio óptico.
- Soporte de cinta adhesiva
NOTA: Esta técnica se suele aplicar en cultivos fúngicos para ser fácilmente utilizados en la sección 6 o no mantenidos, ya que la cinta adhesiva no se puede esterilizar en autoclave, y pueden producirse contaminaciones después de ejecutar el método.- Coloque una gota de un tinte elegido (paso 5.5) en un portaobjetos de vidrio limpio. Corta una tira de cinta adhesiva transparente del tamaño que se ajuste bien al portaobjetos de vidrio y a la caída del tinte central.
- Dirija la superficie pegajosa de la tira hacia la superficie del micelio. No presiones, y trata de recoger algunas hifas sin pegar demasiadas.
- Pega la cinta al portaobjetos de vidrio, asegurándote de que la mancha esté en contacto con las hifas recogidas. Coloque una gota de agua sobre la cinta y coloque un cubreobjetos. Analice el portaobjetos bajo un microscopio óptico.
- Cultivo de diapositivas de hongos filamentosos20
- Dentro de una placa de Petri grande (>9 cm de diámetro, preferiblemente), coloque: papel de filtro en la parte inferior, una varilla de vidrio en forma de U o una adaptación (el objetivo es proporcionar una elevación para el portaobjetos de vidrio), un portaobjetos de vidrio y dos cubreobjetos. Las placas de Petri más altas facilitan la manipulación. Autoclave estos kits de placas de Petri, uno para cada aislado.
- Preparar un pequeño volumen de 39 g/L de PDA o 17 g/L de CMA, añadir 10 g/L de agar bacteriológico y autoclave (el volumen de una placa de Petri es suficiente para más de 30 aislamientos). Los medios de cultivo utilizados en el cultivo de diapositivas deben ser más duros que los medios habituales. Autoclave 100 mL de agua destilada.
- Trabaje dentro de una campana de flujo laminar. Coloque el medio líquido en una placa de Petri, produciendo una capa de alrededor de 0,5 cm de altura. Deja que el medio se solidifique. Una vez sólido, use un bisturí estéril para cortar cuadrados del medio que tengan una dimensión de 1 cm x 1 cm.
- Abra el kit de placa de Petri dentro de una campana de flujo laminar y use pinzas flameadas para organizar el material esterilizado en autoclave dentro de la placa. Coloque el material en el orden numerado, como se ve en la Figura 2A, colocando un cuadrado de medio de cultivo en el portaobjetos de vidrio. Antes de colocar el cubreobjetos sobre el medio, continúe con el paso 5.4.5.
- Use un palillo de dientes esterilizado en autoclave para recuperar algunas hifas de un aislado y frote cuidadosamente las cuatro caras laterales del medio colocado en el portaobjetos de vidrio. Coloque un cubreobjetos esterilizado en autoclave sobre el cuadrado mediano con pinzas flameadas.
- Utilice una punta de pipeta estéril para colocar agua esterilizada en autoclave en el papel de filtro, para crear una cámara húmeda. Utilice un volumen para saturar el papel sin colocar agua en exceso. Selle la placa de Petri con film transparente e identifíquela con el respectivo código de aislamiento. Mantener la vajilla en la oscuridad a 25-27 °C durante 3-7 días.
- Evalúe el crecimiento de las hifas en el portaobjetos de vidrio y en el cubreobjetos. A partir de cada kit de cultivo de portaobjetos se producen dos portaobjetos, uno utilizando el portaobjetos de vidrio con hifas y el cubreobjetos esterilizado en autoclave, el segundo utilizando el portaobjetos con hifas y otro portaobjetos de vidrio.
- Después de observar el crecimiento de las hifas, separe con cuidado el cuadrado de medio del portaobjetos de vidrio (Figura 2B), monte los dos portaobjetos utilizando una tinción elegida (paso 5.5) y analice bajo un microscopio óptico. Recuerde identificar el código de aislamiento en la diapositiva. Para producir portaobjetos semipermanentes, selle el cubreobjetos con esmalte de uñas transparente.
- Para producir portaobjetos permanentes, lave cuidadosamente el tinte de las hifas adheridas después de teñirlo y deje que el portaobjetos se seque. Monte con un medio de montaje rápido (para más detalles, lea Peña-Passos et al.10).
NOTA: El inconveniente de producir un portaobjetos permanente con esta técnica es que las esporas y las estructuras no adheridas al vidrio pueden lavarse.
- Métodos de tinción y observación de hifas
- Azul de algodón de lactofenol (LPCB)21: Agregue 20 ml de ácido láctico, 40 ml de glicerol y 20 ml de agua destilada. Disuelva 20 g de cristales de fenol en esta solución calentando suavemente. Disolver 0,05 g de azul de metilo (azul de algodón o 2 ml de solución acuosa al 1%). El LPCB también se puede comprar fácilmente ya preparado.
PRECAUCIÓN: El fenol es altamente tóxico y volátil; Manipularlo exclusivamente dentro de una campana extractora y usar guantes. - Azul de toluidina O10 (TBO): Preparar TBO al 0,05% en tampón fosfato 0,1 M (pH 6,8).
- Rojo Congo22,23,24: Preparar una solución de rojo Congo al 1% en agua destilada y filtrarla. Incubar durante 5-10 min. Este colorante también se puede aplicar para microscopía de fluorescencia25.
- Al observar y fotografiar hifas fúngicas bajo un microscopio óptico, consulte diversas fuentes bibliográficas para ayudar a identificar estructuras (consulte la discusión).
- Azul de algodón de lactofenol (LPCB)21: Agregue 20 ml de ácido láctico, 40 ml de glicerol y 20 ml de agua destilada. Disuelva 20 g de cristales de fenol en esta solución calentando suavemente. Disolver 0,05 g de azul de metilo (azul de algodón o 2 ml de solución acuosa al 1%). El LPCB también se puede comprar fácilmente ya preparado.

Figura 2: Procedimientos para el cultivo de portaobjetos de hongos filamentosos . (A) Configuración esquemática de un kit de cultivo de portaobjetos, donde los números indican el orden de disposición de los elementos. (B) Se observa el desprendimiento del cuadrado del medio de cultivo después del crecimiento de hifas en el portaobjetos de vidrio y el cubreobjetos. Haga clic aquí para ver una versión más grande de esta figura.
6. Extracción de ADN total de aislados fúngicos (protocolo casero26 con modificaciones 27)
NOTA: Todos los materiales, soluciones y reactivos utilizados en esta sección deben ser estériles. Los que no se pueden comprar ya esterilizados deben esterilizarse en autoclave a 121 °C durante 20 min. Use guantes durante todo el protocolo y realice algunas etapas dentro de una campana extractora.
- Prepare el tampón de extracción: dodecil sulfato de sodio (SDS) al 1%, 250 mM de NaCl, 200 mM de Tris-HCl (pH 8,0) y 25 mM de ácido etilendiaminotetraacético (EDTA). Cultive los aislados purificados en 39 g/L de PDA durante 7-14 días.
- Raspar cuidadosamente el micelio de un aislado con una espátula o una cuchara y transferir los fragmentos a un mortero esterilizado en autoclave, evitando transferir el medio de cultivo con el agregado de hifas. Con un mortero de porcelana, muele el micelio con nitrógeno líquido hasta obtener un polvo fino. No deje que el micelio que se está moliendo se derrita, agregando nitrógeno para evitarlo.
- Agregue 1 ml de tampón de extracción en un tubo de microcentrífuga de 2 ml y coloque la muestra molida hasta la marca de 1,5 ml. Agitar suavemente el contenido de los tubos para homogeneizar.
- Agitar los tubos en un vórtice durante 5 s y colocarlos en un termobloque a 65 °C durante 20 min. Homogeneizar cuidadosamente el contenido por inversión cada 7-10 min.
- Centrifugar los tubos a 10.000 x g durante 10 min a 4 °C. Transfiera 800 μL de la fase superior a un nuevo tubo de microcentrífuga de 2 mL, agregue 800 μL de fenol a los tubos y mezcle el contenido por inversión.
PRECAUCIÓN: El fenol es un reactivo altamente tóxico y volátil; Puede causar eritema, gangrena y necrosis tisular. Cuando se inhala, puede causar disnea y tos. La absorción sistémica puede dañar el hígado, los riñones y el sistema nervioso central. Manipule el fenol exclusivamente dentro de una campana extractora y use guantes.
NOTA: A partir del siguiente paso, use guantes y realice el protocolo dentro de una campana extractora. - Centrifugar los tubos a 10.000 x g durante 10 min a 4 °C y transferir 800 μL de la fase superior a un nuevo tubo de 2 mL, evitando cuidadosamente la transferencia del contenido de la fase inferior.
- Añadir 400 μL de fenol y 400 μL de cloroformo a los tubos y mezclar el contenido por inversión. Centrifugarlos a 10.000 x g durante 10 min a 4 °C.
PRECAUCIÓN: El cloroformo es un reactivo altamente tóxico y volátil; Puede causar irritación y lesiones cuando entra en contacto con la piel y, si se inhala, afecta los sistemas nervioso central y cardiorrespiratorio, el hígado y los riñones. Manipule el cloroformo exclusivamente dentro de una campana extractora y use guantes. - Recupere 800 μL o menos de la fase superior a un nuevo tubo de 2 mL. Añadir 800 μL de cloroformo, mezclar el contenido por inversión y centrifugar a 10.000 x g durante 10 min a 4 °C.
NOTA: En este paso, no se deben transferir residuos de la fase inferior a tubos nuevos. Por lo tanto, tenga mucho cuidado al recuperar la fase superior. - Transfiera 600-800 μL de la fase superior a un nuevo tubo de 1,5 mL y agregue 450 μL de isopropanol. Mezclar el contenido por inversión e incubar a 25 °C durante 5 min.
- Centrifugar los tubos a 10.000 x g durante 5 min a 4 °C y desechar el sobrenadante con una micropipeta. Tenga cuidado de no desechar el gránulo depositado en el fondo del tubo.
- Añadir 500 μL de etanol al 80% y centrifugar durante 5 min. Repita dos veces en caso de que el gránulo no se aclare.
- Retirar el etanol con una micropipeta y secar el pellet a 37 °C durante 30-60 min. Añadir 30-50 μL de agua desionizada, eluyendo el pellet con una micropipeta.
- Mantenga los tubos a 4 °C durante la noche para una elución completa del ADN y congele el contenido a -20 °C.
7. Extracción de ADN total de órganos vegetales para metodología de metabarcoding (kit comercial)
NOTA: Para la siguiente metodología, es necesario adquirir el kit comercial indicado en la Tabla de Materiales como kit de extracción de ADN de suelo. Todos los materiales, soluciones y reactivos utilizados en esta sección deben ser estériles. Los que no se pueden comprar ya esterilizados deben esterilizarse en autoclave a 121 °C durante 20 min. Se recomienda encarecidamente el uso de guantes durante todo el protocolo, y los pasos se pueden realizar dentro de una campana de flujo laminar. El protocolo descrito es una modificación de De Souza et al.12, a partir del protocolo detallado por el fabricante.
- Utilizando morteros de porcelana, muela las raíces recogidas siguiendo el paso 1.2 en nitrógeno líquido, reduciendo las muestras a un polvo fino. Añadir 0,3 g de las muestras molidas a los tubos PowerBead y agitar suavemente para homogeneizar.
- Añadir 60 μL de solución C1 (incluida en el kit) a los tubos PowerBead y mezclar el contenido por inversión. Utilizando un homogeneizador de tejidos y un lisador celular (Tabla de Materiales), ate los tubos firmemente a un soporte adecuado, acople el soporte al vórtice y ponga en marcha el equipo a máxima velocidad durante 10-20 min.
NOTA: En caso de que la solución C1 se precipite, caliéntela a 60 °C hasta su completa disolución. - Centrifugar los tubos a 10.000 x g durante 30 s a 25 °C. Transfiera 500 μL del sobrenadante a tubos de microcentrífuga de 2 mL. El contenido transferido puede ser partículas.
- Añadir 250 μL de solución C2 a los tubos y agitar en un vórtice durante 5 s. Incubar a 4 °C durante 5 min y luego centrifugar a 10.000 x g durante 1-2 min a 25 °C.
- Transfiera más de 600 μL del sobrenadante a nuevos tubos de 2 mL, evitando el pellet. Añadir 200 μL de solución C3 y agitar en un vórtice durante 5 s.
- Incubar a 4 °C durante 5 min y luego centrifugar a 10.000 x g durante 1 min a 25 °C. En esta etapa, asegúrese de que el sobrenadante no sea partícula.
- Transfiera más de 750 μL del sobrenadante a nuevos tubos de 2 mL, evitando el pellet. Homogeneizar bien la solución C4, añadir 1.100 μL de solución C4 al sobrenadante y agitar en un vórtice durante 5 s.
- Cargue 675 μL del contenido de los tubos en las columnas de centrifugación MB, sobre el filtro, y centrifugue a 10.000 x g durante 1 min a 25 °C. Deseche el contenido líquido.
- Repita el paso anterior dos veces hasta que se procese todo el contenido de cada tubo. A continuación, añadir 500 μL de solución C5 en el centro del filtro presente en la columna superior del tubo y centrifugar a 10.000 x g durante 30 s a 25 °C.
- Deseche el contenido líquido y vuelva a centrifugar en las mismas condiciones que en el paso anterior. Transfiera con cuidado la columna superior de la columna giratoria MB a nuevos tubos de microcentrífuga de 2 ml, evitando gotear cualquier contenido líquido en la columna.
- Añadir 85-100 μL de solución C6 en el centro del filtro y esperar 1 min. Centrifugar a 10.000 x g durante 30 s a 25 °C y desechar la columna de centrifugación MB. Almacenar los tubos a -80 °C.
8. Cuantificación de ADN en un espectrofotómetro (consultar la Tabla de Materiales)
- Con el espectrofotómetro indicado, abra el software. Seleccione la opción Ácido nucleico, elija la opción ADN y establezca la concentración en ng/μL.
- Agregue 1 μL de agua desionizada al detector de espectrofotómetro y calibre seleccionando la opción En blanco. Después de leer, limpie suavemente el detector con papel de seda suave.
- Asigne un nombre a la muestra de campo con el código de aislamiento de la muestra a leer y coloque 1 μL de la misma en el detector del equipo. Seleccione la opción Medir; Se genera un gráfico y una tabla con los resultados.
- Repita el paso 8.3 hasta que se hayan leído todas las muestras. Se recomienda guardar la tabla con los resultados generados para su registro y análisis: seleccione la opción Informes y la ubicación donde se guardará el archivo .xml.
- Agregue 5 μL de agua desionizada al detector, espere varios minutos y límpielo suavemente con papel de seda suave.
Resultados
En el protocolo de aislamiento, considerando que existe contaminación por el agua utilizada en el último lavado y que la contaminación también se detecta en las placas de Petri con fragmentos inoculados, se pueden tomar diferentes acciones, dependiendo del tipo de contaminante (Tabla 1). Este procedimiento debe repetirse desde el principio en caso de contaminantes fúngicos altamente esporulantes, que también presentan un crecimiento acelerado, y bacterias de multiplicación intensa, resistentes a los antibióticos elegidos. En cambio, si el contaminante fúngico presenta un crecimiento lento o no esporula, es posible simplemente evitar aislarlo en los pasos posteriores del protocolo. Por último, las contaminaciones con cepas bacterianas que se multiplican lentamente también pueden evitarse fácilmente en los pasos posteriores, especialmente en la purificación, al recuperar las hifas.
En todos los casos presentados anteriormente, el escenario ideal es rehacer el procedimiento de desinfestación utilizando otras muestras e instalar los fragmentos de la planta en un medio de cultivo que combine otros antibióticos, con un espectro de acción más amplio, en los casos de bacterias resistentes que contaminen las primeras placas. Sin embargo, teniendo en cuenta las dificultades que entraña la recolección de nuevas muestras y la obtención de órganos de plantas micoheterótrofas, algunos casos pueden resolverse con éxito en las etapas posteriores del protocolo, como se presenta en la Tabla 1.
| Contaminante | Características del contaminante | Posible consecuencia de la contaminación | Acción requerida | |||
| Gérmenes | Multiplicación intensa | Inhibición total o parcial del crecimiento de los endófitos de interés | Reiniciar el aislamiento desde el principio | |||
| Multiplicación lenta | Aislamiento de la bacteria junto con el endófito de interés | Evite las colonias de bacterias durante la purificación | ||||
| Hongos | Crecimiento vigoroso y/o muy esporulado | Imposibilita el aislamiento y/o purificación de los endófitos de interés | Reiniciar el aislamiento desde el principio | |||
| Crecimiento lento y/o no esporulado | Aislamiento del contaminante por error | Evite el aislamiento del contaminante | ||||
Tabla 1: Descripción de los posibles contaminantes en el proceso de aislamiento de hongos endófitos, consecuencias de la contaminación y procedimientos de mitigación.
Después de 5 días desde la instalación de los fragmentos de órgano en el medio PDA, es posible visualizar el crecimiento de pequeños grupos de micelios de hongos filamentosos, emergiendo del interior de los fragmentos (Figura 3). Cada morfotipo micelial debe dar lugar a un aislado fúngico al final del protocolo de aislamiento, considerando que cada uno de ellos debe ser estriado en una nueva placa de Petri con medio AA para su purificación. Se aconseja que las placas originales de la instalación del fragmento no se desechen antes de completar todo el protocolo de aislamiento. Pueden conservarse refrigerados a 4 °C hasta obtener las colonias purificadas. Además, con respecto a esta parte del protocolo, el análisis de FI puede ser de gran ayuda para evaluar el porcentaje de muestras de tejido que dan lugar a hongos aislados entre el total de muestras instaladas.
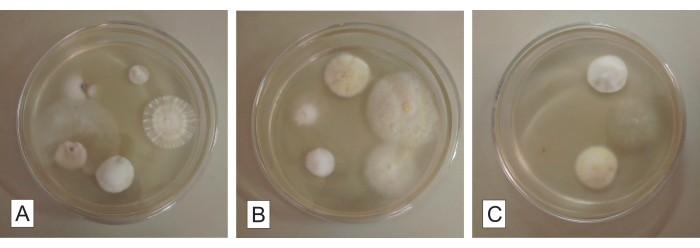
Figura 3: Fragmentos de raíces para aislamiento fúngico en agar papa dextrosa. Hongos endófitos cultivables que crecen a partir de los fragmentos de raíz de una orquídea micoheterótrofa después de unos 5 días de incubación. Cada placa de Petri (A, B y C) contiene cinco fragmentos de raíz. Haga clic aquí para ver una versión más grande de esta figura.
Después de 3 días después de la incubación de las placas AA para la purificación aislada, las diminutas colonias de hifas, casi imperceptibles, deberían haber crecido a partir de las estrías en el medio AA. En esta etapa, en caso de que hubiera alguna contaminación por bacterias de crecimiento lento en las placas de aislamiento, es posible que estas bacterias también se transfirieran a las placas AA con las hifas endófitas fúngicas seleccionadas. Si es así, las bacterias se restringirán a las regiones de las estrías, con poco crecimiento en comparación con el hongo, lo que permitirá la recuperación únicamente del hongo de interés para las nuevas placas de PDA.
Durante 7-14 días de inoculación de los aislados fúngicos purificados en placas de PDA, la colonia pura debe crecer centrífugamente (desde el centro de la placa hasta su periferia), formando un único micelio circular (Figura 4). Las posibles contaminaciones son fácilmente identificables en esta etapa, ya que comprometen considerablemente la homogeneidad de la colonia en cuanto a su crecimiento, forma, aspecto, color, producción de pigmentos en el medio, etc. (Figura 5). Suponiendo que las colonias finales obtenidas no sean puras, los hongos cultivados inicialmente a partir de los fragmentos de órganos deben someterse de nuevo a procedimientos de purificación, mediante estrías y subcultivos (paso 2.4). Es posible recuperar los aislamientos de los órganos instalados inicialmente o de las placas de aislamiento final que contienen cultivos heterogéneos.
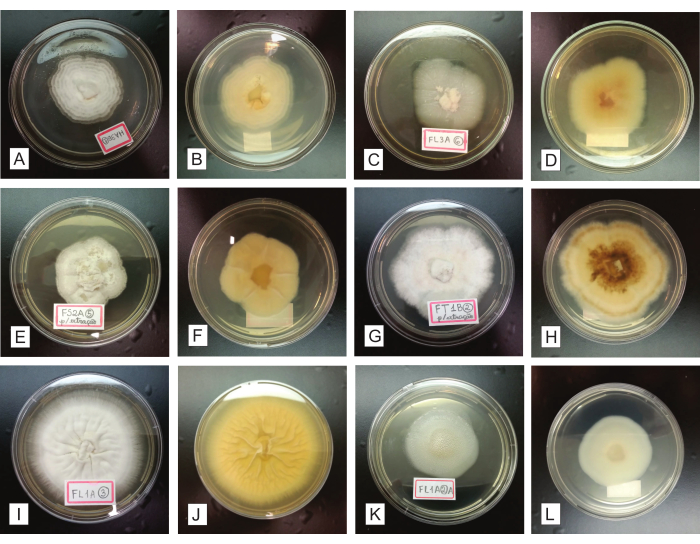
Figura 4: Representación de aislados fúngicos purificados, cultivados durante 7-14 días en un medio de cultivo PDA. En la primera y tercera columna (A, C, E, G, I y K), las colonias registradas se ven desde la parte superior; la segunda y cuarta columnas (B, D, F, H, J y L) presentan las mismas colonias, respectivamente vistas desde la parte inferior. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Representación de aislados de hongos impuros, cultivados durante 7-14 días en medio de cultivo PDA. En la primera columna (A, D y G), se ve una vista general de las colonias desde la parte superior; la segunda columna (B, E y F) muestra las colonias en detalle; la tercera columna (C, F e I) muestra las colonias desde la parte inferior. Los números representan diferentes morfotipos fúngicos presentes en cada plato, y las líneas representan una sutil delimitación entre los diferentes aislados de hongos. Haga clic aquí para ver una versión más grande de esta figura.
La conservación de los aislados no debe realizarse utilizando un único método de almacenamiento, ya que cada hongo puede presentar mayor o menor sensibilidad a cada método descrito. Es muy recomendable elegir al menos dos tipos de almacenamiento para cada aislado de hongo, garantizando más posibilidades de éxito en su conservación. Destacamos que la inviabilidad de los hongos conservados en Castellani o aceite mineral se espera después de 6 meses o menos. Los investigadores deben considerar si estos son los únicos métodos de preservación elegidos, para evitar posibles pérdidas inesperadas de aislados. Para prolongar este período limitado, es posible reactivar los aislados de hongos cultivándolos en el medio PDA (39 g/L) durante 2-3 semanas y luego almacenándolos nuevamente. Después de un período de almacenamiento, se espera que los aislados presenten un crecimiento más lento, en comparación con la tasa de crecimiento observada antes de su preservación, y colonias con aspectos menos vigorosos (es decir, menos densos, de diferente color). El subcultivo unas cuantas veces en un medio de cultivo rico en nutrientes es suficiente para restablecer tales características.
Al evaluar la macromorfología de las colonias obtenidas en el procedimiento de aislamiento, se deben recolectar datos cualitativos, considerando tantas características como sea posible: (a) color de la colonia (parte superior e inferior), que se puede analizar utilizando guías de color impresas (por ejemplo, Rayner28, Kornerup y Wanscher29, Ridgway30); b) opacidad de las colonias: transparente, opaca, translúcida; (c) pigmentos difusibles en el medio31 (presencia/ausencia y color); d) exudados31 (presencia/ausencia, color, aspecto general); (e) estructuras macroscópicas31 (presencia/ausencia, tipo y apariencia; por ejemplo, esclerocios, picnidios); f) micelios aéreos y sumergidos 19,32,21 (presencia/ausencia, apariencia escasa o abundante); g) aspecto de los márgenes19 (color, forma, uniformidad, sumergido o aéreo); y (h) la topografía de la colonia (amontonada, arrugada, crateriforme, plana, etc.), el aspecto general y la textura: algodonosa, aterciopelada, polvorienta, lanosa, sebácea (cerosa), glabra, calcárea, viscosa, coriácea, espinosa, etc.19,21.
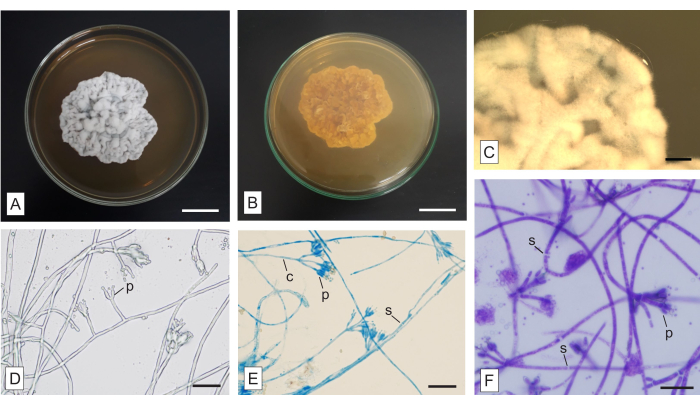
Figura 6: Macro y micromorfología de un hongo no identificado aislado de la orquídea micoheterótrofa Wullschlaegelia aphylla. (A) Aspectos superiores y (B) inferiores de la colonia, (C) vista amplificada de las hifas aéreas (como se ve en un microscopio estereoscópico). Micromorfología de las hifas (D) sin tinción y teñidas con (E) LPCB y (F) TBO. Abreviaturas: c = conidióforo, p = phialide, s = tabique. Barras de escala: A,B = 2 cm; C = 2 mm; D-F = 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
En la Figura 6 se muestra una colonia aislada de las raíces fusiformes de la orquídea micoheterótrofa Wullschlaegelia aphylla, aplicando la metodología descrita. La colonia es de color blanquecino a gris en la parte superior (Figura 6A) y pardusca en la cara inferior (Figura 6B). Es opaco, sin pigmentos difusibles en el medio ni exudados. Los micelios son aéreos y abundantes, los márgenes son irregulares y aéreos, y la colonia tiene una textura aterciopelada y una topografía arrugada (Figura 6A). Las estructuras macroscópicas están ausentes.
El método de cultivo de portaobjetos es tradicional y ventajoso, y aunque requiere mucho tiempo, se puede aplicar para producir portaobjetos permanentes que puedan examinarse posteriormente. Los diferentes colorantes aquí presentados pueden ser utilizados para evidenciar estructuras importantes, y requieren pruebas en las muestras, para determinar el tiempo de incubación más adecuado. El rojo Congo y el TBO son tintes de uso general. El rojo Congo es adecuado para demostrar algunas estructuras delicadas (lectura adicional: Malloch22). El TBO tiñe el contenido celular con mayor intensidad que la pared celular del hongo (Figura 6F). A pesar de ser un colorante habitual para estructuras vegetales y no tan común en microscopía fúngica, el TBO tiene una importante propiedad metacromática33, que favorece otras aplicaciones, como el análisis fúngico en tejidos vegetales10. El LPCB es un colorante ampliamente aplicado en el análisis de hongos, aunque exige precaución debido al fenol. El azul de algodón (sinónimo: azul de metilo) es la mancha, el ácido láctico es un agente de limpieza y el fenol es un agente de muerte21. LPCB tiene afinidad por la quitina y evidencia paredes de esporas y ornamentaciones23,24. Los tabiques fúngicos no se tiñen ni por LPCB (Figura 6E) ni por TBO (Figura 6F), lo que facilita la identificación de dichas estructuras. La aplicación de tinciones es ventajosa para evidenciar estructuras que no se ven fácilmente cuando las hifas no están teñidas (Figura 6D).
Para la extracción de ADN de muestras de hongos y raíces, se debe respetar la cantidad de muestra molida en nitrógeno líquido que se agregará al tampón de extracción, sin exceder la cantidad indicada en el protocolo. Cabe destacar que una gran cantidad de muestra procesada no representa simplemente la obtención de una alta concentración de ADN sin mayores inconvenientes, ya que puede comprometer considerablemente la calidad final del ADN. Esto ocurre cuando se saturan las siguientes etapas de purificación del ADN. Además, la concentración de compuestos putativos producidos por plantas u hongos (por ejemplo, pigmentos, metabolitos secundarios), presentes en la solución final con ADN, puede actuar para reducir la calidad del ADN y/o inhibir la reacción en cadena de la polimerasa (PCR). Otro problema que puede reducir significativamente la calidad del ADN es desechar el medio de cultivo por completo con micelio fúngico y molerlo, lo que debe evitarse encarecidamente. Algunos reactivos del procedimiento de extracción (p. ej., fenol, alcohol etílico, SDS) y otras sustancias (p. ej., pigmentos fúngicos) pueden ser inhibidores que interfieren con la PCR.
En las etapas de limpieza del ADN, cuando se utiliza fenol y cloroformo, el sobrenadante siempre debe representar la fase con menos impurezas, comprendiendo normalmente la fase más clara. Durante tal etapa, la fase inferior no debe recuperarse al aspirar el sobrenadante que se transferirá a nuevos tubos. Al final del protocolo, el gránulo (constituido por ADN) debe presentar un color translúcido (muy deseable) a blanquecino. En la etapa de secado, proceder al secado del ADN en un termobloque es fundamental para evaporar completamente el etanol. Después de 12 h a 4 °C, el pellet debería haber eluido completamente en la solución acuosa añadida a los tubos, no siendo visible después de eso. En caso de que la elución no sea completa, y la concentración de ADN esté muy por debajo de la concentración ideal, es posible mantener la solución refrigerada durante 12 h más, sin pérdidas significativas en su calidad.
Los análisis de ADN en el software generan una tabla, como se representa en la Tabla 2, donde se indican los datos relacionados con la cantidad y calidad del ADN. La cuantificación del ADN viene dada por la concentración de ácidos nucleicos en nanogramos por microlitro de una muestra. Teniendo en cuenta la identificación molecular de hongos aislados, las muestras de ADN deben tener una concentración aproximada de 200 ng/μL, ideal para las etapas de PCR. En el caso de muestras con concentraciones más altas de ácidos nucleicos, se debe diluir una alícuota de la muestra, aproximando la concentración al valor mencionado anteriormente. Las muestras con concentraciones excesivamente altas, como las muestras 1 y 2 de la Tabla 2, pueden sugerir una detección falsa debido a contaminantes en la solución. Mientras tanto, las muestras con concentraciones de ácidos nucleicos más bajas, como la número 7 (Tabla 2), aún pueden ser utilizadas para PCR, siendo importante aplicar un mayor volumen de muestra en las reacciones. En cuanto a los parámetros de calidad, es satisfactorio que los cocientes 260/280 y 260/230 se encuentren entre 1,8 y 2,2, como se indica en las muestras 3, 5 y 8 (Tabla 2). Las muestras que presenten dichos valores (estando muy por debajo o por encima) deben ser sometidas a nuevas extracciones de ADN, como se indica en las muestras 9 y 10 de la Tabla 2. En los casos de muestras con una cantidad adecuada de ADN y que superen mínimamente el rango de calidad ideal, como sugieren los valores de 260/280 y 260/230 en los números 4 y 6 (Tabla 2), la dilución es el procedimiento más beneficioso para hacer que la concentración de ácidos nucleicos sea adecuada para la PCR y diluir posibles inhibidores, como los reactivos vestigiales del procedimiento de extracción. en las muestras.
| Muestra | Ácido nucleico Conc. | Unidad | A260 | A280 | 260/280 | 260/230 | Tipo de muestra |
| 1 | 14491.7 | ng/μL | 289.833 | 141.175 | 2.05 | 1.8 | ADN |
| 2 | 13359.3 | ng/μL | 267.187 | 124.607 | 2.14 | 2.15 | ADN |
| 3 | 1137.6 | ng/μL | 22.751 | 10.574 | 2.15 | 2.13 | ADN |
| 4 | 1472.6 | ng/μL | 29.452 | 13.287 | 2.22 | 2.16 | ADN |
| 5 | 3464.8 | ng/μL | 69.295 | 33.329 | 2.08 | 1.88 | ADN |
| 6 | 1884.2 | ng/μL | 37.684 | 17.912 | 2.1 | 1.78 | ADN |
| 7 | 187.6 | ng/μL | 3.751 | 1.834 | 2.05 | 2.06 | ADN |
| 8 | 1580.3 | ng/μL | 31.607 | 15.281 | 2.07 | 1.98 | ADN |
| 9 | 923.3 | ng/μL | 18.466 | 9.196 | 2.01 | 1.37 | ADN |
| 10 | 2414.4 | ng/μL | 48.287 | 21.008 | 2.3 | 3.45 | ADN |
Tabla 2: Resultados generados en el software a partir de muestras de ADN fúngico analizadas en un espectrofotómetro.
A diferencia de las muestras de ADN de hongos endófitos aislados, la concentración de ADN en las muestras para análisis de metabarcoding debe presentar cantidades masivas de moléculas de ADN extraídas, con alta calidad y peso molecular. Las muestras deben ser evaluadas por electroforesis en gel de agarosa, obteniendo la máxima integridad posible, con poca o ninguna mancha en el gel.
Discusión
La desinfestación superficial de muestras de plantas es una de las etapas más críticas en el protocolo presentado. Es muy deseable que no se contaminen las antenas PDA con gotas del último lavado. Las bacterias se observan con frecuencia como contaminantes en las placas de aislamiento, generalmente más que los hongos esporulantes en el aire, considerando que las bacterias endófitas también son comunes dentro de los tejidos vegetales 3,11. Por lo tanto, la adición de antibióticos en el medio de cultivo al instalar los fragmentos de órgano es esencial. Se obtienen mejores resultados cuando se combinan diferentes tipos de antibióticos, lo que da como resultado un espectro de acción más amplio. Otra consideración importante es el sesgo intrínseco en el aislamiento de los hongos, ya que el uso de PDA y AA inevitablemente seleccionará ciertas especies y desfavorecerá a otras. La combinación de otros medios de cultivo puede minimizar el sesgo, aunque no eliminar tales limitaciones9.
Generalmente, los hongos que producen esporas mientras crecen en placas de Petri presentan mayores tasas de supervivencia en los protocolos de conservación, ya sea a temperaturas más bajas o no, y los aislados no esporulantes pueden no tolerar la preservación de Castellani o aceite mineral34,35,36, por lo que un método de criopreservación puede ser crucial en tales casos. Además, algunos aislados pueden ser sensibles a la congelación, y la adición de crioprotectores ayuda a la preservación, como en el método presentado con vermiculita18. Las características de la colonia y las hifas pueden ser útiles para agrupar los aislados con rasgos similares, facilitando la selección para su uso en tratamientos de germinación simbiótica (como detallan Pena-Passos et al.10). Estas características también contribuyen a la elección de los métodos de conservación a aplicar, especialmente teniendo en cuenta la esporulación o su ausencia.
La caracterización morfológica de los aislados también mejora y complementa las descripciones morfológicas disponibles en la literatura especializada, especialmente cuando se asocian con la caracterización molecular. A pesar de ser un desafío con numerosos aislamientos, la caracterización morfológica de los hongos lleva mucho tiempo y depende de los datos publicados. Aunque solo se planea realizar la caracterización molecular, es recomendable mantener un registro fotográfico de la apariencia de la colonia (macromorfología) y publicarlo siempre que sea posible, para contribuir a futuros procedimientos de identificación. También es importante mantener fotos de las placas donde se instalan los fragmentos, para permitir la comparación entre los aislamientos finales obtenidos y los morfotipos observados creciendo en las placas de instalación. En tal caso, los hongos comparados deben cultivarse en el mismo tipo de medio. En Currah et al.16 se encuentran metodologías adicionales para evaluar hongos micorrícicos comunes de orquídeas, como el cultivo en medio de ácido tánico (TAM) para diferenciar los géneros Epulorhiza (teleomorfo: Tulasnella) y Ceratorhiza (teleomorfo: Ceratobasidium), y el cultivo en CMA para estimular células monilioides para la caracterización de hongos del complejo Rhizoctonia .
Teniendo en cuenta los métodos detallados para observar hifas fúngicas bajo un microscopio óptico, el montaje de carda y el montaje de cinta adhesiva son más rápidos que el cultivo de portaobjetos, aunque el método de montura de carda no es adecuado para analizar esporas y medir los ángulos de las ramas. El montaje con cinta adhesiva conserva la organización del micelio mejor que la técnica de montaje de carda, aunque el uso de cinta adhesiva no es tan fiable como el cultivo de portaobjetos para medir los ángulos de las ramas (observación de los autores). El cultivo de portaobjetos, a pesar de llevar mucho tiempo, es la técnica más adecuada para producir portaobjetos semipermanentes y permanentes. La macro y micromorfología se analizan teniendo en cuenta la literatura. Webster y Weber32 es una extensa fuente general de información y referencias adicionales. Currah et al.16 y Zettler y Corey37 proporcionan información útil sobre los hongos micorrícicos de las orquídeas. A pesar de describir hongos de importancia médica, Walsh et al.21 y McGinnis31 también son útiles sobre las características generales de las colonias de hongos filamentosos, información visual/descriptiva y referencias adicionales para la micromorfología fúngica.
De acuerdo con Yu et al.38, los análisis de cuantificación de ADN en un espectrofotómetro presentan una gran precisión y exactitud, siendo consistentes con los análisis cuantitativos de PCR en tiempo real. Una observación importante es que algunos aislados pueden no proporcionar muestras de ADN adecuadamente puras para las siguientes etapas de identificación molecular, ya que los endófitos fúngicos de los micoheterótrofos son extremadamente diversos, especialmente los asociados con plantas tropicales39, al igual que los metabolitos producidos. Los análisis de espectrofotómetro pueden utilizarse para evaluar estos casos y diferentes protocolos, o se pueden aplicar kits comerciales para la purificación del ADN de las muestras. Las etapas posteriores sugeridas en la identificación molecular de aislados son la amplificación de la región del espaciador transcrito interno (ITS) y la secuenciación. La región ITS es un ADN espaciador de ARN ribosómico ampliamente empleado en la literatura especializada y formalmente aceptado como el principal código de barras para la identificación de hongos 9,40. Puede amplificarse por PCR con los cebadores ITS1 e ITS441, y luego secuenciarse en la plataforma Sanger42.
Los análisis de metabarcódigos, además de permitir la investigación de la riqueza, abundancia y composición taxonómica de microorganismos en muestras ambientales y de plantas, pueden proporcionar resultados de interacciones positivas o negativas entre estos microorganismos12. La etapa de maceración con nitrógeno líquido añadido al protocolo estándar DNeasy PowerSoil tiene como objetivo romper el mayor número posible de células fúngicas en relación con los tejidos vegetales, lo que permite un mejor muestreo de la comunidad fúngica en el tejido macerado11. Las opciones de cebadores para secuencias fúngicas (códigos de barras) en la identificación molecular deben tener en cuenta la interferencia con el ADN de la planta. Aconsejamos el uso de cebadores que amplifiquen regiones variables del gen fúngico 18S, por ejemplo, ITS1-5,8S-ITS243. Tras la obtención de las muestras de ADN, los siguientes pasos son la preparación de las bibliotecas metagenómicas, que dependen de la plataforma de secuenciación elegida, la posterior secuenciación de las librerías y el análisis de los datos obtenidos mediante herramientas bioinformáticas. Recomendamos utilizar la plataforma Illumina MiSeq, que representa una opción menos costosa y de alto rendimiento, generando secuencias de 250 pb en cortos períodos de tiempo11,44.
Divulgaciones
Los autores no tienen nada que revelar ni ningún conflicto de intereses.
Agradecimientos
Agradecemos el financiamiento de la FAPESP (2015/26479-6) y del CNPq (447453/2014-9). JLSM agradece al CNPq por las becas de productividad (303664/2020-7). El MPP agradece a la Capes (beca de maestría, proceso 88887.600591/2021-00) y al CNPq.
Materiales
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Referencias
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , Springer-Verlag. Berlin. 1-13 (2006).
- Smith, S. E., Read, D. J. Mycorrhizal Symbiosis. , Elsevier Science Academic Press. London. (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777(2022).
- Araújo, W. L., et al. Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, Santarém: UFOPA. 257(2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774(2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589(2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069(2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. Larone's Medically Important Fungi: A Guide to Identification. , ASM Press. (2018).
- Malloch, D. Moulds: Isolation, Cultivation, Identification. , Available from: http://website.nbm-mnb.ca/mycologywebpages/Moulds/Moulds.html (1997).
- Smith, P. Microscopy: Chemical Reagents. British Mycological Society. , Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022).
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , Santarém: UFOPA. 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. Methuen Handbook of Colour. Methuen handbook of colour. , Methuen & Co. Ltd. London. (1967).
- Ridgway, R. Color Standards and Color Nomenclature. , Washington, DC. Published by the author (1912).
- McGinnis, M. R. Laboratory Handbook of Medical Mycology. , Elsevier Science. (2012).
- Webster, J., Weber, R. Introduction to Fungi. , Cambridge University Press. Cambridge, UK. (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados