Method Article
Preparação da Amostra e Quantitação Relativa usando Metilação Redutiva de Aminas para Estudos de Peptidômica
Neste Artigo
Resumo
Este artigo descreve um método de preparação amostral baseado na inativação de calor para preservar peptídeos endógenos evitando a degradação pós-morte, seguido de quantitação relativa usando rotulagem isotópica mais LC-MS.
Resumo
A peptidomia pode ser definida como a análise qualitativa e quantitativa dos peptídeos em uma amostra biológica. Suas principais aplicações incluem identificar os biomarcadores de peptídeos de doença ou estresse ambiental, identificar neuropeptídeos, hormônios e peptídeos intracelulares bioativos, descobrir peptídeos antimicrobianos e nutracêuticos a partir de hidrólises proteicos, e pode ser usado em estudos para entender os processos proteolíticos. O recente avanço na preparação da amostra, métodos de separação, técnicas de espectrometria de massa e ferramentas computacionais relacionadas ao sequenciamento de proteínas contribuíram para o aumento do número de peptídeos identificados e peptidomes caracterizados. Estudos peptidômicos frequentemente analisam peptídeos que são gerados naturalmente nas células. Aqui, é descrito um protocolo de preparação de amostras baseado na inativação do calor, que elimina a atividade de protease, e extração com condições leves, portanto não há laços de peptídeos. Além disso, também é mostrada a quantitação relativa de peptídeos usando rotulagem estável de isótopos por metilação redutiva de aminas. Este método de rotulagem tem algumas vantagens, pois os reagentes estão disponíveis comercialmente, baratos em comparação com outros, quimicamente estáveis, e permite a análise de até cinco amostras em uma única corrida LC-MS.
Introdução
As ciências "omics" são caracterizadas pela análise profunda de um conjunto de moléculas, como DNA, RNA, proteínas, peptídeos, metabólitos, etc. Estes conjuntos de dados gerados em larga escala (genômica, transcriômica, proteômica, peptidômica, metabolômica, etc.) revolucionaram a biologia e levaram a uma compreensão avançada dos processos biológicos1. O termo peptidomia começou a ser introduzido no início do século XX, e alguns autores se referem a ele como um ramo da proteômica2. No entanto, a peptidômica possui particularidades distintas, onde o principal interesse é investigar o conteúdo de peptídeos gerados naturalmente durante os processos celulares, bem como a caracterização da atividade biológica dessas moléculas3,4.
Inicialmente, os estudos de peptídeos bioativos eram restritos aos neuropeptídeos e peptídeos hormonais através da degradação de Edman e radioimunessay. No entanto, essas técnicas não permitem uma análise global, dependendo do isolamento de cada peptídeo em altas concentrações, tempo para a geração de anticorpos, além da possibilidade de reatividade cruzada5.
A análise da peptidomia só foi possível após vários avanços na cromatografia líquida acoplado à espectrometria de massa (LC-MS) e projetos de genoma que forneceram pools de dados abrangentes para estudos proteômicos/peptidóticos6,7. Além disso, um protocolo específico de extração de peptídeos para peptidomes precisava ser estabelecido porque os primeiros estudos que analisaram neuropeptídeos globalmente em amostras cerebrais mostraram que a detecção foi afetada pela degradação maciça de proteínas, que ocorrem principalmente neste tecido após 1 min após a morte. A presença desses fragmentos de peptídeo mascarava o sinal de neuropeptídeo e não representava o peptidome in vivo. Esse problema foi resolvido principalmente com a aplicação de inativação de aquecimento rápido de proteases usando irradiação de micro-ondas, o que reduziu drasticamente a presença desses fragmentos de artefato e permitiu não apenas a identificação de fragmentos de neuropeptídeos, mas revelou a presença de um conjunto de peptídeos de proteínas citostómicas, mitocondriais e nucleares, diferentes de degradação6,8,9.
Esses procedimentos metodológicos permitiram uma expansão do peptidome para além dos conhecidos neuropeptídeos, onde centenas de peptídeos intracelulares gerados principalmente pela ação de proteasomes foram identificados em leveduras10, zebrafish11, tecidos de roedores12 e células humanas13. Dezenas desses peptídeos intracelulares têm sido extensivamente mostrados com atividades biológicas e farmacológicas14,15. Além disso, esses peptídeos podem ser usados como biomarcadores de doenças e possivelmente ter significância clínica, como demonstrado no fluido cefalorraquidiano de pacientes com aneurisma saccular intracraniano16.
Atualmente, além da identificação de sequências de peptídeos, é possível através da espectrometria de massa obter dados de quantitação absoluta e relativa. Na quantitação absoluta, os níveis de peptídeos em uma amostra biológica são comparados aos padrões sintéticos, enquanto na quantitação relativa, os níveis de peptídeos são comparados entre duas ou mais amostras17. A quantitação relativa pode ser realizada utilizando as seguintes abordagens: 1) "livre de rótulos"18; 2) rotulagem metabólica in vivo ou 3) rotulagem química. Os dois últimos baseiam-se no uso de formas isotópicas estáveis incorporadas em peptídeos19,20. Na análise sem rótulos, os níveis de peptídeos são estimados considerando a força do sinal (contagem espectral) durante o LC-MS18. No entanto, a rotulagem isotópica pode obter níveis relativos mais precisos de peptídeos.
Muitos estudos peptidômicos utilizaram butiratrato de trimetilamônio (TMAB) rotulando reagentes como rotulagem química, e mais recentemente, a metilação redutiva de Aminas (RMA) com formas deutadas e não deuteradas de reagentes de formaldeído e cianoboroiddeto de sódio foram usados11,21,22. No entanto, os rótulos TMAB não estão disponíveis comercialmente, e o processo de síntese é muito trabalhoso. Por outro lado, na RMA, os reagentes estão disponíveis comercialmente, baratos em comparação com outros rótulos, o procedimento é simples de realizar, e os peptídeos rotulados são estáveis23,24.
O uso de RMA envolve a formação de uma base de Schiff permitindo que os peptídeos reajam com formaldeído, seguido de uma reação de redução através do cianoboroidido. Esta reação causa dimetilação de grupos de amino livres em terminais N e cadeias laterais de lise e monometilados N-terminais prolines. Como os resíduos de prolina são muitas vezes raros no terminal N, praticamente todos os peptídeos com aminas gratuitas no n-terminus são rotulados com dois grupos de metila23,24,25.
Protocolo
O procedimento a seguir para extração de peptídeos e metilação redutiva foi adaptado de procedimentos publicados anteriormente24,25,26,27. Esse protocolo seguiu as diretrizes do Conselho Nacional de Controle de Experimentação Animal (CONCEA) e foi aprovado pela Comissão de Ética para Uso de Animais (CEUA) do Instituto de Biociências da Universidade estadual de São Paulo. As etapas do protocolo são mostradas na Figura 1.
NOTA: Prepare todas as soluções aquosas em água ultrauso.
1. Extração de peptídeo
- Cultura celular
- Cultivar células SHSY5Y em um prato de 15 cm a 37 °C abaixo de 5% de CO2 no meio modificado da Águia de Dulbecco contendo 15% de soro bovino fetal e 1% penicilina-estreptomicina.
- Use 2-3 placas para cada amostra. Aumentar as células para 100% confluência.
- Após o tratamento pretendido, lave as células duas vezes com soro fisiológico tamponado por fosfato. Em seguida, adicione 10 mL de soro fisiológico tamponado com fosfato, raspe as células e colete em um tubo de 15 mL.
- Centrifugar a 800 x g por 5 min e remover o supernatante. Resuspenda a pelota em 1 mL de água deionizada ultra-purificada a 80 °C.
- Transfira o conteúdo do tubo (lisecelular) para um tubo de microfuça de 2 mL.
- Tecidos animais (principalmente tecido nervoso):
- Anestesiar o adulto macho danio rerio (zebrafish) com uma dose letal de MS 222 (100 mg/L) e submetê-lo imediatamente a 8 s de radiação de micro-ondas para inativar peptidase e protease.
NOTA: Pode-se usar um forno de micro-ondas do tipo doméstico. Um micro-ondas de 900 W foi usado para 8 a 10 s em potência total. O micro-ondas utilizado deve ser capaz de elevar a temperatura cerebral para > 80 °C dentro de 10 s. A reprodutibilidade no aquecimento entre as amostras também seria beneficiada colocando o tecido no mesmo local no micro-ondas. - Após a inativação do calor, colete todo o cérebro em um tubo de microfuça de 2 mL e congele a −80 °C até a análise.
- Resuspenda a amostra de tecido em 1 mL de água deionizada ultra-purificada a 80 °C. Sonicar o tecido com uma sonda usando 30 pulsos (4 Hz) de 1 s.
NOTAs: Para tecidos hepáticos e renais, use um homogeneizador mecânico a 10.000-30.000 rpm para 20 s. Para tecidos musculares, triture o tecido em nitrogênio líquido usando um cadinho de porcelana e pilão. As etapas a seguir são as mesmas para o liseto celular ou o tecido homogeneizado.
- Anestesiar o adulto macho danio rerio (zebrafish) com uma dose letal de MS 222 (100 mg/L) e submetê-lo imediatamente a 8 s de radiação de micro-ondas para inativar peptidase e protease.
- Incubar o tecido de lise celular ou homogeneizar a 80 °C por 20 min. Em seguida, esfrie no gelo por 10-30 min.
- Adicione 10 μL de solução de estoque de 1 M HCl para cada 1 mL de volume amostral para obter uma concentração final de 10 mM. Misture por vórtice por 20 s e incuba ainda mais no gelo por 15 minutos.
NOTA: Antes de acidificar, certifique-se de que a amostra esteja completamente resfriada para evitar quebrar as ligações de peptídeos causadas pela acidificação a temperaturas elevadas. - Centrifugar o lisecelular ou o tecido homogeneado a 12.000 x g a 4 °C por 15 min. Colete o supernatante em tubos de microcentrifuuge de proteína de baixa ligação e armazene-o a -80 °C.
- Limpe os dispositivos de ultrafiltração (filtros de corte de 10 kDa) adicionando água e centrífuga a 2300 x g por 3 min. Repita este passo mais duas vezes.
- Coloque o supernatante nos filtros de corte pré-lavados de 10 kDa e centrífuga a 2.300 x g a 4 °C por 50 min em uma centrífuga rereto. O fluxo representa o extrato de peptídeo.
- Desalte as amostras em colunas de limpeza de fase invertida de acordo com as instruções do fabricante usando soluções de acetonitrilo (ACN) e ácido trifluoroacético (TFA), conforme descrito abaixo:
- Equilibre a coluna com 1 mL de 100% ACN.
- Lave a coluna com solução de 1 mL de 5% ACN com 0,1% de TFA.
- Carregue o volume completo da amostra na coluna.
- Lave a coluna com solução de 1 mL de 5% DE ACN com 0,1% de TFA
- Elute os peptídeos da coluna com uma solução de 1,8 mL de 100% ACN com 0,15% TFA em tubos de microcentrifuge de baixa ligação de proteína.
- Seque a amostra completamente em uma centrífuga de vácuo. Defina o método de concentração para solventes orgânicos e temperatura a 30 °C. O tempo de concentração é monitorado em exibição.
- Armazene as amostras a −80°C até o próximo passo.
2. Quantificação de peptídeo com fluorescamina
NOTA: A quantidade de peptídeo pode ser estimada usando fluorescamina no pH 6.8 como descrito anteriormente11,28. Este método consiste na fixação de uma molécula de fluorescamina às aminas primárias presentes nos resíduos de lise (K) e/ou no terminal N de peptídeos. A reação é realizada no pH 6.8 para garantir que a fluorescamina reaja apenas com os grupos amino dos peptídeos e não com aminoácidos livres. A fluorescamina é medida usando um espectrofluorômetro em um comprimento de onda de excitação de 370 nm e um comprimento de onda de emissão de 480 nm.
- Prepare diferentes concentrações do peptídeo padrão (0,05, 0.1, 0.15, 0.2, 0.3, 0.5 e 0,7 μg/μL) e armazene as alíquotas a -20 °C.
NOTA: O peptídeo 5A (LTLRTKL) é sugerido, uma vez que possui uma composição e concentração conhecidas. - Prepare a solução de estoque de fluorescamina (0,3 mg/mL) em acetona. Alíquotar rapidamente em tubos de microcentrifuuge (1 mL), vedação usando parafilme e armazenar a -20 °C no escuro.
- Prepare 0,2 M Tampão fosfato (PB) em pH 6.8.
NOTA: Prepare 0,2 M PB adicionando 0,1 M tampão de fosfato pH 6.8 (26,85 mL de Na2HPO3 1M) e 0,1 M tampão fosfato pH 6.8 (23,15 mL de NaH2PO3 1M) a 250 mL de água - Resuspende as amostras de peptídeos em 100-200 μL de água ultra-purificada.
- Pipeta 2,5 μL das concentrações de peptídeos padrão e amostras na placa branca de 96 poços para ensaios de fluorescência em triplicado. Adicione 25 μL de tampão fosfato de 0,2 M.
- Adicione 12,5 μL de fluorescamina com uma pipeta multicanal. Homogeneize suavemente por 1 min no agitador rotador orbital.
- Em seguida, adicione 110 μL de água com uma pipeta multicanal para parar a reação.
NOTA: Transfira a solução de estoque de fluorescamina e a água ultrauso para dois reservatórios para pipetar essas soluções com uma pipeta multicanal. - Ajuste os seguintes parâmetros de leitura no espectrofluorômetro: Leia as amostras do topo, comprimento de onda de excitação a 370 nm e comprimento de onda de emissão a 480 nm.
- Leia a placa no espectrômetro.
3. Metilação redutiva da rotulagem de aminas
NOTA: Este método de rotulagem isotópica baseia-se na dimetilação de grupos de amina com formas deutadas e não deutadas de reagentes de formaldeído e cianoboroidride de sódio. O produto final desta reação adiciona 28 Da, 30 Da, 32 Da, 34 Da ou 36 Da à massa final de cada peptídeo em cada local de rotulagem disponível (lysine ou N-terminal). Esta reação produz uma diferença m/z nos peptídeos rotulados com diferentes formas observadas no espectro MS (Tabela 1).
ATENÇÃO: Os equipamentos de segurança adequados devem ser usados para manusear esses compostos, e devem ser tomados cuidados para minimizar a exposição. Procedimentos com reagentes de formaldeído e cianoboroidride de sódio devem ser realizados em um capô de fumaça porque são muito tóxicos (incluindo a pesagem do cianoboroidride de sódio). Durante a reação de sacieda e acidificação, um gás tóxico (cianeto de hidrogênio) pode ser gerado.
- Prepare as seguintes soluções frescas do estoque ou reagentes no dia do procedimento em água ultrauso:
- Diluir o estoque de 37% de Formaldeído (CH2O) para 4%.
- Diluir o estoque de 20% de Deuterado de Formaldeído (CD2O) para 4%.
- Diluir o estoque de 20 % Deuterated C13 Formaldeído (13CD2O) para 4%.
- Prepare 0,6 m NaBH3CN.
- Prepare-se 0,6 m naBd3Cn.
- Prepare uma solução de 1% de bicarbonato de amônio.
- Prepare uma solução 5% de ácido fórmico.
NOTA: Quanto à quantitação relativa dos peptídeos, uma vez que diferentes esquemas experimentais podem ser realizados dependendo do número de rótulos utilizados, é necessário atenção durante o procedimento de rotulagem química. Recomenda-se separar pequenas alíquotas dos rótulos em racks separados com as respectivas amostras a serem rotuladas para reduzir o potencial de erro humano na adição do reagente errado aos tubos de amostra.
- Prepare cada amostra contendo até 25 μg do peptídeo. As amostras não devem conter Tris ou Amônio Bicarbonato.
NOTA: As quantidades descritas abaixo são suficientes para cada amostra em um volume de 100 μL.
Prossiga com as etapas 3.3-3.8 em um capô de fumaça. - Adicione 1/10º volume de 1 M TEAB às amostras (concentração final da solução 100 mM de TEAB). Verifique o pH com um papel indicador de pH; deve ser entre 5-8. Ajuste com HCl ou NaOH, se necessário.
- Adicione 4 μL de Formaldeído deuterado ou Formaldeído deuterado C13 de acordo com o esquema de rotulagem estabelecido. Misture por 5 s por vórtice.
- Adicione 4 μL de NaBH3CN (0,6 M) ou NaBD3CN (0,6 M) de acordo com o esquema de rotulagem estabelecido. Misture por 5 s por vórtice.
- Incubar em um capô de fumaça por 2 h à temperatura ambiente, misturando a cada 30 minutos.
- Repita as etapas 3.4 e 3.5. Incubar as amostras em um capô de fumaça durante a noite à temperatura ambiente.
- Adicione 16 μL de bicarbonato de amônio (1%) e misture por vórtice. Coloque a amostra no gelo, adicione 8 μL de ácido fórmico (5%), e misture por vórtice para 5 s.
- Combine as amostras, ajuste o pH para 2-4 e desalva as amostras combinadas em colunas de limpeza de fase invertida, conforme descrito anteriormente na etapa 1.8.
- Seque a amostra completamente em uma centrífuga de vácuo. Defina o método de concentração para solventes orgânicos e temperatura a 30 °C. O tempo de concentração é monitorado em exibição.
- Armazene as amostras a -20 °C.
4. Cromatografia líquida e espectrometria de massa
- Execute a análise LC-MS usando um sistema nanoHPLC acoplado a um instrumento MS compatível com etiquetas de metila.
NOTA: Uma variedade de instrumentos ms são compatíveis com a rotulagem RMA. Um sistema nanoHPLC acoplado ao Orbitrap é normalmente usado para realizar análises de LC-MS através de uma fonte de íon nanoeletríspray. Primeiro, a amostra é carregada em um pré-lombar e peptídeos separados em uma coluna analítica. A elução de peptídeos é realizada utilizando um gradiente linear de 5%-45% de acetonitrilo, em ácido fórmico de 0,1%, durante 90 min, com um fluxo de 200 nL/min. O espectrômetro de massa é definido para funcionar no modo dependente de dados. Cada varredura completa é adquirida a uma intensidade de 10-30 eV, 2,3 Kv, e então os dez picos mais altos são selecionados para a dissociação induzida por colisão (CID). O tempo de injeção é definido na armadilha de íons a 100 ms, e a injeção Fourier Transform (FT)-MS é fixada com uma resolução de 1000 ms 30.000 a m/z 300-1800. Um mínimo de 5000 contagens e uma exclusão dinâmica de 70 s é usado para realizar a varredura de fragmentação.
5. Quantitação relativa de peptídeos
NOTA: Os espectros ms são analisados no software do espectrômetro de massa. Grupos de pico de peptídeos rotulados com diferentes tags são identificados no espectro de MS. A quantitação relativa é calculada pela intensidade de cada pico monoisotópico. Cada grupo tratado é comparado ao respectivo grupo controle.
- Clique duplo direito no arquivo de amostra bruta para abrir o software de análise de espectro. Carregue os cromatogramas de tempo de retenção (RT) e MS (EM) em duas guias, superior e inferior, respectivamente.
- Clique com o botão direito de clicar uma vez sequencialmente nos ícones de opções Display e Mass na barra de ferramentas do software e definir a precisão de massa para quatro decimais.
- Posicione o cursor do mouse em qualquer lugar da guia RT. Procure o tempo de retenção do íon correspondente a ser analisado e clique no botão direito do mouse. O espectro MS do tempo selecionado será mostrado automaticamente na guia EM.
- Posicione o cursor do mouse em qualquer lugar da guia EM. Procure os íons a serem analisados.
- Clique direito e segure-se em uma região adjacente à esquerda perto desses íons. Em seguida, arraste o mouse para a direita na faixa desejada para ampliar a região de interesse.
- Mantenha o mouse posicionado na guia EM e clique nas setas do teclado direito ou esquerdo para definir a faixa de íons a serem analisados.
- Posicione o cursor do mouse novamente no início do intervalo de tempo desejado.
- Clique com o botão direito do mouse e arraste o mouse até o valor da hora escolhida. Deixe o botão. A intensidade acumulada dos íons será mostrada automaticamente na guia EM.
- Colete os dados de intensidade de íons, m/z e íons em uma planilha.
NOTA: A massa monoisotópica de cada peptídeo sem adição de grupos de metila é calculada a partir da seguinte fórmula:
Peptídeo não modificado em massa = (m/z a x z) - (C a x T) - (1.008 x z)
m/zais a massa observada para cobrar valor para o pico monoisotópico para cada peptídeo rotulado com diferentes combinações de tags (a =1, 2, 3, 4 ou 5, correspondente ao número da amostra).
z é estado de carga.
Ca é a massa monoisotópica de um par de grupos de metila:
Para a=1, Ca = 28,0313 (a adição líquida de dois grupos CH3 à amina primária)
Para a=2, Ca = 30,0439 para dois grupos CHD2
Para a=3, Ca = 32,0564 para dois grupos CD2H
Para a=4, Ca= 34,0690 para dois grupos CD3
Para a=5, Ca = 36,0757 para dois 13 grupos CD3
T é o número de pares de grupos de metila incorporados no peptídeo. Isso pode ser calculado a partir da seguinte fórmula quando cinco tags são usadas: T=z*(m/z5 - m/z1)/8. Para peptídeos que contêm uma única amina primária e, portanto, são rotulados com apenas dois grupos de metila apresentam sobreposições de pico no espectro de MS quando são usadas etiquetas adjacentes. O pico de intensidade de cada peptídeo rotulado pode ser corrigido usando as equações descritas por Tashima e Fricker25.
6. Identificação de Peptídeos
- Para identificar peptídeos, analise os dados de MS/MS usando um mecanismo de pesquisa de banco de dados29,30.
- Para calcular a taxa de descoberta falsa (FDR) usando o método de fusão de isca, pesquise um banco de dados de isca.
NOTA: Os parâmetros de pesquisa geralmente utilizados não são especificidade de enzimas; tolerância de massa precursora definida de 15-50 ppm; tolerância de massa de íons fragmentado de 0,5 Da; modificações variáveis: aminas reativas de resíduos de Lys e N-terminus dos rótulos metilados isotópicos de peptídeos (L1 (+28), L2 (+30), L3 (+30 32), L4 (+34) e L5 (+36)), metionina oxidada (+15,99 Da) e acetilação (+42,01 Da). - Em seguida, classifique os peptídeos pela sua média de confiança local para selecionar os melhores espectros para anotar e filtrar por FDR ≤5%.

Figura 1: Estudos peptidômicos do fluxo de trabalho. Etapas de extração de peptídeos e Metilação Redutiva de aminas. Clique aqui para ver uma versão maior desta figura.
Resultados
Os resultados obtidos a partir das corridas realizadas no espectrômetro de massa são armazenados em arquivos de dados brutos que podem ser abertos no software de espectrômetro de massa. Nos espectros de MS, é possível observar grupos de pico representando peptídeos rotulados de acordo com o esquema de rotulagem utilizado, variando de 2-5 rótulos. Por exemplo, na Figura 2, pares de picos detectados em tempo cromatográfico são representados em um experimento onde apenas dois rótulos isotópicos foram usados em duas amostras diferentes na mesma corrida. A Figura 3 mostra outras possibilidades de resultados positivos, utilizando 3 e 4 rótulos diferentes em cada execução de LC-MS. Ao usar 4 ou 5 etiquetas em uma execução LC/MS, pode haver uma sobreposição de picos dos peptídeos rotulados com as diferentes tags que precisam ser corrigidas para obter o valor real de intensidade de cada pico (Figura 4).
A rotulagem isotópica também pode ser usada para mostrar substratos e produtos in vitro para um determinado protease ou peptidase, como mostrado na Figura 5. Finalmente, diferentes softwares podem ser usados para identificar os peptídeos rotulados, como Peaks Studio ou MASCOT. Esses aplicativos de software foram criados para análise proteômica; portanto, os dados de quantitação proteica não devem ser considerados para análise de peptidomia, e cada peptídeo rotulado identificado dentro dos parâmetros de confiabilidade deve ser verificado e, em seguida, quantificado. Se os peptídeos tiverem sido detectados e rotulados com sucesso, esses programas fornecerão uma lista de sequências de peptídeos identificadas contendo os rótulos. A Figura 6 é mostrada como um exemplo da identificação de uma sequência de peptídeos feita pelo programa. Neste caso, apenas 3 formas diferentes dos rótulos (L1, L3 e L5) foram utilizadas para rotular três amostras diferentes separadamente, que foram então misturadas e analisadas por espectrometria de massa em uma única execução.
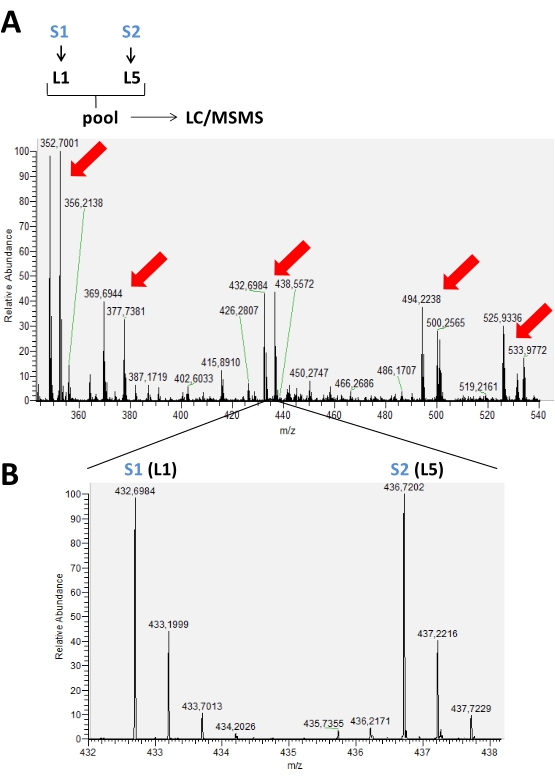
Figura 2: Espectro de MS representativo de um tempo cromatográfico acumulado em um experimento típico de rotulagem usando dimetilação redutiva de aminas. Em (A), as setas vermelhas indicam a presença de pares de picos de diferentes peptídeos rotulados com 2 formas isotópicas (L1 e L5) para comparação entre duas amostras diferentes (S1 e S2). Em (B), uma imagem ampliada de um espectro de MS do mesmo peptídeo mostrando m/z diferente por causa do uso de etiquetas. Neste caso, não houve variação na intensidade máxima para este peptídeo presente nessas amostras. Clique aqui para ver uma versão maior desta figura.

Figura 3: Foi utilizado espectro ms representativo de peptídeos rotulados com metilação redutiva de aminas utilizando diferentes números de etiquetas. (A) Foi utilizada uma rotulagem triplex. É possível observar um espectro ms de um peptídeo presente em 3 amostras diferentes (S1, S2 e S3) rotuladas com etiquetas L1, L3 e L5, respectivamente. Neste caso, o nível do peptídeo rotulado com L5 foi o dobro do nível observado para o mesmo peptídeo rotulado com etiquetas L1 e L3. (B) A rotulagem de quadripex foi realizada utilizando as etiquetas L1, L2, L3 e L4. Neste caso, as amostras de controle (S1 e S3) foram rotuladas com L1 e L3, respectivamente, e comparadas com duas amostras experimentais (S2 e S4) rotuladas com rótulos L2 e L4. Não foram observadas diferenças significativas para este peptídeo entre as amostras. Clique aqui para ver uma versão maior desta figura.

Figura 4: Espectro ms representativo de um peptídeo rotulado apresentando uma sobreposição de pico. Este número mostra o espectro ms de um peptídeo de carga 3, massa de 2098.87 Da com uma única amina primária disponível para rotulagem. A diferença entre os peptídeos rotulados é de apenas 2 Da, causando uma sobreposição quando 4 ou 5 rótulos são usados na mesma linha LC/MS. Usando um modelo baseado em equações polinomiais cúbicas, é possível corrigir essas sobreposições entre os rótulos. No gráfico, as barras vermelhas mostram a média dos valores de intensidade ajustados para este peptídeo em duas corridas. As barras pretas mostram a média dos valores de intensidade sobrepostos. Após a correção, este peptídeo mostrou pouca variação de intensidade entre as amostras (barras vermelhas). Clique aqui para ver uma versão maior desta figura.

Figura 5: Espectro de MS representativo de peptídeos rotulados com metilação redutiva de aminas em estudo de proteólise. Aqui, os extratos de peptídeo foram incubados com neurolises de 200 nM e 20 nM para caracterizar seus substratos e produtos. Para a confirmação do resultado, foram realizadas duas corridas de LC/MS com estratégias de rotulagem Forward e Reverse. No segundo passo, a posição das amostras é alterada no procedimento de rotulagem em relação ao esquema utilizado no primeiro run. Em A, é mostrado um peptídeo que não muda (NC) na presença da enzima neurolise. Em B, um peptídeo que desapareceu na presença de uma alta concentração da enzima (S2) e que apresentou uma pequena redução na baixa concentração da enzima (S4) tanto na rotulagem para a frente quanto na rotulagem inversa. Este peptídeo é considerado um substrato (SB) da enzima. Em C um exemplo de peptídeo que foi considerado um produto (DP), pois sua concentração foi aumentada (S2 e S4) na presença da enzima. Clique aqui para ver uma versão maior desta figura.

Figura 6: Espectro msms representativo e identificação de um peptídeo rotulado realizado por um mecanismo de pesquisa de banco de dados. Neste exemplo, é possível observar 2+ íons com m/z 672.3802, 676.4045 e 680.4241 correspondentes a um peptídeo da mesma massa rotulado com as formas metiladas L1, L3 e L5, respectivamente. Esta sequência ADQVSASLAKQGL foi identificada através do espectro MS/MS como um fragmento da proteína tau isoform X1 associada a microtúbulos. Este peptídeo tem o terminal N e uma lise disponível como sites de rotulagem adicionando uma diferença de massa de 8 Daltons entre os peptídeos rotulados. O espectro MS deste peptídeo é mostrado na Figura 3A. Clique aqui para ver uma versão maior desta figura.
| Amostra | H2CO | D2CO | D213CO | NaBH3CN | NaBD3CN | Etiqueta | Massa adicional |
| 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | código | (Da) | |
| 1 | X | X | L1 | 28.0313 | |||
| 2 | X | X | L2 | 30.0439 | |||
| 3 | X | X | L3 | 32.0564 | |||
| 4 | X | X | L4 | 34.069 | |||
| 5 | X | X | L5 | 36.0757 |
Tabela 1: Reagentes de um experimento típico usando a combinação indicada de formas deutadas e não deutadas de formaldeído e cianoboroidride de sódio.
Discussão
Na maioria dos estudos de peptidóticos, um dos passos críticos é, sem dúvida, a preparação da amostra que deve ser cuidadosamente realizada para evitar a presença de fragmentos de peptídeos gerados por proteases após alguns minutos após a morte. Os estudos iniciais sobre extratos cerebrais preparados a partir de amostras não micro-ondas mostraram um grande número de fragmentos de proteínas presentes nos microfiltratos de 10 kDa. Diferentes abordagens foram descritas para evitar espectros de peptídeos da degradação proteica: sacrifício animal de irradiação de micro-ondas focado6,8, dissecção criostata seguida de um tampão de extração fervente31, e irradiação de micro-ondas pós-sacrifício de tecido usando um forno de micro-ondas tipo doméstico9,26 . Para a cultura celular e alguns tecidos, a inativação protease pode ser feita diretamente adicionando água a 80 °C. No entanto, algumas amostras, como o tecido nervoso, podem ser mais sensíveis à mudança pós-morte, e a inativação protease por irradiação de micro-ondas foi indicada como um método de escolha. Além disso, outro ponto importante durante a extração do peptídeo é garantir que os extratos estejam gelados antes de adicionar ácido para evitar que as ligações ácido-labile quebrem, como o decote das ligações Asp-Pro26.
Diferentes estratégias podem ser utilizadas para a quantificação relativa de peptídeos, mas nenhuma delas pode ser considerada completamente ideal. Para escolher o método a ser utilizado, o pesquisador deve considerar fatores como disponibilidade de horas de uso no espectrômetro de massa, disponibilidade comercial e custos de rotulagem de reagentes e facilidade na análise dos dados obtidos17,25,26,32. O método sem rótulos tem sido amplamente utilizado, mas requer muitas horas no espectrômetro de massa. É necessário injetar réplicas técnicas para cada amostra e depende da reprodutibilidade cromatográfica entre as amostras. Outros reagentes de rotulagem química, por exemplo, ITRAQ (tags isobáricas para quantitação relativa e absoluta) e TMT (tag de massa tandem), são caros e só fornecem quantitação de peptídeos selecionados para análises em MS/MS25,33,34.
A maior limitação da quantificação relativa através da rotulagem química usando RMA é a sobreposição que ocorre para alguns peptídeos ao realizar um protocolo usando 4 ou 5 rótulos na mesma corrida, resultando em diferenças de massa de 2 Da para peptídeos com uma única amina primária e 1 Da para peptídeos com prolina N-terminus e sem resíduos de lisesina interna. No entanto, Tashima e Fricker (2018) desenvolveram um modelo para corrigir a sobreposição isotópica com base nas equações polinomiais cúbicas25, que obtêm a intensidade correta dos peptídeos rotulados nas amostras. Além disso, nem todos os peptídeos podem ser vistos pela RMA. Por exemplo, alguns peptídeos não possuem uma amina livre de terminal N devido à acetilação, pioglutamylation ou outra modificação. Se as lises internas também estiverem ausentes nestes peptídeos, elas não serão rotuladas pelo reagente RMA e aparecerão no espectro m/z como picos únicos inquantificáveis27.
Divulgações
Não existem interesses financeiros concorrentes.
Agradecimentos
O desenvolvimento e o uso das técnicas aqui descritas foram apoiados pelo Conselho Nacional de Pesquisa 420811/2018-4 (LMC); A Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) concede 2019/16023-6 (LMC), 2019/17433-3 (LOF) e 21/01286-1 (MEME). Os financiadores não tiveram papel na concepção do estudo, coleta e análise de dados, decisão de publicar ou elaboração do artigo.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
Referências
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223(1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312(2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150(2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093(2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188(2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321(2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , Chapter 18 13(2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross,, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados