Method Article
Probenvorbereitung und relative Quantifizierung mittels reduktiver Methylierung von Aminen für Peptidomik-Studien
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt eine Probenvorbereitungsmethode, die auf Wärmeinaktivierung basiert, um endogene Peptide zu erhalten und den Abbau post mortem zu vermeiden, gefolgt von einer relativen Quantifizierung mittels Isotopenmarkierung plus LC-MS.
Zusammenfassung
Peptidomics kann als die qualitative und quantitative Analyse von Peptiden in einer biologischen Probe definiert werden. Zu den Hauptanwendungen gehören die Identifizierung der Peptid-Biomarker für Krankheit oder Umweltstress, die Identifizierung von Neuropeptiden, Hormonen und bioaktiven intrazellulären Peptiden, die Entdeckung antimikrobieller und nutrazeutischer Peptide aus Proteinhydrolysaten und können in Studien verwendet werden, um die proteolytischen Prozesse zu verstehen. Der jüngste Fortschritt in der Probenvorbereitung, Trennmethoden, Massenspektrometrietechniken und Berechnungswerkzeugen im Zusammenhang mit der Proteinsequenzierung hat zur Erhöhung der identifizierten Peptidzahl und der charakterisierten Peptidome beigetragen. Peptidomische Studien analysieren häufig Peptide, die auf natürliche Weise in Zellen erzeugt werden. Hier wird ein Probenvorbereitungsprotokoll beschrieben, das auf Wärmeinaktivierung basiert, das die Proteaseaktivität eliminiert, und die Extraktion unter milden Bedingungen, so dass es keine Spaltung der Peptidbindungen gibt. Darüber hinaus wird auch die relative Quantifizierung von Peptiden mittels stabiler Isotopenmarkierung durch reduktive Methylierung von Aminen gezeigt. Diese Markierungsmethode hat einige Vorteile, da die Reagenzien im Handel erhältlich, im Vergleich zu anderen kostengünstig, chemisch stabil sind und die Analyse von bis zu fünf Proben in einem einzigen LC-MS-Lauf ermöglichen.
Einleitung
"Omics" -Wissenschaften zeichnen sich durch die tiefe Analyse eines Molekülsatzes wie DNA, RNA, Proteine, Peptide, Metaboliten usw. aus. Diese generierten groß angelegten Datensätze (Genomik, Transkriptomik, Proteomik, Peptidomik, Metabolomik usw.) haben die Biologie revolutioniert und zu einem fortgeschrittenen Verständnis biologischer Prozesse geführt1. Der Begriff Peptidomik wurde im frühen 20. Jahrhundert eingeführt, und einige Autoren haben ihn als einen Zweig der Proteomik bezeichnet2. Die Peptidomik weist jedoch deutliche Besonderheiten auf, wobei das Hauptinteresse darin besteht, den natürlich erzeugten Peptidgehalt während zellulärer Prozesse sowie die Charakterisierung der biologischen Aktivität dieser Moleküle zu untersuchen3,4.
Zunächst beschränkten sich bioaktive Peptidstudien auf die Neuropeptide und Hormonpeptide durch Edman-Abbau und Radioimmunoassay. Diese Techniken erlauben jedoch keine globale Analyse, abhängig von der Isolierung jedes Peptids in hohen Konzentrationen, Zeit für die Bildung von Antikörpern, neben der Kreuzreaktivitätsmöglichkeit5.
Die Peptidomik-Analyse wurde erst nach mehreren Fortschritten in der Flüssigkeitschromatographie-gekoppelten Massenspektrometrie (LC-MS) und Genomprojekten ermöglicht, die umfassende Datenpools für Proteomik-/Peptidomik-Studien lieferten6,7. Darüber hinaus musste ein spezifisches Peptidextraktionsprotokoll für Peptidome etabliert werden, da die ersten Studien, die Neuropeptide weltweit in Gehirnproben analysierten, zeigten, dass der Nachweis durch den massiven Abbau von Proteinen beeinflusst wurde, die hauptsächlich in diesem Gewebe nach 1 min Post mortem auftreten. Das Vorhandensein dieser Peptidfragmente maskierte das Neuropeptidsignal und stellte das Peptidom in vivo nicht dar. Dieses Problem wurde hauptsächlich durch die Anwendung der schnellen Erwärmungsinaktivierung von Proteasen mittels Mikrowellenbestrahlung gelöst, die das Vorhandensein dieser Artefaktfragmente drastisch reduzierte und nicht nur die Identifizierung von Neuropeptidfragmenten ermöglichte, sondern auch das Vorhandensein einer Reihe von Peptiden aus zytosolischen, mitochondrialen und Kernproteinen aufdeckte, die sich von Degradome unterscheiden6,8,9.
Diese methodischen Verfahren ermöglichten eine Erweiterung des Peptidoms über die bekannten Neuropeptide hinaus, wo Hunderte von intrazellulären Peptiden, die hauptsächlich durch die Wirkung von Proteasomen erzeugt wurden, in Hefe10, Zebrafischen11, Nagetiergeweben12 und menschlichen Zellen13 identifiziert wurden. Dutzende dieser intrazellulären Peptide haben nachweislich sowohl biologische als auch pharmakologische Aktivitäten14,15. Darüber hinaus können diese Peptide als Krankheitsbiomarker verwendet werden und haben möglicherweise klinische Bedeutung, wie in der Zerebrospinalflüssigkeit von Patienten mit intrakraniellen sackförmigen Aneurysmen gezeigt wurde16.
Derzeit ist es neben der Identifizierung von Peptidsequenzen durch Massenspektrometrie möglich, Daten der absoluten und relativen Quantifizierung zu erhalten. Bei der absoluten Quantifizierung werden die Peptidspiegel in einer biologischen Probe mit synthetischen Standards verglichen, während bei der relativen Quantifizierung die Peptidspiegel zwischen zwei oder mehr Proben verglichen werden17. Die relative Quantifizierung kann mit den folgenden Ansätzen durchgeführt werden: 1) "label free"18; 2) in vivo metabolische Markierung oder 3) chemische Markierung. Die letzten beiden basieren auf der Verwendung stabiler Isotopenformen, die in Peptide eingebaut sind19,20. Bei der markierungsfreien Analyse werden die Peptidspiegel unter Berücksichtigung der Signalstärke (Spektralzählungen) während des LC-MS18 geschätzt. Die Isotopenmarkierung kann jedoch genauere relative Konzentrationen von Peptiden erhalten.
Viele peptidomische Studien verwendeten Trimethylammoniumbutyrat (TMAB) Markierungsreagenzien als chemische Markierung, und in jüngerer Zeit wurde die reduktive Methylierung von Aminen (RMA) mit deuterierten und nicht deuterierten Formen von Formaldehyd und Natriumcyanoborhydridreagenzien verwendet11,21,22. Die TMAB-Etiketten sind jedoch nicht im Handel erhältlich und der Syntheseprozess ist sehr aufwendig. Auf der anderen Seite sind die Reagenzien in der RMA kommerziell erhältlich, kostengünstig im Vergleich zu anderen Etiketten, das Verfahren ist einfach durchzuführen und die markierten Peptide sind stabil23,24.
Die Verwendung von RMA beinhaltet die Bildung einer Schiff-Base, indem die Peptide mit Formaldehyd reagieren können, gefolgt von einer Reduktionsreaktion durch das Cyanoborhydrid. Diese Reaktion bewirkt eine Dimethylierung freier Aminogruppen auf N-terminalen und Lysin-Seitenketten und Monomethylaten N-terminalen Prolinen. Da Prolinreste auf dem N-Terminal oft selten sind, sind praktisch alle Peptide mit freien Aminen am N-Terminus mit zwei Methylgruppen markiert23,24,25.
Protokoll
Das folgende Verfahren zur Peptidextraktion und reduktiven Methylierung wurde aus zuvor veröffentlichten Verfahren angepasst24,25,26,27. Dieses Protokoll folgte den Richtlinien des National Council for Animal Experimentation Control (CONCEA) und wurde von der Ethikkommission für Tierversuche (CEUA) am Biowissenschaftlichen Institut der Staatlichen Universität Sao Paulo genehmigt. Die Protokollschritte sind in Abbildung 1 dargestellt.
HINWEIS: Bereiten Sie alle wässrigen Lösungen in Reinstwasser vor.
1. Peptidextraktion
- Zellkultur
- Kultivieren Sie SHSY5Y-Zellen in einer 15 cm großen Schale bei 37 °C unter 5% CO2 in Dulbeccos modifiziertem Eagle's Medium, das 15% fötales Rinderserum und 1% Penicillin-Streptomycin enthält.
- Verwenden Sie 2-3 Platten für jede Probe. Wachsen Sie die Zellen zu 100% Konfluenz.
- Nach der beabsichtigten Behandlung waschen Sie die Zellen zweimal mit phosphatgepufferter Kochsalzlösung. Als nächstes fügen Sie 10 ml phosphatgepufferte Kochsalzlösung hinzu, kratzen Sie die Zellen und sammeln Sie sie in einem 15 ml Röhrchen.
- Zentrifugieren Sie bei 800 x g für 5 min und entfernen Sie den Überstand. Resuspendieren Sie das Pellet in 1 ml ultragereinigtem entionisiertem Wasser bei 80 °C.
- Übertragen Sie den Inhalt des Röhrchens (zelluläres Lysat) in ein 2 ml Mikrofugenröhrchen.
- Tierische Gewebe (hauptsächlich Nervengewebe):
- Betäuben Sie den männlichen Wildtyp Danio rerio (Zebrafisch) mit einer tödlichen Dosis von MS 222 (100 mg / L) und setzen Sie ihn sofort 8 s Mikrowellenstrahlung aus, um Peptidase und Protease zu inaktivieren.
HINWEIS: Ein Haushalts-Mikrowellenherd kann verwendet werden. Eine 900 W Mikrowelle wurde für 8 bis 10 s bei voller Leistung verwendet. Die verwendete Mikrowelle muss in der Lage sein, die Gehirntemperatur innerhalb von 10 s auf > 80 °C zu erhöhen. Die Reproduzierbarkeit beim Erhitzen zwischen den Proben würde auch dadurch begünstigt, dass das Gewebe an derselben Stelle in der Mikrowelle platziert wird. - Nach der Wärmeinaktivierung das gesamte Gehirn in einem 2-ml-Mikrofugenröhrchen sammeln und bis zur Analyse bei −80 °C einfrieren.
- Resuspendieren Sie die Gewebeprobe in 1 ml ultragereinigtem entionisiertem Wasser bei 80 °C. Beschallen Sie das Gewebe mit einer Sonde mit 30 Impulsen (4 Hz) von 1 s.
HINWEISE: Verwenden Sie für Leber- und Nierengewebe einen mechanischen Homogenisator mit 10.000-30.000 U / min für 20 s. Für Muskelgewebe mahlen Sie das Gewebe in flüssigem Stickstoff mit einem Porzellantiegel und Stößel. Die folgenden Schritte sind die gleichen für das zelluläre Lysat oder das homogenate Gewebe.
- Betäuben Sie den männlichen Wildtyp Danio rerio (Zebrafisch) mit einer tödlichen Dosis von MS 222 (100 mg / L) und setzen Sie ihn sofort 8 s Mikrowellenstrahlung aus, um Peptidase und Protease zu inaktivieren.
- Inkubieren Sie das zelluläre Lysat- oder Homogenatgewebe bei 80 °C für 20 min. Als nächstes kühlen Sie es 10-30 Minuten auf Eis ab.
- 10 μL 1 M HCl-Stammlösung für jeweils 1 mL Probenvolumen zugeben, um eine Endkonzentration von 10 mM zu erhalten. Mischen Sie durch Wirbeln für 20 s und inkubieren Sie weiter auf Eis für 15 min.
HINWEIS: Stellen Sie vor dem Ansäuern sicher, dass die Probe vollständig abgekühlt ist, um zu vermeiden, dass die durch Ansäuerung bei erhöhten Temperaturen verursachten Peptidbindungen unterbrochen werden. - Zentrifugieren Sie das zelluläre Lysat oder das homogenate Gewebe bei 12.000 x g bei 4 °C für 15 min. Sammeln Sie den Überstand in mikrozentrifugen Röhrchen mit niedrigem Bindungsprotein und lagern Sie ihn bei -80 °C.
- Reinigen Sie die Ultrafiltrationsgeräte (10 kDa-Trennfilter) durch Zugabe von Wasser und Zentrifuge bei 2300 x g für 3 min. Wiederholen Sie diesen Schritt noch zwei weitere Male.
- Den Überstand in die vorgewaschenen 10 kDa-Trennfilter geben und bei 2.300 x g bei 4 °C für 50 min in einer gekühlten Zentrifuge zentrifugieren. Der Durchfluss stellt den Peptidextrakt dar.
- Entsalzen Sie die Proben auf Umkehrphasen-Reinigungskolonnen gemäß den Anweisungen des Herstellers unter Verwendung von Acetonitril ( ACN) und Trifluoressigsäure (TFA) -Lösungen wie unten beschrieben:
- Gleichsetzen Sie die Säule mit 1 ml 100% ACN.
- Waschen Sie die Säule mit 1 ml Lösung von 5% ACN mit 0,1% TFA.
- Laden Sie das gesamte Volumen der Probe in die Spalte.
- Waschen Sie die Säule mit 1 ml Lösung von 5% ACN mit 0,1% TFA
- Eluieren Sie die Peptide aus der Säule mit einer 1,8 ml Lösung von 100% ACN mit 0,15% TFA in proteinarm bindenden Mikrozentrifugenröhrchen.
- Trocknen Sie die Probe vollständig in einer Vakuumzentrifuge. Die Konzentrationsmethode für organische Lösungsmittel und die Temperatur werden auf 30 °C eingestellt. Die Konzentrationszeit wird auf dem Display überwacht.
- Lagern Sie die Proben bis zum nächsten Schritt bei −80°C.
2. Peptidquantifizierung mit Fluorescamin
ANMERKUNG: Die Menge an Peptid kann unter Verwendung von Fluorescamin bei pH 6,8 wie zuvor beschrieben geschätzt werden11,28. Diese Methode besteht aus der Bindung eines Fluorescaminmoleküls an die primären Amine, die in den Lysin(K)-Resten und/oder dem N-Terminal von Peptiden vorhanden sind. Die Reaktion wird bei pH 6,8 durchgeführt, um zu gewährleisten, dass das Fluorescamin nur mit den Aminogruppen der Peptide und nicht mit freien Aminosäuren reagiert. Das Fluorescamin wird mit einem Spektrofluorometer bei einer Anregungswellenlänge von 370 nm und einer Emissionswellenlänge von 480 nm gemessen.
- Verschiedene Konzentrationen des Standardpeptids (0,05, 0,1, 0,15, 0,2, 0,3, 0,5 und 0,7 μg/μL) herstellen und die Aliquots bei -20 °C lagern.
HINWEIS: Das Peptid 5A (LTLRTKL) wird vorgeschlagen, da es eine bekannte Zusammensetzung und Konzentration aufweist. - Herstellen einer Fluorescamine-Stammlösung (0,3 mg/ml) in Aceton. Schnell in Mikrozentrifugenröhrchen (1 ml) aliquotieren, mit Parafilm verschließen und im Dunkeln bei -20 °C lagern.
- 0,2 M Phosphatpuffer (PB) bei pH 6,8 herstellen.
ANMERKUNG: Herstellen von 0,2 M PB durch Zugabe von 0,1 M Phosphatpuffer pH 6,8 (26,85 ml Na2HPO3 1M) und 0,1 M Phosphatpuffer pH 6,8 (23,15 ml NaH2PO3 1M) zu 250 mL Wasser - Resuspendieren Sie die Peptidproben in 100-200 μL ultragereinigtem Wasser.
- 2,5 μL der Standardpeptidkonzentrationen und -proben werden für Fluoreszenzassays in dreifacher Ausführung auf die weiße 96-Well-Platte pipettiert. Fügen Sie 25 μL 0,2 M Phosphatpuffer hinzu.
- Fügen Sie 12,5 μL Fluorescamin mit einer Mehrkanalpipette hinzu. Homogenisieren Sie vorsichtig für 1 min auf dem Orbital-Rotator-Shaker.
- Als nächstes fügen Sie 110 μL Wasser mit einer Mehrkanalpipette hinzu, um die Reaktion zu stoppen.
HINWEIS: Geben Sie die Fluorescamin-Stammlösung und das Reinstwasser in zwei Reservoirs, um diese Lösungen mit einer Mehrkanalpipette zu pipettieren. - Stellen Sie die folgenden Messparameter auf dem Spektrofluorometer ein: Lesen Sie die Proben von oben, Anregungswellenlänge bei 370 nm und Emissionswellenlänge bei 480 nm.
- Lesen Sie die Platte auf dem Spektrofluorometer.
3. Reduktive Methylierung von Aminen Markierung
HINWEIS: Diese Isotopenmarkierungsmethode basiert auf der Dimethylierung von Amingruppen mit deuterierten und nicht deuterierten Formen von Formaldehyd- und Natriumcyanoborhydridreagenzien. Das Endprodukt dieser Reaktion fügt der Endmasse jedes Peptids an jeder verfügbaren Markierungsstelle (Lysin oder N-terminal) 28 Da, 30 Da, 32 Da, 34 Da oder 36 Da hinzu. Diese Reaktion erzeugt einen m/z-Unterschied in den Peptiden, die mit verschiedenen Formen markiert sind, die im MS-Spektrum beobachtet wurden (Tabelle 1).
VORSICHT: Für den Umgang mit diesen Verbindungen sollte eine geeignete Sicherheitsausrüstung verwendet werden, und es sollte darauf geachtet werden, die Exposition zu minimieren. Verfahren mit Formaldehyd- und Natriumcyanoborhydridreagenzien sollten in einem Abzug durchgeführt werden, da sie sehr giftig sind (einschließlich des Wiegens des Natriumcyanoborhydrids). Während der Abschreckreaktion und Ansäuerung kann ein toxisches Gas (Blausäure) entstehen.
- Bereiten Sie die folgenden frischen Lösungen aus der Brühe oder Reagenzien am Tag des Verfahrens in Reinstwasser vor:
- Verdünnen Sie den Vorrat an 37% Formaldehyd (CH2O) auf 4%.
- Verdünnen Sie den Vorrat an 20% deuteriertem Formaldehyd (CD2O) auf 4%.
- Verdünnen Sie den Vorrat an 20 % deuteriertem C13-Formaldehyd (13CD2O) auf 4 %.
- Bereiten Sie 0,6 m NaBH3CN vor.
- Bereiten Sie 0,6 m NaBD3CN vor.
- Bereiten Sie eine Lösung von 1% Ammoniumbicarbonat vor.
- Bereiten Sie eine Lösung 5% Ameisensäure vor.
HINWEIS: In Bezug auf die relative Quantifizierung von Peptiden, da je nach Anzahl der verwendeten Markierungen unterschiedliche experimentelle Schemata durchgeführt werden können, ist während des chemischen Markierungsverfahrens Aufmerksamkeit erforderlich. Es wird empfohlen, kleine Aliquots der Etiketten in separaten Racks mit den entsprechenden zu etikettierenden Proben zu trennen, um das Potenzial für menschliche Fehler bei der Zugabe des falschen Reagenzes zu den Probenröhrchen zu verringern.
- Bereiten Sie jede Probe vor, die bis zu 25 μg des Peptids enthält. Die Proben dürfen weder Tris noch Ammoniumbicarbonat enthalten.
HINWEIS: Die unten beschriebenen Mengen sind für jede Probe in einem Volumen von 100 μL ausreichend.
Fahren Sie mit den Schritten 3.3-3.8 in einem Abzug fort. - 1/10 Volumen von 1 M TEAB zu den Proben geben (Endkonzentration der Lösung 100 mM TEAB). Überprüfen Sie den pH-Wert mit einem pH-Indikatorpapier; es muss zwischen 5-8 liegen. Bei Bedarf mit HCl oder NaOH einstellen.
- Fügen Sie 4 μL nicht deuteriertes, deuteriertes Formaldehyd oder C13 deuteriertes Formaldehyd gemäß dem festgelegten Kennzeichnungsschema hinzu. Mischen Sie für 5 s durch Vortexing.
- Fügen Sie 4 μL NaBH3CN (0,6 M) oder NaBD3CN (0,6 M) gemäß dem festgelegten Kennzeichnungsschema hinzu. Mischen Sie für 5 s durch Vortexing.
- In einem Abzug für 2 h bei Raumtemperatur inkubieren und alle 30 Minuten mischen.
- Wiederholen Sie die Schritte 3.4 und 3.5. Inkubieren Sie die Proben über Nacht bei Raumtemperatur in einem Abzug.
- Fügen Sie 16 μL Ammoniumbicarbonat (1%) hinzu und mischen Sie durch Vortexing. Legen Sie die Probe auf Eis, geben Sie 8 μL Ameisensäure (5%) hinzu und mischen Sie durch Wirbel für 5 s.
- Kombinieren Sie die Proben, stellen Sie den pH-Wert auf 2-4 ein und entsalzen Sie die kombinierten Proben auf Umkehrphasen-Reinigungssäulen, wie zuvor in Schritt 1.8 beschrieben.
- Trocknen Sie die Probe vollständig in einer Vakuumzentrifuge. Die Konzentrationsmethode für organische Lösungsmittel und die Temperatur werden auf 30 °C eingestellt. Die Konzentrationszeit wird auf dem Display überwacht.
- Lagern Sie die Proben bei -20 °C.
4. Flüssigkeitschromatographie und Massenspektrometrie
- Führen Sie eine LC-MS-Analyse mit einem nanoHPLC-System durch, das mit einem MS-Instrument gekoppelt ist, das mit Methylmarkierungen kompatibel ist.
HINWEIS: Eine Vielzahl von MS-Geräten ist mit der RMA-Kennzeichnung kompatibel. Ein mit Orbitrap gekoppeltes NanoHPLC-System wird typischerweise verwendet, um LC-MS-Analysen durch eine Nanoelektrospray-Ionenquelle durchzuführen. Zunächst wird die Probe in eine Vorsäule geladen und die Peptide in einer analytischen Säule getrennt. Die Elution von Peptiden erfolgt unter Verwendung eines linearen Gradienten von 5% -45% Acetonitril, in 0,1% Ameisensäure, während 90 min, mit einem Fluss von 200 nL / min. Das Massenspektrometer ist so eingestellt, dass es im datenabhängigen Modus funktioniert. Jeder vollständige Scan wird mit einer Intensität von 10-30 eV, 2,3 Kv erfasst, und dann werden die zehn höchsten Peaks für die kollisionsinduzierte Dissoziationsfragmentierung (CID) ausgewählt. Die Injektionszeit wird auf die Ionenfalle auf 100 ms eingestellt, und die Fourier-Transformation (FT)-MS-Injektion wird mit einer Auflösung von 1000 ms 30.000 bei m/z 300-1800 fixiert. Ein Minimum von 5000 Zählungen und ein dynamischer Ausschluss von 70 s wird verwendet, um Fragmentierungsscans durchzuführen.
5. Relative Quantifizierung von Peptiden
HINWEIS: Die MS-Spektren werden in der Massenspektrometer-Software analysiert. Peakgruppen von markierten Peptiden mit unterschiedlichen Tags werden in den MS-Spektren identifiziert. Die relative Quantifizierung wird durch die Intensität jedes monoisotopen Peaks berechnet. Jede behandelte Gruppe wird mit der jeweiligen Kontrollgruppe verglichen.
- Doppelklicken Sie mit der rechten Maustaste auf die Rohprobendatei, um die Spektrumanalysesoftware zu öffnen. Laden Sie die Chromatogramme Retention Time (RT) und MS Spectrum (EM) in zwei Registerkarten, oben bzw. unten.
- Klicken Sie einmal nacheinander mit der rechten Maustaste auf die Symbole Anzeige - und Massenoptionen in der Softwaresymbolleiste und legen Sie die Massengenauigkeit auf vier Dezimalstellen fest.
- Positionieren Sie den Mauszeiger an einer beliebigen Stelle auf der Registerkarte RT. Suchen Sie nach der Retentionszeit des entsprechenden zu analysierenden Ions und klicken Sie mit der rechten Maustaste. Das MS-Spektrum der ausgewählten Zeit wird automatisch im EM-Tab angezeigt.
- Positionieren Sie den Mauszeiger an einer beliebigen Stelle auf der Registerkarte EM. Suchen Sie nach den zu analysierenden Ionen.
- Klicken Sie mit der rechten Maustaste und halten Sie einen benachbarten Bereich links in der Nähe dieser Ionen gedrückt. Ziehen Sie dann die Maus im gewünschten Bereich nach rechts, um den gewünschten Bereich zu vergrößern.
- Halten Sie die Maus auf der Registerkarte EM positioniert und klicken Sie auf die Pfeile der rechten oder linken Tastatur, um den Bereich der zu analysierenden Ionen zu definieren.
- Positionieren Sie den Mauszeiger auf der Registerkarte RT wieder am Anfang des gewünschten Zeitintervalls.
- Klicken Sie mit der rechten Maustaste und ziehen Sie die Maus bis zum gewählten Zeitwert. Lassen Sie die Schaltfläche stehen. Die akkumulierte Intensität der Ionen wird automatisch auf der Registerkarte EM angezeigt.
- Sammeln Sie die m/z-, z- und Ionenintensitätsdaten in einer Tabelle.
ANMERKUNG: Die monoisotope Masse jedes Peptids ohne Zusatz von Methylgruppen wird nach folgender Formel berechnet:
Masse unmodifiziertes Peptid = (m/z a x z) - (C a x T) - (1,008 x z)
m/zaist der beobachtete Masse-Ladungswert für den monoisotopen Peak für jedes Peptid, das mit verschiedenen Kombinationen von Tags markiert ist (a =1, 2, 3, 4 oder 5, entsprechend der Probennummer).
z ist der Ladezustand.
Ca ist die monoisotope Masse eines Paares von Methylgruppen:
Für a=1, Ca = 28,0313 (die Nettoaddition von zwei CH3-Gruppen zum primären Amin)
Für a=2, Ca = 30,0439 für zwei CHD2-Gruppen
Für a=3, Ca = 32,0564 für zwei CD2H-Gruppen
Für a=4, Ca= 34.0690 für zwei CD3-Gruppen
Für a=5, Ca = 36,0757 für zwei 13 CD3-Gruppen
T ist die Anzahl der Paare von Methylgruppen, die in das Peptid eingebaut sind. Dies kann aus der folgenden Formel berechnet werden, wenn fünf Tags verwendet werden: T=z*(m/z5 - m/z1)/8. Für Peptide, die ein einzelnes primäres Amin enthalten und daher mit nur zwei Methylgruppen markiert sind, liegen Peaküberlappungen auf den MS-Spektren vor, wenn benachbarte Markierungen verwendet werden. Die Spitzenintensität jedes markierten Peptids kann mit den von Tashima und Fricker25 beschriebenen Gleichungen korrigiert werden.
6. Peptid-Identifizierung
- Um Peptide zu identifizieren, analysieren Sie die MS/MS-Daten mit einer Datenbanksuchmaschine29,30.
- Um die False Discovery Rate (FDR) mithilfe der Decoy-Fusionsmethode zu berechnen, durchsuchen Sie eine Köderdatenbank.
ANMERKUNG: Die im Allgemeinen verwendeten Suchparameter sind keine Enzymspezifität; Vorstufe Massentoleranz eingestellt 15-50 ppm; Fragmentionenmassentoleranz von 0,5 Da; variable Modifikationen: reaktive Amine aus Lys-Resten und N-Terminus der Peptide isotopmethylierte Markierungen (L1 (+28), L2 (+30), L3 (+32), L4 (+34) und L5 (+36)), oxidiertes Methionin (+15,99 Da) und Acetylierung (+42,01 Da). - Sortieren Sie dann die Peptide nach ihrem durchschnittlichen lokalen Konfidenzwert, um die besten Spektren auszuwählen, um sie zu kommentieren, und filtern Sie sie nach FDR ≤5%.

Abbildung 1: Workflow für Peptidomic-Studien. Schritte der Peptidextraktion und reduktiven Methylierung von Aminen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Die Ergebnisse der auf dem Massenspektrometer durchgeführten Läufe werden in Rohdatendateien gespeichert, die in der Massenspektrometer-Software geöffnet werden können. In den MS-Spektren ist es möglich, Peakgruppen zu beobachten, die markierte Peptide gemäß dem verwendeten Markierungsschema darstellen, das von 2-5 Markierungen reicht. In Abbildung 2 werden beispielsweise Paare von Peaks, die in einer chromatographischen Zeit detektiert wurden, in einem Experiment dargestellt, bei dem nur zwei Isotopenmarkierungen in zwei verschiedenen Proben im selben Durchlauf verwendet wurden. Abbildung 3 zeigt weitere Möglichkeiten positiver Ergebnisse unter Verwendung von 3 und 4 verschiedenen Labels in jedem LC-MS-Lauf. Bei verwendung von 4 oder 5 Markierungen in einem LC/MS-Lauf kann es zu einer Überlappung der Peaks der markierten Peptide mit den verschiedenen Tags kommen, die korrigiert werden müssen, um den tatsächlichen Intensitätswert jedes Peaks zu erhalten (Abbildung 4).
Die Isotopenmarkierung kann auch verwendet werden, um Substrate und Produkte in vitro für eine bestimmte Protease oder Peptidase zu zeigen, wie in Abbildung 5 gezeigt. Schließlich kann verschiedene Software verwendet werden, um die markierten Peptide zu identifizieren, wie Peaks Studio oder MASCOT. Diese Softwareanwendungen wurden für die Proteomanalyse entwickelt; Daher sollten die Proteinquantifizierungsdaten für die Peptidomik-Analyse nicht berücksichtigt werden, und jedes markierte Peptid, das innerhalb der Zuverlässigkeitsparameter identifiziert wurde, muss überprüft und dann quantifiziert werden. Wenn die Peptide erfolgreich nachgewiesen und markiert wurden, liefern diese Programme eine Liste der identifizierten Peptidsequenzen, die die Markierungen enthalten. Abbildung 6 zeigt ein Beispiel für die Identifizierung einer Peptidsequenz durch das Programm. In diesem Fall wurden nur 3 verschiedene Formen der Etiketten (L1, L3 und L5) verwendet, um drei verschiedene Proben separat zu kennzeichnen, die dann gemischt und massenspektrometrisch in einem einzigen Durchlauf analysiert wurden.
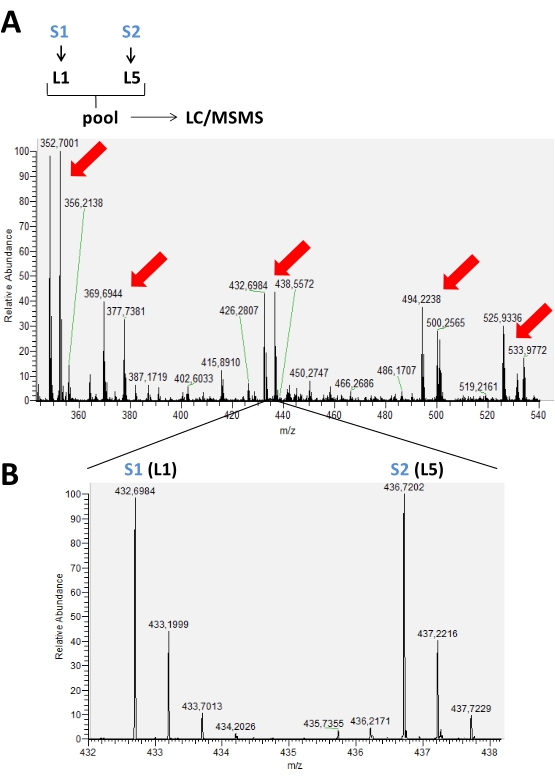
Abbildung 2: MS-Spektrum repräsentativ für eine chromatographische Zeit, die in einem typischen Markierungsexperiment unter Verwendung der reduktiven Dimethylierung von Aminen akkumuliert wurde. In (A) zeigen die roten Pfeile das Vorhandensein von Peakpaaren verschiedener Peptide an, die mit 2 Isotopenformen (L1 und L5) für den Vergleich zwischen zwei verschiedenen Proben (S1 und S2) markiert sind. In (B) ein vergrößertes Bild eines MS-Spektrums desselben Peptids, das aufgrund der Verwendung von Markierungen unterschiedliche m/z zeigt. In diesem Fall gab es keine Variation der Spitzenintensität für dieses Peptid, das in diesen Proben vorhanden war. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Repräsentatives MS-Spektrum markierter Peptide mit reduktiver Methylierung von Aminen unter Verwendung unterschiedlicher Anzahl von Tags. (A) Es wurde eine Triplex-Markierung verwendet. Es ist möglich, ein MS-Spektrum eines Peptids zu beobachten, das in 3 verschiedenen Proben (S1, S2 und S3) vorhanden ist, die mit L1-, L3- bzw. L5-Tags markiert sind. In diesem Fall war das Niveau des markierten Peptids mit L5 doppelt so hoch wie das Niveau, das für dasselbe Peptid beobachtet wurde, das mit L1- und L3-Tags markiert war. (B) Eine Quadripex-Kennzeichnung wurde unter Verwendung der Etiketten L1, L2, L3 und L4 durchgeführt. In diesem Fall wurden Kontrollproben (S1 und S3) mit L1 bzw. L3 markiert und mit zwei experimentellen Proben (S2 und S4) verglichen, die mit L2- und L4-Etiketten gekennzeichnet waren. Für dieses Peptid wurden keine signifikanten Unterschiede zwischen den Proben beobachtet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Repräsentatives MS-Spektrum eines markierten Peptids mit einer Peaküberlappung. Diese Abbildung zeigt das MS-Spektrum eines Peptids der Ladung 3, Masse von 2098,87 Da mit einem einzigen primären Amin, das zur Markierung zur Verfügung steht. Der Unterschied zwischen den markierten Peptiden beträgt nur 2 Da, was zu einer Überlappung führt, wenn 4 oder 5 Etiketten im selben LC / MS-Lauf verwendet werden. Mit einem Modell, das auf kubischen Polynomgleichungen basiert, ist es möglich, diese Überlappungen zwischen den Bezeichnungen zu korrigieren. In der Grafik zeigen die roten Balken den Durchschnitt der für dieses Peptid in zwei Durchläufen eingestellten Intensitätswerte. Die schwarzen Balken zeigen den Durchschnitt der überlappenden Intensitätswerte. Nach der Korrektur zeigte dieses Peptid geringe Unterschiede in der Intensität zwischen den Proben (rote Balken). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Repräsentatives MS-Spektrum markierter Peptide mit reduktiver Methylierung von Aminen in einer Proteolysestudie. Hier wurden Peptidextrakte mit 200 nM und 20 nM Neurolysin inkubiert, um ihre Substrate und Produkte zu charakterisieren. Zur Bestätigung des Ergebnisses wurden zwei LC/MS-Läufe mit Vorwärts- und Rückwärtsbeschriftungsstrategien durchgeführt. Im zweiten Durchlauf wird die Position der Proben im Etikettierverfahren in Bezug auf das im ersten Durchlauf verwendete Schema geändert. In A wird ein Peptid gezeigt, das sich in Gegenwart des Neurolysin-Enzyms nicht verändert (NC). In B ein Peptid, das in Gegenwart einer hohen Konzentration des Enzyms (S2) verschwand und eine geringe Verringerung der niedrigen Konzentration des Enzyms (S4) sowohl in der Vorwärts- als auch in der Rückwärtsmarkierung zeigte. Dieses Peptid gilt als Substrat (SB) des Enzyms. In C ein Beispiel für ein Peptid, das als Produkt (PD) betrachtet wurde, weil seine Konzentration (S2 und S4) in Gegenwart des Enzyms erhöht wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Repräsentatives MSMS-Spektrum und Identifizierung eines markierten Peptids durch eine Datenbanksuchmaschine. In diesem Beispiel ist es möglich, 2+ Ionen mit m/z 672,3802, 676,4045 und 680,4241 zu beobachten, die einem Peptid der gleichen Masse entsprechen, das mit den methylierten Formen L1, L3 bzw. L5 markiert ist. Diese Sequenz ADQVSASLAKQGL wurde über das MS/MS-Spektrum als Fragment des Mikrotubuli-assoziierten Proteins Tau-Isoform X1 identifiziert. Dieses Peptid hat das N-terminale und ein Lysin, das als Markierungsstellen zur Verfügung steht und einen Massenunterschied von 8 Dalton zwischen den markierten Peptiden hinzufügt. Das MS-Spektrum dieses Peptids ist in Abbildung 3A dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Probe | H2CO | D2CO | D213CO | NaBH3CN | NaBD3CN | Etikett | Zusätzliche Masse |
| 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | Code | (Da) | |
| 1 | X | X | L1 | 28.0313 | |||
| 2 | X | X | L2 | 30.0439 | |||
| 3 | X | X | L3 | 32.0564 | |||
| 4 | X | X | L4 | 34.069 | |||
| 5 | X | X | L5 | 36.0757 |
Tabelle 1: Reagenzien eines typischen Experiments unter Verwendung der angegebenen Kombination von deuterierten und nicht deuterierten Formen von Formaldehyd und Natriumcyanoborhydrid.
Diskussion
In den meisten Peptidomik-Studien ist einer der kritischen Schritte zweifellos die Probenvorbereitung, die sorgfältig durchgeführt werden sollte, um das Vorhandensein von Peptidfragmenten zu vermeiden, die von Proteasen nach einigen Minuten post mortem erzeugt werden. Die ersten Studien an Gehirnextrakten, die aus nicht in der Mikrowelle hergestellten Proben hergestellt wurden, zeigten eine große Anzahl von Proteinfragmenten, die in den 10-kDa-Mikrofiltraten vorhanden sind. Es wurden verschiedene Ansätze beschrieben, um Peptidspektren vor dem Proteinabbau zu vermeiden: gezielte Mikrowellenbestrahlung, Tieropfer6,8, Kryostatendissektion gefolgt von einem siedenden Extraktionspuffer31 und Mikrowellenbestrahlung von Gewebe nach dem Opfer mit einem Haushaltsmikrowellenofen9,26 . Bei Zellkulturen und einigen Geweben kann die Proteaseinaktivierung direkt durch Zugabe von Wasser bei 80 °C erfolgen. Einige Proben, wie z. B. Nervengewebe, können jedoch empfindlicher auf postmortale Veränderungen reagieren, und die Proteaseinaktivierung durch Mikrowellenbestrahlung wurde als Wahlmethode angegeben. Darüber hinaus ist ein weiterer wichtiger Punkt während der Peptidextraktion sicherzustellen, dass die Extrakte eiskalt sind, bevor Säure hinzugefügt wird, um zu verhindern, dass Säure-Labile-Bindungen brechen, wie z.B. die Spaltung von Asp-Pro-Bindungen26.
Für die relative Quantifizierung von Peptiden können verschiedene Strategien verwendet werden, aber keine von ihnen kann als völlig ideal angesehen werden. Um die zu verwendende Methode zu wählen, muss der Forscher Faktoren wie die Verfügbarkeit von Nutzungsstunden im Massenspektrometer, kommerziell verfügbar und Kosten für die Kennzeichnung von Reagenzien sowie die einfache Analyse der erhaltenen Daten berücksichtigen17,25,26,32. Die markierungsfreie Methode ist weit verbreitet, benötigt aber viele Stunden im Massenspektrometer. Es ist notwendig, technische Replikate für jede Probe zu injizieren und hängt von der chromatographischen Reproduzierbarkeit zwischen den Proben ab. Andere chemische Markierungsreagenzien, zum Beispiel ITRAQ (isobare Tags für relative und absolute Quantifizierung) und TMT (Tandem Mass Tag), sind teuer und bieten nur eine Quantifizierung von Peptiden, die für die MS/MS-Analyse ausgewählt wurden25,33,34.
Die Haupteinschränkung der relativen Quantifizierung durch chemische Markierung unter Verwendung von RMA ist die Überlappung, die bei einigen Peptiden auftritt, wenn ein Protokoll mit 4 oder 5 Markierungen gleichzeitig durchgeführt wird, was zu Massenunterschieden von 2 Da für Peptide mit einem einzigen primären Amin und 1 Da für Peptide mit N-Terminus-Prolin und ohne interne Lysinreste führt. Tashima und Fricker (2018) entwickelten jedoch ein Modell zur Korrektur der Isotopenüberlappung basierend auf kubischen Polynomgleichungen25, die die korrekte Intensität der markierten Peptide in den Proben erhalten. Darüber hinaus können nicht alle Peptide durch RMA gesehen werden. Zum Beispiel fehlt einigen Peptiden ein N-terminales freies Amin aufgrund von Acetylierung, Pyroglutamylierung oder einer anderen Modifikation. Fehlen auch interne Lysine in diesen Peptiden, werden sie vom RMA-Reagenz nicht markiert und erscheinen auf den m/z-Spektren als nicht quantifizierbare Einzelpeaks27.
Offenlegungen
Es bestehen keine konkurrierenden finanziellen Interessen.
Danksagungen
Die Entwicklung und Anwendung der hier beschriebenen Techniken wurde durch den zuschuss des brasilianischen Nationalen Forschungsrats 420811/2018-4 (LMC) unterstützt; Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) Zuschüsse 2019/16023-6 (LMC), 2019/17433-3 (LOF) und 21/01286-1 (MEME). Die Geldgeber spielten keine Rolle beim Studiendesign, der Datenerhebung und -analyse, der Entscheidung zur Veröffentlichung oder der Vorbereitung des Artikels.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
Referenzen
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223(1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312(2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150(2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093(2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188(2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321(2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , Chapter 18 13(2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross,, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten