Method Article
Préparation des échantillons et quantification relative à l’aide de la méthylation réductrice des amines pour les études peptidomiques
Dans cet article
Résumé
Cet article décrit une méthode de préparation d’échantillons basée sur l’inactivation thermique pour préserver les peptides endogènes en évitant la dégradation post-mortem, suivie d’une quantification relative utilisant le marquage isotopique plus LC-MS.
Résumé
La peptidomique peut être définie comme l’analyse qualitative et quantitative des peptides dans un échantillon biologique. Ses principales applications comprennent l’identification des biomarqueurs peptidiques de la maladie ou du stress environnemental, l’identification des neuropeptides, des hormones et des peptides intracellulaires bioactifs, la découverte de peptides antimicrobiens et nutraceutiques à partir d’hydrolysats de protéines, et peut être utilisé dans des études pour comprendre les processus protéolytiques. Les progrès récents dans la préparation des échantillons, les méthodes de séparation, les techniques de spectrométrie de masse et les outils informatiques liés au séquençage des protéines ont contribué à l’augmentation du nombre de peptides identifiés et des peptidomes caractérisés. Les études peptidomiques analysent fréquemment les peptides naturellement générés dans les cellules. Ici, un protocole de préparation d’échantillon basé sur l’inactivation thermique est décrit, ce qui élimine l’activité de la protéase et l’extraction dans des conditions douces, de sorte qu’il n’y a pas de clivage des liaisons peptidiques. En outre, la quantification relative des peptides à l’aide du marquage isotopique stable par méthylation réductrice des amines est également montrée. Cette méthode d’étiquetage présente certains avantages car les réactifs sont disponibles dans le commerce, peu coûteux par rapport aux autres, chimiquement stables et permettent l’analyse de jusqu’à cinq échantillons en une seule série LC-MS.
Introduction
Les sciences « omiques » sont caractérisées par l’analyse approfondie d’un ensemble de molécules, telles que l’ADN, l’ARN, les protéines, les peptides, les métabolites, etc. Ces ensembles de données générés à grande échelle (génomique, transcriptomique, protéomique, peptidomique, métabolomique, etc.) ont révolutionné la biologie et conduit à une compréhension avancée des processus biologiques1. Le terme peptidomique a commencé à être introduit au début du 20ème siècle, et certains auteurs l’ont appelé une branche de la protéomique2. Cependant, la peptidomique présente des particularités distinctes, où l’intérêt principal est d’étudier la teneur en peptides générés naturellement au cours des processus cellulaires, ainsi que la caractérisation de l’activité biologique de ces molécules3,4.
Initialement, les études sur les peptides bioactifs étaient limitées aux neuropeptides et aux peptides hormonaux par dégradation d’Edman et dosage radioimmunologique. Cependant, ces techniques ne permettent pas une analyse globale, en fonction de l’isolement de chaque peptide à des concentrations élevées, du temps de génération d’anticorps, en dehors de la possibilité de réactivité croisée5.
L’analyse peptidomique n’a été rendue possible qu’après plusieurs avancées dans la spectrométrie de masse couplée par chromatographie liquide (LC-MS) et des projets de génome qui ont fourni des pools de données complets pour les études protéomiques / peptidomics6,7. De plus, un protocole d’extraction peptidique spécifique pour les peptidomes devait être établi car les premières études qui ont analysé les neuropeptides à l’échelle mondiale dans des échantillons de cerveau ont montré que la détection était affectée par la dégradation massive des protéines, qui se produisent principalement dans ce tissu après 1 min post-mortem. La présence de ces fragments peptidiques masquait le signal neuropeptide et ne représentait pas le peptidome in vivo. Ce problème a été résolu principalement avec l’application de l’inactivation rapide par chauffage des protéases à l’aide de l’irradiation par micro-ondes, ce qui a considérablement réduit la présence de ces fragments d’artefacts et a permis non seulement l’identification de fragments de neuropeptides, mais a révélé la présence d’un ensemble de peptides de protéines cytosoliques, mitochondriales et nucléaires, différentes de degradome6,8,9.
Ces procédures méthodologiques ont permis une expansion du peptidome au-delà des neuropeptides bien connus, où des centaines de peptides intracellulaires générés principalement par l’action des protéasomes ont été identifiés dans la levure10, le poisson-zèbre11, les tissus de rongeurs12 et les cellules humaines13. Il a été largement démontré que des dizaines de ces peptides intracellulaires ont des activités biologiques et pharmacologiques14,15. En outre, ces peptides peuvent être utilisés comme biomarqueurs de la maladie et éventuellement avoir une signification clinique, comme démontré dans le liquide céphalo-rachidien chez les patients atteints d’anévrismes sacculaires intracrâniens16.
Actuellement, en plus de l’identification des séquences peptidiques, il est possible par spectrométrie de masse d’obtenir des données de quantification absolue et relative. Dans la quantification absolue, les niveaux de peptides dans un échantillon biologique sont comparés à des étalons synthétiques, tandis que dans la quantification relative, les niveaux de peptides sont comparés entre deux échantillons ou plus17. La quantification relative peut être effectuée à l’aide des approches suivantes : 1) « sans étiquette »18; 2) marquage métabolique in vivo ou 3) marquage chimique. Les deux derniers sont basés sur l’utilisation de formes isotopiques stables incorporées dans des peptides19,20. Dans l’analyse sans étiquette, les niveaux de peptides sont estimés en tenant compte de l’intensité du signal (comptage spectral) pendant le LC-MS18. Cependant, le marquage isotopique peut obtenir des niveaux relatifs plus précis de peptides.
De nombreuses études peptidomiques ont utilisé des réactifs de marquage du butyrate de triméthylammonium (TMAB) comme marquage chimique et, plus récemment, la méthylation réductrice des amines (RMA) avec des formes deutérées et non deutérées de formaldéhyde et de réactifs cyanoborohydrure de sodium ont été utilisées11,21,22. Cependant, les étiquettes TMAB ne sont pas disponibles dans le commerce et le processus de synthèse est très laborieux. D’autre part, dans le RMA, les réactifs sont disponibles dans le commerce, peu coûteux par rapport aux autres étiquettes, la procédure est simple à effectuer et les peptides marqués sont stables23,24.
L’utilisation de RMA consiste à former une base de Schiff en permettant aux peptides de réagir avec le formaldéhyde, suivie d’une réaction de réduction à travers le cyanoborohydrure. Cette réaction provoque la diméthylation des groupes aminés libres sur les chaînes latérales N-terminales et lysine et les monométhylates N-terminaux prolines. Comme les résidus de proline sont souvent rares sur le N-terminal, pratiquement tous les peptides avec des amines libres sur le N-terminus sont marqués avec deux groupes méthyle23,24,25.
Protocole
La procédure suivante pour l’extraction peptidique et la méthylation réductrice a été adaptée des procédures précédemment publiées24,25,26,27. Ce protocole suivait les directives du Conseil national pour le contrôle de l’expérimentation animale (CONCEA) et a été approuvé par la Commission d’éthique pour l’utilisation animale (CEUA) de l’Institut des biosciences de l’Université d’État de Sao Paulo. Les étapes du protocole sont illustrées à la figure 1.
REMARQUE: Préparer toutes les solutions aqueuses dans de l’eau ultrapure.
1. Extraction peptidique
- Culture cellulaire
- Cultiver des cellules SHSY5Y dans un plat de 15 cm à 37 °C sous 5% de CO2 dans le milieu Eagle modifié de Dulbecco contenant 15% de sérum bovin fœtal et 1% de pénicilline-streptomycine.
- Utilisez 2-3 plaques pour chaque échantillon. Cultivez les cellules jusqu’à 100% de confluence.
- Après le traitement prévu, lavez les cellules deux fois avec une solution saline tamponnée au phosphate. Ensuite, ajoutez 10 mL de solution saline tamponnée au phosphate, grattez les cellules et collectez dans un tube de 15 mL.
- Centrifuger à 800 x g pendant 5 min et retirer le surnageant. Remettre en suspension la pastille dans 1 mL d’eau désionisée ultra-purifiée à 80 °C.
- Transférer le contenu du tube (lysat cellulaire) dans un tube microfuge de 2 mL.
- Tissus animaux (principalement les tissus nerveux) :
- Anesthésier le Danio rerio (poisson zèbre) mâle adulte de type sauvage avec une dose létale de MS 222 (100 mg/L) et le soumettre immédiatement à 8 s de rayonnement micro-ondes pour inactiver la peptidase et la protéase.
REMARQUE: Un four à micro-ondes de type domestique peut être utilisé. Un micro-ondes de 900 W a été utilisé pendant 8 à 10 s à pleine puissance. Le micro-ondes utilisé doit pouvoir élever la température du cerveau à > 80 °C en 10 s. La reproductibilité du chauffage entre les échantillons serait également bénéfique en plaçant le tissu au même endroit dans le micro-ondes. - Après l’inactivation par la chaleur, recueillir tout le cerveau dans un tube de microfuge de 2 mL et congeler à −80 °C jusqu’à l’analyse.
- Remettre en suspension l’échantillon de tissu dans 1 mL d’eau désionisée ultra-purifiée à 80 °C. Soniquer le tissu avec une sonde en utilisant 30 impulsions (4 Hz) de 1 s.
NOTES: Pour les tissus hépatiques et rénaux, utilisez un homogénéisateur mécanique à 10 000-30 000 tr / min pendant 20 s. Pour les tissus musculaires, broyer le tissu dans de l’azote liquide à l’aide d’un creuset en porcelaine et d’un pilon. Les étapes suivantes sont les mêmes pour le lysat cellulaire ou le tissu homogénéate.
- Anesthésier le Danio rerio (poisson zèbre) mâle adulte de type sauvage avec une dose létale de MS 222 (100 mg/L) et le soumettre immédiatement à 8 s de rayonnement micro-ondes pour inactiver la peptidase et la protéase.
- Incuber le lysat cellulaire ou homogénérer le tissu à 80 °C pendant 20 min. Ensuite, refroidissez-le sur de la glace pendant 10 à 30 minutes.
- Ajouter 10 μL de solution mère HCl de 1 M pour chaque 1 mL de volume d’échantillon pour obtenir une concentration finale de 10 mM. Mélanger en vortexant pendant 20 s et incuber davantage sur de la glace pendant 15 min.
REMARQUE: Avant de s’acidifier, assurez-vous que l’échantillon est complètement refroidi pour éviter de briser les liaisons peptidiques causées par l’acidification à des températures élevées. - Centrifuger le lysat cellulaire ou le tissu homogénéisé à 12 000 x g à 4 °C pendant 15 min. Recueillir le surnageant dans des tubes de microcentrifugation protéique à faible liaison et le stocker à -80 °C.
- Nettoyez les dispositifs d’ultrafiltration (filtres coupe 10 kDa) en ajoutant de l’eau et centrifugez à 2300 x g pendant 3 min. Répétez cette étape deux fois de plus.
- Placer le surnageant dans les filtres de coupure prélavés de 10 kDa et centrifuger à 2 300 x g à 4 °C pendant 50 min dans une centrifugeuse réfrigérée. Le flow-through représente l’extrait peptidique.
- Dessalez les échantillons sur des colonnes de nettoyage en phase inversée conformément aux instructions du fabricant à l’aide de solutions d’acétonitrile (ACN) et d’acide trifluoroacétique (TFA) comme décrit ci-dessous:
- Équilibrez la colonne avec 1 mL de 100 % d’ACN.
- Laver la colonne avec une solution de 1 mL d’ACN à 5 % avec 0,1 % de TFA.
- Chargez le volume complet de l’échantillon dans la colonne.
- Laver la colonne avec une solution de 1 mL d’ACN à 5 % avec 0,1 % de TFA
- Éluez les peptides de la colonne avec une solution de 1,8 mL d’ACN à 100 % avec 0,15 % de TFA dans des tubes de microcentrifugation à faible liaison protéique.
- Sécher complètement l’échantillon dans une centrifugeuse à vide. Réglez la méthode de concentration des solvants organiques et la température à 30 °C. Le temps de concentration est surveillé à l’écran.
- Conservez les échantillons à −80 °C jusqu’à l’étape suivante.
2. Quantification peptidique avec de la fluorescamine
REMARQUE: La quantité de peptide peut être estimée en utilisant de la fluorescamine à pH 6,8 comme décrit précédemment11,28. Cette méthode consiste en la fixation d’une molécule de fluorescamine aux amines primaires présentes dans les résidus de lysine (K) et/ou le N-terminal des peptides. La réaction est réalisée à un pH de 6,8 pour garantir que la fluorescamine ne réagit qu’avec les groupes aminés des peptides et non avec les acides aminés libres. La fluorescamine est mesurée à l’aide d’un spectrofluoromètre à une longueur d’onde d’excitation de 370 nm et à une longueur d’onde d’émission de 480 nm.
- Préparer différentes concentrations du peptide étalon (0,05, 0,1, 0,15, 0,2, 0,3, 0,5 et 0,7 μg/μL) et conserver les aliquotes à -20 °C.
REMARQUE: Le peptide 5A (LTLRTKL) est suggéré car il a une composition et une concentration connues. - Préparer une solution mère de fluorescamine (0,3 mg/mL) dans de l’acétone. Aliquote rapidement dans des tubes de microcentrifugation (1 mL), sceller à l’aide d’un parafilm et conserver à -20 °C dans l’obscurité.
- Préparer un tampon phosphate (PB) de 0,2 M à un pH de 6,8.
REMARQUE : Préparer 0,2 M PB en ajoutant 0,1 M tampon phosphate pH 6,8 (26,85 mL de Na2HPO3 1M) et 0,1 M tampon phosphate pH 6,8 (23,15 mL de NaH2PO3 1M) à 250 mL d’eau - Remettre en suspension les échantillons peptidiques dans 100-200 μL d’eau ultra-purifiée.
- Pipette 2,5 μL des concentrations peptidiques standard et des échantillons sur la plaque blanche de 96 puits pour les essais de fluorescence en triple. Ajouter 25 μL de tampon phosphate de 0,2 M.
- Ajouter 12,5 μL de fluorescamine à l’aide d’une pipette multicanal. Homogénéiser doucement pendant 1 min sur le secoueur du rotateur orbital.
- Ensuite, ajoutez 110 μL d’eau avec une pipette multicanal pour arrêter la réaction.
REMARQUE: Transférer la solution mère de fluorescamine et l’eau ultrapure dans deux réservoirs pour pipeter ces solutions avec une pipette multicanal. - Ajustez les paramètres de lecture suivants sur le spectrofluoromètre : Lisez les échantillons par le haut, la longueur d’onde d’excitation à 370 nm et la longueur d’onde d’émission à 480 nm.
- Lisez la plaque sur le spectrofluoromètre.
3. Méthylation réductrice du marquage des amines
REMARQUE: Cette méthode de marquage isotopique est basée sur la diméthylation des groupes amines avec des formes deutérées et non deutérées de réactifs de formaldéhyde et de cyanoborohydrure de sodium. Le produit final de cette réaction ajoute 28 Da, 30 Da, 32 Da, 34 Da ou 36 Da à la masse finale de chaque peptide à chaque site de marquage disponible (lysine ou N-terminal). Cette réaction produit une différence m/z dans les peptides marqués avec différentes formes observées dans le spectre de la SEP (tableau 1).
ATTENTION : Un équipement de sécurité approprié doit être utilisé pour manipuler ces composés, et des précautions doivent être prises pour minimiser l’exposition. Les procédures avec du formaldéhyde et des réactifs cyanoborohydrure de sodium doivent être effectuées dans une hotte car ils sont très toxiques (y compris la pesée du cyanoborohydrure de sodium). Au cours de la réaction de trempe et d’acidification, un gaz toxique (cyanure d’hydrogène) peut être généré.
- Préparez les solutions fraîches suivantes à partir du stock ou des réactifs le jour de la procédure dans de l’eau ultrapure:
- Diluer le stock de formaldéhyde (CH2O) à 37%.
- Diluer le stock de formaldéhyde deutéré (CD2O) à 20%.
- Diluer le stock de formaldéhyde C13 deutéré de 20 % (13CD2O) à 4 %.
- Préparer 0,6 M NaBH3CN.
- Préparez 0,6 m de NaBD3CN.
- Préparer une solution de bicarbonate d’ammonium à 1%.
- Préparez une solution à 5% d’acide formique.
REMARQUE: En ce qui concerne la quantification relative des peptides, comme différents schémas expérimentaux peuvent être effectués en fonction du nombre d’étiquettes utilisées, une attention particulière est nécessaire lors de la procédure de marquage chimique. Il est recommandé de séparer les petites aliquotes des étiquettes dans des racks séparés avec les échantillons respectifs à étiqueter afin de réduire le risque d’erreur humaine lors de l’ajout du mauvais réactif aux tubes d’échantillonnage.
- Préparer chaque échantillon contenant jusqu’à 25 μg du peptide. Les échantillons ne doivent pas contenir de tris ou de bicarbonate d’ammonium.
REMARQUE: Les quantités décrites ci-dessous sont suffisantes pour chaque échantillon dans un volume de 100 μL.
Passez aux étapes 3.3 à 3.8 dans une hotte aspirante. - Ajouter 1/10e de volume de 1 M de TEAB aux échantillons (concentration finale de la solution 100 mM de TEAB). Vérifiez le pH avec un papier indicateur de pH; il doit être compris entre 5 et 8. Ajuster avec HCl ou NaOH si nécessaire.
- Ajouter 4 μL de formaldéhyde non deutéré, de deutéré ou de formaldéhyde deutéré en C13 selon le schéma d’étiquetage établi. Mélanger pendant 5 s par vortex.
- Ajouter 4 μL de NaBH3CN (0,6 M) ou de NaBD3CN (0,6 M) selon le schéma d’étiquetage établi. Mélanger pendant 5 s par vortex.
- Incuber dans une hotte pendant 2 h à température ambiante, en mélangeant toutes les 30 min.
- Répétez les étapes 3.4 et 3.5. Incuber les échantillons dans une hotte pendant la nuit à température ambiante.
- Ajouter 16 μL de bicarbonate d’ammonium (1%) et mélanger par vortex. Placer l’échantillon sur de la glace, ajouter 8 μL d’acide formique (5%) et mélanger par vortex pendant 5 s.
- Combinez les échantillons, ajustez le pH à 2-4 et dessalez les échantillons combinés sur des colonnes de nettoyage en phase inversée comme décrit précédemment à l’étape 1.8.
- Sécher complètement l’échantillon dans une centrifugeuse à vide. Réglez la méthode de concentration des solvants organiques et la température à 30 °C. Le temps de concentration est surveillé à l’écran.
- Conserver les échantillons à -20 °C.
4. Chromatographie liquide et spectrométrie de masse
- Effectuer une analyse LC-MS à l’aide d’un système nanoHPLC couplé à un instrument MS compatible avec les étiquettes méthyliques.
REMARQUE: Une variété d’instruments MS sont compatibles avec l’étiquetage RMA. Un système nanoHPLC couplé à Orbitrap est généralement utilisé pour effectuer une analyse LC-MS via une source d’ions de nanoélectropulture. Tout d’abord, l’échantillon est chargé dans une précolonne et les peptides séparés dans une colonne analytique. L’élution des peptides est réalisée en utilisant un gradient linéaire de 5% à 45% d’acétonitrile, dans de l’acide formique à 0,1%, pendant 90 min, avec un débit de 200 nL / min. Le spectromètre de masse est configuré pour fonctionner en mode dépendant des données. Chaque balayage complet est acquis à une intensité de 10-30 eV, 2,3 Kv, puis les dix pics les plus élevés sont sélectionnés pour la fragmentation par dissociation induite par collision (CID). Le temps d’injection est réglé sur le piège à ions à 100 ms, et l’injection de transformée de Fourier (FT)-MS est fixée avec une résolution de 1000 ms 30 000 à m/z 300-1800. Un minimum de 5000 comptes et une exclusion dynamique de 70 s sont utilisés pour effectuer l’analyse de fragmentation.
5. Quantification relative des peptides
REMARQUE: Les spectres MS sont analysés dans le logiciel du spectromètre de masse. Des groupes de pics de peptides marqués avec différentes étiquettes sont identifiés dans les spectres MS. La quantification relative est calculée par l’intensité de chaque pic monoisotopique. Chaque groupe traité est comparé au groupe témoin respectif.
- Double-cliquez avec le bouton droit de la souris sur le fichier d’échantillon brut pour ouvrir le logiciel d’analyse du spectre. Chargez les chromatogrammes temps de rétention (RT) et spectre MS (EM) dans deux onglets, en haut et en bas, respectivement.
- Cliquez une fois séquentiellement avec le bouton droit de la souris sur les icônes Options d’affichage et de masse de la barre d’outils du logiciel et définissez la précision de masse sur quatre décimales.
- Placez le curseur de la souris n’importe où sur l’onglet RT. Recherchez le temps de rétention de l’ion correspondant à analyser et cliquez avec le bouton droit de la souris. Le spectre MS de l’heure sélectionnée sera automatiquement affiché dans l’onglet EM.
- Placez le curseur de la souris n’importe où sur l’onglet EM. Recherchez les ions à analyser.
- Cliquez avec le bouton droit de la souris et maintenez-le enfoncé dans une région adjacente à gauche près de ces ions. Ensuite, faites glisser la souris vers la droite à la plage souhaitée pour zoomer sur la région d’intérêt.
- Gardez la souris positionnée sur l’onglet EM et cliquez sur les flèches droite ou gauche du clavier pour définir la plage d’ions à analyser.
- Placez à nouveau le curseur de la souris sur l’onglet RT au début de l’intervalle de temps souhaité.
- Cliquez avec le bouton droit de la souris et faites glisser la souris jusqu’à la valeur de temps choisie. Laissez le bouton. L’intensité accumulée des ions sera automatiquement affichée sur l’onglet EM.
- Collectez les données d’intensité m/z, z et ionique sur une feuille de calcul.
REMARQUE: La masse monoisotopique de chaque peptide sans groupes méthyle ajoutés est calculée à partir de la formule suivante:
Masse peptide non modifié = (m/z a x z) - (C a x T) - (1,008 x z)
m/zaest la valeur de masse à charge observée pour le pic monoisotopique pour chaque peptide marqué avec différentes combinaisons de marqueurs (a = 1, 2, 3, 4 ou 5, correspondant au numéro de l’échantillon).
z est l’état de charge.
Ca est la masse monoisotopique d’une paire de groupes méthyle :
Pour a=1, Ca = 28,0313 (l’addition nette de deux groupes CH3 à l’amine primaire)
Pour a=2, Ca = 30,0439 pour deux groupes CHD2
Pour a=3, Ca = 32,0564 pour deux groupes CD2H
Pour a=4, Ca= 34,0690 pour deux groupes CD3
Pour a=5, Ca = 36,0757 pour deux groupes de 13 CD3
T est le nombre de paires de groupes méthyle incorporés dans le peptide. Cela peut être calculé à partir de la formule suivante lorsque cinq balises sont utilisées : T=z*(m/z5 - m/z1)/8. Pour les peptides qui contiennent une seule amine primaire et qui sont donc marqués avec seulement deux groupes méthyles présents des chevauchements de pic sur les spectres MS lorsque des étiquettes adjacentes sont utilisées. L’intensité maximale de chaque peptide marqué peut être corrigée à l’aide des équations décrites par Tashima et Fricker25.
6. Identification des peptides
- Pour identifier les peptides, analysez les données MS/MS à l’aide d’un moteur de recherche de base de données29,30.
- Pour calculer le taux de fausse découverte (FDR) à l’aide de la méthode de fusion de leurres, effectuez une recherche dans une base de données de leurres.
REMARQUE: Les paramètres de recherche généralement utilisés ne sont pas spécifiques aux enzymes; tolérance de masse de précurseur fixée à 15-50 ppm; tolérance de masse d’ions fragment de 0,5 Da; modifications variables : amines réactives issues des résidus de Lys et N-terminus des peptides isotopiques méthylés (L1 (+28), L2 (+30), L3 (+32), L4 (+34) et L5 (+36)), méthionine oxydée (+15,99 Da) et acétylation (+42,01 Da). - Ensuite, triez les peptides en fonction de leur moyenne de confiance locale pour sélectionner les meilleurs spectres à annoter et filtrez-les par FDR ≤5%.

Figure 1 : Flux de travail des études peptidomiques. Étapes de l’extraction peptidique et de la méthylation réductrice des amines. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Les résultats obtenus à partir des exécutions effectuées sur le spectromètre de masse sont stockés dans des fichiers de données brutes qui peuvent être ouverts dans le logiciel du spectromètre de masse. Dans les spectres MS, il est possible d’observer des groupes de pics représentant des peptides marqués selon le schéma de marquage utilisé, allant de 2 à 5 étiquettes. Par exemple, dans la figure 2, des paires de pics détectés dans un temps chromatographique sont représentées dans une expérience où seulement deux étiquettes isotopiques ont été utilisées dans deux échantillons différents dans la même série. La figure 3 montre d’autres possibilités de résultats positifs, en utilisant 3 et 4 étiquettes différentes dans chaque exécution LC-MS. Lors de l’utilisation de 4 ou 5 étiquettes dans une série LC/MS, il peut y avoir un chevauchement des pics des peptides marqués avec les différentes étiquettes qui doivent être corrigées pour obtenir la valeur d’intensité réelle de chaque pic (Figure 4).
Le marquage isotopique peut également être utilisé pour montrer des substrats et des produits in vitro pour une protéase ou une peptidase donnée, comme le montre la figure 5. Enfin, différents logiciels peuvent être utilisés pour identifier les peptides étiquetés, tels que Peaks Studio ou MASCOT. Ces applications logicielles ont été créées pour l’analyse protéomique; par conséquent, les données de quantification des protéines ne doivent pas être prises en compte pour l’analyse peptidomique, et chaque peptide marqué identifié dans les paramètres de fiabilité doit être vérifié puis quantifié. Si les peptides ont été détectés et marqués avec succès, ces programmes fourniront une liste de séquences peptidiques identifiées contenant les étiquettes. La figure 6 montre un exemple de l’identification d’une séquence peptidique effectuée par le programme. Dans ce cas, seules 3 formes différentes d’étiquettes (L1, L3 et L5) ont été utilisées pour étiqueter séparément trois échantillons différents, qui ont ensuite été mélangés et analysés par spectrométrie de masse en une seule fois.
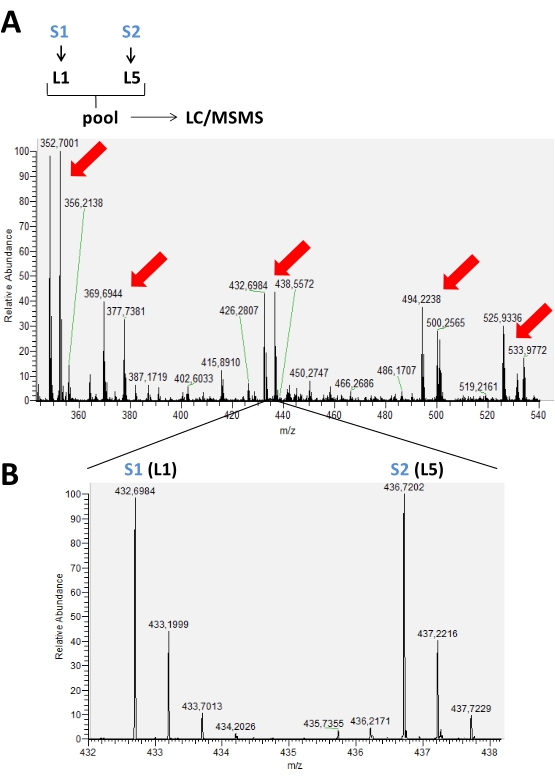
Figure 2 : Spectre MS représentatif d’un temps chromatographique accumulé dans une expérience de marquage typique utilisant la diméthylation réductrice des amines. Dans (A), les flèches rouges indiquent la présence de paires de pics de différents peptides marqués avec 2 formes isotopiques (L1 et L5) pour la comparaison entre deux échantillons différents (S1 et S2). En (B), image agrandie d’un spectre MS du même peptide montrant des m/z différents en raison de l’utilisation d’étiquettes. Dans ce cas, il n’y avait aucune variation dans l’intensité maximale de ce peptide présent dans ces échantillons. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Spectre représentatif de la SEP de peptides marqués avec méthylation réductrice des amines à l’aide de différents nombres de marqueurs. (A) Un marquage triplex a été utilisé. Il est possible d’observer un spectre MS d’un peptide présent dans 3 échantillons différents (S1, S2 et S3) marqués respectivement avec des étiquettes L1, L3 et L5. Dans ce cas, le niveau du peptide marqué avec L5 était deux fois le niveau observé pour le même peptide marqué avec des étiquettes L1 et L3. (B) Un étiquetage quadripex a été effectué à l’aide des étiquettes L1, L2, L3 et L4. Dans ce cas, les échantillons témoins (S1 et S3) ont été étiquetés avec L1 et L3, respectivement, et comparés à deux échantillons expérimentaux (S2 et S4) étiquetés avec des étiquettes L2 et L4. Aucune différence significative n’a été observée pour ce peptide entre les échantillons. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Spectre MS représentatif d’un peptide marqué présentant un chevauchement de pic. Cette figure montre le spectre MS d’un peptide de charge 3, masse de 2098,87 Da avec une seule amine primaire disponible pour le marquage. La différence entre les peptides marqués n’est que de 2 Da, ce qui provoque un chevauchement lorsque 4 ou 5 étiquettes sont utilisées dans la même série LC/MS. En utilisant un modèle basé sur des équations polynomiales cubiques, il est possible de corriger ces chevauchements entre les étiquettes. Dans le graphique, les barres rouges montrent la moyenne des valeurs d’intensité ajustées pour ce peptide en deux séries. Les barres noires montrent la moyenne des valeurs d’intensité qui se chevauchent. Après correction, ce peptide a montré peu de variation d’intensité entre les échantillons (barres rouges). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Spectre MS représentatif de peptides marqués avec méthylation réductrice des amines dans une étude de protéolyse. Ici, des extraits peptidiques ont été incubés avec 200 nM et 20 nM de neurolysine pour caractériser leurs substrats et produits. Pour confirmer le résultat, deux exécutions LC/MS avec des stratégies d’étiquetage forward et Reverse ont été effectuées. Dans la deuxième exécution, la position des échantillons est modifiée dans la procédure d’étiquetage par rapport au schéma utilisé lors de la première exécution. Dans A, on montre un peptide qui ne change pas (NC) en présence de l’enzyme neurolysine. Dans B, un peptide qui a disparu en présence d’une concentration élevée de l’enzyme (S2) et qui a montré une petite réduction de la faible concentration de l’enzyme (S4) dans le marquage avant et arrière. Ce peptide est considéré comme un substrat (SB) de l’enzyme. En C un exemple d’un peptide qui a été considéré comme un produit (PD), car sa concentration a été augmentée (S2 et S4) en présence de l’enzyme. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Spectre représentatif des HARSAH et identification d’un peptide marqué effectuée par un moteur de recherche de base de données. Dans cet exemple, il est possible d’observer 2+ ions avec m/z 672,3802, 676,4045 et 680,4241 correspondant à un peptide de même masse marqué avec les formes méthylées L1, L3 et L5, respectivement. Cette séquence ADQVSASLAKQGL a été identifiée à travers le spectre MS/MS comme un fragment de la protéine tau X1 associée aux microtubules. Ce peptide a le N-terminal et une lysine disponibles comme sites de marquage ajoutant une différence de masse de 8 Daltons entre les peptides marqués. Le spectre de sep-ès de ce peptide est illustré à la figure 3A. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Échantillon | H2CO | D2CO | D213CO | NaBH3CN | NaBD3CN | Étiquette | Masse supplémentaire |
| 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | code | (Da) | |
| 1 | X | X | L1 | 28.0313 | |||
| 2 | X | X | L2 | 30.0439 | |||
| 3 | X | X | L3 | 32.0564 | |||
| 4 | X | X | L4 | 34.069 | |||
| 5 | X | X | L5 | 36.0757 |
Tableau 1 : Réactifs d’une expérience typique utilisant la combinaison indiquée de formes dentérées et non deutérées de formaldéhyde et de cyanoborohydrure de sodium.
Discussion
Dans la plupart des études peptidomiques, l’une des étapes critiques est, sans aucun doute, la préparation de l’échantillon qui doit être soigneusement effectuée pour éviter la présence de fragments peptidiques générés par les protéases après quelques minutes post-mortem. Les premières études sur les extraits cérébraux préparés à partir d’échantillons non micro-ondes ont montré la présence d’un grand nombre de fragments de protéines dans les microfiltrats de 10 kDa. Différentes approches ont été décrites pour éviter les spectres peptidiques de la dégradation des protéines : irradiation micro-ondes ciblée sacrifice d’animal6,8, dissection de cryostat suivie d’un tampon d’extraction bouillant31, et irradiation micro-ondes post-sacrifice des tissus à l’aide d’un four à micro-ondes de type domestique9,26 . Pour la culture cellulaire et certains tissus, l’inactivation de la protéase peut se faire directement en ajoutant de l’eau à 80 °C. Cependant, certains échantillons, tels que le tissu nerveux, peuvent être plus sensibles au changement post-mortem, et l’inactivation de la protéase par irradiation par micro-ondes a été indiquée comme méthode de choix. De plus, un autre point important lors de l’extraction peptidique est de s’assurer que les extraits sont glacés avant d’ajouter de l’acide pour empêcher les liaisons acide-labile de se briser, comme le clivage des liaisons Asp-Pro26.
Différentes stratégies peuvent être utilisées pour la quantification relative des peptides, mais aucune d’entre elles ne peut être considérée comme complètement idéale. Pour choisir la méthode à utiliser, le chercheur doit tenir compte de facteurs tels que la disponibilité des heures d’utilisation dans le spectromètre de masse, la disponibilité commerciale et les coûts d’étiquetage des réactifs, ainsi que la facilité d’analyse des données obtenues17,25,26,32. La méthode sans étiquette a été largement utilisée mais nécessite de nombreuses heures dans le spectromètre de masse. Il est nécessaire d’injecter des répliques techniques pour chaque échantillon et dépend de la reproductibilité chromatographique entre les échantillons. D’autres réactifs de marquage chimique, par exemple l’ITRAQ (étiquettes isobares pour la quantification relative et absolue) et le TMT (étiquette de masse en tandem), sont coûteux et ne fournissent que la quantification des peptides sélectionnés pour l’analyse MS/MS25,33,34.
La principale limite de la quantification relative par marquage chimique à l’aide de RMA est le chevauchement qui se produit pour certains peptides lors de l’exécution d’un protocole utilisant 4 ou 5 étiquettes à la même exécution, ce qui entraîne des différences de masse de 2 Da pour les peptides avec une seule amine primaire et de 1 Da pour les peptides avec de la proline N-terminus et aucun résidu de lysine interne. Cependant, Tashima et Fricker (2018) ont développé un modèle pour corriger le chevauchement isotopique basé sur des équations polynomiales cubiques25, qui obtiennent l’intensité correcte des peptides marqués dans les échantillons. De plus, tous les peptides ne peuvent pas être vus par RMA. Par exemple, certains peptides manquent d’une amine libre N-terminale en raison de l’acétylation, de la pyroglutamylation ou d’une autre modification. Si les lysines internes sont également absentes de ces peptides, elles ne seront pas marquées par le réactif RMA et apparaîtront sur les spectres m/z comme des pics uniques non quantifiables27.
Déclarations de divulgation
Il n’existe pas d’intérêts financiers concurrents.
Remerciements
Le développement et l’utilisation des techniques décrites ici ont été soutenus par la subvention du Conseil national brésilien de la recherche 420811/2018-4 (LMC); Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) subventions 2019/16023-6 (LMC), 2019/17433-3 (LOF) et 21/01286-1 (MEME). Les bailleurs de fonds n’ont joué aucun rôle dans la conception de l’étude, la collecte et l’analyse des données, la décision de publier ou la préparation de l’article.
matériels
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
Références
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223(1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312(2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150(2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093(2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188(2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321(2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , Chapter 18 13(2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross,, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon