Method Article
Preparazione del campione e quantificazione relativa mediante metilazione riduttiva delle ammine per studi peptidomici
In questo articolo
Riepilogo
Questo articolo descrive un metodo di preparazione del campione basato sull'inattivazione termica per preservare i peptidi endogeni evitando la degradazione post-mortem, seguito dalla quantificazione relativa utilizzando l'etichettatura isotopica più LC-MS.
Abstract
La peptidomica può essere definita come l'analisi qualitativa e quantitativa dei peptidi in un campione biologico. Le sue principali applicazioni includono l'identificazione dei biomarcatori peptidici della malattia o dello stress ambientale, l'identificazione di neuropeptidi, ormoni e peptidi intracellulari bioattivi, la scoperta di peptidi antimicrobici e nutraceutici da idrolizzati proteici e può essere utilizzato in studi per comprendere i processi proteolitici. Il recente progresso nella preparazione dei campioni, nei metodi di separazione, nelle tecniche di spettrometria di massa e negli strumenti computazionali relativi al sequenziamento delle proteine ha contribuito all'aumento del numero di peptidi identificati e delle peptidome caratterizzate. Gli studi peptidomici analizzano frequentemente i peptidi che sono generati naturalmente nelle cellule. Qui viene descritto un protocollo di preparazione del campione basato sull'inattivazione del calore, che elimina l'attività della proteasi e l'estrazione con condizioni lievi, quindi non vi è scissione dei legami peptidici. Inoltre, viene mostrata anche la quantificazione relativa dei peptidi utilizzando l'etichettatura isotopica stabile mediante metilazione riduttiva delle ammine. Questo metodo di etichettatura presenta alcuni vantaggi in quanto i reagenti sono disponibili in commercio, poco costosi rispetto ad altri, chimicamente stabili e consentono l'analisi di un massimo di cinque campioni in una singola esecuzione LC-MS.
Introduzione
Le scienze "omiche" sono caratterizzate dall'analisi approfondita di un insieme di molecole, come DNA, RNA, proteine, peptidi, metaboliti, ecc. Questi set di dati generati su larga scala (genomica, trascrittomica, proteomica, peptidomica, metabolomica, ecc.) hanno rivoluzionato la biologia e portato a una comprensione avanzata dei processi biologici1. Il termine peptidomico cominciò ad essere introdotto all'inizio del 20 ° secolo e alcuni autori si sono riferiti ad esso come una branca della proteomica2. Tuttavia, la peptidomica ha peculiarità distinte, dove l'interesse principale è quello di indagare il contenuto di peptidi generati naturalmente durante i processi cellulari, nonché la caratterizzazione dell'attività biologica di queste molecole3,4.
Inizialmente, gli studi sui peptidi bioattivi erano limitati ai neuropeptidi e ai peptidi ormonali attraverso la degradazione di Edman e il test radioimmunologico. Tuttavia, queste tecniche non consentono un'analisi globale, a seconda dell'isolamento di ciascun peptide in alte concentrazioni, del tempo per la generazione di anticorpi, oltre alla possibilità di cross-reattività5.
L'analisi peptidomica è stata resa possibile solo dopo diversi progressi nella spettrometria di massa accoppiata a cromatografia liquida (LC-MS) e nei progetti sul genoma che hanno fornito pool di dati completi per studi di proteomica/peptidomica6,7. Inoltre, è stato necessario stabilire uno specifico protocollo di estrazione peptidica per le peptidome perché i primi studi che hanno analizzato i neuropeptidi a livello globale in campioni di cervello hanno dimostrato che il rilevamento era influenzato dalla massiccia degradazione delle proteine, che si verificano principalmente in questo tessuto dopo 1 minuto post-mortem. La presenza di questi frammenti peptidici mascherava il segnale neuropeptide e non rappresentava il peptidome in vivo. Questo problema è stato risolto principalmente con l'applicazione dell'inattivazione a riscaldamento rapido delle proteasi mediante irradiazione a microonde, che ha ridotto drasticamente la presenza di questi frammenti di artefatti e ha permesso non solo l'identificazione di frammenti neuropeptidici ma ha rivelato la presenza di un insieme di peptidi da proteine citosoliche, mitocondriali e nucleari, diverse da degradome6,8,9.
Queste procedure metodologiche hanno permesso un'espansione del peptidome oltre i ben noti neuropeptidi, dove centinaia di peptidi intracellulari generati principalmente dall'azione dei proteasomi sono stati identificati nel lievito10, nel pesce zebra11, nei tessuti dei roditori12 e nelle cellule umane13. Dozzine di questi peptidi intracellulari hanno ampiamente dimostrato di avere attività sia biologiche che farmacologiche14,15. Inoltre, questi peptidi possono essere utilizzati come biomarcatori di malattia e possibilmente hanno un significato clinico, come dimostrato nel liquido cerebrospinale da pazienti con aneurismi sacculari intracranici16.
Attualmente, oltre all'identificazione di sequenze peptidiche, è possibile attraverso la spettrometria di massa ottenere dati di quantificazione assoluta e relativa. Nella quantificazione assoluta, i livelli peptidici in un campione biologico vengono confrontati con gli standard sintetici, mentre nella relativa quantificazione, i livelli peptidici vengono confrontati tra due o più campioni17. La quantificazione relativa può essere eseguita utilizzando i seguenti approcci: 1) "label free"18; 2) etichettatura metabolica in vivo o 3) etichettatura chimica. Gli ultimi due si basano sull'uso di forme isotopiche stabili incorporate nei peptidi19,20. Nell'analisi senza etichetta, i livelli di peptidi sono stimati considerando la potenza del segnale (conteggi spettrali) durante l'LC-MS18. Tuttavia, l'etichettatura isotopica può ottenere livelli relativi più accurati di peptidi.
Molti studi peptidomici hanno utilizzato reagenti di etichettatura del butirrato trimetilammonio (TMAB) come etichettatura chimica e, più recentemente, sono stati utilizzati la metilazione riduttiva delle ammine (RMA) con forme deuterate e non deuterate di reagenti di formaldeide e cianoboroidruro di sodio11,21,22. Tuttavia, le etichette TMAB non sono disponibili in commercio e il processo di sintesi è molto laborioso. D'altra parte, nell'RMA, i reagenti sono disponibili in commercio, poco costosi rispetto ad altre etichette, la procedura è semplice da eseguire e i peptidi etichettati sono stabili23,24.
L'uso di RMA comporta la formazione di una base di Schiff consentendo ai peptidi di reagire con la formaldeide, seguita da una reazione di riduzione attraverso la cianoboroidruro. Questa reazione provoca la dimetilazione di gruppi amminici liberi su N-terminali e catene laterali di lisina e monometilati Proline N-terminali. Come i residui di prolina sono spesso rari sul N-terminale, praticamente tutti i peptidi con ammine libere sul N-terminus sono etichettati con due gruppi metilici23,24,25.
Protocollo
La seguente procedura per l'estrazione peptidica e la metilazione riduttiva è stata adattata dalle procedure precedentemente pubblicate24,25,26,27. Questo protocollo ha seguito le linee guida del Consiglio nazionale per il controllo della sperimentazione animale (CONCEA) ed è stato approvato dalla Commissione etica per l'uso animale (CEUA) presso l'Istituto di bioscienze dell'Università statale di San Paolo. I passaggi del protocollo sono illustrati nella Figura 1.
NOTA: Preparare tutte le soluzioni acquose in acqua ultrapura.
1. Estrazione peptidica
- Coltura cellulare
- Coltiva le cellule SHSY5Y in un piatto di 15 cm a 37 °C sotto il 5% di CO2 nel mezzo dell'Aquila modificato di Dulbecco contenente il 15% di siero bovino fetale e l'1% di penicillina-streptomicina.
- Utilizzare 2-3 piastre per ogni campione. Far crescere le cellule al 100% di confluenza.
- Dopo il trattamento previsto, lavare le cellule due volte con soluzione salina tamponata con fosfato. Quindi, aggiungere 10 ml di soluzione salina tamponata con fosfato, raschiare le cellule e raccogliere in un tubo da 15 ml.
- Centrifugare a 800 x g per 5 min e rimuovere il surnatante. Risospesare il pellet in 1 mL di acqua deionizzata ultrapurata a 80 °C.
- Trasferire il contenuto del tubo (lisato cellulare) in un tubo da microfuga da 2 ml.
- Tessuti animali (principalmente tessuto nervoso):
- Anestetizzare Danio rerio (zebrafish) maschio selvatico con una dose letale di MS 222 (100 mg/L) e sottoporlo immediatamente a 8 s di radiazioni a microonde per inattivare peptidasi e proteasi.
NOTA: è possibile utilizzare un forno a microonde di tipo domestico. Un forno a microonde da 900 W è stato utilizzato per 8-10 s a piena potenza. Il forno a microonde utilizzato deve essere in grado di aumentare la temperatura cerebrale a > 80 °C entro 10 s. La riproducibilità nel riscaldamento tra i campioni trarrebbe vantaggio anche dal posizionamento del tessuto nella stessa posizione nel microonde. - Dopo l'inattivazione del calore, raccogliere l'intero cervello in un tubo di microfuga da 2 ml e congelare a -80 °C fino all'analisi.
- Sospendere il campione di tessuto in 1 mL di acqua deionizzata ultra-purificata a 80 °C. Sonicare il tessuto con una sonda utilizzando 30 impulsi (4 Hz) di 1 s.
NOTE: Per i tessuti epatici e renali, utilizzare un omogeneizzatore meccanico a 10.000-30.000 giri /min per 20 s. Per i tessuti muscolari, macinare il tessuto in azoto liquido usando un crogiolo di porcellana e un pestello. I seguenti passaggi sono gli stessi per il lisato cellulare o il tessuto omogeneizzato.
- Anestetizzare Danio rerio (zebrafish) maschio selvatico con una dose letale di MS 222 (100 mg/L) e sottoporlo immediatamente a 8 s di radiazioni a microonde per inattivare peptidasi e proteasi.
- Incubare il tessuto cellulare lisato o omogeneizzato a 80 °C per 20 min. Quindi, raffreddarlo sul ghiaccio per 10-30 minuti.
- Aggiungere 10 μL di soluzione madre da 1 M HCl per ogni 1 mL di volume del campione per ottenere una concentrazione finale di 10 mM. Mescolare vorticosamente per 20 s e incubare ulteriormente sul ghiaccio per 15 min.
NOTA: Prima di acidificare, assicurarsi che il campione sia completamente raffreddato per evitare di rompere i legami peptidici causati dall'acidificazione a temperature elevate. - Centrifugare il lisato cellulare o il tessuto omogeneizzato a 12.000 x g a 4 °C per 15 min. Raccogliere il surnatante in provette di microcentrifuga proteica a basso legame e conservarlo a -80 °C.
- Pulire i dispositivi di ultrafiltrazione (filtri cut-off da 10 kDa) aggiungendo acqua e centrifugare a 2300 x g per 3 min. Ripetere questo passaggio altre due volte.
- Posizionare il surnatante nei filtri di taglio prelavati da 10 kDa e centrifugare a 2.300 x g a 4 °C per 50 minuti in una centrifuga refrigerata. Il flow-through rappresenta l'estratto peptidico.
- Dissalare i campioni su colonne di pulizia in fase inversa secondo le istruzioni del produttore utilizzando soluzioni di acetonitrile (ACN) e acido trifluoroacetico (TFA) come descritto di seguito:
- Equilibrare la colonna con 1 mL di 100% ACN.
- Lavare la colonna con 1 mL di soluzione al 5% di ACN con 0,1% di TFA.
- Caricare il volume completo dell'esempio nella colonna.
- Lavare la colonna con 1 mL di soluzione al 5% di ACN con 0,1% di TFA
- Eluire i peptidi dalla colonna con una soluzione da 1,8 mL di ACN al 100% con TFA allo 0,15% in tubi microcentrifuga a basso legame proteico.
- Asciugare completamente il campione in una centrifuga sottovuoto. Impostare il metodo di concentrazione per i solventi organici e la temperatura a 30 °C. Il tempo di concentrazione viene monitorato in mostra.
- Conservare i campioni a -80°C fino al passaggio successivo.
2. Quantificazione peptidica con fluorescamina
NOTA: La quantità di peptide può essere stimata utilizzando fluorescamina a pH 6,8 come descritto in precedenza11,28. Questo metodo consiste nell'attaccamento di una molecola di fluorescamina alle ammine primarie presenti nei residui di lisina (K) e/o nell'N-terminale dei peptidi. La reazione viene eseguita a pH 6,8 per garantire che la fluorescamina reagisca solo con i gruppi amminici dei peptidi e non con gli amminoacidi liberi. La fluorescamina viene misurata utilizzando uno spettrofluorometro a una lunghezza d'onda di eccitazione di 370 nm e una lunghezza d'onda di emissione di 480 nm.
- Preparare diverse concentrazioni del peptide standard (0,05, 0,1, 0,15, 0,2, 0,3, 0,5 e 0,7 μg/μL) e conservare le aliquote a -20 °C.
NOTA: Il peptide 5A (LTLRTKL) è suggerito poiché ha una composizione e una concentrazione note. - Preparare la soluzione madre di fluorescamina (0,3 mg/ml) in acetone. Aliquota rapida in tubi microcentrifuga (1 mL), sigillare con parafilm e conservare a -20 °C al buio.
- Preparare il tampone fosfato 0,2 M (PB) a pH 6,8.
NOTA: Preparare 0,2 M PB aggiungendo 0,1 M tampone fosfato pH 6,8 (26,85 mL di Na2HPO3 1M) e 0,1 M tampone fosfato pH 6,8 (23,15 mL di NaH2PO3 1M) a 250 mL di acqua - Sospendere i campioni peptidici in 100-200 μL di acqua ultra-purificata.
- Pipettare 2,5 μL delle concentrazioni peptidiche standard e campioni sulla piastra bianca a 96 pozzetti per saggi di fluorescenza in triplice copia. Aggiungere 25 μL di tampone fosfato da 0,2 M.
- Aggiungere 12,5 μL di fluorescamina con una pipetta multicanale. Omogeneizzare delicatamente per 1 minuto sullo shaker del rotatore orbitale.
- Quindi, aggiungere 110 μL di acqua con una pipetta multicanale per fermare la reazione.
NOTA: Trasferire la soluzione madre di fluorescamina e l'acqua ultrapura in due serbatoi per pipettare queste soluzioni con una pipetta multicanale. - Regolare i seguenti parametri di lettura sullo spettrofluorometro: leggere i campioni dall'alto, la lunghezza d'onda di eccitazione a 370 nm e la lunghezza d'onda di emissione a 480 nm.
- Leggere la piastra sullo spettrofluorometro.
3. Metilazione riduttiva dell'etichettatura delle ammine
NOTA: Questo metodo di marcatura isotopica si basa sulla dimetilazione di gruppi amminici con forme deuterate e non deuterate di reagenti di formaldeide e cianoboroidruro di sodio. Il prodotto finale di questa reazione aggiunge 28 Da, 30 Da, 32 Da, 34 Da o 36 Da alla massa finale di ciascun peptide in ogni sito di etichettatura disponibile (lisina o N-terminale). Questa reazione produce una differenza m/z nei peptidi marcati con diverse forme osservate nello spettro MS (Tabella 1).
ATTENZIONE: per maneggiare questi composti devono essere utilizzate adeguate attrezzature di sicurezza e si deve prestare attenzione per ridurre al minimo l'esposizione. Le procedure con reagenti di formaldeide e cianoboroidruro di sodio devono essere eseguite in una cappa aspirante perché sono molto tossiche (compresa la pesatura del cianoboroidruro di sodio). Durante la reazione di tempra e acidificazione, può essere generato un gas tossico (acido cianidrico).
- Preparare le seguenti soluzioni fresche dal brodo o dai reagenti il giorno della procedura in acqua ultrapura:
- Diluire lo stock di formaldeide al 37% (CH2O) al 4%.
- Diluire lo stock del 20% di formaldeide deuterata (CD2O) al 4%.
- Diluire lo stock di 20 % di formaldeide C13 deuterata (13CD2O) al 4%.
- Preparare 0,6 M NaBH3CN.
- Preparare 0,6 M NaBD3CN.
- Preparare una soluzione di bicarbonato di ammonio all'1%.
- Preparare una soluzione 5% acido formico.
NOTA: Per quanto riguarda la relativa quantificazione dei peptidi, poiché possono essere eseguiti diversi schemi sperimentali a seconda del numero di etichette utilizzate, è necessaria attenzione durante la procedura di etichettatura chimica. Si raccomanda di separare piccole aliquote delle etichette in rack separati con i rispettivi campioni da etichettare per ridurre il potenziale di errore umano nell'aggiungere il reagente sbagliato alle provette del campione.
- Preparare ogni campione contenente fino a 25 μg del peptide. I campioni non devono contenere Tris o Bicarbonato di Ammonio.
NOTA: Le quantità descritte di seguito sono sufficienti per ciascun campione in un volume di 100 μL.
Procedere con i passaggi 3.3-3.8 in una cappa aspirante. - Aggiungere 1/10 di volume di 1 M TEAB ai campioni (concentrazione finale della soluzione 100 mM di TEAB). Controllare il pH con una carta indicatore di pH; deve essere compreso tra 5-8. Regolare con HCl o NaOH se necessario.
- Aggiungere 4 μL di formaldeide deuterata, deuterata o formaldeide deuterata C13 secondo lo schema di etichettatura stabilito. Mescolare per 5 s per vortice.
- Aggiungere 4 μL di NaBH3CN (0,6 M) o NaBD3CN (0,6 M) secondo lo schema di etichettatura stabilito. Mescolare per 5 s per vortice.
- Incubare in una cappa aspirante per 2 ore a temperatura ambiente, mescolando ogni 30 min.
- Ripetere i passaggi 3.4 e 3.5. Incubare i campioni in una cappa aspirante durante la notte a temperatura ambiente.
- Aggiungere 16 μL di bicarbonato di ammonio (1%) e mescolare a vortice. Posizionare il campione sul ghiaccio, aggiungere 8 μL di acido formico (5%) e mescolare per vortice per 5 s.
- Combinare i campioni, regolare il pH a 2-4 e desalentare i campioni combinati su colonne di pulizia in fase inversa come descritto in precedenza nel passaggio 1.8.
- Asciugare completamente il campione in una centrifuga sottovuoto. Impostare il metodo di concentrazione per i solventi organici e la temperatura a 30 °C. Il tempo di concentrazione viene monitorato in mostra.
- Conservare i campioni a -20 °C.
4. Cromatografia liquida e spettrometria di massa
- Eseguire analisi LC-MS utilizzando un sistema nanoHPLC accoppiato a uno strumento MS compatibile con tag metilici.
NOTA: una varietà di strumenti MS è compatibile con l'etichettatura RMA. Un sistema nanoHPLC accoppiato a Orbitrap viene in genere utilizzato per eseguire analisi LC-MS attraverso una sorgente di ioni nanoelettrospray. In primo luogo, il campione viene caricato in una precolonna e i peptidi separati in una colonna analitica. L'eluizione dei peptidi viene eseguita utilizzando un gradiente lineare del 5%-45% di acetonitrile, in acido formico allo 0,1%, durante 90 min, con un flusso di 200 nL/min. Lo spettrometro di massa è impostato per funzionare in modalità dipendente dai dati. Ogni scansione completa viene acquisita ad un'intensità di 10-30 eV, 2,3 Kv, e quindi i dieci picchi più alti vengono selezionati per la frammentazione della dissociazione indotta da collisione (CID). Il tempo di iniezione è impostato sulla trappola ionica a 100 ms e l'iniezione di trasformata di Fourier (FT)-MS è fissata con una risoluzione di 1000 ms 30.000 a m/z 300-1800. Per eseguire la scansione a frammentazione vengono utilizzati un minimo di 5000 conteggi e un'esclusione dinamica di 70 s.
5. Quantificazione relativa dei peptidi
NOTA: gli spettri MS vengono analizzati nel software dello spettrometro di massa. Gruppi di picco di peptidi marcati con tag diversi sono identificati negli spettri MS. La quantificazione relativa è calcolata dall'intensità di ciascun picco monoisotopico. Ogni gruppo trattato viene confrontato con il rispettivo gruppo di controllo.
- Fare doppio clic con il pulsante destro del mouse sul file di esempio non elaborato per aprire il software di analisi dello spettro. Caricare i cromatogrammi del tempo di ritenzione (RT) e dello spettro MS (EM) in due schede, rispettivamente superiore e inferiore.
- Fare clic con il pulsante destro del mouse una volta in sequenza sulle icone delle opzioni Display e Mass nella barra degli strumenti del software e impostare la precisione di massa su quattro decimali.
- Posizionare il cursore del mouse in un punto qualsiasi della scheda RT. Cercare il tempo di ritenzione dello ione corrispondente da analizzare e fare clic con il pulsante destro del mouse. Lo spettro MS dell'ora selezionata verrà automaticamente visualizzato nella scheda EM.
- Posizionare il cursore del mouse in un punto qualsiasi della scheda EM. Cerca gli ioni da analizzare.
- Fare clic con il pulsante destro del mouse e tenere premuto in una regione adiacente a sinistra vicino a questi ioni. Quindi, trascina il mouse verso destra nell'intervallo desiderato per ingrandire la regione di interesse.
- Tenere il mouse posizionato sulla scheda EM e fare clic sulle frecce della tastiera destra o sinistra per definire la gamma di ioni da analizzare.
- Posizionare nuovamente il cursore del mouse sulla scheda RT all'inizio dell'intervallo di tempo desiderato.
- Fare clic con il pulsante destro del mouse e trascinare il mouse fino al valore temporale scelto. Lascia il pulsante. L'intensità accumulata degli ioni verrà automaticamente mostrata nella scheda EM.
- Raccogli i dati sull'intensità di m/z, z e ioni su un foglio di calcolo.
NOTA: La massa monoisotopica di ciascun peptide senza gruppi metilici aggiunti viene calcolata dalla seguente formula:
Peptide di massa non modificato = (m/z a x z) - (C a x T) - (1,008 x z)
m/zais la massa osservata al valore di carica per il picco monoisotopico per ogni peptide etichettato con diverse combinazioni di tag (a =1, 2, 3, 4 o 5, corrispondente al numero del campione).
z è lo stato di carica.
Ca è la massa monoisotopica di una coppia di gruppi metilici:
Per a=1, Ca = 28,0313 (l'aggiunta netta di due gruppi CH3 all'ammina primaria)
Per a=2, Ca = 30,0439 per due gruppi CHD2
Per a=3, Ca = 32,0564 per due gruppi CD2H
Per a=4, Ca= 34.0690 per due gruppi CD3
Per a=5, Ca = 36,0757 per due 13 gruppi CD3
T è il numero di coppie di gruppi metilici incorporati nel peptide. Questo può essere calcolato dalla seguente formula quando vengono utilizzati cinque tag: T = z * (m / z5 - m / z1) / 8. Per i peptidi che contengono una singola ammina primaria e quindi sono etichettati con solo due gruppi metilici presentano sovrapposizioni di picco sugli spettri MS quando vengono utilizzate etichette adiacenti. L'intensità di picco di ciascun peptide marcato può essere corretta utilizzando le equazioni descritte da Tashima e Fricker25.
6. Identificazione peptidica
- Per identificare i peptidi, analizzare i dati MS/MS utilizzando un motore di ricerca di database29,30.
- Per calcolare il tasso di falsa scoperta (FDR) utilizzando il metodo di fusione esca, cercare in un database esca.
NOTA: I parametri di ricerca generalmente utilizzati non sono specificità enzimatica; set di tolleranza di massa del precursore 15-50 ppm; tolleranza di massa ionica del frammento di 0,5 Da; modificazioni variabili: ammine reattive da residui di Lys e N-terminus delle etichette isotopiche metilate dei peptidi (L1 (+28), L2 (+30), L3 (+32), L4 (+34) e L5 (+36)), metionina ossidata (+15,99 Da) e acetilazione (+42,01 Da). - Quindi, ordina i peptidi in base alla loro media di confidenza locale per selezionare i migliori spettri da annotare e filtrarli per FDR ≤5%.

Figura 1: Flusso di lavoro degli studi peptidomici. Fasi di estrazione peptidica e metilazione riduttiva delle ammine. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
I risultati ottenuti dalle esecuzioni effettuate sullo spettrometro di massa sono memorizzati in file di dati grezzi che possono essere aperti nel software dello spettrometro di massa. Negli spettri MS, è possibile osservare gruppi di picco che rappresentano peptidi marcati secondo lo schema di etichettatura utilizzato, che vanno da 2-5 etichette. Ad esempio, nella Figura 2, coppie di picchi rilevati in un tempo cromatografico sono rappresentate in un esperimento in cui sono state utilizzate solo due etichette isotopiche in due campioni diversi nella stessa esecuzione. La Figura 3 mostra altre possibilità di risultati positivi, utilizzando 3 e 4 etichette diverse in ogni esecuzione LC-MS. Quando si utilizzano 4 o 5 etichette in una corsa LC / MS, potrebbe esserci una sovrapposizione di picchi dei peptidi marcati con i diversi tag che devono essere corretti per ottenere il valore di intensità reale di ciascun picco (Figura 4).
L'etichettatura isotopica può anche essere utilizzata per mostrare substrati e prodotti in vitro per una data proteasi o peptidasi, come mostrato nella Figura 5. Infine, è possibile utilizzare diversi software per identificare i peptidi etichettati, come Peaks Studio o MASCOT. Queste applicazioni software sono state create per l'analisi proteomica; pertanto, i dati di quantificazione delle proteine non devono essere considerati per l'analisi peptidomica e ogni peptide marcato identificato all'interno dei parametri di affidabilità deve essere controllato e quindi quantificato. Se i peptidi sono stati rilevati ed etichettati con successo, questi programmi forniranno un elenco di sequenze peptidiche identificate contenenti le etichette. La Figura 6 è mostrata un esempio dell'identificazione di una sequenza peptidica effettuata dal programma. In questo caso, solo 3 diverse forme delle etichette (L1, L3 e L5) sono state utilizzate per etichettare separatamente tre diversi campioni, che sono stati poi miscelati e analizzati mediante spettrometria di massa in un'unica esecuzione.
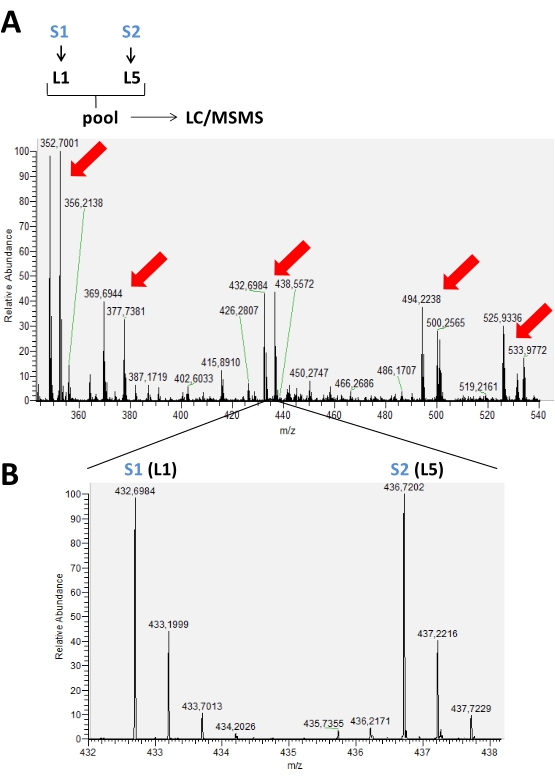
Figura 2: Spettro MS rappresentativo di un tempo cromatografico accumulato in un tipico esperimento di etichettatura utilizzando la dimetilazione riduttiva delle ammine. In (A), le frecce rosse indicano la presenza di coppie di picco di diversi peptidi etichettati con 2 forme isotopiche (L1 e L5) per il confronto tra due diversi campioni (S1 e S2). In (B), un'immagine ingrandita di uno spettro MS dello stesso peptide che mostra diversi m/z a causa dell'uso di etichette. In questo caso, non c'è stata alcuna variazione nell'intensità di picco per questo peptide presente in questi campioni. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Spettro rappresentativo ms di peptidi marcati con metilazione riduttiva delle ammine utilizzando diversi numeri di tag. (A) È stata utilizzata un'etichettatura triplex. È possibile osservare uno spettro MS di un peptide presente in 3 diversi campioni (S1, S2 e S3) etichettati rispettivamente con tag L1, L3 e L5. In questo caso, il livello del peptide etichettato con L5 era il doppio del livello osservato per lo stesso peptide etichettato con tag L1 e L3. (B) Un'etichettatura quadripessa è stata eseguita utilizzando le etichette L1, L2, L3 e L4. In questo caso, i campioni di controllo (S1 e S3) sono stati etichettati rispettivamente con L1 e L3 e confrontati con due campioni sperimentali (S2 e S4) etichettati con etichette L2 e L4. Non sono state osservate differenze significative per questo peptide tra i campioni. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Spettro rappresentativo della SM di un peptide marcato che presenta una sovrapposizione di picco. Questa figura mostra lo spettro MS di un peptide di carica 3, massa di 2098,87 Da con una singola ammina primaria disponibile per l'etichettatura. La differenza tra i peptidi etichettati è solo 2 Da, causando una sovrapposizione quando 4 o 5 etichette vengono utilizzate nella stessa corsa LC / MS. Utilizzando un modello basato su equazioni polinomiali cubiche, è possibile correggere queste sovrapposizioni tra le etichette. Nel grafico, le barre rosse mostrano la media dei valori di intensità regolati per questo peptide in due cicli. Le barre nere mostrano la media dei valori di intensità sovrapposti. Dopo la correzione, questo peptide ha mostrato una piccola variazione di intensità tra i campioni (barre rosse). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Spettro rappresentativo della SM di peptidi marcati con metilazione riduttiva delle ammine in uno studio di proteolisi. Qui, gli estratti peptidici sono stati incubati con 200 nM e 20 nM neurolisina per caratterizzare i loro substrati e prodotti. Per la conferma del risultato, sono state eseguite due esecuzioni LC/MS con strategie di etichettatura Forward e Reverse. Nella seconda esecuzione la posizione dei campioni viene modificata nella procedura di etichettatura in relazione allo schema utilizzato nella prima esecuzione. In A, viene mostrato un peptide che non cambia (NC) in presenza dell'enzima neurolisina. In B, un peptide che è scomparso in presenza di un'alta concentrazione dell'enzima (S2) e che ha mostrato una piccola riduzione della bassa concentrazione dell'enzima (S4) sia nell'etichettatura in avanti che in quella inversa. Questo peptide è considerato un substrato (SB) dell'enzima. In C un esempio di un peptide che è stato considerato un prodotto (PD), perché la sua concentrazione è stata aumentata (S2 e S4) in presenza dell'enzima. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Spettro MSMS rappresentativo e identificazione di un peptide etichettato eseguita da un motore di ricerca di database. In questo esempio, è possibile osservare ioni 2+ con m/z 672.3802, 676.4045 e 680.4241 corrispondenti a un peptide della stessa massa etichettato rispettivamente con le forme metilate L1, L3 e L5. Questa sequenza ADQVSASLAKQGL è stata identificata attraverso lo spettro MS/MS come frammento dell'isoforma X1 della proteina tau associata ai microtubuli. Questo peptide ha l'N-terminale e una lisina disponibili come siti di etichettatura aggiungendo una differenza di massa di 8 Dalton tra i peptidi marcati. Lo spettro MS di questo peptide è mostrato nella Figura 3A. Fare clic qui per visualizzare una versione più grande di questa figura.
| Campione | H2CO · | D2CO · | D213CO · | NaBH3CN | NaBD3CN | Etichetta | Messa aggiuntiva |
| 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | 2 x 4 μL | codice | (Da) | |
| 1 | X | X | L1 · | 28.0313 | |||
| 2 | X | X | L2 · | 30.0439 | |||
| 3 | X | X | L3 · | 32.0564 | |||
| 4 | X | X | L4 · | 34.069 | |||
| 5 | X | X | L5 · | 36.0757 |
Tabella 1: Reagenti di un esperimento tipico che utilizza la combinazione indicata di forme deuterate e non deuterate di formaldeide e cianoboroidruro di sodio.
Discussione
Nella maggior parte degli studi peptidomici, uno dei passaggi critici è, senza dubbio, la preparazione del campione che deve essere eseguita con attenzione per evitare la presenza di frammenti peptidici generati dalle proteasi dopo pochi minuti post-mortem. Gli studi iniziali su estratti cerebrali preparati da campioni non microonde hanno mostrato la presenza di un gran numero di frammenti proteici nei microfiltrati 10-kDa. Sono stati descritti diversi approcci per evitare spettri peptidici dalla degradazione delle proteine: sacrificio animale di irradiazione a microonde focalizzata6,8, dissezione criostatica seguita da un tampone di estrazione bollente31 e irradiazione a microonde post-sacrificio di tessuto utilizzando un forno a microonde di tipo domestico9,26 . Per la coltura cellulare e alcuni tessuti, l'inattivazione della proteasi può essere effettuata direttamente aggiungendo acqua a 80 °C. Tuttavia, alcuni campioni, come il tessuto nervoso, possono essere più sensibili al cambiamento post-mortem e l'inattivazione della proteasi mediante irradiazione a microonde è stata indicata come metodo di scelta. Inoltre, un altro punto importante durante l'estrazione del peptide è assicurarsi che gli estratti siano ghiacciati prima di aggiungere acido per evitare che i legami acido-labile si rompano, come la scissione dei legami Asp-Pro26.
Diverse strategie possono essere utilizzate per la quantificazione relativa dei peptidi, ma nessuna di esse può essere considerata completamente ideale. Per scegliere il metodo da utilizzare, il ricercatore deve considerare fattori come la disponibilità di ore di utilizzo nello spettrometro di massa, disponibili in commercio e i costi di etichettatura dei reagenti, e la facilità nell'analizzare i dati ottenuti17,25,26,32. Il metodo label-free è stato ampiamente utilizzato ma richiede molte ore nello spettrometro di massa. È necessario iniettare repliche tecniche per ciascun campione e dipende dalla riproducibilità cromatografica tra i campioni. Altri reagenti chimici di etichettatura, ad esempio ITRAQ (tag isobarici per la quantificazione relativa e assoluta) e TMT (tag di massa tandem), sono costosi e forniscono solo la quantificazione dei peptidi selezionati per l'analisi MS/MS25,33,34.
Il principale limite della quantificazione relativa attraverso l'etichettatura chimica utilizzando RMA è la sovrapposizione che si verifica per alcuni peptidi quando si esegue un protocollo utilizzando 4 o 5 etichette alla stessa corsa con conseguenti differenze di massa di 2 Da per i peptidi con una singola ammina primaria e 1 Da per i peptidi con prolina N-terminale e nessun residuo interno di lisina. Tuttavia, Tashima e Fricker (2018) hanno sviluppato un modello per correggere la sovrapposizione isotopica basata su equazioni polinomiali cubiche25, che ottengono l'intensità corretta dei peptidi marcati nei campioni. Inoltre, non tutti i peptidi possono essere visti da RMA. Ad esempio, alcuni peptidi mancano di un'ammina libera N-terminale a causa di acetilazione, piroglutamilazione o altra modifica. Se anche le lisine interne sono assenti in questi peptidi, non saranno etichettate dal reagente RMA e appariranno sugli spettri m/z come singoli picchi non quantificabili27.
Divulgazioni
Non esistono interessi finanziari concorrenti.
Riconoscimenti
Lo sviluppo e l'uso delle tecniche qui descritte sono stati supportati dalla sovvenzione del Consiglio Nazionale delle Ricerche brasiliano 420811/2018-4 (LMC); Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) sovvenzioni 2019/16023-6 (LMC), 2019/17433-3 (LOF) e 21/01286-1 (MEME). I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio, nella raccolta e nell'analisi dei dati, nella decisione di pubblicare o nella preparazione dell'articolo.
Materiali
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
Riferimenti
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223(1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312(2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150(2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093(2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188(2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321(2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , Chapter 18 13(2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross,, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon