Method Article
Identificação Global de Redes de Interação Co-Translacional por Perfil Seletivo Ribosome
Neste Artigo
Resumo
As interações co-translacionais desempenham um papel crucial nas modificações da cadeia nascente, na segmentação, na dobra e nas vias de montagem. Aqui, descrevemos o Perfil Ribossomo Seletivo, um método para in vivo, análise direta dessas interações no modelo eucariote Saccharomyces cerevisiae.
Resumo
Nos últimos anos, tornou-se evidente que ribossomos não só decodificam nosso mRNA, mas também guiam o surgimento da cadeia de polipeptídeos no ambiente celular lotado. Os ribossomos fornecem a plataforma para a ligação espacial e cineticamente controlada de fatores de segmentação de membrana, modificando enzimas e acompanhantes dobráveis. Até mesmo a montagem em complexos oligomericos de alta ordem, bem como etapas de formação de rede proteína-proteína, foram recentemente descobertas como coordenadas com síntese.
Aqui, descrevemos o Selective Ribosome Profiling, um método desenvolvido para capturar interações co-translacionais in vivo. Detalharemos as várias etapas de purificação de afinidade necessárias para capturar complexos ribossomo-nascentes, juntamente com os interlaciadores co-translacionais, bem como a extração de mRNA, exclusão de tamanho, transcrição reversa, sequenciamento profundo e etapas de análise de big data, necessárias para decifrar interações co-translacionais em resolução de quase codon.
Introdução
Selective Ribosome Profiling (SeRP) é o único método, até o momento, que captura e caracteriza interações co-translacionais, in vivo, de forma direta 1,2,3,4,5,6. O SeRP permite o perfil global de interações de qualquer fator com a tradução de ribossomos na resolução 2,7 de códon próximo.
O método se baseia no congelamento flash de células em crescimento e na preservação da tradução ativa. Os lises celulares são então tratados com RNase I para digerir todo o mRNA na célula, exceto fragmentos de mRNA protegidos por ribossomos denominados "pegadas ribossósas". A amostra é então dividida em duas partes; uma parte é diretamente usada para o isolamento de todas as pegadas ribossômicas celulares, representando toda a tradução contínua na célula. A segunda parte é utilizada para a afinidade-purificação do subconjunto específico de ribossomos associados a um fator de interesse, por exemplo: modificação de enzimas, fatores de translocação, acompanhantes dobráveis e interações de montagem complexa. As pegadas ribossômicas purificadas por afinidade são coletivamente chamadas de interactome. Em seguida, os mRNAs protegidos por ribossomo são extraídos e usados para a geração de biblioteca cDNA, seguido de sequenciamento profundo.
A análise comparativa das amostras totais de translatome e interactome permite a identificação de todos os orfs que associam ao fator de interesse, bem como a caracterização de cada perfil de interação orf. Este perfil relata as sequências precisas de início e término de engajamento a partir das quais se pode inferir os códons decodificados e os respectivos resíduos da cadeia emergente de polipeptídeos, bem como sobre as variações de velocidade ribossosome durante a interação 7,8. A Figura 1 retrata o protocolo como um esquema.
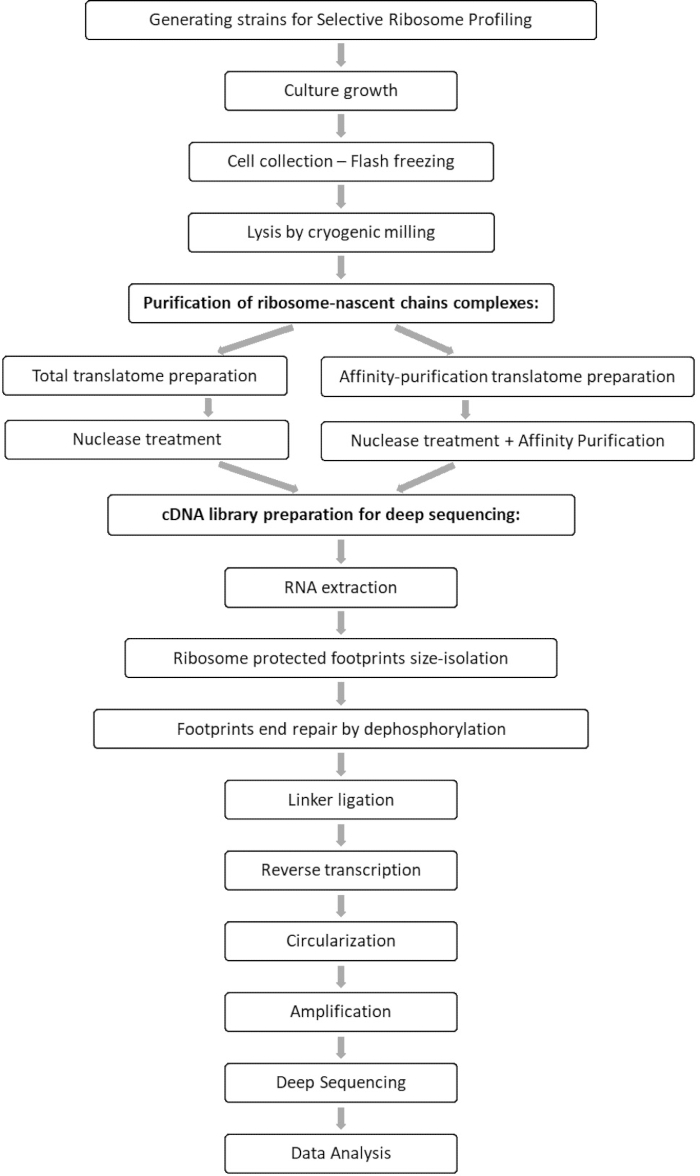
Figura 1: Uma visão geral do protocolo SeRP. Este protocolo pode ser realizado em sua totalidade dentro de 7-10 dias. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Gerando cepas para o perfil seletivo ribossomo
NOTA: Selective Ribosome Profiling (SeRP) é um método que se baseia na purificação de afinidade de fatores de interesse, para avaliar seu modo de interação com complexos de cadeia ribossomos-nascentes. A recombinação homologiosa9, bem como métodos baseados em CRISPR/Cas910 são utilizados para fundir vários fatores de interesse com etiquetas para purificações de afinidade. Tais tags são GFP, para purificações de afinidade GFP-trap, tap-tag para purificações de contas IgG-Sepharose, bem como AVI-Tag purificado por avidin ou streptavidin, para listar alguns exemplos bem-sucedidos dos últimos anos.
- Realizar ensaios de crescimento ou funcional para validar que a marcação não impactou a função das proteínas. A marcação do terminal N' versus C' deve ser avaliada.
NOTA: Os ribossomos (rRNA), bem como muitos domínios de ligação ribossomósmita em vários fatores, são altamente carregados, tornando inpreferíveis tags altamente carregadas (como polihistidina), uma vez que pode levar a falsa descoberta ou modo de ligação alterado.
2. Crescimento da cultura
- Cultivar as culturas de levedura construídas (com base na cepa BY4741), contendo as proteínas marcadas desejadas, em ambos os meios líquidos de levedura-extrato-pepton-dextrose (YPD)- rico, ou em dextrose sintética (SD) meio mínimo (1,7 g/L base de nitrogênio de levedura com sulfato de amônio ou base de nitrogênio de levedura de 1,7 g/L sem sulfato de amônio com ácido glutamico monossódico 1 g/L, 2% de glicose e suplementado com uma mistura completa ou apropriada de aminoácidos).
- Cresça 250-500 mL de cultura celular a um 0,5 OD600 (mid-log), a 30 °C, em um meio apropriado.
3. Coleta celular e lise
- Colete rapidamente células por filtragem de vácuo em uma membrana de manchas de nitrocelulose de 0,45 μm com um sistema de filtragem de vidro (suporte de filtro de vidro com funil de vidro 1 L, base de vácuo e tampa, tela de aço inoxidável, junta e grampo de mola, 90 mm; frasco articular moído 1 L).
- Flash congele as células coletadas, raspando as células pelladas com uma espátula e imediatamente imergindo-as em um tubo líquido cheio de nitrogênio de 50 mL.
PONTO DE PARADA: As células podem ser armazenadas a -80 °C por até 3-4 semanas. - Realize a lise celular por moagem criogênica em um moinho de mistura: duas vezes por 2 min a 30 Hz, com 1 mL do tampão de lise (ver Tabela 1). Esfrie em nitrogênio líquido entre as moagens.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| 10 mg/mL CHX (cicloheximida) | 220 | 0,5 mg/mL |
| 1M Tris-HCl pH 8.0 | 88 | 20 mM |
| 3M KCl | 205.7 | 140 mM |
| 1M MgCl2 | 26.4 | 6 mM |
| 1M PMSF | 4.4 | 1 mM |
| NP-40 | 4.4 | 0.10% |
| Inibidor de protease | 2 comprimidos | |
| DNase I | 8.8 | 0.02 U/mL |
| Volume final | 4,400 |
Tabela 1: Receita para a mistura mestre tampão de lise.
NOTA: O tampão de lise pode ser alterado para conter mais inibidores de protease (como bestatina, leupeptina, aprotinina, etc.) caso a proteína de interesse seja muito instável, mas é importante evitar o EDTA para manter as subunidades pequenas e grandes do ribossomo montadas durante as etapas seguintes. Por razões semelhantes, mantenha sempre pelo menos 6 mM MgCl2 na solução tampão.
ATENÇÃO: O HCL é altamente corrosivo e o PMSF é tóxico. Use luvas e manuseie com cuidado.
- Centrifugar por 2 min a 30.000 x g, 4 °C para limpar o lise e coletar sobrenante.
4. Purificação de complexos de cadeias ribossóficas para SeRP
- Para cada experimento, divida o sobrenante em duas partes; cada um em um tubo de microcentrifuuge diferente: amostra total de RNA (~200 μL) e amostra de imunopurificação (IP) (~700 μL) amostras de translatome.
-
Processando a amostra total de RNA
- Digeste amostra total de RNA usando 10 U de RNase I por 25 min a 4 °C; gire a 30 RPM com um rack de mistura rotativo.
NOTA: As condições de digestão podem ser calibradas usando perfis de pórsome para garantir que não haja excesso ou sub-digestão do pico de monossomos. - Prepare a mistura mestre da almofada de sacarose, conforme descrito na Tabela 2.
- Digeste amostra total de RNA usando 10 U de RNase I por 25 min a 4 °C; gire a 30 RPM com um rack de mistura rotativo.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| 50% de sacarose | 200 | 25% |
| 1M Tris-HCl pH 8.0 | 8 | 20 mM |
| 3M KCl | 18.7 | 140 mM |
| 1M MgCl2 | 4 | 10 mM |
| CHX de 100 mg/mL | 0.4 | 0,1 mg/mL |
| Inibidor de protease | 1 comprimido | |
| Volume final | 400 |
Tabela 2: Receita para mistura master de almofada de sacarose.
- Carregue a amostra em 400 μL da almofada de sacarose e centrífuga em um TLA120-rotor por 90 min a 245.000 x g e a 4 °C.
- Remova o supernatante rapidamente com uma bomba de vácuo e sobrepõe pelotas com um tampão de 150 μL de lise. Resuspengem as pelotas tremendo por 1h a 4 °C e a 300 RPM.
- Resuspenda a pelota residual por pipetação e transferência para um novo tubo de 1,5 mL.
NOTA: 100-200 μg de RNA total é geralmente suficiente para o perfil ribossomo do translaome total. Pode-se adicionar etapa de esgotamento do rRNA a fim de reduzir a contaminação do rRNA, que é o contaminante mais prevalente de cadeias ribossósmos-nascentes complexos purificaçõesde afinidade 11 (ver discussão para mais detalhes).
-
Processamento da amostra de imunopurização
- Lave 100-400 μL da matriz de ligação de afinidade (1:1 contas conjugadas por anticorpos em 70% EtOH) por amostra com tampão de lise de 3 x 1 mL (sem dnase I e inibidores de protease); resuspense a matriz de afinidade no tampão de lise e, em seguida, gire a 30 RPM com um rack de mistura rotativa a 4 °C por 5 min. Precipitar por centrifugação para 30 s a 3.000 x g, 4 °C. Descarte o líquido superior. Repita três vezes.
- Digeste amostras de imunopurificação usando 10 U por unidade A260 nm de RNase I, juntamente com matriz de ligação de afinidade (por exemplo, 100-400 μL de GFP-TRAP por amostra).
- Gire por 25 min a 30 RPM com um rack de mistura rotativa para ligar a proteína à matriz de afinidade, a 4 °C.
- Prepare a mistura mestre do buffer de lavagem conforme detalhado na Tabela 3.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| CHX de 10 mg/mL | 50 | 0,1 mg/mL |
| 1M Tris-HCl pH 8.0 | 100 | 20 mM |
| 3M KCl | 233 | 140 mM |
| 1M MgCl2 | 50 | 10 mM |
| 1M PMSF | 5 | 1 mM |
| NP-40 | 0.5 | 0.01% |
| Inibidor de protease | 2 comprimidos | |
| 50% de Glicerol | 1,000 | 10% |
| Volume final | 5,000 |
Tabela 3: Receita para a mistura mestre do tampão de lavagem.
- Lave a matriz de ligação de afinidade três vezes com 1 mL de tampão de lavagem, cada vez por ~1 min, girando no rack de mistura a 30 RPM, a 4 °C.
- Precipitar por centrifugação a 3.000 x g para 30 s a 4 °C. Descarte o líquido superior.
- Lave mais duas vezes em tampão de lavagem de 1 mL, cada vez por 5 min, girando no rack de mistura a 30 RPM, a 4 °C.
- Precipitar por centrifugação para 30 s a 3.000 x g e 4 °C.
- Use 50 μL de contas para eluição de proteínas com a mesma quantidade de tampão amostral de 2x. Use o resto das contas para extração de RNA.
- Centrifugar por 30 s a 3.000 x g, 4 °C para pelotar as contas e descartar o líquido superior.
- Congele em nitrogênio líquido e armazene a -80 °C. Use estas amostras para extração subsequente de RNA.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C durante a noite ou por mais tempo. Isso pode ser um ponto de parada. - Avalie o sucesso da purificação de afinidade passo a passo da mancha ocidental ou mancha de Coomassie com alíquotas (~10% em volume, após a mistura) de cada etapa. Use sempre o IP simulado em uma cepa WT não marcada como um controle para vinculação não específica à matriz de afinidade.
NOTA: O fundo alto e não específico pode ser superado por etapas adicionais de lavagem com concentrações crescentes de sal/detergente. A interação transitória pode ser estabilizada por vários agentes transfronteiriços, por exemplo, o tratamento paraformaldeído (PFA) de células vivas - adicionando 0,4%-1% pfa à mídia de crescimento por 2-5 min, seguido pela síncina (0,3 M) saciando por 3 min, é altamente recomendado.
ATENÇÃO: Paraformaldeído é um carcinógeno suspeito. Como o paraformaldeído evapora rapidamente e é corrosivo, trabalhe em um capuz de segurança química e use duas camadas de luvas.
5. preparação da biblioteca cDNA para sequenciamento profundo
- Extração de RNA
NOTA: Trabalhe com tubos antiaderente de 1,5 mL livres de RNase para evitar possíveis esgotamentos de RNA ou DNA.- Descongele as amostras das etapas 4.2.5 e 4.3.12 no gelo e resuspende amostras com 10 mM Tris-HCl pH 7.0 para um volume final de 700 μL.
ATENÇÃO: O ácido-fenol e o clorofórmio são voláteis e prejudiciais. Trabalhar em um capuz de segurança química. - Adicione 40 μL de SDS de 20% a 0,7 mL Total de RNA ou eluções IP. Feche e inverta algumas vezes. A precipitação proteica deve tornar as amostras brancas.
- Adicione 0,75 mL de ácido-fenol pré-aquecido:clorofórmio às amostras. Sele os tubos firmemente e agite em uma batedeira térmica a 1.400 RPM por 5 min e a 65 °C. Frio amostras no gelo por 5 minutos.
- Centrifugar o tubo da etapa 5.1.3. a 20.000 x g por 2 min. Transfira a camada superior aquosa para um tubo fresco e adicione a ele 0,7 mL de ácido-fenol:clorofórmio.
- Incubar por 5 minutos à temperatura ambiente, ocasionalmente vórtice. Centrifugar por 2 min a 20.000 x g. Transfira a camada superior aquosa para um tubo fresco e adicione a ele 0,6 mL de clorofórmio e vórtice.
- Centrifugar por 1 min a 20.000 x g. Transfira a camada superior aquosa para um tubo fresco.
- Ácidos nucleicos precipitadores adicionando 78 μL de 3 M NaOAc, pH 5.5, 2 μL de GlycoBlue e 0,75 mL de isopropanol. Vórtice completamente por 5 minutos. Incubar por pelo menos 1 h a -80 °C ou 16 h a -20 °C.
- Centrifugar por 30 min a 20.000 x g e a 4 °C e descartar o supernatante. Lave as pelotas com 0,75 mL de 80% de etanol. Inverta os tubos para uma lavagem completa. Centrifugar a 20.000 x g por 5 min a 4 °C e, em seguida, descartar o sobrenatante.
- Gire a 450 x g, 4 °C para 20 s e retire o etanol restante e descarte os líquidos. Seque a pelota com uma tampa aberta por 5 min a 65 °C. Resuspende as amostras da seguinte forma: para IP ressuspende a amostra em 10 μL de 10 mM Tris-HCl, pH 7.0. Para análise translador total, resuspenncie a amostra em 20 μL de 10 mM Tris-HCl, pH 7.0.
PONTO DE PARADA: O RNA pode ser armazenado a -80 °C por meses.
- Descongele as amostras das etapas 4.2.5 e 4.3.12 no gelo e resuspende amostras com 10 mM Tris-HCl pH 7.0 para um volume final de 700 μL.
- Quantifique a concentração total de RNA por fluorometria
NOTA: Todos os seguintes materiais e superfícies devem ser livres de RNase enquanto preparam a biblioteca cDNA para sequenciamento de próxima geração. Enquanto manuseia amostras de RNA, use luvas.- Diluir 1 μL de RNA total extraído de fenol ácido em 9 μL de 10 mM Tris-HCl, pH 7.0. Quantifique usando um fluorômetro, conforme instruído no site do fabricante.
- Diluir as amostras contendo 50 μg de RNA com 10 μL de 10 mM Tris-HCl, pH 7.0.
NOTA: Não meça amostras de IP, use tudo para o próximo passo.
- Fragmentos de pegada protegida de ribossomo purificadores de gel
- Defina um gel de poliacrilamida de 15% de TBE-ureia e submerse em 1x tampão de execução TBE. Corra por 30 min a 200 V antes do carregamento da amostra. A cada amostra, adicione 20 μL de 2x tampão de amostra de TBE-ureia.
NOTA: O tamanho esperado da banda é em torno de 25-35 nt. - Descongele uma escada de DNA de 10 bps e amostras de desnatura (não escada) a 80 °C por 2 min, e depois esfrie no gelo. Carregue cada amostra em todas as outras pistas. Execute o gel por 50-70 min a 200 V.
- Diluir 6 μL de Ouro SYBR (10.000 x concentrado) em 60 mL de tampão 1x TBE e manchar enquanto balança em caixas protegidas por luz por 15-20 min. Enquanto estiver manchando o gel, prepare um bisturi estéril e tubos de quebra-gel de 0,5 mL em tubos de 1,5 mL rotulados.
- Extirpar as bandas desejadas com um bisturi estéril (use um bem fresco ou limpo entre as amostras) e coloque cada peça de gel em um tubo de quebra-gel.
- Tire uma imagem do gel para garantir que nenhum resíduo amostral seja deixado no gel.
- Centrifugar os tubos contendo fatias cortadas a 20.000 x g por 5 min a 4 °C e transfira as peças de gel restantes do tubo de quebra-gel para o tubo de 1,5 mL.
- Adicione 0,5 mL de 10 mM Tris, pH 7.0. Agite em uma batedeira térmica a 1.400 RPM por 10 min a 70 °C.
- Transfira para uma coluna de acetato de celulose com uma ponta de pipeta larga e centrífuga a 20.000 x g por 3 min a 4 °C.
- Transfira o fluxo para um novo tubo de 1,5 mL e esfrie no gelo.
- Para precipitar os ácidos nucleicos, adicione: 550 μL de IPA, 55 μL de 3 M NaOAc e 2 μL de GlycoBlue e vórtice para misturar bem. Coloque as amostras a -80 °C por pelo menos 1 h.
- Centrifugar por pelo menos 1h a 20.000 x g e 4 °C e descartar o supernatante. Lave as pelotas com 0,75 mL de etanol gelado de 80%. Inverta os tubos para uma lavagem completa até que as pelotas se separem da parte inferior. Centrifugar novamente a 20.000 x g por 5 min a 4 °C e descartar o supernatante.
- Gire a 450 x g, 4 °C para 20 s e retire o etanol restante. Seque as pelotas com tampa aberta por 5 min a 65 °C.
- Adicione 15 μL de 10 mM Tris, pH 7.0 e resuspenda as pelotas completamente. Gire a 450 x g, 4 °C para 20 s e transfira a amostra para um novo tubo de 1,5 mL.
PONTO DE PARADA: O RNA purificado pode ser armazenado a -80 °C por alguns meses.
- Defina um gel de poliacrilamida de 15% de TBE-ureia e submerse em 1x tampão de execução TBE. Corra por 30 min a 200 V antes do carregamento da amostra. A cada amostra, adicione 20 μL de 2x tampão de amostra de TBE-ureia.
- Desfosforilação
- Use 3 μL da seguinte mistura para cada amostra: Adicione 1 μL de inibidor de RNase em 2 μL de 10x T4 tampão de reação de quinase de polinucleotídeo sem ATP. Adicione 2 μL de quinase de polinucleotídeo T4 a cada amostra. Pipeta suavemente para misturar bem e incubar a 37 °C por 2h, sem balançar.
- Para inativar a enzima, incubar a amostra a 75 °C por 10 min e girar para baixo a 450 x g, 4 °C, para 20 s. Adicione 0,5 mL de 10 mM Tris, pH 7.0.
- Para precipitar o ácido nucleico, adicione 2 μL de GlycoBlue, 550 μL de IPA e 55 μL de 3 M NaOAc.
- Vórtice para misturar bem e esfriar as amostras a -80 °C por pelo menos 1 h.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C durante a noite ou por mais tempo. - Repetição passos 5.3.11-5.3.13.
PONTO DE PARADA: O RNA desfosforilado pode ser armazenado a -80 °C por meses.
- Quantificação usando um Bioanalyzer
- Faça uma diluição de 1:4 de cada amostra de RNA misturando 1 μL de amostra e 4 μL de água tratada com DEPC.
ATENÇÃO: O DEPC é um cancerígeno. Use luvas e trabalhe com cuidado. - Execute um Bioanalyzer Small RNA Chip/TapeStation. Siga o protocolo do fabricante.
NOTA: O tamanho esperado do fragmento de RNA protegido por ribossomo é em torno de 28-30 nt.
- Faça uma diluição de 1:4 de cada amostra de RNA misturando 1 μL de amostra e 4 μL de água tratada com DEPC.
- Ligate 3' final com Linker-1
- Diluir 5 pmol de pequenos fragmentos de RNA a 10 μL com 10 mM Tris, pH 7.0. Amostras de desnatura a 80 °C por 2 min e frio no gelo.
- Prepare a mistura mestre conforme detalhado na Tabela 4 e use 29 μL por amostra.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| PEG 8000 filtrado 50% | 16 | 20% |
| DMSO | 4 | 10% |
| Tampão de ligase T4 2 de 10× | 4 | 1x |
| Inibidor de RNase SUPERase-In | 2 | 2 U |
| Linker 3-L1 adenylated de 10 mM | 0.1 | 25 μM |
| Água tratada com DEPC | 2.9 | |
| Volume final | 29 |
Tabela 4: Receita para 3' end ligation master mix.
- Adicione 1 μL de liga ligase T4 RNA 2 e pipeta suavemente para misturar bem. Incubar a 23 °C por 2 h.
- Para precipitar os ácidos nucleicos, adicione: 550 μL de IPA, 500 μL de 10 mM Tris, pH 7.0, 55 μL de 3 M NaOAc e 2 μL de GlycoBlue. Vórtice para misturar bem, e coloque as amostras a -80 °C por pelo menos 1 h.
PONTO DE PARADA: Armazene as amostras a -80 °C durante a noite ou por mais tempo. - Repetição de passos 5.3.2-5.3.12.
- Resuspene a pelota em 6 μL de 10 mM Tris, pH 7.0. Gire a 450 x g, 4 °C para 20 s e transfira a amostra para um novo tubo de 1,5 mL.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C por meses.
-
Purificação de gel de pegadas ligadas a 3'
- Defina um gel de poliacrilamida de 10% TBE-ureia e submerse em 1x tampão de execução TBE. Corra por 30 min a 200 V antes do carregamento da amostra. A cada amostra, adicione 6 μL de 2x tampão de amostra de TBE-uréia.
NOTA: O tamanho esperado da banda é em torno de 71-73 nt. - Repetição de passos 5.3.2-5.3.13.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C por meses.
- Defina um gel de poliacrilamida de 10% TBE-ureia e submerse em 1x tampão de execução TBE. Corra por 30 min a 200 V antes do carregamento da amostra. A cada amostra, adicione 6 μL de 2x tampão de amostra de TBE-uréia.
-
Transcrever reverso 30 fragmentos de pegada ligados para gerar ssDNA
- Prepare uma mistura mestra conforme detalhado na Tabela 5 e use 3 μL por amostra.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| DNTPs de 10 mM | 1 | 0,5 mM |
| 25 μM Linker L(rt) | 0.5 | 625 nM |
| Água tratada com DEPC | 1.5 | |
| Volume final | 3 |
Tabela 5: Receita para a mistura mestre do buffer de transcrição reversa antes da desnaturação dos ácidos nucleicos.
- Vórtice e gire a amostra.
- Incubar amostras a 65 °C por 5 min.
- Amostras de frio no gelo.
- Prepare uma mistura mestra conforme detalhado na Tabela 6 e use 6 μL por amostra. Vórtice e gire a amostra. Adicione 1 μL de Superscript III a cada amostra e pipeta suavemente para misturar bem e incubar por 30 min a 50 °C.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| Tampão FS × 5 | 4 | 1x |
| Inibidor de RNase SUPERase-In | 1 | 2 U |
| DTT 0.1 M | 1 | 5 mM |
| Volume final | 6 |
Tabela 6: Receita para a mistura mestre do buffer de transcrição reversa após a desnaturação dos ácidos nucleicos.
- Adicione 2,3 μL de 1 N NaOH, que hidrolisa RNA e sacia a transcrição reversa.
ATENÇÃO: O NaOH é altamente corrosivo. Use luvas e proteção ocular. - Incubar por 15 min a 95 °C, até que a amostra fique rosa.
- Defina um gel de poliacrilamida de 10% TBE-ureia e submerse em 1x tampão de execução TBE. Corra por 30 min a 200 V antes do carregamento da amostra. A cada amostra, adicione 23 μL de 2x tampão de amostra de TBE-ureia.
NOTA: O tamanho esperado da banda de DNA é de 115-117 nt. - Repetição de passos 5.3.2-5.3.12.
- Resuspene a pelota em 15 μL de 10 mM Tris, pH 8.0. Gire a 450 x g, 4 °C para 20 s e transfira a amostra para um novo tubo de 1,5 mL.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C por meses.
-
circularização ssDNA
- Prepare a seguinte mistura mestre e carga de 4 μL por amostra, conforme detalhado na Tabela 7.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| Tampão CircLigase II de 10× | 2 | 1x |
| 5 M betaine (opcional) | 1 | 0,25 M |
| 50 mM MnCl2 | 1 | 2,5 mM |
| Volume final | 4 |
Tabela 7: Receita para o mix mestre de circularização ssDNA.
- Adicione 1 μL de ligadura circligase II ssDNA a cada amostra e incubar por 1h a 60 °C.
NOTA: A eficiência desta etapa pode ser aumentada adicionando 1 μL de liga ligase de SSNA CircLigase II a cada amostra após a incubação de 1 h. - Inativar a enzima incubando a 80 °C por 10 min.
- Esfrie no gelo e continue a amplificação pcr ou armazene a -80 °C.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C por anos.
-
Amplificação do PCR
- Prepare a seguinte mistura mestre pcr e carregue 82 μL por amostra, conforme detalhado na Tabela 8.
| Reagente | Quantidade por amostra (μL) | Concentração final |
| Água tratada com DEPC | 61.6 | |
| Tampão de reação Phusion HF de 5× | 17.6 | 1x |
| DNTPs de 10 mM | 1.8 | 200 μM |
| Primer para frente pcr de 100 μM | 0.2 | 225 nM |
| HF Phusion polimerase | 0.8 | 1.6 U |
| Volume final | 82 |
Tabela 8: Receita para mistura mestre de amplificação PCR.
- A cada tubo contendo mistura mestra, adicione 5 μL de DNA circularizado.
NOTA: Armazene o restante das amostras de DNA circularizadas a -80 °C. - Adicione um diferente 1 μL de 20 μM PCR primede de código de barras reverso a cada amostra (ver Tabela 9) e vórtice para misturar completamente.
- Alíquotar cada tubo em quatro tubos PCR separados, cada um será usado para um número diferente de ciclos PCR.
- Execute uma reação pcr de acordo com o programa a seguir, conforme detalhado na Tabela 10.
| Ciclo | Denaturação (98 °C) | Anneal (60 °C) | Estender (72 °C) |
| 1 | 30 s | ||
| 2-16 | 10 s | 10 s | 5 s |
Tabela 10: Programa PCR para reação pcr.
- Após os ciclos 8, 9, 10 e 11 remover tubos PCR (para amostras de IP, os ciclos variam de 9 a 15) como primeira tentativa. Após cada ciclo, pausa o programa, pegue uma alíquota e coloque-a no gelo, e então retome rapidamente o programa.
NOTA: O número de ciclos deve ser ajustado com base na quantidade de DNA circularizado em cada reação. Consulte a Figura 4 para um exemplo e esclarecimentos adicionais. - A cada reação de 17 μL, adicione 3,5 μL de corante de carregamento de DNA de 6x.
- Descongele uma escada de DNA de 10 bps.
- Para a separação de tamanho por gel-eletroforese, submersa 8% de poliacrilamida TBE em 1x TBE executando buffer e carregue as amostras de cada número de ciclo diferente em poços adjacentes e execute o gel por 50 min a 180 V.
- Diluir 6 μL de Ouro SYBR (10.000 x concentrado) em 60 mL de tampão 1x TBE e manchar enquanto balança em caixas protegidas por luz por 15-20 min.
- Enquanto estiver manchando o gel, prepare um bisturi estéril e tubos de quebra-gel de 0,5 mL em tubos de 1,5 mL rotulados.
- Tire uma imagem dos ácidos nucleicos manchados.
- Corte a faixa desejada com um tamanho de banda esperado de 174-176 bp com o bisturi estéril e coloque a fatia de gel no tubo preparado de 0,5 mL de quebra-gel (limpe bem entre as amostras e use o agente inativador RNase, ou mude para uma nova lâmina).
- Centrifugar os tubos por 5 min a 20.000 x g e 4 °C, e depois transfira as peças de gel restantes do tubo de quebra-gel de 0,5 mL para o tubo de 1,5 mL.
- Adicione 500 μL de 10 mM Tris, pH 8.0 e agite em uma batedeira térmica a 1.400 RPM por 10 min e 70 °C.
- Transfira o gel dissolvido para uma coluna de acetato de celulose com uma ponta de pipeta larga.
- Centrifugar a coluna por 3 min a 20.000 x g e 4 °C e transferir o fluxo para um novo tubo de 1,5 mL e esfriar no gelo.
- Para precipitar o ácido nucleico, adicione: 550 μL de IPA, 32 μL de 5 M NaCl, 1 μL de 0,5 M EDTA e 2 μL de GlycoBlue e vórtice para misturar bem.
- Mantenha as amostras por pelo menos 1 h a -80 °C, ou -20 °C durante a noite.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C durante a noite ou por mais tempo. - Repetição de passos 5.3.11-5.3.12.
- Resuspend em 11 μL de 10 mM Tris, pH 8.0. Gire a 450 x g, 4 °C para 20 s e transfira a amostra para um novo tubo de 1,5 mL.
PONTO DE PARADA: As amostras podem ser armazenadas a -80 °C por anos.
-
Quantifique a distribuição de tamanho por Bioanalyzer
- Faça uma diluição de 1:4 de cada amostra misturando 1 μL de amostra com 4 μL de água tratada com DEPC.
- Execute o Bioanalyzer Small RNA Chip. Siga o protocolo do fabricante.
NOTA: O comprimento esperado é de 175 ± 5 bp.
-
Quantifique a concentração de DNA por fluorômetro
- Realize uma verificação de concentração de alta sensibilidade dsDNA com um fluorômetro de acordo com as recomendações do fabricante.
- Amostras multiplex e sequência de acordo com as recomendações de Illumina (Index Adapters Pooling Guide12).
6. Análise de dados
- Realizar a análise conforme detalhado no arquivo complementar.
Resultados
Conforme ilustrado no fluxograma deste protocolo (Figura 1), as células foram cultivadas para a fase de registro, e depois coletadas rapidamente pela filtragem e líricas pela moagem criogênica. O lysate foi então dividido em dois: um para pegadas totais de mRNA protegidos por ribossomo e outro para pegadas de mRNA protegidas por ribossomo selecionados, nas quais realizamos purificação de afinidade para puxar para baixo os complexos de cadeias de proteína-ribossomo-nascentes marcadas. Garantimos a expressão proteica marcada e o sucesso da retração pela análise da mancha ocidental, como pode ser visto na Figura 2. Validamos o isolamento de pegadas protegidas por ribossomo, que são tipicamente de 20-45 nt de comprimento por pequena eletroforese de RNA (sistema 2100 BioAnalyzer), permitindo uma mudança de 5-10 nt na detecção de tamanho, de acordo com o manual do sistema (Figura 3). Então, geramos uma biblioteca cDNA para sequenciamento profundo e análise de big data. Ao gerar a biblioteca cDNA, observe que o ciclismo abaixo pode levar a baixo rendimento (como pode ser visto na faixa 2 na Figura 3), mas a ream amplificação é possível para recuperar a biblioteca gerada. O excesso de ciclismo pode ocorrer quando os primers pcr estão esgotados, mas a reação continua. Quando os dNTPs ainda estão presentes, a reação prossegue, gerando artefatos PCR mais longos com sequências quimricas devido aos produtos PCR que se escorram13 (como pode ser visto nas faixas 3-4 na Figura 3, indicadas pela mancha visível). Se a concentração dos dNTPs também se tornar limitante, podem aparecer produtos que indiquem a presença de heterodúplexos compostos apenas de fragmentos de biblioteca parcialmente homólogos. A Figura 4 atua como referência, com a faixa 2 representando a amplificação ideal, e a pista 3 uma amplificação aceitável. As amostras das faixas 4 e 5 (ciclos 10 e 11) não devem ser utilizadas devido à possibilidade de introdução de duplicatas e artefatos pcr. A biblioteca gerada foi ainda validada por eletroforese de DNA de alta sensibilidade (o mesmo sistema BioAnalyzer foi utilizado) para distribuição e quantificação de tamanho exato (Figura 5). Após a ligação de linker final de 3', transcrição reversa e amplificação pcr, uma distribuição de comprimento cDNA como tal é esperado, com um pico acentuado em torno de 175 nt.
Aparamos e removemos adaptadores e códigos de barras da biblioteca sequenciada, e apenas as leituras entre 20 e 45 nt foram selecionadas para análise posterior. A Figura 6 mostra a distribuição de comprimento resultante. As leituras foram divididas em diferentes grupos de: sequências de codificação, introns e sequências intergênicas (Figura 7), e ainda classificadas como mostradas na Figura 8.
A análise final para detecção e caracterização das interações co-translacionais foi realizada com base no enriquecimento de fragmentos de mRNA protegidos por ribossomo, produzindo os gráficos na Figura 9. Comparamos a ocupação ribossoma normalizada (em cada nucleotídeo ao longo de cada orf) do translatome total ao seu translatome selecionado correspondente (nascente-interactome). Por comparação nucleotídea elimina as taxas de tradução artefatos. A reprodutibilidade entre as réplicas biológicas foi avaliada pela correlação de Pearson (limiar > 0,6). Apresentamos Perfis Ribossomos Seletivos, analisando interações co-translacionais do Vma2p com três proteínas: a acompanhante associada ao ribossomo Ssb1p, Pfk2p (Phosphofructokinase) e Fas1p (Sinthase de ácido graxo), com cada proteína C' terminantemente marcada pelo GFP. Executamos o protocolo em réplicas biológicas. Figura 9 A, D e G mostra o esquema experimental de cada purificação de afinidade. Em seguida, mostramos a ocupação ribossa de translatórios totais em comparação com os interactomes Ssb1 ao longo do Vma2p orf, codificando para uma subunidade do vacuolar H+-ATPase (Figura 9 B, E e H). Finalmente, realizamos o perfil de enriquecimento ribossomo baseado em proporção (IP/Total) em cada posição ribossosome em [codon/aa] ao longo do orf (Figura 9 C, F e I). Comparando as interações co-translacionais dessas três proteínas com o Vma2p, que está sendo sintetizado pelo ribossomo, revelou que a acompanhante do Ssb1 envolve o Vma2p nascente em quatro regiões diferentes ao longo do orf, pois identificamos quatro picos significativos de enriquecimento pelo SeRP. Diferentemente, Pfk2p mostra apenas um pico significativo de enriquecimento, conforme identificado pelo SeRP, em uma posição diferente em relação à acompanhante co-translacional Ssb1. A análise das interações co-translacionais de Fas1 com o nascente Vma2p não detectou nenhum enriquecimento significativo. Assim, a comparação desses perfis ribossomos de enriquecimento baseado em proporção demonstra o poder deste protocolo na detecção e caracterização de várias interações co-translacionais em resolução de códons próximos.
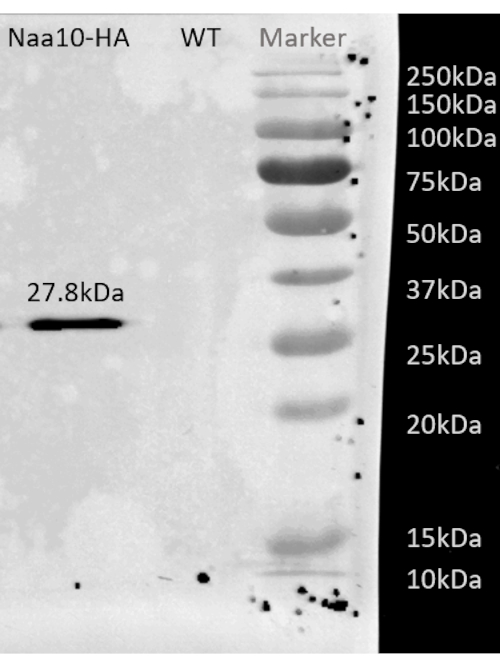
Figura 2: Resultado representativo da mancha ocidental após purificação de afinidade da cepa BY4741 com Naa10 com marca HA. Resultado representativo da mancha ocidental após a purificação de afinidade da cepa BY4741 com Naa10 com marca HA mostrando uma banda em torno de 27,8 kDa, enquanto o tipo selvagem, como um controle negativo, não mostra nenhuma banda. Clique aqui para ver uma versão maior desta figura.

Figura 3: Resultado bioanalyzer representativo após isolamento da pegada e extração de RNA com ácido-fenol:clorofórmio, e um tamanho médio de 25 nt. Por favor clique aqui para ver uma versão maior desta figura.
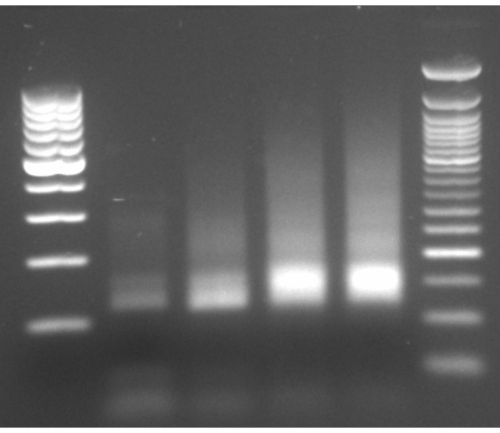
Figura 4: Eletroforese gel representativa da amplificação pcr. Representante gel-eletroforese de amplificação PCR com faixas 2-5 carregadas com produtos PCR dos ciclos 8-11, e escadas em ambos os lados. Clique aqui para ver uma versão maior desta figura.
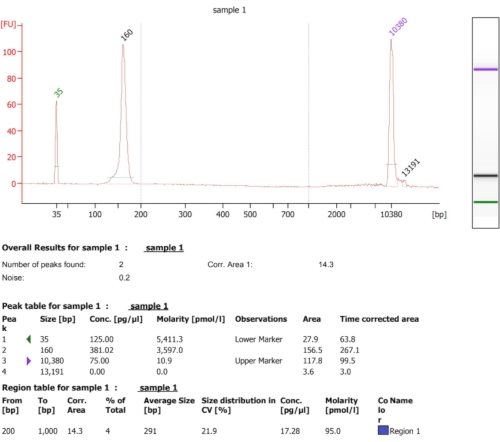
Figura 5: Resultado bioanalyzer representativo obtido após a criação de uma biblioteca cDNA. Clique aqui para ver uma versão maior desta figura.

Figura 6: Distribuição de comprimento esperado de leituras após a remoção dos adaptadores com Cutadapt (removendo leituras menores que 20 ou mais de 45). Clique aqui para ver uma versão maior desta figura.

Figura 7: Porcentagem de sucesso de alinhamento esperado depois de remover leituras de RNA não codificadoras com Bowtie2 e usar TopHat para alinhar as leituras restantes a diferentes organismos. A amostra foi colhida de S. cerevisiae (uma variante mutante de BY4741). Clique aqui para ver uma versão maior desta figura.
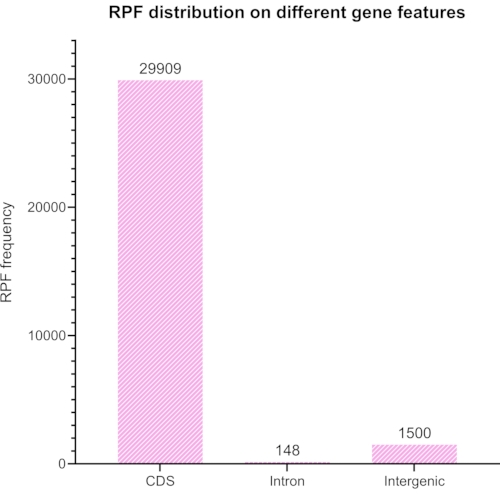
Figura 8: Um gráfico gerado com o RiboToolkit representando a codificação esperada versus a razão não codificação das leituras alinhadas depois de usar o Bowtie2 para remover elementos de rRNA nas leituras. Clique aqui para ver uma versão maior desta figura.
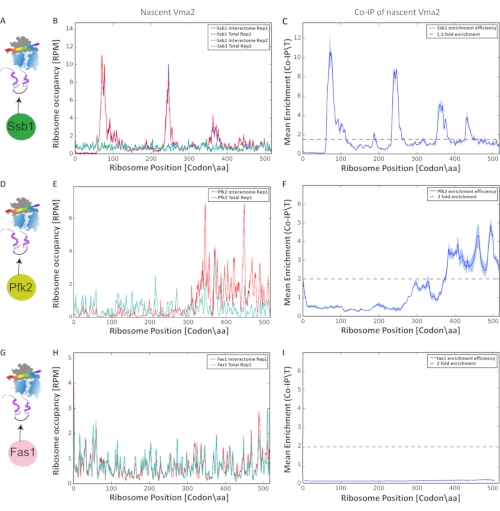
Figura 9: Interações co-translacionais de três proteínas diferentes: Ssb1p, Pfk2p e Fas1p com Vma2p, que está sendo sintetizada pelo ribossomo, analisado pelo SeRP. Todos os eixos y são mostrados em leituras por milhão (RPM) lê.. (A, D, G) Esquema experimental de SeRP de Ssb1p, Pfk2p e Fas1p C' marcados terminantemente pela GFP, respectivamente. (B, E, H) Ocupação ribossa ao longo da orf de translatomes totais em comparação com os interactomes Ssb1, Pfk2p e Fas1p, respectivamente (em réplicas biológicas). (C, F, I) Enriquecimento médio de Ssb1p, Pfk2p e Fas1p (razão IP/Total) em cada posição ribossa em [codon/aa] ao longo do orf, respectivamente. A variação entre as réplicas biológicas é indicada pela área sombreada. Clique aqui para ver uma versão maior desta figura.
Tabela 9: 3' Linker e sequências de primer. 3' Linker L1: Linker 3-L1 com 50 adenylation e 30 identificadores moleculares únicos dideoxy-cytidine ('NN...') (Purificação HPLC livre de RNase; Linker de transcrição reversa: transcrição reversa (L(rt)) com identificadores moleculares únicos e fosforilados (purificação HPLC sem rnase); Primer para frente pcr: PCRf; HPLC purificado. Clique aqui para baixar esta Tabela.
Arquivo Suplementar. Clique aqui para baixar este Arquivo.
Discussão
Aqui, o protocolo detalha a abordagem de Perfil Ribossomo Seletivo para capturar interações co-translacionais em resolução de códon próximo. À medida que o ribossomo surge como um centro para coordenar o surgimento da cadeia nascente no citoplasma lotado, este é um método crucial para identificar e caracterizar as diversas interações co-translacionais necessárias para garantir um proteome funcional, bem como para o estudo de várias doenças. Até o momento, o SeRP é o único método que pode capturar e caracterizar essas interações, de forma direta, in vivo 14,15,16.
O primeiro e mais crítico passo é a coleta de células e a lise celular. É imperativo capturar, em segundos, a tradução contínua, por congelamento de flash seguido de lise em um estado congelado. A coleta de células deve ser feita com pressa para evitar o escoamento ribossômico, bem como induzir respostas translacionais de estresse, que podem ocorrer rapidamente. O segundo passo crítico é o passo de purificação da afinidade. É imprescindível reduzir a vinculação de fundo por meio de uma lavagem rigorosa, garantindo que as interações co-translacionais sejam mantidas, o que pode ser facilitado pelo cruzamento in vivo . Como este protocolo é baseado no fundo alto NGS (Next Generation Sequencing) altamente sensível nos primeiros passos pode ser amplificado nas seguintes etapas de preparação da biblioteca cDNA, levando a baixas relações sinal-ruído.
O tratamento nuclease, para digerir todos os mRNAs não protegidos deve ser avaliado por perfil polisso17 , juntamente com uma avaliação cuidadosa da distribuição isolada de tamanho das pegadas ribossômicas (conforme detalhado acima) para evitar mais ou sob digestão de RNA. A calibração dos tempos de concentração e digestão da Nuclease pode facilitar a recuperação precisa da pegada, pois a digestão excessiva pode levar à digestão de rRNA ribossômico, levando à perda de pegadas protegidas por ribossomos. É importante notar que a sub digestão também pode levar a taxas mais baixas de descoberta de pegadas protegidas por ribossomo, como as etapas de preparação da biblioteca cDNA, bem como as etapas de análise de dados descritas aqui descartam leituras longas e não características.
Embora o esgotamento do RRNA nem sempre constitua um passo crítico e não seja obrigatório, ele tem algumas vantagens, como amostras mais limpas e, portanto, uma taxa maior de leituras mapeadas pelo genoma. Por outro lado, há a possibilidade de vieses, já que muitos protocolos de esgotamento do rRNA também podem causar esgotamento dos fragmentos protegidos por ribossomos desejados. Deve-se também levar em consideração os custos dos kits de esgotamento do rRNA. o esgotamento do rRNA pode ser realizado após a etapa de isolamento da impressão de pé ribossomo ou após a etapa de circularização cDNA.
as etapas de preparação da biblioteca cDNA, conforme descrito aqui, foram otimizadas para entrada de mRNA baixa, pois as etapas de afinidade e purificação ribossomamo reduzem muito a quantidade de entrada de mRNA, em comparação com estudos de expressão RNA-seq. O upscaling da quantidade inicial de culturas celulares pode facilitar muito a geração de bibliotecas cDNA. Alternativamente, qualquer protocolo de biblioteca cDNA de escolha pode se encaixar com os passos de purificação de afinidade e isolamento de pegada descritos aqui. É importante notar que o tratamento Nuclease que gera as pegadas ribossômicas requer o reparo resultante de extremidades mRNA (preparação da biblioteca cDNA, etapa de desfosforilação) para permitir seguir as etapas de ligadura de linker no protocolo cDNA descrito aqui em seu protocolo de escolha.
Durante o sequenciamento, é importante diferenciar o SeRP do RNA-Seq, pois a heterogeneidade das bibliotecas geradas varia muito, dependendo dos fatores marcados pela afinidade. Acompanhantes moleculares e fatores de segmentação são muitas vezes mais promíscuos na vinculação, interagindo com centenas ou milhares de substratos durante a tradução, levando a bibliotecas cDNA altamente diversas. No entanto, interagidores altamente específicos, como os interativos de montagem de complexos co-translacionais, muitas vezes podem levar à geração de bibliotecas cDNA muito menos diversas. O aumento de bibliotecas diversas e não diversas na mesma pista pode melhorar muito o sequenciamento e seguir os resultados da análise de dados.
Outra característica única do SeRP é sua capacidade de capturar variações locais em ocupações ribossósmos ao longo do orf permitindo a descoberta de mudanças ribossômicas na taxa de tradução associadas a cada conjunto de interações. Por isso, é imprescindível comparar ocupações ribossósmos em cada códon ao longo do orf para identificar corretamente o enriquecimento. Utilizar as médias dos orfs pode levar à perda de interações transitórias ou falsa descoberta.
O uso correto do método SeRP abre muitos caminhos co-translacionais para a análise direta, descobrindo novas características mecanicistas, bem como novos fatores associados ao ribossomo, revolucionando o campo proteína-biosíntese.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
Gostaríamos de agradecer a todos os membros do laboratório por discussões frutíferas e Muhammad Makhzumy pela leitura crítica do manuscrito. Este trabalho foi financiado pela isf (Israel Science Foundation) grant 2106/20.
Materiais
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23(2012).
- Guide, P. Illumina Index Adapters - Pooling Guide. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019).
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados