Method Article
Identificación global de redes de interacción co-traslacional mediante perfiles selectivos de ribosomas
En este artículo
Resumen
Las interacciones co-traslacionales juegan un papel crucial en las modificaciones de la cadena naciente, la orientación, el plegado y las vías de ensamblaje. Aquí, describimos el Perfil Selectivo de Ribosomas, un método para el análisis directo in vivo de estas interacciones en el modelo eucariota Saccharomyces cerevisiae.
Resumen
En los últimos años, se ha hecho evidente que los ribosomas no solo decodifican nuestro ARNm, sino que también guían la aparición de la cadena polipeptídica en el entorno celular abarrotado. Los ribosomas proporcionan la plataforma para la unión controlada espacial y cinéticamente de factores dirigidos a la membrana, enzimas modificadoras y chaperonas plegables. Incluso el ensamblaje en complejos oligoméricos de alto orden, así como los pasos de formación de la red proteína-proteína, se descubrieron recientemente que estaban coordinados con la síntesis.
Aquí, describimos el Perfil Selectivo de Ribosomas, un método desarrollado para capturar interacciones co-traslacionales in vivo. Detallaremos los diversos pasos de purificación de afinidad necesarios para capturar complejos de cadena ribosómica-naciente junto con interactores co-traduccionales, así como la extracción de ARNm, la exclusión de tamaño, la transcripción inversa, la secuenciación profunda y los pasos de análisis de big data, necesarios para descifrar las interacciones co-traduccionales en resolución cercana al codón.
Introducción
Elrofiling Ribosome P(SeRP) es el único método, hasta la fecha, que captura y caracteriza las interacciones co-traslacionales, in vivo, de manera directa 1,2,3,4,5,6. SeRP permite el perfil global de las interacciones de cualquier factor con la traducción de ribosomas en resolución de codón cercano 2,7.
El método se basa en la congelación instantánea de las células en crecimiento y la preservación de la traducción activa. Los lisados celulares se tratan con RNasa I para digerir todo el ARNm en la célula, excepto los fragmentos de ARNm protegidos por ribosomas denominados "huellas de ribosomas". La muestra se divide entonces en dos partes; una parte se utiliza directamente para el aislamiento de todas las huellas ribosómicas celulares, que representan toda la traducción en curso en la célula. La segunda parte se utiliza para la afinidad-purificación del subconjunto específico de ribosomas asociados con un factor de interés, por ejemplo: enzimas modificadoras, factores de translocación, chaperonas plegables e interacciones complejo-ensamblaje. Las huellas ribosómicas purificadas por afinidad se denominan colectivamente el interactoma. Luego, los ARNm protegidos por ribosomas se extraen y se utilizan para la generación de bibliotecas de ADNc, seguidos de una secuenciación profunda.
El análisis comparativo de las muestras totales de translatoma e interactoma permite la identificación de todos los orfs que se asocian con el factor de interés, así como la caracterización de cada perfil de interacción orf. Este perfil informa las secuencias precisas de inicio y terminación del compromiso a partir de las cuales se pueden inferir los codones decodificados y los residuos respectivos de la cadena polipeptídica emergente, así como sobre las variaciones de velocidad del ribosoma durante la interacción 7,8. La figura 1 muestra el protocolo como un esquema.
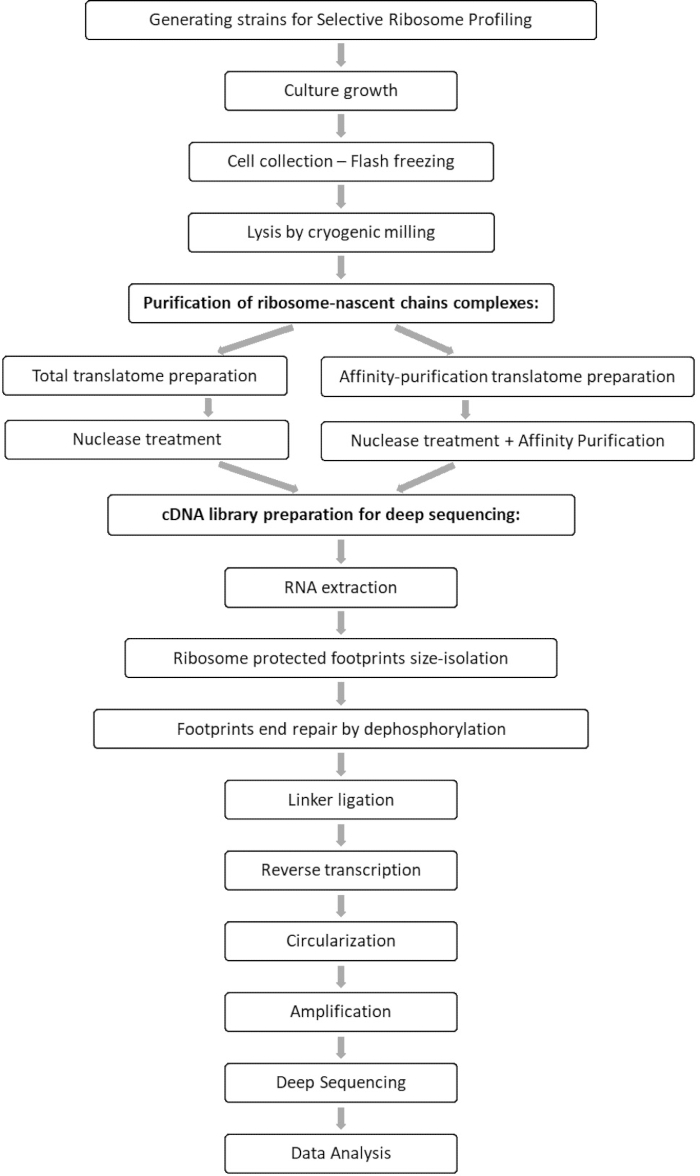
Figura 1: Una visión general del protocolo SeRP. Este protocolo se puede realizar en su totalidad en un plazo de 7-10 días. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Generación de cepas para el perfil selectivo de ribosomas
NOTA: Elrofiling Ribosome P(SeRP) es un método que se basa en la purificación por afinidad de factores de interés, para evaluar su modo de interacción con complejos de cadena ribosomas-nacientes. La recombinación homóloga9, así como los métodos basados en CRISPR / Cas910 se utilizan para fusionar varios factores de interés con etiquetas para purificaciones de afinidad. Tales etiquetas son GFP, para purificaciones de afinidad de trampa GFP, TAP-tag para purificaciones de perlas igG-Sepharose, así como AVI-Tag purificado por avidina o estreptavidina, para enumerar algunos ejemplos exitosos de los últimos años.
- Realizar ensayos funcionales o de crecimiento para validar que el etiquetado no afectó la función de las proteínas. Se debe evaluar el etiquetado terminal N' versus C'.
NOTA: Los ribosomas (ARNr), así como muchos dominios de unión a ribosomas en varios factores, están altamente cargados, lo que hace que las etiquetas altamente cargadas (como la polihistidina) no se puedan usar, ya que puede conducir a un falso descubrimiento o un modo de unión alterado.
2. Crecimiento de la cultura
- Cultivar los cultivos de levadura construidos (basados en la cepa BY4741), que contengan las proteínas marcadas deseadas, ya sea en un medio líquido rico en extracto de levadura-peptona-dextrosa (YPD), o en medio mínimo de dextrosa sintética (SD) (1,7 g/L de base de nitrógeno de levadura con sulfato de amonio o 1,7 g/L de base de nitrógeno de levadura sin sulfato de amonio con 1 g/L de ácido glutámico monosódico, 2% de glucosa y suplementado con una mezcla completa o apropiada de aminoácidos).
- Cultivar 250-500 mL de cultivo celular a 0.5 OD600 (mid-log), a 30 °C, en un medio apropiado.
3. Recolección celular y lisis
- Recolecte rápidamente las células por filtración al vacío en una membrana secante de nitrocelulosa de 0,45 μm con un sistema de filtrado de vidrio (soporte de filtro de vidrio con embudo de vidrio de 1 L, base y tapa de vacío, pantalla de acero inoxidable, junta y abrazadera de resorte, 90 mm; matraz de junta molida 1 L).
- Congele rápidamente las células recolectadas, raspando las células peletizadas con una espátula e inmediatamente sumergiéndolas en un tubo líquido lleno de nitrógeno de 50 ml.
PUNTO DE PARADA: Las células pueden almacenarse a -80 °C durante un máximo de 3-4 semanas. - Realizar lisis celular por molienda criogénica en un molino mezclador: dos veces durante 2 min a 30 Hz, con 1 mL del tampón de lisis (ver Tabla 1). Enfriar en nitrógeno líquido entre moliendas.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| 10 mg/ml de CHX (cicloheximida) | 220 | 0,5 mg/ml |
| 1M Tris-HCl pH 8.0 | 88 | 20 metros |
| 3M KCl | 205.7 | 140 metros |
| 1M MgCl2 | 26.4 | 6 mM |
| 1M PMSF | 4.4 | 1 mM |
| NP-40 | 4.4 | 0.10% |
| Inhibidor de la proteasa | 2 comprimidos | |
| DNasa I | 8.8 | 0,02 U/ml |
| Volumen final | 4,400 |
Tabla 1: Receta para la mezcla maestra de tampón de lisis.
NOTA: El tampón de lisis se puede alterar para contener más inhibidores de la proteasa (como bestatina, leupeptina, aprotinina, etc.) en caso de que la proteína de interés sea muy inestable, pero es importante evitar el EDTA para mantener las subunidades pequeñas y grandes del ribosoma ensambladas durante los siguientes pasos. Por razones similares, mantenga siempre al menos 6 mM MgCl2 en la solución tampón.
PRECAUCIÓN: El HCl es altamente corrosivo y el PMSF es tóxico. Use guantes y manipule con cuidado.
- Centrifugar durante 2 min a 30.000 x g, 4 °C para limpiar el lisado y recoger el sobrenadante.
4. Purificación de complejos de cadenas ribosómicas-nacientes para SeRP
- Para cada experimento, divida el sobrenadante en dos partes; cada uno en un tubo de microcentrífuga diferente: muestra de ARN total (~200 μL) y muestra de inmunopurificación (IP) (~700 μL) muestras de translatoma.
-
Procesamiento de la muestra total de ARN
- Digerir la muestra de ARN total utilizando 10 U de RNasa I durante 25 min a 4 °C; gire a 30 RPM con un bastidor de mezcla giratorio.
NOTA: Las condiciones de digestión se pueden calibrar utilizando perfiles de polisomas para garantizar que no haya un pico de sobre digestión o subdigestión de monosomas. - Prepare la mezcla maestra del cojín de sacarosa como se describe en la Tabla 2.
- Digerir la muestra de ARN total utilizando 10 U de RNasa I durante 25 min a 4 °C; gire a 30 RPM con un bastidor de mezcla giratorio.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| 50% Sacarosa | 200 | 25% |
| 1M Tris-HCl pH 8.0 | 8 | 20 metros |
| 3M KCl | 18.7 | 140 metros |
| 1M MgCl2 | 4 | 10 mM |
| 100 mg/ml CHX | 0.4 | 0,1 mg/ml |
| Inhibidor de la proteasa | 1 comprimido | |
| Volumen final | 400 |
Tabla 2: Receta para la mezcla maestra de cojín de sacarosa.
- Cargue la muestra sobre 400 μL del cojín de sacarosa y la centrífuga en un rotor TLA120 durante 90 min a 245.000 x g y a 4 °C.
- Retire el sobrenadante rápidamente con una bomba de vacío y superponga los gránulos con un tampón de lisis de 150 μL. Resuspendir los pellets agitando durante 1 h a 4 °C y a 300 RPM.
- Resuspend el pellet residual mediante pipeteo y transfiéralo a un nuevo tubo de 1,5 ml.
NOTA: 100-200 μg de ARN total suele ser suficiente para el perfil de ribosoma del translatoma total. Se puede agregar el paso de agotamiento de ARNr para reducir la contaminación de ARNr, que es el contaminante más frecuente de las purificaciones de afinidad de complejos de cadenas ribosoma-nacientes11 (ver discusión para más detalles).
-
Procesamiento de la muestra de inmunopurificación
- Lavar 100-400 μL de la matriz de unión a la afinidad (perlas conjugadas con anticuerpos 1:1 en 70% EtOH) por muestra con 3 x 1 ml tampón de lisis (sin DNasa I e inhibidores de la proteasa); vuelva a suspender la matriz de afinidad en el búfer de lisis y luego gire a 30 RPM con un bastidor de mezcla giratorio a 4 °C durante 5 min. Precipitado por centrifugación durante 30 s a 3.000 x g, 4 °C. Deseche el líquido superior. Repetir tres veces.
- Digerir muestras de inmunopurificación utilizando 10 U por unidad A260 nm de RNasa I, junto con matriz de unión a afinidad (por ejemplo, 100-400 μL de GFP-TRAP por muestra).
- Gire durante 25 min a 30 RPM con un bastidor de mezcla giratorio para unir la proteína a la matriz de afinidad, a 4 °C.
- Prepare la mezcla maestra del tampón de lavado como se detalla en la Tabla 3.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| 10 mg/ml CHX | 50 | 0,1 mg/ml |
| 1M Tris-HCl pH 8.0 | 100 | 20 metros |
| 3M KCl | 233 | 140 metros |
| 1M MgCl2 | 50 | 10 mM |
| 1M PMSF | 5 | 1 mM |
| NP-40 | 0.5 | 0.01% |
| Inhibidor de la proteasa | 2 comprimidos | |
| 50% glicerol | 1,000 | 10% |
| Volumen final | 5,000 |
Tabla 3: Receta para la mezcla maestra del tampón de lavado.
- Lave la matriz de unión por afinidad tres veces con 1 ml de tampón de lavado, cada vez durante ~ 1 min, girando en el bastidor de mezcla a 30 RPM, a 4 ° C.
- Precipitar por centrifugación a 3.000 x g durante 30 s a 4 °C. Deseche el líquido superior.
- Lavar dos veces más en tampón de lavado de 1 ml, cada vez durante 5 min, girando en el estante de mezcla a 30 RPM, a 4 °C.
- Precipitado por centrifugación durante 30 s a 3.000 x g y 4 °C.
- Use 50 μL de perlas para la elución de proteínas con la misma cantidad de 2x tampón de muestra. Utilice el resto de las perlas para la extracción de ARN.
- Centrifugar durante 30 s a 3.000 x g, 4 °C para peletizar las perlas y desechar el líquido superior.
- Congelar en nitrógeno líquido y conservar a -80 °C. Utilice estas muestras para la extracción posterior de ARN.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante la noche o más. Esto puede ser un punto de parada. - Evalúe el éxito del paso de purificación de afinidad mediante western blot o tinción de Coomassie con alícuotas (~ 10% en volumen, después de la mezcla) de cada paso. Utilice siempre IP simulada en una cepa WT no etiquetada como control para el enlace no específico a la matriz de afinidad.
NOTA: El alto fondo no específico se puede superar mediante pasos de lavado adicionales con concentraciones crecientes de sal / detergente. La interacción transitoria puede estabilizarse mediante el tratamiento con varios agentes de reticulación, por ejemplo, el tratamiento con paraformaldehído (PFA) de las células vivas: se recomienda encarecidamente agregar 0.4% -1% de PFA a los medios de crecimiento durante 2-5 min, seguido de un enfriamiento de glicina (0.3 M) durante 3 min.
PRECAUCIÓN: El paraformaldehído es un carcinógeno sospechoso. Dado que el paraformaldehído se evapora rápidamente y es corrosivo, trabaje en una campana de seguridad química y use dos capas de guantes.
5. Preparación de la biblioteca de ADNc para la secuenciación profunda
- Extracción de ARN
NOTA: Trabaje con tubos antiadherentes de 1,5 ml sin RNasa para prevenir el posible agotamiento del ARN o el ADN.- Descongelar las muestras de los pasos 4.2.5 y 4.3.12 en hielo y resuspend muestras con 10 mM Tris-HCl pH 7.0 hasta un volumen final de 700 μL.
PRECAUCIÓN: El ácido-fenol y el cloroformo son volátiles y dañinos. Trabaje en una campana de seguridad química. - Agregue 40 μL de SDS al 20% a 0,7 ml de ELUCIONES TOTALES de ARN o IP. Cerrar e invertir un par de veces. La precipitación de proteínas debe volver las muestras blancas.
- Añadir 0,75 ml de ácido-fenol:cloroformo precalentado a las muestras. Selle bien los tubos y agite en un mezclador térmico a 1.400 RPM durante 5 min y a 65 °C. Enfriar muestras en hielo durante 5 min.
- Centrifugar el tubo a partir del paso 5.1.3. a 20.000 x g durante 2 min. Transfiera la capa acuosa superior a un tubo fresco y agréguele 0,7 ml de ácido-fenol:cloroformo.
- Incubar durante 5 min a temperatura ambiente, ocasionalmente vórtice. Centrifugadora durante 2 min a 20.000 x g. Transfiera la capa acuosa superior a un tubo fresco y agréguele 0,6 ml de cloroformo y vórtice.
- Centrifugadora durante 1 min a 20.000 x g. Transfiera la capa acuosa superior a un tubo fresco.
- Precipitar ácidos nucleicos añadiendo 78 μL de 3 M de NaOAc, pH 5,5, 2 μL de GlycoBlue y 0,75 mL de isopropanol. Vórtice a fondo durante 5 min. Incubar durante al menos 1 h a -80 °C o 16 h a -20 °C.
- Centrifugar durante 30 min a 20.000 x g y a 4 °C y desechar el sobrenadante. Lave los gránulos con 0,75 ml helados de etanol al 80%. Invierta los tubos para un lavado a fondo. Centrifugar a 20.000 x g durante 5 min a 4 °C, y luego desechar el sobrenadante.
- Girar hacia abajo a 450 x g, 4 °C durante 20 s y retirar el etanol restante y desechar los líquidos. Seque el pellet con una tapa abierta durante 5 min a 65 °C. Resuspendir las muestras de la siguiente manera: para IP resuspend la muestra en 10 μL de 10 mM Tris-HCl, pH 7.0. Para el análisis del translatoma total, resuspenda la muestra en 20 μL de 10 mM Tris-HCl, pH 7.0.
PUNTO DE PARADA: El ARN se puede almacenar a -80 °C durante meses.
- Descongelar las muestras de los pasos 4.2.5 y 4.3.12 en hielo y resuspend muestras con 10 mM Tris-HCl pH 7.0 hasta un volumen final de 700 μL.
- Cuantificar la concentración total de ARN mediante fluorometría
NOTA: Todos los siguientes materiales y superficies deben estar libres de RNasa mientras se prepara la biblioteca de ADNc para la secuenciación de próxima generación. Mientras manipula muestras de ARN, use guantes.- Diluir 1 μL de ARN total extraído de fenol ácido en 9 μL de 10 mM Tris-HCl, pH 7.0. Cuantificar usando un fluorómetro, como se indica en el sitio web del fabricante.
- Diluir las muestras que contienen 50 μg de ARN con 10 μL de 10 mM Tris-HCl, pH 7.0.
NOTA: No mida muestras de IP, use todo para el siguiente paso.
- Fragmentos de huella protegida de ribosoma purificador en gel
- Coloque un gel de poliacrilamida de TBE-urea al 15% y sumérjalo en 1x tampón de ejecución de TBE. Funcione durante 30 minutos a 200 V antes de la carga de la muestra. A cada muestra, agregue 20 μL de 2 tampón de muestra de TBE-urea.
NOTA: El tamaño de banda esperado es de alrededor de 25-35 nt. - Descongele una escalera de ADN de 10 pb y desnaturalice las muestras (no la escalera) a 80 ° C durante 2 minutos, y luego enfríe en hielo. Cargue cada muestra en cada otro carril. Haga funcionar el gel durante 50-70 min a 200 V.
- Diluya 6 μL de SYBR Gold (10,000 x concentrado) en 60 mL de 1x tampón TBE y manche mientras agita en cajas protegidas contra la luz durante 15-20 min. Mientras tiñe el gel, prepare un bisturí estéril y tubos rompegeladores de 0,5 ml en tubos etiquetados de 1,5 ml.
- Elimine las bandas deseadas con un bisturí estéril (use uno fresco o limpie bien entre las muestras) y coloque cada pieza de gel en un tubo rompefilulizadores.
- Tome una imagen del gel para asegurarse de que no queden residuos de muestra en el gel.
- Centrifugar los tubos que contienen rodajas cortadas a 20.000 x g durante 5 min a 4 °C y transferir las piezas de gel restantes del tubo rompefilulizadores al tubo de 1,5 ml.
- Agregue 0.5 mL de 10 mM Tris, pH 7.0. Agitar en un mezclador térmico a 1.400 RPM durante 10 min a 70 °C.
- Transfiera a una columna de acetato de celulosa con una punta de pipeta de orificio ancho y centrífuga a 20.000 x g durante 3 min a 4 °C.
- Transfiera el flujo a través de un nuevo tubo de 1,5 ml y enfríe sobre hielo.
- Para precipitar los ácidos nucleicos, agregue: 550 μL de IPA, 55 μL de 3 M NaOAc y 2 μL de GlycoBlue y vórtice para mezclar bien. Coloque las muestras a -80 °C durante al menos 1 h.
- Centrifugar durante al menos 1 h a 20.000 x g y 4 °C y desechar el sobrenadante. Lave los pellets con 0,75 ml de etanol helado al 80%. Invierta los tubos para un lavado a fondo hasta que los gránulos se separen del fondo. Centrifugar de nuevo a 20.000 x g durante 5 min a 4 °C y desechar el sobrenadante.
- Girar hacia abajo a 450 x g, 4 °C durante 20 s y retirar el etanol restante. Seque los gránulos con una tapa abierta durante 5 min a 65 °C.
- Añadir 15 μL de 10 mM Tris, pH 7.0 y resuspendir los pellets a fondo. Gire hacia abajo a 450 x g, 4 °C durante 20 s y transfiera la muestra a un nuevo tubo de 1,5 ml.
PUNTO DE PARADA: El ARN purificado se puede almacenar a -80 °C durante unos meses.
- Coloque un gel de poliacrilamida de TBE-urea al 15% y sumérjalo en 1x tampón de ejecución de TBE. Funcione durante 30 minutos a 200 V antes de la carga de la muestra. A cada muestra, agregue 20 μL de 2 tampón de muestra de TBE-urea.
- Desfosforilación
- Utilice 3 μL de la siguiente mezcla para cada muestra: Agregue 1 μL de inhibidor de la RNasa en 2 μL de tampón de reacción de polinucleótido quinasa T4 T4 sin ATP. Añadir 2 μL de polinucleótido quinasa T4 a cada muestra. Pipetear suavemente para mezclar bien e incubar a 37 °C durante 2 h, sin agitar.
- Para inactivar la enzima, incubar la muestra a 75 °C durante 10 min y girar hacia abajo a 450 x g, 4 °C, durante 20 s. Añadir 0,5 mL de 10 mM Tris, pH 7,0.
- Para precipitar ácido nucleico, agregue 2 μL de GlycoBlue, 550 μL de IPA y 55 μL de 3 M de NaOAc.
- Vórtice para mezclar bien y enfriar las muestras a -80 °C durante al menos 1 h.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante la noche o más. - Repita los pasos 5.3.11-5.3.13.
PUNTO DE PARADA: El ARN desfosforilado puede almacenarse a -80 °C durante meses.
- Cuantificación mediante un bioanalizador
- Haga una dilución 1:4 de cada muestra de ARN mezclando 1 μL de muestra y 4 μL de agua tratada con DEPC.
PRECAUCIÓN: DEPC es un carcinógeno. Use guantes y trabaje con cuidado. - Ejecute un chip/tapeStation de ARN pequeño bioanalizador. Siga el protocolo del fabricante.
NOTA: El tamaño esperado del fragmento de ARN protegido por ribosomas es de alrededor de 28-30 nt.
- Haga una dilución 1:4 de cada muestra de ARN mezclando 1 μL de muestra y 4 μL de agua tratada con DEPC.
- Ligate 3' final con Linker-1
- Diluir 5 pmol de pequeños fragmentos de ARN a 10 μL con 10 mM Tris, pH 7.0. Desnaturalizar las muestras a 80 °C durante 2 min y enfriarlas sobre hielo.
- Prepare la mezcla maestra como se detalla en la Tabla 4 y use 29 μL por muestra.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| 50% PEG 8000 filtrado estéril | 16 | 20% |
| DMSO | 4 | 10% |
| 10× tampón de ARN ligasa 2 T4 | 4 | 1x |
| INHIBIDOR DE LA ARNasa SUPERase-In | 2 | 2 U |
| Enlazador adenilado 3-L1 de 10 mM | 0.1 | 25 μM |
| Agua tratada con DEPC | 2.9 | |
| Volumen final | 29 |
Tabla 4: Receta para la mezcla maestra de ligadura de 3'.
- Añadir 1 μL de ARN ligasa 2 T4 y pipetear suavemente para mezclar bien. Incubar a 23 °C durante 2 h.
- Para precipitar los ácidos nucleicos, añadir: 550 μL de IPA, 500 μL de 10 mM Tris, pH 7.0, 55 μL de 3 M NaOAc y 2 μL de GlycoBlue. Vórtice para mezclar bien, y colocar las muestras a -80 °C durante 1 h como mínimo.
PUNTO DE PARADA: Conservar las muestras a -80 °C durante la noche o más. - Repita los pasos 5.3.2-5.3.12.
- Resuspendir el pellet en 6 μL de 10 mM Tris, pH 7.0. Gire hacia abajo a 450 x g, 4 °C durante 20 s, y transfiera la muestra a un nuevo tubo de 1,5 ml.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante meses.
-
Purificación en gel de huellas unidas a 3'
- Coloque un gel de poliacrilamida de TBE-urea al 10% y sumérjalo en 1x tampón de ejecución de TBE. Funcione durante 30 minutos a 200 V antes de la carga de la muestra. A cada muestra, agregue 6 μL de 2 tampón de muestra de TBE-urea.
NOTA: El tamaño de banda esperado es de alrededor de 71-73 nt. - Repita los pasos 5.3.2-5.3.13.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante meses.
- Coloque un gel de poliacrilamida de TBE-urea al 10% y sumérjalo en 1x tampón de ejecución de TBE. Funcione durante 30 minutos a 200 V antes de la carga de la muestra. A cada muestra, agregue 6 μL de 2 tampón de muestra de TBE-urea.
-
Transcribir inversamente fragmentos de huella vinculados a 3ʹ para generar ssDNA
- Prepare una mezcla maestra como se detalla en la Tabla 5 y use 3 μL por muestra.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| DNTP de 10 mM | 1 | 0,5 mM |
| Enlazador de 25 μM L(rt) | 0.5 | 625 nM |
| Agua tratada con DEPC | 1.5 | |
| Volumen final | 3 |
Tabla 5: Receta para la mezcla maestra tampón de transcripción inversa antes de la desnaturalización de los ácidos nucleicos.
- Vórtice y gire hacia abajo la muestra.
- Incubar muestras a 65 °C durante 5 min.
- Enfriar muestras sobre hielo.
- Prepare una mezcla maestra como se detalla en la Tabla 6 y use 6 μL por muestra. Vórtice y gire hacia abajo la muestra. Añadir 1 μL de Superíndice III a cada muestra y pipetear suavemente para mezclar bien e incubar durante 30 min a 50 °C.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| Búfer FS de 5× | 4 | 1x |
| INHIBIDOR DE LA ARNasa SUPERase-In | 1 | 2 U |
| TDT 0,1 M | 1 | 5 mM |
| Volumen final | 6 |
Tabla 6: Receta para la mezcla maestra tampón de transcripción inversa después de la desnaturalización de los ácidos nucleicos.
- Añadir 2,3 μL de 1 N NaOH, que hidroliza el ARN y apaga la transcripción inversa.
PRECAUCIÓN: El NaOH es altamente corrosivo. Use guantes y protección ocular. - Incubar durante 15 min a 95 °C, hasta que la muestra se vuelva rosada.
- Coloque un gel de poliacrilamida de TBE-urea al 10% y sumérjalo en 1x tampón de ejecución de TBE. Funcione durante 30 minutos a 200 V antes de la carga de la muestra. A cada muestra, agregue 23 μL de 2 tampón de muestra de TBE-urea.
NOTA: El tamaño esperado de la banda de ADN es de 115-117 nt. - Repita los pasos 5.3.2-5.3.12.
- Resuspend el pellet en 15 μL de 10 mM Tris, pH 8.0. Gire hacia abajo a 450 x g, 4 °C durante 20 s y transfiera la muestra a un nuevo tubo de 1,5 ml.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante meses.
-
Circularización ssDNA
- Prepare la siguiente mezcla maestra y cargue 4 μL por muestra, como se detalla en la Tabla 7.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| Búfer CircLigase II de 10× | 2 | 1x |
| 5 M de betaína (opcional) | 1 | 0,25 M |
| 50 mM MnCl2 | 1 | 2,5 mM |
| Volumen final | 4 |
Tabla 7: Receta para la mezcla maestra de circularización ssDNA.
- Añadir 1 μL de CircLigase II ssDNA ligasa a cada muestra e incubar durante 1 h a 60 °C.
NOTA: La eficiencia de este paso se puede aumentar agregando 1 μL de CircLigase II ssDNA ligasa a cada muestra después de 1 h de incubación. - Inactivar la enzima incubando a 80 °C durante 10 min.
- Enfríe sobre hielo y continúe con la amplificación por PCR o almacene a -80 °C.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante años.
-
Amplificación por PCR
- Prepare la siguiente mezcla maestra de PCR y cargue 82 μL por muestra, como se detalla en la Tabla 8.
| Reactivo | Cantidad por muestra (μL) | Concentración final |
| Agua tratada con DEPC | 61.6 | |
| 5× Tampón de reacción Phusion HF | 17.6 | 1x |
| DNTP de 10 mM | 1.8 | 200 μM |
| Imprimación delantera pcr de 100 μM | 0.2 | 225 nM |
| HF Fosfión polimerasa | 0.8 | 1,6 U |
| Volumen final | 82 |
Tabla 8: Receta para la mezcla maestra de amplificación por PCR.
- A cada tubo que contenga la mezcla maestra, agregue 5 μL de ADN circularizado.
NOTA: Guarde el resto de las muestras de ADN circularizadas a -80 °C. - Agregue un cebador de código de barras inverso de PCR de 20 μM diferente a cada muestra (consulte la Tabla 9) y vórtice para mezclar bien.
- Alícuota cada tubo en cuatro tubos de PCR separados, cada uno se utilizará para un número diferente de ciclos de PCR.
- Ejecute una reacción de PCR de acuerdo con el siguiente programa, como se detalla en la Tabla 10.
| Ciclo | Denaturalización (98 °C) | Anneal (60 °C) | Extender (72 °C) |
| 1 | 30 s | ||
| 2-16 | 10 s | 10 s | 5 s |
Tabla 10: Programa de PCR para reacción PCR.
- Después de los ciclos 8, 9, 10 y 11, retire los tubos de PCR (para muestras IP, los ciclos varían de 9 a 15) como primer intento. Después de cada ciclo, haga una pausa en el programa, saque una alícuota y póngala en hielo, y luego reanude rápidamente el programa.
NOTA: El número de ciclos debe ajustarse en función de la cantidad de ADN circularizado en cada reacción. Consulte la Figura 4 para obtener un ejemplo y más aclaraciones. - A cada reacción de 17 μL, agregue 3.5 μL de 6x colorante de carga de ADN.
- Descongelar una escalera de ADN de 10 pb.
- Para la separación de tamaño por electroforesis en gel, sumerja la poliacrilamida TBE al 8% en 1x tampón de funcionamiento TBE y cargue las muestras de cada número de ciclo diferente en pozos adyacentes y ejecute el gel durante 50 min a 180 V.
- Diluya 6 μL de SYBR Gold (10,000 x concentrado) en 60 mL de 1x tampón TBE y manche mientras agita en cajas protegidas contra la luz durante 15-20 min.
- Mientras tiñe el gel, prepare un bisturí estéril y tubos rompegeladores de 0,5 ml en tubos etiquetados de 1,5 ml.
- Tome una imagen de los ácidos nucleicos teñidos.
- Corte la banda deseada con un tamaño de banda esperado de 174-176 pb con el bisturí estéril y coloque la rodaja de gel en el tubo de gel-rompedor de gel de 0,5 ml preparado (limpie a fondo entre las muestras y use un agente inactivador de RNasa, o cambie a una cuchilla nueva).
- Centrifugar los tubos durante 5 min a 20.000 x g y 4 °C, y luego transferir las piezas de gel restantes del tubo de gel-rompedor de 0,5 ml al tubo de 1,5 ml.
- Añadir 500 μL de Tris de 10 mM, pH 8.0 y agitar en una batidora térmica a 1.400 RPM durante 10 min y 70 °C.
- Transfiera el gel disuelto a una columna de acetato de celulosa con una punta de pipeta de orificio ancho.
- Centrifugar la columna durante 3 min a 20.000 x g y 4 °C y transferir el flujo a través a un nuevo tubo de 1,5 ml y enfriar sobre hielo.
- Para precipitar el ácido nucleico, agregue: 550 μL de IPA, 32 μL de 5 M NaCl, 1 μL de 0.5 M EDTA y 2 μL de GlycoBlue y vórtice para mezclar bien.
- Mantenga las muestras durante al menos 1 h a -80 °C, o -20 °C durante la noche.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante la noche o más. - Repita los pasos 5.3.11-5.3.12.
- Resuspend en 11 μL de 10 mM Tris, pH 8.0. Gire hacia abajo a 450 x g, 4 °C durante 20 s y transfiera la muestra a un nuevo tubo de 1,5 ml.
PUNTO DE PARADA: Las muestras se pueden almacenar a -80 °C durante años.
-
Cuantificar la distribución del tamaño por bioanalizador
- Haga una dilución 1:4 de cada muestra mezclando 1 μL de muestra con 4 μL de agua tratada con DEPC.
- Ejecute el chip de ARN pequeño bioanalizador. Siga el protocolo del fabricante.
NOTA: La longitud esperada es de 175 ± 5 pb.
-
Cuantificar la concentración de ADN por fluorómetro
- Realice una comprobación de concentración de alta sensibilidad de dsDNA con un fluorómetro de acuerdo con las recomendaciones del fabricante.
- Muestras multiplex y secuencias según las recomendaciones de Illumina (Index Adapters Pooling Guide12).
6. Análisis de datos
- Realizar el análisis tal y como se detalla en el archivo complementario.
Resultados
Como se ilustra en el diagrama de flujo de este protocolo (Figura 1), las células se cultivaron hasta la fase de registro, y luego se recolectaron rápidamente por filtración y se lisaron mediante molienda criogénica. El lisado se dividió en dos: uno para las huellas totales de ARNm protegido por ribosomas y el otro para las huellas de ARNm protegidas por ribosomas seleccionadas, en las que realizamos la purificación de afinidad para reducir los complejos de cadenas nacientes proteína-ribosoma-naciente marcados. Aseguramos la expresión de proteínas etiquetadas y el éxito del pull-down mediante el análisis de western blot, como se puede ver en la Figura 2. Validamos el aislamiento de huellas protegidas por ribosomas, que suelen tener una longitud de 20-45 nt nt mediante electroforesis de ARN pequeño (sistema BioAnalyzer 2100), lo que permite un cambio de 5-10 nt en la detección de tamaño, según el manual del sistema (Figura 3). Luego, generamos una biblioteca de CDNA para secuenciación profunda y análisis de big data. Al generar la biblioteca de ADNc, tenga en cuenta que el subciclamiento puede conducir a un bajo rendimiento (como se puede ver en el carril 2 de la Figura 3), pero la reamplificación es posible para recuperar la biblioteca generada. El ciclo excesivo puede ocurrir cuando los cebadores de PCR se agotan, pero la reacción continúa. Cuando los dNTP todavía están presentes, la reacción procede, generando artefactos de PCR más largos con secuencias quiméricas debido a que los productos de PCR se ceban a sí mismos13 (como se puede ver en los carriles 3-4 de la Figura 3, indicados por el frotis visible). Si la concentración de dNTP también se vuelve limitante, pueden aparecer productos que indiquen la presencia de heterodúplex compuestos solo parcialmente por fragmentos de biblioteca homólogos. La Figura 4 actúa como referencia, con el carril 2 representando la amplificación óptima, y el carril 3 una amplificación aceptable. No se deben utilizar muestras de los carriles 4 y 5 (ciclos 10 y 11) debido a la posibilidad de introducir duplicados y artefactos de PCR. La biblioteca generada fue validada además por electroforesis de ADN de alta sensibilidad (se utilizó el mismo sistema BioAnalyzer) para la distribución y cuantificación exacta del tamaño (Figura 5). Después de la ligadura del enlazador final de 3', la transcripción inversa y la amplificación por PCR, se espera una distribución de longitud de ADNc como tal, con un pico agudo alrededor de 175 nt.
Recortamos y eliminamos adaptadores y códigos de barras de la biblioteca secuenciada, y solo se seleccionaron las lecturas entre 20 y 45 nt para un análisis posterior. La Figura 6 muestra la distribución de longitud resultante. Las lecturas se dividieron en diferentes grupos de: secuencias codificantes, intrones y secuencias intergénicas (Figura 7), y se clasificaron como se muestra en la Figura 8.
El análisis final para la detección y caracterización de interacciones co-traduccionales se realizó a partir del enriquecimiento de fragmentos de ARNm protegidos por ribosomas, produciendo los gráficos de la Figura 9. Se comparó la ocupación normalizada del ribosoma (en cada nucleótido a lo largo de cada orf) del translatoma total con su correspondiente translatoma seleccionado (interactoma naciente). La comparación por nucleótido elimina los artefactos de las tasas de traducción. La reproducibilidad entre réplicas biológicas se evaluó mediante correlación de Pearson (umbral > 0,6). Presentamos Perfiles Selectivos de Ribosomas, analizando las interacciones co-traduccionales de Vma2p con tres proteínas: la chaperona asociada al ribosoma Ssb1p, Pfk2p (Fosfofructoquinasa) y Fas1p (Ácido graso sintasa), con cada proteína C' terminalmente marcada por GFP. Realizamos el protocolo en réplicas biológicas. Figura 9 A, D y G muestran el esquema experimental de cada purificación de afinidad. A continuación mostramos la ocupación ribosómica de los traducomas totales en comparación con los interactomas Ssb1 a lo largo del orf Vma2p, codificando para una subunidad de la H+-ATPasa vacuolar (Figura 9 B, E y H). Finalmente, se realizó un perfil de enriquecimiento de ribosomas basado en la relación (IP/Total) en cada posición del ribosoma en [codón/aa] a lo largo del orf (Figura 9 C, F e I). La comparación de las interacciones co-traduccionales de estas tres proteínas con Vma2p, que está siendo sintetizado por el ribosoma, reveló que la chaperona Ssb1 involucra al Vma2p naciente en cuatro regiones diferentes a lo largo del orf, ya que identificamos cuatro picos de enriquecimiento significativos por SeRP. De manera diferente, Pfk2p muestra solo un pico de enriquecimiento significativo, según lo identificado por SeRP, en una posición diferente en comparación con el acompañante co-traduccional Ssb1. El análisis de las interacciones co-traduccionales de Fas1 con Vma2p naciente no detectó ningún enriquecimiento significativo. Por lo tanto, la comparación de estos perfiles de ribosomas de enriquecimiento basados en la relación demuestra el poder de este protocolo en la detección y caracterización de diversas interacciones co-traduccionales en resolución cercana al codón.
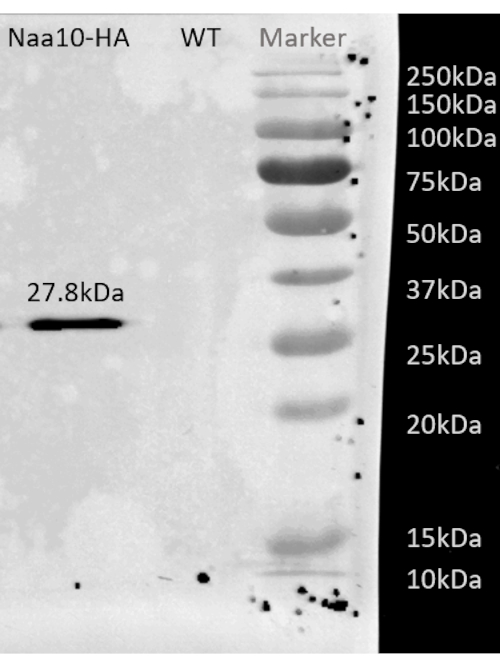
Figura 2: Resultado representativo de western blot después de la purificación por afinidad de la cepa BY4741 con Naa10 marcada con HA. Resultado representativo de western blot después de la purificación por afinidad de la cepa BY4741 con Naa10 marcada con HA que muestra una banda alrededor de 27.8 kDa, mientras que el tipo salvaje, como control negativo, no muestra banda. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Resultado representativo del bioanalizador después del aislamiento de la huella y la extracción de ARN con ácido-fenol: cloroformo, y un tamaño promedio de 25 nt. Haga clic aquí para ver una versión más grande de esta figura.
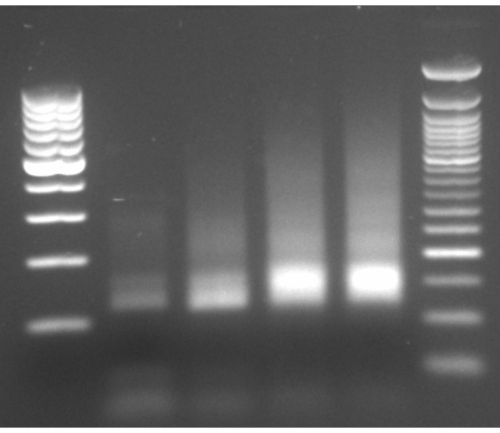
Figura 4: Electroforesis en gel representativa de la amplificación por PCR. Electroforesis en gel representativa de amplificación por PCR con carriles 2-5 cargados con productos de PCR de los ciclos 8-11, y escaleras por ambos lados. Haga clic aquí para ver una versión más grande de esta figura.
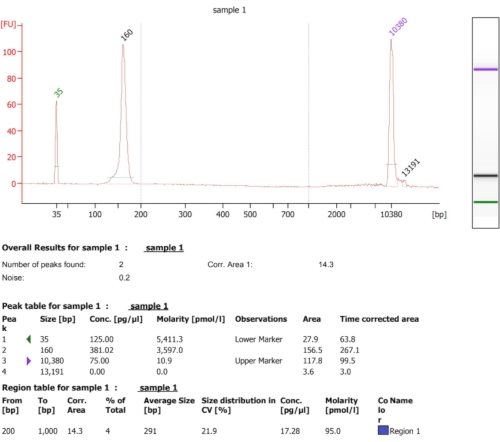
Figura 5: Resultado representativo de BioAnalyzer obtenido tras la creación de una biblioteca de ADNc. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Distribución de longitud esperada de las lecturas después de la extracción de los adaptadores con Cutadapt (eliminación de lecturas inferiores a 20 o superiores a 45). Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Porcentaje de éxito de alineación esperado después de eliminar las lecturas de ARN no codificantes con Bowtie2 y usar TopHat para alinear las lecturas restantes a diferentes organismos. La muestra fue tomada de S. cerevisiae (una variante mutada de BY4741). Haga clic aquí para ver una versión más grande de esta figura.
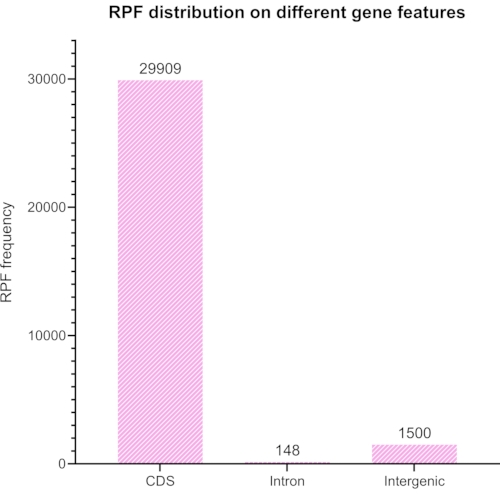
Figura 8: Un gráfico generado con RiboToolkit que representa la relación de codificación esperada versus no codificación de lecturas alineadas después de usar Bowtie2 para eliminar elementos de ARNr en las lecturas. Haga clic aquí para ver una versión más grande de esta figura.
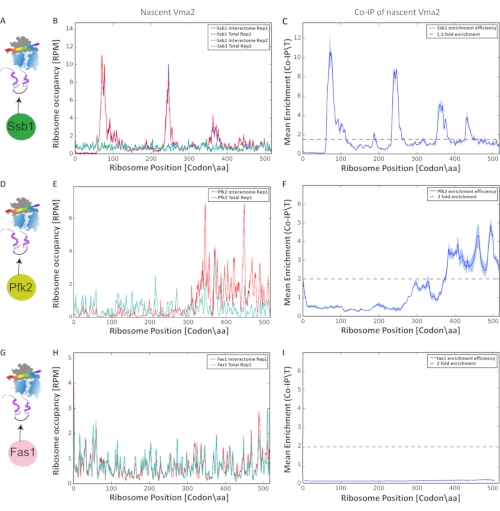
Figura 9: Interacciones cotraduccionales de tres proteínas diferentes: Ssb1p, Pfk2p y Fas1p con Vma2p, que está siendo sintetizado por el ribosoma, analizado por SeRP. Todos los ejes y se muestran en lecturas por millón (RPM). (A, D, G) Esquema experimental de SeRP de Ssb1p, Pfk2p y Fas1p C' etiquetado terminalmente por GFP, respectivamente. (B, E, H) Ocupación de ribosomas a lo largo del orf de los traducomas totales en comparación con los interactomas Ssb1, Pfk2p y Fas1p, respectivamente (en réplicas biológicas). (C, F, I) Enriquecimiento medio de Ssb1p, Pfk2p y Fas1p (relación IP/Total) en cada posición del ribosoma en [codón/aa] a lo largo del orf, respectivamente. La variación entre las réplicas biológicas está indicada por el área sombreada. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 9: 3' Secuencias de enlazador y cebador. 3' Linker L1: Enlazador 3-L1 con adenilación 5ʹ e identificadores moleculares únicos de didesoxi-citidina 3ʹ ('NN...') (Purificación de HPLC sin RNasa; Enlazador de transcripción inversa: transcripción inversa (L(rt)) con identificadores moleculares únicos fosforilados de 5ʹ (purificación hpLC libre de RNasa); Imprimación delantera de PCR: PCRf; HPLC purificado. Haga clic aquí para descargar esta tabla.
Expediente complementario. Haga clic aquí para descargar este archivo.
Discusión
Aquí, el protocolo detalla el enfoque de perfil selectivo de ribosomas para capturar interacciones co-traduccionales en resolución cercana al codón. A medida que el ribosoma se eleva como un centro para coordinar la aparición de la cadena naciente en el citoplasma abarrotado, este es un método crucial para identificar y caracterizar las diversas interacciones co-traduccionales necesarias para garantizar un proteoma funcional, así como para estudiar diversas enfermedades. Hasta la fecha, SeRP es el único método que puede capturar y caracterizar estas interacciones, de manera directa, in vivo 14,15,16.
El primer y más crítico paso es la recolección de células y la lisis. Es imperativo capturar, en cuestión de segundos, la traducción continua, mediante congelación instantánea seguida de lisis en un estado congelado. La recolección de células debe hacerse con prisa para evitar la escorrentía ribosómica, así como inducir respuestas traslacionales de estrés, que pueden ocurrir rápidamente. El segundo paso crítico es el paso de purificación de afinidad. Es imperativo reducir la unión de fondo mediante un lavado estricto al tiempo que se asegura de que se mantengan las interacciones co-traslacionales, lo que puede facilitarse mediante la reticulación in vivo . Como este protocolo se basa en el altamente sensible NGS (Next Generation Sequencing), el alto fondo en los primeros pasos se puede amplificar en los siguientes pasos de preparación de la biblioteca de ADNc, lo que lleva a bajas relaciones señal-ruido.
El tratamiento con nucleasas, para digerir todos los ARNm no protegidos, debe evaluarse mediante el perfil de polisoma17 junto con una evaluación cuidadosa de la distribución del tamaño de las huellas ribosómicas aisladas (como se detalló anteriormente) para evitar la digestión por encima o por debajo del ARN. La calibración de la concentración de nucleasa y los tiempos de digestión pueden facilitar la recuperación precisa de la huella, ya que la sobredigestión puede conducir a la digestión ribosómica de ARNr, lo que lleva a la pérdida de huellas protegidas por ribosomas. Es importante tener en cuenta que la subdigestión también puede conducir a tasas de descubrimiento más bajas de huellas protegidas por ribosomas, ya que los pasos de preparación de la biblioteca de ADNc, así como los pasos de análisis de datos descritos aquí descartan lecturas largas y poco características.
Si bien el agotamiento del ARNr no siempre constituye un paso crítico y no es obligatorio, tiene algunas ventajas, como muestras más limpias y, por lo tanto, una mayor tasa de lecturas mapeadas por el genoma. Por otro lado, existe la posibilidad de sesgos, ya que muchos protocolos de agotamiento de ARNr también podrían causar el agotamiento de los fragmentos protegidos por ribosomas deseados. También se deben tener en cuenta los costos de los kits de agotamiento de ARNr. El agotamiento del ARNr se puede realizar después del paso de aislamiento de la huella del ribosoma del pie o después del paso de circularización del ADNc.
Los pasos de preparación de la biblioteca de aDNc, como se describe aquí, se han optimizado para una baja entrada de ARNm, ya que los pasos de afinidad y purificación de ribosomas reducen en gran medida la cantidad de entrada de ARNm, en comparación con los estudios de expresión de ARN-seq. La ampliación de la cantidad inicial de cultivos celulares puede facilitar en gran medida la generación de bibliotecas de ADNc. Alternativamente, cualquier protocolo de biblioteca de ADNc de elección puede encajar con los pasos de purificación de afinidad y aislamiento de huella descritos aquí. Es importante tener en cuenta que el tratamiento con nucleasa que genera las huellas ribosómicas requiere la reparación final del ARNm resultante (preparación de la biblioteca de ADNc, paso de desfosforilación) para permitir seguir los pasos de ligadura del enlazador en el protocolo de ADNc descrito aquí en su protocolo de elección.
Durante la secuenciación, es importante diferenciar SeRP de RNA-Seq, ya que la heterogeneidad de las bibliotecas generadas varía mucho, dependiendo de los factores de afinidad marcados. Las chaperonas moleculares y los factores de orientación a menudo son más promiscuos en la unión, interactuando con cientos o miles de sustratos durante la traducción, lo que lleva a bibliotecas de ADNc muy diversas. Sin embargo, los interactores altamente específicos, como los interactores de ensamblaje complejos co-traduccionales, a menudo pueden conducir a la generación de bibliotecas de ADNc mucho menos diversas. El pico de bibliotecas diversas y no diversas en el mismo carril puede mejorar en gran medida la secuenciación y el seguimiento de los resultados del análisis de datos.
Otra característica única de SeRP es su capacidad para capturar variaciones locales en las ocupaciones de ribosomas a lo largo del orf , lo que permite el descubrimiento de cambios ribosómicos en la tasa de traslación asociada con cada conjunto de interacciones. Por lo tanto, es imperativo comparar las ocupaciones de ribosomas en cada codón a lo largo del orf para identificar correctamente el enriquecimiento. La utilización de promedios orfs puede conducir a la pérdida de interacciones transitorias o falso descubrimiento.
El uso correcto del método SeRP abre muchas vías de co-traducción al análisis directo, descubriendo nuevas características mecanicistas, así como nuevos factores asociados a ribosomas, revolucionando el campo de la biosíntesis de proteínas.
Divulgaciones
Los autores declaran que no hay conflictos de intereses.
Agradecimientos
Nos gustaría agradecer a todos los miembros del laboratorio por las fructíferas discusiones y a Muhammad Makhzumy por la lectura crítica del manuscrito. Este trabajo fue financiado por la subvención 2106/20 de la ISF (Fundación Israelí para la Ciencia).
Materiales
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referencias
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters - Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados