Method Article
Identification globale des réseaux d’interaction co-translationnelle par profilage sélectif des ribosomes
Dans cet article
Résumé
Les interactions co-traductionnelles jouent un rôle crucial dans les modifications de la chaîne naissante, le ciblage, le pliage et les voies d’assemblage. Nous décrivons ici le profilage sélectif des ribosomes, une méthode d’analyse directe in vivo de ces interactions dans le modèle eucaryote Saccharomyces cerevisiae.
Résumé
Ces dernières années, il est devenu évident que les ribosomes non seulement décodent notre ARNm, mais guident également l’émergence de la chaîne polypeptidique dans l’environnement cellulaire encombré. Les ribosomes fournissent la plate-forme pour la liaison contrôlée spatialement et cinétiquement des facteurs de ciblage membranaire, modifiant les enzymes et pliant les chaperons. Même l’assemblage en complexes oligomères d’ordre élevé, ainsi que les étapes de formation du réseau protéine-protéine, ont récemment été découverts pour être coordonnés avec la synthèse.
Nous décrivons ici le profilage sélectif des ribosomes, une méthode développée pour capturer les interactions co-translationnelles in vivo. Nous détaillerons les différentes étapes de purification d’affinité requises pour capturer les complexes de chaîne naissante de ribosomes avec des interacteurs co-traductionnels, ainsi que les étapes d’extraction de l’ARNm, d’exclusion de taille, de transcription inverse, de séquençage en profondeur et d’analyse de données volumineuses, nécessaires pour déchiffrer les interactions co-traductionnelles en résolution proche du codon.
Introduction
Lerofilage de l’ibosome RR (SeRP) est la seule méthode, à ce jour, qui capture et caractérise les interactions co-translationnelles, in vivo, de manière directe 1,2,3,4,5,6. SeRP permet un profilage global des interactions de n’importe quel facteur avec la traduction des ribosomes en résolution proche du codon 2,7.
La méthode repose sur la congélation éclair des cellules en croissance et la préservation de la traduction active. Les lysats cellulaires sont ensuite traités avec de la RNase I pour digérer tout l’ARNm dans la cellule, à l’exception des fragments d’ARNm protégés par les ribosomes appelés « empreintes de ribosomes ». L’échantillon est ensuite divisé en deux parties; une partie est directement utilisée pour l’isolement de toutes les empreintes ribosomiques cellulaires, représentant toute la traduction en cours dans la cellule. La deuxième partie est utilisée pour la purification d’affinité du sous-ensemble spécifique de ribosomes associés à un facteur d’intérêt, par exemple: enzymes modificatrices, facteurs de translocation, chaperons de repliement et interactions d’assemblage complexe. Les empreintes ribosomiques purifiées par affinité sont collectivement appelées interactome. Ensuite, les ARNm protégés par les ribosomes sont extraits et utilisés pour la génération de bibliothèque d’ADNc, suivie d’un séquençage en profondeur.
L’analyse comparative des échantillons totaux de translatome et d’interactome permet d’identifier tous les orfs associés au facteur d’intérêt, ainsi que la caractérisation de chaque profil d’interaction orf. Ce profil rapporte les séquences précises d’apparition et de terminaison de l’engagement à partir desquelles on peut déduire les codons décodés et les résidus respectifs de la chaîne polypeptidique émergente, ainsi que sur les variations de vitesse des ribosomes au cours de l’interaction 7,8. La figure 1 illustre le protocole sous forme de schéma.
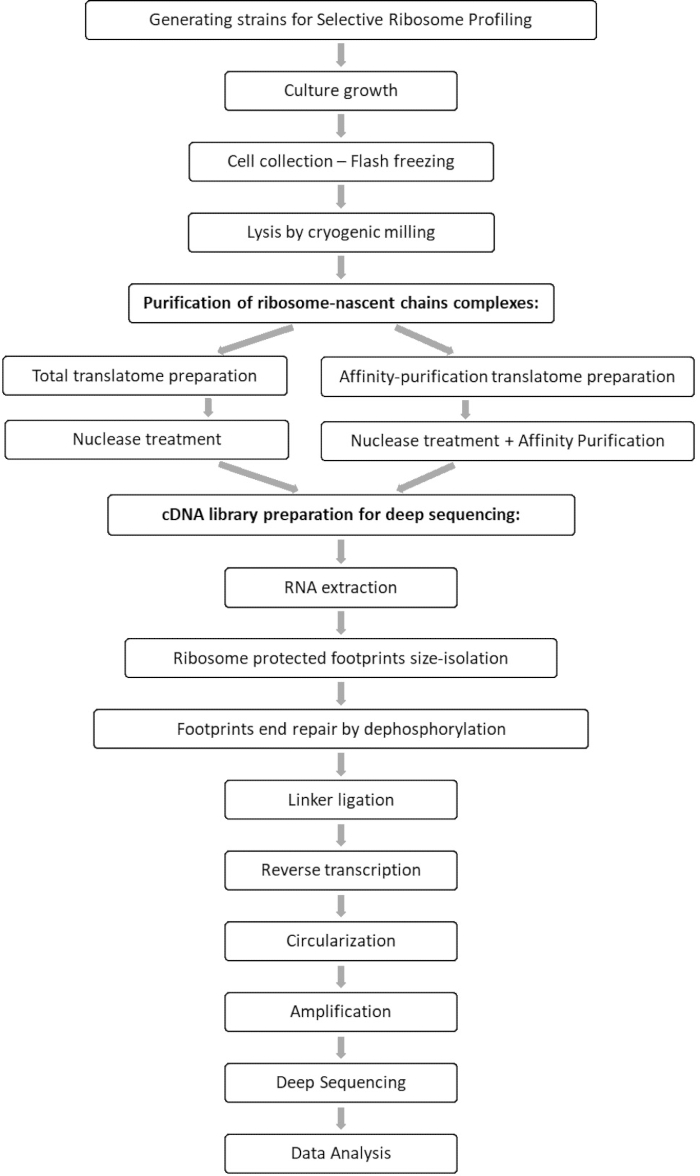
Figure 1 : Vue d’ensemble du protocole SeRP. Ce protocole peut être effectué dans son intégralité dans un délai de 7 à 10 jours. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Génération de souches pour le profilage sélectif des ribosomes
NOTE: Lerofiling Ribosome P(SeRP) est une méthode qui repose sur la purification d’affinité des facteurs d’intérêt, pour évaluer leur mode d’interaction avec les complexes de chaînes naissantes de ribosomes. La recombinaison homologue9, ainsi que les méthodes basées sur CRISPR/Cas910 sont utilisées pour fusionner divers facteurs d’intérêt avec des étiquettes pour les purifications d’affinité. Ces étiquettes sont GFP, pour les purifications d’affinité de piège GFP, TAP-tag pour les purifications de perles d’IgG-Sepharose ainsi que AVI-Tag purifié par avidine ou streptavidine, pour énumérer quelques exemples réussis de ces dernières années.
- Effectuer des tests de croissance ou fonctionnels pour valider que le marquage n’a pas eu d’impact sur la fonction des protéines. L’étiquetage des terminaux N’par rapport à C’doit être évalué.
REMARQUE: Les ribosomes (ARNr), ainsi que de nombreux domaines de liaison aux ribosomes dans divers facteurs, sont très chargés, ce qui rend les étiquettes très chargées (telles que la polyhistidine) impréférables à utiliser, car elles peuvent conduire à une fausse découverte ou à un mode de liaison modifié.
2. Croissance de la culture
- Cultiver les cultures de levure construites (basées sur la souche BY4741), contenant les protéines marquées souhaitées, soit dans un milieu riche en extrait de levure liquide-peptone-dextrose (YPD), soit dans un milieu minimal de dextrose synthétique (SD) (1,7 g/L de base d’azote de levure avec sulfate d’ammonium ou 1,7 g/L de base d’azote de levure sans sulfate d’ammonium avec 1 g/L d’acide glutamique monosodique, 2 % de glucose et complété par un mélange complet ou approprié d’acides aminés).
- Cultiver 250 à 500 mL de culture cellulaire à 0,5 OD600 (mi-log), à 30 °C, dans un milieu approprié.
3. Collecte et lyse des cellules
- Collectez rapidement les cellules par filtration sous vide sur une membrane de buvard de nitrocellulose de 0,45 μm avec un système de filtrage en verre (porte-filtre en verre avec entonnoir en verre de 1 L, base et capuchon en verre, écran en acier inoxydable, joint et pince à ressort, 90 mm; fiole à joint de terre 1 L).
- Flash congèle les cellules collectées, en grattant les cellules granulées avec une spatule et en les immergeant immédiatement dans un tube de 50 mL rempli d’azote liquide.
POINT D’ARRÊT: Les cellules peuvent être conservées à -80 ° C pendant 3-4 semaines. - Effectuer la lyse cellulaire par broyage cryogénique dans un broyeur mélangeur : deux fois pendant 2 min à 30 Hz, avec 1 mL du tampon de lyse (voir tableau 1). Refroidir dans l’azote liquide entre les broyages.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 10 mg/mL DE CHX (cycloheximide) | 220 | 0,5 mg/mL |
| 1M Tris-HCl pH 8,0 | 88 | 20 mM |
| 3M KCl | 205.7 | 140 mM |
| 1M MgCl2 | 26.4 | 6 mM |
| 1M PMSF | 4.4 | 1 mM |
| NP-40 | 4.4 | 0.10% |
| Inhibiteur de la protéase | 2 comprimés | |
| DNase I | 8.8 | 0,02 U/mL |
| Volume final | 4,400 |
Tableau 1 : Recette du mélange maître tampon de lyse.
REMARQUE: Le tampon de lyse peut être modifié pour contenir plus d’inhibiteurs de la protéase (tels que la bestatine, la leupétine, l’aprotinine, etc.) au cas où la protéine d’intérêt serait très instable, mais il est important d’éviter l’EDTA afin de maintenir les petites et grandes sous-unités du ribosome assemblées au cours des étapes suivantes. Pour des raisons similaires, maintenez toujours au moins 6 mM de MgCl2 dans la solution tampon.
ATTENTION : Le HCl est très corrosif et le PMSF est toxique. Portez des gants et manipulez-les avec soin.
- Centrifuger pendant 2 min à 30 000 x g, 4 °C pour éliminer le lysat et recueillir le surnageant.
4. Purification des complexes ribosomes-chaînes naissantes pour SeRP
- Pour chaque expérience, divisez le surnageant en deux parties; chacun dans un tube de microcentrifugation différent : échantillon d’ARN total (~200 μL) et échantillon d’immunopurification (IP) (~700 μL) échantillons de translatome.
-
Traitement de l’échantillon d’ARN total
- Digérer l’échantillon d’ARN total à l’aide de 10 U de RNase I pendant 25 min à 4 °C; tourner à 30 tr/min avec un rack de mélange rotatif.
REMARQUE: Les conditions de digestion peuvent être calibrées à l’aide du profilage des polysomes pour assurer qu’il n’y a pas de pic de sur- ou de sous-digestion des monosomes. - Préparer le mélange principal de coussin de saccharose tel que décrit dans le tableau 2.
- Digérer l’échantillon d’ARN total à l’aide de 10 U de RNase I pendant 25 min à 4 °C; tourner à 30 tr/min avec un rack de mélange rotatif.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 50% de saccharose | 200 | 25% |
| 1M Tris-HCl pH 8,0 | 8 | 20 mM |
| 3M KCl | 18.7 | 140 mM |
| 1M MgCl2 | 4 | 10 mM |
| 100 mg/mL CHX | 0.4 | 0,1 mg/mL |
| Inhibiteur de la protéase | 1 comprimé | |
| Volume final | 400 |
Tableau 2 : Recette du mélange maître de coussin de saccharose.
- Charger l’échantillon sur 400 μL du coussin de saccharose et centrifuger dans un rotor TLA120 pendant 90 min à 245 000 x g et à 4 °C.
- Retirez rapidement le surnageant à l’aide d’une pompe à vide et superposez les pastilles avec un tampon de lyse de 150 μL. Remettre en suspension les granulés en agitant pendant 1 h à 4 °C et à 300 tr/min.
- Remettre en suspension la pastille résiduelle par pipetage et transférer dans un nouveau tube de 1,5 mL.
REMARQUE: 100-200 μg d’ARN total est généralement suffisant pour le profilage des ribosomes du translatome total. On peut ajouter une étape d’épuisement de l’ARNr afin de réduire la contamination par l’ARNr, qui est le contaminant le plus répandu des complexes de chaînes ribosomes-naissantes purifications d’affinité11 (voir la discussion pour plus de détails).
-
Traitement de l’échantillon d’immunopurification
- Laver 100-400 μL de la matrice de liaison d’affinité (billes conjuguées aux anticorps 1:1 dans 70% EtOH) par échantillon avec 3 x 1 mL de tampon de lyse (sans DNase I et inhibiteurs de la protéase); remettez en suspension la matrice d’affinité dans le tampon de lyse, puis faites pivoter à 30 tr/min avec un rack de mélange rotatif à 4 °C pendant 5 min. Précipiter par centrifugation pendant 30 s à 3 000 x g, 4 °C. Jetez le liquide supérieur. Répétez trois fois.
- Digérer des échantillons d’immunopurification en utilisant 10 U par unité A260 nm de RNase I, ainsi qu’une matrice de liaison d’affinité (par exemple, 100-400 μL de GFP-TRAP par échantillon).
- Faire pivoter pendant 25 min à 30 tr/min avec un rack de mélange rotatif pour lier la protéine à la matrice d’affinité, à 4 °C.
- Préparez le mélange maître tampon de lavage comme indiqué dans le tableau 3.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 10 mg/mL CHX | 50 | 0,1 mg/mL |
| 1M Tris-HCl pH 8,0 | 100 | 20 mM |
| 3M KCl | 233 | 140 mM |
| 1M MgCl2 | 50 | 10 mM |
| 1M PMSF | 5 | 1 mM |
| NP-40 | 0.5 | 0.01% |
| Inhibiteur de la protéase | 2 comprimés | |
| 50% Glycérol | 1,000 | 10% |
| Volume final | 5,000 |
Tableau 3 : Recette du mélange maître tampon de lavage.
- Lavez la matrice de liaison d’affinité trois fois avec 1 mL de tampon de lavage, à chaque fois pendant environ 1 min, en tournant dans la grille de mélange à 30 tr / min, à 4 ° C.
- Précipiter par centrifugation à 3 000 x g pendant 30 s à 4 °C. Jetez le liquide supérieur.
- Lavez deux fois de plus dans un tampon de lavage de 1 mL, à chaque fois pendant 5 min, en tournant dans le rack de mélange à 30 tr /min, à 4 °C.
- Précipiter par centrifugation pendant 30 s à 3 000 x g et 4 °C.
- Utilisez 50 μL de billes pour l’élution des protéines avec la même quantité de tampon d’échantillon 2x. Utilisez le reste des perles pour l’extraction de l’ARN.
- Centrifuger pendant 30 s à 3 000 x g, 4 °C pour granuler les billes et jeter le liquide supérieur.
- Congeler dans de l’azote liquide et conserver à -80 °C. Utilisez ces échantillons pour l’extraction ultérieure de l’ARN.
POINT D’ARRÊT : Les échantillons peuvent être conservés à -80 °C pendant la nuit ou plus longtemps. Cela peut être un point d’arrêt. - Évaluez le succès de l’étape de purification d’affinité par western blot ou coloration de Coomassie avec des aliquotes (~10% en volume, après mélange) de chaque étape. Utilisez toujours une adresse IP fictive sur une souche WT non marquée comme contrôle de la liaison non spécifique à la matrice d’affinité.
REMARQUE: Un fond non spécifique élevé peut être surmonté par des étapes de lavage supplémentaires avec des concentrations croissantes de sel / détergent. L’interaction transitoire peut être stabilisée par divers agents de réticulation, par exemple, le traitement par paraformaldéhyde (PFA) des cellules vivantes - l’ajout de 0,4% à 1% de PFA au milieu de croissance pendant 2 à 5 minutes, suivi d’une trempe à la glycine (0,3 M) pendant 3 minutes, est fortement recommandé.
ATTENTION : Le paraformaldéhyde est un cancérogène présumé. Étant donné que le paraformaldéhyde s’évapore rapidement et est corrosif, travaillez dans une hotte de sécurité chimique et portez deux couches de gants.
5. Préparation de la bibliothèque d’ADNc pour le séquençage en profondeur
- Extraction de l’ARN
REMARQUE: Travailler avec des tubes antiadhésifs de 1,5 mL sans RNase pour prévenir un éventuel épuisement de l’ARN ou de l’ADN.- Décongeler les échantillons des étapes 4.2.5 et 4.3.12 sur de la glace et remettre en suspension des échantillons avec 10 mM Tris-HCl pH 7,0 jusqu’à un volume final de 700 μL.
ATTENTION : L’acide-phénol et le chloroforme sont volatils et nocifs. Travailler dans une hotte de sécurité chimique. - Ajouter 40 μL de FDS à 20 % à 0,7 mL d’ARN total ou d’élutions IP. Fermez et inversez plusieurs fois. La précipitation des protéines devrait rendre les échantillons blancs.
- Ajouter 0,75 mL d’acide-phénol:chloroforme préchauffé aux échantillons. Scellez hermétiquement les tubes et secouez-les dans un mélangeur thermique à 1 400 tr/min pendant 5 min et à 65 °C. Refroidir les échantillons sur la glace pendant 5 min.
- Centrifuger le tube à partir de l’étape 5.1.3. à 20 000 x g pendant 2 min. Transférer la couche aqueuse supérieure dans un tube frais et y ajouter 0,7 mL d’acide-phénol:chloroforme.
- Incuber pendant 5 min à température ambiante, parfois en vortexant. Centrifuger pendant 2 min à 20 000 x g. Transférer la couche aqueuse supérieure dans un tube frais et y ajouter 0,6 mL de chloroforme et de vortex.
- Centrifuger pendant 1 min à 20 000 x g. Transférer la couche aqueuse supérieure dans un tube frais.
- Précipiter les acides nucléiques en ajoutant 78 μL de 3 M NaOAc, pH 5,5, 2 μL de GlycoBlue et 0,75 mL d’isopropanol. Vortex à fond pendant 5 min. Incuber pendant au moins 1 h à -80 °C ou 16 h à -20 °C.
- Centrifuger pendant 30 min à 20 000 x g et à 4 °C et jeter le surnageant. Lavez les granulés avec 0,75 mL d’éthanol glacé à 80 %. Inverser les tubes pour un lavage en profondeur. Centrifuger à 20 000 x g pendant 5 min à 4 °C, puis jeter le surnageant.
- Tourner à 450 x g, 4 °C pendant 20 s et retirer l’éthanol restant et jeter les liquides. Sécher la pastille avec un couvercle ouvert pendant 5 min à 65 °C. Remettre en suspension les échantillons comme suit: pour IP, remettre l’échantillon en suspension dans 10 μL de 10 mM Tris-HCl, pH 7,0. Pour l’analyse du translatome total, remettre l’échantillon en suspension dans 20 μL de 10 mM Tris-HCl, pH 7,0.
POINT D’ARRÊT : L’ARN peut être stocké à -80 °C pendant des mois.
- Décongeler les échantillons des étapes 4.2.5 et 4.3.12 sur de la glace et remettre en suspension des échantillons avec 10 mM Tris-HCl pH 7,0 jusqu’à un volume final de 700 μL.
- Quantifier la concentration d’ARN total par fluorométrie
REMARQUE : Tous les matériaux et surfaces suivants doivent être exempts de RNase lors de la préparation de la bibliothèque d’ADNc pour le séquençage de nouvelle génération. Lorsque vous manipulez des échantillons d’ARN, portez des gants.- Diluer 1 μL d’ARN total extrait du phénol acide dans 9 μL de 10 mM de Tris-HCl, pH 7,0. Quantifier à l’aide d’un fluoromètre, comme indiqué sur le site Web du fabricant.
- Diluer les échantillons contenant 50 μg d’ARN avec 10 μL de 10 mM de Tris-HCl, pH 7,0.
REMARQUE: Ne mesurez pas les échantillons IP, utilisez tout pour l’étape suivante.
- Fragments d’empreinte protégés par le ribosome purifiant le gel
- Réglez un gel de polyacrylamide TBE-urée à 15% et immergez-le dans 1x tampon de fonctionnement TBE. Exécuter pendant 30 min à 200 V avant le chargement de l’échantillon. À chaque échantillon, ajoutez 20 μL de 2x tampon d’échantillon d’urée TBE.
REMARQUE: La taille de bande prévue est d’environ 25-35 nt. - Décongeler une échelle d’ADN de 10 pb et dénaturer les échantillons (pas l’échelle) à 80 °C pendant 2 min, puis refroidir sur la glace. Chargez chaque échantillon sur une autre voie. Faites fonctionner le gel pendant 50-70 min à 200 V.
- Diluer 6 μL d’or SYBR (10 000 x concentré) dans 60 mL de tampon 1x TBE et tacher tout en agitant dans des boîtes à l’abri de la lumière pendant 15 à 20 min. Pendant la coloration du gel, préparez un scalpel stérile et des tubes brise-gel de 0,5 mL dans des tubes étiquetés de 1,5 mL.
- Retirez les bandes souhaitées avec un scalpel stérile (utilisez-en un frais ou nettoyez bien entre les échantillons) et placez chaque morceau de gel dans un tube brise-gel.
- Prenez une image du gel pour vous assurer qu’aucun résidu d’échantillon n’est laissé dans le gel.
- Centrifuger les tubes contenant des tranches coupées à 20 000 x g pendant 5 min à 4 °C et transférer les morceaux de gel restants du tube brise-gel vers le tube de 1,5 mL.
- Ajouter 0,5 mL de Tris 10 mM, pH 7,0. Agiter dans un mélangeur thermique à 1 400 tr/min pendant 10 min à 70 °C.
- Transférer dans une colonne d’acétate de cellulose avec une pointe de pipette à large alésage et centrifuger à 20 000 x g pendant 3 min à 4 °C.
- Transférer l’écoulement dans un nouveau tube de 1,5 mL et refroidir sur la glace.
- Afin de précipiter les acides nucléiques, ajoutez: 550 μL d’IPA, 55 μL de 3 M NaOAc et 2 μL de GlycoBlue et de vortex à bien mélanger. Placer les échantillons à -80 °C pendant au moins 1 h.
- Centrifuger pendant au moins 1 h à 20 000 x g et 4 °C et jeter le surnageant. Lavez les granulés avec 0,75 mL d’éthanol glacé à 80 %. Inverser les tubes pour un lavage en profondeur jusqu’à ce que les granulés se séparent du fond. Centrifuger à nouveau à 20 000 x g pendant 5 min à 4 °C et jeter le surnageant.
- Tourner à 450 x g, 4 °C pendant 20 s et retirer l’éthanol restant. Sécher les granulés avec un couvercle ouvert pendant 5 min à 65 °C.
- Ajouter 15 μL de Tris de 10 mM, pH 7,0 et remettre soigneusement les granulés en suspension. Tourner vers le bas à 450 x g, 4 °C pendant 20 s et transférer l’échantillon dans un nouveau tube de 1,5 mL.
POINT D’ARRÊT: L’ARN purifié peut être stocké à -80 ° C pendant quelques mois.
- Réglez un gel de polyacrylamide TBE-urée à 15% et immergez-le dans 1x tampon de fonctionnement TBE. Exécuter pendant 30 min à 200 V avant le chargement de l’échantillon. À chaque échantillon, ajoutez 20 μL de 2x tampon d’échantillon d’urée TBE.
- Déphosphorylation
- Utilisez 3 μL du mélange suivant pour chaque échantillon : Ajouter 1 μL d’inhibiteur de la RNase dans 2 μL de tampon de réaction de polynucléotide kinase T4 sans ATP. Ajouter 2 μL de polynucléotide kinase T4 à chaque échantillon. Pipetter doucement pour bien mélanger et incuber à 37 °C pendant 2 h, sans secouer.
- Pour inactiver l’enzyme, incuber l’échantillon à 75 °C pendant 10 min et tourner vers le bas à 450 x g, 4 °C, pendant 20 s. Ajouter 0,5 mL de 10 mM Tris, pH 7,0.
- Pour précipiter l’acide nucléique, ajoutez 2 μL de GlycoBlue, 550 μL d’IPA et 55 μL de 3 M NaOAc.
- Vortex pour bien mélanger et refroidir les échantillons à -80 °C pendant au moins 1 h.
POINT D’ARRÊT : Les échantillons peuvent être conservés à -80 °C pendant la nuit ou plus longtemps. - Répétez les étapes 5.3.11 à 5.3.13.
POINT D’ARRÊT: L’ARN déphosphorylé peut être stocké à -80 ° C pendant des mois.
- Quantification à l’aide d’un bioanalyseur
- Effectuer une dilution 1:4 de chaque échantillon d’ARN en mélangeant 1 μL d’échantillon et 4 μL d’eau traitée au DEPC.
ATTENTION : Le DEPC est cancérigène. Portez des gants et travaillez avec précaution. - Exécutez un bioanalyseur Small RNA Chip/TapeStation. Suivez le protocole du fabricant.
REMARQUE: La taille attendue des fragments d’ARN protégés par les ribosomes est d’environ 28-30 nt.
- Effectuer une dilution 1:4 de chaque échantillon d’ARN en mélangeant 1 μL d’échantillon et 4 μL d’eau traitée au DEPC.
- Ligate 3' avec Linker-1
- Diluer 5 pmol de petits fragments d’ARN à 10 μL avec 10 mM de Tris, pH 7,0. Dénaturer les échantillons à 80 °C pendant 2 min et refroidir sur la glace.
- Préparer le mélange principal comme indiqué dans le tableau 4 et utiliser 29 μL par échantillon.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 50% de PEG 8000 filtré stérile | 16 | 20% |
| DMSO | 4 | 10% |
| Tampon 10× ARN ligase 2 T4 | 4 | 1x |
| Inhibiteur de la RNase SUPERase-In | 2 | 2 U |
| Linker adénylé 10 mM 3-L1 | 0.1 | 25 μM |
| Eau traitée au DEPC | 2.9 | |
| Volume final | 29 |
Tableau 4 : Recette du mélange maître de ligature finale de 3'.
- Ajouter 1 μL d’ARN T4 ligase 2 et pipeter doucement pour bien mélanger. Incuber à 23 °C pendant 2 h.
- Afin de précipiter les acides nucléiques, ajoutez: 550 μL d’IPA, 500 μL de Tris de 10 mM, pH 7,0, 55 μL de 3 M NaOAc et 2 μL de GlycoBlue. Vortex pour bien mélanger et placer les échantillons à -80 °C pendant au moins 1 h.
POINT D’ARRÊT : Conservez les échantillons à -80 °C pendant la nuit ou plus longtemps. - Répétez les étapes 5.3.2 à 5.3.12.
- Remettre en suspension la pastille dans 6 μL de 10 mM Tris, pH 7,0. Tourner vers le bas à 450 x g, 4 °C pendant 20 s, et transférer l’échantillon dans un nouveau tube de 1,5 mL.
POINT D’ARRÊT: Les échantillons peuvent être conservés à -80 °C pendant des mois.
-
Purification en gel des empreintes liées à 3'
- Réglez un gel de polyacrylamide TBE-urée à 10% et immergez-le dans 1x tampon de fonctionnement TBE. Exécuter pendant 30 min à 200 V avant le chargement de l’échantillon. À chaque échantillon, ajoutez 6 μL de 2x tampon d’échantillon d’urée TBE.
REMARQUE: La taille de bande prévue est d’environ 71-73 nt. - Répétez les étapes 5.3.2 à 5.3.13.
POINT D’ARRÊT: Les échantillons peuvent être conservés à -80 °C pendant des mois.
- Réglez un gel de polyacrylamide TBE-urée à 10% et immergez-le dans 1x tampon de fonctionnement TBE. Exécuter pendant 30 min à 200 V avant le chargement de l’échantillon. À chaque échantillon, ajoutez 6 μL de 2x tampon d’échantillon d’urée TBE.
-
Transcrire à l’envers 3ʹ fragments d’empreinte liés pour générer de l’ADNSs
- Préparer un mélange principal comme indiqué dans le tableau 5 et utiliser 3 μL par échantillon.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 10 mM dNTPs | 1 | 0,5 mM |
| Linker L(rt) de 25 μM | 0.5 | 625 nM |
| Eau traitée au DEPC | 1.5 | |
| Volume final | 3 |
Tableau 5 : Recette du mélange maître tampon de transcription inverse avant la dénaturation des acides nucléiques.
- Vortex et rotation de l’échantillon.
- Incuber des échantillons à 65 °C pendant 5 min.
- Refroidir les échantillons sur la glace.
- Préparer un mélange maître comme indiqué dans le tableau 6 et utiliser 6 μL par échantillon. Vortex et rotation de l’échantillon. Ajouter 1 μL d’exposant III à chaque échantillon et pipeter doucement pour bien mélanger et incuber pendant 30 min à 50 °C.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 5× Tampon FS | 4 | 1x |
| Inhibiteur de la RNase SUPERase-In | 1 | 2 U |
| TNT 0,1 M | 1 | 5 mM |
| Volume final | 6 |
Tableau 6 : Recette du mélange maître tampon de transcription inverse après dénaturation des acides nucléiques.
- Ajouter 2,3 μL de 1 N NaOH, ce qui hydrolyse l’ARN et éteint la transcription inverse.
ATTENTION : Le NaOH est très corrosif. Portez des gants et une protection oculaire. - Incuber pendant 15 min à 95 °C, jusqu’à ce que l’échantillon devienne rose.
- Réglez un gel de polyacrylamide TBE-urée à 10% et immergez-le dans 1x tampon de fonctionnement TBE. Exécuter pendant 30 min à 200 V avant le chargement de l’échantillon. À chaque échantillon, ajoutez 23 μL de 2x tampon d’échantillon d’urée TBE.
REMARQUE: La taille prévue de la bande d’ADN est de 115-117 nt. - Répétez les étapes 5.3.2 à 5.3.12.
- Remettre en suspension la pastille dans 15 μL de 10 mM Tris, pH 8,0. Tourner vers le bas à 450 x g, 4 °C pendant 20 s et transférer l’échantillon dans un nouveau tube de 1,5 mL.
POINT D’ARRÊT: Les échantillons peuvent être conservés à -80 °C pendant des mois.
-
Circularisation de l’ADNs ssDNA
- Préparer le mélange maître suivant et charger 4 μL par échantillon, comme indiqué dans le tableau 7.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| 10× Tampon CircLigase II | 2 | 1x |
| 5 M Bétaïne (facultatif) | 1 | 0,25 m |
| 50 mM MnCl2 | 1 | 2,5 mM |
| Volume final | 4 |
Tableau 7 : Recette du mélange maître de circularisation de l’ADNSS .
- Ajouter 1 μL de circLigase II ssDNA ligase à chaque échantillon et incuber pendant 1 h à 60 °C.
REMARQUE: L’efficacité de cette étape peut être augmentée en ajoutant 1 μL de CircLigase II ssDNA ligase à chaque échantillon après 1 h d’incubation. - Inactiver l’enzyme en incubant à 80 °C pendant 10 min.
- Refroidir sur la glace et continuer à l’amplification PCR ou stocker à -80 °C.
POINT D’ARRÊT: Les échantillons peuvent être conservés à -80 ° C pendant des années.
-
Amplification PCR
- Préparez le mélange maître PCR suivant et chargez 82 μL par échantillon, comme indiqué dans le tableau 8.
| Réactif | Quantité par échantillon (μL) | Concentration finale |
| Eau traitée au DEPC | 61.6 | |
| 5× Tampon de réaction PHusion HF | 17.6 | 1x |
| 10 mM dNTPs | 1.8 | 200 μM |
| Amorce avant PCR 100 μM | 0.2 | 225 nM |
| HF Phusion polymérase | 0.8 | 1,6 U |
| Volume final | 82 |
Tableau 8 : Recette du mélange maître d’amplification PCR.
- À chaque tube contenant le mélange maître, ajouter 5 μL d’ADN circularisé.
REMARQUE: Conservez le reste des échantillons d’ADN circularisés à -80 ° C. - Ajouter un autre 1 μL de 20 μM d’amorce de code-barres inverse PCR à chaque échantillon (voir le tableau 9) et un vortex pour bien mélanger.
- Aliquotez chaque tube dans quatre tubes PCR distincts, chacun sera utilisé pour un nombre différent de cycles PCR.
- Exécutez une réaction PCR selon le programme suivant, comme détaillé dans le tableau 10.
| Cycle | Dénature (98 °C) | Recuit (60 °C) | Étendre (72 °C) |
| 1 | 30 s | ||
| 2-16 | 10 s | 10 s | 5 s |
Tableau 10 : Programme de PCR pour la réaction par PCR.
- Après les cycles 8, 9, 10 et 11, retirez les tubes PCR (pour les échantillons IP, les cycles vont de 9 à 15) comme première tentative. Après chaque cycle, mettez le programme en pause, sortez une aliquote et mettez-la sur la glace, puis reprenez rapidement le programme.
REMARQUE: Le nombre de cycles doit être ajusté en fonction de la quantité d’ADN circularisé dans chaque réaction. Reportez-vous à la figure 4 pour un exemple et des éclaircissements supplémentaires. - À chaque réaction de 17 μL, ajoutez 3,5 μL de colorant à charge d’ADN 6x.
- Décongeler une échelle d’ADN de 10 pb.
- Pour la séparation des tailles par électrophorèse sur gel, immergez 8% de polyacrylamide TBE dans 1x tampon de fonctionnement TBE et chargez les échantillons de chaque numéro de cycle différent dans des puits adjacents et faites couler le gel pendant 50 min à 180 V.
- Diluer 6 μL d’or SYBR (10 000 x concentré) dans 60 mL de tampon 1x TBE et tacher tout en agitant dans des boîtes à l’abri de la lumière pendant 15 à 20 min.
- Pendant la coloration du gel, préparez un scalpel stérile et des tubes brise-gel de 0,5 mL dans des tubes étiquetés de 1,5 mL.
- Prenez une image des acides nucléiques colorés.
- Coupez la bande souhaitée avec une taille de bande prévue de 174-176 bp avec le scalpel stérile et placez la tranche de gel dans le tube de gel de 0,5 mL préparé (nettoyez soigneusement entre les échantillons et utilisez un agent inactivant RNase, ou passez à une nouvelle lame).
- Centrifugez les tubes pendant 5 min à 20 000 x g et 4 °C, puis transférez les morceaux de gel restants du tube de 0,5 mL de brise-gel vers le tube de 1,5 mL.
- Ajouter 500 μL de Tris de 10 mM, pH 8,0 et agiter dans un mélangeur thermique à 1 400 tr/min pendant 10 min et 70 °C.
- Transférer le gel dissous dans une colonne d’acétate de cellulose avec une pointe de pipette à large alésage.
- Centrifuger la colonne pendant 3 min à 20 000 x g et 4 °C et transférer l’écoulement dans un nouveau tube de 1,5 mL et refroidir sur glace.
- Pour précipiter l’acide nucléique, ajoutez : 550 μL d’IPA, 32 μL de 5 M NaCl, 1 μL de 0,5 M EDTA et 2 μL de GlycoBlue et de vortex à bien mélanger.
- Conservez les échantillons pendant au moins 1 h à -80 °C ou -20 °C pendant la nuit.
POINT D’ARRÊT : Les échantillons peuvent être conservés à -80 °C pendant la nuit ou plus longtemps. - Répétez les étapes 5.3.11 à 5.3.12.
- Remettre en suspension dans 11 μL de 10 mM Tris, pH 8,0. Tourner vers le bas à 450 x g, 4 °C pendant 20 s et transférer l’échantillon dans un nouveau tube de 1,5 mL.
POINT D’ARRÊT: Les échantillons peuvent être conservés à -80 ° C pendant des années.
-
Quantifier la distribution de taille par bioanalyseur
- Effectuer une dilution 1:4 de chaque échantillon en mélangeant 1 μL d’échantillon avec 4 μL d’eau traitée au DEPC.
- Exécutez la puce à petit ARN Bioanalyzer. Suivez le protocole du fabricant.
REMARQUE: La longueur prévue est de 175 ± 5 pb.
-
Quantifier la concentration d’ADN par fluoromètre
- Effectuez un contrôle de concentration haute sensibilité dsDNA avec un fluoromètre conformément aux recommandations du fabricant.
- Échantillons multiplex et séquentiels selon les recommandations d’Illumina (Guide de regroupement des adaptateurs d’index12).
6. Analyse des données
- Effectuer l’analyse détaillée dans le fichier supplémentaire.
Résultats
Comme l’illustre l’organigramme de ce protocole (Figure 1), les cellules ont été cultivées en phase logarithmique, puis collectées rapidement par filtration et lysées par broyage cryogénique. Le lysat a ensuite été divisé en deux: l’un pour les empreintes totales d’ARNm protégées par les ribosomes et l’autre pour les empreintes d’ARNm protégées par les ribosomes sélectionnées, sur lesquelles nous avons effectué une purification d’affinité pour abaisser les complexes de chaînes protéine-ribosome-naissantes marqués. Nous avons assuré l’expression des protéines marquées et le succès de l’analyse par transfert western, comme on peut le voir à la figure 2. Nous avons validé l’isolement des empreintes protégées par les ribosomes, qui mesurent généralement de 20 à 45 nt de long par électrophorèse à petit ARN (système 2100 BioAnalyzer), permettant un décalage de taille de 5 à 10 nt, selon le manuel du système (Figure 3). Ensuite, nous avons généré une bibliothèque d’ADNc pour le séquençage en profondeur et l’analyse de données volumineuses. Lors de la génération de la bibliothèque d’ADNc, notez que le sous-cycle peut entraîner un faible rendement (comme on peut le voir dans la voie 2 de la figure 3), mais une réamplification est possible afin de récupérer la bibliothèque générée. Un recyclage excessif peut se produire lorsque les amorces de PCR sont épuisées, mais que la réaction se poursuit. Lorsque les dNTP sont encore présents, la réaction se poursuit, générant des artefacts PCR plus longs avec des séquences chimériques dues aux produits PCR qui s’amorcenteux-mêmes 13 (comme on peut le voir dans les voies 3-4 de la figure 3, indiquée par le frottis visible). Si la concentration des dNTP devient également limitative, des produits indiquant la présence d’hétéroduplexes composés uniquement de fragments de bibliothèque partiellement homologues peuvent apparaître. La figure 4 sert de référence, la voie 2 représentant une amplification optimale et la voie 3 une amplification acceptable. Les échantillons des voies 4 et 5 (cycles 10 et 11) ne devraient pas être utilisés en raison de la possibilité d’introduire des doublons et des artefacts de PCR. La bibliothèque générée a ensuite été validée par électrophorèse de l’ADN à haute sensibilité (le même système de bioanalyseur a été utilisé) pour la distribution et la quantification exactes de la taille (figure 5). Après une ligature de liaison d’extrémité de 3', une transcription inverse et une amplification par PCR, une distribution de longueur d’ADNc en tant que telle est attendue, avec un pic aigu d’environ 175 nt.
Nous avons découpé et retiré les adaptateurs et les codes-barres de la bibliothèque séquencée, et seules les lectures comprises entre 20 et 45 nt ont été sélectionnées pour une analyse plus approfondie. La figure 6 montre la distribution de longueur résultante. Les lectures ont été divisées en différents groupes de : séquences codantes, introns et séquences intergéniques (figure 7), et classées comme illustré à la figure 8.
L’analyse finale pour la détection et la caractérisation des interactions co-translationnelles a été réalisée sur la base de l’enrichissement de fragments d’ARNm protégés par les ribosomes, produisant les graphiques de la figure 9. Nous avons comparé l’occupation normalisée des ribosomes (à chaque nucléotide le long de chaque orf) du translatome total à son translatome sélectionné correspondant (naissant-interactome). La comparaison par nucléotide élimine les artefacts des taux de traduction. La reproductibilité entre les réplicats biologiques a été évaluée par corrélation de Pearson (seuil > 0,6). Nous présentons des profils ribosomes sélectifs, analysant les interactions co-translationnelles de Vma2p avec trois protéines: le chaperon associé aux ribosomes Ssb1p, Pfk2p (Phosphofructokinase) et Fas1p (acide gras synthase), chaque protéine C’étant marquée en phase terminale par GFP. Nous avons effectué le protocole dans des répliques biologiques. Graphique 9 A, D et G montrent le schéma expérimental de chaque purification d’affinité. Nous montrons ensuite l’occupation des ribosomes des translatomes totaux par rapport aux interactomes Ssb1 le long de l’orf Vma2p, codant pour une sous-unité de la H+-ATPase vacuolaire (Figure 9 B, E et H). Enfin, nous avons effectué un profilage de l’enrichissement des ribosomes basé sur le ratio (IP/Total) à chaque position du ribosome dans [codon/aa] le long de l’orf (Figure 9 C, F et I). La comparaison des interactions co-translationnelles de ces trois protéines avec Vma2p, qui est synthétisé par le ribosome, a révélé que le chaperon Ssb1 engage le Vma2p naissant dans quatre régions différentes le long de l’orf, car nous avons identifié quatre pics d’enrichissement significatifs par SeRP. Différemment, Pfk2p ne montre qu’un seul pic d’enrichissement significatif, tel qu’identifié par SeRP, dans une position différente de celle du chaperon co-translationnel Ssb1. L’analyse des interactions co-translationnelles de Fas1 avec Vma2p naissant n’a détecté aucun enrichissement significatif. Ainsi, la comparaison de ces profils de ribosomes d’enrichissement basés sur les ratios démontre la puissance de ce protocole dans la détection et la caractérisation de diverses interactions co-traductionnelles en résolution proche du codon.
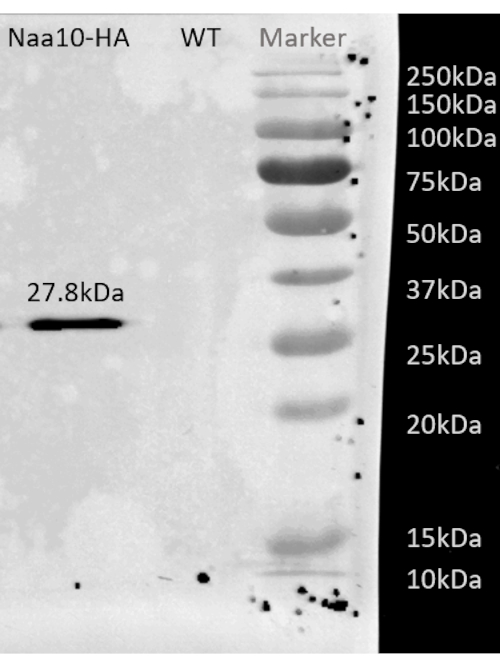
Figure 2 : Résultat représentatif du transfert western après purification par affinité de la souche BY4741 avec Naa10 marqué HA. Résultat de western blot représentatif après purification d’affinité de la souche BY4741 avec Naa10 marqué HA montrant une bande autour de 27,8 kDa, tandis que le type sauvage, en tant que contrôle négatif, ne montre aucune bande. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Résultat représentatif du bioanalyseur après isolement de l’empreinte et extraction de l’ARN avec acide-phénol:chloroforme, et une taille moyenne de 25 nt. Veuillez cliquer ici pour voir une version plus grande de cette figure.
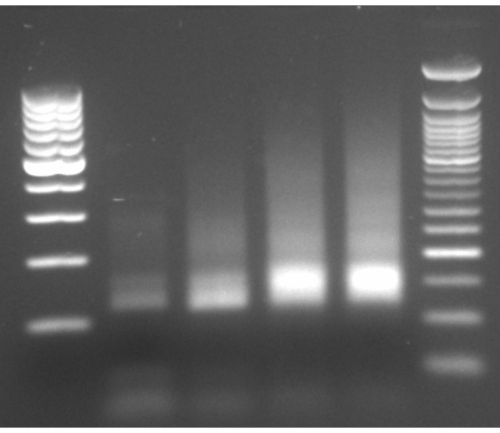
Figure 4 : Électrophorèse sur gel représentative de l’amplification par PCR. Électrophorèse sur gel représentative de l’amplification par PCR avec des voies 2 à 5 chargées de produits PCR des cycles 8 à 11 et des échelles des deux côtés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
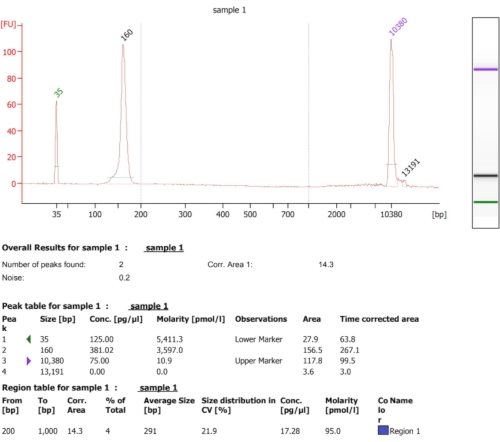
Figure 5 : Résultat représentatif du bioanalyseur obtenu à la suite de la création d’une bibliothèque d’ADNc. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 6 : Distribution attendue de la longueur des lectures après le retrait des adaptateurs avec Cutadapt (retrait des lectures inférieures à 20 ou supérieures à 45). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Pourcentage de réussite de l’alignement attendu après avoir retiré les lectures d’ARN non codantes avec Bowtie2 et utilisé TopHat pour aligner les lectures restantes sur différents organismes. L’échantillon a été prélevé sur S. cerevisiae (une variante mutée de BY4741). Veuillez cliquer ici pour voir une version agrandie de cette figure.
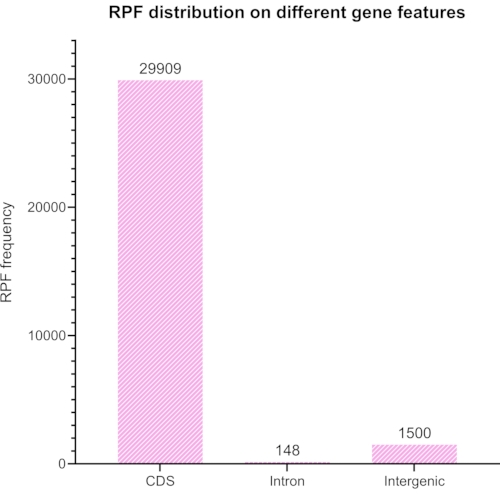
Figure 8 : Un graphique généré avec RiboToolkit représentant le rapport prévu entre le codage et le rapport non codant des lectures alignées après avoir utilisé Bowtie2 pour supprimer les éléments d’ARNr dans les lectures. Veuillez cliquer ici pour voir une version agrandie de cette figure.
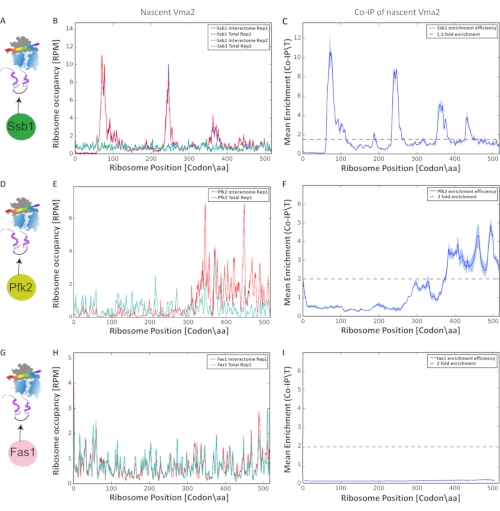
Figure 9 : Interactions co-translationnelles de trois protéines différentes : Ssb1p, Pfk2p et Fas1p avec Vma2p, qui est synthétisé par le ribosome, analysé par SeRP. Tous les axes y sont affichés en lectures par million de lectures (RPM). (A, D, G) Schéma expérimental de SeRP de Ssb1p, Pfk2p et Fas1p C’marqués en terminal par GFP, respectivement. (B, E, H) Occupation des ribosomes le long de l’orf des translatomes totaux par rapport aux interactomes Ssb1, Pfk2p et Fas1p, respectivement (dans les répliques biologiques). (C, F, I) Enrichissement moyen de Ssb1p, Pfk2p et Fas1p (rapport IP/Total) à chaque position de ribosome dans [codon/aa] le long de l’orf, respectivement. La variation entre les répliques biologiques est indiquée par la zone ombragée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 9 : Séquences d’agent de liaison et d’amorce 3'. Linker L1 de 3' : Linker 3-L1 avec adénylation de 5ʹ et identificateurs moléculaires uniques de 3ʹ didéoxy-cytidine (« NN... ») (Purification HPLC sans RNase; Inverseur de transcription : transcription inverse (L(rt)) avec 5ʹ phosphorylés, identificateurs moléculaires uniques (purification HPLC sans RNase) ; Amorce avant PCR: PCRf; HPLC purifié. Veuillez cliquer ici pour télécharger ce tableau.
Dossier supplémentaire. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ici, le protocole détaille l’approche de profilage sélectif des ribosomes pour capturer les interactions co-traductionnelles dans une résolution proche du codon. Au fur et à mesure que le ribosome devient une plaque tournante pour coordonner l’émergence de la chaîne naissante dans le cytoplasme surpeuplé, il s’agit d’une méthode cruciale pour identifier et caractériser les diverses interactions co-translationnelles nécessaires pour assurer un protéome fonctionnel, ainsi que pour étudier diverses maladies. À ce jour, seRP est la seule méthode qui permet de capturer et de caractériser ces interactions, de manière directe, in vivo 14,15,16.
La première et la plus critique étape est la collecte et la lyse des cellules. Il est impératif de capturer, en quelques secondes, la traduction en cours, par congélation éclair suivie d’une lyse à l’état figé. La collecte des cellules doit être effectuée à la hâte afin d’éviter le ruissellement ribosomique ainsi que d’induire des réponses translationnelles de stress, qui peuvent se produire rapidement. La deuxième étape critique est l’étape de purification par affinité. Il est impératif de réduire la liaison de fond par un lavage rigoureux tout en s’assurant que les interactions co-traductionnelles sont maintenues, ce qui peut être facilité par la réticulation in vivo . Comme ce protocole est basé sur le NGS (Next Generation Sequencing) très sensible, l’arrière-plan élevé dans les premières étapes peut être amplifié dans les étapes suivantes de préparation de la bibliothèque d’ADNc, ce qui conduit à de faibles rapports signal/bruit.
Le traitement par nucléases, pour digérer tous les ARNm non protégés, doit être évalué par profilage polysome17 ainsi que par une évaluation minutieuse de la distribution granulométrique des empreintes ribosomiques isolées (comme détaillé ci-dessus) afin d’éviter une digestion excessive ou insuffisante de l’ARN. L’étalonnage de la concentration en nucléases et des temps de digestion peut faciliter une récupération précise de l’empreinte, car une digestion excessive peut entraîner une digestion de l’ARNr ribosomique, entraînant la perte d’empreintes protégées par les ribosomes. Il est important de noter que la sous-digestion peut également entraîner des taux de découverte plus faibles d’empreintes protégées par les ribosomes, car les étapes de préparation de la bibliothèque d’ADNc, ainsi que les étapes d’analyse des données décrites ici éliminent les lectures longues et inhabituelles.
Bien que l’épuisement de l’ARNr ne constitue pas toujours une étape critique et n’est pas obligatoire, il présente certains avantages tels que des échantillons plus propres et, par conséquent, un taux plus élevé de lectures cartographiées par le génome. D’autre part, il existe la possibilité de biais, car de nombreux protocoles d’épuisement de l’ARNr pourraient également entraîner l’épuisement des fragments protégés par les ribosomes souhaités. Il faut également prendre en considération les coûts des kits d’épuisement de l’ARNr. L’épuisement de l’ARNr peut être effectué après l’étape d’isolement de l’empreinte ribosome-pied ou après l’étape de circularisation de l’ADNc.
Les étapes de préparation de la bibliothèque d’ADNc décrites ici ont été optimisées pour une faible entrée d’ARNm, car les étapes d’affinité et de purification des ribosomes réduisent considérablement la quantité d’entrée d’ARNm, par rapport aux études d’expression de l’ARN-seq. La mise à l’échelle de la quantité initiale de cultures cellulaires peut grandement faciliter la génération de bibliothèque d’ADNc. Alternativement, n’importe quel protocole de bibliothèque d’ADNc de choix peut s’adapter aux étapes de purification d’affinité et d’isolation de l’empreinte décrites ici. Il est important de noter que le traitement par nucléase générant les empreintes ribosomiques nécessite la réparation des extrémités d’ARNm résultante (préparation de la bibliothèque d’ADNc, étape de déphosphorylation) pour permettre les étapes de ligature de liaison suivantes dans le protocole d’ADNc décrit ici dans le protocole de votre choix.
Pendant le séquençage, il est important de différencier SeRP de RNA-Seq, car l’hétérogénéité des bibliothèques générées varie considérablement, en fonction des facteurs marqués d’affinité. Les chaperons moléculaires et les facteurs de ciblage sont souvent plus promiscuités dans la liaison, interagissant avec des centaines ou des milliers de substrats pendant la traduction, ce qui conduit à des bibliothèques d’ADNc très diverses. Cependant, des interacteurs très spécifiques, tels que les interacteurs d’assemblage complexes co-translationnels, peuvent souvent conduire à la génération de bibliothèques d’ADNc beaucoup moins diversifiées. L’augmentation du nombre de bibliothèques diverses et non diversifiées sur la même voie peut grandement améliorer le séquençage et le suivi des résultats de l’analyse des données.
Une autre caractéristique unique de SeRP est sa capacité à capturer les variations locales dans les occupations de ribosomes le long de l’orf permettant la découverte de changements ribosomiques dans le taux de translation associés à chaque ensemble d’interactions. Il est donc impératif de comparer les occupations de ribosomes dans chaque codon le long de l’orf pour identifier correctement l’enrichissement. L’utilisation de moyennes orfs peut entraîner la perte d’interactions transitoires ou de fausses découvertes.
L’utilisation correcte de la méthode SeRP ouvre de nombreuses voies co-traductionnelles vers l’analyse directe, découvrant de nouvelles caractéristiques mécanistes ainsi que de nouveaux facteurs associés aux ribosomes, révolutionnant le domaine de la biosynthèse des protéines.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous tenons à remercier tous les membres du laboratoire pour les discussions fructueuses et Muhammad Makhzumy pour la lecture critique du manuscrit. Ce travail a été financé par la subvention 2106/20 de l’ISF (Israeli Science Foundation).
matériels
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Références
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters - Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon