Method Article
選択的リボソームプロファイリングによる共翻訳相互作用ネットワークのグローバル同定
要約
共翻訳相互作用は、新生鎖修飾、標的化、折り畳み、およびアセンブリ経路において重要な役割を果たします。ここで、選択的リボソームプロファイリング、 in vivo、モデル真核生物 サッカロミセス・セレビシエにおけるこれらの相互作用の直接分析のための方法について説明する。
要約
近年、リボソームが私たちのmRNAを解読するだけでなく、混雑した細胞環境へのポリペプチド鎖の出現を導くことが明らかになりました。リボソームは、膜標的因子の空間的および速度論的に制御された結合、修飾酵素、および折り畳みシャペロンのためのプラットフォームを提供する。高次オリゴマー複合体への集合、ならびにタンパク質間ネットワーク形成ステップでさえ、合成と協調することが最近発見された。
ここでは、 インビボでの共翻訳相互作用を捕捉するために開発された方法である選択的リボソームプロファイリングについて説明する。リボソーム-新生鎖複合体を共翻訳インタラクターとともに捕捉するために必要なさまざまなアフィニティー精製ステップ、ならびにコドンに近い分解能で共翻訳相互作用を解読するために必要なmRNA抽出、サイズ排除、逆転写、ディープシーケンシング、ビッグデータ解析ステップについて詳述します。
概要
SeレクティブリボソームPロファイリング(SeRP)は、今日まで、共翻訳相互作用をインビボで、直接的に捕捉し、特徴付ける唯一の方法である1、2、3、4、5、6。SeRPは、任意の因子と翻訳リボソームとの相互作用のグローバルプロファイリングをコドン分解能2,7に近い状態で可能にする。
この方法は、増殖する細胞のフラッシュ凍結および活性翻訳の保存に依存している。次いで、細胞溶解物をRNase Iで処理して、「リボソームフットプリント」と呼ばれるリボソーム保護mRNA断片を除く細胞内のすべてのmRNAを消化する。その後、サンプルは 2 つの部分に分割されます。1つの部分は、すべての細胞リボソームフットプリントの単離に直接使用され、細胞内で進行中のすべての翻訳を表す。第2の部分は、例えば修飾酵素、転座因子、折り畳みシャペロン、および複合体集合相互作用など、関心のある因子に関連するリボソームの特定のサブセットの親和性精製に使用される。親和性精製されたリボソームフットプリントは、総称してインターアクトームと呼ばれる。次に、リボソームで保護されたmRNAを抽出し、cDNAライブラリーの生成に使用し、続いてディープシーケンシングを行います。
全トランスレートームサンプルとインターアクトームサンプルの比較分析により、関心のある因子に関連するすべてのorfの同定、および各orf相互作用プロファイルの特性評価が可能になります。このプロファイルは、解読されたコドンおよび新興ポリペプチド鎖のそれぞれの残基を推測できる正確な関与開始および終結配列、ならびに相互作用中のリボソーム速度変動を報告する7、8。図1は、このプロトコルを回路図として示しています。
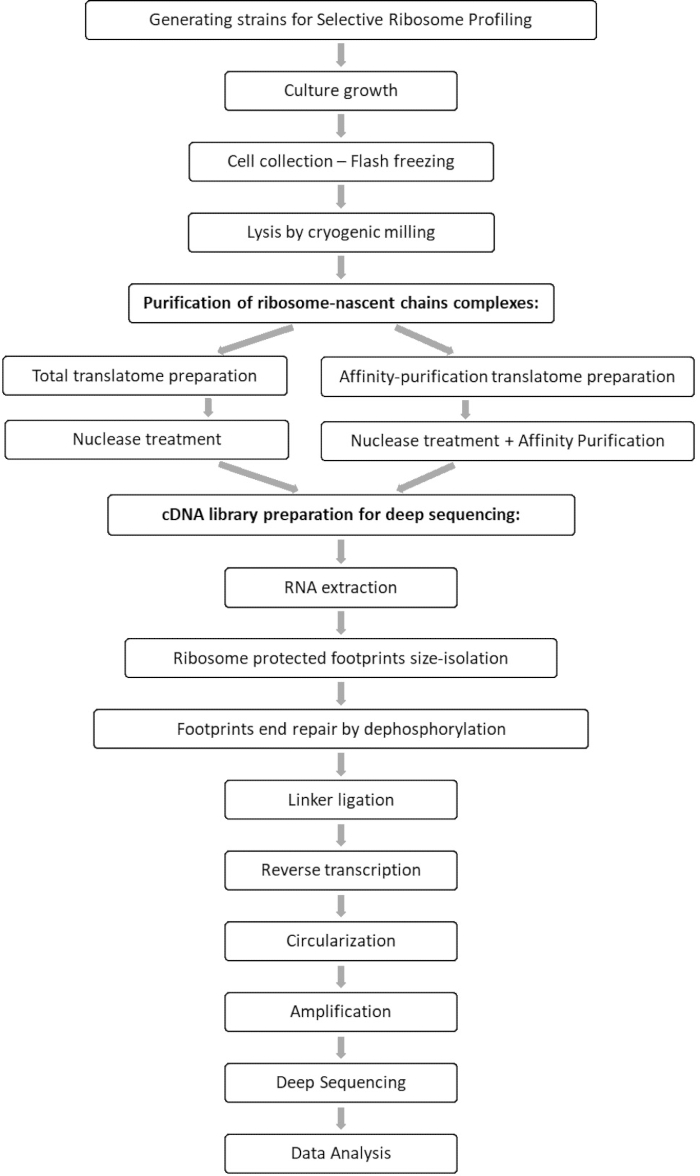
図 1: SeRP プロトコルの概要 このプロトコルは、7〜10日以内にその全体を実行することができます。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
1. 選択的リボソームプロファイリングのための菌株の生成
注:SeレクティブリボソームPロファイリング(SeRP)は、リボソーム - 新生鎖複合体との相互作用の様式を評価するために、関心のある因子のアフィニティー精製に依存する方法である。相同組換え9、ならびにCRISPR/Cas910ベースの方法が、関心のある様々な因子をアフィニティー精製のためのタグと融合させるために利用される。このようなタグは、GFP、GFP−trapアフィニティー精製のための、TAP−タグ、アビジンまたはストレプトアビジンによって精製されたIgG−セファロースビーズ精製ならびにAVI−Tagのための、近年からのいくつかの成功例を列挙する。
- 成長アッセイまたは機能アッセイを実行して、タグ付けがタンパク質の機能に影響を与えなかったことを検証します。N' 端末と C' 端末のタグ付けは評価する必要があります。
注:リボソーム(rRNA)は、様々な要因における多くのリボソーム結合ドメインと同様に、高度に荷電しており、誤った発見または結合モードの変更につながる可能性があるため、高度に荷電したタグ(ポリヒスチジンなど)を使用することは好ましくない。
2. 文化の成長
- 所望のタグ付きタンパク質を含む構築された酵母培養物(株BY4741に基づく)を、液体酵母エキス - ペプトン - デキストロース(YPD)リッチ培地、または合成デキストロース(SD)最小培地(硫酸アンモニウムを含む1.7g / L酵母窒素塩基または硫酸アンモニウムを含まない1.7g / L酵母窒素塩基と1g / Lグルタミン酸一ナトリウム、2%グルコース、およびアミノ酸の完全または適切な混合物)で培養する。
- 250-500 mLの細胞培養物を0.5OD600 (ミッドログ)になるまで、30°Cで、適切な培地中で増殖させる。
3. 細胞の収集と溶解
- ガラスろ過システムを備えた0.45 μmニトロセルロースブロッティング膜(1 Lガラス漏斗付きガラスフィルターホルダー、真空ベースとキャップ、ステンレススチールスクリーン、ガスケットとスプリングクランプ、90 mm、粉砕ジョイントフラスコ1 L)で真空ろ過により細胞を迅速に収集します。
- 回収した細胞をフラッシュフリーズし、ペレット状の細胞をヘラで掻き取り、直ちに液体窒素充填50mLチューブに浸漬した。
停止点:細胞は、-80°Cで最大3〜4週間保存することができる。 - ミキサーミル中で極低温粉砕により細胞溶解を行う:30Hzで2分間2回、1mLの溶解緩衝液を用いて( 表1参照)。フライス加工間で液体窒素中で冷やします。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 10 mg/mL CHX (シクロヘキシミド) | 220 | 0.5 ミリグラム/mL |
| 1M トリス塩酸 pH 8.0 | 88 | 20ミリオンメートル |
| 3M KCl | 205.7 | 140ミリオンメートル |
| 1M ミリグラムCl2 | 26.4 | 6ミリオン |
| 1M PMSF | 4.4 | 1 ミリオン |
| NP-40 | 4.4 | 0.10% |
| プロテアーゼ阻害剤 | 2錠 | |
| DNase I | 8.8 | 0.02 U/mL |
| 最終巻 | 4,400 |
表 1: 溶解バッファーマスターミックスのレシピ。
注:目的のタンパク質が非常に不安定な場合に備えて、溶解バッファーはより多くのプロテアーゼ阻害剤(ベスタチン、リューペプチン、アプロチニンなど)を含むように変更することができますが、次の手順で組み立てられたリボソームの大小のサブユニットを維持するためにEDTAを避けることが重要です。同様の理由から、緩衝液中で常に少なくとも6mMMgCl2 を維持する。
警告: HCl は腐食性が高く、PMSF は有毒です。手袋を着用し、取り扱いには注意が必要です。
- 30,000 x g、4°Cで2分間遠心分離し、溶解液を透明化し、上清を回収した。
4. SeRPに対するリボソーム-新生鎖複合体の精製
- 実験ごとに、上清を2つの部分に分割します。それぞれ異なる微量遠心管:全RNAサンプル(〜200μL)および免疫精製(IP)サンプル(〜700μL)トランスラトームサンプル。
-
全RNAサンプルの処理
- 10UのRNase Iを用いて4°Cで25分間トータルRNAサンプルを消化する;回転ミックスラックで30 RPMで回転します。
注:消化条件は、ポリソームプロファイリングを使用して較正し、モノソームピークの過剰または過小消化がないようにすることができます。 - 表2に記載したようにショ糖クッションマスターミックスを準備する。
- 10UのRNase Iを用いて4°Cで25分間トータルRNAサンプルを消化する;回転ミックスラックで30 RPMで回転します。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 50% スクロース | 200 | 25% |
| 1M トリス塩酸 pH 8.0 | 8 | 20ミリオンメートル |
| 3M KCl | 18.7 | 140ミリオンメートル |
| 1M ミリグラムCl2 | 4 | 10ミリオンメートル |
| 100 ミリグラム/mL CHX | 0.4 | 0.1 ミリグラム/mL |
| プロテアーゼ阻害剤 | 1錠 | |
| 最終巻 | 400 |
表2:スクロースクッションマスターミックスのレシピ。
- サンプルを 400 μL のスクロースクッションにロードし、TLA120 ローターで遠心分離機を 245,000 x g および 4 °C で 90 分間ロードします。
- 真空ポンプで上清をすばやく取り除き、ペレットを150 μLの溶解バッファーでオーバーレイします。4°Cおよび300RPMで1時間振とうすることによってペレットを再懸濁する。
- ピペッティングにより残留ペレットを再懸濁し、新しい1.5mLチューブに移す。
注:100~200μgの総RNAは、通常、全トランスレートームのリボソームプロファイリングに十分である。リボソーム新生鎖複合体アフィニティー精製11 の最も一般的な夾雑物であるrRNA汚染を低減するためにrRNA枯渇工程を追加することができる(さらなる詳細については議論を参照されたい)。
-
免疫精製サンプルの処理
- 1サンプルあたり100~400 μLの親和性結合マトリックス(70%EtOH中の1:1抗体結合ビーズ)を3 x 1mL溶解バッファー(DNase Iおよびプロテアーゼ阻害剤なし)で洗浄する。アフィニティーマトリックスを溶解バッファーに再懸濁し、次いで回転ミックスラックを4°Cで5分間回転させて30RPMで回転させる。沈殿物を3,000 x g、4°Cで30秒間遠心分離した。 上の液体を捨てる。これを3回繰り返します。
- 免疫精製サンプルを、A260 nm単位あたり10 UのRNase Iを親和性結合マトリックス(例えば、サンプルあたり100〜400 μLのGFP-TRAPなど)とともに消化する。
- 回転ミックスラックで30RPMで25分間回転させ、タンパク質をアフィニティーマトリックスに結合させ、4°Cで行います。
- 洗浄バッファーマスターミックスを 表 3 に詳述するように準備します。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 10 ミリグラム/ミリリットル CHX | 50 | 0.1 ミリグラム/mL |
| 1M トリス塩酸 pH 8.0 | 100 | 20ミリオンメートル |
| 3M KCl | 233 | 140ミリオンメートル |
| 1M ミリグラムCl2 | 50 | 10ミリオンメートル |
| 1M PMSF | 5 | 1 ミリオン |
| NP-40 | 0.5 | 0.01% |
| プロテアーゼ阻害剤 | 2錠 | |
| 50%グリセロール | 1,000 | 10% |
| 最終巻 | 5,000 |
表3:洗浄バッファーマスターミックスのレシピ。
- 親和性結合マトリックスを1mLの洗浄緩衝液で3回洗浄し、毎回〜1分間、ミックスラック内で30RPMで回転させ、4°Cで。
- 沈殿物を3,000 x g で4°Cで30秒間遠心分離した。 上の液体を捨てる。
- 1mL洗浄緩衝液でさらに2回洗浄し、その都度5分間、ミックスラック内で30RPMで回転させ、4°Cで洗浄する。
- 沈殿物を3,000 x g および4°Cで30秒間遠心分離することにより得る。
- タンパク質溶出には 50 μL のビーズを使用し、同量の 2x サンプルバッファーを使用します。残りのビーズをRNA抽出に使用します。
- 3,000 x g、4°Cで30秒間遠心分離し、ビーズをペレット化し、上液を捨てた。
- 液体窒素中で凍結し、-80°Cで保存する。 これらのサンプルは、その後の RNA 抽出に使用します。
停止点:サンプルは-80°Cで一晩以上保存できます。これは停止点になる可能性があります。 - 各ステップのアリコートによるウェスタンブロットまたはクーマシー染色(〜10体積%、混合後)によるアフィニティー精製ステップの成功を評価する。タグなしWT株では、アフィニティーマトリックスへの非特異的結合のコントロールとして、常にモックIPを使用してください。
注:高い非特異的バックグラウンドは、塩/洗剤濃度の増加を伴う追加の洗浄ステップによって克服することができます。一過性相互作用は、種々の架橋剤処理、例えば、生細胞のパラホルムアルデヒド(PFA)処理−増殖培地に0.4%〜1%PFAを2〜5分間添加し、続いてグリシン(0.3M)クエンチを3分間行うことを強く推奨する。
警告: パラホルムアルデヒドは発がん性物質の疑いがあります。パラホルムアルデヒドはすぐに蒸発し、腐食性であるため、化学安全フードで作業し、2層の手袋を着用してください。
5. ディープシーケンシングのためのcDNAライブラリー調製
- RNA抽出
注: RNase フリーのノンスティック 1.5 mL チューブを使用して、RNA または DNA の枯渇の可能性を防ぎます。- ステップ 4.2.5 および 4.3.12 のサンプルを氷上で解凍し、10 mM Tris-HCl pH 7.0 でサンプルを最終容量 700 μL に再懸濁します。
警告: 酸フェノールとクロロホルムは揮発性で有害です。化学薬品の安全フードで作業してください。 - 40 μL の 20% SDS を 0.7 mL のトータル RNA 溶出液または IP 溶出液に加えます。数回閉じて反転します。タンパク質沈殿はサンプルを白くするはずです。
- 0.75 mLの予め加温した酸 - フェノール:クロロホルムをサンプルに加える。チューブをしっかりと密封し、サーマルミキサーで1,400 RPMで5分間、65°Cで振ってください。 サンプルを氷上で5分間冷やします。
- ステップ5.1.3からチューブを遠心分離する。20,000 x g で2分間。上部の水層を新鮮なチューブに移し、それに0.7mLの酸 - フェノール:クロロホルムを加える。
- 室温で5分間インキュベートし、時にはボルテックスする。20,000 x gで2分間遠心分離 する。上部の水層を新鮮なチューブに移し、それに0.6mLのクロロホルムとボルテックスを加える。
- 20,000 x gで1分間遠心分離 する。上部の水層を新鮮なチューブに移す。
- 78 μL の 3 M NaOAc、pH 5.5、2 μL の GlycoBlue、および 0.75 mL のイソプロパノールを加えて核酸を沈殿させます。5分間徹底的に渦巻きます。-80°Cで少なくとも1時間、または-20°Cで16時間インキュベートする。
- 20,000 x g および4°Cで30分間遠心分離し、上清を捨てる。ペレットを氷冷0.75mLの80%エタノールで洗浄する。チューブを反転させて徹底的に洗浄します。20,000 x g で4°Cで5分間遠心分離し、上清を捨てた。
- 450 x g、4 °Cで20秒間スピンダウンし、残りのエタノールを除去して液体を捨てます。開いた蓋でペレットを65°Cで5分間乾燥させる。 以下のようにサンプルを再懸濁する:IPについては、10 μLの10 mM Tris-HCl、pH 7.0でサンプルを再懸濁する。全トランスレートーム分析では、サンプルを20 μLの10 mM Tris-HCl、pH 7.0に再懸濁します。
停止点:RNAは-80°Cで数ヶ月間保存することができます。
- ステップ 4.2.5 および 4.3.12 のサンプルを氷上で解凍し、10 mM Tris-HCl pH 7.0 でサンプルを最終容量 700 μL に再懸濁します。
- 蛍光測定による総RNA濃度の定量
注:次世代シーケンシング用のcDNAライブラリを準備する際には、以下のすべての材料および表面をRNaseフリーにする必要があります。RNAサンプルを取り扱うときは、手袋を着用してください。- 1 μL の酸性フェノール抽出トータル RNA を 9 μL の 10 mM Tris-HCl、pH 7.0 に希釈します。製造元のWebサイトの指示に従って、蛍光光度計を使用して定量します。
- 50 μg の RNA を含むサンプルを 10 μL の 10 mM Tris-HCl、pH 7.0 で希釈します。
メモ:IPサンプルは測定せず、次のステップですべてを使用してください。
- リボソーム保護フットプリント断片をゲル精製
- 15%TBE-尿素ポリアクリルアミドゲルをセットし、1x TBEランニングバッファーに沈めます。サンプルローディングの前に200Vで30分間実行します。各サンプルに、20 μL の 2x TBE-尿素サンプルバッファーを追加します。
メモ: 予想されるバンドサイズは約 25 ~ 35 nt です。 - 10 bp の DNA はしごを解凍し、サンプル (はしごではない) を 80 °C で 2 分間変性させた後、氷上で冷やします。各サンプルを他のすべてのレーンにロードします。ゲルを200Vで50〜70分間実行します。
- 60 mL の 1x TBEバッファーで 6 μL の SYBR Gold (10,000 x 濃縮物) を希釈し、光で保護されたボックス内で 15 ~ 20 分間振盪しながら染色します。ゲルを染色しながら、滅菌メスおよび標識された1.5 mLチューブに0.5 mLゲルブレーカーチューブを調製する。
- 滅菌メスで目的のバンドを切除し(新鮮なものを使用するか、サンプル間でよくきれいにしてください)、各ゲル片をゲルブレーカーチューブに入れます。
- ゲルの画像を撮影して、ゲルにサンプル残渣が残っていないことを確認します。
- 切断スライスを含むチューブを20,000 x g で4°Cで5分間遠心分離し、残りのゲル片をゲルブレーカーチューブから1.5 mLチューブに移します。
- 0.5 mLの10 mMトリス、pH 7.0を加える。サーマルミキサーで1,400 RPMで70°Cで10分間振る。
- ワイドボアピペットチップを備えたセルロースアセテートカラムに移し、20,000 x g で4°Cで3分間遠心分離した。
- フロースルーを新しい1.5mLチューブに移し、氷上で冷やします。
- 核酸を沈殿させるために、550 μLのIPA、55 μLの3 M NaOAc、および2 μLのGlycoBlueと渦を加えて、十分に混合する。サンプルを-80°Cで少なくとも1時間置く。
- 20,000 x g および4°Cで少なくとも1時間遠心分離し、上清を捨てる。ペレットを0.75mLの氷冷80%エタノールで洗浄する。ペレットが底部から離れるまで徹底的に洗浄するためにチューブを反転させる。再び20,000 x g で4°Cで5分間遠心分離し、上清を捨てた。
- 450 x g、4 °Cで20秒間スピンダウンし、残りのエタノールを除去します。開いた蓋でペレットを65°Cで5分間乾燥させる。
- 15 μLの10 mM Tris、pH 7.0を加え、ペレットを完全に再懸濁する。450 x g、4 °C で 20 秒間スピンダウンし、サンプルを新しい 1.5 mL チューブに移します。
停止点:精製されたRNAは、-80°Cで数ヶ月間保存することができます。
- 15%TBE-尿素ポリアクリルアミドゲルをセットし、1x TBEランニングバッファーに沈めます。サンプルローディングの前に200Vで30分間実行します。各サンプルに、20 μL の 2x TBE-尿素サンプルバッファーを追加します。
- 脱リン酸化
- 各サンプルに3 μLの以下のミックスを使用する:ATPを含まない2 μLの10x T4ポリヌクレオチドキナーゼ反応バッファーに1 μLのRNase阻害剤を加える。各サンプルに2μLのT4ポリヌクレオチドキナーゼを加える。ピペットで優しくよく混ぜ、振とうせずに37°Cで2時間インキュベートします。
- 酵素を失活させるには、サンプルを75°Cで10分間インキュベートし、450 x g、4°C、20秒間スピンダウンします。
- 核酸を沈殿させるには、2 μL の GlycoBlue、550 μL の IPA、および 55 μL の 3 M NaOAc を加えます。
- ボルテックスで十分に混合し、サンプルを-80°Cで少なくとも1時間冷却する。
停止点:サンプルは-80°Cで一晩以上保存できます。 - 手順 5.3.11 ~ 5.3.13 を繰り返します。
停止点:脱リン酸化RNAは、-80°Cで数ヶ月間保存することができます。
- バイオアナライザーを用いた定量
- 1 μLのサンプルと4 μLのDEPC処理水を混合して、各RNAサンプルを1:4に希釈する。
注意:DEPCは発癌物質です。手袋を着用し、慎重に作業してください。 - バイオアナライザースモールRNAチップ/テープステーションを実行します。製造元のプロトコルに従います。
注:予想されるリボソーム保護RNA断片サイズは約28〜30 ntである。
- 1 μLのサンプルと4 μLのDEPC処理水を混合して、各RNAサンプルを1:4に希釈する。
- リンカ-1で3'終了をリゲート
- 5 pmol の低分子 RNA 断片を 10 mM トリス、pH 7.0 で 10 μL に希釈します。サンプルを80°Cで2分間変性させ、氷上で冷やす。
- 表 4 に詳述されているようにマスターミックスを調製し、サンプルあたり 29 μL を使用します。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 50%滅菌ろ過PEG 8000 | 16 | 20% |
| ティッカー | 4 | 10% |
| 10× T4 RNAリガーゼ2バッファー | 4 | 1倍速 |
| スーパーアーゼインヒビター | 2 | 2 U |
| 10mM 腺付加リンカー 3-L1 | 0.1 | 25 μM |
| デップ処理水 | 2.9 | |
| 最終巻 | 29 |
表4:3'末端ライゲーションマスターミックスのレシピ。
- T4 RNAリガーゼ2を1 μL加え、ピペットを軽く加えてよく混ぜる。23°Cで2時間インキュベートする。
- 核酸を沈殿させるために、550 μLのIPA、500 μLの10 mM Tris、pH 7.0、55 μLの3 M NaOAc、および2 μLのGlycoBlueを加える。渦を十分に混合し、試料を-80°Cで少なくとも1時間置いた。
停止点:サンプルを-80°Cで一晩以上保管してください。 - 手順 5.3.2 ~ 5.3.12 を繰り返します。
- ペレットを6 μLの10 mM Tris、pH 7.0に再懸濁する。450 x g、4 °C で 20 秒間スピンダウンし、サンプルを新しい 1.5 mL チューブに移します。
停止点:サンプルは-80°Cで数ヶ月間保存することができます。
-
3'リンクされたフットプリントのゲル精製
- 10%TBE-尿素ポリアクリルアミドゲルをセットし、1x TBEランニングバッファーに沈めます。サンプルローディングの前に200Vで30分間実行します。各サンプルに、6 μL の 2x TBE-尿素サンプルバッファーを追加します。
メモ: 予想されるバンドサイズは約 71 ~ 73 nt です。 - 手順 5.3.2 ~ 5.3.13 を繰り返します。
停止点:サンプルは-80°Cで数ヶ月間保存することができます。
- 10%TBE-尿素ポリアクリルアミドゲルをセットし、1x TBEランニングバッファーに沈めます。サンプルローディングの前に200Vで30分間実行します。各サンプルに、6 μL の 2x TBE-尿素サンプルバッファーを追加します。
-
3'リンクされたフットプリント断片を逆転写してssDNAを生成する
- 表 5 に詳述されているようにマスターミックスを調製し、サンプルあたり 3 μL を使用します。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 10 mM dNTP | 1 | 0.5ミリアンペア時 |
| 25 μM リンカー L(rt) | 0.5 | 625 ナノメートル |
| デップ処理水 | 1.5 | |
| 最終巻 | 3 |
表5:核酸の変性前の逆転写バッファーマスターミックスのレシピ。
- 渦を巻き起こし、サンプルをスピンダウンします。
- サンプルを65°Cで5分間インキュベートします。
- 氷の上でサンプルを冷やす。
- 表6に詳述されているようにマスターミックスを調製し、サンプルあたり6μLを使用する。渦を巻き起こし、サンプルをスピンダウンします。各サンプルに1 μLの上付き文字IIIを加え、ピペットで軽く混合し、50°Cで30分間インキュベートします。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 5× FS バッファ | 4 | 1倍速 |
| スーパーアーゼインヒビター | 1 | 2 U |
| DTT 0.1 M | 1 | 5ミリオン |
| 最終巻 | 6 |
表6:核酸の変性後の逆転写バッファーマスターミックスのレシピ。
- 2.3 μLの1 N NaOHを加え、RNAを加水分解し、逆転写を消光する。
警告: NaOH は腐食性が高いです。手袋を着用し、目の保護具を着用してください。 - サンプルがピンク色に変わるまで、95°Cで15分間インキュベートする。
- 10%TBE-尿素ポリアクリルアミドゲルをセットし、1x TBEランニングバッファーに沈めます。サンプルローディングの前に200Vで30分間実行します。各サンプルに、23 μL の 2x TBE-尿素サンプルバッファーを追加します。
注: 予想される DNA バンド サイズは 115 ~ 117 nt です。 - 手順 5.3.2 ~ 5.3.12 を繰り返します。
- ペレットを15 μLの10 mM Tris、pH 8.0に再懸濁する。450 x g、4 °C で 20 秒間スピンダウンし、サンプルを新しい 1.5 mL チューブに移します。
停止点:サンプルは-80°Cで数ヶ月間保存することができます。
-
ssDNA環状化
- 以下のマスターミックスを調製し、 表7に詳述されているように、サンプルあたり4μLをロードします。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| 10× サークリガーゼII緩衝液 | 2 | 1倍速 |
| 5 M ベタイン (オプション) | 1 | 0.25メートル |
| 50 ミリグラム MnCl2 | 1 | 2.5ミリオン |
| 最終巻 | 4 |
表7:ssDNA環状化マスターミックスのレシピ。
- 各サンプルに1 μLのCircLigase II ssDNAリガーゼを加え、60°Cで1時間インキュベートします。
注:このステップの効率は、1時間のインキュベーション後に各サンプルに1μLのCircLigase II ssDNAリガーゼを添加することによって増加させることができる。 - 80°Cで10分間インキュベートすることにより酵素を失活させる。
- 氷上で冷やし、PCR増幅を続けるか、-80°Cで保存する。
停止点:サンプルは-80°Cで何年も保存できます。
-
PCR増幅
- 以下のPCRマスターミックスを調製し、表8に詳述されているように、サンプルあたり 82 μLをロードします。
| 試薬 | 1サンプルあたりの量(μL) | 最終濃度 |
| デップ処理水 | 61.6 | |
| 5× フジオンHF反応バッファー | 17.6 | 1倍速 |
| 10 mM dNTP | 1.8 | 200 μM |
| 100 μM PCR フォワードプライマー | 0.2 | 225 ナノメートル |
| HFフシオンポリメラーゼ | 0.8 | 1.6 U |
| 最終巻 | 82 |
表 8: PCR 増幅マスターミックスのレシピ。
- マスターミックスを含む各チューブに、5μLの環状化DNAを加える。
注:残りの円形化DNAサンプルを-80°Cで保管してください。 - 異なる1 μLの20 μM PCRリバースバーコードプライマーを各サンプル( 表9参照)に加え、ボルテックスを加えて十分に混合します。
- 各チューブを4つの別々のPCRチューブにアリコートし、それぞれが異なる数のPCRサイクルに使用されます。
- 表10に詳述するように、以下のプログラムに従ってPCR反応を実行する。
| サイクル | 変性(98°C) | アニール(60°C) | 延長 (72 °C) |
| 1 | 30秒 | ||
| 2-16 | 10秒 | 10秒 | 5 秒 |
表10:PCR反応用PCRプログラム
- サイクル 8、9、10、および 11 の後、最初の試行として PCR チューブを取り外します (IP サンプルの場合、サイクルの範囲は 9 ~ 15 です)。各サイクルの後、プログラムを一時停止し、アリコートを1つ取り出して氷の上に置き、すぐにプログラムを再開します。
注:サイクル数は、各反応における環状化DNAの量に基づいて調整する必要があります。例とさらなる説明については、 図 4 を参照してください。 - 各 17 μL の反応に、3.5 μL の 6 x DNA ローディング色素を加えます。
- 10 bp の DNA はしごを解凍します。
- ゲル電気泳動によるサイズ分離のために、8%TBEポリアクリルアミドを1x TBEランニングバッファーに沈め、各サイクル番号のサンプルを隣接するウェルにロードし、ゲルを180Vで50分間実行します。
- 60 mL の 1x TBEバッファーで 6 μL の SYBR Gold (10,000 x 濃縮物) を希釈し、光で保護されたボックス内で 15 ~ 20 分間振盪しながら染色します。
- ゲルを染色しながら、滅菌メスおよび標識された1.5 mLチューブに0.5 mLゲルブレーカーチューブを調製する。
- 染色された核酸の画像を撮影する。
- 滅菌メスで174-176 bpの予想されるバンドサイズで所望のバンドを切断し、調製した0.5mLゲルブレーカーチューブにゲルスライスを置く(サンプル間の十分に洗浄し、RNase不活性化剤を使用するか、新しいブレードに切り替える)。
- チューブを 20,000 x g および 4 °C で 5 分間遠心分離し、残りのゲル片を 0.5 mL ゲルブレーカーチューブから 1.5 mL チューブに移します。
- 500 μL の 10 mM Tris、pH 8.0 を加え、サーマルミキサーで 1,400 RPM で 10 分間、70 °C で振とうします。
- 溶解したゲルを、広いボアピペットチップを備えたセルロースアセテートカラムに移す。
- カラムを 20,000 x g および 4 °C で 3 分間遠心分離し、フロースルーを新しい 1.5 mL チューブに移し、氷上で冷やします。
- 核酸を沈殿させるには、IPA 550 μL、5 M NaCl 32 μL、0.5 M EDTA 1 μL、および GlycoBlue と Vortex 2 μL を加えて完全に混合します。
- サンプルを-80°Cまたは-20°Cで一晩少なくとも1時間保管してください。
停止点:サンプルは-80°Cで一晩以上保存できます。 - 手順 5.3.11 ~ 5.3.12 を繰り返します。
- 11 μL の 10 mM トリス、pH 8.0 で再懸濁します。450 x g、4 °C で 20 秒間スピンダウンし、サンプルを新しい 1.5 mL チューブに移します。
停止点:サンプルは-80°Cで何年も保存できます。
-
バイオアナライザーによるサイズ分布の定量化
- 1 μLのサンプルと4 μLのDEPC処理水を混合して、各サンプルの1:4希釈を行います。
- バイオアナライザースモールRNAチップを実行します。製造元のプロトコルに従います。
メモ: 想定される長さは 175 ± 5 bp です。
-
蛍光光度計によるDNA濃度の定量
- dsDNAの高感度濃度チェックは、メーカーの推奨に従って蛍光光度計で行います。
- Illuminaの推奨事項(インデックスアダプタプーリングガイド12)に従ったマルチプレックスおよびシーケンスサンプル。
6. データ解析
- 補足ファイルに詳述されているように分析を実行します。
結果
このプロトコールのフローチャート(図1)に例示されるように、細胞を対数期まで増殖させ、次いで濾過によって迅速に回収し、極低温粉砕によって溶解した。次に、ライセートを2つに分けました:1つはリボソームで保護されたmRNAフットプリント用、もう1つは選択されたリボソーム保護mRNAフットプリント用で、アフィニティー精製を実施してタグ付きタンパク質-リボソーム-新生鎖複合体をプルダウンしました。 図2に見られるように、タグ付きタンパク質発現とウェスタンブロット分析によるプルダウンの成功を確認しました。リボソームで保護されたフットプリント(通常20~45 ntの長さ)の単離を、スモールRNA電気泳動(2100 BioAnalyzerシステム)によって検証し、システムマニュアルに従ってサイズ検出の5~10 ntシフトを可能にしました(図3)。次に、ディープシーケンシングとビッグデータ解析のためのcDNAライブラリを生成しました。cDNAライブラリの生成中は、アンダーサイクリングは低収率( 図3のレーン2に見られるように)につながる可能性があるが、生成されたライブラリを回復するために再増幅が可能であることに注意してください。PCRプライマーが枯渇しても反応が継続すると、オーバーサイクルが発生することがあります。dNTPsがまだ存在する場合、反応は進行し、PCR産物がそれ自体13 をプライミングすることによるキメラ配列を有するより長いPCRアーティファクトを生成する( 図3のレーン3〜4に見られるように、目に見えるスミアによって示される)。dNTPsの濃度も制限的になると、部分的に相同なライブラリー断片のみからなるヘテロ二本鎖の存在を示す産物が出現し得る。 図4 は基準として機能し、レーン2は最適な増幅を表し、レーン3は許容可能な増幅を表します。レーン 4 および 5 (サイクル 10 および 11) からのサンプルは、PCR の複製物およびアーティファクトを導入する可能性があるため、使用しないでください。生成されたライブラリーを、正確なサイズ分布および定量のために、高感度DNA電気泳動(同じBioAnalyzerシステムを使用した)によってさらに検証した(図5)。3'末端リンカーライゲーション、逆転写およびPCR増幅の後、cDNA長分布が期待され、175nt付近に鋭いピークを有する。
シーケンスされたライブラリからアダプターとバーコードをトリミングして削除し、20 ~ 45 nt の読み取りのみがさらなる分析のために選択されました。 図 6 に、結果の長さ分布を示します。読み取りを、コード配列、イントロン、および遺伝子間配列(図7)の異なるグループに分け、さらに 図8に示すように分類した。
共翻訳相互作用の検出および特徴付けのための最終分析を、リボソーム保護mRNA断片の富化に基づいて実施し、図9のグラフを作製した。我々は、全トランスレイトームの正規化されたリボソーム占有率(各orfに沿った各ヌクレオチドにおける)を、対応する選択されたトランスレートーム(新生児インターアクトーム)と比較した。ヌクレオチドごとの比較は、翻訳速度アーチファクトを排除する。生物学的反復間の再現性は、ピアソン相関によって評価した(閾値>0.6)。我々は、Vma2pと3つのタンパク質(リボソーム関連シャペロンSsb1p、Pfk2p(ホスホフルクトキナーゼ)およびFas1p(脂肪酸合成酵素)との共翻訳相互作用を分析し、各タンパク質C'末端にGFPによってタグ付けされた選択的リボソームプロファイルを提示する。我々は、生物学的複製においてプロトコルを実施した。図9A、D及びGは、それぞれアフィニティー精製の実験スキームを示す。次に、液胞H+-ATPaseのサブユニットをコードするVma2p orfに沿ったSsb1インターアクトームと比較した全トランスレートームのリボソーム占有率を示す(図9 B、EおよびH)。最後に、オルフに沿った[コドン/aa]の各リボソーム位置で、比率ベースのリボソーム富化プロファイリング(IP/Total)を行った(図9 C、FおよびI)。これら3つのタンパク質とリボソームによって合成されているVma2pとの共翻訳相互作用を比較すると、SeRPによって4つの有意な濃縮ピークを同定したため、Ssb1シャペロンがorfに沿った4つの異なる領域で新生Vma2pと係合することが明らかになった。異なることに、Pfk2pは、SeRPによって同定されたように、共翻訳シャペロンSsb1と比較して異なる位置に1つの有意な濃縮ピークのみを示す。したがって、これらの比率ベースの濃縮リボソームプロファイルの比較は、コドン分解能に近い様々な共翻訳相互作用の検出および特性評価におけるこのプロトコルの力を実証する。
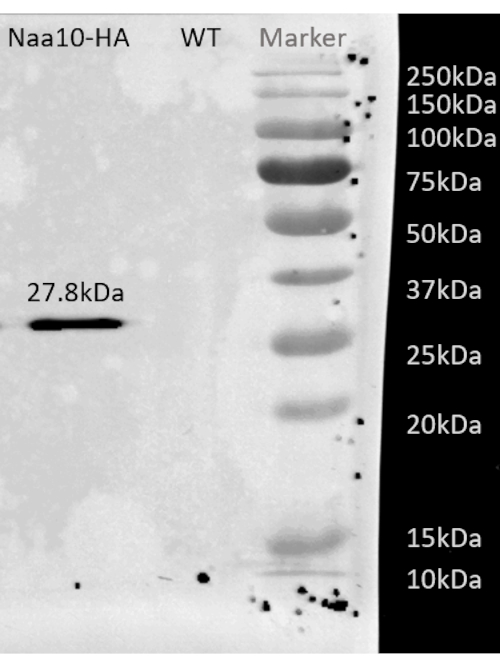
図2:HAタグNaa10によるBY4741株のアフィニティー精製後の代表的なウェスタンブロット結果。 HAタグ付きNaa10を用いたBY4741株のアフィニティー精製後の代表的なウェスタンブロット結果は、27.8kDa付近のバンドを示し、一方、野生型は、ネガティブコントロールとして、バンドを示さない。 この図の拡大版を表示するには、ここをクリックしてください。

図3:フットプリントの単離と酸-フェノール:クロロホルムによるRNA抽出後の代表的なBioAnalyzerの結果、および平均サイズは25 ntです。この図の拡大版を表示するには、ここをクリックしてください。
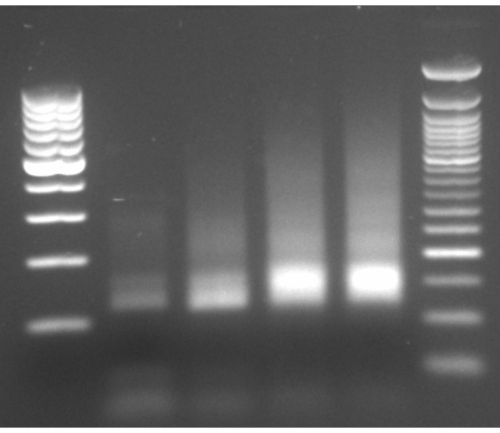
図4:PCR増幅の代表的なゲル電気泳動。 サイクル8〜11からのPCR産物をロードしたレーン2〜5、および両側にはしごを用いたPCR増幅の代表的なゲル電気泳動。 この図の拡大版を表示するには、ここをクリックしてください。
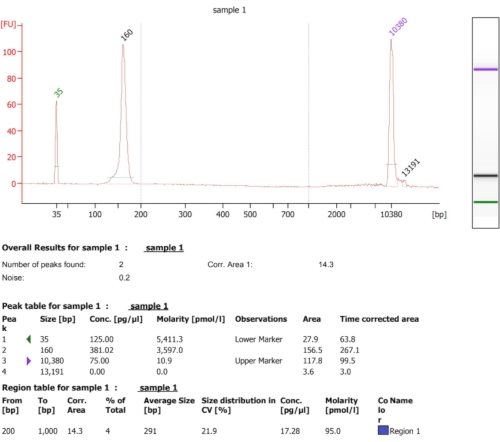
図5:cDNAライブラリの作成後に得られた代表的なBioAnalyzerの結果。 この図の拡大版を表示するには、ここをクリックしてください。

図6:Cutadaptを使用してアダプタを取り外した後の読み取りの予想される長さ分布 (20より短い読み取りまたは45より長い読み取りの除去)。 この図の拡大版を表示するには、ここをクリックしてください。

図7:Bowtie2で非コードRNA読み取りを除去し、TopHatを使用して残りの読み取りを異なる生物にアラインメントした後の予想されるアラインメント成功率。 サンプルは 、S. cerevisiae (BY4741の変異変異体)から採取した。 この図の拡大版を表示するには、ここをクリックしてください。
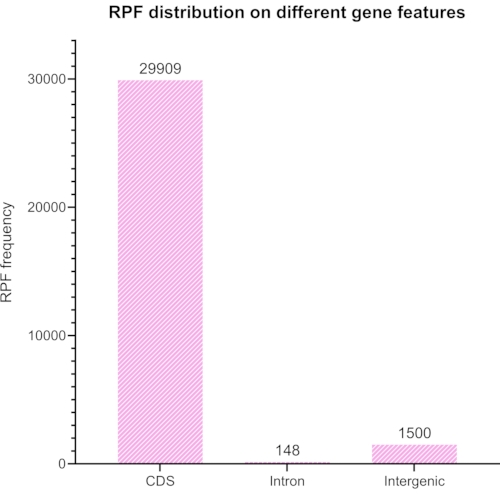
図8:RiboToolkitで生成されたグラフは、Bowtie2を使用して読み取り中のrRNA要素を除去した後のアライメントされた読み取りの予想されるコーディング対非コーディングの比率を表しています。 この図の拡大版を表示するには、ここをクリックしてください。
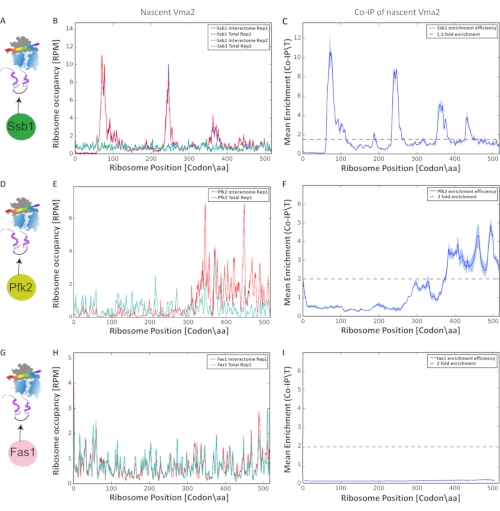
図9:Ssb1p、Pfk2p、およびFas1pの3つの異なるタンパク質と、SeRPによって分析されたリボソームによって合成されているVma2pとの共翻訳相互作用。 すべての y 軸は、100 万回あたりの読み取り数 (RPM) で示されます。(A, D, G)Ssb1p、Pfk2p、およびFas1p C'のSeRPの実験スキームは、それぞれGFPによって末端タグが付けられた。(B、E、H)Ssb1、Pfk2p、およびFas1pインターアクトームと比較した全翻訳体の orf に沿ったリボソーム占有率(生物学的反復において)。(C, F, I) それぞれ、orfに沿った[コドン/aa]の各リボソーム位置におけるSsb1p、Pfk2p、およびFas1pの平均濃縮度(IP/総比)。生物学的反復間の変動は、陰影付き領域によって示される。 この図の拡大版を表示するには、ここをクリックしてください。
表9:3'リンカーおよびプライマー配列。 3'リンカーL1:5'アデニル化および3'ジデオキシ-シチジンの一意の分子識別子('NN...')を有するリンカー3-L1(RNaseフリーHPLC精製;逆転写リンカー:5'リン酸化された一意の分子識別子を有する逆転写(L(rt))(RNaseフリーHPLC精製);PCRフォワードプライマー:PCRf;HPLC精製。 この表をダウンロードするには、ここをクリックしてください。
ディスカッション
ここで、プロトコルは、コドン分解能に近い共翻訳相互作用を捕捉するための選択的リボソームプロファイリングアプローチを詳述する。リボソームが混雑した細胞質への新生鎖出現を調整するためのハブとして上昇するにつれて、これは機能的なプロテオームを確保するために必要な様々な共翻訳相互作用を同定し、特徴付けるために、そして様々な疾患を研究するための重要な方法である。今日まで、SeRPは、インビボで、直接的に、これらの相互作用を捕捉し、特徴付けることができる唯一の方法である14、15、16。
最初の最も重要なステップは、細胞の収集と溶解です。数秒以内に、フラッシュフリーズに続いて凍結状態で溶解することによって、進行中の翻訳をキャプチャすることが不可欠です。細胞収集は、リボソーム流出を回避し、急速に起こり得るストレス翻訳応答を誘導するために、急いで行わなければならない。第2の重要な工程は、アフィニティー精製工程である。共翻訳相互作用が維持されていることを確認しながら、ストリンジェントな洗浄によってバックグラウンド結合を減少させることが不可欠であり、これは in vivo 架橋によって促進され得る。このプロトコルは高感度NGS(次世代シーケンシング)に基づいているため、最初のステップで高バックグラウンドで、以下のcDNAライブラリ調製ステップで増幅することができ、低いシグナル対ノイズ比につながります。
ヌクレアーゼ処理は、すべての非保護mRNAを消化するために、RNA消化の過または下を避けるために単離されたリボソームフットプリントサイズ分布(上記で詳述したように)の慎重な評価と共にポリソームプロファイリング17 によって評価されるべきである。ヌクレアーゼ濃度と消化時間の較正は、過剰消化がリボソームrRNA消化につながり、リボソームで保護されたフットプリントの損失につながる可能性があるため、正確なフットプリントの回復を促進することができます。ここで説明するcDNAライブラリの準備ステップおよびデータ分析ステップは、長くて特徴のない読み取りを破棄するため、消化不足はリボソームで保護されたフットプリントの発見率を低下させる可能性があることに注意することが重要です。
rRNAの枯渇は必ずしも重要なステップを構成するわけではなく、必須ではありませんが、サンプルの洗浄やゲノムマッピング読み取り率の向上など、いくつかの利点があります。一方、多くのrRNA枯渇プロトコルも所望のリボソーム保護断片の枯渇を引き起こす可能性があるため、バイアスの可能性がある。また、rRNA枯渇キットのコストも考慮する必要があります。rRNA枯渇は、リボソーム・フットプリント単離工程後またはcDNA環状化工程後に行うことができる。
cDNAライブラリー調製ステップは、ここに記載されるように、低mRNAインプットに対して最適化されており、親和性およびリボソーム精製ステップがmRNAインプット量を高度に減少させるほど、RNA-seq発現研究と比較して。細胞培養の初期量をアップスケーリングすると、cDNAライブラリーの生成が大幅に促進されます。あるいは、選択した任意のcDNAライブラリプロトコルは、ここで説明するアフィニティー精製およびフットプリント分離ステップに適合させることができます。リボソームフットプリントを生成するヌクレアーゼ処理では、選択したプロトコルでここで説明するcDNAプロトコルで次のリンカーライゲーションステップを可能にするために、結果として生じるmRNA末端の修復(cDNAライブラリ調製、脱リン酸化ステップ)が必要であることに注意することが重要です。
シーケンシング中は、生成されたライブラリの不均一性がアフィニティータグ付き因子によって大きく異なるため、SeRPをRNA-Seqと区別することが重要です。分子シャペロンおよび標的因子は、しばしば結合においてより乱雑であり、翻訳中に数百または数千の基質と相互作用し、非常に多様なcDNAライブラリーをもたらす。しかし、共翻訳複合体アセンブリインタラクターなどの高度に特異的な相互作用因子は、しばしばはるかに多様性の低いcDNAライブラリーの生成につながる可能性があります。同じレーン上の多様なライブラリと非多様なライブラリのスパイクは、シーケンシングとデータ解析結果の追跡を大幅に改善することができます。
SeRPのもう1つのユニークな特徴は、 ORF に沿ったリボソーム占有率の局所的な変動を捕捉する能力であり、相互作用の各セットに関連する翻訳速度のリボソームシフトの発見を可能にする。したがって、エンリッチメントを正しく識別するために、 orf に沿った各コドンのリボソーム占有率を比較することが不可欠です。 ORF の平均を利用すると、一時的な相互作用の損失や誤った発見につながる可能性があります。
SeRP法を正しく使用すると、多くの共翻訳経路が直接解析に開かれ、新しい機構的特徴と新しいリボソーム関連因子が発見され、タンパク質生合成分野に革命がもたらされます。
開示事項
著者らは利益相反がないと宣言しています。
謝辞
実りある議論をしてくれた研究室のメンバー全員と、原稿を批判的に読んでくれたムハンマド・マフズミーに感謝します。この研究は、ISF(イスラエル科学財団)助成金2106/20によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
参考文献
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23(2012).
- Guide, P. Illumina Index Adapters - Pooling Guide. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019).
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820(2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved