Method Article
זיהוי גלובלי של רשתות אינטראקציה בין תרגומים משותפים על ידי פרופיל ריבוזום סלקטיבי
In This Article
Summary
אינטראקציות של תרגום משותף ממלאות תפקיד מכריע בשינויים בשרשרת המתהווה, במיקוד, בקיפול ובמסלולי הרכבה. כאן, אנו מתארים פרופיל ריבוזום סלקטיבי, שיטה לניתוח ישיר של in vivo, של אינטראקציות אלה במודל eukaryote Saccharomyces cerevisiae.
Abstract
בשנים האחרונות התברר כי ריבוזומים לא רק מפענחים את ה-mRNA שלנו אלא גם מנחים את הופעתה של שרשרת הפוליפפטידים לסביבה התאית הצפופה. ריבוזומים מספקים את הפלטפורמה לקשירה מבוקרת מרחבית וקינטית של גורמי מיקוד ממברנה, אנזימים משנים ומלווים מתקפלים. אפילו ההרכבה לקומפלקסים אוליגומריים מסדר גבוה, כמו גם לשלבי היווצרות רשת חלבונים-חלבונים, התגלו לאחרונה כמתואמים עם סינתזה.
כאן אנו מתארים פרופיל ריבוזום סלקטיבי, שיטה שפותחה כדי ללכוד אינטראקציות תרגום משותף in vivo. אנו נפרט את שלבי טיהור הזיקה השונים הנדרשים ללכידת קומפלקסים של שרשרת ריבוזום-מתהווה יחד עם אינטראקציות תרגום משותף, כמו גם את שלבי מיצוי ה-mRNA, הרחקת הגודל, השעתוק ההפוך, הריצוף העמוק וניתוח הביג-דאטה, הנדרשים כדי לפענח אינטראקציות של תרגום משותף ברזולוציה של כמעט קודון.
Introduction
Selective Ribosome Profiling (SeRP) היא השיטה היחידה, עד כה, הלוכדת ומאפיינת אינטראקציות תרגום משותף, in vivo, באופן ישיר 1,2,3,4,5,6. SeRP מאפשר פרופיל גלובלי של אינטראקציות של כל גורם עם תרגום ריבוזומים ברזולוציה של כמעט קודון 2,7.
השיטה מסתמכת על הקפאת הבזק של תאים גדלים ושמירה על תרגום פעיל. לאחר מכן, ליזטים של תאים מטופלים באמצעות RNase I כדי לעכל את כל ה-mRNA בתא, למעט שברי mRNA המוגנים על-ידי ריבוזומים המכונים "עקבות ריבוזומים". לאחר מכן מתפצלים המדגם לשני חלקים; חלק אחד משמש ישירות לבידוד של כל עקבות הריבוזומליות התאיות, המייצגות את כל התרגום המתמשך בתא. החלק השני משמש לטיהור זיקה של תת-קבוצה ספציפית של ריבוזומים הקשורים לגורם עניין, לדוגמה: שינוי אנזימים, גורמי טרנסלוקציה, מלווים מתקפלים ואינטראקציות של הרכבה מורכבת. עקבות הריבוזומליות המטוהרות זיקה נקראות באופן קולקטיבי האינטראקציה. לאחר מכן, ה-mRNA המוגנים בריבוזום מופקים ומשמשים ליצירת ספריית cDNA, ולאחר מכן לריצוף עמוק.
ניתוח השוואתי של סך כל דגימות התרגום והאינטראקציה מאפשר זיהוי של כל האורפים המתקשרים עם גורם העניין, כמו גם אפיון של כל פרופיל אינטראקציה של orf. פרופיל זה מדווח על רצפי ההתחלה והסיום המדויקים של ההתקשרות שמהם ניתן להסיק את הקודונים המפוענחים ואת השאריות המתאימות של שרשרת הפוליפפטידים המתהווה, כמו גם על שינויי מהירות הריבוזומים במהלך האינטראקציה 7,8. איור 1 מתאר את הפרוטוקול כשרטוט.
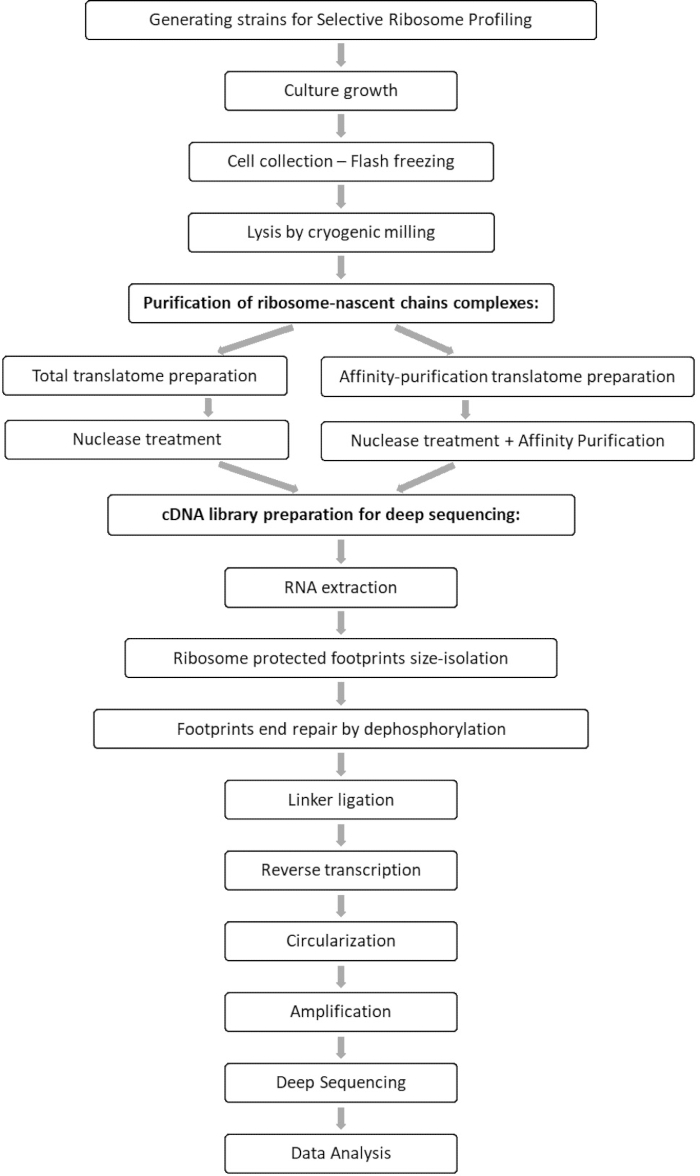
איור 1: סקירה כללית של פרוטוקול SeRP. פרוטוקול זה יכול להתבצע בשלמותו תוך 7-10 ימים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Protocol
1. יצירת זנים עבור פרופיל ריבוזום סלקטיבי
הערה: Selective Ribosome Profiling (SeRP) היא שיטה המסתמכת על טיהור זיקה של גורמים מעניינים, כדי להעריך את אופן האינטראקציה שלהם עם קומפלקסים של שרשרת ריבוזומים-מתהווים. רקומבינציה הומולוגית9, כמו גם שיטות מבוססות CRISPR/Cas910 משמשות כדי למזג גורמים שונים של עניין עם תגים לטיהור זיקה. תגים כאלה הם GFP, עבור טיהורי זיקה של מלכודת GFP, תג TAP עבור טיהורי חרוזי IgG-Sepharose וכן AVI-Tag המטוהר על ידי אבידין או סטרפטווידין, כדי למנות כמה דוגמאות מוצלחות מהשנים האחרונות.
- בצע בדיקות גדילה או בדיקות פונקציונליות כדי לאמת שהתיוג לא השפיע על תפקוד החלבונים. יש להעריך את התיוג הטרמינלי של N' לעומת C'.
הערה: הריבוזומים (rRNA), כמו גם תחומים רבים של קשירת ריבוזומים בגורמים שונים, הם טעונים מאוד, מה שהופך תגים טעונים מאוד (כגון polyhistidine) לבלתי ניתנים לשימוש, מכיוון שהם עלולים להוביל לגילוי שגוי או למצב קשירה שונה.
2. צמיחת תרבות
- טפחו את תרביות השמרים הבנויות (המבוססות על הזן BY4741), המכילות את החלבונים המתויגים הרצויים, במדיום עשיר בתמצית שמרים נוזלית-פפטון-דקסטרוז (YPD), או במדיום מינימלי של דקסטרוז סינתטי (SD) (1.7 גרם/ל' בסיס חנקן שמרים עם אמוניום סולפט או 1.7 גרם/ל' בסיס חנקן שמרים ללא אמוניום סולפט עם חומצה גלוטמית מונוסודיום 1 גרם/ל', 2% גלוקוז ותוספת לתערובת שלמה או מתאימה של חומצות אמינו).
- לגדל 250-500 מ"ל של תרבית תאים ל-0.5 OD600 (אמצע יומן), ב-30 מעלות צלזיוס, במדיום מתאים.
3. איסוף תאים וליזיס
- אספו במהירות תאים על ידי סינון ואקום על קרום כתם ניטרוצלולוז 0.45 מיקרומטר עם מערכת סינון זכוכית (מחזיק מסנן זכוכית עם משפך זכוכית 1 ליטר, בסיס ואקום וכובע, מסך נירוסטה, אטם ומהדק קפיץ, 90 מ"מ; בקבוק מפרק טחון 1 ליטר).
- הבזק להקפיא את התאים שנאספו, על ידי גירוד התאים הכדוריים עם מרית ומיד טבילתם בצינור 50 מ"ל מלא חנקן נוזלי.
נקודת עצירה: ניתן לאחסן את התאים בטמפרטורה של -80 מעלות צלזיוס למשך עד 3-4 שבועות. - בצע תזה של תאים על ידי שחיקה קריוגנית במפעל מיקסר: פעמיים במשך 2 דקות ב-30 הרץ, עם 1 מ"ל של מאגר הליזות (ראו טבלה 1). מצננים בחנקן נוזלי בין כרסומים.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 10 מ"ג/מ"ל CHX (ציקלוהקסימיד) | 220 | 0.5 מ"ג/מ"ל |
| 1M Tris-HCl pH 8.0 | 88 | 20 mM |
| 3M KCl | 205.7 | 140 mM |
| 1M MgCl2 | 26.4 | 6 מ"מ |
| 1M PMSF | 4.4 | 1 מ"מ |
| NP-40 | 4.4 | 0.10% |
| מעכב פרוטאז | 2 טבליות | |
| DNase I | 8.8 | 0.02 U/מ"ל |
| הכרך הסופי | 4,400 |
טבלה 1: מתכון לתערובת המאסטר של מאגר הליזיס.
הערה: ניתן לשנות את מאגר ה-Lysis כך שיכיל יותר מעכבי פרוטאזות (כגון בסטטין, לאופפטין, אפרוטינין וכו') במקרה שהחלבון המעניין מאוד לא יציב, אך חשוב להימנע מ-EDTA על מנת לשמור על תת-היחידות הקטנות והגדולות של הריבוזום שהורכבו במהלך השלבים הבאים. מסיבות דומות, תמיד לשמור על לפחות 6 mM MgCl2 בתמיסת המאגר.
אזהרה: HCl הוא מאוד קורוזיבי ו-PMSF רעיל. ללבוש כפפות וטפל בזהירות.
- צנטריפוגה במשך 2 דקות ב 30,000 x g, 4 °C כדי לנקות את lysate ולאסוף supernatant.
4. טיהור קומפלקסים של שרשראות ריבוזום-מתהוות עבור SeRP
- עבור כל ניסוי, חלקו את הסופרנטנט לשני חלקים; כל אחד בצינור מיקרו-סנטריפוג' אחר: דגימת RNA כוללת (~200 μL) ודגימת אימונופוריפיקציה (IP) (~700 μL) דגימות תרגום.
-
עיבוד דגימת הרנ"א הכוללת
- לעכל דגימת RNA כוללת באמצעות 10 U של RNase I במשך 25 דקות ב 4 °C ; סובב ב-30 סל"ד עם מתלה מיקס מסתובב.
הערה: ניתן לכייל את תנאי העיכול באמצעות פרופיל פוליאזום כדי להבטיח שלא יהיה שיא עיכול יתר או תת-עיכול של מונוזומים. - הכן את תערובת המאסטר של כרית הסוכרוז כמתואר בטבלה 2.
- לעכל דגימת RNA כוללת באמצעות 10 U של RNase I במשך 25 דקות ב 4 °C ; סובב ב-30 סל"ד עם מתלה מיקס מסתובב.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 50% סוכרוז | 200 | 25% |
| 1M Tris-HCl pH 8.0 | 8 | 20 mM |
| 3M KCl | 18.7 | 140 mM |
| 1M MgCl2 | 4 | 10 mM |
| 100 מ"ג/מ"ל CHX | 0.4 | 0.1 מ"ג/מ"ל |
| מעכב פרוטאז | 1 טבליה | |
| הכרך הסופי | 400 |
טבלה 2: מתכון לתערובת מאסטר כרית סוכרוז.
- טען את הדגימה על 400 μL של כרית הסוכרוז והצנטריפוגה ברוטור TLA120 ברוטור TLA120 למשך 90 דקות ב- 245,000 x g וב- 4 ° C.
- הסר את ה-supernatant במהירות באמצעות משאבת ואקום וכיסוי כדורי-על עם מאגר ליזיס של 150 μL. החיזרו את הכדורים על ידי טלטולים במשך שעה אחת ב-4 מעלות צלזיוס וב-300 סל"ד.
- בצעו החייאה של הכדור השיורי על ידי צנרת והעברתם לצינור חדש של 1.5 מ"ל.
הערה: 100-200 מיקרוגרם של רנ"א כולל מספיק בדרך כלל ליצירת פרופיל ריבוזומים של התרגום הכולל. ניתן להוסיף את שלב דלדול ה-rRNA על מנת להפחית את זיהום ה-rRNA, שהוא המזהם הנפוץ ביותר של קומפלקסים של שרשראות ריבוזום-מתהוות11 ( ראו דיון לפרטים נוספים).
-
עיבוד דגימת האימונופוריפיקציה
- לשטוף 100-400 μL של מטריצת קשירת זיקה (1:1 חרוזים מצומדים נוגדנים ב 70% EtOH) לכל דגימה עם 3 x 1 מ"ל lysis buffer (ללא DNase I ומעכבי פרוטאז); בצעו מחדש את מטריצת הזיקה במאגר הליזיס, ולאחר מכן סובבו ב-30 סל"ד עם מתלה תערובת מסתובב ב-4 מעלות צלזיוס למשך 5 דקות. זרז על ידי צנטריפוגה במשך 30 שניות ב 3,000 x g, 4 °C (4 °F). יש להשליך את הנוזל העליון. חזרו על הפעולה שלוש פעמים.
- לעכל דגימות אימונופוריפיקציה באמצעות 10 U ליחידת A260 ננומטר של RNase I, יחד עם מטריצת קשירת זיקה (לדוגמה, 100-400 μL של GFP-TRAP לכל דגימה).
- סובבו במשך 25 דקות ב-30 סל"ד עם מתלה תערובת מסתובב כדי לקשור את החלבון למטריצת הזיקה, ב-4 מעלות צלזיוס.
- הכינו את התערובת הראשית של מאגר הכביסה כמפורט בטבלה 3.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 10 מ"ג/מ"ל CHX | 50 | 0.1 מ"ג/מ"ל |
| 1M Tris-HCl pH 8.0 | 100 | 20 mM |
| 3M KCl | 233 | 140 mM |
| 1M MgCl2 | 50 | 10 mM |
| 1M PMSF | 5 | 1 מ"מ |
| NP-40 | 0.5 | 0.01% |
| מעכב פרוטאז | 2 טבליות | |
| 50% גליצרול | 1,000 | 10% |
| הכרך הסופי | 5,000 |
טבלה 3: מתכון לתערובת המאסטר של מאגר הכביסה.
- שטפו את מטריצת קשירת הזיקה שלוש פעמים עם 1 מ"ל של מאגר שטיפה, בכל פעם למשך כדקה אחת, תוך סיבוב בארון התקשורת של התערובת ב-30 סל"ד, ב-4 מעלות צלזיוס.
- לזרז על ידי צנטריפוגה ב 3,000 x g עבור 30 s ב 4 °C (64 °F). יש להשליך את הנוזל העליון.
- יש לשטוף פעמיים נוספות במאגר שטיפה של 1 מ"ל, בכל פעם למשך 5 דקות, תוך סיבוב במדף התערובת ב-30 סל"ד, ב-4 מעלות צלזיוס.
- לזרז על ידי צנטריפוגה במשך 30 שניות ב 3,000 x g ו 4 °C (74 °F).
- השתמש ב- 50 μL של חרוזים עבור elution חלבון עם אותה כמות של 2x מאגר דגימה. השתמש בשאר החרוזים למיצוי RNA.
- צנטריפוגה במשך 30 שניות ב-3,000 x גרם, 4 מעלות צלזיוס כדי להעיף את החרוזים ולהשליך את הנוזל העליון.
- להקפיא בחנקן נוזלי ולאחסן בטמפרטורה של -80 מעלות צלזיוס. השתמש בדגימות אלה לצורך מיצוי RNA לאחר מכן.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 מעלות צלזיוס למשך הלילה או יותר. זו יכולה להיות נקודת עצירה. - הערך את ההצלחה של שלב טיהור הזיקה על ידי כתם מערבי או צביעת Coomassie עם aliquots (~ 10% לפי נפח, לאחר ערבוב) של כל צעד. השתמש תמיד ב- IP מדומה על זן WT שאינו מתויג כבקרה עבור קשירה לא ספציפית למטריצת הזיקה.
הערה: ניתן להתגבר על רקע גבוה שאינו ספציפי על ידי שלבי שטיפה נוספים עם עלייה בריכוזי המלח/חומרי ניקוי. ניתן לייצב אינטראקציה חולפת על ידי טיפול בחומרים שונים של קישורים צולבים, לדוגמה, טיפול פרפורמלדהיד (PFA) בתאים חיים - הוספת 0.4%-1% PFA למדיית הגדילה למשך 2-5 דקות, ואחריה מרווה של גליצין (0.3 M) למשך 3 דקות, מומלצת מאוד.
אזהרה: פרפורמלדהיד הוא חשוד כמסרטן. מכיוון שהפרפורמלדהיד מתאדה במהירות והוא קורוזיבי, יש לעבוד על מכסה מנוע בטיחותי כימי וללבוש שתי שכבות של כפפות.
5. הכנת ספריית cDNA לריצוף עמוק
- מיצוי RNA
הערה: עבוד עם צינורות 1.5 מ"ל שאינם מקלים ללא RNase כדי למנוע דלדול אפשרי של RNA או DNA.- להפשיר את הדגימות משלבים 4.2.5 ו-4.3.12 על דגימות קרח והחייאה עם 10 mM Tris-HCl pH 7.0 לנפח סופי של 700 μL.
אזהרה: חומצה-פנול וכלורופורם הם נדיפים ומזיקים. עבודה במכסה בטיחות כימי. - הוסף 40 μL של 20% SDS ל 0.7 מ"ל סה"כ RNA או IP elutions. סגור והתהפך כמה פעמים. משקעי חלבון צריכים להפוך את הדגימות ללבנים.
- הוסיפו 0.75 מ"ל של חומצה-פנול:כלורופורם שחוממה מראש לדגימות. אטמו את הצינורות בחוזקה ורעדו במיקסר תרמי ב-1,400 סל"ד למשך 5 דקות וב-65 מעלות צלזיוס. מצננים דגימות על קרח במשך 5 דקות.
- צנטריפוגה הצינור משלב 5.1.3. ב 20,000 x g במשך 2 דקות. מעבירים את השכבה המימית העליונה לצינור טרי ומוסיפים לה 0.7 מ"ל של חומצה-פנול:כלורופורם.
- דגירה במשך 5 דקות בטמפרטורת החדר, מדי פעם מערבולת. צנטריפוגה למשך 2 דקות ב 20,000 x g. מעבירים את השכבה המימית העליונה לצינור טרי ומוסיפים לה 0.6 מ"ל כלורופורם ומערבולת.
- צנטריפוגה למשך דקה אחת ב 20,000 x g. מעבירים את השכבה המימית העליונה לצינור טרי.
- לזרז חומצות גרעין על ידי הוספת 78 μL של 3 M NaOAc, pH 5.5, 2 μL של GlycoBlue, ו 0.75 מ"ל של איזופרופנול. מערבולת ביסודיות במשך 5 דקות. דגירה למשך שעה אחת לפחות בטמפרטורה של -80 מעלות צלזיוס או 16 שעות בטמפרטורה של -20 מעלות צלזיוס.
- צנטריפוגה במשך 30 דקות ב 20,000 x g וב 4 °C ו להשליך את supernatant. לשטוף את הכדורים עם קר כקרח 0.75 מ"ל של 80% אתנול. הפוך את הצינורות לשטיפה יסודית. צנטריפוגה ב 20,000 x g במשך 5 דקות ב 4 ° C, ולאחר מכן להשליך את supernatant.
- סובבו כלפי מטה ב-450 x גרם, 4 מעלות צלזיוס במשך 20 שניות והסירו את האתנול הנותר והשליכו את הנוזלים. מייבשים את הכדור עם מכסה פתוח למשך 5 דקות בטמפרטורה של 65 מעלות צלזיוס. בצעו החייאה של הדגימות באופן הבא: עבור IP החייאת הדגימה ב-10 μL של 10 mM Tris-HCl, pH 7.0. עבור ניתוח תרגום כולל, החייאת הדגימה ב-20 μL של 10 mM Tris-HCl, pH 7.0.
נקודת עצירה: ניתן לאחסן RNA בטמפרטורה של -80 מעלות צלזיוס במשך חודשים.
- להפשיר את הדגימות משלבים 4.2.5 ו-4.3.12 על דגימות קרח והחייאה עם 10 mM Tris-HCl pH 7.0 לנפח סופי של 700 μL.
- לכמת את ריכוז הרנ"א הכולל על ידי פלואורומטריה
הערה: כל החומרים והמשטחים הבאים צריכים להיות ללא RNase בעת הכנת ספריית cDNA לריצוף הדור הבא. בעת הטיפול בדגימות RNA, יש ללבוש כפפות.- דילול 1 μL של RNA כולל המופק על ידי חומצה פנול ב-9 μL של 10 mM Tris-HCl, pH 7.0. לכמת באמצעות פלואורומטר, כפי שהונחה באתר היצרן.
- דיללו את הדגימות המכילות 50 מיקרוגרם של רנ"א עם 10 מיקרול של 10 mM Tris-HCl, pH 7.0.
הערה: אל תמדוד דגימות IP, השתמש בכל דבר לשלב הבא.
- שברי טביעת רגל מוגנים של ריבוזום ג'ל-טיהור
- הגדר ג'ל פוליאקרילאמיד TBE-urea 15% ושקע במאגר ריצה TBE 1x. הפעל במשך 30 דקות ב- 200 V לפני טעינת הדגימה. לכל דגימה, הוסף 20 μL של מאגר דגימות TBE-urea 2x.
הערה: גודל הרצועה הצפוי הוא בסביבות 25-35 nt. - להפשיר סולם דנ"א של 10 bp ודגימות דנטורציה (לא סולם) בטמפרטורה של 80 מעלות צלזיוס למשך 2 דקות, ולאחר מכן להצטנן על קרח. טען כל דגימה על כל נתיב אחר. הפעל את הג'ל במשך 50-70 דקות ב 200 V.
- דילול 6 μL של זהב SYBR (10,000 x תרכיז) ב-60 מ"ל של 1x TBE חיץ וכתם תוך כדי רעד בקופסאות מוגנות אור למשך 15-20 דקות. בעת צביעת הג'ל, הכינו אזמל סטרילי וצינורות שוברי ג'ל 0.5 מ"ל בצינורות המסומנים ב-1.5 מ"ל.
- הוציאו את הרצועות הרצויות עם אזמל סטרילי (השתמשו באזמל טרי או נקו היטב בין דגימות) והניחו כל חתיכת ג'ל בצינור שובר ג'ל.
- צלם תמונה של הג'ל כדי לוודא שלא נותרו שאריות דגימה בג'ל.
- צנטריפוגות הצינורות המכילים פרוסות חתוכות ב 20,000 x g במשך 5 דקות ב 4 ° C ולהעביר את חתיכות הג'ל הנותרות מן צינור שובר ג'ל לצינור 1.5 מ"ל.
- הוסף 0.5 מ"ל של 10 mM Tris, pH 7.0. יש לנער במיקסר תרמי ב-1,400 סל"ד למשך 10 דקות ב-70 מעלות צלזיוס.
- מעבירים לעמוד אצטט תאית עם קצה פיפטה רחב, וצנטריפוגה ב-20,000 x גרם למשך 3 דקות ב-4 מעלות צלזיוס.
- מעבירים את הזרימה לצינור חדש של 1.5 מ"ל וצוננים על קרח.
- על מנת לזרז את חומצות הגרעין, להוסיף: 550 μL של IPA, 55 μL של 3 M NaOAc, ו 2 μL של GlycoBlue ומערבולת לערבב ביסודיות. הניחו את הדגימות בטמפרטורה של -80 מעלות צלזיוס למשך שעה אחת לפחות.
- צנטריפוגה במשך שעה אחת לפחות ב-20,000 x גרם ו-4 מעלות צלזיוס והשליכו את ה-supernatant. לשטוף את הכדורים עם 0.75 מ"ל של קר כקרח 80% אתנול. הופכים את הצינורות לשטיפה יסודית עד שהכדורים נפרדים מהתחתית. צנטריפוגה שוב ב 20,000 x g במשך 5 דקות ב 4 ° C ולהשליך את supernatant.
- סובבו כלפי מטה ב-450 x גרם, 4 מעלות צלזיוס במשך 20 שניות והסירו את האתנול הנותר. מייבשים את הכדורים עם מכסה פתוח למשך 5 דקות בטמפרטורה של 65 מעלות צלזיוס.
- הוסיפו 15 μL של 10 mM Tris, pH 7.0 והחזירו את הכדורים ביסודיות. סובב מטה ב 450 x g, 4 ° C עבור 20 שניות ולהעביר את הדגימה לצינור חדש 1.5 מ"ל.
נקודת עצירה: ניתן לאחסן RNA מטוהר בטמפרטורה של -80 מעלות צלזיוס למשך מספר חודשים.
- הגדר ג'ל פוליאקרילאמיד TBE-urea 15% ושקע במאגר ריצה TBE 1x. הפעל במשך 30 דקות ב- 200 V לפני טעינת הדגימה. לכל דגימה, הוסף 20 μL של מאגר דגימות TBE-urea 2x.
- דפוספורילציה
- השתמש ב- 3 μL של התערובת הבאה עבור כל דגימה: הוסף 1 μL של מעכב RNase לתוך 2 μL של 10x T4 מאגר תגובת פולינוקלאוטיד קינאז ללא ATP. הוסף 2 μL של T4 פולינוקלאוטיד קינאז לכל דגימה. פיפט בעדינות כדי לערבב היטב ולדגום ב 37 °C (74 °F) במשך 2 שעות, מבלי לרעוד.
- כדי להשבית את האנזים, לדגום את הדגימה בטמפרטורה של 75 מעלות צלזיוס למשך 10 דקות ולהסתחרר כלפי מטה ב-450 x גרם, 4 מעלות צלזיוס, במשך 20 שניות. הוסף 0.5 מ"ל של 10 mM Tris, pH 7.0.
- כדי לזרז חומצת גרעין, הוסיפו 2 μL של GlycoBlue, 550 μL של IPA ו-55 μL של 3 M NaOAc.
- מערבולת כדי לערבב ביסודיות ולצנן את הדגימות ב -80 מעלות צלזיוס למשך שעה אחת לפחות.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 מעלות צלזיוס למשך הלילה או יותר. - חזור על שלבים 5.3.11-5.3.13.
נקודת עצירה: ניתן לאחסן RNA דה-פוספורילט בטמפרטורה של -80 מעלות צלזיוס במשך חודשים.
- כימות באמצעות ביואנליזר
- בצע דילול של 1:4 של כל דגימת RNA על ידי ערבוב של 1 μL של דגימה ו-4 μL של מים שטופלו ב-DEPC.
אזהרה: DEPC הוא מסרטן. ללבוש כפפות ולעבוד בזהירות. - הפעל שבב RNA קטן של ביואנלייזר / טייפסטיישן. עקוב אחר פרוטוקול היצרן.
הערה: גודל שבר הרנ"א הצפוי המוגן בריבוזומים הוא בסביבות 28-30 nt.
- בצע דילול של 1:4 של כל דגימת RNA על ידי ערבוב של 1 μL של דגימה ו-4 μL של מים שטופלו ב-DEPC.
- סיום ליגייט 3' עם לינקר-1
- דילול 5 pmol של שברי RNA קטנים ל-10 μL עם 10 mM Tris, pH 7.0. דנטורציה של דגימות בטמפרטורה של 80 מעלות צלזיוס למשך 2 דקות וקירור על קרח.
- הכן את התערובת הראשית כמפורט בטבלה 4 והשתמש ב- 29 μL לכל דגימה.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 50% PEG 8000 מסונן סטרילי | 16 | 20% |
| DMSO | 4 | 10% |
| 10× T4 RNA ליגאז 2 חיץ | 4 | 1x |
| מעכב SUPERase-in RNase | 2 | 2 U |
| 10 mM adenylated linker 3-L1 | 0.1 | 25 מיקרומטר |
| מים שטופלו ב-DEPC | 2.9 | |
| הכרך הסופי | 29 |
טבלה 4: מתכון לתערובת מאסטר של קשירת סוף 3 אינץ'.
- מוסיפים 1 μL של T4 RNA ליגאז 2 ופיפט בעדינות כדי לערבב היטב. אינקובציה בטמפרטורה של 23 מעלות צלזיוס למשך 2 שעות.
- על מנת לזרז את חומצות הגרעין, הוסף: 550 μL של IPA, 500 μL של 10 mM Tris, pH 7.0, 55 μL של 3 M NaOAc, ו 2 μL של GlycoBlue. מערבולת כדי לערבב ביסודיות, ולמקם את הדגימות על -80 מעלות צלזיוס למשך שעה אחת לפחות.
נקודת עצירה: אחסן את הדגימות בטמפרטורה של -80 מעלות צלזיוס למשך הלילה או יותר. - חזור על שלבים 5.3.2-5.3.12.
- בצעו החייאה של הכדור ב-6 מיקרו-ל' של 10 מ"מ טריס, עמ' 7.0. סובבו כלפי מטה ב-450 x גרם, 4 מעלות צלזיוס למשך 20 שניות, והעבירו את הדגימה לצינור חדש של 1.5 מ"ל.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 °C (80 °F) למשך חודשים.
-
טיהור ג'ל של עקבות מקושרות 3'
- הגדר ג'ל פוליאקרילאמיד TBE-urea 10% ושקע במאגר ריצה TBE 1x. הפעל במשך 30 דקות ב- 200 V לפני טעינת הדגימה. לכל דגימה, הוסף 6 μL של מאגר דגימות TBE-urea 2x.
הערה: גודל הפס הצפוי הוא בסביבות 71-73 nt. - חזור על שלבים 5.3.2-5.3.13.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 °C (80 °F) למשך חודשים.
- הגדר ג'ל פוליאקרילאמיד TBE-urea 10% ושקע במאגר ריצה TBE 1x. הפעל במשך 30 דקות ב- 200 V לפני טעינת הדגימה. לכל דגימה, הוסף 6 μL של מאגר דגימות TBE-urea 2x.
-
הפוך תעתיק 3ʹ מקטעי טביעת רגל מקושרים ליצירת ssDNA
- הכינו תמהיל ראשי כמפורט בטבלה 5 והשתמשו ב-3 μL לכל מדגם.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 10 mM dNTPs | 1 | 0.5 מ"מ |
| 25 μM Linker L(rt) | 0.5 | 625 ננומטר |
| מים שטופלו ב-DEPC | 1.5 | |
| הכרך הסופי | 3 |
טבלה 5: מתכון לתערובת הראשית של מאגר השעתוק ההפוך לפני דנטורציה של חומצות גרעין.
- מערבולת ולסובב את הדגימה.
- דגימות דגירה בטמפרטורה של 65 מעלות צלזיוס למשך 5 דקות.
- מצננים דגימות על קרח.
- הכן תערובת ראשית כמפורט בטבלה 6 והשתמש ב- 6 μL לכל מדגם. מערבולת ולסובב את הדגימה. הוסיפו 1 μL של כתב עילי III לכל דגימה ופיפט בעדינות כדי לערבב היטב ולדגום היטב למשך 30 דקות בטמפרטורה של 50 מעלות צלזיוס.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 5× מאגר FS | 4 | 1x |
| מעכב SUPERase-in RNase | 1 | 2 U |
| DTT 0.1 M | 1 | 5 מ"מ |
| הכרך הסופי | 6 |
טבלה 6: מתכון לתערובת הראשית של מאגר השעתוק ההפוך לאחר דנטורציה של חומצות גרעין.
- הוסיפו 2.3 μL של 1 N NaOH, אשר עושה הידרוליזה של RNA ומרווה את השעתוק ההפוך.
אזהרה: NaOH הוא מאוד קורוזיבי. לבשו כפפות והגנת עיניים. - דגירה למשך 15 דקות בטמפרטורה של 95 מעלות צלזיוס, עד שהמדגם הופך ורוד.
- הגדר ג'ל פוליאקרילאמיד TBE-urea 10% ושקע במאגר ריצה TBE 1x. הפעל במשך 30 דקות ב- 200 V לפני טעינת הדגימה. לכל דגימה, הוסף 23 μL של מאגר דגימות TBE-urea 2x.
הערה: גודל רצועת הדנ"א הצפוי הוא 115-117 nt. - חזור על שלבים 5.3.2-5.3.12.
- בצעו החייאה של הכדור ב-15 מיקרול'ל של 10 mM Tris, pH 8.0. סובב מטה ב 450 x g, 4 ° C עבור 20 שניות ולהעביר את הדגימה לצינור חדש 1.5 מ"ל.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 °C (80 °F) למשך חודשים.
-
מעגליות ssDNA
- הכן את תערובת המאסטר הבאה וטען 4 μL לכל דגימה, כמפורט בטבלה 7.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| 10× חיץ CircLigase II | 2 | 1x |
| 5 M בטאין (אופציונלי) | 1 | 0.25 מ' |
| 50 mM MnCl2 | 1 | 2.5 מ"מ |
| הכרך הסופי | 4 |
טבלה 7: מתכון לתערובת מאסטר של מעגליות ssDNA .
- הוסף 1 μL של CircLigase II ssDNA ליגאז לכל דגימה ודגירה במשך שעה אחת ב 60 °C (60 °F).
הערה: ניתן להגדיל את היעילות של שלב זה על ידי הוספת 1 μL של CircLigase II ssDNA ליגאז לכל דגימה לאחר דגירה של שעה אחת. - להשבית את האנזים על ידי דגירה ב 80 מעלות צלזיוס במשך 10 דקות.
- צוננים על הקרח וממשיכים להגברת PCR או מאחסנים בטמפרטורה של -80 מעלות צלזיוס.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 מעלות צלזיוס במשך שנים.
-
הגברה PCR
- הכן את תערובת האב הבאה של PCR וטען 82 μL לכל דגימה, כמפורט בטבלה 8.
| מגיב | כמות לדוגמה (μL) | ריכוז סופי |
| מים שטופלו ב-DEPC | 61.6 | |
| 5× מאגר התגובה של Phusion HF | 17.6 | 1x |
| 10 mM dNTPs | 1.8 | 200 מיקרומטר |
| 100 μM PCR פריימר קדימה | 0.2 | 225 ננומטר |
| HF Phusion פולימראז | 0.8 | 1.6 U |
| הכרך הסופי | 82 |
טבלה 8: מתכון לתערובת מאסטר להגברת PCR.
- לכל צינור המכיל תערובת מאסטר, הוסיפו 5 μL של דנ"א מעגלי.
הערה: אחסן את שאר דגימות הדנ"א המעגליות בטמפרטורה של -80 °C (80 °F). - הוסף פריימר ברקוד הפוך של 1 μL של 20 μM PCR לכל דגימה (ראה טבלה 9) ומערבולת כדי לערבב ביסודיות.
- Aliquot כל צינור לתוך ארבעה צינורות PCR נפרדים, כל אחד ישמש עבור מספר שונה של מחזורי PCR.
- הפעל תגובת PCR בהתאם לתוכנית הבאה, כמפורט בטבלה 10.
| מחזור | דנטורציה (98 °C) | חישול (60 °C) | הרחבה (72 °C) |
| 1 | 30 שניות | ||
| 2-16 | 10 שניות | 10 שניות | 5 שניות |
טבלה 10: תוכנית PCR לתגובת PCR.
- לאחר מחזורים 8, 9, 10 ו-11 מסירים צינורות PCR (עבור דגימות IP, המחזורים נעים בין 9 ל-15) כניסיון ראשון. לאחר כל מחזור, השהו את התוכנית, הוציאו אליקו אחד והניחו אותו על הקרח, ואז חידשו במהירות את התוכנית.
הערה: יש להתאים את מספר המחזורים בהתבסס על כמות הדנ"א המעגלי בכל תגובה. עיין באיור 4 לקבלת דוגמה והבהרה נוספת. - לכל תגובה של 17 μL, הוסיפו 3.5 μL של 6x צבע טעינת DNA.
- להפשיר סולם דנ"א של 10 bp.
- להפרדת גודל על ידי ג'ל-אלקטרופורזה, הטביעו 8% פוליאקרילאמיד TBE ב-1x TBE וטענו את הדגימות של כל מספר מחזור שונה לבארות סמוכות והריצו את הג'ל במשך 50 דקות ב-180 וולט.
- דילול 6 μL של זהב SYBR (10,000 x תרכיז) ב-60 מ"ל של 1x TBE חיץ וכתם תוך כדי רעד בקופסאות מוגנות אור למשך 15-20 דקות.
- בעת צביעת הג'ל, הכינו אזמל סטרילי וצינורות שוברי ג'ל 0.5 מ"ל בצינורות המסומנים ב-1.5 מ"ל.
- צלם תמונה של חומצות הגרעין המוכתמות.
- חותכים את הרצועה הרצויה עם גודל פס צפוי של 174-176 כ"ס עם האזמל הסטרילי ומניחים את פרוסת הג'ל בצינור מפסק הג'ל המוכן 0.5 מ"ל (נקו היטב בין דגימות והשתמשו בחומר ההשבתה RNase, או עברו ללהב חדש).
- צנטריפוגה של הצינורות במשך 5 דקות ב-20,000 x g ו-4°C, ולאחר מכן העבר את חלקי הג'ל הנותרים מצינור שובר הג'ל 0.5 מ"ל לצינור 1.5 מ"ל.
- הוסף 500 μL של 10 mM Tris, pH 8.0 ולנער במיקסר תרמי ב 1,400 סל"ד למשך 10 דקות ו 70 ° C.
- מעבירים את הג'ל המומס לעמוד אצטט תאית עם קצה פיפטה רחב.
- צנטריפוגה של העמודה במשך 3 דקות ב-20,000 x g ו-4°C והעבירה את הזרימה לצינור חדש של 1.5 מ"ל וצינון על קרח.
- כדי לזרז את חומצת הגרעין, הוסף: 550 μL של IPA, 32 μL של 5 M NaCl, 1 μL של 0.5 M EDTA, ו 2 μL של GlycoBlue ומערבולת כדי לערבב ביסודיות.
- שמור את הדגימות לפחות שעה אחת ב -80 °C (80 °F), או -20 °C (80 °F) במהלך הלילה.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 מעלות צלזיוס למשך הלילה או יותר. - חזור על שלבים 5.3.11-5.3.12.
- Resuspend ב 11 μL של 10 mM Tris, pH 8.0. סובב מטה ב 450 x g, 4 ° C עבור 20 שניות ולהעביר את הדגימה לצינור חדש 1.5 מ"ל.
נקודת עצירה: ניתן לאחסן דגימות בטמפרטורה של -80 מעלות צלזיוס במשך שנים.
-
כימות התפלגות הגודל על ידי Bioanalyzer
- בצע דילול של 1:4 של כל דגימה על ידי ערבוב של 1 μL של דגימה עם 4 μL של מים שטופלו ב- DEPC.
- הפעל את שבב ה-RNA הקטן של Bioanalyzer. עקוב אחר פרוטוקול היצרן.
הערה: האורך הצפוי הוא 175 ± 5 bp.
-
לכמת את ריכוז הדנ"א על ידי פלואורומטר
- בצע בדיקת ריכוז dsDNA ברגישות גבוהה עם פלואורומטר בהתאם להמלצות היצרן.
- ריבוי דוגמאות ורצף על פי המלצות אילומינה (מדריך איגום מתאמי אינדקס12).
6. ניתוח נתונים
- בצע ניתוח כמפורט בקובץ המשלים.
תוצאות
כפי שמודגם בתרשים הזרימה של פרוטוקול זה (איור 1), התאים גודלו לפאזה של רישום, ולאחר מכן נאספו במהירות על ידי סינון ונשחקו על ידי שחיקה קריוגנית. לאחר מכן חולק הליסאט לשניים: האחד עבור עקבות mRNA כוללות המוגנות על-ידי ריבוזומים והשני עבור עקבות mRNA נבחרות המוגנות על-ידי ריבוזומים, שבהן ביצענו טיהור זיקה כדי למשוך מטה את קומפלקסי השרשראות המתהוות של חלבון-ריבוזום-מתהווה. הבטחנו את ביטוי החלבון המתויג ואת הצלחת המשיכה כלפי מטה על ידי ניתוח כתמים מערבי, כפי שניתן לראות באיור 2. אימתנו את הבידוד של עקבות מוגנות ריבוזומים, שבדרך כלל הן באורך של 20-45 nt על ידי אלקטרופורזה קטנה של RNA (מערכת BioAnalyzer 2100), מה שמאפשר שינוי גודל של 5-10 nt, על פי מדריך המערכת (איור 3). לאחר מכן, יצרנו ספריית cDNA לריצוף עמוק וניתוח ביג דאטה. בעת יצירת ספריית cDNA, שימו לב שתת-מחזוריות יכולה להוביל לתפוקה נמוכה (כפי שניתן לראות בנתיב 2 באיור 3), אך הגברה מחדש אפשרית כדי לשחזר את הספרייה שנוצרה. רכיבה על אופניים יתר עלולה להתרחש כאשר פריימרים PCR מתרוקנים אך התגובה נמשכת. כאשר dNTPs עדיין קיימים, התגובה ממשיכה, ומייצרת ממצאי PCR ארוכים יותר עם רצפים כימריים עקב מוצרי PCR המגדירים את עצמם13 (כפי שניתן לראות בנתיבים 3-4 באיור 3, המסומן על ידי המריחה הנראית לעין). אם גם הריכוז של ה-dNTPs הופך להיות מגביל, יכולים להופיע מוצרים המעידים על נוכחות של הטרודופלקסים המורכבים מקטעי ספרייה הומולוגיים חלקיים בלבד. איור 4 משמש כאסמכתא, כאשר נתיב 2 מייצג הגברה אופטימלית, ונתיב 3 הוא הגברה מקובלת. אין להשתמש בדגימות מנתיבים 4 ו-5 (מחזורים 10 ו-11) בשל האפשרות להציג כפילויות וממצאים של PCR. הספרייה שנוצרה אומתה עוד יותר על ידי אלקטרופורזה של דנ"א ברגישות גבוהה (אותה מערכת BioAnalyzer שימשה) לצורך התפלגות גודל וכימות מדויקים (איור 5). לאחר קשירת קישור קצה של 3', שעתוק הפוך והגברת PCR, צפויה התפלגות אורך cDNA ככזו, עם שיא חד סביב 175 nt.
קיצצנו והסרנו מתאמים וברקודים מהספרייה הרצופה, ורק הקריאות בין 20 ל-45 nt נבחרו לניתוח נוסף. איור 6 מציג את התפלגות האורך המתקבלת. הקריאות חולקו לקבוצות שונות של: רצפי קידוד, אינטרונים ורצפים אינטרגניים (איור 7), וסווגו עוד יותר כפי שמוצג באיור 8.
ניתוח סופי לאיתור ואפיון של אינטראקציות תרגום משותף בוצע על סמך העשרת שברי mRNA המוגנים על ידי ריבוזומים, והפיק את הגרפים באיור 9. השווינו את תפוסת הריבוזומים המנורמלת (בכל נוקלאוטיד לאורך כל אורף) של התרגום הכולל לתרגום הנבחר המתאים לו (nascent-interactome). לכל השוואת נוקלאוטידים מבטלת את שיעורי התרגום. יכולת השכפול בין שכפול ביולוגי הוערכה על ידי מתאם פירסון (סף > 0.6). אנו מציגים פרופילי ריבוזומים סלקטיביים, המנתחים אינטראקציות תרגומיות משותפות של Vma2p עם שלושה חלבונים: המלווה הקשור לריבוזום Ssb1p, Pfk2p (Phosphofructokinase) ו- Fas1p (חומצת שומן סינתאז), כאשר כל חלבון C' מתויג באופן סופי על ידי GFP. ביצענו את הפרוטוקול בשכפולים ביולוגיים. תרשים 9 A, D ו- G מראים את הסכימה הניסויית של כל טיהור זיקה. לאחר מכן אנו מראים את תפוסת הריבוזומים של סך התרגומים בהשוואה לאינטראקציות Ssb1 לאורך ה-Vma2p orf, המקודדות עבור תת-יחידה של ה-vacuolar H+-ATPase (איור 9 B, E ו-H). לבסוף, ביצענו פרופיל העשרת ריבוזומים מבוסס יחס (IP/Total) בכל מיקום ריבוזום ב-[codon/aa] לאורך ה-orf (איור 9 C, F ו-I). השוואת האינטראקציות התרגומיות המשותפות של שלושת החלבונים הללו עם Vma2p, המסונתז על ידי הריבוזום, גילתה כי מלווה Ssb1 מפעיל את ה-Vma2p המתהווה בארבעה אזורים שונים לאורך האורף, כפי שזיהינו ארבעה שיאי העשרה משמעותיים על ידי SeRP. באופן שונה, Pfk2p מראה רק שיא העשרה משמעותי אחד, כפי שזוהה על ידי SeRP, בעמדה שונה בהשוואה למלווה התרגום המשותף Ssb1. ניתוח של אינטראקציות התרגום המשותף של Fas1 עם Vma2p המתהווה לא זיהה העשרה משמעותית. לפיכך, ההשוואה בין פרופילי ריבוזום העשרה מבוססי יחס אלה מדגימה את כוחו של פרוטוקול זה בזיהוי ואפיון של אינטראקציות תרגום משותפות שונות ברזולוציה כמעט קודונית.
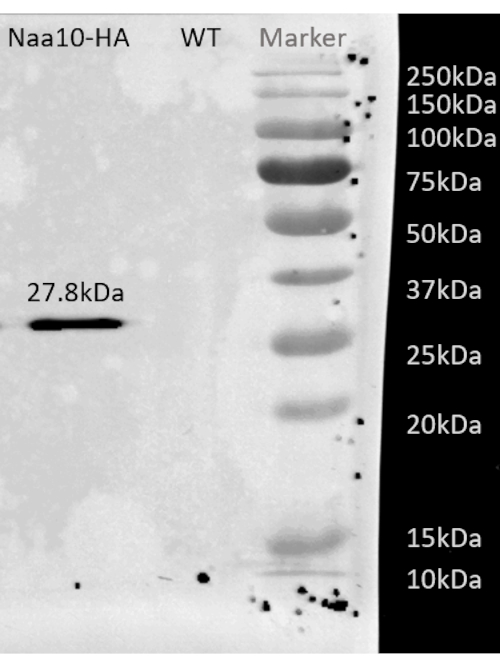
איור 2: תוצאה מייצגת של כתם מערבי לאחר טיהור זיקה של זן BY4741 עם Naa10 המתויג כ-HA. תוצאת כתם מערבי מייצגת לאחר טיהור זיקה של זן BY4741 עם Naa10 מתויג HA המציג רצועה סביב 27.8 kDa, בעוד שהסוג הפראי, כבקרה שלילית, אינו מראה רצועה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 3: תוצאת BioAnalyzer מייצגת לאחר בידוד טביעת רגל ומיצוי RNA עם חומצה-פנול:כלורופורם, וגודל ממוצע של 25 nt. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
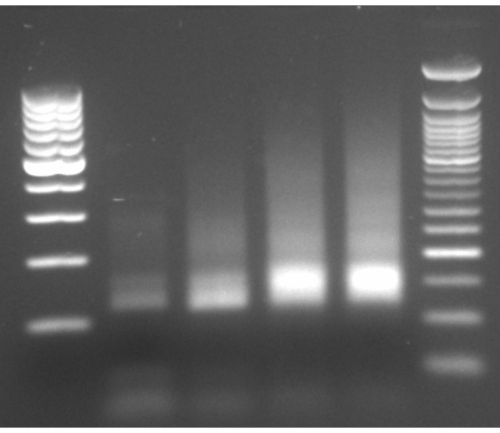
איור 4: אלקטרופורזה מייצגת של ג'ל-אלקטרופורזה של PCR. ג'ל-אלקטרופורזה מייצגת של הגברת PCR עם נתיבים 2-5 עמוסים במוצרי PCR ממחזורים 8-11, וסולמות משני הצדדים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
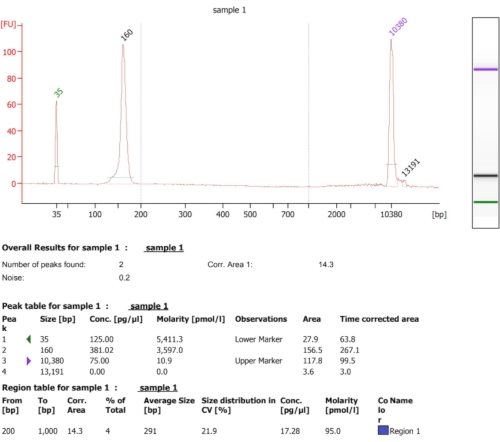
איור 5: תוצאת BioAnalyzer מייצגת שהתקבלה בעקבות יצירת ספריית cDNA. לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 6: התפלגות האורך הצפויה של הקריאות לאחר הסרת המתאמים עם Cutadapt (הסרת קריאות קצרות מ-20 או יותר מ-45). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 7: אחוזי ההצלחה הצפויים ביישור לאחר הסרת קריאות RNA שאינן מקודדות עם Bowtie2 ושימוש ב-TopHat כדי ליישר את הקריאות הנותרות לאורגניזמים שונים. הדגימה נלקחה מ- S. cerevisiae (גרסה מוטציה של BY4741). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
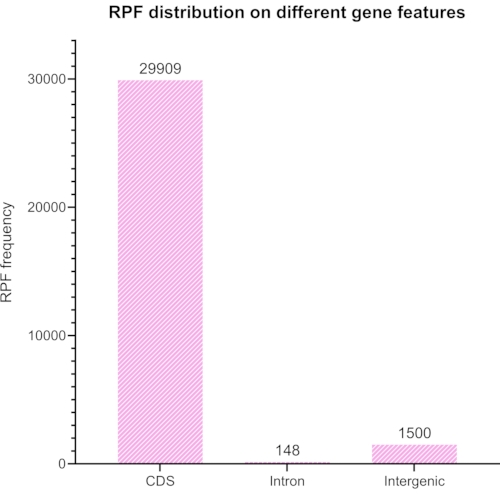
איור 8: גרף שנוצר עם RiboToolkit המייצג קידוד צפוי לעומת יחס שאינו מקודד של קריאות מיושרות לאחר שימוש ב-Bowtie2 כדי להסיר רכיבי rRNA בקריאות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
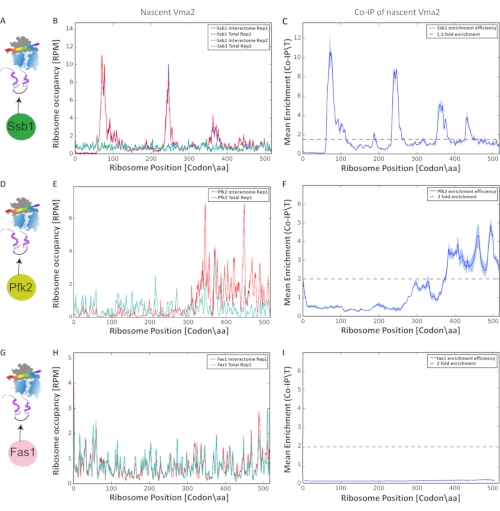
איור 9: אינטראקציות תרגום משותפות של שלושה חלבונים שונים: Ssb1p, Pfk2p ו-Fas1p עם Vma2p, אשר מסונתז על-ידי הריבוזום, מנותח על-ידי SeRP. כל צירי ה-y מוצגים בקריאה למיליון (סל"ד) קריאות. (א, ד, ז) סכימה ניסיונית של SeRP של Ssb1p, Pfk2p ו- Fas1p C' מתויגת באופן סופי על ידי GFP, בהתאמה. (ב, ה, ח) תפוסת ריבוזום לאורך ה-orf של סך התרגומים בהשוואה לאינטראקציות Ssb1, Pfk2p ו-Fas1p, בהתאמה (בשכפולים ביולוגיים). (ג, פ, א) העשרה ממוצעת של Ssb1p, Pfk2p ו-Fas1p (יחס IP/Total) בכל מיקום ריבוזום ב-[קודון/aa] לאורך ה-orf, בהתאמה. השונות בין השכפולים הביולוגיים מסומנת על ידי האזור המוצל. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
טבלה 9: 3' רצפי קישור ופריימר. 3' Linker L1: Linker 3-L1 עם אדנילציה של 5ʹ ו-3ʹ דידאוקסי-ציטידין מזהים מולקולריים ייחודיים ('NN...') (טיהור HPLC ללא RNase; מקשר תעתיק הפוך: שעתוק הפוך (L(rt)) עם 5ʹ זרחן, מזהים מולקולריים ייחודיים (טיהור HPLC ללא RNase); פריימר קדמי PCR: PCRf; HPLC מטוהר. אנא לחץ כאן כדי להוריד טבלה זו.
קובץ משלים. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
כאן, הפרוטוקול מפרט את גישת פרופיל הריבוזומים הסלקטיבית ללכידת אינטראקציות של תרגום משותף ברזולוציה של כמעט קודון. ככל שהריבוזום עולה כמרכז לתיאום הופעת השרשרת המתהווה לתוך הציטופלסמה הצפופה, זוהי שיטה חיונית לזיהוי ואפיון האינטראקציות השונות של התרגום המשותף הנדרשות כדי להבטיח פרוטאום תפקודי, כמו גם לחקר מחלות שונות. נכון להיום, SeRP היא השיטה היחידה שיכולה ללכוד ולאפיין את האינטראקציות הללו, באופן ישיר, in vivo 14,15,16.
השלב הראשון והקריטי ביותר הוא איסוף תאים וליזיס. חובה ללכוד, תוך שניות, תרגום מתמשך, על ידי הקפאת הבזק ואחריה תזה במצב קפוא. איסוף התאים חייב להיעשות בחיפזון על מנת למנוע נגר ריבוזומלי וכן לגרום לתגובות תרגום מתח, שיכולות להתרחש במהירות. השלב הקריטי השני הוא שלב טיהור הזיקה. זה הכרחי כדי להפחית את קשירת הרקע על ידי שטיפה קפדנית תוך הקפדה על אינטראקציות תרגום משותף נשמרות, אשר ניתן להקל על ידי in vivo cross-linking. מכיוון שפרוטוקול זה מבוסס על הרקע הגבוה הרגיש ביותר של NGS (ריצוף הדור הבא) בשלבים הראשונים ניתן להגביר בשלבי ההכנה הבאים של ספריית cDNA, מה שמוביל ליחסי אות לרעש נמוכים.
יש להעריך את הטיפול בנוקלאז, כדי לעכל את כל ה-mRNA שאינם מוגנים על ידי פרופיל פוליאזום17 יחד עם הערכה זהירה של התפלגות גודל העקבות הריבוזומליות המבודדות (כמפורט לעיל) כדי למנוע עיכול RNA מעל או תחת. כיול ריכוז הנוקלאז וזמני העיכול יכול להקל על התאוששות מדויקת של טביעת הרגל, שכן עיכול יתר עלול להוביל לעיכול rRNA ריבוזומלי, מה שיוביל לאובדן עקבות מוגנות ריבוזום. חשוב לציין שתת-עיכול יכול גם להוביל לשיעורי גילוי נמוכים יותר של עקבות מוגנות ריבוזומים, שכן שלבי ההכנה של ספריית cDNA, כמו גם שלבי ניתוח הנתונים המתוארים כאן, מבטלים קריאות ארוכות ולא אופייניות.
בעוד שדלדול rRNA לא תמיד מהווה צעד קריטי ואינו חובה, יש לו כמה יתרונות כגון דגימות נקיות יותר, ולכן שיעור גבוה יותר של קריאות ממופות גנום. מצד שני, קיימת אפשרות של הטיות, שכן פרוטוקולים רבים של דלדול rRNA עלולים גם לגרום לדלדול של השברים המוגנים בריבוזומים הרצויים. יש לקחת בחשבון גם את העלויות של ערכות דלדול ה-rRNA. ניתן לבצע דלדול rRNA לאחר שלב הבידוד של הדפסת ריבוזום-רגל או לאחר שלב העגליזציה של cDNA.
שלבי ההכנה של ספריית cDNA כמתואר כאן, הותאמו לקלט mRNA נמוך, מכיוון ששלבי הזיקה וטיהור הריבוזומים מפחיתים מאוד את כמות קלט ה-mRNA, בהשוואה למחקרי ביטוי RNA-seq. הגדלת הכמות הראשונית של תרביות תאים יכולה להקל מאוד על יצירת ספריית cDNA. לחלופין, כל פרוטוקול בחירת ספריית cDNA יכול להתאים לשלבי טיהור הזיקה ובידוד טביעת הרגל המתוארים כאן. חשוב לציין כי הטיפול בנוקלאז המייצר את טביעות הרגל הריבוזומליות דורש את תיקון קצוות ה-mRNA שנוצר כתוצאה מכך (הכנת ספריית cDNA, שלב Dephosphorylation) כדי לאפשר ביצוע שלבי קשירת קישור בפרוטוקול cDNA המתואר כאן בפרוטוקול שבחרת.
במהלך הריצוף, חשוב להבדיל בין SeRP ל-RNA-Seq, שכן ההטרוגניות של הספריות שנוצרו משתנה מאוד, בהתאם לגורמי הזיקה המתויגים. מלווים מולקולריים וגורמי מיקוד הם לעתים קרובות מופקרים יותר בקשירתם, ומקיימים אינטראקציה עם מאות או אלפי מצעים במהלך התרגום, מה שמוביל לספריות cDNA מגוונות ביותר. עם זאת, אינטראקציות ספציפיות מאוד, כגון אינטראקציות הרכבה מורכבות של תרגום משותף, יכולות לעתים קרובות להוביל ליצירת ספריות cDNA הרבה פחות מגוונות. עלייה חדה בספריות מגוונות ולא מגוונות באותו נתיב יכולה לשפר מאוד את הריצוף ואת תוצאות ניתוח הנתונים.
תכונה ייחודית נוספת של SeRP היא היכולת שלו ללכוד וריאציות מקומיות בתפוסות ריבוזומים לאורך האורף ומאפשרת גילוי של תזוזות ריבוזומליות בקצב התרגום הקשורות לכל קבוצת אינטראקציות. לכן חובה להשוות את תפוסת הריבוזומים בכל קודון לאורך האורף כדי לזהות נכון את ההעשרה. שימוש בממוצעי אורפס עלול להוביל לאובדן אינטראקציות חולפות או לגילוי שגוי.
שימוש נכון בשיטת SeRP פותח מסלולים רבים של תרגום משותף לניתוח ישיר, ומגלה תכונות מכניסטיות חדשניות כמו גם גורמים חדשים הקשורים לריבוזומים, ומחולל מהפכה בתחום החלבון-ביוסינתזה.
Disclosures
המחברים מצהירים על היעדר ניגודי עניינים.
Acknowledgements
ברצוננו להודות לכל חברי המעבדה על הדיונים הפוריים ולמוחמד מחזומי על הקריאה הביקורתית בכתב היד. עבודה זו מומנה על ידי מענק ISF (הקרן הישראלית למדע) 2106/20.
Materials
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
References
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters - Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved