Method Article
Identificazione globale delle reti di interazione co-traslazionale mediante profilazione selettiva dei ribosomi
In questo articolo
Riepilogo
Le interazioni co-traslazionali svolgono un ruolo cruciale nelle modifiche della catena nascente, nel targeting, nella piegatura e nei percorsi di assemblaggio. Qui, descriviamo il Selective Ribosome Profiling, un metodo per l'analisi diretta in vivo di queste interazioni nel modello eucariota Saccharomyces cerevisiae.
Abstract
Negli ultimi anni, è diventato evidente che i ribosomi non solo decodificano il nostro mRNA, ma guidano anche l'emergere della catena polipeptidica nell'ambiente cellulare affollato. I ribosomi forniscono la piattaforma per il legame controllato spazialmente e cineticamente di fattori di targeting della membrana, enzimi modificanti e chaperoni pieghevoli. Anche l'assemblaggio in complessi oligomerici di alto ordine, così come le fasi di formazione della rete proteina-proteina, sono stati recentemente scoperti per essere coordinati con la sintesi.
Qui, descriviamo il Selective Ribosome Profiling, un metodo sviluppato per catturare le interazioni co-traduzionali in vivo. Descriveremo in dettaglio le varie fasi di purificazione dell'affinità necessarie per catturare complessi ribosomi-catena nascente insieme agli interattori co-traduzionali, nonché l'estrazione dell'mRNA, l'esclusione delle dimensioni, la trascrizione inversa, il sequenziamento profondo e le fasi di analisi dei big data, necessarie per decifrare le interazioni co-traduzionali nella risoluzione quasi codone.
Introduzione
Selective Ribosome Profiling (SeRP) è l'unico metodo, ad oggi, che cattura e caratterizza le interazioni co-traslazionali, in vivo, in modo diretto 1,2,3,4,5,6. SeRP consente la profilazione globale delle interazioni di qualsiasi fattore con la traduzione dei ribosomi nella risoluzione vicino al codone 2,7.
Il metodo si basa sul congelamento flash delle cellule in crescita e sulla conservazione della traduzione attiva. I lisati cellulari vengono quindi trattati con RNasi I per digerire tutto l'mRNA nella cellula ad eccezione dei frammenti di mRNA protetti dai ribosomi chiamati "impronte di ribosomi". Il campione viene quindi diviso in due parti; una parte viene utilizzata direttamente per l'isolamento di tutte le impronte ribosomiali cellulari, rappresentando tutta la traduzione in corso nella cellula. La seconda parte viene utilizzata per l'affinità-purificazione del sottoinsieme specifico di ribosomi associati a un fattore di interesse, ad esempio: modifica di enzimi, fattori di traslocazione, chaperoni di piegatura e interazioni complesso-assemblaggio. Le impronte ribosomiali purificate dall'affinità sono collettivamente chiamate interattoma. Quindi, gli mRNA protetti dai ribosomi vengono estratti e utilizzati per la generazione di librerie di cDNA, seguiti dal sequenziamento profondo.
L'analisi comparativa dei campioni totali di translatome e interactome consente l'identificazione di tutti gli orfs che si associano al fattore di interesse, nonché la caratterizzazione di ciascun profilo di interazione orf. Questo profilo riporta le precise sequenze di insorgenza e terminazione dell'incarico da cui si possono dedurre i codoni decodificati e i rispettivi residui della catena polipeptidica emergente, nonché sulle variazioni di velocità del ribosoma durante l'interazione 7,8. La Figura 1 illustra il protocollo come schematico.
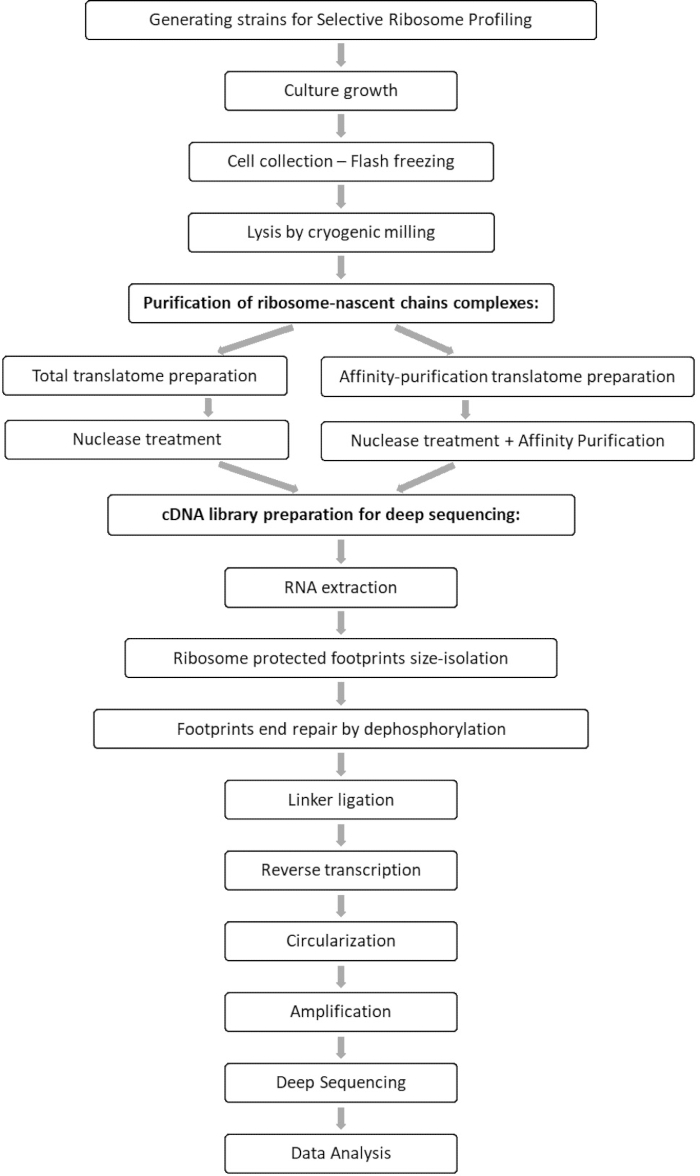
Figura 1: Panoramica del protocollo SeRP. Questo protocollo può essere eseguito nella sua interezza entro 7-10 giorni. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Generazione di ceppi per la profilazione selettiva dei ribosomi
NOTA: Selective Ribosome Profiling (SeRP) è un metodo che si basa sulla purificazione dell'affinità dei fattori di interesse, per valutare la loro modalità di interazione con i complessi di catene ribosomi-nascenti. La ricombinazione omologa9 e i metodi basati su CRISPR / Cas910 vengono utilizzati per fondere vari fattori di interesse con tag per purificazioni di affinità. Tali tag sono GFP, per le purificazioni di affinità GFP-trap, TAP-tag per le purificazioni di perline IgG-Sepharose e AVI-Tag purificato da avidina o streptavidina, per elencare alcuni esempi di successo degli ultimi anni.
- Eseguire test di crescita o funzionali per convalidare che il tagging non abbia avuto un impatto sulla funzione delle proteine. Il tagging terminale N' versus C' dovrebbe essere valutato.
NOTA: I ribosomi (rRNA), così come molti domini di legame dei ribosomi in vari fattori, sono altamente carichi, rendendo i tag altamente carichi (come la poliistidina) non prefaducibili da usare, poiché possono portare a false scoperte o modalità di legame alterata.
2. Crescita della cultura
- Coltivare le colture di lievito costruite (basate sul ceppo BY4741), contenenti le proteine marcate desiderate, in un mezzo liquido ricco di estratto di lievito-peptone-destrosio (YPD) o in un mezzo minimo di destrosio sintetico (SD) (1,7 g/L di base di azoto di lievito con solfato di ammonio o 1,7 g/L di lievito a base di azoto senza solfato di ammonio con 1 g/L di acido glutammico monosodico, 2% di glucosio e integrato con una miscela completa o appropriata di aminoacidi).
- Coltivare 250-500 mL di coltura cellulare fino a 0,5 OD600 (mid-log), a 30 °C, in un mezzo appropriato.
3. Raccolta e lisi cellulare
- Raccogliere rapidamente le celle mediante filtrazione sotto vuoto su una membrana di blotting nitrocellulosa da 0,45 μm con un sistema di filtraggio in vetro (portafiltro in vetro con imbuto di vetro da 1 L, base e tappo sottovuoto, schermo in acciaio inossidabile, guarnizione e morsetto a molla, 90 mm; pallone giunto rettificato 1 L).
- Flash congelare le cellule raccolte, raschiando le cellule pellettate con una spatola e immergendole immediatamente in un tubo da 50 ml pieno di azoto liquido.
PUNTO DI ARRESTO: Le cellule possono essere conservate a -80 °C per un massimo di 3-4 settimane. - Eseguire la lisi cellulare mediante macinazione criogenica in un mulino miscelatore: due volte per 2 minuti a 30 Hz, con 1 mL del tampone di lisi (vedere Tabella 1). Raffreddare in azoto liquido tra le fresature.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 10 mg/mL CHX (cicloeximide) | 220 | 0,5 mg/mL |
| 1M Tris-HCl pH 8,0 | 88 | 20 mM |
| 3M KCl | 205.7 | 140 metri quadrati |
| 1M MgCl2 | 26.4 | 6 mM |
| 1 milione di PMSF | 4.4 | 1 mM |
| NP-40 · | 4.4 | 0.10% |
| Inibitore della proteasi | 2 compresse | |
| DNasi I | 8.8 | 0,02 U/mL |
| Volume finale | 4,400 |
Tabella 1: Ricetta per la miscela master del buffer di lisi.
NOTA: Il tampone di lisi può essere modificato per contenere più inibitori della proteasi (come bestatina, leupeptina, aprotinina, ecc.) nel caso in cui la proteina di interesse sia molto instabile, ma è importante evitare l'EDTA per mantenere le piccole e grandi subunità del ribosoma assemblate durante i passaggi successivi. Per ragioni simili, mantenere sempre almeno 6 mM MgCl2 nella soluzione tampone.
ATTENZIONE: HCl è altamente corrosivo e PMSF è tossico. Indossare guanti e manico con cura.
- Centrifugare per 2 min a 30.000 x g, 4 °C per eliminare il lisato e raccogliere il surnatante.
4. Purificazione di complessi ribosomi-catene nascenti per SeRP
- Per ogni esperimento, dividere il surnatante in due parti; ciascuno in una diversa provetta di microcentrifuga: campione totale di RNA (~ 200 μL) e campione di immunopurificazione (IP) (~ 700 μL) campioni di translatoma.
-
Elaborazione del campione totale di RNA
- Digerire il campione totale di RNA utilizzando 10 U di RNasi I per 25 minuti a 4 °C; ruotare a 30 RPM con un rack di miscelazione rotante.
NOTA: le condizioni di digestione possono essere calibrate utilizzando la profilatura dei polisomi per garantire che non ci sia sovra o sottodigestione del picco dei monosomi. - Preparare la miscela principale del cuscino di saccarosio come descritto nella Tabella 2.
- Digerire il campione totale di RNA utilizzando 10 U di RNasi I per 25 minuti a 4 °C; ruotare a 30 RPM con un rack di miscelazione rotante.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 50% Saccarosio | 200 | 25% |
| 1M Tris-HCl pH 8,0 | 8 | 20 mM |
| 3M KCl | 18.7 | 140 metri quadrati |
| 1M MgCl2 | 4 | 10 mM |
| 100 mg/ml di CHX | 0.4 | 0,1 mg/mL |
| Inibitore della proteasi | 1 compressa | |
| Volume finale | 400 |
Tabella 2: Ricetta per il master mix di cuscini di saccarosio.
- Caricare il campione su 400 μL del cuscino di saccarosio e della centrifuga in un rotore TLA120 per 90 minuti a 245.000 x g e a 4 °C.
- Rimuovere rapidamente il surnatante con una pompa per vuoto e sovrapporre i pellet con un tampone di lisi da 150 μL. Risospese il pellet agitando per 1 ora a 4 °C e a 300 giri/min.
- Risospesso il pellet residuo mediante pipettaggio e trasferimento in un nuovo tubo da 1,5 ml.
NOTA: 100-200 μg di RNA totale sono solitamente sufficienti per la profilazione del ribosoma del translatoma totale. Si può aggiungere la fase di deplezione dell'rRNA al fine di ridurre la contaminazione da rRNA, che è il contaminante più diffuso dei complessi ribosomi-catene nascenti purificazioni di affinità11 (vedi discussione per ulteriori dettagli).
-
Elaborazione del campione di immunopurazione
- Lavare 100-400 μL della matrice legante l'affinità (perline coniugate con anticorpi 1:1 in EtOH al 70%) per campione con tampone di lisi 3 x 1 mL (senza DNasi I e inibitori della proteasi); risospesere la matrice di affinità nel buffer di lisi e quindi ruotare a 30 RPM con un rack di miscelazione rotante a 4 °C per 5 minuti. Precipitato per centrifugazione per 30 s a 3.000 x g, 4 °C. Scartare il liquido superiore. Ripeti tre volte.
- Digerire i campioni di immunopurificazione utilizzando 10 U per unità A260 nm di RNasi I, insieme alla matrice di legame di affinità (ad esempio, 100-400 μL di GFP-TRAP per campione).
- Ruotare per 25 minuti a 30 RPM con un rack di miscelazione rotante per legare la proteina alla matrice di affinità, a 4 °C.
- Preparare la miscela principale del tampone di lavaggio come descritto nella Tabella 3.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 10 mg/mL CHX | 50 | 0,1 mg/mL |
| 1M Tris-HCl pH 8,0 | 100 | 20 mM |
| 3M KCl | 233 | 140 metri quadrati |
| 1M MgCl2 | 50 | 10 mM |
| 1 milione di PMSF | 5 | 1 mM |
| NP-40 · | 0.5 | 0.01% |
| Inibitore della proteasi | 2 compresse | |
| 50% Glicerolo | 1,000 | 10% |
| Volume finale | 5,000 |
Tabella 3: Ricetta per il mix master del tampone di lavaggio.
- Lavare la matrice legante l'affinità tre volte con 1 mL di tampone di lavaggio, ogni volta per ~ 1 min, ruotando nel rack di miscelazione a 30 RPM, a 4 °C.
- Precipitare per centrifugazione a 3.000 x g per 30 s a 4 °C. Scartare il liquido superiore.
- Lavare altre due volte in 1 mL di tampone di lavaggio, ogni volta per 5 minuti, ruotando nel rack di miscelazione a 30 RPM, a 4 °C.
- Precipitato per centrifugazione per 30 s a 3.000 x g e 4 °C.
- Utilizzare 50 μL di perline per l'eluizione proteica con la stessa quantità di 2x tampone campione. Utilizzare il resto delle perline per l'estrazione dell'RNA.
- Centrifugare per 30 s a 3.000 x g, 4 °C per pellettizzare le perline ed scartare il liquido superiore.
- Congelare in azoto liquido e conservare a -80 °C. Utilizzare questi campioni per la successiva estrazione dell'RNA.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C durante la notte o più a lungo. Questo può essere un punto di arresto. - Valutare il successo della purificazione dell'affinità passo per western blot o colorazione Coomassie con aliquote (~ 10% in volume, dopo la miscelazione) di ogni fase. Utilizzare sempre l'IP fittizio su un ceppo WT non contrassegnato come controllo per il legame non specifico alla matrice di affinità.
NOTA: l'elevato background non specifico può essere superato con ulteriori fasi di lavaggio con l'aumentare delle concentrazioni di sale / detersivo. L'interazione transitoria può essere stabilizzata da vari trattamenti con agenti reticolanti, ad esempio il trattamento con paraformaldeide (PFA) delle cellule viventi - l'aggiunta dello 0,4% -1% di PFA ai mezzi di crescita per 2-5 minuti, seguita da spegnimento della glicina (0,3 M) per 3 minuti, è altamente raccomandata.
ATTENZIONE: La paraformaldeide è un sospetto cancerogeno. Poiché la paraformaldeide evapora rapidamente ed è corrosiva, lavorare in una cappa di sicurezza chimica e indossare due strati di guanti.
5. Preparazione della libreria cDNA per il sequenziamento profondo
- Estrazione dell'RNA
NOTA: Lavorare con tubi antiaderenti da 1,5 mL privi di RNasi per prevenire possibili deplezioni di RNA o DNA.- Scongelare i campioni dai passaggi 4.2.5 e 4.3.12 sul ghiaccio e risospese i campioni con 10 mM Tris-HCl pH 7.0 a un volume finale di 700 μL.
ATTENZIONE: L'acido-fenolo e il cloroformio sono volatili e dannosi. Lavora in una cappa di sicurezza chimica. - Aggiungere 40 μL di SDS al 20% a 0,7 mL di RNA totale o eluizioni IP. Chiudi e inverti un paio di volte. La precipitazione delle proteine dovrebbe rendere i campioni bianchi.
- Aggiungere 0,75 ml di acido-fenolo preriscaldato:cloroformio ai campioni. Sigillare saldamente i tubi e agitare in un miscelatore termico a 1.400 giri/min per 5 minuti e a 65 °C. Raffreddare i campioni sul ghiaccio per 5 minuti.
- Centrifugare il tubo dal punto 5.1.3. a 20.000 x g per 2 min. Trasferire lo strato acquoso superiore in un tubo fresco e aggiungere ad esso 0,7 ml di acido-fenolo:cloroformio.
- Incubare per 5 minuti a temperatura ambiente, occasionalmente vorticosamente. Centrifuga per 2 min a 20.000 x g. Trasferire lo strato acquoso superiore in un tubo fresco e aggiungere ad esso 0,6 ml di cloroformio e vortice.
- Centrifuga per 1 min a 20.000 x g. Trasferire lo strato acquoso superiore in un tubo fresco.
- Precipitare gli acidi nucleici aggiungendo 78 μL di 3 M NaOAc, pH 5,5, 2 μL di GlycoBlue e 0,75 mL di isopropanolo. Vortice completo per 5 min. Incubare per almeno 1 ora a -80 °C o 16 ore a -20 °C.
- Centrifugare per 30 min a 20.000 x g e a 4 °C ed eliminare il surnatante. Lavare il pellet con 0,75 ml ghiacciati di etanolo all'80%. Invertire i tubi per un lavaggio accurato. Centrifugare a 20.000 x g per 5 minuti a 4 °C, quindi scartare il surnatante.
- Girare verso il basso a 450 x g, 4 °C per 20 s e rimuovere l'etanolo rimanente ed eliminare i liquidi. Asciugare il pellet con un coperchio aperto per 5 minuti a 65 °C. Sospendere i campioni come segue: per IP risospese il campione in 10 μL di 10 mM Tris-HCl, pH 7.0. Per l'analisi del trasloma totale, risospesare il campione in 20 μL di 10 mM Tris-HCl, pH 7,0.
PUNTO DI ARRESTO: L'RNA può essere conservato a -80 °C per mesi.
- Scongelare i campioni dai passaggi 4.2.5 e 4.3.12 sul ghiaccio e risospese i campioni con 10 mM Tris-HCl pH 7.0 a un volume finale di 700 μL.
- Quantificare la concentrazione totale di RNA mediante fluorometria
NOTA: tutti i seguenti materiali e superfici devono essere privi di RNasi durante la preparazione della libreria di cDNA per il sequenziamento di nuova generazione. Durante la manipolazione di campioni di RNA, indossare guanti.- Diluire 1 μL di RNA totale estratto da fenolo acido in 9 μL di 10 mM Tris-HCl, pH 7,0. Quantificare utilizzando un fluorometro, come indicato sul sito Web del produttore.
- Diluire i campioni contenenti 50 μg di RNA con 10 μL di 10 mM Tris-HCl, pH 7,0.
NOTA: non misurare campioni IP, utilizzare tutto per il passaggio successivo.
- Frammenti di impronta protetta dal ribosoma purificante in gel
- Impostare un gel di poliacrilammide TBE-urea al 15% e immergerlo in 1x TBE running buffer. Eseguire per 30 minuti a 200 V prima del caricamento del campione. A ciascun campione, aggiungere 20 μL di 2x tbe-urea tampone campione.
NOTA: la dimensione della banda prevista è di circa 25-35 nt. - Scongelare una scala di DNA da 10 bp e denaturare i campioni (non la scala) a 80 °C per 2 minuti, quindi raffreddare sul ghiaccio. Carica ogni campione su ogni altra corsia. Eseguire il gel per 50-70 minuti a 200 V.
- Diluire 6 μL di SYBR Gold (10.000 x concentrato) in 60 mL di 1x tbE tampone e macchiare mentre si agita in scatole protette dalla luce per 15-20 min. Durante la colorazione del gel, preparare un bisturi sterile e tubi rompi gel da 0,5 ml in tubi etichettati da 1,5 ml.
- Asportare le fasce desiderate con un bisturi sterile (usarne uno fresco o pulire bene tra i campioni) e posizionare ogni pezzo di gel in un tubo rompi-gel.
- Scatta un'immagine del gel per assicurarti che nel gel non rimangano residui di campioni.
- Centrifugare i tubi contenenti fette tagliate a 20.000 x g per 5 minuti a 4 °C e trasferire i restanti pezzi di gel dal tubo rompi gel al tubo da 1,5 ml.
- Aggiungere 0,5 mL di 10 mM Tris, pH 7,0. Agitare in un miscelatore termico a 1.400 giri/min per 10 minuti a 70 °C.
- Trasferire su una colonna di acetato di cellulosa con punta a pipetta a foro largo e centrifugare a 20.000 x g per 3 minuti a 4 °C.
- Trasferire il flusso attraverso un nuovo tubo da 1,5 ml e raffreddare su ghiaccio.
- Per precipitare gli acidi nucleici, aggiungere: 550 μL di IPA, 55 μL di 3 M NaOAc e 2 μL di GlycoBlue e vortice per mescolare accuratamente. Posizionare i campioni a -80 °C per almeno 1 ora.
- Centrifugare per almeno 1 ora a 20.000 x g e 4 °C ed eliminare il surnatante. Lavare il pellet con 0,75 ml di etanolo all'80% ghiacciato. Capovolgere i tubi per un lavaggio accurato fino a quando i pellet non si separano dal fondo. Centrifugare nuovamente a 20.000 x g per 5 minuti a 4 °C ed eliminare il surnatante.
- Girare verso il basso a 450 x g, 4 °C per 20 s e rimuovere l'etanolo rimanente. Asciugare il pellet con un coperchio aperto per 5 minuti a 65 °C.
- Aggiungere 15 μL di 10 mM Tris, pH 7,0 e risospescire accuratamente il pellet. Girare verso il basso a 450 x g, 4 °C per 20 s e trasferire il campione in un nuovo tubo da 1,5 ml.
PUNTO DI ARRESTO: L'RNA purificato può essere conservato a -80 °C per alcuni mesi.
- Impostare un gel di poliacrilammide TBE-urea al 15% e immergerlo in 1x TBE running buffer. Eseguire per 30 minuti a 200 V prima del caricamento del campione. A ciascun campione, aggiungere 20 μL di 2x tbe-urea tampone campione.
- Defosforilazione
- Utilizzare 3 μL della seguente miscela per ciascun campione: Aggiungere 1 μL di inibitore della RNasi in 2 μL di 10x T4 tampone di reazione polinucleotide chinasi senza ATP. Aggiungere 2 μL di polinucleotide chinasi T4 a ciascun campione. Pipettare delicatamente per mescolare bene e incubare a 37 °C per 2 ore, senza agitare.
- Per inattivare l'enzima, incubare il campione a 75 °C per 10 minuti e ruotare verso il basso a 450 x g, 4 °C, per 20 s. Aggiungere 0,5 mL di 10 mM Tris, pH 7,0.
- Per precipitare l'acido nucleico, aggiungere 2 μL di GlycoBlue, 550 μL di IPA e 55 μL di 3 M NaOAc.
- Vortice per mescolare accuratamente e raffreddare i campioni a -80 °C per almeno 1 ora.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C durante la notte o più a lungo. - Ripetere i passaggi 5.3.11-5.3.13.
PUNTO DI ARRESTO: L'RNA defosforilato può essere conservato a -80 °C per mesi.
- Quantificazione mediante bioanalizzatore
- Effettuare una diluizione 1:4 di ciascun campione di RNA mescolando 1 μL di campione e 4 μL di acqua trattata con DEPC.
ATTENZIONE: DEPC è cancerogeno. Indossare guanti e lavorare con attenzione. - Eseguire un Bioanalyzer Small RNA Chip/TapeStation. Seguire il protocollo del produttore.
NOTA: La dimensione prevista del frammento di RNA protetto da ribosomi è di circa 28-30 nt.
- Effettuare una diluizione 1:4 di ciascun campione di RNA mescolando 1 μL di campione e 4 μL di acqua trattata con DEPC.
- Ligate 3' fine con Linker-1
- Diluire 5 pmol di piccoli frammenti di RNA a 10 μL con 10 mM Tris, pH 7,0. Denaturare i campioni a 80 °C per 2 minuti e raffreddare su ghiaccio.
- Preparare la miscela principale come descritto nella Tabella 4 e utilizzare 29 μL per campione.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 50% PEG 8000 con filtro sterile | 16 | 20% |
| DMSO | 4 | 10% |
| 10× T4 RNA ligasi 2 tampone | 4 | 1x |
| Inibitore della RNasi SUPERase-In | 2 | 2 U |
| 10 mM linker adenilato 3-L1 | 0.1 | 25 μM |
| Acqua trattata con DEPC | 2.9 | |
| Volume finale | 29 |
Tabella 4: Ricetta per 3' end ligation master mix.
- Aggiungere 1 μL di T4 RNA ligasi 2 e pipettare delicatamente per mescolare bene. Incubare a 23 °C per 2 ore.
- Per precipitare gli acidi nucleici, aggiungere: 550 μL di IPA, 500 μL di 10 mM Tris, pH 7,0, 55 μL di 3 M NaOAc e 2 μL di GlycoBlue. Vortice per mescolare accuratamente e posizionare i campioni a -80 °C per almeno 1 ora.
PUNTO DI ARRESTO: Conservare i campioni a -80 °C durante la notte o più a lungo. - Ripetere i passaggi 5.3.2-5.3.12.
- Risospendare il pellet in 6 μL di 10 mM Tris, pH 7.0. Girare verso il basso a 450 x g, 4 °C per 20 s, e trasferire il campione in un nuovo tubo da 1,5 ml.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C per mesi.
-
Purificazione in gel di impronte collegate a 3'
- Impostare un gel di poliacrilammide TBE-urea al 10% e immergerlo in 1x TBE running buffer. Eseguire per 30 minuti a 200 V prima del caricamento del campione. A ciascun campione, aggiungere 6 μL di 2x tbe-urea tampone campione.
NOTA: la dimensione della banda prevista è di circa 71-73 nt. - Ripetere i passaggi 5.3.2-5.3.13.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C per mesi.
- Impostare un gel di poliacrilammide TBE-urea al 10% e immergerlo in 1x TBE running buffer. Eseguire per 30 minuti a 200 V prima del caricamento del campione. A ciascun campione, aggiungere 6 μL di 2x tbe-urea tampone campione.
-
Trascrivere inversamente frammenti di footprint collegati a 3ʹ per generare ssDNA
- Preparare una miscela principale come descritto nella Tabella 5 e utilizzare 3 μL per campione.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 10 mM dNTPs | 1 | 0,5 mM |
| 25 μM Linker L(rt) | 0.5 | 625 nM |
| Acqua trattata con DEPC | 1.5 | |
| Volume finale | 3 |
Tabella 5: Ricetta per la miscela master del buffer di trascrizione inversa prima della denaturazione degli acidi nucleici.
- Vortice e rotazione verso il basso del campione.
- Incubare i campioni a 65 °C per 5 min.
- Raffreddare i campioni sul ghiaccio.
- Preparare una miscela principale come descritto nella Tabella 6 e utilizzare 6 μL per campione. Vortice e rotazione verso il basso del campione. Aggiungere 1 μL di Apice III a ciascun campione e pipettare delicatamente per mescolare bene e incubare per 30 minuti a 50 °C.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 5× Buffer FS | 4 | 1x |
| Inibitore della RNasi SUPERase-In | 1 | 2 U |
| DTT 0,1 M | 1 | 5 mM |
| Volume finale | 6 |
Tabella 6: Ricetta per il master mix del buffer di trascrizione inversa dopo la denaturazione degli acidi nucleici.
- Aggiungere 2,3 μL di 1 N NaOH, che idrolizza l'RNA e spegne la trascrizione inversa.
ATTENZIONE: NaOH è altamente corrosivo. Indossare guanti e protezione per gli occhi. - Incubare per 15 minuti a 95 °C, fino a quando il campione diventa rosa.
- Impostare un gel di poliacrilammide TBE-urea al 10% e immergerlo in 1x TBE running buffer. Eseguire per 30 minuti a 200 V prima del caricamento del campione. A ciascun campione, aggiungere 23 μL di 2x tbe-urea tampone campione.
NOTA: la dimensione prevista della banda del DNA è 115-117 nt. - Ripetere i passaggi 5.3.2-5.3.12.
- Risospendare il pellet in 15 μL di 10 mM Tris, pH 8,0. Girare verso il basso a 450 x g, 4 °C per 20 s e trasferire il campione in un nuovo tubo da 1,5 ml.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C per mesi.
-
Circolarizzazione ssDNA
- Preparare la seguente miscela principale e caricare 4 μL per campione, come descritto nella Tabella 7.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| 10× Tampone CircLigase II | 2 | 1x |
| 5 M Betaina (opzionale) | 1 | 0,25 m |
| 50 mM MnCl2 | 1 | 2,5 mM |
| Volume finale | 4 |
Tabella 7: Ricetta per il master mix di circolarizzazione ssDNA.
- Aggiungere 1 μL di CircLigase II ssDNA ligasi a ciascun campione e incubare per 1 ora a 60 °C.
NOTA: L'efficienza di questa fase può essere aumentata aggiungendo 1 μL di CircLigase II ssDNA ligasi a ciascun campione dopo 1 ora di incubazione. - Inattivare l'enzima incubando a 80 °C per 10 min.
- Raffreddare su ghiaccio e continuare ad amplificare PCR o conservare a -80 °C.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C per anni.
-
Amplificazione PCR
- Preparare la seguente miscela master PCR e caricare 82 μL per campione, come dettagliato nella Tabella 8.
| Reagente | Quantità per campione (μL) | Concentrazione finale |
| Acqua trattata con DEPC | 61.6 | |
| 5× Tampone di reazione Phusion HF | 17.6 | 1x |
| 10 mM dNTPs | 1.8 | 200 μM |
| Primer anteriore PCR da 100 μM | 0.2 | 225 nM |
| HF Phusion polimerasi | 0.8 | 1,6 U |
| Volume finale | 82 |
Tabella 8: Ricetta per il master mix di amplificazione PCR.
- Ad ogni tubo contenente miscela master, aggiungere 5 μL di DNA circolarizzato.
NOTA: Conservare il resto dei campioni di DNA circolarizzati a -80 °C. - Aggiungere un diverso primer per codice a barre inverso PCR da 1 μL da 20 μM a ciascun campione (vedere Tabella 9) e un vortice per mescolare accuratamente.
- Aliquotare ogni tubo in quattro tubi PCR separati, ciascuno dei quali sarà utilizzato per un diverso numero di cicli pcr.
- Eseguire una reazione PCR secondo il seguente programma, come dettagliato nella Tabella 10.
| Ciclo | Denaturazione (98 °C) | Ricottura (60 °C) | Estensione (72 °C) |
| 1 | 30 anni | ||
| 2-16 | 10 s | 10 s | 5 s |
Tabella 10: Programma PCR per la reazione PCR.
- Dopo i cicli 8, 9, 10 e 11 rimuovere i tubi PCR (per i campioni IP, i cicli vanno da 9 a 15) come primo tentativo. Dopo ogni ciclo, metti in pausa il programma, togli un'aliquota e mettila sul ghiaccio, quindi riprendi rapidamente il programma.
NOTA: Il numero di cicli deve essere regolato in base alla quantità di DNA circolarizzato in ogni reazione. Fare riferimento alla Figura 4 per un esempio e ulteriori chiarimenti. - Ad ogni reazione da 17 μL, aggiungere 3,5 μL di colorante a carico di DNA 6x.
- Scongelare una scala del DNA da 10 bp.
- Per la separazione dimensionale mediante elettroforesi su gel, immergere l'8% di poliacrilammide TBE in 1x TBE running buffer e caricare i campioni di ogni diverso numero di ciclo in pozzi adiacenti ed eseguire il gel per 50 minuti a 180 V.
- Diluire 6 μL di SYBR Gold (10.000 x concentrato) in 60 mL di 1x tbE tampone e macchiare mentre si agita in scatole protette dalla luce per 15-20 min.
- Durante la colorazione del gel, preparare un bisturi sterile e tubi rompi gel da 0,5 ml in tubi etichettati da 1,5 ml.
- Scatta un'immagine degli acidi nucleici colorati.
- Tagliare la fascia desiderata con una dimensione prevista della banda di 174-176 bp con il bisturi sterile e posizionare la fetta di gel nel tubo gel-breaker preparato da 0,5 ml (pulire accuratamente tra i campioni e utilizzare l'agente inattivante RNase o passare a una nuova lama).
- Centrifugare i tubi per 5 minuti a 20.000 x g e 4 °C, quindi trasferire i restanti pezzi di gel dal tubo rompi gel da 0,5 mL al tubo da 1,5 ml.
- Aggiungere 500 μL di 10 mM Tris, pH 8,0 e agitare in un miscelatore termico a 1.400 RPM per 10 min e 70 °C.
- Trasferire il gel disciolto su una colonna di acetato di cellulosa con una punta a pipetta a foro largo.
- Centrifugare la colonna per 3 minuti a 20.000 x g e 4 °C e trasferire il flusso attraverso un nuovo tubo da 1,5 ml e raffreddare su ghiaccio.
- Per precipitare l'acido nucleico, aggiungere: 550 μL di IPA, 32 μL di 5 M NaCl, 1 μL di 0,5 M EDTA e 2 μL di GlycoBlue e vortice per mescolare accuratamente.
- Conservare i campioni per almeno 1 ora a -80 °C o -20 °C durante la notte.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C durante la notte o più a lungo. - Ripetere i passaggi 5.3.11-5.3.12.
- Risospeso in 11 μL di 10 mM Tris, pH 8,0. Girare verso il basso a 450 x g, 4 °C per 20 s e trasferire il campione in un nuovo tubo da 1,5 ml.
PUNTO DI ARRESTO: I campioni possono essere conservati a -80 °C per anni.
-
Quantificare la distribuzione dimensionale mediante Bioanalyzer
- Effettuare una diluizione 1:4 di ciascun campione mescolando 1 μL di campione con 4 μL di acqua trattata con DEPC.
- Esegui il Bioanalyzer Small RNA Chip. Seguire il protocollo del produttore.
NOTA: la lunghezza prevista è di 175 ± 5 bp.
-
Quantificare la concentrazione di DNA mediante fluorometro
- Eseguire un controllo della concentrazione ad alta sensibilità del dsDNA con un fluorometro secondo le raccomandazioni del produttore.
- Multiplex e campioni di sequenza secondo le raccomandazioni Illumina (Index Adapters Pooling Guide12).
6. Analisi dei dati
- Eseguire l'analisi come dettagliato nel file supplementare.
Risultati
Come illustrato nel diagramma di flusso di questo protocollo (Figura 1), le cellule sono state coltivate fino alla fase di log, e poi raccolte rapidamente per filtrazione e lisate mediante macinazione criogenica. Il lisato è stato poi diviso in due: uno per le impronte totali di mRNA protette dai ribosomi e l'altro per impronte di mRNA protette da ribosomi selezionate, su cui abbiamo eseguito la purificazione dell'affinità per abbattere i complessi di catene proteiche-ribosomi-nascenti marcati. Abbiamo assicurato l'espressione proteica etichettata e il successo del pull-down mediante analisi western blot, come si può vedere nella Figura 2. Abbiamo convalidato l'isolamento delle impronte protette dai ribosomi, che sono in genere lunghe 20-45 nt da una piccola elettroforesi a RNA (sistema BioAnalyzer 2100), consentendo uno spostamento di 5-10 nt nel rilevamento delle dimensioni, secondo il manuale del sistema (Figura 3). Quindi, abbiamo generato una libreria di cDNA per il sequenziamento profondo e l'analisi dei big data. Durante la generazione della libreria cDNA, si noti che il sottociclismo può portare a un basso rendimento (come si può vedere nella corsia 2 nella Figura 3), ma la ri-amplificazione è possibile per recuperare la libreria generata. Il ciclo eccessivo può verificarsi quando i primer PCR sono esauriti, ma la reazione continua. Quando i dNTP sono ancora presenti, la reazione procede, generando artefatti PCR più lunghi con sequenze chimeriche dovute a prodotti PCR che si innescano13 (come si può vedere nelle corsie 3-4 in Figura 3, indicate dallo striscio visibile). Se anche la concentrazione dei dNTP diventa limitante, possono comparire prodotti che indicano la presenza di eteroduplex composti da frammenti di libreria solo parzialmente omologhi. La figura 4 funge da riferimento, con la corsia 2 che rappresenta l'amplificazione ottimale e la corsia 3 un'amplificazione accettabile. I campioni delle corsie 4 e 5 (cicli 10 e 11) non devono essere utilizzati a causa della possibilità di introdurre duplicati e artefatti della PCR. La libreria generata è stata ulteriormente convalidata mediante elettroforesi del DNA ad alta sensibilità (è stato utilizzato lo stesso sistema BioAnalyzer) per la distribuzione e la quantificazione delle dimensioni esatte (Figura 5). Dopo la legatura del linker finale di 3', la trascrizione inversa e l'amplificazione PCR, si prevede una distribuzione della lunghezza del cDNA in quanto tale, con un picco acuto intorno a 175 nt.
Abbiamo tagliato e rimosso adattatori e codici a barre dalla libreria sequenziata e solo le letture tra 20 e 45 nt sono state selezionate per ulteriori analisi. La Figura 6 mostra la distribuzione della lunghezza risultante. Le letture sono state suddivise in diversi gruppi di: sequenze codificanti, introni e sequenze intergeniche (Figura 7) e ulteriormente classificate come mostrato nella Figura 8.
L'analisi finale per il rilevamento e la caratterizzazione delle interazioni co-traduzionali è stata eseguita sulla base dell'arricchimento di frammenti di mRNA protetti da ribosomi, producendo i grafici in Figura 9. Abbiamo confrontato l'occupazione normalizzata del ribosoma (ad ogni nucleotide lungo ogni orf) del translatome totale con il corrispondente translatome selezionato (nascente-interactome). Il confronto per nucleotide elimina gli artefatti dei tassi di traduzione. La riproducibilità tra le repliche biologiche è stata valutata mediante correlazione di Pearson (soglia > 0,6). Presentiamo i profili selettivi dei ribosomi, analizzando le interazioni co-traduzionali di Vma2p con tre proteine: lo chaperone associato al ribosoma Ssb1p, Pfk2p (Fosfofruttochinasi) e Fas1p (Fatty acid synthase), con ogni proteina C' taggata terminalmente da GFP. Abbiamo eseguito il protocollo in repliche biologiche. Figura 9 A, D e G mostrano lo schema sperimentale di ogni purificazione di affinità. Successivamente mostriamo l'occupazione ribosomica dei trasdudemi totali rispetto agli interattomi Ssb1 lungo l'orf Vma2p, codificando per una subunità della vacuolare H+-ATPasi (Figura 9 B, E e H). Infine, abbiamo eseguito il profilo di arricchimento dei ribosomi basato sul rapporto (IP/ Totale) in ogni posizione del ribosoma in [codone/ aa] lungo l'orf (Figura 9 C, F e I). Confrontando le interazioni co-traslazionali di queste tre proteine con Vma2p, che viene sintetizzato dal ribosoma, ha rivelato che l'chaperone Ssb1 impegna il nascente Vma2p in quattro diverse regioni lungo l'orf, poiché abbiamo identificato quattro picchi di arricchimento significativi da Parte della SeRP. Diversamente, Pfk2p mostra solo un picco di arricchimento significativo, come identificato da SeRP, in una posizione diversa rispetto all'accompagnatore co-traduzionale Ssb1. L'analisi delle interazioni co-traduzionali di Fas1 con vma2p nascente non ha rilevato alcun arricchimento significativo. Pertanto, il confronto di questi profili di ribosomi di arricchimento basati sul rapporto dimostra il potere di questo protocollo nel rilevamento e nella caratterizzazione di varie interazioni co-traslazionali in quasi risoluzione codone.
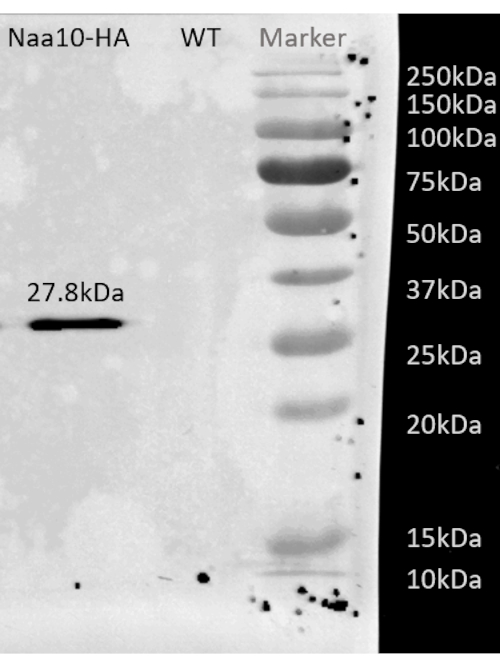
Figura 2: Risultato rappresentativo della macchia occidentale dopo la purificazione di affinità del ceppo BY4741 con Naa10 con tag HA. Risultato rappresentativo del western blot dopo la purificazione di affinità del ceppo BY4741 con Naa10 con tag HA che mostra una banda intorno a 27,8 kDa, mentre il wild-type, come controllo negativo, non mostra alcuna banda. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Risultato rappresentativo del bioanalizzatore dopo l'isolamento dell'impronta e l'estrazione dell'RNA con acido-fenolo: cloroformio e una dimensione media di 25 nt. Fare clic qui per visualizzare una versione più grande di questa figura.
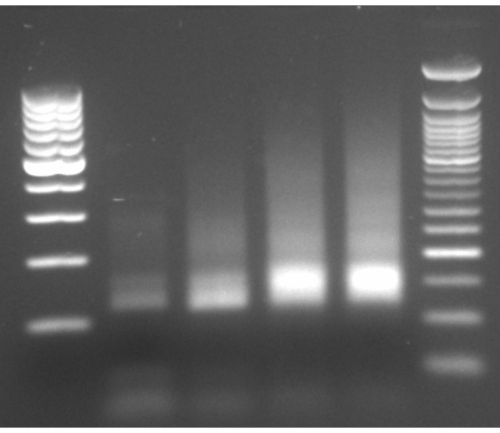
Figura 4: Elettroforesi su gel rappresentativa dell'amplificazione PCR. Elettroforesi gel rappresentativa dell'amplificazione PCR con corsie 2-5 caricate con prodotti PCR dei cicli 8-11 e scale su entrambi i lati. Fare clic qui per visualizzare una versione più grande di questa figura.
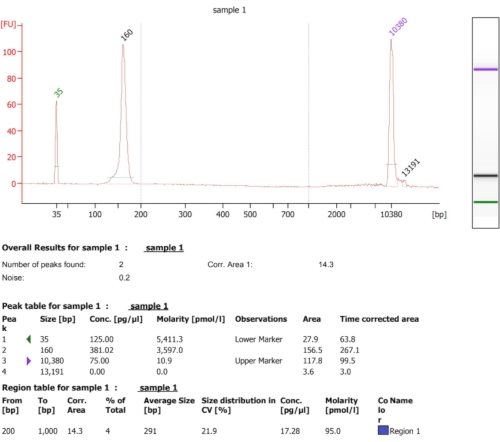
Figura 5: Risultato rappresentativo di BioAnalyzer ottenuto in seguito alla creazione di una libreria di cDNA. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Distribuzione della lunghezza prevista delle letture dopo la rimozione degli adattatori con Cutadapt (rimozione di letture inferiori a 20 o superiori a 45). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 7: Percentuale di successo dell'allineamento prevista dopo la rimozione delle letture dell'RNA non codificante con Bowtie2 e l'utilizzo di TopHat per allineare le letture rimanenti a diversi organismi. Il campione è stato prelevato da S. cerevisiae (una variante mutata di BY4741). Fare clic qui per visualizzare una versione più grande di questa figura.
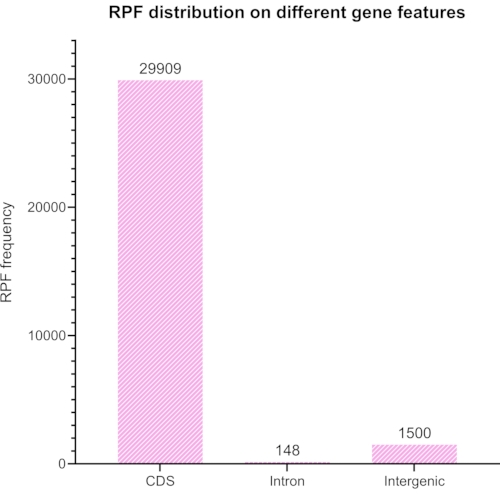
Figura 8: Un grafico generato con RiboToolkit che rappresenta la codifica prevista rispetto al rapporto non codificante delle letture allineate dopo aver usato Bowtie2 per rimuovere gli elementi rRNA nelle letture. Fare clic qui per visualizzare una versione più grande di questa figura.
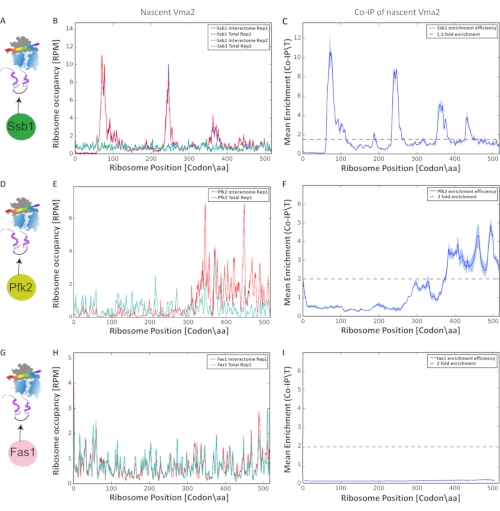
Figura 9: Interazioni co-traduzionali di tre diverse proteine: Ssb1p, Pfk2p e Fas1p con Vma2p, che viene sintetizzato dal ribosoma, analizzato da SeRP. Tutti gli assi y sono mostrati in letture per milione (RPM). (A, D, G) Schema sperimentale di SeRP di Ssb1p, Pfk2p e Fas1p C' terminalmente taggati da GFP, rispettivamente. (B, E, H) Occupazione di ribosomi lungo l'orf dei trasdudemi totali rispetto agli interattomi Ssb1, Pfk2p e Fas1p, rispettivamente (nelle repliche biologiche). (C, F, I) Arricchimento medio di Ssb1p, Pfk2p e Fas1p (rapporto IP/Totale) in ciascuna posizione del ribosoma in [codone/aa] lungo l'orf, rispettivamente. La variazione tra le repliche biologiche è indicata dall'area ombreggiata. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 9: 3' Linker e sequenze di primer. 3' Linker L1: Linker 3-L1 con adenilazione 5ʹ e identificatori molecolari unici dideossi-citidina 3ʹ ('NN...') (Purificazione HPLC senza RNasi; Linker di trascrizione inversa: trascrizione inversa (L(rt)) con identificatori molecolari univoci fosforilati a 5ʹ (purificazione HPLC senza RNasi); Primer anteriore PCR: PCRf; HPLC purificato. Fare clic qui per scaricare questa tabella.
File integrativo. Fare clic qui per scaricare questo file.
Discussione
Qui, il protocollo descrive in dettaglio l'approccio Selective Ribosome Profiling per l'acquisizione di interazioni co-traslazionali in risoluzione quasi codone. Mentre il ribosoma sale come hub per coordinare l'emergenza della catena nascente nel citoplasma affollato, questo è un metodo cruciale per identificare e caratterizzare le varie interazioni co-traduzionali necessarie per garantire un proteoma funzionale, nonché per studiare varie malattie. Ad oggi, la SeRP è l'unico metodo in grado di catturare e caratterizzare queste interazioni, in modo diretto, in vivo 14,15,16.
Il primo e più critico passo è la raccolta e la lisi cellulare. È imperativo catturare, in pochi secondi, la traduzione in corso, mediante congelamento flash seguito da lisi in uno stato congelato. La raccolta delle cellule deve essere effettuata in fretta al fine di evitare il deflusso ribosomiale e di indurre risposte traslazionali allo stress, che possono verificarsi rapidamente. Il secondo passo critico è la fase di purificazione dell'affinità. È imperativo ridurre il legame di fondo mediante un lavaggio rigoroso, assicurandosi al contempo che le interazioni co-traslazionali siano mantenute, il che può essere facilitato dalla reticolazione in vivo . Poiché questo protocollo si basa sull'alta sensibilità NGS (Next Generation Sequencing), lo sfondo alto nei primi passi può essere amplificato nelle seguenti fasi di preparazione della libreria cDNA, portando a bassi rapporti segnale-rumore.
Il trattamento con nucleasi, per digerire tutti gli mRNA non protetti, deve essere valutato mediante profilo polisomico17 insieme ad un'attenta valutazione della distribuzione dimensionale delle impronte ribosomiali isolate (come descritto sopra) per evitare la digestione sopra o sotto l'RNA. La calibrazione della concentrazione di nucleasi e dei tempi di digestione può facilitare un accurato recupero dell'impronta, poiché la digestione eccessiva può portare alla digestione dell'rRNA ribosomiale, portando alla perdita di impronte protette dai ribosomi. È importante notare che la sotto-digestione può anche portare a tassi di scoperta più bassi di impronte protette dai ribosomi, poiché le fasi di preparazione della libreria cDNA, così come le fasi di analisi dei dati qui descritte scartano letture lunghe e insolite.
Mentre l'esaurimento dell'rRNA non costituisce sempre un passaggio critico e non è obbligatorio, presenta alcuni vantaggi come campioni più puliti e, quindi, un tasso più elevato di letture mappate sul genoma. D'altra parte, c'è la possibilità di pregiudizi, poiché molti protocolli di esaurimento dell'rRNA potrebbero anche causare l'esaurimento dei frammenti protetti dal ribosoma desiderati. Si dovrebbe anche prendere in considerazione i costi dei kit di esaurimento dell'rRNA. L'esaurimento dell'rRNA può essere eseguito dopo la fase di isolamento dell'impronta del ribosoma o dopo la fase di circolarizzazione del cDNA.
Le fasi di preparazione della libreria di cDNA, come descritto qui, sono state ottimizzate per un basso input di mRNA, poiché le fasi di purificazione dell'affinità e del ribosoma riducono notevolmente la quantità di input di mRNA, rispetto agli studi di espressione RNA-seq. L'upscaling della quantità iniziale di colture cellulari può facilitare notevolmente la generazione di librerie di cDNA. In alternativa, qualsiasi protocollo di libreria cDNA di scelta può adattarsi ai passaggi di purificazione dell'affinità e isolamento dell'impronta descritti qui. È importante notare che il trattamento con nucleasi che genera le impronte ribosomiali richiede la riparazione delle estremità dell'mRNA risultante (preparazione della libreria di cDNA, fase di defosforilazione) per consentire le seguenti fasi di legatura del linker nel protocollo cDNA descritto qui nel protocollo di scelta.
Durante il sequenziamento, è importante differenziare la SeRP dall'RNA-Seq, poiché l'eterogeneità delle librerie generate varia notevolmente, a seconda dei fattori contrassegnati dall'affinità. Gli chaperoni molecolari e i fattori di targeting sono spesso più promiscui nel legame, interagendo con centinaia o migliaia di substrati durante la traduzione, portando a librerie di cDNA altamente diversificate. Tuttavia, gli interattori altamente specifici, come gli interattori di assembly complessi co-traduzionali, possono spesso portare alla generazione di librerie di cDNA molto meno diversificate. Il picco di librerie diverse e non diverse sulla stessa corsia può migliorare notevolmente il sequenziamento e i risultati dell'analisi dei dati successivi.
Un'altra caratteristica unica della SeRP è la sua capacità di catturare le variazioni locali nelle occupazioni di ribosomi lungo l'orf consentendo la scoperta di spostamenti ribosomiali nella velocità di traduzione associati a ciascun insieme di interazioni. È quindi imperativo confrontare le occupazioni di ribosomi in ciascun codone lungo l'orf per identificare correttamente l'arricchimento. L'utilizzo di medie orfs può portare alla perdita di interazioni transitorie o a false scoperte.
L'uso corretto del metodo SeRP apre molti percorsi co-traslazionali all'analisi diretta, scoprendo nuove caratteristiche meccanicistiche e nuovi fattori associati ai ribosomi, rivoluzionando il campo della biosintesi proteica.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Vorremmo ringraziare tutti i membri del laboratorio per le fruttuose discussioni e Muhammad Makhzumy per la lettura critica del manoscritto. Questo lavoro è stato finanziato dalla sovvenzione ISF (Israeli Science Foundation) 2106/20.
Materiali
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Riferimenti
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23(2012).
- Guide, P. Illumina Index Adapters - Pooling Guide. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019).
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820(2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon