Method Article
Globale Identifizierung von co-translationalen Interaktionsnetzwerken durch selektives Ribosomenprofiling
In diesem Artikel
Zusammenfassung
Co-translationale Interaktionen spielen eine entscheidende Rolle bei Modifikationen, Targeting-, Faltungs- und Montagepfaden der entstehenden Kette. Hier beschreiben wir Selective Ribosome Profiling, eine Methode zur in vivo, direkten Analyse dieser Wechselwirkungen im Modell Eukaryote Saccharomyces cerevisiae.
Zusammenfassung
In den letzten Jahren hat sich gezeigt, dass Ribosomen nicht nur unsere mRNA entschlüsseln, sondern auch die Entstehung der Polypeptidkette in die überfüllte zelluläre Umgebung leiten. Ribosomen bieten die Plattform für die räumlich und kinetisch kontrollierte Bindung von Membran-Targeting-Faktoren, die Modifikation von Enzymen und die Faltung von Chaperonen. Sogar die Montage zu oligomeren Komplexen höherer Ordnung sowie Protein-Protein-Netzwerkbildungsschritte wurden kürzlich entdeckt, um mit der Synthese koordiniert zu sein.
Hier beschreiben wir das selektive Ribosom-Profiling, eine Methode, die entwickelt wurde, um co-translationale Interaktionen in vivo zu erfassen. Wir werden die verschiedenen Affinitätsreinigungsschritte detailliert beschreiben, die für die Erfassung von Ribosomen-Nascent-Chain-Komplexen zusammen mit co-translationalen Interaktoren erforderlich sind, sowie die mRNA-Extraktions-, Größenausschluss-, Reverse-Transkriptions-, Deep-Sequencing- und Big-Data-Analyseschritte, die erforderlich sind, um co-translationale Interaktionen in nahezu Codon-Auflösung zu entschlüsseln.
Einleitung
Selective RIbosom PRofiling (SeRP) ist die bisher einzige Methode, die co-translationale Interaktionen in vivo auf direkte Weise erfasst und charakterisiert 1,2,3,4,5,6. SeRP ermöglicht die globale Profilerstellung von Wechselwirkungen eines beliebigen Faktors mit der Übersetzung von Ribosomen in nahezu Codon-Auflösung 2,7.
Die Methode beruht auf dem Einfrieren von Schockzellen und der Erhaltung der aktiven Translation. Zelllysate werden dann mit RNase I behandelt, um die gesamte mRNA in der Zelle mit Ausnahme der Ribosomen-geschützten mRNA-Fragmente, die als "Ribosomen-Fußabdrücke" bezeichnet werden, zu verdauen. Die Probe wird dann in zwei Teile geteilt; Ein Teil wird direkt für die Isolierung aller zellulären ribosomalen Fußabdrücke verwendet, die die gesamte laufende Translation in der Zelle darstellen. Der zweite Teil wird für die Affinitätsreinigung der spezifischen Teilmenge von Ribosomen verwendet, die mit einem interessierenden Faktor assoziiert sind, zum Beispiel: Modifikation von Enzymen, Translokationsfaktoren, faltenden Chaperonen und komplexen Assemblierungsinteraktionen. Die affinitätsgereinigten ribosomalen Fußabdrücke werden zusammenfassend als Interaktom bezeichnet. Dann werden die Ribosomen-geschützten mRNAs extrahiert und für die cDNA-Bibliotheksgenerierung verwendet, gefolgt von einer tiefen Sequenzierung.
Die vergleichende Analyse der gesamten Translatom- und Interaktomproben ermöglicht die Identifizierung aller Orfs, die mit dem interessierenden Faktor assoziiert sind, sowie die Charakterisierung jedes ORF-Interaktionsprofils. Dieses Profil berichtet über die genauen Engagement-Beginn- und Terminierungssequenzen, aus denen man auf die dekodierten Codons und die jeweiligen Reste der entstehenden Polypeptidkette schließen kann, sowie über die Ribosomengeschwindigkeitsschwankungen während der Wechselwirkung 7,8. Abbildung 1 zeigt das Protokoll als Schaltplan.
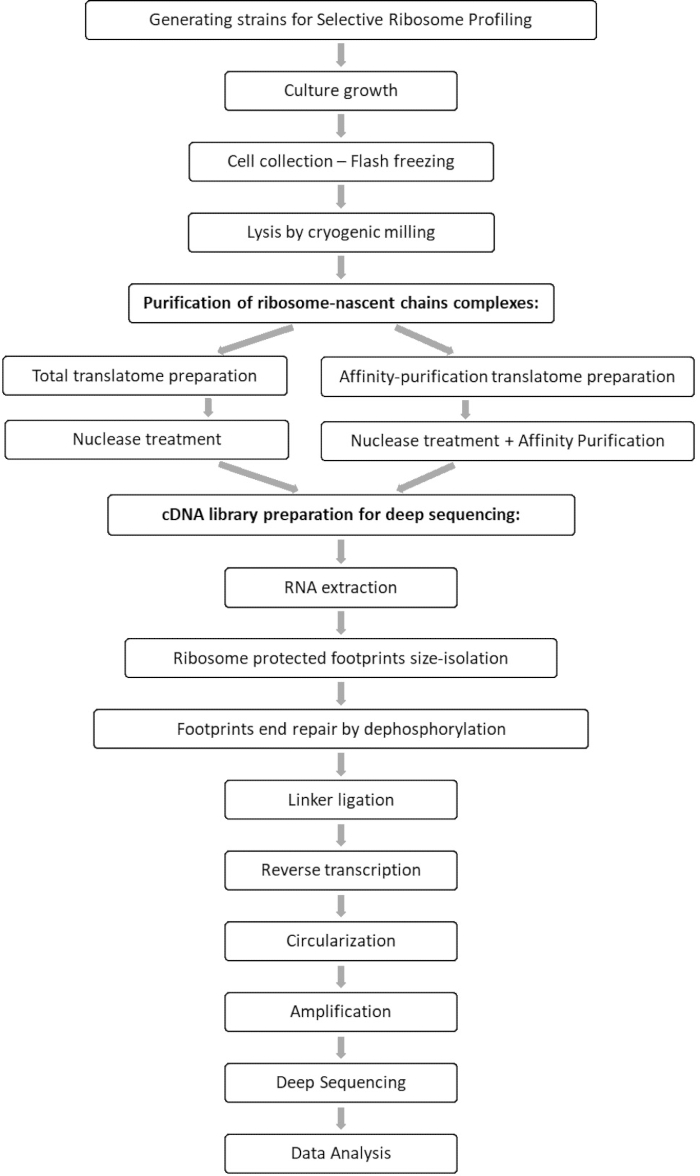
Abbildung 1: Eine Übersicht über das SeRP-Protokoll. Dieses Protokoll kann in seiner Gesamtheit innerhalb von 7-10 Tagen durchgeführt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
1. Generieren von Stämmen für die selektive Ribosomenprofilierung
HINWEIS: Selektives RIbosom PRofiling (SeRP) ist eine Methode, die auf der Affinitätsreinigung von Faktoren von Interesse beruht, um ihre Art der Interaktion mit Ribosomen-naszierenden Kettenkomplexen zu bewerten. Homologe Rekombination9 sowie CRISPR / Cas910-basierte Methoden werden verwendet, um verschiedene Faktoren von Interesse mit Tags für Affinitätsreinigungen zu verschmelzen. Solche Tags sind GFP, für GFP-Fallen-Affinitätsreinigungen, TAP-Tag für IgG-Sepharus-Perlenreinigungen sowie AVI-Tag, gereinigt durch Avidin oder Streptavidin, um nur einige erfolgreiche Beispiele aus den letzten Jahren aufzulisten.
- Führen Sie Wachstums- oder Funktionstests durch, um zu validieren, dass das Tagging die Proteinfunktion nicht beeinträchtigt hat. N' versus C' Terminal Tagging sollte evaluiert werden.
HINWEIS: Die Ribosomen (rRNA) sowie viele Ribosomenbindungsdomänen in verschiedenen Faktoren sind hoch geladen, was die Verwendung von hochgeladenen Tags (wie Polyhistidin) unbevorzugt macht, da dies zu einer falschen Entdeckung oder einem veränderten Bindungsmodus führen kann.
2. Kulturwachstum
- Kultivieren Sie die konstruierten Hefenkulturen (basierend auf dem Stamm BY4741), die die gewünschten markierten Proteine enthalten, entweder in flüssigem Hefeextrakt-Pepton-Dextrose (YPD)-reichem Medium oder in synthetischem Dextrose (SD) minimalem Medium (1,7 g/L Hefestickstoffbase mit Ammoniumsulfat oder 1,7 g/L Hefestickstoffbase ohne Ammoniumsulfat mit 1 g/L Mononatriumglutaminsäure, 2% Glucose und ergänzt mit einer vollständigen oder geeigneten Mischung von Aminosäuren).
- Züchten Sie 250-500 ml Zellkultur auf 0,5 OD600 (Mid-Log) bei 30 °C in einem geeigneten Medium.
3. Zellentnahme und Lyse
- Schnelles Sammeln von Zellen durch Vakuumfiltration auf einer 0,45 μm Nitrocellulose-Löschmembran mit einem Glasfiltersystem (Glasfilterhalter mit 1 L Glastrichter, Vakuumboden und -kappe, Edelstahlsieb, Dichtung und Federklemme, 90 mm; geschliffener Fugenkolben 1 L).
- Schockgefrieren Sie die gesammelten Zellen, indem Sie die pelletierten Zellen mit einem Spatel abkratzen und sofort in ein mit flüssigem Stickstoff gefülltes 50-ml-Röhrchen tauchen.
HALTEPUNKT: Die Zellen können bis zu 3-4 Wochen bei -80 °C gelagert werden. - Zelllyse durch kryogene Vermahlung in einer Mischermühle: zweimal für 2 min bei 30 Hz, mit 1 ml des Lysepuffers (siehe Tabelle 1). Zwischen den Mühlen in flüssigem Stickstoff abkühlen.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 10 mg/ml CHX (Cycloheximid) | 220 | 0,5 mg/ml |
| 1M Tris-HCl pH 8,0 | 88 | 20 mM |
| 3 Mio. KCl | 205.7 | 140 mM |
| 1 Mio. mgCl2 | 26.4 | 6 mM |
| 1 Mio. PMSF | 4.4 | 1 mM |
| NP-40 | 4.4 | 0.10% |
| Proteasehemmer | 2 Tabletten | |
| DNase I | 8.8 | 0,02 U/ml |
| Letzter Band | 4,400 |
Tabelle 1: Rezept für den Lysepuffer-Mastermix.
HINWEIS: Der Lysepuffer kann geändert werden, um mehr Proteasehemmer (wie Bestatin, Leupeptin, Aprotinin usw.) zu enthalten, falls das interessierende Protein sehr instabil ist, aber es ist wichtig, EDTA zu vermeiden, um die kleinen und großen Untereinheiten des Ribosoms zu erhalten, die während der folgenden Schritte zusammengesetzt sind. Aus ähnlichen Gründen mindestens 6 mMMgCl2 in der Pufferlösung halten.
ACHTUNG: HCl ist stark korrosiv und PMSF ist giftig. Tragen Sie Handschuhe und behandeln Sie sie mit Sorgfalt.
- Zentrifüte für 2 min bei 30.000 x g, 4 °C, um das Lysat zu reinigen und Überstände zu sammeln.
4. Reinigung von Ribosomen-Naszenzketten-Komplexen für SeRP
- Teilen Sie für jedes Experiment den Überstand in zwei Teile; jeweils in einem anderen Mikrozentrifugenröhrchen: Gesamt-RNA-Probe (~ 200 μL) und Immunreinigungsprobe (IP) (~ 700 μL) Translatomproben.
-
Verarbeitung der gesamten RNA-Probe
- Aufschluss der gesamten RNA-Probe unter Verwendung von 10 HE RNase I für 25 min bei 4 °C; Drehen Sie sich mit einem rotierenden Mix-Rack mit 30 U / min.
HINWEIS: Die Aufschlussbedingungen können mithilfe von Polysomenprofilen kalibriert werden, um sicherzustellen, dass die Monosomenspitze nicht über- oder unterverdaut wird. - Bereiten Sie die Saccharose-Kissen-Mastermischung wie in Tabelle 2 beschrieben vor.
- Aufschluss der gesamten RNA-Probe unter Verwendung von 10 HE RNase I für 25 min bei 4 °C; Drehen Sie sich mit einem rotierenden Mix-Rack mit 30 U / min.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 50% Saccharose | 200 | 25% |
| 1M Tris-HCl pH 8,0 | 8 | 20 mM |
| 3 Mio. KCl | 18.7 | 140 mM |
| 1 Mio. mgCl2 | 4 | 10 mM |
| 100 mg/ml CHX | 0.4 | 0,1 mg/ml |
| Proteasehemmer | 1 Tablette | |
| Letzter Band | 400 |
Tabelle 2: Rezept für Saccharose-Kissen-Mastermix.
- Laden Sie die Probe auf 400 μL des Saccharosekissens und der Zentrifuge in einem TLA120-Rotor für 90 min bei 245.000 x g und bei 4 °C.
- Entfernen Sie den Überstand schnell mit einer Vakuumpumpe und überlagern Sie Pellets mit einem 150 μL Lysepuffer. Resuspendieren Sie die Pellets durch Schütteln für 1 h bei 4 °C und bei 300 U / min.
- Resuspendiert das Restpellet durch Pipettieren und Transfer in ein neues 1,5-ml-Rohr.
HINWEIS: 100-200 μg Gesamt-RNA sind normalerweise ausreichend für die Ribosomenprofilierung des Gesamttranslatoms. Man kann den rRNA-Depletionsschritt hinzufügen, um die rRNA-Kontamination zu reduzieren, die die häufigste Verunreinigung der Ribosomen-naszierenden Kettenkomplexe Affinitätsreinigungen11 ist (siehe Diskussion für weitere Details).
-
Verarbeitung der Immunreinigungsprobe
- Waschen Sie 100-400 μL der affinitätsbindenden Matrix (1:1 Antikörper-konjugierte Kügelchen in 70% EtOH) pro Probe mit 3 x 1 ml Lysepuffer (ohne DNase I und Proteasehemmer); Schwebe die Affinitätsmatrix im Lysepuffer und drehe sie dann bei 30 U/min mit einem rotierenden Mix-Rack bei 4 °C für 5 min. Ausfällung durch Zentrifugation für 30 s bei 3.000 x g, 4 °C. Entsorgen Sie die obere Flüssigkeit. Wiederholen Sie dies dreimal.
- Aufschluss von Immunreinigungsproben mit 10 HE pro A260-nm-Einheit RNase I, zusammen mit einer affinitätsbindenden Matrix (z. B. 100-400 μL GFP-TRAP pro Probe).
- 25 min bei 30 U/min mit einem rotierenden Mix-Rack drehen, um das Protein bei 4 °C an die Affinitätsmatrix zu binden.
- Bereiten Sie die Waschpuffer-Mastermischung wie in Tabelle 3 beschrieben vor.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 10 mg/ml CHX | 50 | 0,1 mg/ml |
| 1M Tris-HCl pH 8,0 | 100 | 20 mM |
| 3 Mio. KCl | 233 | 140 mM |
| 1 Mio. mgCl2 | 50 | 10 mM |
| 1 Mio. PMSF | 5 | 1 mM |
| NP-40 | 0.5 | 0.01% |
| Proteasehemmer | 2 Tabletten | |
| 50% Glycerin | 1,000 | 10% |
| Letzter Band | 5,000 |
Tabelle 3: Rezept für die Waschpuffer-Mastermischung.
- Waschen Sie die affinitätsbindende Matrix dreimal mit 1 ml Waschpuffer, jedes Mal für ~ 1 min, und drehen Sie sich im Mix-Rack bei 30 U / min bei 4 ° C.
- Ausfällung durch Zentrifugation bei 3.000 x g für 30 s bei 4 °C. Entsorgen Sie die obere Flüssigkeit.
- Noch zweimal in 1 ml Waschpuffer waschen, jedes Mal für 5 min, im Mix-Rack bei 30 U / min bei 4 ° C drehen.
- Ausfällung durch Zentrifugation für 30 s bei 3.000 x g und 4 °C.
- Verwenden Sie 50 μL Perlen für die Proteinelution mit der gleichen Menge an 2x Probenpuffer. Verwenden Sie den Rest der Perlen für die RNA-Extraktion.
- Zentrifuge für 30 s bei 3.000 x g, 4 °C, um die Perlen zu pelletieren und die obere Flüssigkeit zu entsorgen.
- In flüssigem Stickstoff einfrieren und bei -80 °C lagern. Verwenden Sie diese Proben für die anschließende RNA-Extraktion.
HALTEPUNKT: Proben können über Nacht oder länger bei -80 °C gelagert werden. Dies kann ein Haltepunkt sein. - Beurteilen Sie den Erfolg des Affinitätsreinigungsschrittes durch Western Blot oder Coomassie Färbung mit Aliquots (~ 10% nach Volumen, nach dem Mischen) jedes Schrittes. Verwenden Sie immer Mock-IP auf einer nicht getaggten WT-Dehnung als Kontrolle für unspezifische Bindung an die Affinitätsmatrix.
HINWEIS: Hohe unspezifische Hintergründe können durch zusätzliche Waschschritte mit steigenden Salz-/Waschmittelkonzentrationen überwunden werden. Die transiente Interaktion kann durch die Behandlung verschiedener Vernetzungsmittel stabilisiert werden, z. B. die Behandlung lebender Zellen mit Paraformaldehyd (PFA) - die Zugabe von 0,4% -1% PFA zu den Wachstumsmedien für 2-5 min, gefolgt von Glycin (0,3 M) Abschreckung für 3 min, wird dringend empfohlen.
ACHTUNG: Paraformaldehyd ist ein mutmaßliches Karzinogen. Da Paraformaldehyd schnell verdunstet und korrosiv ist, arbeiten Sie in einer chemischen Sicherheitshaube und tragen Sie zwei Lagen Handschuhe.
5. Vorbereitung der cDNA-Bibliothek für die Tiefensequenzierung
- RNA-Extraktion
HINWEIS: Arbeiten Sie mit RNase-freien Antihaft-1,5-ml-Röhrchen, um eine mögliche RNA- oder DNA-Erschöpfung zu verhindern.- Die Proben aus den Schritten 4.2.5 und 4.3.12 werden auf Eis aufgetaut und die Proben mit 10 mM Tris-HCl pH 7.0 auf ein Endvolumen von 700 μL resuspendiert.
ACHTUNG: Säurephenol und Chloroform sind flüchtig und schädlich. Arbeiten Sie in einer Chemikaliensicherheitshaube. - Fügen Sie 40 μL 20% SDS zu 0,7 ml Gesamt-RNA- oder IP-Elutions hinzu. Schließen und umkehren Sie einige Male. Die Proteinfällung sollte die Proben weiß machen.
- 0,75 ml vorgewärmtes Säurephenol:Chloroform in die Proben geben. Die Rohre dicht verschließen und in einem Thermomischer bei 1.400 U/min 5 min und bei 65 °C schütteln. Proben 5 min auf Eis kühlen.
- Das Röhrchen aus Schritt 5.1.3 zentrifugieren. bei 20.000 x g für 2 min. Die oberste wässrige Schicht in ein frisches Röhrchen geben und 0,7 ml Säurephenol:Chloroform hinzufügen.
- Inkubieren Sie für 5 Minuten bei Raumtemperatur und wirbeln Sie gelegentlich. Zentrifuge für 2 min bei 20.000 x g. Die oberste wässrige Schicht in ein frisches Rohr geben und 0,6 ml Chloroform und Wirbel hinzufügen.
- Zentrifuge für 1 min bei 20.000 x g. Übertragen Sie die obere wässrige Schicht auf ein frisches Röhrchen.
- Nukleinsäuren durch Zugabe von 78 μL 3 M NaOAc, pH 5,5, 2 μL GlycoBlue und 0,75 ml Isopropanol ausfallen. Wirbel gründlich für 5 min. Inkubieren Sie mindestens 1 h bei -80 °C oder 16 h bei -20 °C.
- 30 min bei 20.000 x g und bei 4 °C zentrifugieren und den Überstand entsorgen. Waschen Sie die Pellets mit eiskalten 0,75 ml 80% Ethanol. Drehen Sie die Rohre für eine gründliche Wäsche um. Bei 20.000 x g für 5 min bei 4 °C zentrifugieren und dann den Überstand verwerfen.
- Bei 450 x g, 4 °C für 20 s nach unten drehen und das restliche Ethanol entfernen und die Flüssigkeiten entsorgen. Trocknen Sie das Pellet mit einem offenen Deckel für 5 min bei 65 °C. Resuspendieren Sie die Proben wie folgt: für IP resuspendiert die Probe in 10 μL von 10 mM Tris-HCl, pH 7,0. Für die Gesamttranslatomanalyse wird die Probe in 20 μL von 10 mM Tris-HCl, pH 7,0, resuspendiert.
HALTEPUNKT: RNA kann monatelang bei -80 °C gelagert werden.
- Die Proben aus den Schritten 4.2.5 und 4.3.12 werden auf Eis aufgetaut und die Proben mit 10 mM Tris-HCl pH 7.0 auf ein Endvolumen von 700 μL resuspendiert.
- Quantifizierung der gesamten RNA-Konzentration durch Fluorometrie
HINWEIS: Alle folgenden Materialien und Oberflächen sollten RNase-frei sein, während die cDNA-Bibliothek für die Sequenzierung der nächsten Generation vorbereitet wird. Tragen Sie beim Umgang mit RNA-Proben Handschuhe.- Verdünnen Sie 1 μL Säurephenol-extrahierte Gesamt-RNA in 9 μL von 10 mM Tris-HCl, pH 7,0. Quantifizieren Sie mit einem Fluorometer, wie auf der Website des Herstellers angegeben.
- Verdünnen Sie die Proben, die 50 μg RNA enthalten, mit 10 μL von 10 mM Tris-HCl, pH 7,0.
HINWEIS: Messen Sie keine IP-Proben, sondern verwenden Sie alles für den nächsten Schritt.
- Ribosomen-geschützte Fußabdruckfragmente mit Gelreinigung
- Setzen Sie ein 15% TBE-Harnstoff-Polyacrylamid-Gel und tauchen Sie in 1x TBE-Laufpuffer ein. 30 Minuten lang bei 200 V laufen, bevor die Probe geladen wird. Zu jeder Probe fügen Sie 20 μL 2x TBE-Harnstoff-Probenpuffer hinzu.
HINWEIS: Die erwartete Bandgröße liegt bei 25-35 nt. - Tauen Sie eine 10 bp DNA-Leiter auf und denaturieren Sie Proben (nicht Leiter) bei 80 ° C für 2 Minuten und kühlen Sie dann auf Eis. Laden Sie jede Probe auf jede andere Spur. Lassen Sie das Gel für 50-70 min bei 200 V laufen.
- Verdünnen Sie 6 μL SYBR Gold (10.000 x Konzentrat) in 60 ml 1x TBE-Puffer und färben Sie, während Sie in lichtgeschützten Boxen für 15-20 min schütteln. Bereiten Sie beim Färben des Gels ein steriles Skalpell und 0,5 ml Gelbrecherröhrchen in etikettierten 1,5 ml Röhrchen vor.
- Entfernen Sie die gewünschten Bänder mit einem sterilen Skalpell (verwenden Sie ein frisches oder reinigen Sie es gut zwischen den Proben) und legen Sie jedes Gelstück in ein Gelbrecherröhrchen.
- Machen Sie ein Bild des Gels, um sicherzustellen, dass keine Probenrückstände im Gel verbleiben.
- Die Röhrchen mit geschnittenen Scheiben bei 20.000 x g zentrifugieren Sie 5 min bei 4 °C und geben die restlichen Gelstücke aus dem Gelbrecherröhrchen in das 1,5 ml Röhrchen.
- Fügen Sie 0,5 ml von 10 mM Tris, pH 7,0 hinzu. In einem Thermomischer bei 1.400 U/min 10 min bei 70 °C schütteln.
- Transfer in eine Celluloseacetatsäule mit breiter Pipettenspitze und Zentrifuge bei 20.000 x g für 3 min bei 4 °C.
- Übertragen Sie den Durchfluss auf ein neues 1,5-ml-Rohr und kühlen Sie es auf Eis.
- Um die Nukleinsäuren auszufällen, fügen Sie hinzu: 550 μL IPA, 55 μL 3 M NaOAc und 2 μL GlycoBlue und Vortex zum gründlichen Mischen. Legen Sie die Proben für mindestens 1 h auf -80 °C.
- Zentrifen Sie für mindestens 1 h bei 20.000 x g und 4 °C und entsorgen Sie den Überstand. Waschen Sie die Pellets mit 0,75 ml eiskaltem 80% Ethanol. Drehen Sie die Rohre für eine gründliche Wäsche um, bis sich die Pellets vom Boden trennen. Zentrifugieren Sie erneut bei 20.000 x g für 5 min bei 4 °C und entsorgen Sie den Überstand.
- Bei 450 x g, 4 °C für 20 s nach unten drehen und das restliche Ethanol entfernen. Trocknen Sie die Pellets mit einem offenen Deckel für 5 min bei 65 °C.
- Fügen Sie 15 μL von 10 mM Tris, pH 7,0 hinzu und resuspendieren Sie die Pellets gründlich. Bei 450 x g, 4 °C für 20 s nach unten drehen und die Probe in ein neues 1,5-ml-Röhrchen überführen.
HALTEPUNKT: Gereinigte RNA kann einige Monate bei -80 °C gelagert werden.
- Setzen Sie ein 15% TBE-Harnstoff-Polyacrylamid-Gel und tauchen Sie in 1x TBE-Laufpuffer ein. 30 Minuten lang bei 200 V laufen, bevor die Probe geladen wird. Zu jeder Probe fügen Sie 20 μL 2x TBE-Harnstoff-Probenpuffer hinzu.
- Dephosphorylierung
- Verwenden Sie 3 μL der folgenden Mischung für jede Probe: Fügen Sie 1 μL RNase-Inhibitor in 2 μL 10x T4-Polynukleotidkinase-Reaktionspuffer ohne ATP hinzu. Geben Sie 2 μL T4-Polynukleotidkinase zu jeder Probe. Vorsichtig gut vermischen und 2 h bei 37 °C inkubieren, ohne zu schütteln.
- Um das Enzym zu inaktivieren, inkubieren Sie die Probe bei 75 °C für 10 min und drehen Sie sie bei 450 x g, 4 °C, für 20 s nach unten. Fügen Sie 0,5 ml von 10 mM Tris, pH 7,0 hinzu.
- Um Nukleinsäure auszufällen, fügen Sie 2 μL GlycoBlue, 550 μL IPA und 55 μL 3 M NaOAc hinzu.
- Vortex, um die Proben gründlich zu mischen und bei -80 °C für mindestens 1 h zu kühlen.
HALTEPUNKT: Proben können über Nacht oder länger bei -80 °C gelagert werden. - Wiederholen Sie die Schritte 5.3.11-5.3.13.
HALTEPUNKT: Dephosphorylierte RNA kann monatelang bei -80 °C gelagert werden.
- Quantifizierung mit einem Bioanalyzer
- Machen Sie eine 1: 4-Verdünnung jeder RNA-Probe, indem Sie 1 μL Probe und 4 μL DEPC-behandeltes Wasser mischen.
ACHTUNG: DEPC ist ein Karzinogen. Tragen Sie Handschuhe und arbeiten Sie sorgfältig. - Führen Sie einen Bioanalyzer Small RNA Chip/TapeStation aus. Befolgen Sie das Protokoll des Herstellers.
HINWEIS: Die erwartete Ribosomen-geschützte RNA-Fragmentgröße liegt bei etwa 28-30 nt.
- Machen Sie eine 1: 4-Verdünnung jeder RNA-Probe, indem Sie 1 μL Probe und 4 μL DEPC-behandeltes Wasser mischen.
- Ligate 3' Ende mit Linker-1
- Verdünnen Sie 5 pmol kleiner RNA-Fragmente auf 10 μL mit 10 mM Tris, pH 7,0. Denaturieren Sie die Proben bei 80 °C für 2 Minuten und kühlen Sie sie auf Eis.
- Bereiten Sie die Mastermischung wie in Tabelle 4 beschrieben vor und verwenden Sie 29 μL pro Probe.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 50% sterilfiltriertes PEG 8000 | 16 | 20% |
| DMSO | 4 | 10% |
| 10× T4 RNA Ligase 2 Puffer | 4 | 1x |
| SUPERase-In RNase-Inhibitor | 2 | 2 HE |
| 10 mM adenylierter Linker 3-L1 | 0.1 | 25 μM |
| DEPC-behandeltes Wasser | 2.9 | |
| Letzter Band | 29 |
Tabelle 4: Rezept für 3' End Ligation Master Mix.
- Fügen Sie 1 μL T4-RNA-Ligase 2 hinzu und pipettieren Sie vorsichtig, um gut zu mischen. Bei 23 °C für 2 h inkubieren.
- Um die Nukleinsäuren auszufällen, fügen Sie hinzu: 550 μL IPA, 500 μL 10 mM Tris, pH 7,0, 55 μL 3 M NaOAc und 2 μL GlycoBlue. Vortex gründlich mischen und die Proben mindestens 1 h bei -80 °C platzieren.
HALTEPUNKT: Lagern Sie die Proben über Nacht oder länger bei -80 °C. - Wiederholen Sie die Schritte 5.3.2-5.3.12.
- Resuspendiert das Pellet in 6 μL von 10 mM Tris, pH 7,0. Bei 450 x g, 4 °C für 20 s nach unten drehen und die Probe in ein neues 1,5-ml-Röhrchen überführen.
HALTEPUNKT: Proben können monatelang bei -80 °C gelagert werden.
-
Gelreinigung von 3' verknüpften Fußabdrücken
- Setzen Sie ein 10% FSME-Harnstoff-Polyacrylamid-Gel ein und tauchen Sie in 1x TBE-Laufpuffer ein. 30 Minuten lang bei 200 V laufen, bevor die Probe geladen wird. Zu jeder Probe fügen Sie 6 μL 2x TBE-Harnstoff-Probenpuffer hinzu.
HINWEIS: Die erwartete Bandgröße liegt bei 71-73 nt. - Wiederholen Sie die Schritte 5.3.2-5.3.13.
HALTEPUNKT: Proben können monatelang bei -80 °C gelagert werden.
- Setzen Sie ein 10% FSME-Harnstoff-Polyacrylamid-Gel ein und tauchen Sie in 1x TBE-Laufpuffer ein. 30 Minuten lang bei 200 V laufen, bevor die Probe geladen wird. Zu jeder Probe fügen Sie 6 μL 2x TBE-Harnstoff-Probenpuffer hinzu.
-
Reverse Transkribieren von 3ʹ verknüpften Footprint-Fragmenten zur Generierung von ssDNA
- Bereiten Sie eine Mastermischung wie in Tabelle 5 beschrieben vor und verwenden Sie 3 μL pro Probe.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 10 mM dNTPs | 1 | 0,5 mM |
| 25 μM Linker L(rt) | 0.5 | 625 nM |
| DEPC-behandeltes Wasser | 1.5 | |
| Letzter Band | 3 |
Tabelle 5: Rezeptur für die Reverse-Transkriptionspuffer-Mastermischung vor der Denaturierung von Nukleinsäuren.
- Wirbeln Sie die Probe ein und drehen Sie sie nach unten.
- Proben bei 65 °C für 5 min inkubieren.
- Proben auf Eis kühlen.
- Bereiten Sie eine Mastermischung wie in Tabelle 6 beschrieben vor und verwenden Sie 6 μL pro Probe. Wirbeln Sie die Probe ein und drehen Sie sie nach unten. 1 μL Superscript III in jede Probe geben und vorsichtig pipettieren, um gut zu mischen und 30 min bei 50 °C zu inkubieren.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 5× FS-Puffer | 4 | 1x |
| SUPERase-In RNase-Inhibitor | 1 | 2 HE |
| DTT 0,1 Mio. | 1 | 5 mM |
| Letzter Band | 6 |
Tabelle 6: Rezept für den Reverse Transcription Buffer Master Mix nach der Denaturierung von Nukleinsäuren.
- Fügen Sie 2,3 μL von 1 N NaOH hinzu, wodurch RNA hydrolysiert und die umgekehrte Transkription gelöscht wird.
ACHTUNG: NaOH ist stark korrosiv. Tragen Sie Handschuhe und Augenschutz. - 15 min bei 95 °C inkubieren, bis die Probe rosa wird.
- Setzen Sie ein 10% FSME-Harnstoff-Polyacrylamid-Gel ein und tauchen Sie in 1x TBE-Laufpuffer ein. 30 Minuten lang bei 200 V laufen, bevor die Probe geladen wird. Zu jeder Probe fügen Sie 23 μL 2x TBE-Harnstoff-Probenpuffer hinzu.
HINWEIS: Die erwartete DNA-Bandgröße beträgt 115-117 nt. - Wiederholen Sie die Schritte 5.3.2-5.3.12.
- Resuspendiert das Pellet in 15 μL von 10 mM Tris, pH 8,0. Bei 450 x g, 4 °C für 20 s nach unten drehen und die Probe in ein neues 1,5-ml-Röhrchen überführen.
HALTEPUNKT: Proben können monatelang bei -80 °C gelagert werden.
-
ssDNA-Zirkularisation
- Bereiten Sie die folgende Mastermischung vor und laden Sie 4 μL pro Probe, wie in Tabelle 7 beschrieben.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| 10× CircLigase II Puffer | 2 | 1x |
| 5 M Betain (optional) | 1 | 0,25 m |
| 50 mM MnCl2 | 1 | 2,5 mM |
| Letzter Band | 4 |
Tabelle 7: Rezept für den ssDNA-Zirkularisierungs-Mastermix .
- 1 μL CircLigase II ssDNA Ligase in jede Probe geben und für 1 h bei 60 °C inkubieren.
HINWEIS: Die Effizienz dieses Schritts kann durch Zugabe von 1 μL CircLigase II ssDNA Ligase zu jeder Probe nach 1 Stunde Inkubation erhöht werden. - Inaktivieren Sie das Enzym durch Inkubation bei 80 °C für 10 min.
- Auf Eis kühlen und mit der PCR-Verstärkung fortfahren oder bei -80 °C lagern.
HALTEPUNKT: Proben können jahrelang bei -80 °C gelagert werden.
-
PCR-Verstärkung
- Bereiten Sie den folgenden PCR-Master-Mix vor und laden Sie 82 μL pro Probe, wie in Tabelle 8 beschrieben.
| Reagenz | Menge pro Probe (μL) | Endkonzentration |
| DEPC-behandeltes Wasser | 61.6 | |
| 5× Phusion HF Reaktionspuffer | 17.6 | 1x |
| 10 mM dNTPs | 1.8 | 200 μM |
| 100 μM PCR Vorwärtsprimer | 0.2 | 225 nM |
| HF-Phusionspolymerase | 0.8 | 1,6 HE |
| Letzter Band | 82 |
Tabelle 8: Rezept für PCR-Verstärkungs-Mastermix.
- Zu jedem Röhrchen, das die Mastermischung enthält, fügen Sie 5 μL zirkularisierte DNA hinzu.
HINWEIS: Lagern Sie den Rest der zirkularisierten DNA-Proben bei -80 °C. - Fügen Sie jeder Probe (siehe Tabelle 9) einen anderen 1 μL 20 μM PCR-Reverse-Barcode-Primer hinzu (siehe Tabelle 9) und wirbeln Sie, um gründlich zu mischen.
- Aliquot jede Röhre in vier separate PCR-Röhren, die jeweils für eine unterschiedliche Anzahl von PCR-Zyklen verwendet werden.
- Führen Sie eine PCR-Reaktion gemäß dem folgenden Programm aus, wie in Tabelle 10 beschrieben.
| Zyklus | Denaturierung (98 °C) | Glühen (60 °C) | Ausfahren (72 °C) |
| 1 | 30 Sek. | ||
| 2-16 | 10 s | 10 s | 5 s |
Tabelle 10: PCR-Programm für die PCR-Reaktion.
- Nach den Zyklen 8, 9, 10 und 11 werden PCR-Röhrchen (bei IP-Proben reichen die Zyklen von 9 bis 15) als ersten Versuch entfernt. Nach jedem Zyklus pausieren Sie das Programm, nehmen Sie ein Aliquot heraus und legen Sie es auf Eis, und setzen Sie das Programm dann schnell fort.
HINWEIS: Die Anzahl der Zyklen sollte basierend auf der Menge der zirkularisierten DNA in jeder Reaktion angepasst werden. Ein Beispiel und weitere Erläuterungen finden Sie in Abbildung 4. - Zu jeder 17-μL-Reaktion fügen Sie 3,5 μL 6x DNA-Belastungsfarbstoff hinzu.
- Tauen Sie eine 10 bp DNA-Leiter auf.
- Für die Größentrennung durch Gelelektrophorese tauchen Sie 8% FSME-Polyacrylamid in 1x TBE-Laufpuffer und laden die Proben jeder verschiedenen Zykluszahl in benachbarte Vertiefungen und lassen das Gel für 50 min bei 180 V laufen.
- Verdünnen Sie 6 μL SYBR Gold (10.000 x Konzentrat) in 60 ml 1x TBE-Puffer und färben Sie, während Sie in lichtgeschützten Boxen für 15-20 min schütteln.
- Bereiten Sie beim Färben des Gels ein steriles Skalpell und 0,5 ml Gelbrecherröhrchen in etikettierten 1,5 ml Röhrchen vor.
- Machen Sie ein Bild der gefärbten Nukleinsäuren.
- Schneiden Sie das gewünschte Band mit einer erwarteten Bandgröße von 174-176 bp mit dem sterilen Skalpell ab und legen Sie die Gelscheibe in das vorbereitete 0,5 ml Gelbrecherröhrchen (reinigen Sie gründlich zwischen den Proben und verwenden Sie RNase-Inaktivierungsmittel oder wechseln Sie zu einer neuen Klinge).
- Zentrifugieren Sie die Röhrchen für 5 min bei 20.000 x g und 4 °C und übertragen Sie dann die verbleibenden Gelstücke aus dem 0,5 ml Gelbrecherröhrchen in das 1,5 ml Rohr.
- 500 μL 10 mM Tris, pH 8,0 hinzufügen und in einem Thermomischer bei 1.400 U/min für 10 min und 70 °C schütteln.
- Übertragen Sie das gelöste Gel auf eine Celluloseacetatsäule mit einer breiten Pipettenspitze.
- Zentrifugieren Sie die Kolonne für 3 min bei 20.000 x g und 4 °C und übertragen Sie den Durchfluss auf ein neues 1,5-ml-Rohr und kühlen Sie es auf Eis.
- Um die Nukleinsäure auszufällen, fügen Sie hinzu: 550 μL IPA, 32 μL 5 M NaCl, 1 μL 0,5 M EDTA und 2 μL GlycoBlue und Vortex, um gründlich zu mischen.
- Bewahren Sie die Proben mindestens 1 h bei -80 °C oder -20 °C über Nacht auf.
HALTEPUNKT: Proben können über Nacht oder länger bei -80 °C gelagert werden. - Wiederholen Sie die Schritte 5.3.11-5.3.12.
- Resuspend in 11 μL von 10 mM Tris, pH 8,0. Bei 450 x g, 4 °C für 20 s nach unten drehen und die Probe in ein neues 1,5-ml-Röhrchen überführen.
HALTEPUNKT: Proben können jahrelang bei -80 °C gelagert werden.
-
Quantifizierung der Größenverteilung durch Bioanalyzer
- Nehmen Sie eine 1:4-Verdünnung jeder Probe vor, indem Sie 1 μL Probe mit 4 μL DEPC-behandeltem Wasser mischen.
- Führen Sie den Bioanalyzer Small RNA Chip aus. Befolgen Sie das Protokoll des Herstellers.
HINWEIS: Die erwartete Länge beträgt 175 ± 5 bp.
-
Quantifizierung der DNA-Konzentration durch Fluorometer
- Führen Sie eine dsDNA-Hochempfindlichkeitskonzentrationsprüfung mit einem Fluorometer gemäß den Empfehlungen des Herstellers durch.
- Multiplex- und Sequenzproben gemäß den Illumina-Empfehlungen (Index Adapters Pooling Guide12).
6. Datenanalyse
- Führen Sie die Analyse wie in der Zusatzdatei beschrieben durch.
Ergebnisse
Wie im Flussdiagramm dieses Protokolls dargestellt (Abbildung 1), wurden Zellen in die Log-Phase gezüchtet und dann schnell durch Filtration gesammelt und durch kryogenes Mahlen lysiert. Das Lysat wurde dann in zwei Teile geteilt: einen für die gesamten Ribosomen-geschützten mRNA-Fußabdrücke und den anderen für ausgewählte Ribosomen-geschützte mRNA-Fußabdrücke, an denen wir eine Affinitätsreinigung durchführten, um die markierten Protein-Ribosomen-naszierenden Kettenkomplexe herunterzuziehen. Wir haben die markierte Proteinexpression und den Erfolg des Pulldowns durch die Western-Blot-Analyse sichergestellt, wie in Abbildung 2 zu sehen ist. Wir validierten die Isolierung von Ribosomen-geschützten Fußabdrücken, die typischerweise 20-45 t lang sind, durch kleine RNA-Elektrophorese (2100 BioAnalyzer-System), was eine 5-10 nt Verschiebung der Größenerkennung gemäß dem Systemhandbuch ermöglicht (Abbildung 3). Dann generierten wir eine cDNA-Bibliothek für Deep Sequencing und Big-Data-Analyse. Beachten Sie beim Generieren der cDNA-Bibliothek, dass Undercycling zu einer geringen Ausbeute führen kann (wie in Bahn 2 in Abbildung 3 zu sehen ist), aber eine erneute Verstärkung ist möglich, um die generierte Bibliothek wiederherzustellen. Overcycling kann auftreten, wenn PCR-Primer erschöpft sind, aber die Reaktion anhält. Wenn noch dNTPs vorhanden sind, verläuft die Reaktion und erzeugt längere PCR-Artefakte mit chimären Sequenzen, da sich PCR-Produkte auf13 vorbereiten (wie in den Bahnen 3-4 in Abbildung 3 zu sehen ist, was durch den sichtbaren Abstrich angezeigt wird). Wenn auch die Konzentration der dNTPs einschränkend wird, können Produkte auftreten, die auf das Vorhandensein von Heteroduplexen hinweisen, die nur teilweise aus homologen Bibliotheksfragmenten bestehen. Abbildung 4 fungiert als Referenz, wobei Bahn 2 die optimale Verstärkung und Spur 3 eine akzeptable Verstärkung darstellt. Proben aus den Bahnen 4 und 5 (Zyklen 10 und 11) sollten aufgrund der Möglichkeit, PCR-Duplikate und Artefakte einzuführen, nicht verwendet werden. Die generierte Bibliothek wurde durch hochempfindliche DNA-Elektrophorese (das gleiche BioAnalyzer-System wurde verwendet) für die genaue Größenverteilung und Quantifizierung weiter validiert (Abbildung 5). Nach 3'-End-Linker-Ligatur, Reverse-Transkription und PCR-Amplifikation wird eine cDNA-Längenverteilung als solche erwartet, mit einem scharfen Peak um 175 nt.
Wir haben Adapter und Barcodes aus der sequenzierten Bibliothek beschnitten und entfernt, und nur die Lesevorgänge zwischen 20 und 45 NT wurden für die weitere Analyse ausgewählt. Abbildung 6 zeigt die resultierende Längenverteilung. Die Lesevorgänge wurden in verschiedene Gruppen unterteilt: Kodierungssequenzen, Introns und intergene Sequenzen (Abbildung 7) und weiter klassifiziert, wie in Abbildung 8 gezeigt.
Die endgültige Analyse zum Nachweis und zur Charakterisierung von co-translationalen Interaktionen wurde basierend auf der Anreicherung von Ribosomen-geschützten mRNA-Fragmenten durchgeführt, wodurch die Diagramme in Abbildung 9 erzeugt wurden. Wir verglichen die normalisierte Ribosomenbelegung (an jedem Nukleotid entlang jedes Orf) des Gesamttranslatoms mit seinem entsprechenden ausgewählten Translatom (Nasenz-Interactom). Der Vergleich pro Nukleotid eliminiert Artefakte der Translationsraten. Die Reproduzierbarkeit zwischen biologischen Replikaten wurde durch Pearson-Korrelation (Schwellenwert > 0,6) bewertet. Wir präsentieren selektive Ribosom-Profile und analysieren co-translationale Interaktionen von Vma2p mit drei Proteinen: dem Ribosomen-assoziierten Chaperon Ssb1p, Pfk2p (Phosphofructokinase) und Fas1p (Fettsäuresynthase), wobei jedes Protein C' von GFP endgültig markiert wird. Wir haben das Protokoll in biologischen Replikaten durchgeführt. Abbildung 9 A, D und G zeigen das experimentelle Schema jeder Affinitätsreinigung. Als nächstes zeigen wir die Ribosomenbelegung der gesamten Translatome im Vergleich zu Ssb1-Interactomen entlang des Vma2p-Orf, kodiert für eine Untereinheit der vakuolären H+-ATPase (Abbildung 9 B, E und H). Schließlich führten wir an jeder Ribosomenposition in [codon/aa] entlang des ORF ein ratiobasiertes Ribosomen-Enrichment-Profiling (IP/Total) durch (Abbildung 9 C, F und I). Der Vergleich der co-translationalen Wechselwirkungen dieser drei Proteine mit Vma2p, das vom Ribosom synthetisiert wird, ergab, dass Ssb1-Chaperon das entstehende Vma2p an vier verschiedenen Regionen entlang des Orf angreift, da wir vier signifikante Anreicherungspeaks durch SeRP identifiziert haben. Anders zeigt Pfk2p nur einen signifikanten Anreicherungspeak, wie von SeRP identifiziert, in einer anderen Position als das co-translationale Chaperon Ssb1. Die Analyse der co-translationalen Wechselwirkungen von Fas1 mit dem entstehenden Vma2p ergab keine signifikante Anreicherung. Der Vergleich dieser quotenbasierten Anreicherungsribosomenprofile zeigt somit die Leistungsfähigkeit dieses Protokolls bei der Erkennung und Charakterisierung verschiedener co-translationaler Wechselwirkungen in nahezu Codon-Auflösung.
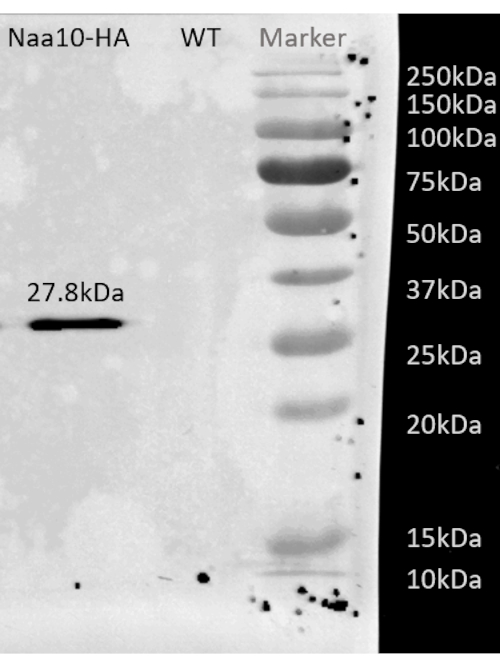
Abbildung 2: Repräsentatives Western-Blott-Ergebnis nach Affinitätsreinigung des BY4741-Stammes mit HA-markiertem Naa10. Repräsentatives Western-Blot-Ergebnis nach Affinitätsreinigung des BY4741-Stammes mit HA-markiertem Naa10, das ein Band um 27,8 kDa zeigt, während der Wild-Typ als Negativkontrolle kein Band zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Repräsentatives BioAnalyzer-Ergebnis nach Footprint-Isolierung und RNA-Extraktion mit Säurephenol:Chloroform und einer durchschnittlichen Größe von 25 nt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
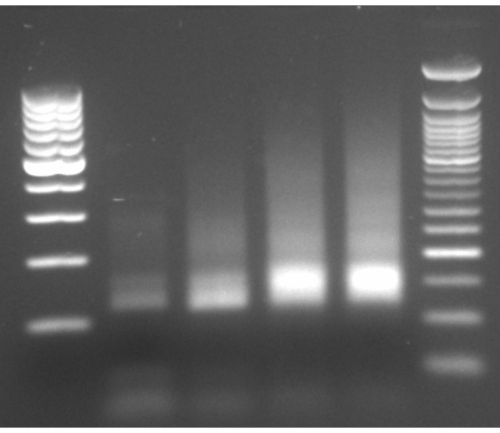
Abbildung 4: Repräsentative Gelelektrophorese der PCR-Amplifikation. Repräsentative Gelelektrophorese der PCR-Amplifikation mit Bahnen 2-5, die mit PCR-Produkten aus den Zyklen 8-11 beladen sind, und Leitern auf beiden Seiten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
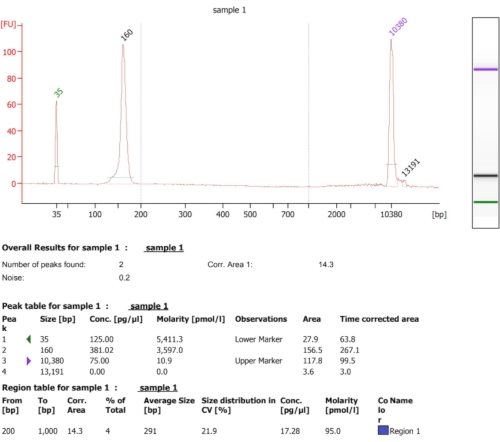
Abbildung 5: Repräsentatives BioAnalyzer-Ergebnis, das nach der Erstellung einer cDNA-Bibliothek erzielt wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 6: Erwartete Längenverteilung der Lesevorgänge nach dem Entfernen der Adapter mit Cutadapt (Entfernen von Lesevorgängen, die kürzer als 20 oder länger als 45 sind). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 7: Erwarteter Prozentsatz des Ausrichtungserfolgs nach dem Entfernen nicht-kodierender RNA-Lesevorgänge mit Bowtie2 und der Verwendung von TopHat zum Ausrichten der verbleibenden Lesevorgänge an verschiedene Organismen. Die Probe wurde aus S. cerevisiae (einer mutierten Variante von BY4741) entnommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
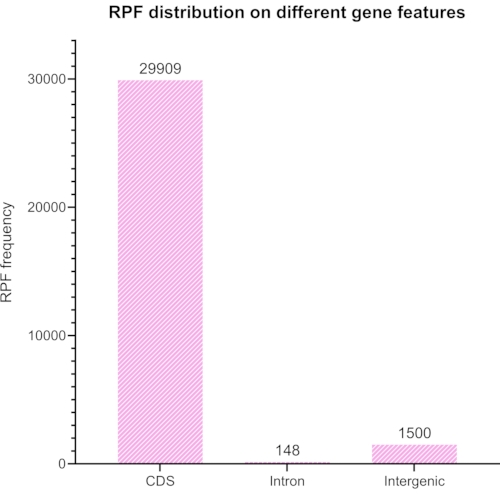
Abbildung 8: Ein mit RiboToolkit generiertes Diagramm, das das erwartete Verhältnis von Codierung zu Nichtcodierung der ausgerichteten Lesevorgänge nach der Verwendung von Bowtie2 zum Entfernen von rRNA-Elementen in den Lesevorgängen darstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
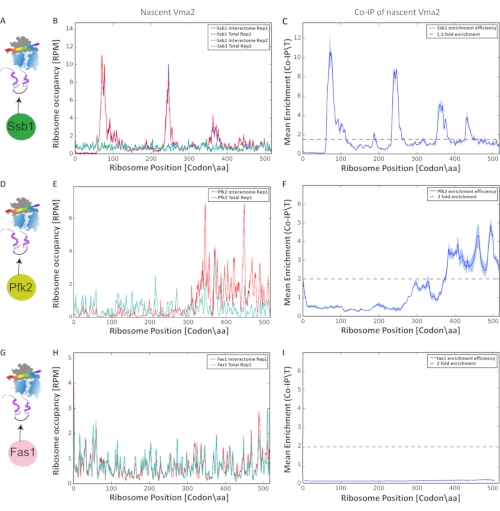
Abbildung 9: Co-translationale Wechselwirkungen von drei verschiedenen Proteinen: Ssb1p, Pfk2p und Fas1p mit Vma2p, das vom Ribosom synthetisiert und von SeRP analysiert wird. Alle y-Achsen werden in Lesevorgängen pro Million (RPM) angezeigt. (A, D, G) Experimentelles Schema von SeRP von Ssb1p, Pfk2p und Fas1p C', das jeweils von GFP endgültig markiert wurde. (B, E, H) Ribosomenbelegung entlang der Orf von Gesamttranslatomen im Vergleich zu Ssb1-, Pfk2p- bzw. Fas1p-Interaktomen (in biologischen Replikaten). (C, F, I) Mittlere Anreicherung von Ssb1p, Pfk2p und Fas1p (IP/Gesamtverhältnis) an jeder Ribosomenposition in [codon/aa] entlang des orf. Die Variation zwischen biologischen Replikaten wird durch den schattierten Bereich angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 9: 3' Linker- und Primersequenzen 3' Linker L1: Linker 3-L1 mit 5' Adenylierung und 3' Dideoxy-Cytidin unique molecular identifiers ('NN...') (RNase-freie HPLC-Reinigung; Reverse-Transkriptions-Linker: Reverse Transkription (L(rt)) mit 5' phosphorylierten, eindeutigen molekularen Identifikatoren (RNase-freie HPLC-Aufreinigung); PCR-Vorwärtsprimer: PCRf; HPLC gereinigt. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Datei. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Hier beschreibt das Protokoll den Selective Ribosome Profiling-Ansatz zur Erfassung co-translationaler Interaktionen in nahezu Codon-Auflösung. Da das Ribosom als Drehscheibe für die Koordination der Entstehung der entstehenden Kette in das überfüllte Zytoplasma aufsteigt, ist dies eine entscheidende Methode, um die verschiedenen co-translationalen Wechselwirkungen zu identifizieren und zu charakterisieren, die zur Gewährleistung eines funktionellen Proteoms sowie zur Untersuchung verschiedener Krankheiten erforderlich sind. Bis heute ist SeRP die einzige Methode, die diese Interaktionen in vivo14,15,16 direkt erfassen und charakterisieren kann.
Der erste und kritischste Schritt ist die Zellsammlung und Lyse. Es ist unerlässlich, innerhalb von Sekunden die fortlaufende Übersetzung durch Einfrieren von Flammen zu erfassen, gefolgt von einer Lyse in einem eingefrorenen Zustand. Die Zellentnahme muss mit Eile erfolgen, um einen ribosomalen Abfluss zu vermeiden und Stresstranslationsreaktionen zu induzieren, die schnell auftreten können. Der zweite kritische Schritt ist der Affinitätsreinigungsschritt. Es ist unerlässlich, die Hintergrundbindung durch stringentes Waschen zu reduzieren und gleichzeitig sicherzustellen, dass die co-translationalen Wechselwirkungen erhalten bleiben, was durch In-vivo-Vernetzung erleichtert werden kann. Da dieses Protokoll auf dem hochempfindlichen NGS (Next Generation Sequencing) basiert, kann ein hoher Hintergrund in den ersten Schritten in den folgenden cDNA-Bibliotheksvorbereitungsschritten verstärkt werden, was zu einem niedrigen Signal-Rausch-Verhältnis führt.
Die Nuklasbehandlung, um alle nicht geschützten mRNAs zu verdauen, sollte durch Polysomenprofiling17 zusammen mit einer sorgfältigen Bewertung der isolierten ribosomalen Fußabdrücke Größenverteilung (wie oben beschrieben) bewertet werden, um eine Über- oder Unter-RNA-Verdauung zu vermeiden. Die Kalibrierung der Nuklaspenkonzentration und der Verdauungszeiten kann eine genaue Wiederherstellung des Fußabdrucks erleichtern, da eine Überverdauung zu einer ribosomalen rRNA-Verdauung führen kann, was zum Verlust von Ribosomen-geschützten Fußabdrücken führt. Es ist wichtig zu beachten, dass eine Unterverdauung auch zu niedrigeren Entdeckungsraten von Ribosomen-geschützten Fußabdrücken führen kann, da die hier beschriebenen Schritte zur Vorbereitung der cDNA-Bibliothek sowie die hier beschriebenen Datenanalyseschritte lange, uncharakteristische Lesevorgänge verwerfen.
Während die rRNA-Erschöpfung nicht immer einen kritischen Schritt darstellt und nicht obligatorisch ist, hat sie einige Vorteile wie sauberere Proben und daher eine höhere Rate an genomkartierten Lesevorgängen. Auf der anderen Seite besteht die Möglichkeit von Verzerrungen, da viele rRNA-Depletionsprotokolle auch zu einer Erschöpfung der gewünschten Ribosomen-geschützten Fragmente führen können. Man sollte auch die Kosten der rRNA-Depletion-Kits berücksichtigen. Die rRNA-Depletion kann nach dem Ribosomen-Fuß-Druck-Isolationsschritt oder nach dem cDNA-Zirkularisierungsschritt durchgeführt werden.
Die hier beschriebenen Schritte zur Vorbereitung der cDNA-Bibliothek wurden für einen niedrigen mRNA-Input optimiert, da die Affinitäts- und Ribosomenreinigungsschritte die mRNA-Eingangsmenge im Vergleich zu RNA-Seq-Expressionsstudien stark reduzieren. Das Hochskalieren der anfänglichen Menge an Zellkulturen kann die Generierung von cDNA-Bibliotheken erheblich erleichtern. Alternativ kann jedes cDNA-Bibliotheksprotokoll der Wahl mit den hier beschriebenen Schritten zur Affinitätsreinigung und Fußabdruckisolierung passen. Es ist wichtig zu beachten, dass die Nuclease-Behandlung, die die ribosomalen Fußabdrücke erzeugt, die resultierende mRNA-Endreparatur (cDNA-Bibliotheksvorbereitung, Dephosphorylierungsschritt) erfordert, um die folgenden Linker-Ligationsschritte in dem hier beschriebenen cDNA-Protokoll in Ihrem Protokoll Ihrer Wahl zu ermöglichen.
Während der Sequenzierung ist es wichtig, SeRP von RNA-Seq zu unterscheiden, da die Heterogenität der generierten Bibliotheken stark variiert, abhängig von den Affinitäts-markierten Faktoren. Molekulare Chaperone und Targeting-Faktoren sind bei der Bindung oft promiskuitiver und interagieren während der Translation mit Hunderten oder Tausenden von Substraten, was zu sehr unterschiedlichen cDNA-Bibliotheken führt. Hochspezifische Interaktoren, wie z.B. co-translationale komplexe Assemblierungsinteraktoren, können jedoch oft zur Generierung von viel weniger unterschiedlichen cDNA-Bibliotheken führen. Ein Anstieg der verschiedenen und nicht unterschiedlichen Bibliotheken auf derselben Spur kann die Sequenzierung und das Verfolgen von Datenanalyseergebnissen erheblich verbessern.
Ein weiteres einzigartiges Merkmal von SeRP ist seine Fähigkeit, lokale Variationen der Ribosomenbelegungen entlang des ORF zu erfassen, was die Entdeckung ribosomaler Verschiebungen der Translationsrate ermöglicht, die mit jedem Satz von Interaktionen verbunden sind. Es ist daher unerlässlich, die Ribosomenbelegungen in jedem Codon entlang des Orf zu vergleichen, um die Anreicherung korrekt zu identifizieren. Die Verwendung von ORFS-Durchschnittswerten kann zum Verlust vorübergehender Interaktionen oder zu falschen Entdeckungen führen.
Die korrekte Anwendung der SeRP-Methode öffnet viele co-translationale Wege zur direkten Analyse und entdeckt neue mechanistische Merkmale sowie neuartige Ribosomen-assoziierte Faktoren, die das Protein-Biosynthese-Feld revolutionieren.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir danken allen Labormitgliedern für die fruchtbaren Diskussionen und Muhammad Makhzumy für die kritische Lektüre des Manuskripts. Diese Arbeit wurde durch den ISF (Israeli Science Foundation) Grant 2106/20 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * - for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referenzen
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23(2012).
- Guide, P. Illumina Index Adapters - Pooling Guide. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019).
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820(2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten