Method Article
Imagem de super-resolução para estudar co-localização de proteínas e marcadores sinápticos em neurônios primários
Neste Artigo
Resumo
Este protocolo mostra como empregar microscopia de super-resolução para estudar a co-localização de proteínas em culturas neuronais primárias.
Resumo
Sinapses são os elementos funcionais dos neurônios e seus defeitos ou perdas são a base de várias doenças neurodegenerativas e neurológicas. Estudos de imagem são amplamente utilizados para investigar sua função e plasticidade em condições fisiológicas e patológicas. Devido ao seu tamanho e estrutura, estudos de localização de proteínas requerem técnicas de imagem de alta resolução. Neste protocolo, descrevemos um procedimento para estudar em neurônios primários a co-localização de proteínas-alvo com marcadores sinápticos em um nível de super-resolução utilizando microscopia de iluminação estruturada (SIM). SIM é uma técnica de iluminação de luz padronizada que dobra a resolução espacial da microscopia de campo largo, atingindo um detalhe de cerca de 100 nm. O protocolo indica os controles e configurações necessários para estudos robustos de co-localização e uma visão geral dos métodos estatísticos para analisar adequadamente os dados de imagem.
Introdução
A compreensão e visão da sinapse mudou enormemente desde sua primeira descrição por1Foster e Sherrington em 18971 . Desde então, nosso conhecimento da comunicação neuronal e dos processos moleculares por trás dela tem crescido exponencialmente2. Tornou-se claro que as sinapses podem ser consideradas como um sistema de dois compartimentos: um compartimento pré-sináptico contendo vesículas para a liberação de neurotransmissores e um compartimento pós-sináptico com receptores3. Essa visão simplista, nos últimos vinte anos, evoluiu para uma complexa rede de proteínas necessárias para transdutor de transmissão ligado à sinalização4.
Os ganhos no entendimento se devem, em parte, às técnicas de super-resolução que superaram o limite de difração da microscopia de luz convencional para se adequar melhor à dimensão das sinapses5,,6,,7,,8,,9,,10. Devido ao limite de difração, um microscópio óptico não pode atingir uma resolução acima de 200 nm lateralmente11,12. Para contornar esse limite, foram criadas técnicas de super-resolução, utilizando diferentes abordagens e atingindo diferentes resoluções de limite de subditração: SIM, STED (Estimulada Microscopia de Esgotamento de Emissões), PALM (Microscopia de Localização FotoAtivada) e STORM (Microscopia óptica de Reconstrução Estocástica)13,14. O SIM dobra a resolução espacial de sistemas de microscopia de campo largo baseados em laser inserindo uma grade de difração no feixe de excitação15. A grade móvel difunde os raios laser para criar um padrão de iluminação conhecido, geralmente listras. Este padrão de luz estruturado propositalmente é sobreposto à distribuição espacial desconhecida do corante fluorescente (da amostra). As franjas de interferência formadas pelos dois padrões codificam para detalhes finos indistinguíveis com microscopia de campo largo normal. A imagem super-resolvida final é obtida pela combinação e decodificação com métodos matemáticos várias imagens brutas da mesma amostra obtidas pelas traduções e rotações da grade de difração. A resolução das imagens super-resolvidas atinge 100 nm na lateral e 500 nm nas direções axial para 2D-SIM15 ou 100 nm na lateral e 250 nm nas direções axial para 3D-SIM16.
O novo entendimento da sinapse é ainda mais importante à luz das muitas doenças neurológicas onde a disfunção sináptica desempenha um papel importante no início e progressão17,18. Doença de Alzheimer, Síndrome de Down, Doença de Parkinson, doenças de príon, epilepsia, transtornos do espectro autista e síndrome do X frágil, entre outros, têm sido associadas a anormalidades na composição sináptica, morfologia e função19,,20,,21,22.
Recentemente, utilizando um conjunto de anticorpos específicos do SUMH, usamos o SIM para mostrar a co-localização nos neurônios hipocampais primários das proteínas SUMO com os marcadores pré e pós-sináptico sinaptophysin e PSD95 no nível de super resolução23. Isso nos permitiu confirmar evidências de microscopia bioquímica e confocal da localização do SUM nos neurônios.
Aqui, descrevemos um protocolo para estudar a localização de proteínas em neurônios primários hipocampais de camundongos. Ao mesmo tempo, este protocolo pode ser adaptado a diferentes tipos de culturas neuronais primárias.
Protocolo
1. Culturas primárias
- Neurônios primários hipocampais de rato de cultura em tampas câmaras (como Ibidi μ-Slide 8 Bem ou Nunc Lab-Tek Chambered Coverglass) que correspondem ao requisito objetivo para #1,5 (0,17 mm) de espessura de deslizamento.
- Tampas de câmara de revestimento com 100 μL de poli-L-lisina (100 μg/mL).
- No dia seguinte, lave as tampas câmara duas vezes com soro fisco estéril tamponado (PBS).
- Para obter neurônios primários do camundongo, isole o hipocampi dos filhotes P1-P423.
- Coloque hipocampi dissecado em 10 mL de Mídia de Dissecção (Tabela 1) e deixe-os depositar na parte inferior do tubo.
- Usando uma pipeta estéril, remova cuidadosamente a Mídia de Dissecção, deixando o hipocampi intacto na parte inferior do tubo.
- Adicione 10 mL de Media 1 (Tabela 1) ao hipocampi e incubar por 30 minutos a 37 °C.
- Usando uma pipeta estéril, remova cuidadosamente a Media 1, deixando o hipocampi intacto na parte inferior do tubo.
- Adicione 10 mL de Media 2(Tabela 1) e deixe o tubo de centrífuga (tampado) sob o capô horizontalmente por 45 minutos.
- Deixe o tubo de centrífugas ficar verticalmente para permitir que o tecido se instale na parte inferior do tubo.
- Usando uma pipeta estéril, remova cuidadosamente a Media 2, deixando o hipocampi intacto na parte inferior do tubo centrífuga.
- Adicionar 2 mL de Media 3(Tabela 1).
- Usando uma pipeta p1000 com ponta filtrada, dissociar mecanicamente células do tecido.
- Transfira o supernante, no qual estão localizados os neurônios isolados, para um tubo de centrífuga de 15 mL.
- Centrifugar a suspensão celular por 2 min a 300 x g em temperatura ambiente (RT).
- Após a centrifugação, as células estão localizadas na parte inferior do tubo de centrífuga. Usando uma pipeta estéril, descarte o supernaspeso.
- Células resuspend em 1 mL de Media 4.
- Use um filtro de 70 μm para eliminar células não dissociadas.
- Conte células viáveis em uma câmara de Bürker adicionando 1 μL de solução azul trypan de 0,4 % a 19 μL da suspensão celular.
- Células de placa a 70.000 células/bem em um volume de 200 μL por poço.
- Deixe as células anexar por 2h em uma incubadora umidificada a 37 °C e 5% de CO2.
- Retire as tampas câmaradas da incubadora e substitua cuidadosamente o meio por 200 μL de Mídia de Cultura.
- Deixe as tampas em uma incubadora umidificada a 37 °C e 5% de CO2.
- Substitua um terço do meio por novas mídias culturais a cada 5-7 dias.
- Espere até que os neurônios primários hipocampais estejam totalmente maduros (12-14 dias após o revestimento) para realizar estudos de co-localização.
2. Coloração de imunofluorescência
- Pegue as tampas de câmara da incubadora.
- Remova o meio.
- Lave rapidamente os poços com 200 μL de PBS.
- Adicione 4% de paraformaldeído (PFA) em PBS (200 μL/bem) aos neurônios para corrigi-los rapidamente.
- Incubar as células por 15 minutos na RT.
- Remova a solução PFA.
- Permeabilize as células adicionando PBS com Triton X-100 (200 μL/well).
- Incubar por 1 min na RT.
- Remova a solução e incuba as amostras com 1% de albumina de soro bovino (BSA) em PBS (200 μL/well) por 1 h em RT para cobrir passivamente todas as superfícies de ligação livre da placa com uma proteína irrelevante para a análise. Um buffer de bloqueio baseado em BSA sem Triton X-100 reduz o fundo do anticorpo de forma mais eficiente do que o mesmo buffer com Triton X-100 de 0,2%.
- Remova a solução.
- Adicione o anticorpo primário de escolha diluído em uma solução PBS contendo 1% BSA e 0,2% Triton X-100 (120-200 μL/bem, dependendo da diluição e disponibilidade de anticorpos). Incubar as amostras por 2h.
- Como controle negativo, não adicione nenhum anticorpo primário a um dos poços. Vários anticorpos de diferentes espécies contra alvos diferentes podem ser usados ao mesmo tempo. Use um anticorpo contra MAP2 (um marcador neuronal) criado em frango, um anticorpo contra PSD95 ou sinaptofisina criada em camundongos, e um anticorpo contra uma proteína alvo levantada em coelho. Isso permite análises sim de três cores.
- Lave rapidamente os poços três vezes com PBS (200 μL/well).
- Adicione anticorpos secundários (anticorpos secundários dyLight e Alexa podem ser ambos diluídos) diluídos em uma solução PBS contendo 1% BSA e 0,2% Triton X-100 (200 μL/well). Incubar as amostras por 1h na RT.
- Lave rapidamente os poços três vezes com PBS (200 μL/well).
- Adicione corante Hoechst a uma concentração de 1 μg/mL diluída em PBS (200 μL/well) para colorir núcleos. Incubar as amostras por 10 minutos na RT.
- Lave rapidamente os poços duas vezes com PBS.
- Monte células usando um montador compatível com SIM. Use 10 μL/well de ProLong Glass Antifade Mountant.
- Cubra e proteja as células com um óculos de cobertura (por exemplo, um copo de cobertura redondo com um diâmetro de 8 mm). Os quadrados também podem ser usados.
- Armazene as tampas em câmara no RT e espere pelo menos 48h antes de adquirir imagens. A Diamond Glass requer pelo menos dois dias de cura antes das aquisições de super resolução.
3. Controle de especificidade de anticorpos
NOTA: Use duas estratégias para garantir a especificidade do anticorpo. A primeira estratégia é usar pelo menos dois anticorpos diferentes visando o mesmo substrato. A segunda estratégia é a neutralização de anticorpos por incubação com o alvo proteico purificado ou o epítope usado para elevar o anticorpo.
- Incubar o anticorpo de escolha com cinco vezes mais do alvo recombinante ou epítope por 1 h em RT em 1% de BSA na PBS.
- Após a incubação, use o anticorpo neutralizado na concentração usual para coloração conforme descrito acima de 2,11.
4. Calibração do microscópio
NOTA: Usamos rotineiramente um sistema de microscópio de super resolução N-SIM fabricado pela Nikon para os estudos de super-resolução. No entanto, várias outras empresas também oferecem microscópios de super resolução em seus catálogos. Embora as indicações específicas para o sistema N-SIM da Nikon sejam descritas, as instruções a seguir podem ser generalizadas para outros sistemas. Antes da aquisição de imagens SIM, o sistema requer uma calibração adequada com contas fluorescentes específicas de tamanho de sub-resolução. Um exemplo são as microesferas tetraspeck. Estas contas são manchadas com diferentes corantes fluorescentes para permitir a calibração de diferentes lasers com uma amostra.
- Em um banho de água sonicate em torno de 1,8 x 108 microesferas fluorescentes por 10 minutos. O sistema N-SIM da Nikon requer uma amostra de contas multicoloridas pouco povoadas para a calibração. Isso pode diferir para outros sistemas que requerem uma densa camada única de contas fluorescentes de tamanho de sub-resolução. Ajuste o número de partículas fluorescentes de acordo.
- Diluir as microesferas fluorescentes 1:500 em água dupla destilada.
- Sonicate uma segunda vez por mais 10 minutos.
- Pipet 15 μL das contas diluídas em um poço de um deslizamento de cobertura câmara.
- Deixe a solução secar por 5 minutos na RT.
- Adicione 10 μL da solução de montagem e coloque uma tampa de 8 mm por cima.
- Aguarde pelo menos 48 horas para permitir a cura adequada.
- Ligue o microscópio e os lasers.
- Deixe o sistema aquecer para alcançar o equilíbrio térmico de todos os componentes do microscópio. O sistema de microscópio de super-resolução N-SIM requer pelo menos 3 horas.
- Selecione o objetivo de 100x.
- Inicie a calibração alinhando os lasers ao centro do bloco de grade de difração. No sistema N-SIM, um botão de micrômetro e uma câmera dedicada permitem o centro dos feixes de luz para o alvo.
- Insira a tampa de câmara no microscópio para visualização. Ajuste o sistema para a espessura da tampa de câmara, ajustando a coleira de correção objetiva. O software NIS, o software proprietário fornecido com sistemas de microscópio de super-resolução N-SIM, tem uma função automática para regular colares de correção.
- Ajuste o foco do bloco de grade para cada canal para garantir a iluminação de padrão estruturado focalizada na amostra. O software NIS fornece uma função automática para esta tarefa.
- Em seguida, adquira imagens 3D-SIM brutas das microesferas multicoloridas. Reconstrua as imagens brutas para obter uma imagem super-resolvida usando o software de microscópio ou a plataforma de software de código aberto para a análise de imagens biológicas ImageJ24 e o plugin fairSIM25.
- Calcule, para cada comprimento de onda separado, a transformação fourier da imagem super-resolvida obtida em 4.14. Se a imagem transformada não conseguir um padrão correto semelhante a uma flor, reinicie a calibração a partir de 4.11, uma vez que a super-resolução não foi alcançada.
- Na imagem super resolvida, selecione uma única microesfera e calcule seu perfil de intensidade para cada canal para medir a resolução alcançada. Agora deve estar perto de 100 nm lateralmente.
- Em seguida, realize o registro do canal sobrepondo uma aquisição multicanal das microesferas. O objetivo é reunir todos os sinais do canal lateralmente e axily. Isso eliminará as aberrações cromáticas devido ao desalinhamento dos diferentes canais e ajudará na análise de co-localização.
- Confirme a qualidade da calibração usando as funções "Passos de Fase de Iluminação" e "Foco de Padrão de Iluminação" do SIMcheck26, um conjunto de plugins para o aplicativo de código aberto ImageJ. Para isso, prepare um clipe de cobertura com câmara para obter uma densa camada única de microesferas TetraSpeck e adquira uma imagem 3D-SIM da amostra. Analise a imagem no ImageJ e, se forem detectadas aberrações, reinicie a calibração do microscópio a partir da etapa 4.11.
5. Aquisição
- Comece a analisar a amostra usando um objetivo de 40x no modo confocal ou widefield. Isso permite a navegação para a amostra, mantendo bons detalhes e um grande campo de visão.
- Use o sinal de anticorpos MAP2 para identificar uma área que represente processos neuronais.
- Adquira imagens da amostra no modo confocal para determinar a qualidade da coloração. A má qualidade confocal refletirá na má qualidade do SIM, exigindo, portanto, que as amostras sejam descartadas.
- Se a área e a qualidade das imagens forem satisfatórias, mude o objetivo para 100x.
- Aplique óleo ao objetivo de 100x.
- Adquira uma imagem widefield ou confocal que será usada posteriormente para avaliar a qualidade da imagem super-resolvida (Figura 1A,B).
- Mude para o modo 3D-SIM.
- Usando janelas de diálogo para definir parâmetros para aquisição, selecione a configuração de profundidade de bit mais alta disponível para maximizar as informações de cores. Normalmente, 16 bits é a escolha padrão. Além disso, para melhorar a relação sinal-ruído, selecione um baixo valor de frequência para aquisição, como 1 MHz.
- Usando janelas histogramas, defina lasers para obter uma resposta linear do sinal. Para evitar a perda de informações, limite pixels saturados nas imagens. O sistema N-SIM usa uma câmera Andor iXon3. Ao trabalhar em 16 bits, escolha uma intensidade de destino de 16.000 para garantir a resposta linear da câmera. Alternativamente, escolha uma faixa entre 30.000-45.000 para maximizar o alcance dinâmico da aquisição.
- Defina a potência laser entre 0,1% e 50% ao fotografar as amostras e os tempos de exposição entre 50 e 2 s. Potências a laser acima de 50% podem causar fotobleachamento rápido dos fluoroforos em uso.
- Comece a adquirir as imagens no modo 3D-SIM.
- Use o SIMcheck, um conjunto de plugins gratuitos para ImageJ, para avaliar a qualidade da aquisição das imagens brutas.
- Se o SIMCheck não detectar quaisquer artefatos ou problemas de qualidade, adquira um mínimo de 10 imagens de 4 réplicas técnicas para permitir a análise estatística.
6. Pós-produção: reconstrução de imagem
NOTA: As imagens adquiridas 3D-SIM são imagens brutas que precisam ser processadas para obter imagens super-resolvidas reconstruídas. A reconstrução incorreta de imagens brutas pode levar a artefatos que afetariam a análise das amostras. Deve-se, portanto, prestar muita atenção à escolha adequada dos parâmetros de reconstrução.
- Processe as imagens brutas usando o software de análise de reconstrução do microscópio para obter uma imagem super-resolvida(Figura 1C). Alternativamente, use o plugin ImageJ livremente disponível fairSIM para reconstruir imagens brutas.
- Calcule a transformação Fourier das imagens super-resolvidas usando o software de reconstrução do microscópio ou o plugin ImageJ SIMCheck. Uma boa imagem reconstruída deve retornar, para cada canal, uma imagem semelhante a uma flor. Se as imagens reconstruídas não recriarem uma forma semelhante a uma flor, reiniciem a partir das imagens brutas e reconstruam-nas modificando os parâmetros de reconstrução, como filtragem de Wiener, apodização e supressão de ordem zero27. No software NIS, usando a visualização para monitorar como a alteração dos parâmetros afeta a imagem final resolvida, modifique os parâmetros i) Contraste de Modulação de Iluminação, ii) Supressão de Ruído de Alta Resolução e iii) Supressão de Foco.
- Em seguida, analise a imagem reconstruída para detectar artefatos imparcialmente usando NanoJ-SQUIRREL28, um plugin baseado em ImageJ para avaliar a qualidade de imagens super-resolvidas.
- Se o NanoJ-SQUIRREL detectar artefatos, reinicie-os a partir das imagens brutas e reconstrua-os modificando os parâmetros de reconstrução, como filtragem de Wiener, apodização e supressão de ordem zero. No software NIS, usando a visualização para monitorar como a alteração dos parâmetros afeta a imagem final resolvida, modifique os parâmetros Contraste de Modulação de Iluminação, Supressão de Ruído de Alta Resolução e Supressão de Foco.
- Use as imagens super resolvidas para calcular o perfil de co-localização e/ou os coeficientes de Pearson e Mander.
7. Co-localização com análise de perfil
NOTA: Como primeiro passo para estudar a co-localização entre marcadores sinápticos e uma proteína de interesse, pegue uma imagem super resolvida e analise um único lócus para determinar a sobreposição de sinal.
- Identifique um único lócus na imagem super resolvida.
- Obtenha os perfis de intensidade dos sinais fluorescentes do lócus de interesse.
- Exportar os dados.
- Use o GraphPad Prism, ou um software de análise semelhante, para normalizar todos os picos de sinal e obter intensidades de sinal comparáveis para cada canal com o objetivo final de determinar a co-localização específica do lócus.
8. Quantificação dos coeficientes de Pearson e Mander
NOTA: Se a análise do perfil tiver sugerido a co-localização do locus único, uma análise mais geral de toda a imagem pode ser realizada calculando os coeficientes de Pearson e Mander29,30.
- Use o JACoP31, um plug-in ImageJ, para determinar os dois parâmetros de co-localização: Pearson's e Mander's.
9. Análise estatística
- Use o GraphPad Prism, ou um software de análise e gráfico semelhante, para processar dados coletados com JACoP.
- Utilize pelo menos 40 imagens SIM para cada condição analisada para obtenção de gráficos e para relevância estatística.
Resultados
Apresentamos aqui o fluxo de trabalho padrão para estudar a co-localização das proteínas neuronais. Primeiro calibramos o microscópio e, em seguida, realizamos a análise sim das amostras. Para calibrar o sistema, utilizamos microesferas fluorescentes de 0,1 μm de diâmetro. Ao obter imagens 3D-SIM super resolvidas das contas, os dados de imagem subjacentes são transformados em Fourier para recon converter-nas em uma representação de frequência espacial. Na Figura 2A,o padrão de flores distinto é apresentado como uma indicação de níveis de detalhes de super-resolução. Em seguida, medimos a resolução alcançada calculando a largura total pela metade máxima (FWHM) do pico do perfil de intensidade de uma única conta(Figura 2B,C). Finalmente, corrigimos a aberração cromática por registro de canal, novamente utilizando microesferas fluorescentes(Figura 3A,B). Em seguida, começamos a analisar a amostra com um objetivo de 100x e adquirimos imagens 3D-SIM. Utilizamos o SIMCheck para avaliar a adequação da iluminação de campo ou movimento durante a aquisição(Figura 4A). Verificamos diferenças de intensidade entre ângulos de padrão de iluminação(Figura 4B)e calculamos a razão do contraste modulação ao ruído para medir o contraste da faixa local(Figura 4C). Por fim, estimamos a efetiva resolução da reconstrução (Figura 4D).
Em seguida, confirmamos a qualidade das imagens super resolvidas usando NanoJ-SQUIRREL. Na primeira imagem reconstruída(Figura 5A),NanoJ-SQUIRREL detectou a presença de artefatos(Figura 5C). Mudamos os parâmetros de reconstrução para obter uma nova imagem super-resolvida(Figura 5B) e NanoJ-SQUIRREL confirmou a falta de artefatos(Figura 5D). Depois de calibrar o sistema e avaliar a qualidade das imagens reconstruídas, passamos a analisar as culturas neuronais primárias manchadas com um anticorpo contra MAP2, um marcador neuronal, PSD95, um marcador pós-sináptico e a proteína alvo SUMO1. Primeiro analisamos a amostra realizando microscopia confocal de quatro canais com um objetivo de 40x (Figura 6A). Ao selecionar uma área representando processos neuronais, mudamos para um objetivo de 100x. Adquirimos imagens confocal e SIM da mesma área para avaliar a qualidade da reconstrução com NanoJ-SQUIRREL e realizar análises de co-localização. Na Figura 6B,mostramos a imagem 3D-SIM super resolvida dos neurônios manchados para SENP1 e drebrin. A co-localização em imagens super resolvidas pode ser analisada com análise de perfil(Figura 7A) e quantificação dos coeficientes de Pearson e Mander(Figura 7B).

Figura 1: Comparação de aquisições widefield, confocal e SIM. (A) Imagem widefield dos neurônios hipocampais primários imunossuados para SENP1 (verde), drebrina (vermelho) e MAP2 (mauve). Dapi foi usado para manchar núcleos. Barra de escala 5 μm. (B) Imagem confocal da mesma amostra do painel A. (C) Imagem SIM da mesma amostra dos painéis A e B. Clique aqui para ver uma versão maior desta figura.

Figura 2: Análise de uma imagem 3D-SIM de microesferas para calibração de microscópio. (A) Transformação fast fourier de uma aquisição de microesferas com sua forma de flor. (B) Seleção de uma única microesfera para determinar a resolução espacial lateral. (C) Perfil de intensidade da microesfera única em B com a medição de seu FWHM. Os valores representam a resolução alcançada pelo instrumento. Clique aqui para ver uma versão maior desta figura.

Figura 3: Registro de três canais. (A) Aquisição de microesferas multicoloridas (comprimentos de onda 488nm, 555nm e 647nm) Microesferas TetraSpeck antes do registro. (B) Aquisição da mesma amostra após a calibração. Clique aqui para ver uma versão maior desta figura.
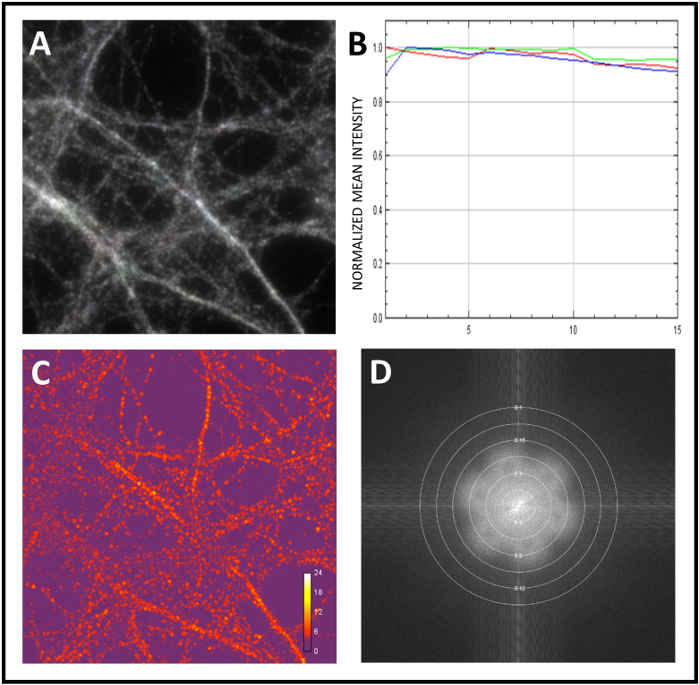
Figura 4: Avaliação de qualidade de imagens brutas e reconstruídas utilizando SIMcheck. (A) Análise de variação de movimento e iluminação usando SIMCheck. O sinal de cinza para branco representa iluminação homogênea e ausência de movimento durante a aquisição. (B) Perfil de Intensidade do Canal obtido pela análise da imagem bruta. Neste exemplo, a variação de intensidade é mínima, para sugerir falta ou branqueamento ou flutuações. (C) Contraste de Modulação Bruta para calcular a razão do contraste modulação-ruído dentro da imagem. O mapa de calor mostra variações de contraste de modulação. (D) Trama fourier reconstruída para analisar o espectro fourier amplitude para determinar a resolução efetiva da reconstrução. Clique aqui para ver uma versão maior desta figura.

Figura 5: Avaliação da qualidade da imagem de super resolução usando NanoJ-SQUIRREL. (A) Referência de imagem de super-resolução com artefatos. (B) Referência imagem de super-resolução de boa qualidade. (C) Imagem representando o mapa de erro NanoJ-SQUIRREL de A. Áreas mais claras representam artefatos em grande escala, enquanto os mais escuros representam a reconstrução correta. (D) Mapa de erro NanoJ-SQUIRREL de B. Clique aqui para ver uma versão maior desta figura.

Figura 6: Amostra de imagem SIM. (A) Microscopia confocal de neurônios primários. Foi escolhido um objetivo 40X para obter uma visão geral da amostra, mantendo uma boa resolução. As células foram imunos detidas para SUMO1 (verde), PSD95 (vermelho) e MAP2 (mauve). Dapi foi usado para manchar núcleos. Barra de escala 50 μm. As imagens foram exibidas como projeção Z. (B) Imagens SIM para SUMO1 e PSD95 na área destacada na caixa verde no painel A utilizando um objetivo de 100X. Pontas de flechas vermelhas indicam a posição do inset mostrado em A usado para calcular o perfil de intensidade. Barra de escala 5 μm. Clique aqui para ver uma versão maior desta figura.
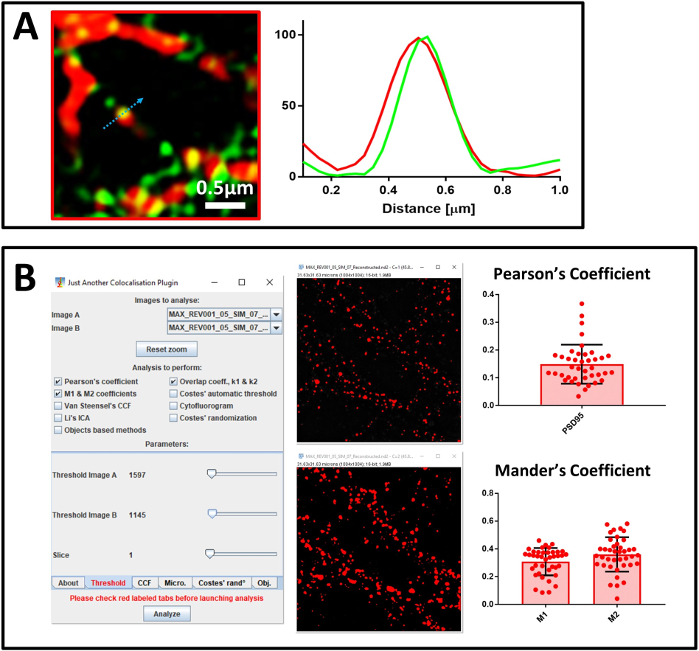
Figura 7: Análise de co-localização. (A) Imagem super-resolvida de neurônios primários manchados com um anticorpo contra SENP1 (em verde) e drebrin (em vermelho), barra de escala 0,5 μm, e seu perfil de intensidade. Os valores do gráfico foram normalizados para cada canal para 100 (unidade arbitrária) e correspondem à intensidade do pixel mostrada pela seta azul. (B) Análise utilizando JACoP para calcular o Coeficiente de Correlação de Pearson e o coeficiente de Mander entre SENP1 e drebrin. Janelas do plug-in configurado e limiar visual são mostrados. O coeficiente de Mander é expresso por dois valores – fração SENP1 que co-localiza com drebrin (M1) e a fração drebrina que co-localiza com SENP1 (M2). Clique aqui para ver uma versão maior desta figura.
Discussão
Elucidar a estrutura e a composição da sinapse é crucial para a compreensão dos processos fisiológicos e patológicos que regulam a memória e a cognição. Enquanto no estado normal, as sinapses são os blocos de construção da memória, elas também estão por trás de distúrbios neurológicos complexos, como a doença de Alzheimer32. O protocolo descrito aqui serve para estudar a co-localização de proteínas neuronais com uma técnica de microscopia de super resolução chamada SIM. Utilizando um padrão particular de iluminação, o SIM pode atingir uma resolução de cerca de 0,1 μm, que é adequada para o estudo de sinapses, que normalmente medem entre 0,03 e 0,15 μm. Para maiores detalhes, outras técnicas de super-resolução, como STED (Estimulada Microscopia de Esgotamento de Emissões), PALM (PhotoActivated Localization Microscopy) ou STORM (Microscopia de Reconstrução Óptica Estocástica), que podem atingir uma resolução de 10-20 nm, podem ser aplicadas33.
Aqui, descrevemos a análise da co-localização de proteínas-alvo com marcadores sinápticos em neurônios primários. O protocolo pode ser aplicado a qualquer cultura primária de células neuronais, como neurônios hipocampais, cerebelares ou corticais e até mesmo a culturas de neurônios primários que não pertencem ao sistema nervoso central, como neurônios do sistema nervoso entérico. A chave para a análise em nível de super-resolução, no entanto, são os reagentes utilizados durante a aquisição, como tampas de câmara e soluções de montagem compatíveis com o índice de difração do objetivo. Usamos tampas de câmara para sua facilidade de uso, mas o método clássico e mais barato de cultivar neurônios primários em vidro de cobertura revestido é, no entanto, válido, particularmente com um vidro de cobertura de alta precisão #1,5H (0,17 mm). Além disso, deve ser utilizado um meio de comunicação de montagem que possa atingir um índice de refração o mais próximo possível do índice de refração do vidro (1,52) e um óleo de imersão para o objetivo de 100x com um índice de refração de 1.515. A temperatura ambiente constante e tabelas estabilizadas também são obrigatórias para garantir a precisão das aquisições.
Usamos anticorpos secundários dyLight e Alexa nos estudos sim. Devido aos seus picos estreitos de excitação e emissão e bom rendimento quântico, eles são indicados para técnicas de super-resolução que requerem a melhor relação sinal/ruído. Dempsey et al. compararam Alexa, dyLight e outros fluoroforos para imagens de super resolução34.
Durante a aquisição, rotineiramente definimos a câmera em 1 MHz acima de 10 MHz. 1 MHz, graças a uma velocidade de aquisição mais lenta, dá às imagens mais precisão e menos ruído do que 10 MHz. O modo de leitura de 1 MHz também pode gravar com um pouco de profundidade de 16 bits (em comparação com o máximo de 14 bits de 10 MHz), dando mais informações de cor e um gradiente de cores mais preciso para as imagens. No entanto, 10 MHz, com sua velocidade, é útil para imagens ao vivo. Para evitar branqueamento e preservar o fluoróforo, também estabelecemos o poder laser o mais baixo possível. Para melhorar a intensidade do sinal, os valores de ganho podem ser aumentados. Vale ressaltar que ganhos mais baixos garantem imagens mais limpas sem aumentar o ruído. Em geral, os melhores resultados são obtidos enquanto a imagem dentro de 7 μm da parte inferior do deslizamento de cobertura câmara. Isso é especialmente importante quando se usa um objetivo de 100x com imersão em óleo. Se for necessária uma aquisição mais profunda entre as células/tecidos, uma escolha melhor pode ser o uso de um objetivo de 60x com imersão na água.
Um dos principais desafios na realização de estudos sim é a reconstrução de imagem35. A obtenção de imagens super-resolvidas sem artefatos e aberração requer não apenas o uso de condições experimentais ad-hoc, mas também a calibração cuidadosa do sistema e otimização dos parâmetros para obter as imagens finais. No protocolo, descrevemos como evitar alguns dos erros mais comuns, avaliando a calibração do sistema e a análise de qualidade de imagens cruas e reconstruídas. Especificamente, descrevemos o uso dos plugins ImageJ SIMCheck e NanoJ-SQUIRREL para garantir as configurações corretas do instrumento para evitar artefatos comuns de imagens super-resolvidas. Os aplicativos permitem uma avaliação imparcial da qualidade das imagens finais que não se baseia no benchmarking subjetivo dos resultados em relação ao conhecimento prévio das estruturas de estudo.
Sugerimos o uso de sinaptofisina e PSD95 ou drebrin como marcadores pré e pós-sináptico, embora outros marcadores também sejam válidos. Um enorme corpo de literatura descreve proteínas como fagote como marcadores sinápticos36,37. Vale ressaltar que os marcadores pré e postssináspicos estão presentes em toda a célula, excluindo o núcleo. Grande parte de seu sinal não é sináptico, mas representa proteínas no transporte ou degradação, fundo ou outros artefatos. Por isso, é importante escolher cuidadosamente a área da análise. Usamos sinal de anticorpos MAP2 para escolher terminais de axônio e dendrítica.
Na análise da co-localização utilizamos duas abordagens. A primeira é uma abordagem visual, baseada na análise de perfil que mostra eventos únicos de co-localização e identifica a contribuição de cada canal. Uma ressalva dessa abordagem, no entanto, é o fraco poder estatístico. Por essa razão, decidimos também utilizar um segundo método baseado na análise de um número maior de eventos representativos de todo o campo de cada imagem. Este método baseia-se no cálculo do coeficiente de correlação do Pearson e dos coeficientes M1 e M2 de Mander. Usamos o coeficiente de correlação do Pearson para descrever a sobreposição de sinais na imagem e o M1 e M2 de Mander para descrever a co-localização recíproca entre sinais de interesse38. Para o cálculo, empregamos o plugin ImageJ JACoP, pois possui um recurso que permite definir um limiar manual para descartar qualquer contribuição de fundo para a análise, especialmente fundamental para a análise de Mander.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem a Edoardo Micotti pela crítica construtiva ao manuscrito. Este estudo foi apoiado pela BrightFocus A2019296F, por Fondo di Beneficenza - Gruppo Intesa Sanpaolo (LC), pela Fondazione Regionale per la Ricerca Biomedica (Care4NeuroRare CP_20/2018) (CN) e pela Rede de Treinamento Inovador Marie Skłodowska-Curie (JK).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan blue solution | Thermo Fisher Scientific | 15250061 | Chemical |
| 70 µm filter | Corning | 352350 | Equiment |
| Alexa | Thermo Fisher Scientific | - | Antibody |
| Antibody SENP1 | Santa Cruz | sc-271360 | Antibody |
| B27 Supplement | Life Technologies | 17504044 | Chemical |
| Bovine serum albumin | Merck | 5470 | Chemical |
| CaCl2 | Merck Life Science | 21115 | Chemical |
| Chambered coverslips | Ibidi | 80826 | Equiment |
| DyLight | Thermo Fisher Scientific | - | Antibody |
| FBS (Hyclone) | GIBCO | SH3007002 (CHA1111L) | Serum |
| FluoSpheres carboxylate-modified microspheres, 0.1 μm, yellow–green fluorescent | Thermo Fisher Scientific | F8803 | Equiment |
| Glucose | Merck Life Science | G8769 | Chemical |
| Glutamax | GIBCO | 35050061 | Chemical |
| HEPES | Merck Life Science | H3537 | Chemical |
| L-Cystein | Merck Life Science | C6852-25g | Chemical |
| MAP2 | Merck | AB15452 | Antibody |
| MEM | Life Technologies | 21575022 | Medium |
| MgCl | Merck Life Science | M8266 | Chemical |
| NaOH | VWR International | 1,091,371,000 | Chemical |
| Neurobasal A | Life Technologies | 10888022 | Medium |
| N-SIM Super Resolution Microscope | Nikon | - | Instrument |
| Papain | Merck Life Science | P-3125 | Chemical |
| paraformaldehyde | Thermo Fisher Scientific | 28908 | Chemical |
| Pen/Strep 10x | Life Technologies | 15140122 | Chemical |
| phosphate-buffered saline | Gibco | 10010023 | Chemical |
| Poly-L lysine | Sigma | P2636 | Chemical |
| ProLong Diamond Glass Antifade Mountant | Thermo Fisher Scientific | P36970 | Chemical |
| PSD95 | NeuroMab | K28/43 | Antibody |
| Round coverglass | Thermo | 12052712 | Equiment |
| SUMO1 | Abcam | ab32058 | Antibody |
| Synaptophysin | Merck | S5768 | Antibody |
| Triton X-100 | Merck | T8787 | Chemical |
| Trypsin inhibitor | Merck Life Science | T9003-500MG | Chemical |
Referências
- Foster, M., Sherrington, C. S. . A textbook of physiology, part three: The central nervous system (7th ed.). , (1897).
- Choquet, D., Triller, A. The Dynamic Synapse. Neuron. 80 (3), 691-703 (2013).
- McAllister, A. K. Dynamic Aspects of CNS Synapse Formation. Annual Review of Neuroscience. 30 (1), 425-450 (2007).
- Yuzaki, M. Two Classes of Secreted Synaptic Organizers in the Central Nervous System. Annual Review of Physiology. 80 (1), 243-262 (2018).
- Baddeley, D., Bewersdorf, J. Biological Insight from Super-Resolution Microscopy: What We Can Learn from Localization-Based Images. Annual Review of Biochemistry. 87 (1), 965-989 (2018).
- Sigal, Y. M., Zhou, R., Zhuang, X. Visualizing and discovering cellular structures with super-resolution microscopy. Science. 361 (6405), 880-887 (2018).
- Vangindertael, J., et al. An introduction to optical super-resolution microscopy for the adventurous biologist. Methods and Applications in Fluorescence. 6 (2), 022003 (2018).
- Badawi, Y., Nishimune, H. Super-resolution microscopy for analyzing neuromuscular junctions and synapses. Neuroscience Letters. 715, 134644 (2020).
- Scalisi, S., Barberis, A., Petrini, E. M., Zanacchi, F. C., Diaspro, A. Unveiling the Inhibitory Synapse Organization Using Superresolution Microscopy. Biophysical Journal. 116 (3), 133 (2019).
- Yang, X., Specht, C. G. Subsynaptic Domains in Super-Resolution Microscopy: The Treachery of Images. Frontiers in Molecular Neuroscience. 12, (2019).
- Monro, T. Beyond the diffraction limit. Nature Photonics. 3 (7), 361 (2009).
- Won, R. Eyes on super-resolution. Nature Photonics. 3 (7), 368-369 (2009).
- Wegel, E., et al. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific Reports. 6 (1), 27290 (2016).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Gustafsson, M. G. L., et al. Three-Dimensional Resolution Doubling in Wide-Field Fluorescence Microscopy by Structured Illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Brose, N., O'Connor, V., Skehel, P. Synaptopathy: dysfunction of synaptic function. Biochemical Society Transactions. 38 (2), 443-444 (2010).
- Tyebji, S., Hannan, A. J. Synaptopathic mechanisms of neurodegeneration and dementia: Insights from Huntington's disease. Progress in Neurobiology. 153, 18-45 (2017).
- Won, H., Mah, W., Kim, E. Autism spectrum disorder causes, mechanisms, and treatments: focus on neuronal synapses. Frontiers in Molecular Neuroscience. 6, (2013).
- Pfeiffer, B. E., Huber, K. M. The State of Synapses in Fragile X Syndrome. The Neuroscientist. 15 (5), 549-567 (2009).
- Pavlowsky, A., Chelly, J., Billuart, P. Emerging major synaptic signaling pathways involved in intellectual disability. Molecular Psychiatry. 17 (7), 682-693 (2012).
- Senatore, A., Restelli, E., Chiesa, R. Synaptic dysfunction in prion diseases: a trafficking problem. International Journal of Cell Biology. 2013, 543803 (2013).
- Colnaghi, L., et al. Super Resolution Microscopy of SUMO Proteins in Neurons. Frontiers in Cellular Neuroscience. 13, (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Müller, M., Mönkemöller, V., Hennig, S., Hübner, W., Huser, T. Open-source image reconstruction of super-resolution structured illumination microscopy data in ImageJ. Nature Communications. 7 (1), 10980 (2016).
- Ball, G., et al. SIMcheck: a Toolbox for Successful Super-resolution Structured Illumination Microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Schaefer, L. H., Schuster, D., Schaffer, J. Structured illumination microscopy: artefact analysis and reduction utilizing a parameter optimization approach. Journal of Microscopy. 216 (2), 165-174 (2004).
- Culley, S., et al. NanoJ-SQUIRREL: quantitative mapping and minimisation of super-resolution optical imaging artefacts. Nature Methods. 15 (4), 263-266 (2018).
- Manders, E. M. M., Verbeek, F. J., Aten, J. A. Measurement of co-localization of objects in dual-colour confocal images. Journal of Microscopy. 169 (3), 375-382 (1993).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander’s overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
- Bolte, S., Cordelières, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224, 213-232 (2006).
- Bae, J. R., Kim, S. H. Synapses in neurodegenerative diseases. BMB Reports. 50 (5), 237-246 (2017).
- Godin, A. G., Lounis, B., Cognet, L. Super-resolution Microscopy Approaches for Live Cell Imaging. Biophysical Journal. 107 (8), 1777-1784 (2014).
- Dempsey, G. T., Vaughan, J. C., Chen, K. H., Bates, M., Zhuang, X. Evaluation of fluorophores for optimal performance in localization-based super-resolution imaging. Nature Methods. 8 (12), 1027-1036 (2011).
- Karras, C., et al. Successful optimization of reconstruction parameters in structured illumination microscopy - A practical guide. Optics Communications. 436, 69-75 (2019).
- Bereczki, E., et al. Synaptic markers of cognitive decline in neurodegenerative diseases: a proteomic approach. Brain: A Journal of Neurology. 141 (2), 582-595 (2018).
- Gilestro, G. F., Tononi, G., Cirelli, C. Widespread Changes in Synaptic Markers as a Function of Sleep and Wakefulness in Drosophila. Science. 324 (5923), 109-112 (2009).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander's overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados