Method Article
Imagerie super-résolution pour étudier la co-localisation des protéines et des marqueurs synaptiques dans les neurones primaires
Dans cet article
Résumé
Ce protocole montre comment utiliser la microscopie à super résolution pour étudier la co-localisation des protéines dans les cultures neuronales primaires.
Résumé
Les synapses sont les éléments fonctionnels des neurones et leurs défauts ou pertes sont à la base de plusieurs troubles neurodégénératifs et neurologiques. Les études d’imagerie sont largement utilisées pour étudier leur fonction et leur plasticité dans des conditions physiologiques et pathologiques. En raison de leur taille et de leur structure, les études de localisation des protéines nécessitent des techniques d’imagerie à haute résolution. Dans ce protocole, nous décrivons une procédure d’étude dans les neurones primaires la co-localisation des protéines cibles avec des marqueurs synaptiques à un niveau de super-résolution utilisant la microscopie d’illumination structurée (SIM). SIM est une technique d’éclairage à motifs qui double la résolution spatiale de la microscopie à large champ, atteignant un détail d’environ 100 nm. Le protocole indique les contrôles et les paramètres requis pour des études de co-localisation robustes et une vue d’ensemble des méthodes statistiques pour analyser correctement les données d’imagerie.
Introduction
La compréhension et la vue de la synapse a énormément changé depuis sa première description par Foster et Sherrington en 18971. Depuis lors, notre connaissance de la communication neuronale et les processus moléculaires derrière elle a augmenté de façon exponentielle2. Il est devenu clair que les synapses peuvent être considérées comme un système à deux compartiments : un compartiment pré-synaptique contenant des vésicules pour la libération de neurotransmetteurs et un compartiment post-synaptique avec des récepteurs3. Cette vision simpliste, au cours des vingt dernières années, s’est transformée en un réseau complexe de protéines nécessaires pour transduire la liaison des émetteurs en signalisation4.
Les gains dans la compréhension sont en partie dus aux techniques de super-résolution qui ont surmonté la limite de diffraction de la microscopie légère conventionnelle pour convenir à la dimension des synapses mieux5,6,7,8,9,10. En raison de la limite de diffraction, un microscope optique ne peut pas atteindre une résolution supérieure à 200 nm par la suite11,12. Pour contourner cette limite, des techniques de superrésolution ont été créées, en utilisant différentes approches et en atteignant différentes résolutions limites de sous-diffraction : SIM, STED (Stimated Emission Ecution Exceed Microscopy), PALM (PhotoActivated Localization Microscopy) et STORM (Stochastic Optical Reconstruction Microscopy)13,14. SIM double la résolution spatiale des systèmes de microscopie à large champ à base de laser en insérant une grille de diffraction dans le chemin de faisceaud’excitation 15. La grille mobile diffracts les faisceaux laser pour créer un modèle d’éclairage connu, généralement rayures. Ce motif de lumière volontairement structuré est superposé à la distribution spatiale inconnue du colorant fluorescent (de l’échantillon). Les franges d’interférence formées par les deux modèles codent pour des détails fins autrement indiscernables avec la microscopie normale de large champ. L’image finale super-résolue est obtenue en combinant et décodage avec des méthodes mathématiques plusieurs images brutes du même échantillon obtenus par les traductions et les rotations de la grille de diffraction. La résolution des images super-résolues atteint 100 nm dans le côté et 500 nm dans les directions axiales pour 2D-SIM15 ou 100 nm dans le latéral et 250 nm dans les directions axiales pour 3D-SIM16.
La nouvelle compréhension de la synapse est encore plus importante à la lumière des nombreux troubles neurologiques où le dysfonctionnement synaptique joue un rôle majeur dans l’apparition et la progression17,18. La maladie d’Alzheimer, le syndrome de Down, la maladie de Parkinson, les maladies à prions, l’épilepsie, les troubles du spectre autistique et le syndrome fragile de X, entre autres, ont été liés à des anomalies dans la composition synaptique, la morphologie et la fonction19,20,21,22.
Récemment, à l’aide d’un ensemble d’anticorps spécifiques à sumo, nous avons utilisé sim pour montrer la co-localisation dans les neurones hippocampaux primaires des protéines SUMO avec les marqueurs pré- et post-synaptiques synaptophysine et PSD95 au niveau de super-résolution23. Cela nous a permis de confirmer les preuves biochimiques et confocales de microscopie de la localisation sumo dans les neurones.
Ici, nous décrivons un protocole pour étudier la localisation des protéines dans les neurones primaires hippocampaux de souris. En même temps, ce protocole peut être adapté à différents types de cultures neuronales primaires.
Protocole
1. Cultures primaires
- Les neurones primaires hippocampaux de souris de culture dans les couvertures chambrées (telles que Ibidi μ-Slide 8 Well ou Nunc Lab-Tek Chambered Coverglass) qui correspondent à l’exigence objective pour #1,5 (0,17 mm) couvrent l’épaisseur.
- Pelage de couvercles en chambre avec 100 μL de poly-L-lysine (100 μg/mL).
- Le lendemain, lavez deux fois les couvercles chambrelés à l’une de la solution saline stérile tamponnée au phosphate (PBS).
- Pour obtenir des neurones primaires de souris, isoler hippocampi des chiots P1-P423.
- Placez les hippocampes disséqués dans 10 ml de Dissection Media (Tableau 1) et laissez-les déposer au fond du tube.
- À l’aide d’une pipette stérile, retirez soigneusement le support de dissection, laissant l’hippocampe intact au fond du tube.
- Ajouter 10 mL de Media 1 (Tableau 1) à l’hippocampe et incuber pendant 30 minutes à 37 °C.
- À l’aide d’une pipette stérile, retirez soigneusement le média 1, laissant l’hippocampe intact au fond du tube.
- Ajouter 10 ml de media 2 (tableau 1) et laisser le tube de centrifugeuse (plafonné) sous le capot horizontalement pendant 45 minutes.
- Laissez le tube de centrifugeuse reposer verticalement pour permettre au tissu de s’installer au fond du tube.
- À l’aide d’une pipette stérile, retirez soigneusement le média 2, laissant l’hippocampe intact au fond du tube de centrifugeuse.
- Ajouter 2 mL de Media 3 (Tableau 1).
- À l’aide d’une pipette p1000 à pointe filtrée, dissocier mécaniquement les cellules du tissu.
- Transférer le supernatant, dans lequel se trouvent les neurones isolés, dans un tube de centrifugeuse de 15 mL.
- Centrifugez la suspension cellulaire pendant 2 min à 300 x g à température ambiante (RT).
- Après la centrifugeuse, les cellules sont situées au fond du tube de centrifugeuse. À l’aide d’une pipette stérile jeter le supernatant.
- Resuspender les cellules dans 1 mL de Media 4.
- Utilisez un filtre de 70 μm pour éliminer les cellules non dissociées.
- Compter les cellules viables dans une chambre Bürker en ajoutant 1 μL de 0,4 % de solution bleue Trypan à 19 μL de la suspension cellulaire.
- Plaquez des cellules à 70 000 cellules/puits dans un volume de 200 μL par puits.
- Laisser les cellules s’attacher pendant 2 h dans un incubateur humidifié à 37 °C et 5 % de CO2.
- Sortez les couvercles chambrelés de l’incubateur et remplacez soigneusement le support par 200 μL de Culture Media.
- Laissez les couvercles chambrelés dans un incubateur humidifié à 37 °C et 5% CO2.
- Remplacez un tiers du milieu par des supports de culture frais tous les 5-7 jours.
- Attendez que les neurones primaires hippocampaux soient pleinement mûris (12-14 jours après le placage) pour effectuer des études de co-localisation.
2. Coloration d’immunofluorescence
- Prenez les couvertures chambred de l’incubateur.
- Retirer le milieu.
- Lavez rapidement les puits avec 200 μL de PBS.
- Ajouter 4 % de paraformaldéhyde (PFA) dans pbs (200 μL/puits) aux neurones pour les réparer rapidement.
- Incuber les cellules pendant 15 min à RT.
- Retirez la solution PFA.
- Permeabiliser les cellules en ajoutant PBS avec 0,2% Triton X-100 (200 μL/puits).
- Incuber pendant 1 min à RT.
- Retirer la solution et incuber les échantillons avec 1% d’albumine de sérum bovin (BSA) en PBS (200 μL/well) pendant 1 h à RT pour couvrir passivement toutes les surfaces de liaison libre de la plaque avec une protéine non pertinente pour l’analyse. Un tampon de blocage basé sur BSA sans Triton X-100 réduit le fond d’anticorps plus efficacement que le même tampon avec 0,2% Triton X-100.
- Retirez la solution.
- Ajouter l’anticorps primaire de choix dilué dans une solution PBS contenant 1% de BSA et 0,2% de Triton X-100 (120-200 μL/puits, selon la dilution et la disponibilité des anticorps). Incuber les échantillons pendant 2 h.
- En tant que contrôle négatif, n’ajoutez aucun anticorps primaire à l’un des puits. Plusieurs anticorps de différentes espèces contre différentes cibles peuvent être utilisés en même temps. Utilisez un anticorps contre le MAP2 (un marqueur neuronal) élevé dans le poulet, un anticorps contre le PSD95 ou la synaptophysine élevé dans la souris, et un anticorps contre une protéine cible élevée chez le lapin. Cela permet des analyses SIM tricolores.
- Laver rapidement les puits trois fois avec pbs (200 μL/puits).
- Ajouter des anticorps secondaires (dyLight et Alexa anticorps secondaires peuvent tous deux être utilisés) dilués dans une solution PBS contenant 1% BSA et 0,2% Triton X-100 (200 μL/bien). Incuber les échantillons pendant 1 h à RT.
- Laver rapidement les puits trois fois avec pbs (200 μL/puits).
- Ajouter le colorant Hoechst à une concentration de 1 μg/mL diluée en PBS (200 μL/puits) pour tacher les noyaux. Incuber les échantillons pendant 10 minutes à RT.
- Lavez rapidement les puits deux fois avec PBS.
- Montez des cellules à l’aide d’un supporteur compatible SIM. Utilisez 10 μL/puits de ProLong Glass Antifade Mountant.
- Couvrir et protéger les cellules à l’aide d’un verre à couverture (p. ex., un verre rond d’un diamètre de 8 mm). Les carrés peuvent également être utilisés.
- Stockez les couvercles en chambre à RT et attendez au moins 48 h avant d’acquérir des images. Diamond Glass nécessite au moins deux jours de séchage avant les acquisitions de super-résolution.
3. Contrôle de la spécificité des anticorps
REMARQUE : Utilisez deux stratégies pour assurer la spécificité des anticorps. La première stratégie consiste à utiliser au moins deux anticorps différents ciblant le même substrat. La deuxième stratégie est la neutralisation des anticorps par incubation avec la cible protéique purifiée ou l’épitope utilisé pour élever l’anticorps.
- Incuber l’anticorps de choix avec cinq fois plus de la cible recombinante ou épitope pendant 1 h à RT dans 1% BSA dans PBS.
- Après l’incubation, utilisez l’anticorps neutralisé à la concentration habituelle pour la coloration comme décrit ci-dessus à partir de 2,11.
4. Étalonnage du microscope
REMARQUE : Nous utilisons régulièrement un système de microscope à super résolution N-SIM fabriqué par Nikon pour les études de super-résolution. Cependant, plusieurs autres entreprises offrent également des microscopes super-résolution dans leurs catalogues. Bien que des indications spécifiques pour le système N-SIM de Nikon soient décrites, les instructions qui suivent peuvent être généralisées à d’autres systèmes. Avant l’acquisition d’images SIM, le système nécessite un étalonnage approprié avec des perles fluorescentes de taille de sous-résolution spécifiques. Un exemple est les microsphères TetraSpeck. Ces perles sont tachées de différents colorants fluorescents pour permettre l’étalonnage de différents lasers avec un seul échantillon.
- Dans un bain d’eau sonicate autour de 1,8 x 108 microsphères fluorescentes pendant 10 minutes. Le système N-SIM de Nikon nécessite un échantillon de perles multicolores peu peuplé pour l’étalonnage. Cela pourrait différer pour d’autres systèmes qui nécessitent une couche dense unique de perles fluorescentes de taille sous-résolution. Ajuster le nombre de particules fluorescentes en conséquence.
- Diluer les microsphères fluorescentes 1:500 dans de l’eau double distillée.
- Soniment une deuxième fois pendant 10 minutes supplémentaires.
- Pipet 15 μL des perles diluées dans un puits d’un couvercle chambré.
- Laissez sécher la solution pendant 5 minutes à RT.
- Ajouter 10 μL de la solution de montage et placer une housse de 8 mm sur le dessus.
- Attendez au moins 48 heures pour permettre un traitement approprié.
- Allumez le microscope et les lasers.
- Laissez le système se réchauffer pour atteindre l’équilibre thermique de tous les composants du microscope. Le système de microscope super-résolution N-SIM nécessite au moins 3 heures.
- Sélectionnez l’objectif 100x.
- Démarrez l’étalonnage en alignant les lasers au centre du bloc de grille de diffraction. Dans le système N-SIM, un bouton micromètre et une caméra dédiée permettent de centrer les faisceaux lumineux vers la cible.
- Insérez le couvercle chambrelé dans le microscope pour la visualisation. Réglez le système sur l’épaisseur du couvercle en chambre en ajustant le collier de correction objectif. Le logiciel NIS, le logiciel propriétaire fourni avec les systèmes de microscope à super résolution N-SIM, a une fonction automatique pour réguler les colliers de correction.
- Ajuster la mise au point de bloc de grille pour chaque canal afin d’assurer l’éclairage structuré concentré sur l’échantillon. Le logiciel NIS fournit une fonction automatique pour cette tâche.
- Ensuite, acquérir des images brutes 3D-SIM des microsphères multicolores. Reconstruire les images brutes pour obtenir une image super-résolu à l’aide du logiciel microscope ou de la plate-forme logicielle open-source pour l’analyse des images biologiques ImageJ24 et le plugin fairSIM25.
- Calculer, pour chaque longueur d’onde séparée, la transformation Fourier de l’image super-résolue obtenue en 4.14. Si l’image transformée ne parvient pas à obtenir un modèle de fleur correct, redémarrez l’étalonnage à partir de 4.11 puisque la super-résolution n’a pas été réalisée.
- Dans l’image super-résolue, sélectionnez une seule microsphère et calculez son profil d’intensité pour chaque canal afin de mesurer la résolution obtenue. Il doit maintenant être proche de 100 nm ultérieurement.
- Ensuite, effectuer l’enregistrement des canaux en superposant une acquisition multicanal des microsphères. Le but est de coluler tous les signaux de canal ultérieurement et axialement. Cela permettra d’éliminer les aberrations chromatiques dues au désalignement des différents canaux et d’aider l’analyse de co-localisation.
- Confirmez la qualité de l’étalonnage à l’aide des fonctions « Étapes de phase d’illumination » et « Mise au point de modèle d’illumination » de SIMcheck26, une suite de plugins pour l’application open-source ImageJ. À cette fin, préparez un couvercle de chambre pour obtenir une couche unique dense de microsphères TetraSpeck et acquérir une image 3D-SIM de l’échantillon. Analyser l’image dans ImageJ et, si des aberrations sont détectées, redémarrez l’étalonnage du microscope à partir de l’étape 4.11.
5. Acquisition
- Commencez à analyser l’échantillon à l’aide d’un objectif 40x en mode confocal ou widefield. Cela permet la navigation à l’échantillon, en maintenant de bons détails et un grand champ de vision.
- Utilisez le signal d’anticorps MAP2 pour identifier une zone représentant les processus neuronaux.
- Acquérir des images de l’échantillon en mode confocal pour déterminer la qualité de la coloration. Une mauvaise qualité confocale se reflétera dans la mauvaise qualité de LA CARTE SIM, ce qui nécessite que les échantillons soient jetés.
- Si la surface et la qualité des images sont satisfaisantes, passez l’objectif à 100x.
- Appliquer de l’huile sur l’objectif 100x.
- Acquérir une image large ou confocale qui sera utilisée plus tard pour évaluer la qualité de l’image super-résolue (Figure 1A,B).
- Passez au mode 3D-SIM.
- À l’aide de fenêtres de dialogue pour définir des paramètres d’acquisition, sélectionnez le paramètre de profondeur de bit le plus élevé disponible pour maximiser les informations sur les couleurs. En règle générale, 16 bits est le choix standard. De plus, pour améliorer le rapport signal/bruit, sélectionnez une valeur basse fréquence pour l’acquisition, comme 1 MHz.
- À l’aide de fenêtres histogrammes, définissez la puissance des lasers pour obtenir une réponse linéaire du signal. Pour éviter la perte d’informations, limitez les pixels saturés dans les images. Le système N-SIM utilise une caméra Andor iXon3. Lorsque vous travaillez à 16 bits, choisissez une intensité cible de 16 000 pour assurer la réponse linéaire de la caméra. Sinon, choisissez une fourchette comprise entre 30 000 et 45 000 pour maximiser la gamme dynamique de l’acquisition.
- Réglez la puissance laser entre 0,1 % et 50 % lors de l’imagerie des échantillons et les temps d’exposition entre 50 ms et 2 s. Les puissances laser supérieures à 50% peuvent causer le photobleaching rapide des fluorophores en usage.
- Commencez à acquérir les images en mode 3D-SIM.
- Utilisez SIMcheck, une suite de plugins gratuits pour ImageJ, pour évaluer la qualité de l’acquisition des images brutes.
- Si SIMCheck ne détecte pas d’artefacts ou de problèmes de qualité, obtenez un minimum de 10 images à partir de 4 répliques techniques pour permettre l’analyse statistique.
6. Post-production : reconstruction d’images
REMARQUE : Les images acquises par 3D-SIM sont des images brutes qui doivent être traitées pour obtenir des images reconstruites super-résolues. Une reconstitution incorrecte d’images brutes peut mener à des artefacts qui affecteraient l’analyse des échantillons. Une grande attention devrait donc être accordée au choix dû des paramètres de reconstruction.
- Traiter les images brutes à l’aide du logiciel d’analyse de reconstruction du microscope pour obtenir une image super-résolue (Figure 1C). Vous pouvez également utiliser le plugin fairSIM photoJ librement disponible pour reconstruire des images brutes.
- Calculez la transformation Fourier des images super-résolues à l’aide du logiciel de reconstruction du microscope ou du plugin SimCheck d’ImageJ. Une bonne image reconstruite doit rapporter, pour chaque canal, une image fleurie. Si les images reconstruites ne parviennent pas à recréer une forme de fleur, redémarrez à partir des images brutes et reconstruisez-les en modifiant les paramètres de reconstruction tels que le filtrage Wiener, l’apodisation et la suppression de l’ordre zéro27. Dans le logiciel NIS, en utilisant l’aperçu pour surveiller comment la modification des paramètres affecte l’image finale résolue, modifier les paramètres i) Contraste de modulation de l’éclairage, ii) Suppression du bruit à haute résolution et iii) Suppression hors mise au point.
- Ensuite, analyser l’image reconstruite pour détecter de manière impartiale les artefacts à l’aide de NanoJ-SQUIRREL28, un plugin basé sur ImageJ pour évaluer la qualité des images super-résolues.
- Si NanoJ-SQUIRREL détecte des artefacts, redémarrez à partir des images brutes et reconstruisez-les en modifiant les paramètres de reconstruction tels que le filtrage Wiener, l’apodisation et la suppression de l’ordre zéro. Dans le logiciel NIS, à l’aide de l’aperçu pour surveiller comment la modification des paramètres affecte l’image finale résolue, modifiez les paramètres Du contraste de modulation d’illumination, suppression du bruit à haute résolution et suppression de la mise au point.
- Utilisez les images super-résolues pour calculer le profil de co-localisation et/ou les coefficients de Pearson et Mander.
7. Co-localisation avec analyse de profil
REMARQUE : Dans un premier temps pour étudier la co-localisation entre les marqueurs synaptiques et une protéine d’intérêt, prenez une image super-résolue et analysez un seul locus pour déterminer le chevauchement du signal.
- Identifiez un seul locus sur l’image super-résolue.
- Obtenez les profils d’intensité des signaux fluorescents du lieu d’intérêt.
- Exporter les données.
- Utilisez GraphPad Prism, ou un logiciel d’analyse similaire, pour normaliser tous les pics de signal et obtenir des intensités de signal comparables pour chaque canal dans le but final de déterminer la co-localisation spécifique au locus.
8. Quantification des coefficients de Pearson et Mander
REMARQUE : Si l’analyse de profil a suggéré une co-localisation de locus unique, une analyse plus générale de l’image entière peut être effectuée en calculant les coefficients de Pearson et de Mander29,30.
- Utilisez JACoP31, un plug-in ImageJ, pour déterminer les deux paramètres de la co-localisation : Pearson et Mander’s.
9. Analyse statistique
- Utilisez GraphPad Prism, ou un logiciel d’analyse et de graphique similaire, pour traiter les données collectées avec JACoP.
- Utilisez au moins 40 images SIM pour chaque condition analysée pour obtenir des graphiques et pour la pertinence statistique.
Résultats
Nous présentons ici le flux de travail standard pour étudier la co-localisation des protéines neuronales. Nous avons d’abord calibré le microscope et ensuite nous avons effectué l’analyse SIM des échantillons. Pour calibrer le système, nous avons utilisé des microsphères fluorescentes de 0,1 μm de diamètre. Lors de l’obtention d’images 3D-SIM super-résolues des perles, les données d’image sous-jacentes sont fourier-transformés pour les re-convertir en une représentation de fréquence spatiale. Dans la figure 2A, le motif de fleur distinct est présenté comme une indication des niveaux de détail de super-résolution. Nous avons ensuite mesuré la résolution obtenue en calculant la largeur complète à la moitié maximale (FWHM) du pic du profil d’intensité d’une seule perle (Figure 2B,C). Enfin, nous avons corrigé l’aberration chromatique par enregistrement des canaux, encore une fois à l’aide de microsphères fluorescentes (Figure 3A,B). Ensuite, nous avons commencé à analyser l’échantillon avec un objectif 100x et nous avons acquis des images 3D-SIM. Nous avons utilisé SIMCheck pour évaluer l’égalité de l’éclairage ou du mouvement sur le terrain lors del’acquisition ( Figure 4A). Nous avons vérifié les différences d’intensité entre les angles de motif d’éclairage (figure 4B)et nous avons calculé le rapport entre le contraste de modulation et le bruit pour mesurer le contraste de bande locale (figure 4C). Enfin, nous avons estimé la résolution effective de la reconstruction (Figure 4D).
Nous avons ensuite confirmé la qualité des images super-résolues en utilisant NanoJ-SQUIRREL. Dans la première image reconstruite (Figure 5A), NanoJ-SQUIRREL a détecté la présence d’artefacts (Figure 5C). Nous avons modifié les paramètres de reconstruction pour obtenir une nouvelle image super-résolue (Figure 5B) et NanoJ-SQUIRREL a confirmé le manque d’artefacts (Figure 5D). Après avoir calibré le système et évalué la qualité des images reconstruites, nous avons ensuite commencé à analyser les cultures neuronales primaires tachées d’un anticorps contre MAP2, un marqueur neuronal, PSD95, un marqueur post-synaptique et la protéine cible SUMO1. Nous avons d’abord analysé l’échantillon effectuant une microscopie confocale à quatre canaux avec un objectif de 40x (Figure 6A). Lors de la sélection d’une zone représentant les processus neuronaux, nous sommes passés à un objectif 100x. Nous avons acquis des images confocales et SIM d’une même zone pour évaluer la qualité de la reconstruction avec NanoJ-SQUIRREL et effectuer des analyses de co-localisation. Dans la figure 6B, nous montrons l’image 3D-SIM super-résolue des neurones tachés pour SENP1 et drebrin. La co-localisation dans les images super-résolues peut être analysée avec l’analyse de profil (figure 7A) et la quantification des coefficients de Pearson et de Mander (figure 7B).

Figure 1 : Comparaison des acquisitions de terrains larges, confocales et SIM. (A)Image de widefield des neurones hippocampaux primaires immunostained pour SERP1 (vert), drebrin (rouge) et MAP2 (mauve). DAPI a été utilisé pour tacher les noyaux. Barre d’échelle 5 μm. (B) Image confocale du même échantillon du panneau A. (C) Image SIM du même échantillon de panneau A et B. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Analyse d’une image 3D-SIM de microsphères pour l’étalonnage du microscope. (A) Fast Fourier transformer d’une acquisition de microsphères avec sa forme de fleur. (B) Sélection d’une microsphère unique pour déterminer la résolution spatiale latérale. (C) Profil d’intensité de la microsphère unique en B avec la mesure de son FWHM. Les valeurs représentent la résolution obtenue par l’instrument. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Enregistrement à trois canaux. (A) Acquisition de micro-phères TetraSpeck multicolores (longueurs d’onde 488nm, 555nm et 647nm) TetraSpeck avant inscription. (B) Acquisition du même échantillon après étalonnage. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
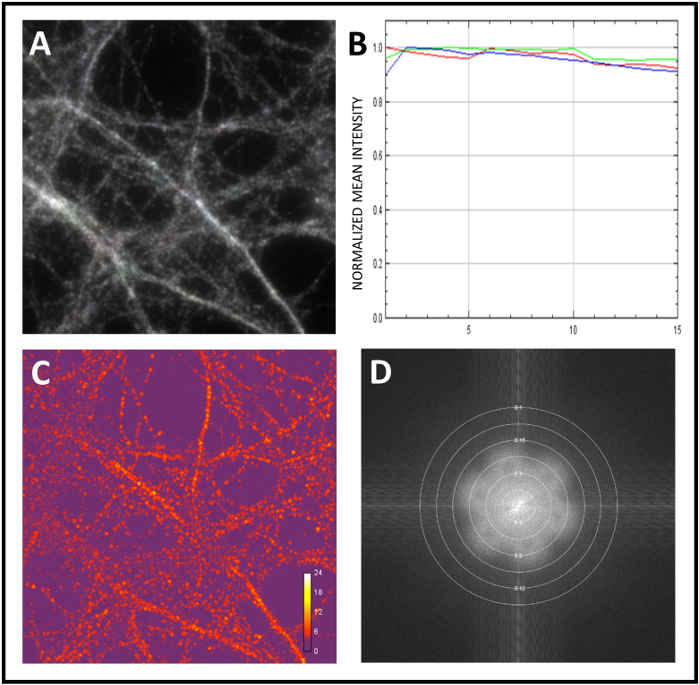
Figure 4 : Évaluation de la qualité des images brutes et reconstruites à l’aide de SIMcheck. (A) Analyse de variation de mouvement et d’illumination à l’aide de SIMCheck. Le signal gris à blanc représente l’illumination homogène et l’absence de mouvement lors de l’acquisition. (B) Profil d’intensité de canal obtenu en analysant l’image brute. Dans cet exemple, il y a une variation d’intensité minime, pour suggérer un manque ou un blanchiment ou des fluctuations. (C) Modulation brute Contraste pour calculer le rapport de la modulation contraste-bruit dans l’image. La carte thermique montre des variations de contraste de modulation. (D) Reconstruit la parcelle fourier pour analyser le spectre de l’amplitude Fourier afin de déterminer la résolution efficace de la reconstruction. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 5 : Évaluation de la qualité de l’image à super résolution à l’aide de NanoJ-SQUIRREL. (A) Image de référence de super-résolution avec des artefacts. (B) Image de référence de super-résolution de bonne qualité. (C) Image représentant la carte d’erreur NanoJ-SQUIRREL de A. Les zones plus claires représentent des artefacts à grande échelle, tandis que les plus sombres représentent une reconstruction correcte. (D) Carte d’erreur NanoJ-SQUIRREL de B. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 6 : exemple d’image SIM. (A) Microscopie confocale des neurones primaires. Un objectif 40X a été choisi pour obtenir une vue d’ensemble de l’échantillon tout en maintenant une bonne résolution. Les cellules ont été immunostained pour SUMO1 (vert), PSD95 (rouge) et MAP2 (mauve). DAPI a été utilisé pour tacher les noyaux. Barre d’échelle 50 μm. Les images ont été affichées sous forme de projection Z. (B) images SIM pour SUMO1 et PSD95 sur la zone mise en évidence dans la boîte verte dans le panneau A en utilisant un objectif 100X. Les pointes de flèche rouges indiquent la position de l’ensemble indiqué dans a utilisé pour calculer le profil d’intensité. Barre d’échelle 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
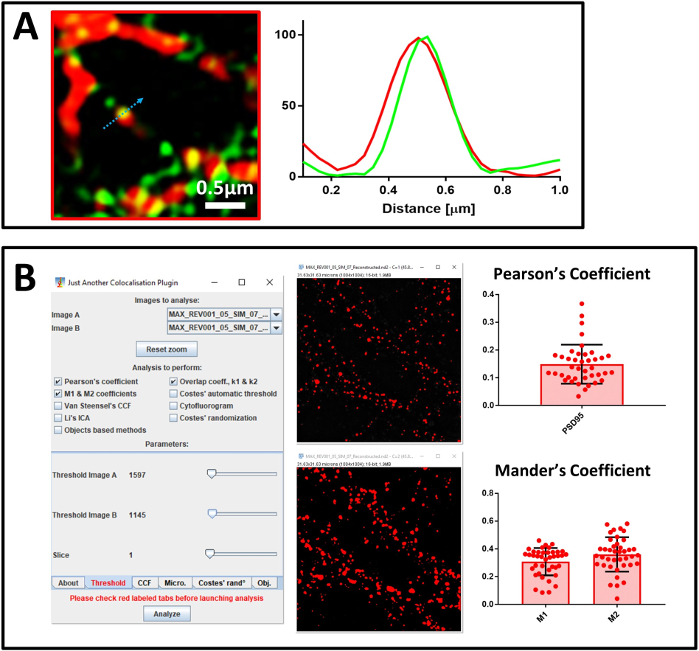
Figure 7 : Analyse de co-localisation. (A) Image super-résolue des neurones primaires tachés d’un anticorps contre SENP1 (en vert) et drebrin (en rouge), barre d’échelle 0,5 μm, et son profil d’intensité. Les valeurs du graphique ont été normalisées pour chaque canal à 100 (unité arbitraire) et correspondent à l’intensité du pixel indiquée par la flèche bleue. (B) Analyse utilisant JACoP pour calculer le coefficient de corrélation de Pearson et le coefficient de Mander entre SERP1 et drebrin. Les fenêtres de la configuration du plug-in et du seuil visuel sont affichées. Le coefficient de Mander est exprimé par deux valeurs – la fraction SERP1 qui co-localise avec la drebrine (M1) et la fraction de dérébrrine qui co-localise avec SERP1 (M2). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
L’élucidation de la structure et de la composition de la synapse est cruciale pour comprendre les processus physiologiques et pathologiques qui régulent la mémoire et la cognition. Alors que dans l’état normal, les synapses sont les éléments constitutifs de la mémoire, ils sont également à la base de troubles neurologiques complexes tels que la maladie d’Alzheimer32. Le protocole décrit ici sert à étudier la co-localisation des protéines neuronales avec une technique de microscopie super-résolution appelée SIM. En utilisant un modèle particulier d’éclairage, la SIM peut atteindre une résolution d’environ 0,1 μm, qui convient à l’étude des synapses, qui mesurent normalement entre 0,03 et 0,15 μm. Pour plus de détails encore, d’autres techniques de super-résolution telles que STED (Stimulated Emission Ecuttement Microscopy), PALM (PhotoActivated Localization Microscopy) ou STORM (Stochastic Optical Reconstruction Microscopy), qui peuvent atteindre une résolution de 10-20 nm, peuvent être appliquées33.
Ici, nous décrivons l’analyse de la co-localisation des protéines cibles avec des marqueurs synaptiques dans les neurones primaires. Le protocole peut être appliqué à n’importe quelle culture primaire des cellules neuronales, telles que les neurones hippocampal, cérebelaires ou corticaux et même aux cultures de neurones primaires qui n’appartiennent pas au système nerveux central, tels que les neurones du système nerveux entérique. La clé de l’analyse au niveau de la super-résolution, cependant, sont les réactifs utilisés lors de l’acquisition, tels que les couvercles en chambre et les solutions de montage compatibles avec l’indice de diffraction de l’objectif. Nous avons utilisé des couvercles de chambre pour leur facilité d’utilisation, mais la méthode classique et moins coûteuse de culture des neurones primaires sur le verre de couverture enduit est néanmoins valable, en particulier avec une haute précision #1,5 H (0,17 mm) de verre de couverture. En outre, un milieu de montage qui peut atteindre un indice de réfraction aussi proche que possible de l’indice de réfraction du verre (1,52) et une huile d’immersion pour l’objectif 100x avec un indice de réfraction de 1.515 devrait être utilisé. La température ambiante constante et les tables stabilisées sont également obligatoires pour garantir l’exactitude des acquisitions.
Nous utilisons à la fois dyLight et Alexa anticorps secondaires dans les études SIM. En raison de leurs pics étroits d’excitation et d’émission et de leur bon rendement quantique, ils sont indiqués pour les techniques de super-résolution qui exigent le meilleur rapport signal/bruit. Dempsey et coll. ont comparé Alexa, dyLight et d’autres fluorophores pour l’imagerie super-résolution34.
Lors de l’acquisition, nous avons régulièrement réglé l’appareil photo à 1 MHz sur 10 MHz. 1 MHz, grâce à une vitesse d’acquisition plus lente, donne aux images plus de précision et moins de bruit que 10 MHz. 1 MHz mode de lecture peut également enregistrer avec une profondeur un peu de 16 bits (par rapport à la maximum 14 bits de 10 MHz), donnant plus d’informations de couleur et un gradient de couleur plus précis pour les images. Cependant, 10 MHz, avec sa vitesse, est utile pour les images en direct. Pour éviter le blanchiment et préserver le fluorophore, nous mettons également la puissance laser aussi faible que possible. Pour améliorer l’intensité du signal, les valeurs de gain peuvent être augmentées. Il est à noter que le gain inférieur garantit des images plus propres sans augmenter le bruit. En général, les meilleurs résultats sont obtenus lors de l’imagerie dans un rayon de 7 μm à partir du bas du couvercle chambrelé. Ceci est particulièrement important lors de l’utilisation d’un objectif 100x avec immersion à l’huile. Si une acquisition plus profonde à travers les cellules/tissus est nécessaire, un meilleur choix peut être l’utilisation d’un objectif 60x avec immersion dans l’eau.
L’un des principaux défis dans l’exécution des études SIM est la reconstruction d’image35. L’obtention d’images super-résolues sans artefacts et aberration nécessite non seulement l’utilisation de conditions expérimentales ad hoc, mais aussi un étalonnage minutieux du système et l’optimisation des paramètres pour obtenir les images finales. Dans le protocole, nous décrivons comment éviter certaines des erreurs les plus courantes en évaluant l’étalonnage du système et l’analyse de la qualité des images brutes et reconstruites. Plus précisément, nous décrivons l’utilisation des plugins ImageJ SIMCheck et NanoJ-SQUIRREL pour assurer les paramètres corrects de l’instrument afin d’éviter les artefacts communs d’images super-résolues. Les applications permettent une évaluation impartiale de la qualité des images finales qui n’est pas basée sur l’analyse subjective des résultats par rapport à la connaissance préalable des structures d’étude.
Nous suggérons d’utiliser la synaptophysine et le PSD95 ou la drebrine comme marqueurs pré et post-synaptiques, bien que d’autres marqueurs soient également valides. Un énorme corpus de littérature décrit des protéines telles que le basson comme marqueurs synaptiques36,37. Il est à noter que les marqueurs pré- et postsynaptiques sont toutefois présents dans toute la cellule, à l’exclusion du noyau. Une grande partie de leur signal est non synaptique, mais représente des protéines dans le transport ou la dégradation, le fond ou d’autres artefacts. Il est donc important de choisir soigneusement le domaine de l’analyse. Nous utilisons le signal d’anticorps MAP2 pour choisir des terminaux axones et dendritiques.
Dans l’analyse de la co-localisation, nous utilisons deux approches. La première est une approche visuelle, basée sur une analyse de profil qui montre des événements uniques de co-localisation et identifie la contribution de chaque canal. Une mise en garde de cette approche, cependant, est la faible puissance statistique. Pour cette raison, nous avons également décidé d’utiliser une deuxième méthode basée sur l’analyse d’un plus grand nombre d’événements représentatifs de l’ensemble du champ de chaque image. Cette méthode est basée sur le calcul du coefficient de corrélation du Pearson et des coefficients M1 et M2 de Mander. Nous utilisons le coefficient de corrélation de Pearson pour décrire le chevauchement des signaux dans l’image et M1 et M2 de Mander pour décrire la co-localisation réciproque entre les signaux d’intérêt38. Pour le calcul, nous utilisons le plugin ImageJ JACoP, car il dispose d’une fonctionnalité qui vous permet de définir un seuil manuel pour écarter toute contribution de fond à l’analyse, particulièrement critique pour l’analyse de Mander.
Déclarations de divulgation
Les auteurs n’ont rien à révéler.
Remerciements
Les auteurs tiennent à remercier Edoardo Micotti pour la critique constructive du manuscrit. Cette étude a été soutenue par BrightFocus A2019296F, par Fondo di Beneficenza - Gruppo Intesa Sanpaolo (LC), par Fondazione Regionale per la Ricerca Biomedica (Care4NeuroRare CP_20/2018) (CN) et par le Sk’odowska-Curie Innovative Training Network (JK).
matériels
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan blue solution | Thermo Fisher Scientific | 15250061 | Chemical |
| 70 µm filter | Corning | 352350 | Equiment |
| Alexa | Thermo Fisher Scientific | - | Antibody |
| Antibody SENP1 | Santa Cruz | sc-271360 | Antibody |
| B27 Supplement | Life Technologies | 17504044 | Chemical |
| Bovine serum albumin | Merck | 5470 | Chemical |
| CaCl2 | Merck Life Science | 21115 | Chemical |
| Chambered coverslips | Ibidi | 80826 | Equiment |
| DyLight | Thermo Fisher Scientific | - | Antibody |
| FBS (Hyclone) | GIBCO | SH3007002 (CHA1111L) | Serum |
| FluoSpheres carboxylate-modified microspheres, 0.1 μm, yellow–green fluorescent | Thermo Fisher Scientific | F8803 | Equiment |
| Glucose | Merck Life Science | G8769 | Chemical |
| Glutamax | GIBCO | 35050061 | Chemical |
| HEPES | Merck Life Science | H3537 | Chemical |
| L-Cystein | Merck Life Science | C6852-25g | Chemical |
| MAP2 | Merck | AB15452 | Antibody |
| MEM | Life Technologies | 21575022 | Medium |
| MgCl | Merck Life Science | M8266 | Chemical |
| NaOH | VWR International | 1,091,371,000 | Chemical |
| Neurobasal A | Life Technologies | 10888022 | Medium |
| N-SIM Super Resolution Microscope | Nikon | - | Instrument |
| Papain | Merck Life Science | P-3125 | Chemical |
| paraformaldehyde | Thermo Fisher Scientific | 28908 | Chemical |
| Pen/Strep 10x | Life Technologies | 15140122 | Chemical |
| phosphate-buffered saline | Gibco | 10010023 | Chemical |
| Poly-L lysine | Sigma | P2636 | Chemical |
| ProLong Diamond Glass Antifade Mountant | Thermo Fisher Scientific | P36970 | Chemical |
| PSD95 | NeuroMab | K28/43 | Antibody |
| Round coverglass | Thermo | 12052712 | Equiment |
| SUMO1 | Abcam | ab32058 | Antibody |
| Synaptophysin | Merck | S5768 | Antibody |
| Triton X-100 | Merck | T8787 | Chemical |
| Trypsin inhibitor | Merck Life Science | T9003-500MG | Chemical |
Références
- Foster, M., Sherrington, C. S. . A textbook of physiology, part three: The central nervous system (7th ed.). , (1897).
- Choquet, D., Triller, A. The Dynamic Synapse. Neuron. 80 (3), 691-703 (2013).
- McAllister, A. K. Dynamic Aspects of CNS Synapse Formation. Annual Review of Neuroscience. 30 (1), 425-450 (2007).
- Yuzaki, M. Two Classes of Secreted Synaptic Organizers in the Central Nervous System. Annual Review of Physiology. 80 (1), 243-262 (2018).
- Baddeley, D., Bewersdorf, J. Biological Insight from Super-Resolution Microscopy: What We Can Learn from Localization-Based Images. Annual Review of Biochemistry. 87 (1), 965-989 (2018).
- Sigal, Y. M., Zhou, R., Zhuang, X. Visualizing and discovering cellular structures with super-resolution microscopy. Science. 361 (6405), 880-887 (2018).
- Vangindertael, J., et al. An introduction to optical super-resolution microscopy for the adventurous biologist. Methods and Applications in Fluorescence. 6 (2), 022003 (2018).
- Badawi, Y., Nishimune, H. Super-resolution microscopy for analyzing neuromuscular junctions and synapses. Neuroscience Letters. 715, 134644 (2020).
- Scalisi, S., Barberis, A., Petrini, E. M., Zanacchi, F. C., Diaspro, A. Unveiling the Inhibitory Synapse Organization Using Superresolution Microscopy. Biophysical Journal. 116 (3), 133 (2019).
- Yang, X., Specht, C. G. Subsynaptic Domains in Super-Resolution Microscopy: The Treachery of Images. Frontiers in Molecular Neuroscience. 12, (2019).
- Monro, T. Beyond the diffraction limit. Nature Photonics. 3 (7), 361 (2009).
- Won, R. Eyes on super-resolution. Nature Photonics. 3 (7), 368-369 (2009).
- Wegel, E., et al. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific Reports. 6 (1), 27290 (2016).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Gustafsson, M. G. L., et al. Three-Dimensional Resolution Doubling in Wide-Field Fluorescence Microscopy by Structured Illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Brose, N., O'Connor, V., Skehel, P. Synaptopathy: dysfunction of synaptic function. Biochemical Society Transactions. 38 (2), 443-444 (2010).
- Tyebji, S., Hannan, A. J. Synaptopathic mechanisms of neurodegeneration and dementia: Insights from Huntington's disease. Progress in Neurobiology. 153, 18-45 (2017).
- Won, H., Mah, W., Kim, E. Autism spectrum disorder causes, mechanisms, and treatments: focus on neuronal synapses. Frontiers in Molecular Neuroscience. 6, (2013).
- Pfeiffer, B. E., Huber, K. M. The State of Synapses in Fragile X Syndrome. The Neuroscientist. 15 (5), 549-567 (2009).
- Pavlowsky, A., Chelly, J., Billuart, P. Emerging major synaptic signaling pathways involved in intellectual disability. Molecular Psychiatry. 17 (7), 682-693 (2012).
- Senatore, A., Restelli, E., Chiesa, R. Synaptic dysfunction in prion diseases: a trafficking problem. International Journal of Cell Biology. 2013, 543803 (2013).
- Colnaghi, L., et al. Super Resolution Microscopy of SUMO Proteins in Neurons. Frontiers in Cellular Neuroscience. 13, (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Müller, M., Mönkemöller, V., Hennig, S., Hübner, W., Huser, T. Open-source image reconstruction of super-resolution structured illumination microscopy data in ImageJ. Nature Communications. 7 (1), 10980 (2016).
- Ball, G., et al. SIMcheck: a Toolbox for Successful Super-resolution Structured Illumination Microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Schaefer, L. H., Schuster, D., Schaffer, J. Structured illumination microscopy: artefact analysis and reduction utilizing a parameter optimization approach. Journal of Microscopy. 216 (2), 165-174 (2004).
- Culley, S., et al. NanoJ-SQUIRREL: quantitative mapping and minimisation of super-resolution optical imaging artefacts. Nature Methods. 15 (4), 263-266 (2018).
- Manders, E. M. M., Verbeek, F. J., Aten, J. A. Measurement of co-localization of objects in dual-colour confocal images. Journal of Microscopy. 169 (3), 375-382 (1993).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander’s overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
- Bolte, S., Cordelières, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224, 213-232 (2006).
- Bae, J. R., Kim, S. H. Synapses in neurodegenerative diseases. BMB Reports. 50 (5), 237-246 (2017).
- Godin, A. G., Lounis, B., Cognet, L. Super-resolution Microscopy Approaches for Live Cell Imaging. Biophysical Journal. 107 (8), 1777-1784 (2014).
- Dempsey, G. T., Vaughan, J. C., Chen, K. H., Bates, M., Zhuang, X. Evaluation of fluorophores for optimal performance in localization-based super-resolution imaging. Nature Methods. 8 (12), 1027-1036 (2011).
- Karras, C., et al. Successful optimization of reconstruction parameters in structured illumination microscopy - A practical guide. Optics Communications. 436, 69-75 (2019).
- Bereczki, E., et al. Synaptic markers of cognitive decline in neurodegenerative diseases: a proteomic approach. Brain: A Journal of Neurology. 141 (2), 582-595 (2018).
- Gilestro, G. F., Tononi, G., Cirelli, C. Widespread Changes in Synaptic Markers as a Function of Sleep and Wakefulness in Drosophila. Science. 324 (5923), 109-112 (2009).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander's overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon