Method Article
Imágenes de Superresoridad para estudiar la co-localización de proteínas y marcadores sinápticos en neuronas primarias
En este artículo
Resumen
Este protocolo muestra cómo emplear la microscopía de superresorción para estudiar la co-localización de proteínas en cultivos neuronales primarios.
Resumen
Las sinapsis son los elementos funcionales de las neuronas y sus defectos o pérdidas están en la base de varios trastornos neurodegenerativos y neurológicos. Los estudios por imágenes se utilizan ampliamente para investigar su función y plasticidad en condiciones fisiológicas y patológicas. Debido a su tamaño y estructura, los estudios de localización de proteínas requieren técnicas de imagen de alta resolución. En este protocolo, describimos un procedimiento para estudiar en las neuronas primarias la co-localización de proteínas diana con marcadores sinápticos a nivel de superresción utilizando microscopía de iluminación estructurada (SIM). SIM es una técnica de iluminación de luz estanco que duplica la resolución espacial de la microscopía de campo ancho, alcanzando un detalle de alrededor de 100 nm. El protocolo indica los controles y la configuración necesarios para estudios de co-localización robustos y una visión general de los métodos estadísticos para analizar los datos de imagen correctamente.
Introducción
La comprensión y la visión de la sinapsis ha cambiado enormemente desde su primera descripción por Foster y Sherrington en 18971. Desde entonces, nuestro conocimiento de la comunicación neuronal y los procesos moleculares detrás de ella ha crecido exponencialmente2. Ha quedado claro que las sinapsis pueden considerarse como un sistema de dos compartimentos: un compartimento presináptico que contiene vesículas para la liberación de neurotransmisores y un compartimento postsináptico con receptores3. Esta visión simplista, en los últimos veinte años, se ha convertido en una compleja red de proteínas necesarias para transducir la unión del transmisor a la señalización4.
Las ganancias en el entendimiento se deben en parte a las técnicas de superrescencia que superaron el límite de difracción de la microscopía de luz convencional para adaptarse a la dimensión de las sinapsis mejores5,6,7,8,9,10. Debido al límite de difracción, un microscopio óptico no puede alcanzar una resolución superior a 200 nm lateralmente11,12. Para eludir este límite, se crearon técnicas de superrescencia, utilizando diferentes enfoques y alcanzando diferentes resoluciones de límite de subdifacción: SIM, STED (Microscopía de Agotamiento de Emisiones Estimuladas), PALM (Microscopía de Localización Fotoactiva) y STORM (Microscopía de Reconstrucción Óptica Estocástica)13,,14. SIM duplica la resolución espacial de los sistemas de microscopía de campo ancho basados en láser mediante la inserción de una rejilla de difracción en la trayectoria del haz de excitación15. La rejilla móvil difracta los rayos láser para crear un patrón de iluminación conocido, generalmente rayas. Este patrón de luz estructurado específicamente se superpone a la distribución espacial desconocida del tinte fluorescente (de la muestra). Los flecos de interferencia formados por los dos patrones codifican para detalles finos indistinguibles con microscopía de campo ancho normal. La imagen superconsfectada final se obtiene combinando y decodificando con métodos matemáticos varias imágenes crudas de la misma muestra obtenidas por las traducciones y rotaciones de la rejilla de difracción. La resolución de las imágenes superconsueltas alcanza los 100 nm en las direcciones lateral y 500 nm en las direcciones axiales para 2D-SIM15 o 100 nm en las direcciones lateral y 250 nm en las direcciones axiales para 3D-SIM16.
La nueva comprensión de la sinapsis es aún más importante a la luz de los muchos trastornos neurológicos donde la disfunción sináptica juega un papel importante en la aparición y progresión17,,18. Enfermedad de Alzheimer, Síndrome de Down, Enfermedad de Parkinson, enfermedades priónicas, epilepsia, trastornos del espectro autista y síndrome X frágil entre otros se han relacionado con anomalías en la composición sináptica, morfología y función19,,20,,21,,22.
Recientemente, utilizando un conjunto de anticuerpos específicos de SUMO, utilizamos SIM para mostrar la co-localización en las neuronas primarias del hipocampo de las proteínas SUMO con los marcadores pre y postsinápticos sinaptofisina y PSD95 en el nivel de superrescresía23. Esto nos permitió confirmar la evidencia de microscopía bioquímica y confocal de la localización SUMO en las neuronas.
Aquí, describimos un protocolo para estudiar la localización de proteínas en las neuronas primarias del hipocampo de ratón. Al mismo tiempo, este protocolo puede adaptarse a diferentes tipos de cultivos neuronales primarios.
Protocolo
1. Culturas primarias
- Neuronas primarias de hipocampo de ratón de cultivo en cubreobjetos con cámara (como Ibidi μ-Slide 8 Well o Nunc Lab-Tek Chambered Coverglass) que coinciden con el requisito objetivo para el espesor de la cubierta de #1,5 (0,17 mm).
- Cubiertas con cámara de abrigo con 100 ml de poli-L-lisina (100 g/ml).
- Al día siguiente, lave los cubreobjetos a cámara dos veces con solución salina estéril tamponada por fosfato (PBS).
- Para obtener neuronas primarias de ratón, aislar hipocampos de cachorros P1-P423.
- Coloque el hipocampo diseccionado en 10 ml de medios de disección (Tabla 1) y déjelos depositar en la parte inferior del tubo.
- Con una pipeta estéril, retire cuidadosamente el soporte de disección, dejando el hipocampo inalterado en la parte inferior del tubo.
- Añadir 10 ml de Media 1 (Tabla 1) al hipocampo e incubar durante 30 minutos a 37oC.
- Con una pipeta estéril, retire cuidadosamente el Media 1, dejando el hipocampo inalterado en la parte inferior del tubo.
- Añadir 10 ml de Media 2 (Tabla 1) y dejar el tubo centrífugo (tapado) debajo del capó horizontalmente durante 45 minutos.
- Deje reposar el tubo de centrífuga verticalmente para permitir que el tejido se asiente en la parte inferior del tubo.
- Con una pipeta estéril, retire cuidadosamente media 2, dejando el hipocampo inalterado en la parte inferior del tubo centrífugo.
- Añadir 2 mL de Medios 3 (Tabla 1).
- Usando una pipeta p1000 con una punta filtrada, disocia mecánicamente las células del tejido.
- Transfiera el sobrenadante, en el que se encuentran las neuronas aisladas, a un tubo centrífugo de 15 ml.
- Centrifugar la suspensión celular durante 2 min a 300 x g a temperatura ambiente (RT).
- Después de la centrifugación, las células se encuentran en la parte inferior del tubo centrífugo. Con una pipeta estéril, deseche el sobrenadante.
- Células resuspendidas en 1 ml de Medios 4.
- Utilice un filtro de 70 m para eliminar las celdas no disociadas.
- Contar las células viables en una cámara de B-rker añadiendo 1 l de 0,4 % de solución azul de Trippan a 19 l de la suspensión celular.
- Células de placa a 70.000 células/pozo en un volumen de 200 l por pocócil.
- Dejar que las células se adhieran durante 2 h en una incubadora humidificada a 37oC y 5% co2.
- Saque los cubreobjetos con cámara de la incubadora y sustituya cuidadosamente el medio por 200 l de medios de cultivo.
- Dejar los cubreobjetos con cámara en una incubadora humidificada a 37oC y 5% de CO2.
- Sustituya un tercio del medio por medios de cultivo frescos cada 5-7 días.
- Espere hasta que las neuronas primarias del hipocampo estén completamente maduradas (12-14 días después del enchapado) para realizar estudios de co-localización.
2. Tinción de inmunofluorescencia
- Tome los cubreobjetos con cámara de la incubadora.
- Retire el medio.
- Lave rápidamente los pozos con 200 ml de PBS.
- Añadir 4% de paraformaldehído (PFA) en PBS (200 l/pozo) a las neuronas para fijarlas rápidamente.
- Incubar las células durante 15 minutos en RT.
- Retire la solución PFA.
- Permeabilizar las células mediante la adición de PBS con 0.2% Tritón X-100 (200 l/pozo).
- Incubar durante 1 min en RT.
- Retirar la solución e incubar las muestras con albúmina sérica bovina (BSA) en PBS (200 l/pozo) durante 1 h en RT para cubrir pasivamente todas las superficies de unión libres de la placa con una proteína irrelevante para el análisis. Un búfer de bloqueo basado en BSA sin Triton X-100 reduce el fondo de anticuerpos de manera más eficiente que el mismo búfer con 0,2% Triton X-100.
- Retire la solución.
- Añadir el anticuerpo primario de elección diluido en una solución de PBS que contenga 1% de BSA y 0,2% Tritón X-100 (120-200 l/pozo, dependiendo de la dilución y disponibilidad del anticuerpo). Incubar las muestras durante 2 h.
- Como control negativo, no agregue ningún anticuerpo primario a uno de los pozos. Múltiples anticuerpos de diferentes especies contra diferentes objetivos se pueden utilizar al mismo tiempo. Utilice un anticuerpo contra el MAP2 (un marcador neuronal) criado en el pollo, un anticuerpo contra el PSD95 o la sinaptofisina levantado en el ratón y un anticuerpo contra una proteína diana criada en el conejo. Esto permite análisis SIM de tres colores.
- Lave rápidamente los pozos tres veces con PBS (200 l/pozo).
- Añadir anticuerpos secundarios (se pueden utilizar anticuerpos secundarios dyLight y Alexa) diluidos en una solución de PBS que contenga 1% de BSA y 0,2% tritón X-100 (200 l/pozo). Incubar las muestras durante 1 h en RT.
- Lave rápidamente los pozos tres veces con PBS (200 l/pozo).
- Añadir el tinte Hoechst a una concentración de 1 g/ml diluido en PBS (200 l/pozo) a los núcleos de tinción. Incubar las muestras durante 10 minutos en RT.
- Lave rápidamente los pozos dos veces con PBS.
- Monte las celdas con un montura compatible con SIM. Utilice 10 l/pozo de soporte antifada de vidrio ProLong.
- Cubra y proteja las celdas con una cubierta (por ejemplo, una cubierta redonda con un diámetro de 8 mm). También se pueden utilizar plazas.
- Guarde los cubreobjetos con cámara en RT y espere al menos 48 horas antes de adquirir imágenes. Diamond Glass requiere al menos dos días de curado antes de las adquisiciones de superresor resolución.
3. Control de especificidad de anticuerpos
NOTA: Utilice dos estrategias para asegurar la especificidad de los anticuerpos. La primera estrategia es utilizar al menos dos anticuerpos diferentes dirigidos al mismo sustrato. La segunda estrategia es la neutralización de anticuerpos por incubación con el objetivo de proteína purificada o el epítopo utilizado para aumentar el anticuerpo.
- Incubar el anticuerpo de elección con cinco veces el exceso del objetivo recombinante o epítopo durante 1 h en RT en 1% de BSA en PBS.
- Después de la incubación, utilice el anticuerpo neutralizado a la concentración habitual para la tinción como se describió anteriormente a partir de 2.11.
4. Calibración del microscopio
NOTA: Utilizamos rutinariamente un sistema de microscopio de superconorción N-SIM fabricado por Nikon para los estudios de super resolución. Sin embargo, varias otras empresas también ofrecen microscopios de superrescresía en sus catálogos. Aunque se describen indicaciones específicas para el sistema N-SIM de Nikon, las instrucciones siguientes se pueden generalizar en otros sistemas. Antes de la adquisición de imágenes SIM, el sistema requiere una calibración adecuada con perlas fluorescentes de tamaño de subrescresación específicas. Un ejemplo son las microesferas TetraSpeck. Estas perlas están teñidas con diferentes tintes fluorescentes para permitir la calibración de diferentes láseres con una sola muestra.
- En un baño de agua sonicar alrededor de 1.8 x 108 microesferas fluorescentes durante 10 minutos. El sistema N-SIM de Nikon requiere una muestra de perlas multicolores escasamente pobladas para la calibración. Esto podría diferir para otros sistemas que requieren una capa única densa de perlas fluorescentes de tamaño de subresión. Ajuste el número de partículas fluorescentes en consecuencia.
- Diluir las microesferas fluorescentes 1:500 en agua destilada doble.
- Sonicar una segunda vez durante 10 minutos adicionales.
- Pipetear 15 l de las perlas diluidas en un pozo de un cubreobjetos con cámara.
- Deje secar la solución durante 5 minutos en RT.
- Añada 10 l de la solución de montaje y coloque un cubreobjetos de 8 mm en la parte superior.
- Espere al menos 48 horas para permitir el curado adecuado.
- Encienda el microscopio y los láseres.
- Deje que el sistema se caliente para alcanzar el equilibrio térmico de todos los componentes del microscopio. El sistema de microscopio de superconorción N-SIM requiere al menos 3 horas.
- Seleccione el objetivo 100x.
- Inicie la calibración alineando los láseres con el centro del bloque de rejilla de difracción. En el sistema N-SIM, una perilla de micrómetros y una cámara dedicada permiten centrar los haces de luz al objetivo.
- Inserte el cubreobjetos con cámara en el microscopio para su visualización. Ajuste el sistema al espesor de la cubierta con cámara ajustando el collar de corrección objetiva. El software NIS, el software propietario proporcionado con los sistemas de microscopio de superresorción N-SIM, tiene una función automática para regular los collares de corrección.
- Ajuste el enfoque del bloque de rejilla para cada canal para garantizar una iluminación de patrón estructurado enfocada en la muestra. El software NIS proporciona una función automática para esta tarea.
- A continuación, adquiera imágenes 3D-SIM en bruto de las microesferas multicolores. Reconstruya las imágenes en bruto para obtener una imagen superconsuelta utilizando el software del microscopio o la plataforma de software de código abierto para el análisis de imágenes biológicas ImageJ24 y el plugin fairSIM25.
- Calcular, para cada longitud de onda separada, la transformación de Fourier de la imagen superconsuelta obtenida en 4.14. Si la imagen transformada no puede obtener un patrón correcto de flores, reinicie la calibración desde 4.11 ya que no se ha logrado la super-resolución.
- En la imagen superconsútil, seleccione una sola microesfera y calcule su perfil de intensidad para cada canal para medir la resolución alcanzada. Ahora debe estar cerca de 100 nm lateralmente.
- A continuación, realice el registro de canales superponiendo una adquisición multicanal de las microesferas. El objetivo es colisionar todas las señales de canal de forma lateral y axial. Esto eliminará las aberraciones cromáticas debido a la desalineación de los diferentes canales y ayudará al análisis de la co-localización.
- Confirme la calidad de la calibración utilizando las funciones "Illumination Phase Steps" y "Illumination Pattern Focus" de SIMcheck26, un conjunto de plugins para la aplicación de código abierto ImageJ. Con este fin, prepare un cubreobjetos con cámara para obtener una sola capa densa de microesferas TetraSpeck y adquiera una imagen 3D-SIM de la muestra. Analice la imagen en ImageJ y, si se detectan aberraciones, reinicie la calibración del microscopio desde el paso 4.11.
5. Adquisición
- Comience a analizar la muestra utilizando un objetivo 40x en modo confocal o de campo ancho. Esto permite la navegación a la muestra, manteniendo buenos detalles y un gran campo de visión.
- Utilice la señal de anticuerpos MAP2 para identificar un área que represente procesos neuronales.
- Adquiera imágenes de la muestra en modo confocal para determinar la calidad de la tinción. La mala calidad confocal se reflejará en la mala calidad de la SIM, por lo que se requiere que las muestras se desechan.
- Si el área y la calidad de las imágenes son satisfactorias, cambie el objetivo a 100x.
- Aplique aceite al objetivo 100x.
- Adquirir una imagen de campo ancho o confocal que se utilizará más adelante para evaluar la calidad de la imagen superconstionada (Figura 1A,B).
- Cambie al modo 3D-SIM.
- Mediante las ventanas de diálogo para establecer parámetros para la adquisición, seleccione la configuración de profundidad de bits más alta disponible para maximizar la información de color. Normalmente, 16 bits es la opción estándar. Además, para mejorar la relación señal-ruido, seleccione un valor de baja frecuencia para la adquisición, como 1 MHz.
- Usando ventanas de histograma, ajuste la potencia de los láseres para obtener una respuesta lineal de la señal. Para evitar la pérdida de información, limite los píxeles saturados en las imágenes. El sistema N-SIM utiliza una cámara Andor iXon3. Cuando trabaje a 16 bits, elija una intensidad objetivo de 16.000 para garantizar la respuesta lineal de la cámara. Alternativamente, elija un rango entre 30,000-45,000 para maximizar el rango dinámico de la adquisición.
- Establezca la potencia del láser entre 0,1% y 50% al tomar imágenes de las muestras y los tiempos de exposición entre 50 ms y 2 s. Las potencias láser superiores al 50% pueden causar un fotoblanqueo rápido de los fluoróforos en uso.
- Comience a adquirir las imágenes en modo 3D-SIM.
- Utilice SIMcheck, un conjunto de plugins gratuitos para ImageJ, para evaluar la calidad de adquisición de las imágenes sin procesar.
- Si SIMCheck no detecta artefactos ni problemas de calidad, adquiera un mínimo de 10 imágenes de 4 réplicas técnicas para permitir el análisis estadístico.
6. Postproducción: reconstrucción de imágenes
NOTA: Las imágenes adquiridas en 3D-SIM son imágenes sin procesar que deben procesarse para obtener imágenes superconsueltas reconstruidas. La reconstrucción incorrecta de imágenes sin procesar puede dar lugar a artefactos que afectarían al análisis de las muestras. Por lo tanto, se debe prestar una gran atención a la elección adecuada de los parámetros de reconstrucción.
- Procesar las imágenes sin procesar utilizando el software de análisis de reconstrucción del microscopio para obtener una imagen superconsuel(Figura 1C). Alternativamente, utilice el plugin gratuito ImageJ fairSIM para reconstruir imágenes sin procesar.
- Calcule la transformación de Fourier de las imágenes superconsueltas utilizando el software de reconstrucción del microscopio o el plugin ImageJ SIMCheck. Una buena imagen reconstruida debe devolver, para cada canal, una imagen similar a una flor. Si las imágenes reconstruidas no pueden recrear una forma similar a una flor, reinicie desde las imágenes sin procesar y reconstruya modificando los parámetros de reconstrucción como el filtrado De Wiener, la apodización y la supresión de orden cero27. En el software NIS, utilizando la vista previa para supervisar cómo el cambio de los parámetros afecta a la imagen final resuelta, modifique los parámetros i) Contraste de modulación de iluminación, ii) Supresión de ruido de alta resolución y iii) Supresión de de enfoque.
- A continuación, analiza la imagen reconstruida para detectar de forma imparcial artefactos mediante NanoJ-SQUIRREL28, un plugin basado en ImageJ para evaluar la calidad de las imágenes superconsúcedas.
- Si NanoJ-SQUIRREL detecta artefactos, reinicie desde las imágenes sin procesar y reconstruyalos modificando los parámetros de reconstrucción, como el filtrado Wiener, la apodización y la supresión de orden cero. En el software NIS, utilizando la vista previa para supervisar cómo el cambio de los parámetros afecta a la imagen final resuelta, modifique los parámetros Contraste de modulación de iluminación, Supresión de ruido de alta resolución y Supresión de desenfoque.
- Utilice las imágenes superconsueltos para calcular el perfil de co-localización y/o los coeficientes de Pearson y Mander.
7. Co-localización con análisis de perfil
NOTA: Como primer paso para estudiar la co-localización entre marcadores sinápticos y una proteína de interés, tome una imagen superconsuelta y analice un solo locus para determinar la superposición de la señal.
- Identifique un solo locus en la imagen superconsuel.
- Obtener los perfiles de intensidad de las señales fluorescentes del locus de interés.
- Exporte los datos.
- Utilice GraphPad Prism, o un software de análisis similar, para normalizar todos los picos de señal y obtener intensidades de señal comparables para cada canal con el objetivo final de determinar la co-localización específica del locus.
8. Cuantificación de los coeficientes de Pearson y Mander
NOTA: Si el análisis de perfil ha sugerido una sola co-localización de locus, se puede realizar un análisis más general de toda la imagen calculando los coeficientes29,,30de Pearson y Mander.
- Utilice JACoP31, un plug-in ImageJ, para determinar los dos parámetros de la co-localización: Pearson's y Mander's.
9. Análisis estadístico
- Utilice GraphPad Prism, o un software de análisis y gráficos similar, para procesar los datos recopilados con JACoP.
- Utilice al menos 40 imágenes SIM para cada condición analizada para obtener gráficos y para la relevancia estadística.
Resultados
Aquí presentamos el flujo de trabajo estándar para estudiar la co-localización de proteínas neuronales. Primero calibramos el microscopio y luego realizamos el análisis SIM de las muestras. Para calibrar el sistema, utilizamos microesferas fluorescentes de 0,1 m de diámetro. Al obtener imágenes 3D-SIM superconsúcesas de los cordones, los datos de imagen subyacentes se transforman en Fourier para volver a convertirlas en una representación de frecuencia espacial. En la Figura 2A,el patrón de flores distintivo se presenta como una indicación de los niveles de detalle de super-resolución. A continuación medimos la resolución alcanzada calculando la anchura completa a la mitad máxima (FWHM) del pico del perfil de intensidad de un solo cordón(Figura 2B,C). Finalmente, corregimos la aberración cromática por registro de canales, utilizando de nuevo microesferas fluorescentes (Figura 3A,B). A continuación, comenzamos a analizar la muestra con un objetivo de 100x y adquirimos imágenes 3D-SIM. Usamos SIMCheck para evaluar la nieriedad de la iluminación de campo o el movimiento durante la adquisición (Figura 4A). Comprobamos las diferencias de intensidad entre los ángulos del patrón de iluminación (Figura 4B) y calculamos la relación entre el contraste de modulación y el ruido para medir el contraste de la franja local (Figura 4C). Por último, estimamos la resolución efectiva de la reconstrucción(Figura 4D).
A continuación confirmamos la calidad de las imágenes superconsuels mediante el uso de NanoJ-SQUIRREL. En la primera imagen reconstruida (Figura 5A), NanoJ-SQUIRREL detectó la presencia de artefactos (Figura 5C). Cambiamos los parámetros de reconstrucción para obtener una nueva imagen superconsuelbra (Figura 5B) y NanoJ-SQUIRREL confirmó la falta de artefactos (Figura 5D). Después de haber calibrado el sistema y evaluado la calidad de las imágenes reconstruidas, comenzamos a analizar los cultivos neuronales primarios manchados con un anticuerpo contra MAP2, un marcador neuronal, PSD95, un marcador postsináptico y la proteína diana SUMO1. Primero analizamos la muestra realizando microscopía confocal de cuatro canales con un objetivo 40x (Figura 6A). Al seleccionar un área que representa los procesos neuronales, cambiamos a un objetivo 100x. Adquirimos imágenes confocales y SIM de la misma zona para evaluar la calidad de la reconstrucción con NanoJ-SQUIRREL y realizar análisis de co-localización. En la Figura 6B,mostramos la imagen 3D-SIM superconsúctela de las neuronas manchadas para SENP1 y drebrin. La co-localización en imágenes superconsueltas se puede analizar con análisis de perfil(Figura 7A) y cuantificación de los coeficientes de Pearson y Mander (Figura 7B).

Figura 1: Comparación de adquisiciones de campo ancho, confocal y SIM. (A) Imagen de campo amplio de las neuronas primarias del hipocampo inmunotendida para SENP1 (verde), drebrin (rojo) y MAP2 (malva). DAPI se utilizó para manchar núcleos. Barra de escala 5 m. (B) Imagen confocal de la misma muestra del panel A. (C) Imagen SIM de la misma muestra de los paneles A y B. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Análisis de una imagen 3D-SIM de microesferas para calibración del microscopio. (A) Transformación rápida de Fourier de una adquisición de microesferas con su forma floral. (B) Selección de una sola microesfera para determinar la resolución espacial lateral. (C) Perfil de intensidad de la microesfera única en B con la medición de su FWHM. Los valores representan la resolución alcanzada por el instrumento. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Registro de tres canales. (A) Adquisición de microesferas TetraSpeck multicolor (longitudes de onda 488nm, 555nm y 647nm) antes del registro. (B) Adquisición de la misma muestra después de la calibración. Haga clic aquí para ver una versión más grande de esta figura.
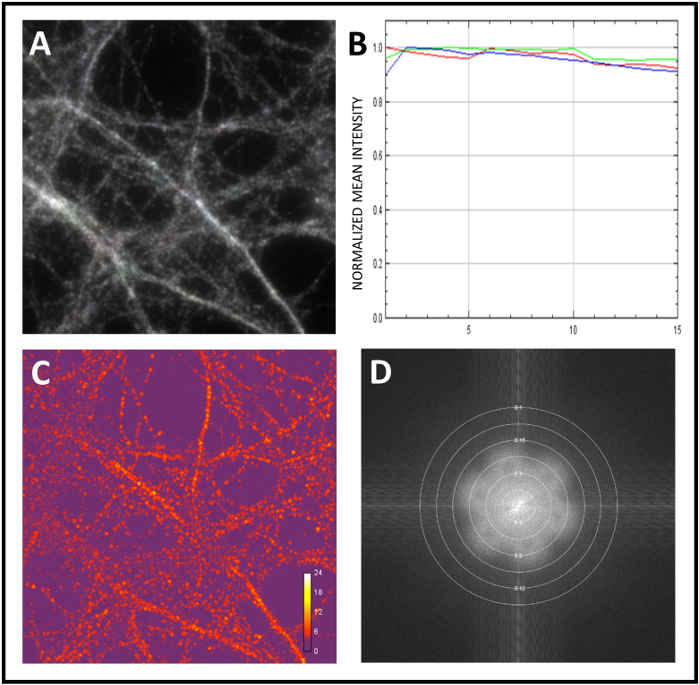
Figura 4: Evaluación de la calidad de las imágenes sin procesar y reconstruidas mediante SIMcheck. (A) Análisis de variación de movimiento e iluminación mediante SIMCheck. El gris de la señal al blanco representa la iluminación homogénea y la ausencia de movimiento durante la adquisición. (B) Perfil de intensidad de canal obtenido mediante el análisis de la imagen sin procesar. En este ejemplo hay variación de intensidad es mínima, para sugerir falta o blanqueo o fluctuaciones. (C) Contraste de modulación sin procesar para calcular la relación entre el contraste-ruido de modulación dentro de la imagen. El mapa de calor muestra variaciones de contraste de modulación. (D) Trazado Fourier reconstruido para analizar la amplitud del espectro de Fourier para determinar la resolución efectiva de la reconstrucción. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Evaluación de la calidad de imagen super resolución mediante NanoJ-SQUIRREL. (A) Referencia de imagen de superresorción con artefactos. (B) Imagen de super resolución de referencia de buena calidad. (C) Imagen que representa el mapa de errores NanoJ-SQUIRREL de A. Las áreas más claras representan artefactos a gran escala, mientras que las más oscuras representan la reconstrucción correcta. (D) Mapa de errores NanoJ-SQUIRREL de B. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Muestra de imagen SIM. (A) Microscopía confocal de neuronas primarias. Se eligió un objetivo 40X para obtener una visión general de la muestra mientras se mantenía una buena resolución. Las células fueron inmunotendéchadas para SUMO1 (verde), PSD95 (rojo) y MAP2 (malva). DAPI se utilizó para manchar núcleos. Barra de escala de 50 m. Las imágenes se mostraban como proyección Z. (B) Imágenes SIM para SUMO1 y PSD95 en el área resaltada en el cuadro verde en el panel A utilizando un objetivo 100X. Las puntas de flecha rojas indican la posición del recuadro que se muestra en A utilizada para calcular el perfil de intensidad. Barra de escala de 5 m. Haga clic aquí para ver una versión más grande de esta figura.
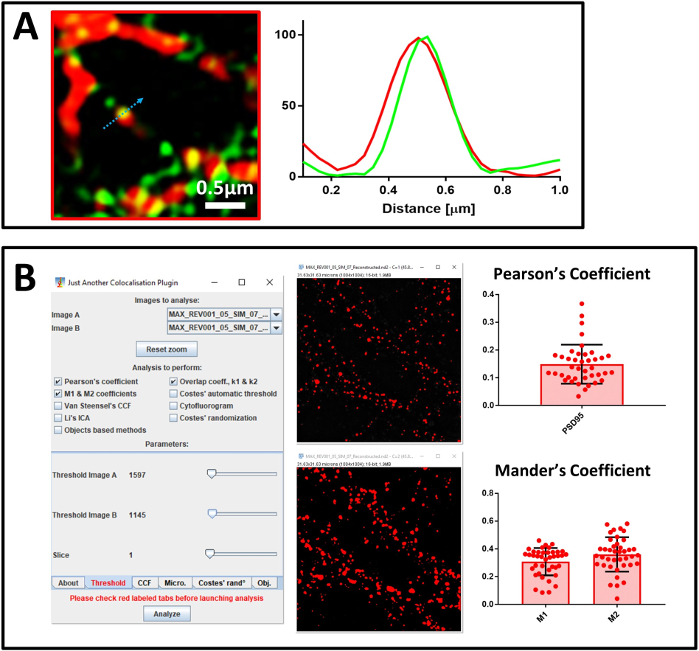
Figura 7: Análisis de co-localización. (A) Imagen supercontenida de las neuronas primarias manchadas con un anticuerpo contra SENP1 (en verde) y drebrin (en rojo), la barra de escala de 0,5 m y su perfil de intensidad. Los valores del gráfico se normalizaron para cada canal a 100 (unidad arbitraria) y corresponden a la intensidad de píxeles mostrada por la flecha azul. (B) Análisis utilizando JACoP para calcular el coeficiente de correlación de Pearson y el coeficiente de Mander entre SENP1 y drebrin. Se muestran las ventanas de la configuración del complemento y el umbral visual. El coeficiente de Mander se expresa por dos valores: fracción SENP1 que co-localiza con drebrin (M1) y la fracción de drebrin que co-localiza con SENP1 (M2). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Elucidar la estructura y composición de la sinapsis es crucial para entender los procesos fisiológicos y patológicos que regulan la memoria y la cognición. Mientras que en el estado normal, las sinapsis son los bloques de construcción de la memoria, también subyacen a trastornos neurológicos complejos como la enfermedad de Alzheimer32. El protocolo descrito aquí sirve para estudiar la co-localización de proteínas neuronales con una técnica de microscopía de superresor resolución llamada SIM. Usando un patrón particular de iluminación, SIM puede alcanzar una resolución de aproximadamente 0,1 m, que es adecuada para el estudio de las sinapsis, que normalmente miden entre 0,03 y 0,15 m. Para un mayor detalle, se pueden aplicar otras técnicas de superrescía como STED (Microscopía de Agotamiento de Emisiones Estimuladas), PALM (Microscopía de Localización Fotoactivada) o STORM (Microscopía de Reconstrucción óptica estocástica), que pueden alcanzar una resolución de 10-20 nm,33.
Aquí, describimos el análisis de la co-localización de proteínas diana con marcadores sinápticos en neuronas primarias. El protocolo se puede aplicar a cualquier cultivo primario de células neuronales, como las neuronas hipocampales, cerebelosas o corticales e incluso a cultivos de neuronas primarias que no pertenecen al sistema nervioso central, como las neuronas del sistema nervioso entérico. La clave para el análisis a nivel de superrescencia, sin embargo, son los reactivos utilizados durante la adquisición, tales como cubiertas de cámara y soluciones de montaje compatibles con el índice de difracción del objetivo. Utilizamos cubreobjetos con cámara por su facilidad de uso, pero el método clásico y más barato de cultivar neuronas primarias en tapa recubierta es sin embargo válido, particularmente con una cubierta de alta precisión #1.5H (0,17 mm). Además, se debe utilizar un medio de montaje que pueda alcanzar un índice de refracción lo más cercano posible al índice de refracción del vidrio (1.52) y un aceite de inmersión para el objetivo 100x con un índice de refracción de 1.515. La temperatura ambiente constante y las mesas estabilizadas también son obligatorias para garantizar la precisión de las adquisiciones.
Utilizamos anticuerpos secundarios dyLight y Alexa en los estudios SIM. Debido a sus estrechos picos de excitación y emisión y buen rendimiento cuántico, están indicados para técnicas de superrescía que requieren la mejor relación señal-ruido. Comparó Alexa, dyLight y otros fluoróforos para imágenes de super resolución34.
Durante la adquisición, establecemos rutinariamente la cámara a 1 MHz sobre 10 MHz. 1 MHz, gracias a una velocidad de adquisición más lenta, da a las imágenes más precisión y menos ruido que 10 MHz. El modo de lectura de 1 MHz también puede grabar con una profundidad de bits de 16 bits (en comparación con el máximo de 14 bits de 10 MHz), dando más información de color y un gradiente de color más preciso a las imágenes. Sin embargo, 10 MHz, con su velocidad, es útil para imágenes en vivo. Para evitar el blanqueo y preservar el fluoróforo, también establecemos la potencia del láser lo más baja posible. Para mejorar la intensidad de la señal, se pueden aumentar los valores de ganancia. Vale la pena señalar que una menor ganancia garantiza imágenes más limpias sin mejorar el ruido. En general, se obtienen los mejores resultados mientras se toma imágenes dentro de 7 m desde la parte inferior del cubreobjetos con cámara. Esto es especialmente importante cuando se utiliza un objetivo 100x con inmersión en aceite. Si se requiere una adquisición más profunda a través de las células / tejidos, una mejor opción puede ser el uso de un objetivo 60x con la inmersión en agua.
Uno de los principales desafíos en la realización de estudios SIM es la reconstrucción de imágenes35. La obtención de imágenes superconstionadas sin artefactos y aberración requiere no sólo el uso de condiciones experimentales ad hoc, sino también una cuidadosa calibración del sistema y la optimización de parámetros para obtener las imágenes finales. En el protocolo, describimos cómo evitar algunos de los errores más comunes mediante la evaluación de la calibración del sistema y el análisis de calidad de las imágenes crudas y reconstruidas. Específicamente, describimos el uso de los plugins ImageJ SIMCheck y NanoJ-SQUIRREL para asegurar la configuración correcta del instrumento para evitar artefactos comunes de imágenes superconsueltas. Las aplicaciones permiten una evaluación imparcial de la calidad de las imágenes finales que no se basa en la evaluación subjetiva de los resultados frente al conocimiento previo de las estructuras de estudio.
Sugerimos el uso de sinaptofisina y PSD95 o drebrin como marcadores pre y post-sinápticos, aunque otros marcadores son válidos también. Un enorme cuerpo de literatura describe proteínas como el faón como marcadores sinápticos36,,37. Vale la pena señalar que los marcadores pre y postsinápticos están sin embargo presentes en toda la célula, excluyendo el núcleo. Gran parte de su señal no es sináptica, pero representa proteínas en el transporte o degradación, antecedentes u otros artefactos. Por lo tanto, es importante elegir cuidadosamente el área del análisis. Utilizamos la señal de anticuerpos MAP2 para elegir terminales de axón y dendríticos.
En el análisis de la co-localización utilizamos dos enfoques. El primero es un enfoque visual, basado en el análisis de perfil que muestra eventos individuales de co-localización e identifica la contribución de cada canal. Sin embargo, una advertencia de este enfoque es el pobre poder estadístico. Por esta razón, decidimos también utilizar un segundo método basado en el análisis de un mayor número de eventos representativos de todo el campo de cada imagen. Este método se basa en el cálculo del coeficiente de correlación de Pearson y los coeficientes M1 y M2 de Mander. Utilizamos el coeficiente de correlación de Pearson para describir la superposición de señales en la imagen y M1 y M2 de Mander para describir la co-localización recíproca entre las señales de interés38. Para el cálculo, empleamos el plugin ImageJ JACoP, ya que tiene una característica que le permite establecer un umbral manual para descartar cualquier contribución de fondo al análisis, especialmente crítico para el análisis de Mander.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores quieren agradecer a Edoardo Micotti por la crítica constructiva del manuscrito. Este estudio fue apoyado por BrightFocus A2019296F, por Fondo di Beneficenza - Gruppo Intesa Sanpaolo (LC), por la Fondazione Regionale per la Ricerca Biomedica (Care4NeuroRare CP_20/2018) (CN) y por la Red de Formación Innovadora Marie Sk-odowska-Curie (JK).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan blue solution | Thermo Fisher Scientific | 15250061 | Chemical |
| 70 µm filter | Corning | 352350 | Equiment |
| Alexa | Thermo Fisher Scientific | - | Antibody |
| Antibody SENP1 | Santa Cruz | sc-271360 | Antibody |
| B27 Supplement | Life Technologies | 17504044 | Chemical |
| Bovine serum albumin | Merck | 5470 | Chemical |
| CaCl2 | Merck Life Science | 21115 | Chemical |
| Chambered coverslips | Ibidi | 80826 | Equiment |
| DyLight | Thermo Fisher Scientific | - | Antibody |
| FBS (Hyclone) | GIBCO | SH3007002 (CHA1111L) | Serum |
| FluoSpheres carboxylate-modified microspheres, 0.1 μm, yellow–green fluorescent | Thermo Fisher Scientific | F8803 | Equiment |
| Glucose | Merck Life Science | G8769 | Chemical |
| Glutamax | GIBCO | 35050061 | Chemical |
| HEPES | Merck Life Science | H3537 | Chemical |
| L-Cystein | Merck Life Science | C6852-25g | Chemical |
| MAP2 | Merck | AB15452 | Antibody |
| MEM | Life Technologies | 21575022 | Medium |
| MgCl | Merck Life Science | M8266 | Chemical |
| NaOH | VWR International | 1,091,371,000 | Chemical |
| Neurobasal A | Life Technologies | 10888022 | Medium |
| N-SIM Super Resolution Microscope | Nikon | - | Instrument |
| Papain | Merck Life Science | P-3125 | Chemical |
| paraformaldehyde | Thermo Fisher Scientific | 28908 | Chemical |
| Pen/Strep 10x | Life Technologies | 15140122 | Chemical |
| phosphate-buffered saline | Gibco | 10010023 | Chemical |
| Poly-L lysine | Sigma | P2636 | Chemical |
| ProLong Diamond Glass Antifade Mountant | Thermo Fisher Scientific | P36970 | Chemical |
| PSD95 | NeuroMab | K28/43 | Antibody |
| Round coverglass | Thermo | 12052712 | Equiment |
| SUMO1 | Abcam | ab32058 | Antibody |
| Synaptophysin | Merck | S5768 | Antibody |
| Triton X-100 | Merck | T8787 | Chemical |
| Trypsin inhibitor | Merck Life Science | T9003-500MG | Chemical |
Referencias
- Foster, M., Sherrington, C. S. . A textbook of physiology, part three: The central nervous system (7th ed.). , (1897).
- Choquet, D., Triller, A. The Dynamic Synapse. Neuron. 80 (3), 691-703 (2013).
- McAllister, A. K. Dynamic Aspects of CNS Synapse Formation. Annual Review of Neuroscience. 30 (1), 425-450 (2007).
- Yuzaki, M. Two Classes of Secreted Synaptic Organizers in the Central Nervous System. Annual Review of Physiology. 80 (1), 243-262 (2018).
- Baddeley, D., Bewersdorf, J. Biological Insight from Super-Resolution Microscopy: What We Can Learn from Localization-Based Images. Annual Review of Biochemistry. 87 (1), 965-989 (2018).
- Sigal, Y. M., Zhou, R., Zhuang, X. Visualizing and discovering cellular structures with super-resolution microscopy. Science. 361 (6405), 880-887 (2018).
- Vangindertael, J., et al. An introduction to optical super-resolution microscopy for the adventurous biologist. Methods and Applications in Fluorescence. 6 (2), 022003 (2018).
- Badawi, Y., Nishimune, H. Super-resolution microscopy for analyzing neuromuscular junctions and synapses. Neuroscience Letters. 715, 134644 (2020).
- Scalisi, S., Barberis, A., Petrini, E. M., Zanacchi, F. C., Diaspro, A. Unveiling the Inhibitory Synapse Organization Using Superresolution Microscopy. Biophysical Journal. 116 (3), 133 (2019).
- Yang, X., Specht, C. G. Subsynaptic Domains in Super-Resolution Microscopy: The Treachery of Images. Frontiers in Molecular Neuroscience. 12, (2019).
- Monro, T. Beyond the diffraction limit. Nature Photonics. 3 (7), 361 (2009).
- Won, R. Eyes on super-resolution. Nature Photonics. 3 (7), 368-369 (2009).
- Wegel, E., et al. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific Reports. 6 (1), 27290 (2016).
- Galbraith, C. G., Galbraith, J. A. Super-resolution microscopy at a glance. Journal of Cell Science. 124 (10), 1607-1611 (2011).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Gustafsson, M. G. L., et al. Three-Dimensional Resolution Doubling in Wide-Field Fluorescence Microscopy by Structured Illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Brose, N., O'Connor, V., Skehel, P. Synaptopathy: dysfunction of synaptic function. Biochemical Society Transactions. 38 (2), 443-444 (2010).
- Tyebji, S., Hannan, A. J. Synaptopathic mechanisms of neurodegeneration and dementia: Insights from Huntington's disease. Progress in Neurobiology. 153, 18-45 (2017).
- Won, H., Mah, W., Kim, E. Autism spectrum disorder causes, mechanisms, and treatments: focus on neuronal synapses. Frontiers in Molecular Neuroscience. 6, (2013).
- Pfeiffer, B. E., Huber, K. M. The State of Synapses in Fragile X Syndrome. The Neuroscientist. 15 (5), 549-567 (2009).
- Pavlowsky, A., Chelly, J., Billuart, P. Emerging major synaptic signaling pathways involved in intellectual disability. Molecular Psychiatry. 17 (7), 682-693 (2012).
- Senatore, A., Restelli, E., Chiesa, R. Synaptic dysfunction in prion diseases: a trafficking problem. International Journal of Cell Biology. 2013, 543803 (2013).
- Colnaghi, L., et al. Super Resolution Microscopy of SUMO Proteins in Neurons. Frontiers in Cellular Neuroscience. 13, (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Müller, M., Mönkemöller, V., Hennig, S., Hübner, W., Huser, T. Open-source image reconstruction of super-resolution structured illumination microscopy data in ImageJ. Nature Communications. 7 (1), 10980 (2016).
- Ball, G., et al. SIMcheck: a Toolbox for Successful Super-resolution Structured Illumination Microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Schaefer, L. H., Schuster, D., Schaffer, J. Structured illumination microscopy: artefact analysis and reduction utilizing a parameter optimization approach. Journal of Microscopy. 216 (2), 165-174 (2004).
- Culley, S., et al. NanoJ-SQUIRREL: quantitative mapping and minimisation of super-resolution optical imaging artefacts. Nature Methods. 15 (4), 263-266 (2018).
- Manders, E. M. M., Verbeek, F. J., Aten, J. A. Measurement of co-localization of objects in dual-colour confocal images. Journal of Microscopy. 169 (3), 375-382 (1993).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander’s overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
- Bolte, S., Cordelières, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224, 213-232 (2006).
- Bae, J. R., Kim, S. H. Synapses in neurodegenerative diseases. BMB Reports. 50 (5), 237-246 (2017).
- Godin, A. G., Lounis, B., Cognet, L. Super-resolution Microscopy Approaches for Live Cell Imaging. Biophysical Journal. 107 (8), 1777-1784 (2014).
- Dempsey, G. T., Vaughan, J. C., Chen, K. H., Bates, M., Zhuang, X. Evaluation of fluorophores for optimal performance in localization-based super-resolution imaging. Nature Methods. 8 (12), 1027-1036 (2011).
- Karras, C., et al. Successful optimization of reconstruction parameters in structured illumination microscopy - A practical guide. Optics Communications. 436, 69-75 (2019).
- Bereczki, E., et al. Synaptic markers of cognitive decline in neurodegenerative diseases: a proteomic approach. Brain: A Journal of Neurology. 141 (2), 582-595 (2018).
- Gilestro, G. F., Tononi, G., Cirelli, C. Widespread Changes in Synaptic Markers as a Function of Sleep and Wakefulness in Drosophila. Science. 324 (5923), 109-112 (2009).
- Adler, J., Parmryd, I. Quantifying colocalization by correlation: the Pearson correlation coefficient is superior to the Mander's overlap coefficient. Cytometry. Part A: The Journal of the International Society for Analytical Cytology. 77 (8), 733-742 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados