Method Article
Visualização cinética de interespécies interespécies unicelulares interespécies
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo de imagem celular live-bacteria permite a visualização de interações entre múltiplas espécies bacterianas no nível de célula única ao longo do tempo. A imagem de lapso de tempo permite a observação de cada espécie bacteriana em monocultura ou cocultura para interrogar interações entre espécies em comunidades bacterianas multiespécies, incluindo motilidade e viabilidade celular individual.
Resumo
As comunidades polimicrobianas são de natureza onipresente, mas estudar suas interações no nível de célula única é difícil. Assim, um método baseado em microscopia foi desenvolvido para observar interações entre duas espécies bacterianas. O uso deste método para interrogar interações entre um patógeno Gram-negativo, Pseudomonas aeruginosa e um patógeno gram-positivo não-motile, Staphylococcus aureus é demonstrado aqui. Este protocolo consiste em co-vacinar cada espécie entre um deslizamento de cobertura e uma almofada de agarose, que mantém as células em um único plano e permite a visualização de comportamentos bacterianos tanto no espaço quanto no tempo.
Além disso, a microscopia de lapso de tempo demonstrada aqui é ideal para visualizar as interações precoces que ocorrem entre duas ou mais espécies bacterianas, incluindo alterações na motilidade das espécies bacterianas na monocultura e na cocultura com outras espécies. Devido à natureza do espaço amostral limitado na configuração da microscopia, este protocolo é menos aplicável para estudar interações posteriores entre espécies bacterianas, uma vez que as populações de células são muito altas. No entanto, existem várias aplicações diferentes do protocolo que incluem o uso de coloração para imagens de células bacterianas vivas e mortas, quantificação da expressão genética ou proteica através de repórteres fluorescentes, e rastreamento do movimento celular bacteriano em experimentos de espécies únicas e multiespécies.
Introdução
Comunidades polimicrobianas são comuns na natureza, abrangendo uma variedade de nichos ambientais1,,2,,3 e humanos4,,5. Algumas das infecções polimicrobianas mais notórias na doença humana incluem biofilmes dentários6, feridas crônicas7,,8, e infecções respiratórias em indivíduos com doença pulmonar obstrutiva crônica9,pneumonia associada ao ventilador10e fibrose cística da doença genética (CF)11,12. Essas infecções são frequentemente compostas de diversas espécies microbianas; no entanto, surgiu um foco recente nas interações entre a bactéria Gram-positive Staphylococcus aureus e a bactéria Gram-negativa Pseudomonas aeruginosa. Estudos que incluem pacientes coinfectados com esses organismos e análises in vitro revelam interações competitivas e cooperativas que podem ter influências profundas e inesperadas na progressão da doença, sobrevivência microbiana, virulência, metabolismo e suscetibilidade a antibióticos (revisados em detalhes por Hotterbeekx et al. 201713 e Limoli e Hoffman 20193).
O crescente interesse pelas interações entre espécies durante a infecção tem gerado uma variedade de métodos para estudar comportamentos da comunidade polimicrobiana. Tipicamente, esses estudos têm utilizado ensaios à base de chapas ou caldos para investigar as diferenças fenotípicas entre monocultura e cocultura. Por exemplo, coculto P. aeruginosa e S. aureus em superfícies sólidas permitiu a visualização da inibição do crescimento ou alterações na produção de fenótipo, pigmento ou polissacarídeo da colônia14,,15,,16. Biofilmes de espécies mistas, em superfícies bióticas ou abióticas, bem como coculpando espécies bacterianas na cultura líquida também permitiram a medição de mudanças no crescimento, metabolismo, tolerância a antibióticos, competição e viabilidade, além da expressão genética e proteica17,18. Embora esses experimentos de cultura em massa tenham revelado uma visão de como P. aeruginosa e S. aureus podem influenciar uns aos outros na escala da comunidade, eles são incapazes de responder a perguntas importantes sobre as interações que ocorrem no nível de célula única. O método aqui apresentado se soma às abordagens para estudar interações entre espécies, focando nas mudanças de movimento, viabilidade celular e expressão genética de células únicas dentro de uma comunidade coculturada ao longo do tempo.
As interações unicelulares podem diferir amplamente das interações que ocorrem em uma comunidade em massa de células. Através da análise unicelular, a heterogeneidade dentro de uma comunidade pode ser quantificada para estudar como a colocação espacial das células influencia a dinâmica da comunidade ou como os níveis de expressão genética e proteica mudam dentro de membros individuais de um grupo. O rastreamento de células também pode fornecer informações sobre como células únicas se movem e se comportam ao longo do tempo, através de várias gerações. Ao rastrear o movimento celular e as alterações na expressão genética simultaneamente, correlações podem ser feitas entre flutuações genéticas e fenótipos correspondentes. Foram descritos protocolos anteriores para o estudo de espécies bacterianas individuais no nível de células únicas. Esses estudos aproveitam as células de imagem ao vivo ao longo do tempo em um único plano, e têm sido úteis para observar fenótipos como divisão celular e suscetibilidade a antibióticos19,,20. Foram utilizadas microscopia suplementar de imagem viva para monitorar o crescimento, a motilidade, a colonização superficial e a formação de biofilmes de espécies bacterianas únicas21,,22,,23. No entanto, embora esses estudos tenham sido perspicazes para entender a fisiologia das bactérias na monocultura, os experimentos para observar o comportamento unicelular de múltiplas espécies bacterianas ao longo do tempo na cocultura são limitados.
Aqui, protocolos anteriores usados para imagens uni espécies foram adaptados para estudar interações entre P. aeruginosa e S. aureus. Esses organismos podem ser diferenciados sob contraste de fase com base em suas morfologias celulares(P. aeruginosa são bacilos e S. aureus são cocci). O desenvolvimento deste método recentemente possibilitou a visualização de comportamentos de motilidade não atribuídos anteriormente não atribuídos de P. aeruginosa na presença de S. aureus24. P. aeruginosa foi encontrado capaz de sentir S. aureus à distância e responder com movimentos de célula única aumentada e direcional em direção a aglomerados de células S. aureus. O movimento P. aeruginosa em direção a S. aureus foi encontrado para exigir o tipo IV pili (TFP), projeções semelhantes ao cabelo cuja extensão coordenada e retração geram um movimento chamado motilidade contração25.
Esses estudos demonstram a utilidade deste método para interrogar interações anteriores entre espécies. No entanto, a imagem em altas densidades celulares em pontos de tempo de interação posteriores é difícil, dado que camadas únicas de células não podem mais ser identificadas, o que coloca principalmente problemas durante a análise pós-imagem. Dada essa limitação, o método é mais adequado para interações anteriores que poderiam ser seguidas com ensaios macroscópicos tradicionais em densidades celulares mais altas representativas de interações posteriores. Limitações adicionais deste método incluem a natureza de baixo rendimento, uma vez que apenas uma amostra pode ser imageda de cada vez e o custo do microscópio, câmera e câmara ambiental. Além disso, a microscopia de fluorescência representa riscos para as células bacterianas como fototoxicidade e fotobleaching, limitando assim a frequência que as imagens de fluorescência podem ser adquiridas. Por fim, as almofadas de agarose utilizadas neste método são altamente suscetíveis a mudanças no ambiente, tornando-se crítico controlar condições como temperatura e umidade, dado que as almofadas podem começar a encolher ou expandir se as condições não estiverem corretas. Finalmente, embora este método não imite o ambiente hospedeiro, ele fornece pistas de como diferentes espécies bacterianas respondem em superfícies, que podem ser acompanhadas em ensaios projetados para imitar condições ambientais/hospedeiras.
Este método difere de estudos anteriores que rastreiam o movimento unicelular, na parte em que as células são inoculadas entre um deslizamento de tampa e uma almofada de agarose, restringindo as células à superfície. Isso permite o rastreamento de células ao longo do tempo em um único plano; no entanto, limita ciclos de engajamento transitório da superfície observados quando as células estão submersas no líquido26. Um benefício adicional das bactérias de imagem sob uma almofada de agarose é que ela imita ensaios de inoculação sub-superfície macroscópicas à base de placas usadas para examinar a motilidade de contração de P. aeruginosa 25. Neste ensaio, as células bacterianas são inoculadas entre o fundo de uma placa de Petri e o ágar, mantendo as células em um único plano enquanto se movem através do fundo do prato para fora do ponto de inoculação, assim como este protocolo de microscopia.
O protocolo de microscopia de lapso de tempo para visualização de interespécies interespécies aqui apresentadas consiste em 1) preparar a amostra bacteriana e a almofada de agarose, 2) selecionar configurações de microscópio para aquisição de imagens e 3) análise pós-imagem. A visualização detalhada do movimento e rastreamento do celular pode ser realizada por aquisição de imagens em intervalos curtos de tempo por contraste de fase. A microscopia de fluorescência também pode ser utilizada para determinar a viabilidade celular ou a expressão genética ao longo do tempo. Aqui, mostramos um exemplo de adaptação para microscopia de fluorescência através da adição de corantes de viabilidade às almofadas agarose.
Protocolo
NOTA: Uma descrição completa e números de catálogo para todos os suprimentos neste protocolo podem ser encontrados na Tabela de Materiais.
1. Preparação de mídia mínima M8T
- Prepare 1 L de base de sais mínimos M8 (5x) dissolvendo 64 g de Na2HPO4·7H2O, 15 g de KH2PO4e 2,5 g NaCl em 800 mL de água ultrauso (UPW, resistência 18 MΩ/cm) em uma garrafa de 2 L. pH para 7,6. Volume completo para 1 L com UPW. Autoclave por 45 min.
- Prepare 500 mL de uma solução de glicose (20% c/v) dissolvendo 100 g de glicose em 400 mL de UPW em uma garrafa de 1 L. Solução completa para 500 mL com UPW. Autoclave por 45 min.
- Prepare 500 mL de uma solução de triptona (20% w/v) dissolvendo 100 g de triptona em 400 mL de UPW. Complete até 500 mL com UPW. Autoclave por 45 min.
- Prepare 1 L de M8 + 10% de triptona (M8T) de mídia mínima adicionando 200 mL de sais mínimos 5x M8 (1x final), 10 mL de 20% de glicose (0.2% final), 1 mL de 1 M MgSO4 (final de 1 mM) e 50 mL de 20% de triptona (1% final) a 600 mL de UPW. Complete a 1 L com UPW. Filtre através de um filtro estéril de 0,2 μm em uma garrafa de armazenamento de mídia estéril de 500 mL.
Dia 1º:
2. Preparação de culturas bacterianas durante a noite
- Inocular 5 mL de mídia mínima M8T com uma única colônia de P. aeruginosa ou S. aureus (incluindo antibióticos quando apropriado) e incubar durante a noite a 37 °C com aeração por não mais do que 16 h.
NOTA: Os patógenos bacterianos P. aeruginosa e S. aureus foram utilizados para este método, pois são comumente coisolados de infecções crônicas, e estudar suas interações é importante para entender como eles contribuem para os desfechos dos pacientes durante infecções polimicrobianas. Outras espécies bacterianas poderiam ser utilizadas dependendo do foco do estudo.
Dia 2:
3. Subcultura de cepas bacterianas
- Subcultura P. aeruginosa 1:500 e S. aureus 1:1000 em 5 mL de M8T fresco (inclua antibióticos quando apropriado). Incubar com aeração a 37 °C até que as culturas atinjam a fase de registro médio (OD600 = ~0,3 - 0,5).
4. Preparação de materiais para moldes de almofadas
- Prepare espátulas metálicas aquecendo a extremidade de uma espátula de laboratório plana e arredondada com um queimador de Bunsen até que metade da extremidade plana possa ser dobrada para um ângulo de 90°. Aqueça a extremidade de outra espátula de laboratório plana e arredondada e dobre ligeiramente os últimos 10 mm para um ângulo de 45 °C.
- Corte os quatro cantos dos moldes de silicone para que os moldes se encaixem dentro de um prato de 35 mm.
- Esterilize as espátulas e um par de pinças adicionando 70% de etanol e passando-as através da chama do queimador Bunsen.
- Limpe o prato e os moldes de silicone com 70% de etanol e seque-os com lenços de linlho.
5. Preparação de almofadas de agarose
NOTA: As almofadas são preparadas com a mídia mínima M8T como fonte de nutrientes para as bactérias utilizadas neste protocolo. No entanto, os nutrientes utilizados nas almofadas podem ser modificados para diferentes organismos.
- Derreta 2% de baixa derretimento em 10 mL de M8T em um frasco limpo de 50 mL Erlenmeyer. Micro-ondas em intervalos curtos (2-5 s) até que a agarose esteja em solução, a fim de evitar que o conteúdo do frasco ebuleque. Uma vez derretido, deixe esfriar em um banho de água de 50 °C por pelo menos 15 min.
- Prepare os moldes alinhando o recorte de silicone com a abertura na antena de 35 mm e bata levemente com espátula para fixar o silicone no prato, e para remover todas as bolhas de ar entre o molde e o prato.
- Uma vez fria, pipeta 915 μL de agarose derretida no molde. Deixe a tampa entreaberta e deixe a almofada secar à temperatura ambiente por 30 minutos.
- Cubra o prato com a tampa e deixe em temperatura ambiente por mais 2 h.
- Prepare os limpadores de umidade enrolando firmemente um lenço sem fiapos. Coloque o lenço enrolado dentro de uma placa de Petri estéril e adicione uniformemente 500 μL de água estéril através do lenço. Aqueça a 37 °C por 1h.
- Aqueça a almofada e um prato estéril de 35 mm até 37 °C por 1h.
6. Preparação de células bacterianas e almofadas inoculantes
- Meça o OD600 de cada subcultura e dilua P. aeruginosa a um OD600 = 0,03 e S. aureus a um OD600 = 0,10 em M8T prewarmed a 37 °C. Misture P. aeruginosa e S. aureus em uma proporção de 1:1 e vórtice.
NOTA: Se as cepas necessitarem de antibióticos, os antibióticos podem ser adicionados à subcultura durante a noite, mas não devem ser adicionados ao misturar espécies para imagens se afetar as outras espécies na cocultura. A estabilidade plasmida e os efeitos dos antibióticos em todas as espécies da cocultura precisarão ser determinados para cada organismo/plasmídeo que está sendo usado. - Pipeta 1 μL de cocultura uniformemente através do fundo de um prato de deslizamento de vidro pré-aquecido e estéril de 35 mm.
- Remova o recorte de silicone do molde usando pinças estéreis.
- Retire a almofada do prato usando espátulas estéreis.
- Deslize a espátula ligeiramente dobrada sob a borda da almofada, enquanto segura o molde de cabeça para baixo, para deixá-la em uma tampa de placa de Petri estéril.
NOTA: Tome cuidado para não forçar o bloco para fora ou ele vai rasgar. Mantenha o controle de que lado da almofada está o fundo.
- Deslize a espátula ligeiramente dobrada sob a borda da almofada, enquanto segura o molde de cabeça para baixo, para deixá-la em uma tampa de placa de Petri estéril.
- Transfira a almofada para o prato com as células bacterianas, de baixo para baixo, deslizando a espátula angular de 90° sob a almofada e colocando-a em cima do deslizamento de cobertura inoculado. Use a espátula angular de 90° para fazer a almofada lavar contra a mancha de cobertura e pressionar suavemente quaisquer bolhas de ar.
- Remova o excesso de umidade dos lenços umedecidos e coloque ao redor da borda do prato, certificando-se de que ele não toque na almofada. A amostra está pronta para a imagem.
7. Configuração do microscópio para imagens ao vivo
- Câmara ambiental quente a 37 °C pelo menos 2 h antes da configuração experimental.
- Ligue todos os componentes, incluindo microscópio, computador e fontes de luz para imagens de brightfield e fluorescência.
- Abra o software de imagem e certifique-se de que as fontes de luz estejam conectadas e funcionando.
- Execute a iluminação Köhler27 para alinhar todos os planos de imagem.
- Primeiro, trazer coberturas marcadas em foco com um objetivo de 20x usando um prato "manequim". Certifique-se de que a torre condensadora está definida na posição "aberta".
NOTA: A iluminação de Köhler deve ser realizada para cada objetivo/amostra único para alinhar adequadamente os planos de imagem. No entanto, o foco e o alinhamento com o prato "manequim" na ampliação mais baixa acelera a configuração em amostras ao vivo em maior ampliação para limitar o tempo entre o tempo de configuração e o início do experimento. - Concentre o condensador.
- Feche o diafragma de campo.
NOTA: Uma abertura em forma de octógono deve aparecer. Se o condensador estiver completamente fora de foco, todo o campo de visão (FOV) parecerá escuro. - Gire os botões de foco do condensador até que as bordas do octógono fiquem nítidas.
NOTA: A intensidade da luz aumentará à medida que os planos de imagem se aproximarem do alinhamento correto.
- Feche o diafragma de campo.
- Alinhe o condensador.
- Centralizar o condensador de campo ajustando os botões de alinhamento.
NOTA: O octógono deve ser centrado no meio do FOV. Os botões de alinhamento diferem dependendo do microscópio. Por exemplo, alguns condensadores de microscópio têm botões, enquanto outros têm parafusos que requerem uma chave de fenda.
- Centralizar o condensador de campo ajustando os botões de alinhamento.
- Concentre o filamento da lâmpada e ajuste a abertura do condensador.
- Coloque um telescópio de fase ou lente Bertrand no caminho da luz para observar o plano focal traseiro do objetivo.
NOTA: Deve haver dois círculos concêntricos. - Gire o botão de foco da lente Bertrand até que os anéis pareçam nítidos.
- Remova a lente Bertrand do caminho da luz.
- Coloque um telescópio de fase ou lente Bertrand no caminho da luz para observar o plano focal traseiro do objetivo.
- Abra o diafragma de campo até que o octógono esteja fora do FOV.
- Mude para o objetivo de 100x e deslize o anel de fase correspondente no lugar antes de adicionar uma gota de óleo de imersão, e coloque o prato de amostra preparado por cima.
- Realize a iluminação Köhler no objetivo de 100x com o prato de amostra bacteriana.
- Primeiro, trazer coberturas marcadas em foco com um objetivo de 20x usando um prato "manequim". Certifique-se de que a torre condensadora está definida na posição "aberta".
- Concentre-se apenas nas bactérias usando o ajuste fino. Uma vez que as bactérias no FOV estejam focadas através da ocular, mude o caminho da luz para a câmera pressionando o botão da câmera no microscópio.
- Clique na opção Fase no software de imagem.
- Ajuste a porcentagem de luz LED DIA emitida selecionando a fonte de luz no software e digitando manualmente a porcentagem de luz desejada a ser usada ou deslizando a barra na escala de porcentagem de luz.
- Ajuste o tempo de exposição da luz LED DIA clicando na fonte de luz e digitando manualmente o tempo de exposição desejado ou selecionando um tempo de exposição do menu suspenso fornecido.
NOTA: O tempo de exposição varia dependendo da câmera que está sendo usada. - Se estiver usando fluorescência, ajuste as configurações da câmera em cada canal correspondente (ou seja, TxRed, GFP), clicando no canal fluorescente de interesse.
- Defina a porcentagem de luz de fluorescência emitida e ajuste o tempo de exposição (conforme realizado nas etapas 7.7 e 7.8 para luz LED DIA).
- Alternativamente, altere a profundidade do bit para ajustar o intervalo dinâmico selecionando uma das outras opções de profundidade de bit no menu suspenso dos controles de visualização.
- Escolha as posições de interesse XY clicando na opção XY no menu de controles de aquisição.
- Mova a posição do palco com o joy stick, ou clicando e arrastando o FOV na tela, e clique na caixa vazia para salvar as coordenadas X e Y de uma posição específica.
NOTA: A seleção de não mais do que três posições XY diferentes, o mais próxima possível, é recomendada para aquisição de lapso de tempo.
- Mova a posição do palco com o joy stick, ou clicando e arrastando o FOV na tela, e clique na caixa vazia para salvar as coordenadas X e Y de uma posição específica.
- Ligue o sistema de foco perfeito (PFS) clicando na caixa PFS na guia XY do menu de controle de aquisição ou pressionando o botão PFS no painel de controle joy stick.
- Gire os botões de ajuste fino para focar nas células bacterianas.
- Clique no botão PFS para cada posição XY, uma vez que as células estejam no plano de foco desejado.
NOTA: O PFS compensa a deriva no eixo Z durante experimentos de lapso de tempo. Isso é necessário para manter o foco das células bacterianas ao longo do tempo. Diferentes fabricantes têm diferentes sistemas de compensação. - Escolha as condições de aquisição de imagem, incluindo intervalo de aquisição e frequência para cada canal (por exemplo, contraste de fase e cada fluoróforo) selecionando entre as opções no menu de controles de aquisição.
NOTA: Para os experimentos aqui apresentados, as imagens de contraste de fase são adquiridas em intervalos a cada 5-10 s, enquanto as imagens de fluorescência nos canais GFP e TxRed são adquiridas a cada 20 minutos. - Comece a fotografar assim que as configurações do microscópio e da aquisição forem configuradas.
8. Opcional: Modificações para imagens ao vivo/mortas
- Derreta 2% de agarose em 10 mL de M8T e deixe esfriar em banho de água de 50 °C por pelo menos 15 min.
- Adicione 1 mM de iodeto de propidium à agarose derretida.
- Uma vez resfriado, pipeta 915 μL de agarose no molde preparado.
- Deixe a tampa entreaberta e deixe o bloco secar à temperatura ambiente por 30 minutos. Proteja-se contra a luz.
- Cubra o prato com a tampa e deixe em temperatura ambiente por mais 2 h.
- Prepare os lenços de umidade.
- Enrole um papel sem fiapos para limpar firmemente.
- Coloque o lenço em uma placa de Petri estéril e adicione 500 μL de água estéril ao lenço.
- Incubar a 37 °C por 1 h.
- Incubar a almofada e um prato estéril de 35 mm a 37 °C por 1h.
- Prossiga para vacinar o prato conforme indicado na seção 6: Preparação de células bacterianas e almofadas inoculantes.
9. Análise de dados
- Identificação de células
- Abra o arquivo de imagem no software de imagem e corte o arquivo para incluir apenas os quadros a serem usados para rastreamento, ampliado para as células de interesse e apenas no canal de contraste de fase.
NOTA: O arquivo cortado pode ser salvo como um novo arquivo no qual os dados de rastreamento podem ser armazenados sem interferir no arquivo original. - Selecione a opção de escolher regiões de interesse (ROI) no menu de controles de análise e defina ROIs traçando células bacterianas individuais ou aglomerados de células no primeiro quadro a ser usado para análise.
NOTA: Os ROIs podem ser definidos manualmente ou como binários.- Definir manualmente o ROI
- Rastreie o perímetro de cada célula bacteriana individual ou aglomerados de células para definir manualmente os ROIs.
NOTA: No software de análise, P. aeruginosa, ou outras células em forma de vara, podem ser traçadas manualmente selecionando o ROI de Elipse e desenhando uma elipse ajustada ao tamanho da célula bacteriana. Alternativamente, a opção ROI polygon pode ser selecionada para rastrear ROIs não-tradicionais, como clusters de células.
- Rastreie o perímetro de cada célula bacteriana individual ou aglomerados de células para definir manualmente os ROIs.
- Identificar ROIs binários
- Clique na opção Binário para ROI para definir ROIs binários.
NOTA: Os objetos são definidos em uma camada binária baseada na separação das células bacterianas pixeladas mais escuras do fundo pixelado mais claro no canal de contraste de fase. - Para limiar das células, selecione a opção Limiar no menu de controles de análise. Selecione o canal de interesse e deslize as barras no histograma de fluorescência para ajustar os valores do intervalo de limiar.
NOTA: A identificação binária de objetos para ROIs também pode ser definida em imagens fluorescentes, cortando as células bacterianas. O limiar estabelece quais intensidades de fluorescência são consideradas objetos e quais intensidades de fluorescência constituem o fundo.
- Clique na opção Binário para ROI para definir ROIs binários.
- Definir manualmente o ROI
- Abra o arquivo de imagem no software de imagem e corte o arquivo para incluir apenas os quadros a serem usados para rastreamento, ampliado para as células de interesse e apenas no canal de contraste de fase.
- Rastreamento celular
- Para acompanhar manualmente os ROIs, selecione o próximo quadro na sequência de imagens e ajuste a posição dos ROIs clicando e arrastando cada ROI para se alinhar com a nova posição da célula bacteriana original.
NOTA: Se as células bacterianas não mudaram de posição, os ROIs não precisam ser movidos. - Repita em todos os quadros sequenciais para quais células devem ser rastreadas.
- À medida que as células se dividem, defina as células filhas como novos ROIs, como descrito na etapa 9.1, e comece a rastrear as células recém-divididas.
NOTA: Se as células forem identificadas como binárias, use a função Track Binaries para rastrear automaticamente os ROIs em quadros selecionados. - Uma vez que as células tenham sido rastreadas através de todos os quadros selecionados, exporte os dados a serem analisados.
- Abra a planilha de dados e identifique as medidas necessárias para análise (ou seja, velocidade do objeto, aceleração ou comprimento do caminho).
NOTA: No caso de medir a direcionamento, são necessárias as medidas de comprimento do caminho e comprimento da linha. O Comprimento da Linha é uma medida da distância euclidiana, ou a distância reta da origem da pista até a borda da colônia S. aureus. O Comprimento do Caminho é uma medição da distância acumulada, ou a soma dos segmentos de faixa de todos os quadros. A direção pode ser calculada como uma razão da distância euclidiana, D(E), (Comprimento da Linha), sobre a distância acumulada, D(A), (Comprimento do Caminho).
- Para acompanhar manualmente os ROIs, selecione o próximo quadro na sequência de imagens e ajuste a posição dos ROIs clicando e arrastando cada ROI para se alinhar com a nova posição da célula bacteriana original.
- Quantificação da fluorescência
- Defina ROIs para células bacterianas fluorescentes conforme descrito na etapa 9.1.
- Repita o rastreamento ou o movimento de ROIs para células bacterianas ou aglomerados nos quadros fluorescentes restantes.
- Exporte a tabela gerada para um arquivo de planilha para análise da intensidade fluorescente.
- Na planilha de dados, procure a coluna com "Intensidade Média" que representa a intensidade média de fluorescência para o ROI de células bacterianas rastreadas.
- Gráfico dos valores de intensidade média para olhar para as mudanças na fluorescência ao longo do tempo.
NOTA: As mudanças na fluorescência ao longo do tempo representam a flutuação da expressão genética para o gene de interesse fluorescentemente rotulado.
Resultados
O uso bem-sucedido do método descrito resultará em uma série de quadros que geram um vídeo no qual as interações entre espécies podem ser observadas ao longo do tempo. O esquema na Figura 1 fornece um visual para destacar os principais passos envolvidos na preparação de materiais para imagem. O uso deste método permitiu a demonstração de células P. aeruginosa exibindo diferentes comportamentos na monocultura versus na cocultura com S. aureus. Em comparação com as células P. aeruginosa na monocultura que permanecem agrupadas em jangadas, quando em cocultura com S. aureus, P. aeruginosa aumentou a motilidade unicelular em relação às colônias de S. aureus (Figura 2A-2B). Cepas bacterianas fluorescentes também permitem visualizar populações mistas de espécies bacterianas. O uso da P. aeruginosa com a etiqueta GFP permitiu a observação de que após p. aeruginosa únicas células aumentarem na motilidade, elas cercarão e eventualmente invadirão colônias de S. aureus (Figura 2C). A utilização de células fluorescentes também permite a visualização da invasão celular P. aeruginosa nas colônias S. aureus pela primeira vez(Figura 2C, inferior).
Embora o protocolo produza imagens de alta qualidade para observar interações entre espécies, existem vários problemas comuns que podem levar a sequências de quadros de baixa qualidade. Umidade inconsistente ao longo da duração do experimento e almofadas secas incorretamente são dois problemas comuns que levam à deriva resultante da mudança da almofada e arrastando as células com ele para fora do FOV (Figura 3A). As mudanças na umidade afetam o ressecamento das almofadas agarose. O aumento da umidade deixa as almofadas muito molhadas, permitindo que a umidade se instale entre a almofada e o fundo do prato de fundo de vidro. A umidade deixa uma camada de líquido espessa o suficiente para as células motil se aglomerarem ou nadarem e não mantém as células bacterianas em um único plano. Enquanto isso, a diminuição da umidade seca o bloco mais rápido, o que faz com que as células desocerem no início. Outro erro comum ao usar este método com fluorescência é adquirir imagens fluorescentes com muita frequência ou expor células bacterianas à luz fluorescente por muito tempo. Curtos intervalos de aquisição para imagens fluorescentes excitam repetidamente fluoroforos com luz de alta intensidade de um comprimento de onda específico. Fluoroforos excitados então reagem com oxigênio, causando degradação do fluoróforo. O fotobleaching resultante esgota a fluorescência até que mais do fluoróforo possa ser expresso e dobrado, mas não prejudica as próprias células bacterianas(Figura 3B). A perda de fluorescência, no entanto, interfere com as medidas da expressão genética/proteína, deixando as células sem marcador até que o fluoróforo possa ser totalmente sintetizado e animado novamente. Além disso, o oxigênio interagindo com fluoroforos excitados pode formar espécies reativas de oxigênio (ROS). Esses radicais ros então danificam as células bacterianas, tornando-se, em última análise, tóxicos e resultando em morte celular dentro de algumas divisões celulares(Figura 3C). Os processos de fotobleachamento e fototoxicidade podem ser facilmente vistos, pois as células perderão sua fluorescência completamente, e em quadros subsequentes, as células deixarão de se dividir e poderão eventualmente morrer(Figura 3B-3C). Uma última questão comum ao configurar o experimento é começar com muitas células por FOV ou com células muito próximas umas das outras, que geralmente são menos de 20 μm de distância. Células lotadas no primeiro quadro produzirão aglomerados de células divisórias que simplesmente se fundem umas nas outras à medida que crescem em vez de interagir. Além disso, as células que começam muito próximas podem não ter tempo suficiente para estabelecer um gradiente de sinais secretos aos quais as outras espécies podem responder(Figura 3D), enquanto células bacterianas distantes podem não ter a chance de se encontrarem durante a duração do experimento.
A Figura 4 mostra um exemplo de como a viabilidade celular bacteriana pode ser visualizada com este protocolo adicionando iodeto de propídio às almofadas de agarose. O iodeto de propidium é impermeável às células vivas, mas pode entrar em células com membranas danificadas e se ligar a ácidos nucleicos. Aqui o WT S. aureus, no meio do registro, foi tratado apenas com mídia ou com supernanato livre de células derivado de P. aeruginosa e imagem imediatamente após o tratamento. Três canais diferentes foram visualizados: Phase, TxRed e GFP. Células verdes brilhantes indicam células vivas expressando ativamente GFP como visto para o S. aureus tratado apenas com mídia (Figura 4A), e células vermelhas indicam células S. aureus manchadas de propidium morto, após serem tratadas com P. aeruginosa supernasal(Figura 4B). Embora apenas um ponto de tempo seja mostrado, este método pode ser adaptado para determinar a viabilidade celular durante a imagem ao vivo de lapso de tempo.
Várias análises pós-imagem podem ser realizadas para quantificar aspectos das interações entre espécies. Por exemplo, o rastreamento de células pode fornecer medidas para a direção dos movimentos de célula única P. aeruginosa em direção a um aglomerado de S. aureus. Os movimentos das células P. aeruginosa individuais são rastreados a partir da estrutura uma célula deixa a jangada através da estrutura em que a célula atinge o aglomerado S. aureus (Figura 5A). A distância entre a jangada P. aeruginosa e o aglomerado S. aureus proporciona a distância euclidiana, D(E), enquanto os comprimentos totais da faixa proporcionam a distância acumulada, D(A) (Figura 5B). A direção de cada célula é calculada como uma razão de D(E)/D(A). Nos experimentos de cocultura, WT P. aeruginosa moveu-se em direção ao WT S. aureus com direção significativamente maior do que em direção a S. aureus ΔagrBDCA, um mutante sem fatores secretos regulamentados por Agr, previamente determinados a ser necessário para a motilidade direcional em relação a S. aureus24 (Figura 5C).
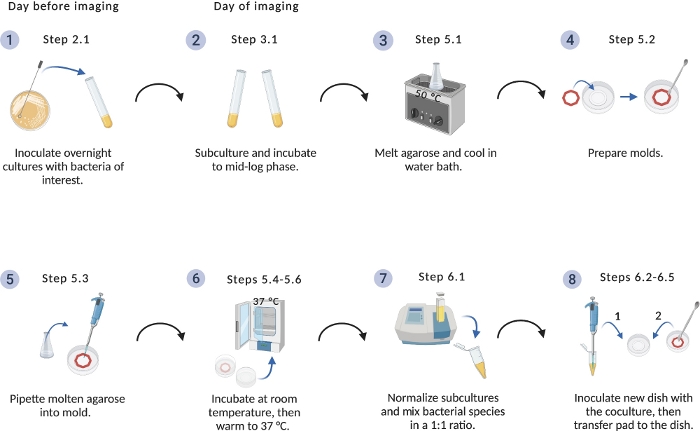
Figura 1: Esquema do protocolo de configuração de imagens.
Visão geral das etapas críticas para a preparação de culturas bacterianas e almofadas de agarose. Criado com BioRender.com. Clique aqui para ver uma versão maior desta figura.
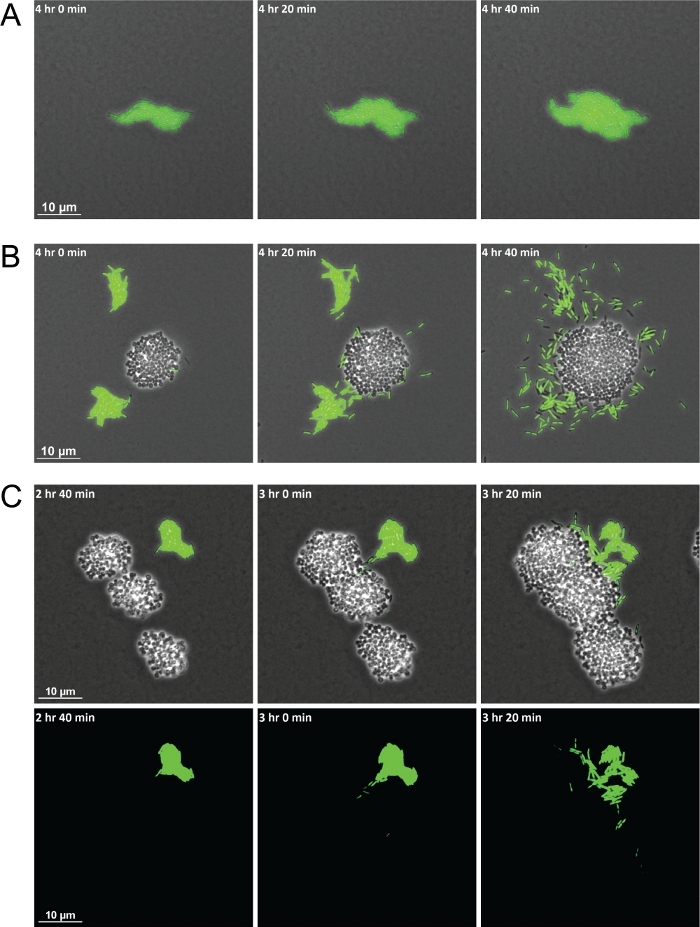
Figura 2: Microscopia de imagem ao vivo e de lapso de tempo mostra diferenças no comportamento de P. aeruginosa quando coculturada com S. aureus.
Fotos de p. aeruginosa (bacilos, verdes) na monocultura (A) e em cocultura com S. aureus (cocci, não marcado) (B). (C) Bactérias fluorescentes rotuladas permitem a visualização de células únicas P. aeruginosa invadindo aglomerados S. aureus. Contraste de fase e sobreposição de canal GFP (superior) e canal GFP sozinho (inferior). Clique aqui para ver uma versão maior desta figura.
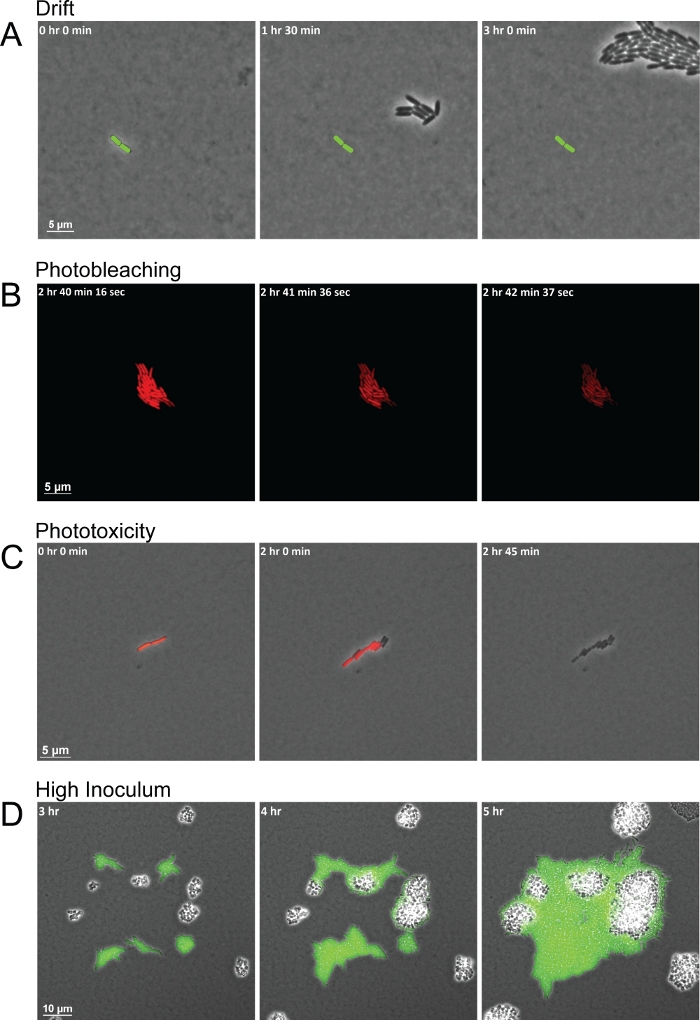
Figura 3: Fotos de fotos de fotos comuns que levam à má aquisição de imagens.
(A) Almofadas secas inadequadamente e umidade inconsistente levam à deriva das células através do FOV durante a duração da imagem. A posição da célula fundadora está marcada em cada quadro (haste verde). (B) Fotobleaching da exposição à luz por muito tempo esgotará os níveis detectáveis de fluorescência por um período de tempo, mas não mata as células. (C) Fototoxicidade como resultado da exposição frequente à luz leva à morte celular. Os primeiros sinais de fototoxicidade são vistos quando as células param de fluorescing e não se dividem. (D) Alto inóculo inicial lota células no FOV e impede a observação de interações entre espécies. Clique aqui para ver uma versão maior desta figura.
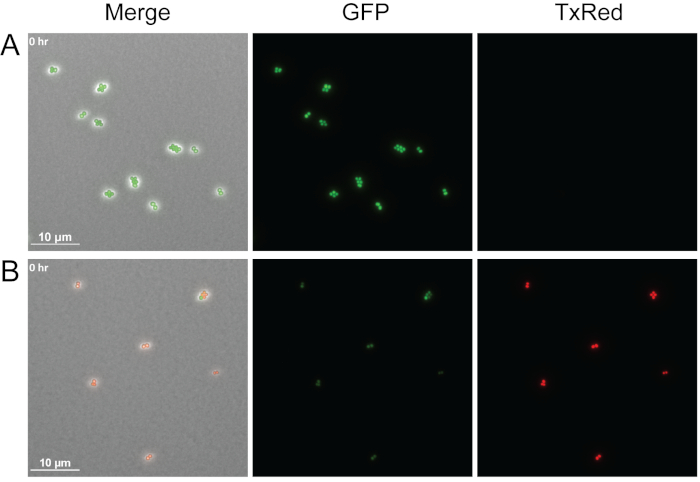
Figura 4: Comparação de células vivas versus s. aureus mortas.
Fotos de snap representativas para WT S. aureus com células rotuladas de GFP tratadas apenas com médio(A) ou supernasal P. aeruginosa livre de células(B). As células foram imediatamente imagens após o tratamento. Células vivas (verdes) expressam ativamente o GFP e excluem o iodeto propidium, enquanto as células mortas (vermelhas) perdem fluorescência GFP e permeabilização da membrana permite que o iodeto de propidium entre nas células e ligue ácidos nucleicos. Clique aqui para ver uma versão maior desta figura.

Figura 5: Análise de rastreamento celular de P. aeruginosa em cocultura com S. aureus.
Anteriormente, este método foi utilizado para realizar o rastreamento celular de WT P. aeruginosa em cocultura com WT ou ΔagrBDCA S. aureus. (A) Representação de P. aeruginosa trilhas unicelulares em uma cocultura com S. aureus ΔagrBDCA. (B) Esquema para as medições de distância euclidiana (D(E)) e distância acumulada(D (A)) utilizadas para determinar a direcionamento ((D(E)/D(A)). (C) Medições de direcionamento de células únicas WT P. aeruginosa em cocultura com WT e ΔagrBDCA S. aureus. Este número foi modificado a partir de Limoli et al. 201924. Clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar. Clique aqui para ver uma versão maior desta figura.
Discussão
Os métodos aqui apresentados descrevem um protocolo para imagens de células vivas de interações de espécies bacterianas no nível unicelular com modificações para outras aplicações, incluindo rastreamento celular e viabilidade celular de monitoramento. Este método abre novos caminhos para o estudo de comportamentos unicelulares de microrganismos na cocultura com outras espécies ao longo do tempo. Especificamente, o protocolo demonstra a utilidade deste método de cocultura na observação de comportamentos de superfície bacteriana, particularmente ao estudar organismos que possuem apêndices associados à superfície e ao líquido para a motilidade. Por exemplo, limitando o movimento bacteriano a uma superfície em um único plano de foco, a motilidade pili-mediada aumentada e direcional de P. aeruginosa em resposta a S. aureus pode ser visualizada.
Como mencionado anteriormente, alcançar resultados ideais com este método de imagem requer a consideração de várias condições, incluindo temperatura e umidade dos instrumentos de amostra e imagem (ou seja, objetivos e estágio). Para obter mais dicas e solução de problemas, consulte o Arquivo Suplementar 1. Um passo crítico no uso bem-sucedido do protocolo é a preparação das almofadas agarose, já que a secagem inadequada é um dos problemas mais comuns. Como mostrado na Figura 3A, se as almofadas não estiverem secas por tempo suficiente, a deriva aparece no início da imagem de lapso de tempo, enquanto as almofadas que são secas por muito tempo começam a encolher e as células saem do FOV algumas horas em imagens. Garantir que todos os materiais sejam pré-aquecidos e mantidos à temperatura e umidade uniformes durante a duração do experimento, através do uso de uma incubadora de topo de palco e lenços umedecidos sem fiapos úmidos, ajudará a reduzir a deriva. Também é aconselhável que um bloco de backup seja sempre feito no caso de a primeira almofada não estar seca corretamente ou lágrimas durante a transferência do molde para o prato de amostra. Além disso, é importante usar um meio de baixa autofluorescência, tanto para culturas bacterianas crescentes quanto para fazer as almofadas, a fim de minimizar a fluorescência de fundo do meio ao fotografar as células. Recomenda-se o uso de mídia mínima para microscopia, uma vez que a mídia rica muitas vezes tem alta autofluorescência. Começando com um inóculo de baixa densidade e até mesmo distribuição espacial de células no FOV também são fatores-chave neste método. Especificamente, em estudos anteriores que avaliavam as interações de P. aeruginosa e S. aureus, isso permitiu que as bactérias gerassem um gradiente suficiente de fatores secretados que podem então ser detectados pelas outras espécies presentes (Figura 2B).
Apesar dos benefícios desse método, há também limitações, incluindo seu preço, natureza de baixo rendimento, restrições de fluorescência e alta dependência do controle das condições ambientais. Figura 3B-3C mostram as principais limitações do uso de microscopia de fluorescência. Como discutido na seção de resultados, se as imagens fluorescentes forem capturadas em intervalos curtos, podem ocorrer fotobleaching e fototoxicidade. Para evitar esses dois desfechos, os intervalos de imagem fluorescentes devem ser retirados o suficiente e com o tempo de exposição à luz fluorescente o mais baixo possível para visualizar adequadamente o fluoróforo. Além disso, ao determinar o intervalo para a imagem de fluorescência, é importante considerar o tempo de maturação de cada fluoróforo. Os fluoroforos utilizados nas cepas P. aeruginosa e S. aureus mostradas nas Cepas Figura 2, Figura 3 e Figura 4, por exemplo, têm um tempo de maturação de cerca de 20 minutos e, portanto, podem ser animados a cada 20 minutos sem preocupação com possíveis efeitos de fotobleaching. Enquanto isso, outra desvantagem deste método é que ele não permite observações de interações interespécies tardias uma vez que as células atingem uma alta densidade celular. Para visualizar células bacterianas individuais, elas devem permanecer em um único plano de foco. No entanto, uma vez que a população atinge uma alta densidade celular, as células começam a crescer em mais de um plano.
Este método pode ser modificado para estudar diferentes fenótipos, como viabilidade celular(Figura 4) e expressão de genes de interesse (dados não mostrados). A Figura 4 mostra um exemplo de como o método foi adaptado para visualizar a viabilidade bacteriana adicionando iodeto de propídio às almofadas de agarose. Outra aplicação deste método é medir a expressão genética/proteína bacteriana na cocultura com outro organismo através de repórteres fluorescentes. Por exemplo, fluoroforos múltiplos podem ser incorporados em um vetor plasmídeo ou no cromossomo bacteriano para estudar simultaneamente a expressão de diferentes genes ou proteínas. Aqui é importante selecionar fluoroforos que não tenham espectros de excitação e emissão sobrepostos. Por fim, o uso do rastreamento de células bacterianas na análise pós-imagem permite que a direcionalidade (Figura 5),velocidade e aceleração, entre outras medidas, sejam calculadas também24.
No geral, este método de imagem de cocultura adaptado de protocolos de monocultura descritos anteriormente aumenta a capacidade de visualizar comportamentos de múltiplas espécies bacterianas na cocultura. Este método oferece a oportunidade de estudar micróbios de uma perspectiva de cultura mista, o que aumentará a compreensão de como cada espécie altera seus comportamentos de forma unicelular, fornecendo, em última análise, uma nova visão sobre como as espécies bacterianas interagem em ambientes polimicrobianos.
Divulgações
Os autores declaram que não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado por financiamento do Prêmio de Transição Pós-Doutor-Faculdade da Fundação Cystic Pós-Docente LIMOLI18F5 (DHL), Prêmio de Recrutamento de Faculdades Juniores da Fundação Cística Júnior LIMOLI19R3 (DHL) e Bolsa de Treinamento NIH T32 5T32HL007638-34 (ASP). Agradecemos a Jeffrey Meisner, Minsu Kim e Ethan Garner por compartilharem protocolos iniciais e conselhos para imagens e fazer almofadas.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose pads | |||
| 35 mm Glass Bottom Dish with 20 mm Micro-well #1.5 Cover Glass | Cellvis | D35-20-1.5-N | One for agarose pad molds, one for experiment |
| KimWipes | Kimberly-Clark Professional | 06-666A | |
| Low-Melt Agarose | Nu-Sieve GTG/Lonza | 50081 | For making agarose pads |
| Round-Bottom Spatulas | VWR | 82027-492 | |
| Round-Tapered Spatulas | VWR | 82027-530 | |
| Silicon Isolators, Press-to-Seal, 1 well, D diameter 2.0 mm 20 mm, silicone/adhesive | Sigma-Aldrich | S6685-25EA | For agarose pad molds |
| Sterile Petri Plates, 85 mm | Kord-Valmark /sold by RPI | 2900 | |
| Tweezers | VWR | 89259-944 | |
| M8T Minimal Media | |||
| D (+) Glucose | RPI | G32045 | |
| KH2PO4 | RPI | P250500 | |
| MgSO4 | Sigma-Aldrich | 208094 | |
| NaCl | RPI | S23025 | |
| Na2HPO4.7H2O | Sigma-Aldrich | 230391 | |
| Tryptone | BD Biosciences | DF0123173 | |
| Microscope | |||
| Andor Sona 4.2B-11 | Andor | 77026135 | Camera. 4.2 Megapixel Back-illuminated sCMOS, 11 μm pixel, 95% QE, 48 fps, USB 3.0, F-mount. |
| Filter Cube GFP | Nikon | 96372 | Filter cube |
| Filter Cube TxRed | Nikon | 96375 | Filter cube |
| H201-NIKON-TI-S-ER | Okolab | 77057447 | Stagetop incubator |
| Nikon NIS-Elements AR with GA3 and 2D and 3D tracking | Nikon | 77010609, MQS43110, 77010603, MQS42950 | Software for data analysis |
| Nikon Ti2 Eclipse | Nikon | Model Ti2-E | Microscope |
| CFI Plan Apo ƛ20x objective (0.75NA) | Nikon | MRD00205 | Objective |
| CFI Plan Apo ƛ100x oil Ph3 DM objective (1.45NA) | Nikon | MRD31905 | Objective |
| ThermoBox with built-in fan heaters | Tokai Hit | TI2TB-E-BK | Enclosure |
| Bacterial Strains | |||
| Pseudomonas aeruginosa PA14 (WT) | PMID: 7604262 | Non-mucoid prototroph | |
| Pseudomonas aeruginosa PA14 (WT) pSMC21 (Ptac-GFP) | PMID: 9361441 | ||
| Pseudomonas aeruginosa PAO1 (WT) pPrpoD-mKate2 | PMID: 26041805 | ||
| Staphylococcus aureus USA300 LAC (WT) | PMID: 23404398 | USA300 CA-Methicillin resistant strain LAC without plasmids | |
| Staphylococcus aureus USA300 LAC (WT) pCM29 (sarAP1-sGFP) | PMID: 20829608 | ||
| Staphylococcus aureus USA300 LAC ΔagrBDCA | PMID: 31713513 | ||
| Viability Stain | |||
| Propidium Iodide | Invitrogen | L7012 | LIVE/DEAD™ BacLight™ Bacterial Viability Kit |
Referências
- Lamichhane, J. R., Venturi, V. Synergisms between microbial pathogens in plant disease complexes: a growing trend. Frontiers in Plant Science. 6, 385 (2015).
- Cursino, L., et al. Identification of an operon, Pil-Chp, that controls twitching motility and virulence in Xylella fastidiosa. Molecular Plant-Microbe Interactions. 24 (10), 1198-1206 (2011).
- Limoli, D. H., Hoffman, L. R. Help, hinder, hide and harm: what can we learn from the interactions between Pseudomonas aeruginosa and Staphylococcus aureus during respiratory infections. Thorax. 74, 684-692 (2019).
- Gabrilska, R. A., Rumbaugh, K. P. Biofilm models of polymicrobial infection. Future Microbiology. 10 (12), 1997-2015 (2015).
- Nobile, C. J., Mitchell, A. P. Microbial biofilms: e pluribus unum. Current Biology. 17 (10), 349-353 (2007).
- Marino, P. J., et al. Community analysis of dental plaque and endotracheal tube biofilms from mechanically ventilated patients. Journal of Critical Care. 39, 149-155 (2017).
- Frank, D. N., et al. Microbial diversity in chronic open wounds. Wound Repair and Regeneration. 17, 163-172 (2009).
- Fazli, M., et al. Nonrandom distribution of Pseudomonas aeruginosa and Staphylococcus aureus in chronic wounds. Journal of Clinical Microbiology. 47 (12), 4084-4089 (2009).
- Shimizu, K., et al. Pathogens in COPD exacerbations identified by comprehensive real-time PCR plus older methods. International Journal of Chronic Obstructive Pulmonary Disease. 10, 2009-2016 (2015).
- Behnia, M., Logan, S. C., Fallen, L., Catalano, P. Nosocomial and ventilator-associated pneumonia in a community hospital intensive care unit: a retrospective review and analysis. BMC Research Notes. 7, 232 (2014).
- Maliniak, M. L., Stecenko, A. A., McCarty, N. A. A longitudinal analysis of chronic MRSA and Pseudomonas aeruginosa co-infection in cystic fibrosis: a single-center study. Journal of Cystic Fibrosis. 15 (3), 350-356 (2016).
- Limoli, D. H., et al. Staphylococcus aureus and Pseudomonas aeruginosa co-infection is associated with cystic fibrosis-related diabetes and poor clinical outcomes. European Journal of Clinical Microbiology & Infectious Diseases. 35 (6), 947-953 (2016).
- Hotterbeekx, A., Kumar-Singh, S., Goossens, H., Malhotra-Kumar, S. In vivo and in vitro interactions between Pseudomonas aeruginosa and Staphylococcus spp. Frontiers in Cellular and Infection Microbiology. 7 (106), (2017).
- Smith, K., et al. Aspergillus fumigatus enhances elastase production in Pseudomonas aeruginosa co-cultures. Medical Mycology. 53, 645-655 (2015).
- Michelson, C. F., et al. Staphylococcus aureus alters growth activity, autolysis, and antibiotic tolerance in a human host-adapted Pseudomonas aeruginosa lineage. Journal of Bacteriology. 196 (22), 3903-3911 (2014).
- Ngamdee, W., et al. Competition between Burkholderia pseudomallei and B. thailandensis. BMC Microbiology. 15, 56 (2015).
- Heir, E., Møretrø, T., Simessen, A., Langsrud, S. Listeria monocytogenes strains show larger variations in competitive growth in mixed culture biofilms and suspensions with bacteria from food processing environments. International Journal of Food Microbiology. 275, 46-55 (2018).
- Lutz, C., Thomas, T., Steinberg, P., Kjelleberg, S., Egan, S. Effect of interspecific competition on trait variation in Phaeobacter inhibens biofilms. Environmental Microbiology. 18 (5), 1635-1645 (2016).
- Meisner, J., et al. FtsEX is required for CwlO peptidoglycan hydrolase activity during cell wall elongation in Bacillus subtilis. Molecular Microbiology. 89 (6), 1069-1083 (2013).
- Coates, J., et al. Antibiotic-induced population fluctuations and stochastic clearance of bacteria. eLife. 7, 32976 (2018).
- Korber, D. R., Lawrence, J. R., Sutton, B., Caldwell, D. E. Effect of laminar flow velocity on the kinetics of surface recolonization by Mot+ and Mot- Pseudomonas fluorescens. Microbial Ecology. 18, 1-19 (1989).
- Lawrence, J. R., Korber, D. R., Caldwell, D. E. Behavioral analysis of Vibrio parahaemolyticus variants in high- and low- viscosity microenvironments by use of digital image processing. Journal of Bacteriology. 174 (17), 5732-5739 (1992).
- Lawrence, J. R., Wolfaardt, G. M., Korber, D. R. Determination of diffusion coefficients in biofilms by confocal laser microscopy. Applied and Environmental Microbiology. 60 (4), 1166-1173 (1994).
- Limoli, D. H., et al. Interspecies interactions induce exploratory motility in Pseudomonas aeruginosa. eLife. 8, 47365 (2019).
- Burrows, L. Pseudomonas aeruginosa twitching motility: type IV pili in action. Annual Review of Microbiology. 66 (1), 493-520 (2012).
- Lee, C. K., et al. Multigenerational memory and adaptive adhesion in early bacterial biofilm communities. PNAS. 115 (17), 4471-4476 (2018).
- Tolosa, A., et al. Enhanced field-of-view integral imaging display using multi-Köhler illumination. Optical Society of America. 22 (26), 31853-31863 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados