Method Article
Visualización cinética de interacciones bacterianas entre especies de una sola célula
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo de imágenes celulares de células bacterianas vivas permite visualizar las interacciones entre múltiples especies bacterianas a nivel de una sola célula a lo largo del tiempo. Las imágenes de lapso de tiempo permiten la observación de cada especie bacteriana en monocultivo o cocultura para interrogar las interacciones entre especies en comunidades bacterianas multiespecies, incluida la motilidad celular individual y la viabilidad.
Resumen
Las comunidades polimicrobianas son omnipresentes en la naturaleza, pero estudiar sus interacciones a nivel de una sola célula es difícil. Por lo tanto, se ha desarrollado un método basado en microscopía para observar las interacciones entre especies entre dos patógenos bacterianos. El uso de este método para interrogar las interacciones entre un patógeno Gram-negativo móvil, Pseudomonas aeruginosa y un patógeno Gram-positivo no móvil, Staphylococcus aureus se muestra aquí. Este protocolo consiste en co-inocular cada especie entre un cubreobjetos y una almohadilla de agarosa, que mantiene las células en un solo plano y permite la visualización de comportamientos bacterianos tanto en el espacio como en el tiempo.
Además, la microscopía de lapso de tiempo que se ha demostrado aquí es ideal para visualizar las interacciones tempranas que tienen lugar entre dos o más especies bacterianas, incluidos los cambios en la motilidad de las especies bacterianas en el monocultivo y en el cocultivo con otras especies. Debido a la naturaleza del espacio limitado de la muestra en la configuración de la microscopía, este protocolo es menos aplicable para estudiar las interacciones posteriores entre las especies bacterianas una vez que las poblaciones celulares son demasiado altas. Sin embargo, hay varias aplicaciones diferentes del protocolo que incluyen el uso de tinción para imágenes de células bacterianas vivas y muertas, la cuantificación de la expresión génica o proteica a través de reporteros fluorescentes, y el seguimiento del movimiento celular bacteriano en experimentos de una sola especie y multiespecie.
Introducción
Las comunidades polimicrobianas son de naturaleza común, abarcando una variedad de ambientales1,,2,3 y nichos humanos4,5. Algunas de las infecciones polimicrobianas más notorias en enfermedades humanas incluyen biopelículas dentales6, heridas crónicas7,8, e infecciones respiratorias en individuos con enfermedad pulmonar obstructiva crónica9, neumonía asociada al respirador10, y la enfermedad genética fibrosis quística (CF)11,12. Estas infecciones a menudo se componen de diversas especies microbianas; sin embargo, ha surgido un enfoque reciente en las interacciones entre la bacteria Gram-positiva Staphylococcus aureus y la bacteria Gram-negativa Pseudomonas aeruginosa. Los estudios que incluyen pacientes coinfectados con estos organismos y análisis in vitro revelan interacciones competitivas y cooperativas que pueden tener influencias profundas e inesperadas en la progresión de la enfermedad, la supervivencia microbiana, la virulencia, el metabolismo y la susceptibilidad a los antibióticos (revisados en detalle por Hotterbeekx et al. 201713 y Limoli y Hoffman 20193).
El creciente interés en las interacciones entre especies durante la infección ha dado lugar a una variedad de métodos para estudiar los comportamientos de la comunidad polimicrobiana. Típicamente, estos estudios han utilizado ensayos a base de placas o caldos para investigar las diferencias fenotípicas entre el monocultivo y la cocultura. Por ejemplo, el coculturing P. aeruginosa y S. aureus en superficies sólidas ha permitido la visualización de la inhibición del crecimiento o cambios en la producción de fenotipo de colonia, pigmento o polisacárido14,,15,,16. Las biopelículas de especies mixtas, en superficies bióticas o abióticas, así como la coculturización de especies bacterianas en cultivo líquido también han permitido la medición de los cambios en el crecimiento, el metabolismo, la tolerancia a los antibióticos, la competencia y la viabilidad, además de la expresión génica y proteica17,18. Si bien estos experimentos de cultivo a granel han revelado una visión de cómo P. aeruginosa y S. aureus podrían influir mutuamente a escala comunitaria, son incapaces de responder preguntas importantes sobre las interacciones que tienen lugar a nivel de una sola célula. El método presentado aquí se suma a los enfoques para estudiar las interacciones entre especies centrándose en los cambios en el movimiento, la viabilidad celular y la expresión génica de células individuales dentro de una comunidad cocultivo a lo largo del tiempo.
Las interacciones de una sola célula pueden diferir ampliamente de las interacciones que tienen lugar en una comunidad masiva de células. A través del análisis de una sola célula, la heterogeneidad dentro de una comunidad se puede cuantificar para estudiar cómo la colocación espacial de las células influye en la dinámica de la comunidad o cómo los niveles de expresión de genes y proteínas cambian dentro de los miembros individuales de un grupo. Las celdas de seguimiento también pueden proporcionar información sobre cómo las células individuales se mueven y se comportan a lo largo del tiempo, a través de varias generaciones. Mediante el seguimiento simultáneo del movimiento celular y los cambios en la expresión génica, se pueden hacer correlaciones entre las fluctuaciones genéticas y los fenotipos correspondientes. Se han descrito protocolos previos para el estudio de especies bacterianas individuales a nivel de una sola célula. Estos estudios aprovechan las células de imagen en vivo a lo largo del tiempo en un solo plano, y han sido útiles para observar fenotipos como la división celular y la susceptibilidad a los antibióticos19,,20. Se ha utilizado una microscopía adicional de imágenes en vivo para monitorear el crecimiento, la motilidad, la colonización superficial y la formación de biopelículas de especies bacterianas únicas21,22,,23. Sin embargo, si bien estos estudios han sido perspicaces para entender la fisiología de las bacterias en el monocultivo, los experimentos para observar el comportamiento de una sola célula de múltiples especies bacterianas a lo largo del tiempo en la cocultura son limitados.
Aquí, los protocolos anteriores utilizados para la imagen de una sola especie se han adaptado para estudiar las interacciones entre P. aeruginosa y S. aureus. Estos organismos se pueden diferenciar bajo contraste de fase basado en sus morfologías celulares(P. aeruginosa son bacilos y S. aureus son cocci). El desarrollo de este método ha permitido recientemente la visualización de comportamientos de motilidad previamente no descritos de P. aeruginosa en presencia de S. aureus24. Se encontró que P. aeruginosa era capaz de detectar S. aureus desde la distancia y responder con movimientos de una sola célula crecientes y direccionales hacia grupos de células de S. aureus. Se encontró que el movimiento P. aeruginosa hacia S. aureus requería el pili tipo IV (TFP), proyecciones similares al cabello cuya extensión coordinada y retracción generan un movimiento llamado motilidad tembloroso25.
Estos estudios demuestran la utilidad de este método para interrogar interacciones anteriores entre especies. Sin embargo, las imágenes con densidades celulares altas en puntos de tiempo de interacción posteriores son difíciles dado que ya no se pueden identificar capas individuales de células, lo que principalmente plantea problemas durante el análisis posterior a la imagen. Dada esta limitación, el método es el más adecuado para interacciones anteriores que luego podrían ser seguidas con ensayos macroscópicos tradicionales a densidades celulares más altas representativas de interacciones posteriores. Las limitaciones adicionales de este método incluyen la naturaleza de bajo rendimiento, ya que solo se puede tomar una imagen de una muestra a la vez y el costo del microscopio, la cámara y la cámara ambiental. Además, la microscopía de fluorescencia plantea riesgos para las células bacterianas como la fototoxicidad y el fotoblancaring, limitando así la frecuencia con la que se pueden adquirir imágenes de fluorescencia. Por último, las almohadillas de agarosa utilizadas en este método son altamente susceptibles a cambios en el entorno, por lo que es crítico controlar para condiciones como la temperatura y la humedad, dado que las almohadillas pueden comenzar a encogerse o expandirse si las condiciones no son correctas. Por último, aunque este método no imita el entorno anfitrión, proporciona pistas sobre cómo las diferentes especies bacterianas responden en las superficies, que se pueden hacer un seguimiento en ensayos diseñados para imitar las condiciones ambientales/de acogida.
Este método difiere de estudios anteriores que rastrean el movimiento de una sola célula, en que las células se inoculan entre un cubreobjetos y una almohadilla de agarosa, restringiendo las células a la superficie. Esto permite el seguimiento de celdas a lo largo del tiempo en un solo plano; sin embargo, limita los ciclos de compromiso de superficie transitoria observados cuando las células se sumergen en líquido26. Un beneficio adicional de las bacterias de la imagen bajo una almohadilla de agarosa es que imita los ensayos macroscópicos de inoculación de subsuelo basados en placas clásicamente utilizados para examinar la motilidad temblorosa de P. aeruginosa 25. En este ensayo, las células bacterianas se inoculan entre la parte inferior de un plato de Petri y el agar, manteniendo las células en un solo plano a medida que se mueven a través de la parte inferior del plato hacia afuera desde el punto de inoculación, al igual que este protocolo de microscopía.
El protocolo de microscopía de lapso de tiempo para visualizar las interacciones entre especies presentadas aquí consiste en 1) preparar la muestra bacteriana y la almohadilla de agarosa, 2) seleccionar la configuración del microscopio para la adquisición de imágenes y 3) el análisis post-imagen. La visualización detallada del movimiento y seguimiento celular se puede realizar mediante la adquisición de imágenes a intervalos de tiempo cortos por contraste de fase. La microscopía de fluorescencia también se puede utilizar para determinar la viabilidad celular o la expresión génica con el tiempo. Aquí, mostramos un ejemplo de adaptación para la microscopía de fluorescencia a través de la adición de tintes de viabilidad a las almohadillas de agarosa.
Protocolo
NOTA: Puede encontrar una descripción completa y números de catálogo para todos los suministros de este protocolo en la Tabla de materiales.
1. Preparación de medios mínimos M8T
- Preparar 1 L de base de sales mínimas M8 (5x) disolviendo 64 g de Na2HPO4a7H 2O, 15 g de KH2PO4y 2,5 g de NaCl en 800 ml de agua ultrapura (UPW, resistividad 18 M/cm) en una botella de 2 L. pH a 7,6. Volumen completo a 1 L con UPW. Autoclave durante 45 min.
- Preparar 500 ml de una solución de glucosa (20% p/v) disolviendo 100 g de glucosa en 400 ml de UPW en una botella de 1 L. Solución completa a 500 mL con UPW. Autoclave durante 45 min.
- Preparar 500 ml de una solución de triptona (20% p/v) disolviendo 100 g de triptona en 400 ml de UPW. Completo a 500 mL con UPW. Autoclave durante 45 min.
- Preparar 1 L de M8 + 10% de triptona (M8T) medios mínimos añadiendo 200 ml de 5 sales mínimas M8 (1x final), 10 ml de 20% glucosa (20% 0,2% final), 1 ml de 1 M MgSO4 (1 mM final), y 50 ml de 20% de triptona (1% final) a 600 mL de UPW. Completa a 1 L con UPW. Filtrar a través de un filtro estéril de 0,2 m en una botella de almacenamiento de medios estéril de 500 ml.
Día 1:
2. Preparación de cultivos bacterianos durante la noche
- Inocular 5 ml de medios mínimos M8T con una sola colonia de P. aeruginosa o S. aureus (incluidos los antibióticos cuando proceda) e incubar durante la noche a 37oC con aireación durante no más de 16 h.
NOTA: Los patógenos bacterianos P. aeruginosa y S. aureus se utilizaron para este método, ya que son comúnmente coisolados a partir de infecciones crónicas, y el estudio de sus interacciones es importante para entender cómo contribuyen a los resultados de los pacientes durante las infecciones polimicrobianas. Otras especies bacterianas podrían utilizarse dependiendo del enfoque del estudio.
Día 2:
3. Subcultura de cepas bacterianas
- Subcultura P. aeruginosa 1:500 y S. aureus 1:1000 en 5 ml de M8T fresco (incluya antibióticos cuando sea apropiado). Incubar con aireación a 37oC hasta que los cultivos alcancen la fase de registro medio (OD600 a 0,3 - 0,5).
4. Preparación de materiales para moldes de almohadillas
- Prepare espátulas metálicas calentando el extremo de una espátula plana y redondeada de laboratorio con un quemador Bunsen hasta que la mitad del extremo plano se pueda doblar en un ángulo de 90o. Calienta el final de otra espátula plana y redondeada de laboratorio y dobla ligeramente los últimos 10 mm hasta un ángulo de 45 oC.
- Corte las cuatro esquinas de los moldes de silicona para que los moldes encajen dentro de un plato de 35 mm.
- Esterilice las espátulas y un par de pinzas añadiendo un 70% de etanol y pasándolas a través de la llama del quemador Bunsen.
- Limpie el plato y los moldes de silicona con 70% de etanol y séquelos con toallitas sin pelusas.
5. Preparación de almohadillas de agarosa
NOTA: Las almohadillas se preparan con el medio mínimo M8T como fuente de nutrientes para las bacterias utilizadas en este protocolo. Sin embargo, los nutrientes utilizados en las almohadillas se pueden modificar para diferentes organismos.
- Derretir 2% de agarosa de baja fusión en 10 ml de M8T en un matraz Erlenmeyer limpio de 50 ml. Microondas en intervalos cortos (2-5 s) hasta que la agarosa esté en solución para evitar que el contenido del matraz hierva. Una vez derretido, dejar enfriar en un baño de agua a 50 oC durante al menos 15 minutos.
- Prepare los moldes alineando el recorte de silicona con la abertura en el plato de 35 mm y toque ligeramente con la espátula para fijar la silicona al plato, y para eliminar todas las burbujas de aire entre el molde y el plato.
- Una vez enfríe, la pipeta 915 l de agarosa fundida entra en el molde. Deje la tapa entreabierta y deje que la almohadilla se seque a temperatura ambiente durante 30 min.
- Cubra el plato con la tapa y déjelo a temperatura ambiente durante 2 h adicionales.
- Prepare las toallitas de humedad enrollando firmemente una toallita sin pelusas. Coloque la toallita enrollada dentro de un plato estéril de Petri y agregue uniformemente 500 l de agua estéril a través de la toallita. Cálido a 37oC durante 1 h.
- Caliente la almohadilla y un plato estéril de 35 mm a 37 oC durante 1 h.
6. Preparación de células bacterianas y almohadillas inoculantes
- Mida el OD600 de cada subcultura y diluya P. aeruginosa a un OD600 a 0,03 y S. aureus a un ODde 600 a 0,10 en M8T precalificado a 37 oC. Mezclar P. aeruginosa y S. aureus en una proporción de 1:1 y vórtice.
NOTA: Si las cepas requieren antibióticos, los antibióticos se pueden añadir a la noche y a la subcultura, pero no deben añadirse al mezclar especies para la toma de imágenes si afecta a las otras especies de la cocultura. La estabilidad del plásmido y los efectos de los antibióticos en todas las especies de la cocultura deberán determinarse para cada organismo/plásmido que se utilice. - Pipetear 1 l de cocultura uniformemente en la parte inferior de un plato de tapapso de vidrio estéril precalegado y estéril de 35 mm.
- Retire el recorte de silicona del molde con pinzas estériles.
- Retire la almohadilla del plato con espátulas estériles.
- Deslice la espátula ligeramente doblada debajo del borde de la almohadilla, mientras sostiene el molde boca abajo, para dejarla caer sobre una tapa de placa Petri estéril.
NOTA: Tenga cuidado de no forzar la almohadilla o se rasgará. Lleve un registro de qué lado de la almohadilla es la parte inferior.
- Deslice la espátula ligeramente doblada debajo del borde de la almohadilla, mientras sostiene el molde boca abajo, para dejarla caer sobre una tapa de placa Petri estéril.
- Transfiera la almohadilla al plato con las células bacterianas, de abajo hacia abajo, deslizando la espátula en ángulo de 90o debajo de la almohadilla y colocándola en la parte superior del cubreobjetos inoculado. Utilice la espátula en ángulo de 90o para hacer que la almohadilla se enjuague contra el cubreobjetos y presione suavemente cualquier burbuja de aire.
- Retire el exceso de humedad de las toallitas húmedas y luego colóquela alrededor del borde del plato, asegurándose de que no toque la almohadilla. La muestra ya está lista para la toma de imágenes.
7. Configuración del microscopio para imágenes en vivo
- Cámara ambiental cálida a 37oC al menos 2 h antes de la instalación experimental.
- Encienda todos los componentes, incluidos el microscopio, la computadora y las fuentes de luz para imágenes de campo brillante y fluorescencia.
- Abra el software de imágenes y asegúrese de que las fuentes de luz estén conectadas y en funcionamiento.
- Realice la iluminaciónK-hler 27 para alinear todos los planos de imagen.
- En primer lugar, pon el cubreobjeto marcado en el foco con un objetivo de 20x usando un plato "ficticio". Asegúrese de que la torreta del condensador esté ajustada a la posición "abierta".
NOTA: Para cada objetivo/muestra único se debe realizar la iluminación de K-hler para alinear correctamente los planos de imagen. Sin embargo, el enfoque y la alineación con el plato "ficticio" en la reducción de la ampliación acelera la configuración de muestras en vivo con un aumento más alto para limitar el tiempo entre la configuración y la hora de inicio del experimento. - Enfoque el condensador.
- Cierre el diafragma de campo.
NOTA: Debería aparecer una abertura en forma de octágono. Si el condensador está completamente desenfocado, todo el campo de visión (FOV) aparecerá oscuro. - Gire las perillas de enfoque del condensador hasta que los bordes del octágono estén nítidos.
NOTA: La intensidad de la luz aumentará a medida que los planos de imagen se acerquen a la alineación correcta.
- Cierre el diafragma de campo.
- Alinee el condensador.
- Centre el condensador de campo ajustando los mandos de alineación.
NOTA: El octágono debe centrarse en la mitad del FOV. Las perillas de alineación varían en función del microscopio. Por ejemplo, algunos condensadores de microscopio tienen perillas, mientras que otros tienen tornillos que requieren un destornillador.
- Centre el condensador de campo ajustando los mandos de alineación.
- Enfoque el filamento de la lámpara y ajuste la abertura del condensador.
- Coloque un telescopio de fase o una lente Bertrand en el camino de la luz para observar el plano focal posterior del objetivo.
NOTA: Debe haber dos círculos concéntricos. - Gire la perilla de enfoque de la lente Bertrand hasta que los anillos se vean nítidos.
- Retire la lente Bertrand de la trayectoria de la luz.
- Coloque un telescopio de fase o una lente Bertrand en el camino de la luz para observar el plano focal posterior del objetivo.
- Abra el diafragma de campo hasta que el octágono esté justo fuera del FOV.
- Cambie al objetivo de 100x y deslice el anillo de fase que coincida en su lugar antes de agregar una gota de aceite de inmersión, y coloque el plato de muestra preparado en la parte superior.
- Realice la iluminación de K-hler en el objetivo 100x con el plato de muestra bacteriano.
- En primer lugar, pon el cubreobjeto marcado en el foco con un objetivo de 20x usando un plato "ficticio". Asegúrese de que la torreta del condensador esté ajustada a la posición "abierta".
- Concéntrese en las bacterias usando sólo el ajuste fino. Una vez que las bacterias en el FOV se enfocan a través del ocular, cambie la trayectoria de la luz a la cámara presionando el botón de la cámara en el microscopio.
- Haga clic en la opción Fase del software de imágenes.
- Ajuste el porcentaje de luz LED DIA emitida seleccionando la fuente de luz en el software e introduciendo manualmente el porcentaje de luz deseado que se utilizará o deslizando la barra en la escala de porcentaje de luz.
- Ajuste el tiempo de exposición de la luz LED DIA haciendo clic en la fuente de luz y introduciendo manualmente el tiempo de exposición deseado o seleccionando un tiempo de exposición en el menú desplegable proporcionado.
NOTA: El tiempo de exposición variará dependiendo de la cámara que se esté utilizando. - Si utiliza fluorescencia, ajuste la configuración de la cámara en cada canal correspondiente (es decir, TxRed, GFP), haciendo clic en el canal fluorescente de interés.
- Ajuste el porcentaje de luz de fluorescencia emitida y, a continuación, ajuste el tiempo de exposición (como se realiza en los pasos 7.7 y 7.8 para la luz DIA LED).
- También puede cambiar la profundidad de bits para ajustar el rango dinámico seleccionando una de las otras opciones de profundidad de bits en el menú desplegable Controles de visualización.
- Elija las posiciones de interés XY haciendo clic en la opción XY en el menú de controles de adquisición.
- Mueva la posición del escenario con el joy stick, o haciendo clic y arrastrando el FOV en la pantalla, y haga clic en el cuadro vacío para guardar las coordenadas X e Y de una posición específica.
NOTA: Se recomienda seleccionar no más de tres posiciones XY diferentes, lo más juntas posibles, para la adquisición de lapso de tiempo.
- Mueva la posición del escenario con el joy stick, o haciendo clic y arrastrando el FOV en la pantalla, y haga clic en el cuadro vacío para guardar las coordenadas X e Y de una posición específica.
- Encienda el sistema de enfoque perfecto (PFS) haciendo clic en el cuadro PFS en la pestaña XY del menú de control de adquisición o presionando el botón PFS en el panel de control joy stick.
- Gire las perillas de ajuste fino para centrarse en las células bacterianas.
- Haga clic en el botón PFS para cada posición XY, una vez que las celdas estén en el plano de enfoque deseado.
NOTA: PFS compensa la deriva en el eje Z durante los experimentos de lapso de tiempo. Esto es necesario para mantener el foco de las células bacterianas a lo largo del tiempo. Diferentes fabricantes tienen diferentes sistemas de compensación. - Elija las condiciones de adquisición de imagen, incluido el intervalo de adquisición y la frecuencia de cada canal (por ejemplo, contraste de fase y cada fluoróforo) seleccionando en las opciones del menú de controles de adquisición.
NOTA: Para los experimentos presentados aquí, las imágenes de contraste de fase se adquieren a intervalos cada 5-10 s, mientras que las imágenes de fluorescencia en los canales GFP y TxRed se adquieren cada 20 minutos. - Comience la toma de imágenes una vez que se configuren los ajustes de microscopio y adquisición.
8. Opcional: Modificaciones para imágenes vivas/muertas
- Derretir un 2% de agarosa en 10 ml de M8T y dejar enfriar en un baño de agua a 50oC durante al menos 15 min.
- Añadir 1 mM de yoduro de propidio a la agarosa fundida.
- Una vez enfriado, pipeta 915 l de agarosa en el molde preparado.
- Deje la tapa entreabierta y deje secar la almohadilla a temperatura ambiente durante 30 min. Proteger de la luz.
- Cubra el plato con la tapa y déjelo a temperatura ambiente durante 2 h adicionales.
- Prepare toallitas de humedad.
- Enrolle bien el papel sin pelusas.
- Coloque la toallita en un plato estéril de Petri y agregue 500 l de agua estéril a la toallita.
- Incubar a 37oC durante 1 h.
- Incubar la almohadilla y un plato estéril de 35 mm a 37oC durante 1 h.
- Proceder a inocular el plato como se indica en la sección 6: Preparación de células bacterianas y almohadillas de inoculación.
9. Análisis de datos
- Identificación de células
- Abra el archivo de imagen en el software de imágenes y recorte el archivo para incluir solo los fotogramas que se utilizarán para el seguimiento, acercarse a las celdas de interés y solo en el canal de contraste de fase.
NOTA: El archivo recortado se puede guardar como un nuevo archivo en el que los datos de seguimiento se pueden almacenar sin interferir con el archivo original. - Seleccione la opción para elegir regiones de interés (ROI) en el menú de controles de análisis y defina ROI trazando celdas bacterianas individuales o grupos de celdas en el primer fotograma que se utilizarán para el análisis.
NOTA: Los ROI se pueden definir manualmente o como binarios.- Definir manualmente el ROI
- Rastree el perímetro de cada célula bacteriana individual o grupos de células para definir manualmente los ROI.
NOTA: En el software de análisis, P. aeruginosa,u otras celdas en forma de varilla, se puede trazar manualmente seleccionando el ROI de Elipse y dibujando una elipse ajustada al tamaño de la célula bacteriana. Como alternativa, se puede seleccionar la opción Roi de polígono para rastrear ROI no con forma tradicional, como clústeres de celdas.
- Rastree el perímetro de cada célula bacteriana individual o grupos de células para definir manualmente los ROI.
- Identificar ROI binarios
- Haga clic la opción binaria al ROI para definir los ROI binarios.
NOTA: Los objetos se definen en una capa binaria basada en la separación de las células bacterianas más oscuras pixeladas del fondo pixelado más claro en el canal de contraste de fase. - Para umbralar las celdas, seleccione la opción Umbral en el menú de controles de análisis. Seleccione el canal de interés y deslice las barras en el histograma de fluorescencia para ajustar los valores de intervalo de umbral.
NOTA: La identificación de objetos binarios para ROI también se puede definir en imágenes fluorescentes mediante el umbral de las células bacterianas. El umbral establece qué intensidades de fluorescencia se consideran objetos y qué intensidades de fluorescencia constituyen el fondo.
- Haga clic la opción binaria al ROI para definir los ROI binarios.
- Definir manualmente el ROI
- Abra el archivo de imagen en el software de imágenes y recorte el archivo para incluir solo los fotogramas que se utilizarán para el seguimiento, acercarse a las celdas de interés y solo en el canal de contraste de fase.
- Seguimiento de celdas
- Para realizar un seguimiento manual de los ROI, seleccione el siguiente fotograma de la secuencia de imágenes y ajuste la posición de los ROI haciendo clic y arrastrando cada ROI para alinearse con la nueva posición de la célula bacteriana original.
NOTA: Si las células bacterianas no han cambiado de posición, no es necesario mover los ROI. - Repita el proceso en todos los fotogramas secuenciales para los que se va a realizar un seguimiento de las celdas.
- A medida que las células se dividen, defina las celdas hijas como nuevos ROI, como se describe en el paso 9.1, y comience a rastrear las celdas recién divididas.
NOTA: Si las celdas se identifican como binarios, utilice la función Binarios de pista para realizar un seguimiento automático de los ROI en los fotogramas seleccionados. - Una vez que se haya realizado un seguimiento de las celdas a través de todos los fotogramas seleccionados, exporte los datos que se van a analizar.
- Abra la hoja de cálculo de datos e identifique las medidas necesarias para el análisis (es decir, velocidad del objeto, aceleración o longitud de ruta).
NOTA: En el caso de la medición de la dirección, se requieren las mediciones de longitud de trayecto y longitud de línea. La Longitud de Línea es una medida de la distancia euclidiana, o la distancia recta desde el origen de la pista hasta el borde de la colonia de S. aureus. La Longitud de trayecto es una medida de la distancia acumulada o la suma de los segmentos de pista de todos los fotogramas. La dirección se puede calcular como una relación de la distancia euclidiana, D(E), (Longitud de línea), sobre la distancia acumulada, D(A), (Longitud de trayecto).
- Para realizar un seguimiento manual de los ROI, seleccione el siguiente fotograma de la secuencia de imágenes y ajuste la posición de los ROI haciendo clic y arrastrando cada ROI para alinearse con la nueva posición de la célula bacteriana original.
- Cuantificación de la fluorescencia
- Defina los ROI para las células bacterianas fluorescentes como se describe en el paso 9.1.
- Repita el trazado o el movimiento de ROI para células bacterianas o racimos en los marcos fluorescentes restantes.
- Exporte la tabla generada a un archivo de hoja de cálculo para el análisis de la intensidad fluorescente.
- En la hoja de cálculo de datos, busque la columna con "Intensidad media" que representa la intensidad media de fluorescencia para el ROI de las células bacterianas trazadas.
- Graficar los valores de Intensidad media para ver los cambios en la fluorescencia a lo largo del tiempo.
NOTA: Los cambios en la fluorescencia a lo largo del tiempo representan la fluctuación de la expresión génica para el gen de interés con etiqueta fluorescente.
Resultados
El uso correcto del método descrito dará lugar a una serie de fotogramas que generan un vídeo en el que las interacciones entre especies se pueden observar a lo largo del tiempo. El esquema de la Figura 1 proporciona un objeto visual para resaltar los pasos clave implicados en la preparación de materiales para la creación de imágenes. El uso de este método ha permitido la demostración de células P. aeruginosa exhibiendo diferentes comportamientos en monocultivo versus en cocultura con S. aureus. En comparación con las células de P. aeruginosa en monocultivo que permanecen agrupadas en balsas, cuando están en cocultura con S. aureus, P. aeruginosa ha aumentado la motilidad de una sola célula hacia las colonias de S. aureus (Figura 2A-2B). Las cepas bacterianas con etiqueta fluorescente también permiten visualizar poblaciones mixtas de especies bacterianas. El uso de P. aeruginosa con etiqueta GFP permitió la observación de que después de que las células individuales de P. aeruginosa aumentaran en la motilidad, rodearán y eventualmente invadirán las colonias de S. aureus (Figura 2C). La utilización de células con marcas fluorescentes también permite la visualización de la invasión celular de P. aeruginosa en las colonias de S. aureus por primera vez(Figura 2C,inferior).
Mientras que el protocolo produce imágenes de alta calidad para observar interacciones entre especies, hay varios problemas comunes que pueden conducir a secuencias de fotogramas de mala calidad. La humedad incoherente durante la duración del experimento y las almohadillas secas incorrectamente son dos problemas comunes que conducen a la deriva resultante de la almohadilla de desplazamiento y arrastrar las células con él fuera del FOV (Figura 3A). Los cambios en la humedad afectan la sequedad de las almohadillas de agarosa. El aumento de la humedad hace que las almohadillas estén demasiado húmedas, lo que permite que la humedad se asiente entre la almohadilla y la parte inferior de la placa de fondo de vidrio. La humedad deja una capa de líquido lo suficientemente gruesa como para que las células motiles se enjambren o naden y no mantiene las células bacterianas en un solo plano. Mientras tanto, las disminuciones de humedad secan la almohadilla más rápido, lo que hace que las células se derraquen desde el principio. Otro error común al usar este método con fluorescencia es adquirir imágenes fluorescentes con demasiada frecuencia o exponer las células bacterianas a la luz fluorescente durante demasiado tiempo. Los intervalos cortos de adquisición de imágenes fluorescentes excitan repetidamente fluoróforos con luz de alta intensidad de una longitud de onda específica. Los fluoróforos excitados reaccionan con oxígeno, causando la degradación del fluoróforo. El fotoblanchado resultante agota la fluorescencia hasta que se puede expresar y plegar más del fluoróforo, pero no daña las propias células bacterianas (Figura 3B). La pérdida de fluorescencia, sin embargo, interfiere con las mediciones de la expresión génica/proteína, dejando las células sin marcador hasta que el fluoróforo puede ser completamente sintetizado y excitado de nuevo. Además, el oxígeno que interactúa con los fluoróforos excitados puede formar especies reactivas de oxígeno (ROS). Estos radicales ROS entonces dañan las células bacterianas, en última instancia, convirtiéndose en tóxicos y resultando en la muerte celular dentro de unas pocas divisiones celulares (Figura 3C). Los procesos de fotoblanqueo y fototoxicidad se pueden ver fácilmente ya que las células primero perderán su fluorescencia por completo, y en marcos posteriores, las células dejarán de dividirse y eventualmente podrán morir(Figura 3B-3C). Un último problema común al configurar el experimento es comenzar con demasiadas células por FOV o con células demasiado cerca unas de otras, lo que generalmente está a menos de 20 m de distancia. Las celdas abarrotadas en el primer fotograma producirán grupos de celdas divisorias que simplemente se fusionan entre sí a medida que crecen en lugar de interactuar. Además, las células que comienzan demasiado cerca pueden no tener tiempo suficiente para establecer un gradiente de señales secretas a las que las otras especies pueden responder (Figura 3D) mientras que las células bacterianas distantes pueden no tener la oportunidad de encontrarse entre sí dentro de la duración del experimento.
La Figura 4 muestra un ejemplo de cómo la viabilidad de las células bacterianas se puede visualizar con este protocolo mediante la adición de yoduro de propidio a las almohadillas de agarosa. El yoduro de propidium es impermeable a las células vivas, pero puede entrar en las células con membranas dañadas y unirse a los ácidos nucleicos. Aquí, el WT S. aureus con etiqueta GFP de registro medio fue tratado con medios solos o con sobrenadante libre de células derivado de P. aeruginosa e inmediatamente después del tratamiento. Se crearon tres canales diferentes: Phase, TxRed y GFP. Las células verdes brillantes indican que las células vivas expresan activamente la GFP como se ve para el S. aureus tratado sólo con medios (Figura 4A), y los glóbulos rojos indican células muertas de yoduro propidium S. aureus, después de ser tratadas con P. aeruginosa sobrenadante (Figura 4B). Aunque solo se muestra un punto de tiempo, este método se puede adaptar para determinar la viabilidad celular durante las imágenes en vivo de lapso de tiempo.
Se pueden realizar varios análisis post-imágenes para cuantificar aspectos de las interacciones entre especies. Por ejemplo, el seguimiento celular puede proporcionar mediciones para la dirección de los movimientos de células individuales de P. aeruginosa hacia un grupo de S. aureus. Los movimientos de las células individuales de P. aeruginosa se rastrean desde el marco que una celda sale de la balsa a través del marco en el que la célula alcanza el cúmulo de S. aureus (Figura 5A). La distancia entre la balsa P. aeruginosa y el cluster S. aureus proporciona la distancia euclidiana, D(E),mientras que las longitudes totales de la vía proporcionan la distancia acumulada, D(A) (Figura 5B). La dirección de cada celda se calcula como una relación de D(E)/D(A). En los experimentos de cocultura, WT P. aeruginosa se movió hacia WT S. aureus con una dirección significativamente mayor que hacia S. aureus agrBDCA, un mutante que carecía de factores secretados regulados por Agr, previamente determinado para la direccionalidad mot hacia S. aureus24 (Figura 5C).
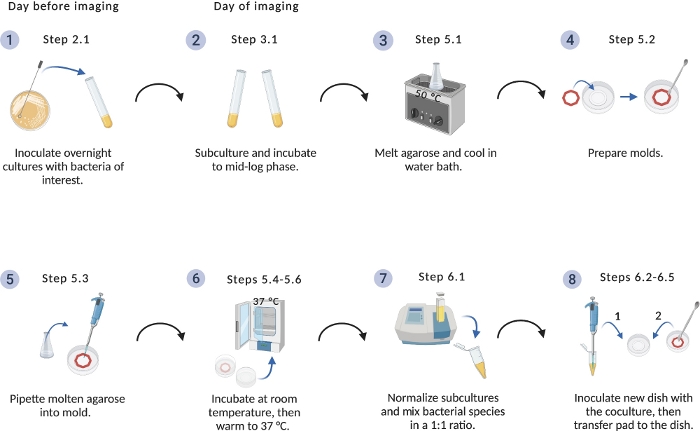
Figura 1: Esquema del protocolo de configuración de imágenes.
Visión general de los pasos críticos para la preparación de cultivos bacterianos y almohadillas de agarosa. Creado con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
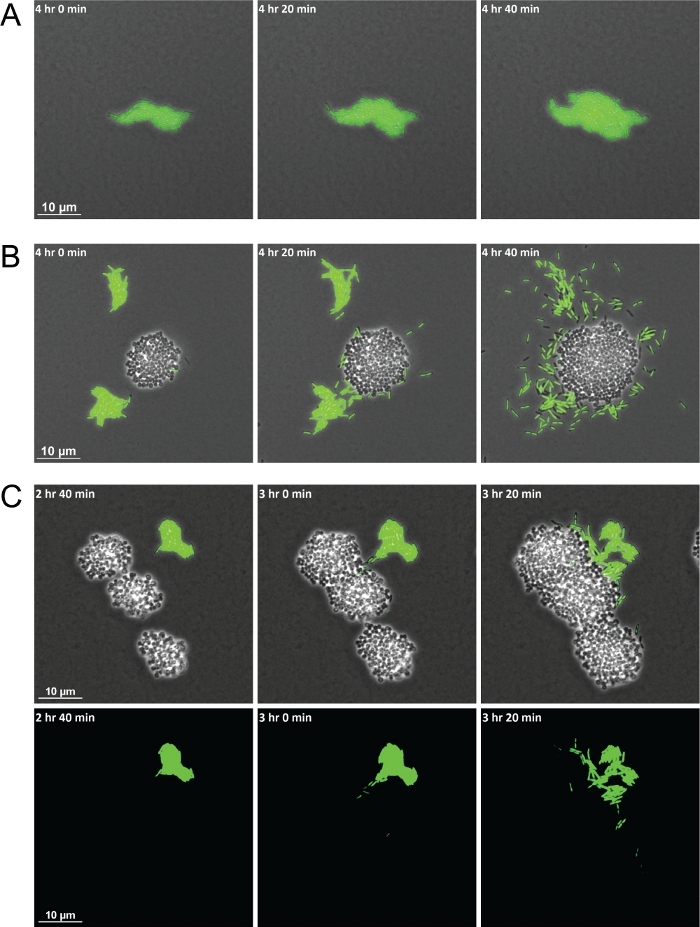
Figura 2: La microscopía de imágenes en vivo de lapso de tiempo muestra diferencias en el comportamiento de P. aeruginosa cuando se coculta con S. aureus.
Tomas de fotos representativas de P. aeruginosa (bacilos, verde) en monocultivo (A) y en cocultura con S. aureus (cocci, sin marcar) (B). (C) Las bacterias con etiqueta fluorescente permiten la visualización de células individuales de P. aeruginosa que invaden los racimos de S. aureus. Contraste de fase y superposición de canales GFP (arriba) y canal GFP solo (abajo). Haga clic aquí para ver una versión más grande de esta figura.
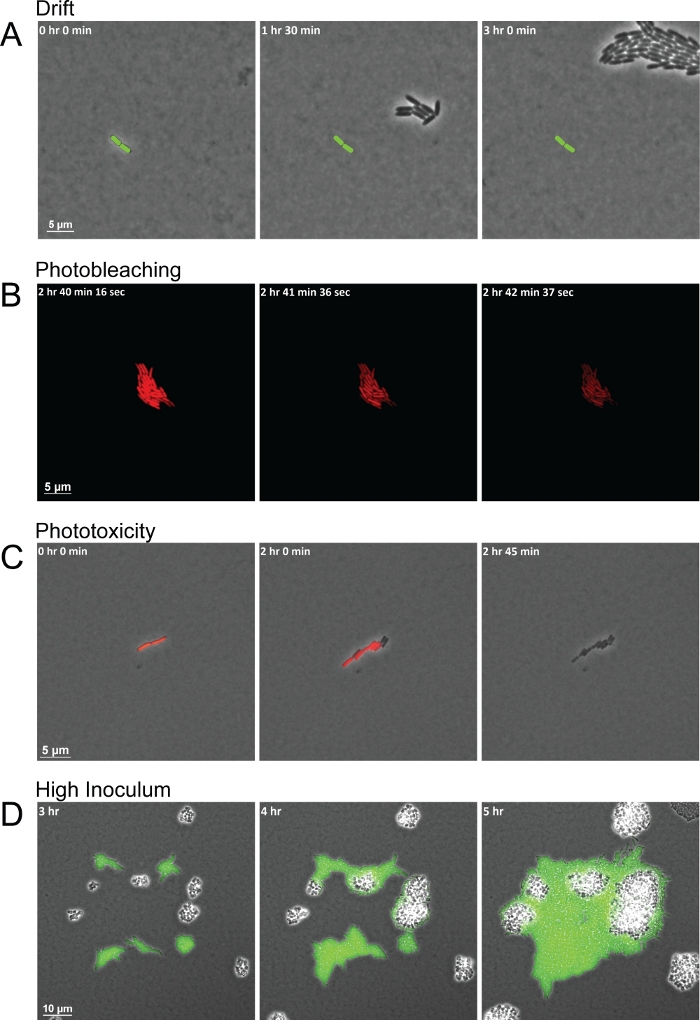
Figura 3: Tomas instantáneas representativas de problemas comunes de creación de imágenes que conducen a una mala adquisición de imágenes.
(A) Las almohadillas secas incorrectamente y la humedad inconsistente conducen a la deriva de las células a través del FOV durante la duración de la toma de imágenes. La posición de la celda fundadora está marcada en cada fotograma (barra verde). (B) El fotoblancar por la exposición a la luz durante demasiado tiempo agotará los niveles detectables de fluorescencia durante un período de tiempo, pero no mata las células. (C) La fototoxicidad como resultado de la exposición frecuente a la luz conduce a la muerte celular. Los primeros signos de fototoxicidad se observan cuando las células dejan de fluorescing y no se dividen. (D) El inóculo inicial alto amontona las células en el FOV y evita la observación de interacciones entre especies. Haga clic aquí para ver una versión más grande de esta figura.
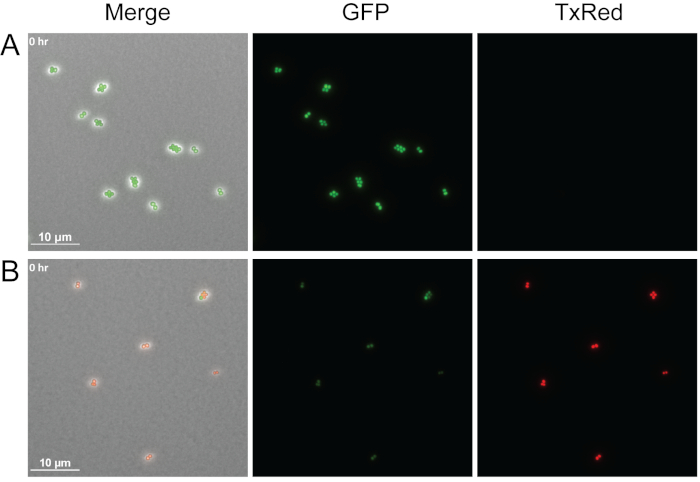
Figura 4: Comparación de células S. aureus vivas frente a muertas.
Tomas de presión representativas para WT S. aureus con etiqueta GFP tratadas con un medio solo (A) o sin células P. aeruginosa supernadante (B). Las células fueron inmediatamente imágenes después del tratamiento. Las células vivas (verdes) expresan activamente la GFP y excluyen el yoduro propidium, mientras que las células muertas (rojas) pierden fluorescencia GFP y la permeabilización de la membrana permite que el yoduro de propidium entre en las células y adhiera a los ácidos nucleicos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Análisis de seguimiento celular de P. aeruginosa en cocultura con S. aureus.
Anteriormente, este método se utilizaba método para realizar el seguimiento celular de WT P. aeruginosa en cocultura con WT oagrBDCA S. aureus. (A) Representación de las pistas de una sola célula de P. aeruginosa en una cocultura con S. aureus agrBDCA. (B) Esquema para las mediciones de distancia euclidiana (D(E)) y distancia acumulada (D(A)) utilizadas para determinar la dirección ((D(E)/D(A)). (C) Mediciones de la dirección de células únicas WT P. aeruginosa en cocultura con WT yagrBDCA S. aureus. Esta cifra ha sido modificada de Limoli et al. 201924. Haga clic aquí para ver una versión más grande de esta figura.
Archivo Suplementario. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los métodos presentados aquí describen un protocolo para la toma de imágenes de células vivas de interacciones de especies bacterianas a nivel de una sola célula con modificaciones para otras aplicaciones, incluido el seguimiento celular y la supervisión de la viabilidad celular. Este método abre nuevas vías para estudiar los comportamientos unicelulares de los microorganismos en la cocultura con otras especies a lo largo del tiempo. Específicamente, el protocolo demuestra la utilidad de este método de cocultura en la observación de comportamientos superficiales bacterianos, particularmente cuando se estudian organismos que tienen apéndices tanto superficiales como líquidos para la motilidad. Por ejemplo, al limitar el movimiento bacteriano a una superficie en un solo plano de enfoque, se puede visualizar la mayor y direccional motilidad mediada por pili de P. aeruginosa en respuesta a S. aureus.
Como se mencionó anteriormente, lograr resultados óptimos con este método de imagen requiere la consideración de varias condiciones, incluyendo la temperatura y la humedad de la muestra y los instrumentos de imagen (es decir, objetivos y etapa). Para obtener más consejos y solucionar problemas, consulte el archivo suplementario 1. Un paso crítico en el uso exitoso del protocolo es la preparación de las almohadillas de agarosa, ya que el secado inadecuado de las almohadillas es uno de los problemas más comunes. Como se muestra en la Figura 3A, si las almohadillas no se secan el tiempo suficiente, la deriva aparece al principio de la imagen de lapso de tiempo, mientras que las almohadillas que se secan durante demasiado tiempo comienzan a encogerse y las células se desvieron del FOV unas horas en la toma de imágenes. Asegurarse de que todos los materiales se precalentan y mantengan a una temperatura y humedad uniformes durante la duración del experimento, mediante el uso de una incubadora de etapa y toallitas húmedas sin pelusas, ayudará a reducir la deriva. También se recomienda que siempre se haga una almohadilla de respaldo en caso de que la primera almohadilla no se seque correctamente o se desgarros durante la transferencia del molde al plato de muestra. Además, es importante utilizar un medio de baja autofluorescencia, tanto para cultivos bacterianos en crecimiento como para hacer las almohadillas con el fin de minimizar la fluorescencia de fondo del medio al tomar imágenes de las células. Se recomienda utilizar medios mínimos para la microscopía, ya que los medios enriquecidos a menudo tienen alta autofluorescencia. A partir de un inóculo de baja densidad e incluso la distribución espacial de las células en el FOV también son factores clave en este método. Específicamente, en estudios previos que evaluaron las interacciones de P. aeruginosa y S. aureus, esto permitió a las bacterias generar un gradiente suficiente de factores secretados que luego pueden ser detectados por las otras especies presentes (Figura 2B).
A pesar de los beneficios de este método, también hay limitaciones que incluyen su precio, naturaleza de bajo rendimiento, restricciones de fluorescencia y alta dependencia de las condiciones ambientales de control. La Figura 3B-3C muestra las principales limitaciones del uso de la microscopía de fluorescencia. Como se explica en la sección de resultados, si las imágenes fluorescentes se capturan en intervalos cortos, se pueden producir fotoblanchas y fototoxicidad. Con el fin de evitar estos dos resultados, los intervalos de imagen fluorescentes deben desmontarse lo suficiente y con el tiempo de exposición a la luz fluorescente lo más bajo posible para visualizar adecuadamente el fluoróforo. Además, al determinar el intervalo para la toma de imágenes por fluorescencia, es importante tener en cuenta el tiempo de maduración de cada fluoróforo. Los fluoróforos utilizados en las cepas P. aeruginosa y S. aureus que se muestran en la Figura 2, Figura 3 y Figura 4, por ejemplo, tienen un tiempo de maduración de unos 20 minutos y por lo tanto pueden excitarse cada 20 minutos sin preocupación de posibles efectos de fotoblancada. Mientras tanto, otra desventaja de este método es que no permite observaciones de interacciones entre especies tardías una vez que las células alcanzan una alta densidad celular. Para visualizar las células bacterianas individuales, deben permanecer en un único plano de enfoque. Sin embargo, una vez que la población alcanza una alta densidad celular, las células comienzan a crecer en más de un plano.
Este método se puede modificar para estudiar diferentes fenotipos como la viabilidad celular (Figura 4) y la expresión de genes de interés (datos no mostrados). La Figura 4 muestra un ejemplo de cómo el método se ha adaptado para visualizar la viabilidad bacteriana mediante la adición de yoduro de propidio a las almohadillas de agarosa. Otra aplicación de este método es medir la expresión bacteriana de genes/proteínas en la cocultura con otro organismo a través de reporteros fluorescentes. Por ejemplo, se pueden incorporar múltiples fluoróforos en un vector plásmido o en el cromosoma bacteriano para estudiar simultáneamente la expresión de diferentes genes o proteínas. Aquí es importante seleccionar fluoróforos que no tienen espectros de excitación y emisión superpuestos. Por último, el uso del seguimiento de células bacterianas en el análisis post-imagen permite calcular la direccionalidad (Figura 5), la velocidad y la aceleración, entre otras mediciones, así24.
En general, este método de imagen de cocultivo adaptado a partir de protocolos de monocultivo descritos anteriormente mejora la capacidad de visualizar comportamientos de múltiples especies bacterianas en la cocultura. Este método ofrece la oportunidad de estudiar microbios desde una perspectiva de cultivo mixto, lo que aumentará la comprensión de cómo cada especie altera sus comportamientos de una sola célula, proporcionando en última instancia una nueva visión de cómo las especies bacterianas interactúan en ambientes polimicrobianos.
Divulgaciones
Los autores declaran que no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la financiación del Premio de Transición Postdoc-to-Faculty de la Fundación De la Cis fibrosis LIMOLI18F5 (DHL), el Premio de Reclutamiento de la Facultad Juvenil de la Fundación de Fibrosis Quística LIMOLI19R3 (DHL), y la Beca de Formación NIH T32 5T32HL007638-34 (ASP). Agradecemos a Jeffrey Meisner, Minsu Kim y Ethan Garner por compartir los protocolos iniciales y consejos para la creación de imágenes y la fabricación de almohadillas.
Materiales
| Name | Company | Catalog Number | Comments |
| Agarose pads | |||
| 35 mm Glass Bottom Dish with 20 mm Micro-well #1.5 Cover Glass | Cellvis | D35-20-1.5-N | One for agarose pad molds, one for experiment |
| KimWipes | Kimberly-Clark Professional | 06-666A | |
| Low-Melt Agarose | Nu-Sieve GTG/Lonza | 50081 | For making agarose pads |
| Round-Bottom Spatulas | VWR | 82027-492 | |
| Round-Tapered Spatulas | VWR | 82027-530 | |
| Silicon Isolators, Press-to-Seal, 1 well, D diameter 2.0 mm 20 mm, silicone/adhesive | Sigma-Aldrich | S6685-25EA | For agarose pad molds |
| Sterile Petri Plates, 85 mm | Kord-Valmark /sold by RPI | 2900 | |
| Tweezers | VWR | 89259-944 | |
| M8T Minimal Media | |||
| D (+) Glucose | RPI | G32045 | |
| KH2PO4 | RPI | P250500 | |
| MgSO4 | Sigma-Aldrich | 208094 | |
| NaCl | RPI | S23025 | |
| Na2HPO4.7H2O | Sigma-Aldrich | 230391 | |
| Tryptone | BD Biosciences | DF0123173 | |
| Microscope | |||
| Andor Sona 4.2B-11 | Andor | 77026135 | Camera. 4.2 Megapixel Back-illuminated sCMOS, 11 μm pixel, 95% QE, 48 fps, USB 3.0, F-mount. |
| Filter Cube GFP | Nikon | 96372 | Filter cube |
| Filter Cube TxRed | Nikon | 96375 | Filter cube |
| H201-NIKON-TI-S-ER | Okolab | 77057447 | Stagetop incubator |
| Nikon NIS-Elements AR with GA3 and 2D and 3D tracking | Nikon | 77010609, MQS43110, 77010603, MQS42950 | Software for data analysis |
| Nikon Ti2 Eclipse | Nikon | Model Ti2-E | Microscope |
| CFI Plan Apo ƛ20x objective (0.75NA) | Nikon | MRD00205 | Objective |
| CFI Plan Apo ƛ100x oil Ph3 DM objective (1.45NA) | Nikon | MRD31905 | Objective |
| ThermoBox with built-in fan heaters | Tokai Hit | TI2TB-E-BK | Enclosure |
| Bacterial Strains | |||
| Pseudomonas aeruginosa PA14 (WT) | PMID: 7604262 | Non-mucoid prototroph | |
| Pseudomonas aeruginosa PA14 (WT) pSMC21 (Ptac-GFP) | PMID: 9361441 | ||
| Pseudomonas aeruginosa PAO1 (WT) pPrpoD-mKate2 | PMID: 26041805 | ||
| Staphylococcus aureus USA300 LAC (WT) | PMID: 23404398 | USA300 CA-Methicillin resistant strain LAC without plasmids | |
| Staphylococcus aureus USA300 LAC (WT) pCM29 (sarAP1-sGFP) | PMID: 20829608 | ||
| Staphylococcus aureus USA300 LAC ΔagrBDCA | PMID: 31713513 | ||
| Viability Stain | |||
| Propidium Iodide | Invitrogen | L7012 | LIVE/DEAD™ BacLight™ Bacterial Viability Kit |
Referencias
- Lamichhane, J. R., Venturi, V. Synergisms between microbial pathogens in plant disease complexes: a growing trend. Frontiers in Plant Science. 6, 385 (2015).
- Cursino, L., et al. Identification of an operon, Pil-Chp, that controls twitching motility and virulence in Xylella fastidiosa. Molecular Plant-Microbe Interactions. 24 (10), 1198-1206 (2011).
- Limoli, D. H., Hoffman, L. R. Help, hinder, hide and harm: what can we learn from the interactions between Pseudomonas aeruginosa and Staphylococcus aureus during respiratory infections. Thorax. 74, 684-692 (2019).
- Gabrilska, R. A., Rumbaugh, K. P. Biofilm models of polymicrobial infection. Future Microbiology. 10 (12), 1997-2015 (2015).
- Nobile, C. J., Mitchell, A. P. Microbial biofilms: e pluribus unum. Current Biology. 17 (10), 349-353 (2007).
- Marino, P. J., et al. Community analysis of dental plaque and endotracheal tube biofilms from mechanically ventilated patients. Journal of Critical Care. 39, 149-155 (2017).
- Frank, D. N., et al. Microbial diversity in chronic open wounds. Wound Repair and Regeneration. 17, 163-172 (2009).
- Fazli, M., et al. Nonrandom distribution of Pseudomonas aeruginosa and Staphylococcus aureus in chronic wounds. Journal of Clinical Microbiology. 47 (12), 4084-4089 (2009).
- Shimizu, K., et al. Pathogens in COPD exacerbations identified by comprehensive real-time PCR plus older methods. International Journal of Chronic Obstructive Pulmonary Disease. 10, 2009-2016 (2015).
- Behnia, M., Logan, S. C., Fallen, L., Catalano, P. Nosocomial and ventilator-associated pneumonia in a community hospital intensive care unit: a retrospective review and analysis. BMC Research Notes. 7, 232 (2014).
- Maliniak, M. L., Stecenko, A. A., McCarty, N. A. A longitudinal analysis of chronic MRSA and Pseudomonas aeruginosa co-infection in cystic fibrosis: a single-center study. Journal of Cystic Fibrosis. 15 (3), 350-356 (2016).
- Limoli, D. H., et al. Staphylococcus aureus and Pseudomonas aeruginosa co-infection is associated with cystic fibrosis-related diabetes and poor clinical outcomes. European Journal of Clinical Microbiology & Infectious Diseases. 35 (6), 947-953 (2016).
- Hotterbeekx, A., Kumar-Singh, S., Goossens, H., Malhotra-Kumar, S. In vivo and in vitro interactions between Pseudomonas aeruginosa and Staphylococcus spp. Frontiers in Cellular and Infection Microbiology. 7 (106), (2017).
- Smith, K., et al. Aspergillus fumigatus enhances elastase production in Pseudomonas aeruginosa co-cultures. Medical Mycology. 53, 645-655 (2015).
- Michelson, C. F., et al. Staphylococcus aureus alters growth activity, autolysis, and antibiotic tolerance in a human host-adapted Pseudomonas aeruginosa lineage. Journal of Bacteriology. 196 (22), 3903-3911 (2014).
- Ngamdee, W., et al. Competition between Burkholderia pseudomallei and B. thailandensis. BMC Microbiology. 15, 56 (2015).
- Heir, E., Møretrø, T., Simessen, A., Langsrud, S. Listeria monocytogenes strains show larger variations in competitive growth in mixed culture biofilms and suspensions with bacteria from food processing environments. International Journal of Food Microbiology. 275, 46-55 (2018).
- Lutz, C., Thomas, T., Steinberg, P., Kjelleberg, S., Egan, S. Effect of interspecific competition on trait variation in Phaeobacter inhibens biofilms. Environmental Microbiology. 18 (5), 1635-1645 (2016).
- Meisner, J., et al. FtsEX is required for CwlO peptidoglycan hydrolase activity during cell wall elongation in Bacillus subtilis. Molecular Microbiology. 89 (6), 1069-1083 (2013).
- Coates, J., et al. Antibiotic-induced population fluctuations and stochastic clearance of bacteria. eLife. 7, 32976 (2018).
- Korber, D. R., Lawrence, J. R., Sutton, B., Caldwell, D. E. Effect of laminar flow velocity on the kinetics of surface recolonization by Mot+ and Mot- Pseudomonas fluorescens. Microbial Ecology. 18, 1-19 (1989).
- Lawrence, J. R., Korber, D. R., Caldwell, D. E. Behavioral analysis of Vibrio parahaemolyticus variants in high- and low- viscosity microenvironments by use of digital image processing. Journal of Bacteriology. 174 (17), 5732-5739 (1992).
- Lawrence, J. R., Wolfaardt, G. M., Korber, D. R. Determination of diffusion coefficients in biofilms by confocal laser microscopy. Applied and Environmental Microbiology. 60 (4), 1166-1173 (1994).
- Limoli, D. H., et al. Interspecies interactions induce exploratory motility in Pseudomonas aeruginosa. eLife. 8, 47365 (2019).
- Burrows, L. Pseudomonas aeruginosa twitching motility: type IV pili in action. Annual Review of Microbiology. 66 (1), 493-520 (2012).
- Lee, C. K., et al. Multigenerational memory and adaptive adhesion in early bacterial biofilm communities. PNAS. 115 (17), 4471-4476 (2018).
- Tolosa, A., et al. Enhanced field-of-view integral imaging display using multi-Köhler illumination. Optical Society of America. 22 (26), 31853-31863 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados