Method Article
Visualização e Análise de Artérias ariníngeos usando Imunohistoquímica de montagem integral e Reconstrução 3D
Neste Artigo
Resumo
Aqui, descrevemos um protocolo para visualizar e analisar as artérias do arco faríngeo 3, 4 e 6 dos embriões de camundongos usando imunofluorescência de montagem total, limpeza de tecidos, microscopia confocal e reconstrução 3D.
Resumo
A formação inadequada ou a remodelação das artérias do arco faríngeo (PAAs) 3, 4 e 6 contribuem para algumas das formas mais graves de doença cardíaca congênita. Para estudar a formação de PAAs, desenvolvemos um protocolo utilizando imunofluorescência de montagem completa juntamente com a limpeza de tecido de álcool benzílico/benzoato de benzílico (BABB) e microscopia confocal. Isso permite a visualização do endotélio do arco faríngeo em uma resolução celular fina, bem como a conectividade 3D da vasculatura. Usando software, estabelecemos um protocolo para quantificar o número de células endoteliais (CEIs) em PAAs, bem como o número de CE dentro do plexo vascular em torno dos PAAs dentro dos arcos faríngea3, 4 e 6. Quando aplicada a todo o embrião, essa metodologia proporciona uma visualização abrangente e análise quantitativa da vasculatura embrionária.
Introdução
Durante a embriogênese do camundongo, as artérias arqueína faríngeas (PAAs) surgem como pares bilaterais simétricos de artérias que conectam o coração com a aortae dorsal1. À medida que o embrião se desenvolve, o primeiro e o segundo pares de PAAs regredem, enquanto os3º,4ºe6º PAAs passam por uma série de eventos de remodelação assimétrica para formar as artérias arqueoréticas2.
Os PAAs 3, 4 e 6 desenvolvem-se através da vasculogênese, que é a formação de novo dos vasos sanguíneos3. Defeitos na formação ou remodelação dessas artérias arqueais dão origem a vários defeitos cardíacos congênitos, como os observados em pacientes com Síndrome de DiGeorge4,5. Portanto, a compreensão de mecanismos que regulam o desenvolvimento de PAAs pode levar a uma melhor compreensão da etiologia da doença cardíaca congênita (CHD).
As abordagens atuais para visualizar e analisar o desenvolvimento do PAA incluem imunofluorescência de seções teciduais, moldes vasculares, injeção de tinta índia, microscopia episcópica de alta resolução e/ou imunohistoquímica de montagem total1,,4,,5,6,7. Aqui, descrevemos um protocolo que combina imunofluorescência total, microscopia confocal e renderização de imagem 3D, a fim de coletar, analisar e quantificar dados volumétricas, conectividade vascular e identidade celular. Além disso, detalhamos um método para compartimentar e quantificar os números de CE em cada arco faríngeo como forma de estudar a formação do plexo vascular do arco faríngeo e sua remodelação nos PAAs. Embora este protocolo seja projetado para analisar o desenvolvimento do PAA, ele pode ser usado para analisar outras redes vasculares em desenvolvimento.
Protocolo
O uso e os procedimentos de uso de animais foram aprovados pelo Comitê de Cuidados e Uso de Animais Institucionais da Universidade Rutgers.
1. Elaboração de soluções
- Prepare 1 L de solução salina tamponada com 0,1% Triton-X-100 (PBST) e esterilização do filtro. Esta solução pode ser armazenada à temperatura ambiente (RT) por pelo menos um ano.
- Prepare 600 μL de tampão de bloqueio composto por 10% do soro de burro normal em PBST. Faça esta solução fresca cada vez.
- Prepare 50 mL das seguintes diluições de metanol (MeOH) em uma capa de fluxo: 25% MeOH em água deionizada (dH2O), 50% MeOH em dH2O e 75% MeOH em dH2O. Vortex para misturar. Loja na RT.
- Prepare 50 mL das seguintes soluções de benzoilal-benzoato (BABB) em tubos cônicos de 50 mL.
- Para 100% BABB, adicione 32 mL de benzoato de bentil a 16 mL de álcool benzílico (2:1 volume por relação de volume).
- Para 50% BABB, adicione 16 mL de benzoileato e 8 mL de álcool benzílico a 24 mL de MeOH.
- Cubra tubos cônicos em papel alumínio para proteger da luz. Essas soluções podem ser armazenadas no RT por até um ano.
ATENÇÃO: Babb é tóxico e corrosivo. Deve ser manuseado e descartado de acordo com o MSDS.
2. Dissecção e fixação de embriões
NOTA: Este protocolo é adequado para embriões de rato E9.5 e E10.5 (masculino ou feminino) isolados de qualquer cepa de rato. Para embriões mais jovens e mais velhos, os tempos de incubação devem ser experimentalmente determinados para maximizar a relação de sinal para ruído do sinal de fluorescência.
- Encha um de 35 mm e outro de 60 mm de placas de Petri com 1x PBS e coloque no gelo até que seja necessário.
- Eutanie um rato grávida por inalação de CO2. Realizar a luxação cervical como medida secundária de eutanásia.
- Limpe a área abdominal da represa com 70% de etanol. Aperte a área abdominal usando fórceps e faça uma incisão semelhante a V usando uma tesoura cirúrgica a partir da base da parede abdominal na linha média; continuar a abrir a cavidade torácica. Levante o tecido abdominal e mova os intestinos para o lado para expor os chifres uterinos.
- Faça um corte na base do canal vaginal, e com fórceps, puxe o útero para longe da represa. Faça um corte adicional em cada ovário para libertar o útero. Transfira o útero para uma das placas de Petri de 60 mm contendo 1x PBS frio.
- Usando uma tesoura reta, corte a parede uterina entre cada local de implantação. Pegue um decidua com uma pipeta de vidro e transfira para a placa de Petri de 35 mm com 1x PBS. um microscópio de dissecção, insira uma tesoura reta no espaço entre a decidua e a parede uterina. Corte e remova a parede uterina.
- Com fórceps finos, remova as membranas de decidua e reichert do embrião fazendo cuidadosamente incisões transversais ao longo do tecido e puxando o tecido para longe do saco de gema. Remova o saco de gema e o saco amniótico puxando cuidadosamente o tecido para longe do embrião e fazendo cortes na allantois e veia umbilical.
NOTA: Sacos de gema podem ser usados para genotipar embriões. - Transfira cada embrião com uma tubulação de vidro para tubos individuais de 2 mL preenchidos com 1 mL de 1x PBS. Rotular cada tubo com um identificador único.
- Para corrigir embriões, remova cuidadosamente o 1x PBS e adicione a solução de 4% de paraformaldeído (PFA) em 1x PBS. Incubar a 4 °C com agitação suave durante a noite.
NOTA: A fixação de 4% de PFA é adequada para os anticorpos mencionados neste protocolo. No entanto, os procedimentos de fixação devem ser otimizados para anticorpos adicionais.
3. Coloração de embriões
NOTA: Nesta seção, os embriões são permeabilizados e manchados com anticorpos primários e secundários. Como o desenvolvimento do PAA prossegue rapidamente, as diferenças no estágio embrionário afetarão muito a análise a jusante. Portanto, os embriões devem ser compatíveis com a idade, contando cuidadosamente somitas para combinar com o controle e pares mutantes antes de outras manipulações.
- Para lavar embriões, remova cuidadosamente 4% de PFA e adicione 1x PBS. Inverta suavemente o tubo várias vezes. Coloque o(s) lado direito do lado direito para cima e deixe o embrião afundar. Repita a lavagem 3 vezes. Coloque os tubos com embriões no gelo.
NOTA: (Ponto de parada opcional) Após as lavadas, os embriões podem ser desidratados em séries classificadas de MeOH por 30 minutos por diluição, como na seção 1.3, e armazenados a -20 °C em 100% MeOH para uso posterior por até 6 meses. - Para embriões E10.5, use uma pipeta de vidro para transferir um embrião para uma placa de Petri de 35 mm cheia de 1x PBS refrigerado. Aperte cuidadosamente o embrião logo acima do membro traseiro com fórceps finos e faça um corte transversal para remover a metade posterior do embrião. Isso permite que o embrião fique deitado em uma posição sagital para a etapa 4.2. Coloque o embrião de volta no tubo de 2 mL com PBS fresco 1x.
NOTA: Um embrião de controle e mutante pode ser emparelhado e manchado com a mesma solução de anticorpos em um tubo, para as etapas 3.3 a 3.8.- Se colorir dois embriões juntos, corte a cabeça de um embrião acima do primeiro arco faríngeo, beliscando com fórceps finos para fazer um corte transversal. Isso distinguirá embriões de dois genótipos diferentes dentro de cada tubo.
- Para permeabilizar os embriões, pipeta 1x PBS do tubo, tomando cuidado para não tocar no embrião(s). Adicione 1 mL de PBST. Coloque o tubo a 4 °C com agitação suave durante a noite.
NOTA: (Ponto de parada opcional) Os embriões podem ser mantidos na solução PBST a 4 °C por vários dias. - Para evitar a ligação não específica de anticorpos, primeiro remova o PBST do tubo, tomando cuidado para não tocar no embrião( s). Adicione 600 μL de solução de tampão de bloqueio aos embriões. Bloqueie os embriões a 4 °C com agitação suave durante a noite.
NOTA: A solução de bloqueio precisa ser girada em alta velocidade em uma centrífuga de bancada imediatamente antes de ser usada para remover detritos. - Para manchar e quantificar as CE, utilize anticorpos contra VEGFR2 e ERG. Soluções de anticorpos são feitas no buffer de bloqueio. O anticorpo anti-VEGFR2 é diluído 1:200 e o anticorpo ERG é diluído 1:1000.
NOTA: As soluções de anticorpos precisam ser giradas em alta velocidade em uma centrífuga de bancada imediatamente antes de serem usados para remover partículas.- Para incubar embriões com anticorpos primários, remova a solução tampão de bloqueio do tubo, tomando cuidado para não tocar nos embriões. Adicione 600 μL de solução primária de anticorpos a cada tubo. Incubar embriões a 4 °C com agitação suave durante 4-5 dias.
- Para lavar os embriões da solução de anticorpos, primeiro remova a solução primária de anticorpos do tubo. Lave embriões a cada hora com 1 mL de PBST à temperatura ambiente (RT) com agitação suave. Lave os embriões 4-5 vezes durante o dia e depois incuba a 4 °C com agitação suave durante a noite. Repita as lavadas no dia seguinte.
- Faça soluções secundárias de anticorpos diluindo o anti-cabra Alexa Fluor 488 e anti-mouse Alexa Fluor 555 1:300 no buffer de bloqueio. Diluir o estoque DAPI 1:1000 no buffer de bloqueio.
NOTA: As soluções de anticorpos devem ser giradas em alta velocidade em uma centrífuga de bancada imediatamente antes de serem usadas para remover partículas. Além disso, outros corantes Alexa Fluor podem ser usados em vez de 488 ou 555.- Para incubar os embriões com anticorpos secundários, remova o PBST do tubo. Adicione 600 μL de solução de anticorpos secundários a cada tubo. Incubar embriões a 4 °C com agitação suave durante 4-5 dias.
- Para lavar os embriões da solução de anticorpos, primeiro remova a solução de anticorpos secundários do tubo. Lave os embriões a cada hora com 1 mL de PBST em RT com agitação suave. Lave os embriões 4-5 vezes durante o dia e depois incuba a 4 °C com agitação suave durante a noite. Repita as lavadas no dia seguinte.
4. Incorporando embriões em agarose
NOTA: Na seção 4, os embriões serão incorporados em agarose. Este processo de incorporação serve a dois propósitos: orientar adequadamente o embrião antes da imagem e auxiliar na localização do embrião após ter sido limpo em BABB (etapas 5.2.2 - 5.3.2).
- Prepare 200 mL de solução de agarose de 1%, adicionando 2 g de agarose a 200 mL de dH2O. Microondas até que toda a agarose seja dissolvida.
NOTA: A agarose restante pode ser armazenada a 4 °C e reaquecida para usoposterior. - Usando um molde de parafina de plástico e pipeta de vidro, transfira suavemente um embrião para o molde. Remova cuidadosamente o PBST do embrião. Coloque o embrião em uma posição sagital. Rapidamente, adicione cerca de 0,5 mL de agarose quente ao molde - apenas o suficiente para cobrir o embrião e preencher o molde. Certifique-se de que não há bolhas de ar cercando o embrião.
- Coloque o molde no gelo e cubra com papel alumínio até que a agarose se solidificou.
NOTA: Não permita que o embrião seque após a remoção do PBST. A solução de agarose deve ser quente o suficiente para permanecer líquida quando está sendo adicionada ao embrião. Adicione apenas agarose suficiente para cobrir o embrião, mas não muito, caso contrário será difícil de imaginar. A profundidade da imagem é determinada em parte pela distância de trabalho do objetivo.
5. Desidratação e limpeza de tecidos
NOTA: Nesta seção, os embriões são desidratados utilizando séries de metanol, depois limpos no solvente orgânico, BABB, e montados entre dois deslizamentos separados por um espaçador de borracha; neste protocolo, são utilizados espaçadores de borracha Fast Well. O para-choque Fast Well tem uma superfície adesivo de dupla lateral. O espaçador é necessário para criar um poço, no qual o embrião será colocado e mantido entre dois deslizamentos de cobertura.
-
Desidratação do metanol
- Rotular novos tubos de 2 mL, um por embrião. Adicione 1 mL de 25% de MeOH por tubo.
- Usando um bisturi limpo, corte suavemente a agarose ao redor do embrião, deixando o suficiente ao redor do embrião para que possa ser recolhido por fórceps. Use fórceps finos para pegar suavemente a agarose com o embrião incorporado e colocá-lo no tubo rotulado com 25% de MeOH. Não permita que os fórceps toquem no embrião.
- Incubar embriões em RT com agitação suave por 1 hora no escuro.
- Remova 25% de MeOH do tubo, tomando cuidado para não tocar no embrião. Adicione 1 mL de 50% de MeOH por tubo. Incubar no RT com agitação suave por 1 hora no escuro.
- Remova 50% de MeOH do tubo, tomando cuidado para não tocar no embrião. Adicione 1 mL de 75% de MeOH por tubo. Incubar no RT com agitação suave por 1 hora no escuro.
- Remova 75% de MeOH do tubo, tomando cuidado para não tocar no embrião. Adicione 1 mL de 100% De MeOH por tubo. Incubar no RT com agitação suave por 1 hora no escuro. Repita 100% MeOH lavar duas vezes.
-
Limpando com BABB
- Remova 100% MeOH do tubo, tomando cuidado para não tocar no embrião. Adicione 1 mL de 50% BABB por tubo. Incubar no RT com agitação suave por 1 hora no escuro.
- Remova 50% BABB do tubo, tomando cuidado para não tocar no embrião. Adicione 1 mL de 100% BABB por tubo. Incubar no RT com agitação suave por 1 hora no escuro. Repita 100% BABB lavar duas vezes.
NOTA: (Ponto de parada opcional) Os embriões podem permanecer em 100% BABB em tubos por cerca de uma semana. O armazenamento mais longo fará com que o BABB dissolva o plástico dos tubos.
-
Montagem de embriões para imagem
- Coloque um pára-choque fast well em um deslizamento de tampa de vidro de 24 mm x 60 mm #1,5, descascando o adesivo plástico de um lado. Certifique-se de que não há bolhas de ar entre o deslizamento de cobertura e o pára-choque, aplicando pressão suave no adesivo de plástico em cima do pára-choque de borracha. Rotule o deslizamento de cobertura de acordo com o número do embrião, genótipo e anticorpos usados para coloração.
NOTA: Qualquer espaçador pode ser colocado entre as tampas, desde que seja espesso o suficiente para evitar esmagar ou esmagar um embrião. Usamos espaçadores Fast Well devido à sua espessura e conveniência, que inclui superfícies adesivos em ambos os lados do espaçador para fixá-lo para tampar. - Retire cuidadosamente o tubo e descarte o BABB 100% do tubo. Depois de visualizar o embrião embutido em agarose no tubo, use fórceps finos para pegar a agarose e cuidadosamente transferir o embrião para o deslizamento de cobertura dentro do Poço Rápido - não permita que os fórceps toquem no embrião.
- Retire o segundo adesivo de plástico do para-choque e coloque o segundo deslizamento de cobertura em cima. Remova as bolhas de ar pressionando suavemente a tampa. Tenha cuidado para não quebrar o vidro.
NOTA: As amostras podem ser armazenadas planas em um suporte de slides no escuro em RT por até um ano se o selo estiver apertado.
- Coloque um pára-choque fast well em um deslizamento de tampa de vidro de 24 mm x 60 mm #1,5, descascando o adesivo plástico de um lado. Certifique-se de que não há bolhas de ar entre o deslizamento de cobertura e o pára-choque, aplicando pressão suave no adesivo de plástico em cima do pára-choque de borracha. Rotule o deslizamento de cobertura de acordo com o número do embrião, genótipo e anticorpos usados para coloração.
6. Aquisição de dados
NOTA: Nas etapas seguintes, o endotélio dos arcos faríngeas 3, 4 e 6 será imagem do microscopia confocal.
-
Posicionamento de slides no estágio do microscópio
- Para imagem embriões, utilize um microscópio confocal equipado com um objetivo de imersão de água de 20x, abertura numérica 0,95, distância de trabalho de 0,95 mm e o software NIS-Elements AR 5.11.01 de 64 bits.
- Usando fluorescência de campo largo, localize visualmente os arcos faríngeas. Centralize a visão de campo em torno do4º PAA.
- Se o campo de visão do objetivo não capturar toda a área do arco faríngeo, pegue e costure um grande painel de imagens com sobreposição de 1%. Para evitar o movimento da amostra durante a aquisição da imagem grande, fixe suavemente o conjunto de deslizamento de cobertura ao palco usando argila de moldagem.
-
Definição de parâmetros de aquisição
- Defina o tamanho do pinhole para 1.0.
- Na guia Nd Acquisition, defina os limites superior e inferior da imagem usando o ajuste grosseiro. Defina o tamanho da etapa Z de acordo com as especificações do software. Determine a espessura que pode ser imagem da distância de trabalho do objetivo e da clareza da amostra.
- Devido à espessura do embrião, ajuste o ganho ao longo da pilha Z. Defina a intensidade do laser e ganhe no meio da pilha Z para cada canal (405, 488 e 555) e atribua valores a guia Correção de Intensidade Z. Defina os mesmos valores para a fatia inferior.
- Role pelo embrião até que os sinais de fluorescência comecem a parecer mais fracos. Aumente o ganho de cada canal até que a intensidade do sinal pareça semelhante ao segmento anterior. Atribua o novo valor na guia Correção de Intensidade Z. Repita até que a pilha z esteja concluída. Configurações de importação de volta à Aquisição de ND.
- Executar varredura usando a opção Correção de Execução Z.
7. Análise usando o software Imaris
NOTA: Nestas etapas, as imagens confocais serão analisadas utilizando-se o software de análise de imagens de microscopia, versão 9.2.0 do Imaris. Durante esta análise, selecionaremos primeiro regiões de interesse a serem analisadas pela criação de superfícies. Em seguida, usaremos a função Máscara para separar visualmente essas regiões. Finalmente, usaremos a função Spot para quantificar o número de CE dentro de cada região de interesse.
- Dependendo do software de imagem usado na etapa 6, converta imagens para .ims usando Imaris File Converter.
- Abra os arquivos .ims. Defina a imagem para Ortogonal em Camera/Labels | Painel tipo câmera.
- Localize os PAAs e oriente a imagem para o aparecimento.
NOTA: Quando os arquivos forem abertos pela primeira vez, eles aparecerão como uma compilação 3D de todas as fatias imagemdas. Nesta etapa, os PAAs serão localizados fazendo a imagem 3D em uma imagem 2D. A imagem 2D permite, então, que os PAAs sejam devidamente orientados para análise.- No painel Propriedades, desligue o volume. No painel Propriedades, clique em Adicionar novo orto slicer. Definir orientação de fatia para o plano XY. Use a posição de fatia para percorrer a imagem até encontrar os PAAs.
- Se os PAAs não forem paralelos à parte superior e inferior da imagem, gire livremente a imagem usando o cursor do mouse para que os PAAs correm da esquerda para a direita através da tela. No menu suspenso processamento de imagem, selecione 'Girar livre' e clique em OK.
- Surgindo o 3º Arco Faríngeo (Figura 2A, B - B")
NOTA: Nestas etapas, os arcos faríngeos e paas serão rastreados usando a ferramenta Superfície para gerar uma região de interesse 'superficial'. Isso permitirá que cada região de interesse seja visualmente isolada do tecido circundante. Aqui descrevemos os passos à superfície e analisamos osrd componentes endoteliais do arco 3 faríngeo. Os arcos faríngeas 4 e 6 são analisados da mesma forma.- Para superfície do endotélio em todo o arco3º faríngeo, clique no botão Adicionar nova superfície localizado o painel Propriedades. Clique duas vezes na Superfície 1 e renomeie a nova superfície para"3º Arco Faríngeo".
- Selecione Pular criação automática, editar manualmente. Definir orientação de superfície para o plano YZ (orientação coronal). Use a posição de fatia para colocar o plano de superfície do arco 3da faríngea ao lugar onde o3º PAA e a Aorta Dorsal se conectam.
- Gire a imagem de modo que oplano de superfície do arco 3 da faríngea esteja à vista. Desligue o Ortho Slicer 1.
- o Sorteio | Contorno | Guia de modo, selecione a função Modo de desenho de distância. Ajuste as configurações dos parâmetros, se necessário. Manter parâmetros de superfície consistentes entre as amostras. Para este exemplo, o espaçamento do Vértice é de 10 μm.
- Para começar a aparecer, pressione a tecla Esc e clique no botão Desenhar. Rastreie o perímetro do arco 3º faríngeo com o cursor do rato. Use a posição de fatia para mover de 10 a 25 fatias. Rastreie o perímetro do arco faríngeo. Repita até que o arco faríngeo esteja totalmente traçado.
- Para gerar a superfície da região traced, selecione o botão Criar superfície no painel Propriedades.
- Surgindo o 3º PAA (Figura 2C -C")
- Para superfície do endotélio do3º PAA, primeiro desligue a região superficial da etapa 7.4, desssincando a caixa de superfície do Arco 3da Faringe. Em seguida, clique no botão Adicionar nova superfície novamente. Clique duas vezes na Superfície 1 e renomeie a nova superfície para "3rd PAA".
- Selecione Pular criação automática, editar manualmente. Definir orientação de superfície para o plano YZ (orientação coronal). Use a posição de fatia para colocar o plano de superfície 3rd PAA ao lugar onde o3º PAA e a Aorta Dorsal se conectam e, em seguida, repita os passos 7.4.
- Mascaramento de estruturas superficiais
NOTA: Nas etapas seguintes, cada região de interesse será mascarada. O mascaramento permite que a região de interesse seja visualmente distinta do resto do tecido imagemlinso e permite a quantificação dessas distintas estruturas de interesse. Abaixo, descrevemos os passos em Imaris para visualizar e analisar o endotélio PAA, bem como o plexo - a vasculatura menor em torno dos PAAs dentro dos arcos faríngeos. Nestas etapas, a superfície3ª PAA será mascarada para visualizar e realizar análises somente nos PAAs usando a função Spot descrita na Seção 7.7.- Selecione Volume para visualizar todos os canais mascarados. Pressione a tecla Esc para girar a imagem e posicionar a imagem em uma posição XY.
- Na guia Editar, selecione Seleção de Máscaras para o3º PAA. Selecione o canal DAPI e clique em OK. Repita para os demais canais.
- No teclado, pressione Ctrl + D para visualizar o painel Ajustes de exibição. Selecione cada novo canal e renomeie-os para deixar claro o que cada canal mostra. Por exemplo, isso levará a três novos canais: "3rd PAA DAPI", "3rd PAA ERG" e "3rd PAA VEGFR2".
NOTA: Nas etapas 7.6.4-7.6.7 criaremos canais apenas para plexo do arco faríngeo. - Para visualizar o plexo endotelial separadamente do PAA, primeiro selecionaremos A Seleção de Máscaras para a superfície 3ª PAA. Selecione o canal DAPI. Desmarcar Selecione voxels externos à superfície e verifique Selecione voxels dentro da superfície para botões, defina Os voxels selecionados dentro da superfície para zero. Clique em OK.
- Repita para os demais canais. Esta operação excluirá a região que contém o PAA dos novos canais mascarados. Renomeie os canais para deixar claro o que cada canal mostra. Por exemplo, isso levará a três novos canais: "NÃO-PAA DAPI", "Non-PAA ERG" e "Non-PAA VEGFR2".
- Para visualizar o plexo endotelial dentro do arcofaríngeo 3, selecione Seleção de Máscaras na guia Editar, para a superfície do Arco3 faríngeo. Selecione o canal DAPI não-PAA. Clique em OK.
- Repita para os demais canais não-PAA. Renomeie os canais para deixar claro o que cada canal mostra. Por exemplo, isso levará a três novos canais: "Plexus DAPI", "Plexus ERG" e "Plexus VEGFR2".
- Quantificação dos números da CE
NOTA: A expressão dos núcleos erg marca núcleos endoteliais tornando conveniente quantificar os números da CE. Nestas etapas, o número de CeS será quantificado usando a função Spot para gerar um ponto para cada CE marcado pela expressão ERG no PAA e plexo. Na seção 7.7, serão geradas manchas para cada célula ERG positiva no PAA mascarado, seguida da desseleção de manchas em células ERG-positivas, VEGFR2-negativas.- No teclado, pressione Ctrl + D para visualizar o painel Ajustes de exibição. Desligue todos os canais, exceto paa erg.
- Na guia Propriedades, clique no botão Adicionar novos pontos. Clique em Pontos 1 e renomeie-o para "PAA Total Number of ECs". Clique no botão de seta azul. Para o Canal fonte,selecione o canal PAA ERG. Ajuste o diâmetro XY estimado para 4 μm. Vá para o próximo painel clicando no botão de seta azul.
- Ajuste o número de pontos vistos usando a escala deslizante, para garantir que cada núcleo CE (marcado pela expressão ERG) seja representado por um ponto. Clique no botão de seta dupla verde.
- Desligue o canal PAA ERG no ajuste de exibição. Ligue o canal PAA VEGFR2 para visualizar o endotélio PAA.
- Para quantificar com precisão o número de CE, garantimos que cada ponto expresse os marcadores CE, ERG e VEGFR2. Para fazer isso, selecione Superfície de Objeto na guia Editar | Painel Adicionar/Excluir. Pressione a tecla Esc e exclua quaisquer pontos que não sejam VEGFR2 positivos, segurando o shift e selecionando o ponto.
NOTA: Na etapa seguinte, serão geradas manchas para cada célula ERG-positiva no plexo mascarado, seguida seleção de manchas em células ERG-positivas, VEGFR2-negativas. - Na guia Propriedades, clique no botão Adicionar novos pontos. Selecione Pontos 1 e renomeie para "Plexus Total Number of ECs". Clique no botão de seta azul. Para o Canal fonte, selecione o canal Plexus ERG. Ajuste o diâmetro XY estimado para 4 μm. Repita as etapas 7.7.1 - 7.7.5 para plexus número total de ECs.
- Clique na guia Estatísticas de cada função spot para determinar o número total de CeS no3º PAA e no plexo do arco faríngeo.
- Repita as etapas 7.7.1 - 7.7.6 para os PAAs restantes e plexo do arco faríngeo.
Resultados
O protocolo de imunofluorescência de montagem completa aqui apresentado produz resultados claros e limpos, permitindo a reconstrução 3D do endotélio do arco faríngeo, como visto na Figura 1A. É importante incubar embriões por tempo suficiente em cada solução de anticorpos para garantir a penetração completa através da amostra, bem como, lavar completamente os embriões após a incubação de anticorpos. Na Figura 1B,pontos grandes e brilhantes aparecem como resultado de partículas nas soluções de anticorpos ou de tampão de bloqueio. Descobrimos que centrifugar cada solução antes do uso e períodos mais longos de lavadas pbst após cada incubação de anticorpos resolve esse problema.
A Figura 2 ilustra o processo utilizado para a superfície de um arco faríngeo e um PAA para análise conforme descrito na seção 7 do protocolo. Usando a função mascarada, o software Imaris permite que regiões superficiais sejam visualmente separadas e analisadas independentemente.
A Figura 3 demonstra o mascaramento individual de diferentes compartimentos vasculares nos arcos faríngeos: o PAA (Figura 3A, B, C) e o plexo(Figura 3A', B', C'). O mascaramento permite a análise e quantificação dos números da CE em cada estrutura separadamente. Na Figura 3C-C', o recurso Spot é usado para quantificar o número total de CeS tanto no PAA quanto no plexo, atribuindo um único ponto para cada núcleo que expressa erg. É importante notar que o algoritmo usado para a função Spot foi projetado para gerar um ponto para qualquer pixel de um tamanho especificado. Erg, que é usado aqui como marcador de núcleos CE, também é expresso em células de crista neural8; células de crista neural não expressam VEGFR2. A Figura 3D ilustra um exemplo de um ponto ERG-positivo (verde), VEGFR2-negativo (rosa) que foi gerado pela função Imaris Spot. Como resultado, é essencial verificar se cada ponto representa uma única CE e é rotulado com erg e VEGFR2.
| Passo | Tempo | Temperatura | |
| 1 | Lavagem/Permeabilização do PBST | 24 h ou O/N | 4 °C |
| 2 | Tampão de bloqueio | 25 h ou O/N | 4 °C |
| 3 | Anticorpo Primário | 4-5 dias | 4 °C |
| 4 | Lavagem PBST | 4-5 vezes por dia durante 2 dias | RT (ou 4 °C se O/N) |
| 5 | Anticorpo Secundário | 4-5 dias | 4 °C |
| 6 | Lavagem PBST | 4-5 vezes por dia durante 2 dias | RT (ou 4 °C se O/N) |
| 7 | Incorporar | N/A | Rt |
| 8 | Desidratação de Metanol e BABB | 1 hora por etapa | Rt |
Tabela 1: Visão geral de todo o protocolo de imunofluorescência do monte. O/N - durante a noite; RT - temperatura ambiente.
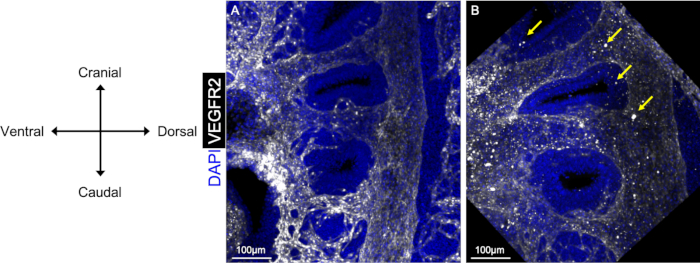
Figura 1: Comparação de imagens limpas e sujas após a imunofluorescência de montagem total. As visões sagitais do embrião E10.5 mostram o uso de anticorpo anti-VEGFR2 (branco) para visualizar o endotélio PAA. Os embriões completamente lavados com incubações de anticorpos post PBST(A)têm uma maior relação sinal-ruído e produzem uma imagem mais limpa, quando comparados com embriões que não são completamente lavados(B). As setas em B mostram áreas de ruído/sujeira que apareceram na imagem quando um embrião não é completamente lavado ou a solução de anticorpos não foi centrifugada. Clique aqui para ver uma versão maior desta figura.

Figura 2: Superfície do arco faríngeo (PA) e PAA. Uma visão sagital 2D (A) é usada para identificar a localização dos PAAs na imagem confocal. Um ortocortador de ortocoroa(A, linha amarela) é colocado através dos PAAs. O arco faríngeo(B)e paa(C)são então superfícies na orientação coronal usando a ferramenta Desenho de Distância em Imaris. A ferramenta Desenho de Distância, definida como 10 μm, é usada para traçar o perímetro do arco3º faríngeo(B)ou do PAA(C). Os contornos são desenhados a cada 10-25 fatias através de todo o arco(B', C'). Os contornos são combinados para gerar uma superfície 3D do arco faríngeo(B"ou do PAA(C). Clique aqui para ver uma versão maior desta figura.
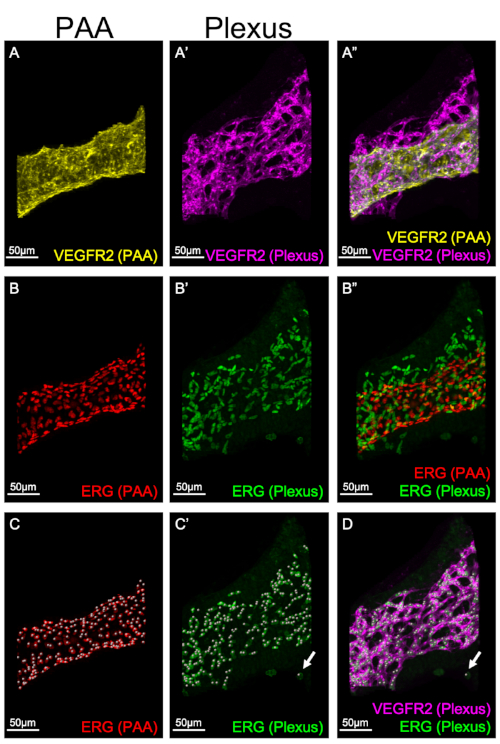
Figura 3: Quantificação de números CE em um PAA e um plexo. Reconstruções 3D são usadas para visualizar a estrutura e a expressão dos marcadores CE em um PAA ou em um plexo CE separadamente. Os painéis A-A' mostram a expressão de VEGFR2 no PAA (A, amarelo) e no plexo(A', rosa). O painel A" ilustra uma mesclagem da expressão PAA e plexus VEGFR2. Os painéis B-B' mostram a expressão do ERG no PAA(B, vermelho) e no plexo(B', verde). O painel B" ilustra a fusão do PAA e do plexo. C-C'. A função Spot em Imaris é usada para quantificar o número de CeS no PAA ou plexo. Cada célula ERG-positiva no PAA(C, vermelho) ou plexo(C', verde)é atribuída a um único ponto para marcar uma única CE. A seta em C'-D mostra um exemplo erg-positivo, vegfr2-negativo no plexo que foi gerado pela função Imaris Spot. Este ponto está excluído da quantificação. Clique aqui para ver uma versão maior desta figura.
Discussão
A capacidade de visualizar o endotélio em embriões de camundongos em 3D forneceu novas percepções sobre seu desenvolvimento3. Aqui apresentamos um protocolo que permite imagens 3D de alta resolução de embriões, visualização da conectividade vascular e análises quantitativas da formação de PAA. Este protocolo pode ser empregado para ver como alterações genéticas ou insultos ambientais afetam o desenvolvimento do PAA. O procedimento aqui relatado utiliza anticorpos contra VEGFR2 e ERG para visualizar a formação de PAA e quantificar o número da CE; no entanto, anticorpos adicionais podem ser usados para visualizar e analisar outros aspectos do desenvolvimento da artéria da arqueada, como o recrutamento da crista neural ou a diferenciação de células musculares lisas. Se este procedimento for utilizado em estágios iniciais de embriogênese, é importante notar que alguns antígenos (por exemplo, ERG) detectados neste protocolo podem ainda não ser expressos. Outras manchas nucleares, como DAPI ou DRAQ5 ou rotulagem de linhagem com rastreadores com etiquetanuclear, podem ser usadas para quantificar o número da CE.
Existem várias etapas críticas dentro do protocolo: garantir que 1) embriões não fiquem dessecados entre as mudanças de solução; 2) os embriões são completamente lavados após incubações de anticorpos; e 3) que os embriões estão completamente desidratados com MeOH antes de limpar os tecidos com BABB.
As lavadas de metanol antes da limpeza tecidual servem a dois propósitos: eliminar a fluorescência devido à expressão de proteínas fluorescentes (por exemplo, a expressão de EGFP ou tdTomato usado para rastreamento de linhagem) no embrião, e desidratar o tecido. A eliminação da fluorescência de proteínas fluorescentes permite o uso de qualquer combinação de fluoróforos para imagem. Anticorpos contra EGFP e TdTomato (cereja) podem ser usados para visualizar a expressão dessas proteínas fluorescentes. Alternativamente, o MeOH pode ser substituído por tetrahidrofurano para preservar a fluorescência das proteínas fluorescentes9.
Descobrimos que embriões que não foram devidamente desidratados antes da limpeza babb são difíceis de imaginar devido à dispersão de luz. Babb é uma solução hidrofóbica que requer desidratação completa em um solvente orgânico, a fim de limpar o tecido opaco. A limpeza completa garante a capacidade de obter imagens nos níveis mais profundos possíveis dentro do embrião10,11. Neste protocolo, utilizou-se um objetivo de imersão de 20x de água, devido à sua longa distância de trabalho e disponibilidade no momento de nossos experimentos. Os objetivos de imersão do óleo são mais adequados para este protocolo, pois babb e óleo têm índices refrativos mais próximos do que a água e babb. No entanto, apesar da diferença no índice de refração, o objetivo de imersão da água utilizado neste protocolo proporcionou excelente qualidade de imagem.
Existem algumas limitações deste protocolo. A limpeza babb utilizada aqui é tóxica e corrosiva11,,12,,13. Babb dissolve cola e plásticos. Se as amostras não forem manuseadas corretamente durante a imagem, a lente objetiva do microscópio pode ser danificada por BABB que pode escapar da amostra através de rachaduras na tampa ou uma vedação quebrada entre o pára-choque fast well e o deslizamento de cobertura. Métodos de compensação que não utilizam solventes orgânicos, como o CLARITY, podem ser utilizados como alternativas10,,11,,14. A solução de correspondência de índice de refração da CLARITY tem um índice de refração semelhante ao da água, o que o torna um método de compensação adequado se usar um objetivo de imersão de água. Uma limitação adicional deste protocolo é que ele só pode ser realizado em tecidos não vivos, impedindo assim sua aplicação para imagens vivas.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Agradecemos a Brianna Alexander, Caolan O'Donnell e Michael Warkala pela leitura e edição cuidadosa deste manuscrito. Este trabalho foi apoiado pelo financiamento do National Heart, Lung and Blood Institute do NIH R01 HL103920, R01 HL134935, R21 OD025323-01 para SA; A AJR é apoiada pelo NHLBI HL103920-08S1 e pelo Instituto Nacional de Artrite e Treinamento de Doenças Musculoesqueléticas e de Pele grant T32052283-11.
Materiais
| Name | Company | Catalog Number | Comments |
| 10x PBS | MP Biomedicals | PBS10X02 | |
| 20x water immersion objective | Nikon | MRD77200 | |
| Agarose | Bio-Rad Laboratories | 1613101 | |
| Alexa Fluor 488 anti-goat | Invitrogen | A-11055 | |
| Alexa Fluor 555 anti-mouse | Invitrogen | A-31570 | |
| Analysis Software | Imaris 9.2.0 | ||
| Benzyl Alcohol | Sigma-Aldrich | 305197 | |
| Benzyl Benzoate | Sigma-Aldrich | 8.18701.0100 | |
| Cover Slips | VWR | 16004-312 | |
| DAPI (5 mg/mL stock) | Fisher Scientific | D3571 | |
| Eppendorf Tubes (2.0 mL) | Fisher Scientific | 05-408-138 | |
| Ethanol | VWR | 89370-084 | |
| Falcon tubes (50 mL) | Corning | 352098 | |
| Fast wells | Grace Bio Labs | 664113 | |
| Forceps | Roboz | RS-5015 | |
| Goat anti-VEGFR2 | R&D Systems, Inc. | AF644 | |
| Methanol | VWR | BDH1135-4LP | |
| Microscope | Nikon | A1HD25 | |
| Mouse anti-ERG | Abcam | ab214341 | |
| Normal Donkey Serum | Sigma-Aldrich | D9663 | |
| Paraformaldehyde | Electron Microscopy Sciences | 15710 | |
| Pasteur pipets | Fisher Scientific | 13-678-20D | |
| Petri dishes (35 mm) | Genesee Scientific | 32-103 | |
| Petri dishes (60 mm) | Genesee Scientific | 32-105 | |
| Plastic Molds | VWR | 18000-128 | |
| Scapels | Exelint International Co. | 29552 | |
| Triton-X-100 | Fisher Scientific | BP 151-500 |
Referências
- Hiruma, T., Nakajima, Y., Nakamura, H. Development of pharyngeal arch arteries in early mouse embryo. Journal of Anatomy. 201 (1), 15-29 (2002).
- Hutson, M. R., Kirby, M. L. Model systems for the study of heart development and disease Cardiac neural crest and conotruncal malformations. Seminars in Cell & Developmental Biology. 18 (1), 101-110 (2007).
- Wang, X., et al. Endothelium in the pharyngeal arches 3, 4 and 6 is derived from the second heart field. Developmental Biology. 421 (2), 108-117 (2017).
- Jerome, L. A., Papaioannou, V. E. DiGeorge syndrome phenotype in mice mutant for the T-box gene, Tbx1. Nature Genetics. 27 (3), 286-291 (2001).
- Lindsay, E. A., et al. Tbx1 haploinsufficieny in the DiGeorge syndrome region causes aortic arch defects in mice. Nature. 410 (6824), 97-101 (2001).
- Weninger, W., et al. Visualising the Cardiovascular System of Embryos of Biomedical Model Organisms with High Resolution Episcopic Microscopy (HREM). Journal of Cardiovascular Development and Disease. 5 (4), 58 (2018).
- Phillips, H. M., et al. Pax9 is required for cardiovascular development and interacts with Tbx1 in the pharyngeal endoderm to control 4th pharyngeal arch artery morphogenesis. Development. 146 (18), (2019).
- Vlaeminck-Guillem, V., et al. The Ets family member Erg gene is expressed in mesodermal tissues and neural crests at fundamental steps during mouse embryogenesis. Mechanisms of Development. 91 (1-2), 331-335 (2000).
- Ertürk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-217 (2012).
- Azaripour, A., et al. A survey of clearing techniques for 3D imaging of tissues with special reference to connective tissue. Progress in Histochemistry and Cytochemistry. 51 (2), 9-23 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying Tissue Clearing. Cell. 162 (2), 246-257 (2015).
- Becker, K., Jährling, N., Saghafi, S., Weiler, R., Dodt, H. U. Chemical Clearing and Dehydration of GFP Expressing Mouse Brains. PLoS One. 7 (3), e33916 (2012).
- Ertürk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados