Method Article
Visualización y análisis de arterias de arco faríngeo utilizando inmunohistoquímica de montaje completo y reconstrucción 3D
En este artículo
Resumen
Aquí, describimos un protocolo para visualizar y analizar las arterias del arco faríngeo 3, 4 y 6 de embriones de ratón utilizando inmunofluorescencia de montaje completo, limpieza de tejidos, microscopía confocal y reconstrucción 3D.
Resumen
La formación o remodelación inadecuadas de las arterias del arco faríngeo (PAA) 3, 4 y 6 contribuyen a algunas de las formas más graves de cardiopatía congénita. Para estudiar la formación de PAA, desarrollamos un protocolo utilizando inmunofluorescencia integral junto con la limpieza de tejido de alcohol bencílico/benzilo bencílico (BABB), y microscopía confocal. Esto permite la visualización del endotelio del arco faríngeo a una resolución celular fina, así como la conectividad 3D de la vasculatura. Utilizando software, hemos establecido un protocolo para cuantificar el número de células endoteliales (ECs) en PAA, así como el número de ECs dentro del plexo vascular que rodea a los PAA dentro de los arcos faríngeos 3, 4 y 6. Cuando se aplica a todo el embrión, esta metodología proporciona una visualización integral y un análisis cuantitativo de la vasculatura embrionaria.
Introducción
Durante la embriogénesis del ratón, las arterias del arco faríngeo (PAA) surgen como pares simétricos, bilaterales de arterias que conectan el corazón con las aortas dorsales1. A medida que el embrión se desarrolla, el primer y segundo par de PAA retroceden, mientras que losPAA 3,4y 6 se someten a una serie de eventos de remodelación asimétrica para formar las arterias del arco aórtico2.
Los PAA 3, 4 y 6 se desarrollan a través de vasculogénesis, que es la formación de novo de los vasos sanguíneos3. Los defectos en la formación o remodelación de estas arterias del arco dan lugar a diversos defectos cardíacos congénitos, como los observados en pacientes con Síndrome de DiGeorge4,5. Por lo tanto, comprender los mecanismos que regulan el desarrollo de los PAA puede conducir a una mejor comprensión de la etiología de las enfermedades cardíacas congénitas ( CHD).
Los enfoques actuales para visualizar y analizar el desarrollo de PAA incluyen inmunofluorescencia de secciones tisulares, moldes vasculares, inyección de tinta de la India, microscopía episcópica de alta resolución y/o inmunohistoquímica de montaje completo1,4,5,6,7. Aquí, describimos un protocolo que combina inmunofluorescencia de montaje completo, microscopía confocal y renderizado de imágenes 3D con el fin de recopilar, analizar y cuantificar datos volumétricos, conectividad vascular e identidad celular. Además, detallamos un método para compartimentar y cuantificar el número de CE en cada arco faríngeo como un medio para estudiar la formación del arco faríngeo plexo vascular y su remodelación en los PAA. Si bien este protocolo está diseñado para analizar el desarrollo de PAA, se puede utilizar para analizar otras redes vasculares en desarrollo.
Protocolo
El uso y los procedimientos de los animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Rutgers.
1. Preparación de soluciones
- Preparar 1 L de solución salina tamponada de fosfato con 0.1% Triton-X-100 (PBST) y filtrar esterilizar. Esta solución se puede almacenar a temperatura ambiente (RT) durante al menos un año.
- Preparar 600 l de tampón de bloqueo que consiste en 10% del suero de burro normal en PBST. Haga que esta solución sea fresca cada vez.
- Preparar 50 ml de las siguientes diluciones de metanol (MeOH) en una campana de flujo: 25% MeOH en agua desionizada (dH2O), 50% MeOH en dH2O, y 75% MeOH en dH2O. Vortex para mezclar. Almacenar en RT.
- Preparar 50 ml de las siguientes soluciones de benzoato de alcohol bencílico (BABB) en tubos cónicos de 50 ml.
- Para 100% BABB, añadir 32 ml de benzoato de bencilo a 16 ml de alcohol bencílico (2:1 volumen por relación de volumen).
- Para 50% BABB, añadir 16 ml de benzoato de bencilo y 8 ml de alcohol bencílico a 24 ml de MeOH.
- Cubra los tubos cónicos en papel de aluminio para protegerlos de la luz. Estas soluciones se pueden almacenar en RT durante un año.
PRECAUCION: BABB es tóxico y corrosivo. Debe manipularse y desecharse de acuerdo con MSDS.
2. Disección y fijación de embriones
NOTA: Este protocolo es adecuado para embriones de ratón E9.5 y E10.5 (hombres o mujeres) aislados de cualquier cepa de ratón. Para los embriones más jóvenes y mayores, los tiempos de incubación deben determinarse experimentalmente para maximizar la relación señal-ruido de la señal de fluorescencia.
- Llene uno de 35 mm y uno de 60 mm de platos Petri con 1 PBS y colóquelo sobre hielo hasta que sea necesario.
- Euthanizar un ratón embarazada por inhalación de CO2. Realizar la luxación cervical como una medida secundaria de la eutanasia.
- Limpie la zona abdominal de la presa con un 70% de etanol. Pellizcar el área abdominal usando fórceps y hacer una incisión en Forma de V usando tijeras quirúrgicas a partir de la base de la pared abdominal en la línea media; seguir abrióndo la cavidad torácica. Levante el tejido abdominal y mueva los intestinos hacia un lado para exponer los cuernos uterinos.
- Haga un corte en la base del canal vaginal, y con fórceps, tire del útero lejos de la presa. Haga un corte adicional en cada ovario para liberar el útero. Transfiera el útero a uno de los platos Petri de 60 mm que contienen frío 1x PBS.
- Con tijeras rectas, corte la pared uterina entre cada sitio de implantación. Recoja una decidua con un pipeta de vidrio y transfiera a la placa Petri de 35 mm con 1pbS. Bajo un microscopio de disección, inserte tijeras rectas en el espacio entre la decidua y la pared uterina. Cortar y quitar la pared uterina.
- Con fórceps finos, retire la decidua y las membranas de Reichert del embrión haciendo cuidadosamente incisiones transversales a lo largo del tejido y sacando el tejido lejos del saco de yema. Retire el saco de yema y el saco amniótico tirando cuidadosamente del tejido del embrión y haciendo cortes en la alantois y la vena umbilical.
NOTA: Los sacos de yema se pueden utilizar para genotipar embriones. - Transfiera cada embrión con un pipeta de vidrio en tubos individuales de 2 ml llenos de 1 ml de 1pbS. Etiquete cada tubo con un identificador único.
- Para fijar embriones, retire cuidadosamente el 1x PBS y agregue 4% de solución de paraformaldehído (PFA) en 1x PBS. Incubar a 4oC con agitación suave durante la noche.
NOTA: La fijación del 4% de PFA es adecuada para los anticuerpos mencionados en este protocolo. Sin embargo, los procedimientos de fijación deben optimizarse para anticuerpos adicionales.
3. Tinción de embriones
NOTA: En esta sección, los embriones se permeabilian y se tiñen con anticuerpos primarios y secundarios. Debido a que el desarrollo de PAA continúa rápidamente, las diferencias en la etapa embrionaria afectarán en gran medida el análisis aguas abajo. Por lo tanto, los embriones deben ser emparejados por la edad contando cuidadosamente las somitas para que coincidan con el control y los pares mutantes antes de más manipulaciones.
- Para lavar los embriones, retire cuidadosamente el 4% de PFA y agregue 1 x PBS. Invierta suavemente el(los) tubo(s) varias veces. Coloque el tubo o tubos del lado derecho hacia arriba y permita que los embriones se hundan. Repita el lavado 3 veces. Coloque el tubo(s) con embriones sobre hielo.
NOTA: (Punto de parada opcional) Después de los lavados, los embriones pueden deshidratarse en serie calificada de MeOH durante 30 minutos por dilución, como en la sección 1.3, y almacenarse a -20 oC en 100% MeOH para su uso posterior durante un máximo de 6 meses. - Para embriones E10.5, utilice un pipeta de vidrio para transferir un embrión a una placa Petri de 35 mm llena de 1PBS refrigerado 1x. Pellizca cuidadosamente el embrión justo por encima de la extremidad posterior con fórceps finos y haz un corte transversal para eliminar la mitad posterior del embrión. Esto permite que el embrión se auna en una posición sagital para el paso 4.2. Vuelva a colocar el embrión en el tubo de 2 ml con PBS 1x fresco.
NOTA: Un embrión de control y mutante se puede emparejar y teñir con la misma solución de anticuerpos en un tubo, para los pasos 3.3 a 3.8.- Si se tiñen dos embriones juntos, corte la cabeza de un embrión por encima del primer arco faríngeo pellizcando con fórceps finos para hacer un corte transversal. Esto distinguirá embriones de dos genotipos diferentes dentro de cada tubo.
- Para permeabilizar el embrión, pipetee 1x PBS del tubo, teniendo cuidado de no tocar el(los) embrión(s). Agregue 1 mL de PBST. Colocar el tubo a 4oC con una suave agitación durante la noche.
NOTA: (Punto de parada opcional) Los embriones se pueden mantener en solución PBST a 4 oC durante varios días. - Para evitar la unión inespecífica de anticuerpos, primero retire el PBST del tubo, teniendo cuidado de no tocar el(los) embrión(s). Añadir 600 l de solución tampón de bloqueo al embrión o embriones. Bloquear el embrión a 4oC con una agitación suave durante la noche.
NOTA: La solución de bloqueo debe girarse a máxima velocidad en una centrífuga de sobremesa inmediatamente antes de usarla para eliminar los desechos. - Para manchar y cuantificar los CE, utilice anticuerpos contra VEGFR2 y ERG. Las soluciones de anticuerpos se realizan en el búfer de bloqueo. El anticuerpo anti-VEGFR2 se diluye 1:200 y el anticuerpo ERG se diluye 1:1000.
NOTA: Las soluciones de anticuerpos deben girarse a máxima velocidad en una centrífuga de sobremesa inmediatamente antes de su uso para eliminar las partículas.- Para incubar embriones con anticuerpos primarios, elimine la solución tampón de bloqueo del tubo, teniendo cuidado de no tocar el(los) embrión(s). Añadir 600 ml de solución de anticuerpos primarios a cada tubo. Incubar embriones a 4oC con agitación suave durante 4-5 días.
- Para lavar el embrión o embriones de la solución de anticuerpos, primero retire la solución primaria de anticuerpos del tubo. Lavar los embriones cada hora con 1 ml de PBST a temperatura ambiente (RT) con agitación suave. Lavar los embriones 4-5 veces durante el día y luego incubar a 4 oC con agitación suave durante la noche. Repetir lavados al día siguiente.
- Haga soluciones secundarias de anticuerpos diluyendo Alexa Fluor 488 anticabra y Alexa Fluor 555 1:300 anti-ratón en búfer de bloqueo. Diluir el stock DAPI 1:1000 en el búfer de bloqueo.
NOTA: Las soluciones de anticuerpos deben girarse a máxima velocidad en una centrífuga de sobremesa inmediatamente antes de usarlas para eliminar las partículas. Además, otros tintes Alexa Fluor se pueden utilizar en lugar de 488 o 555.- Para incubar el embrión con anticuerpos secundarios, retire el PBST del tubo. Añadir 600 ml de solución secundaria de anticuerpos a cada tubo. Incubar embriones a 4oC con agitación suave durante 4-5 días.
- Para lavar el embrión o embriones de la solución de anticuerpos, primero retire la solución secundaria de anticuerpos del tubo. Lave el(los) embrión(es) cada hora con 1 ml de PBST a RT con agitación suave. Lavar los embriones 4-5 veces durante el día y luego incubar a 4 oC con agitación suave durante la noche. Repetir lavados al día siguiente.
4. Incrustar embriones en agarosa
NOTA: En la sección 4, los embriones se incrustarán en agarosa. Este proceso de incrustación tiene dos propósitos: orientar adecuadamente el embrión antes de la toma de imágenes, y ayudar a localizar el embrión después de que haya sido despejado en BABB (pasos 5.2.2 - 5.3.2).
- Preparar 200 ml de solución de agarosa al 1% añadiendo 2 g de agarosa a 200 ml de dH2O. Microondas hasta que se disuelva toda la agarosa.
NOTA: La agarosa restante se puede almacenar a 4 oC y se puede recalentar para usos posteriores. - Usando un molde de parafina de plástico y un pipeteo de vidrio, transfiera suavemente un embrión al molde. Retire cuidadosamente el PBST del embrión. Coloque el embrión en una posición sagital. Rápidamente, agregue alrededor de 0,5 ml de agarosa caliente al molde - lo suficiente para cubrir el embrión y llenar el molde. Asegúrese de que ninguna burbuja de aire rodee el embrión.
- Coloque el molde sobre hielo y cúbralo con papel de aluminio hasta que la agarosa se haya solidificado.
NOTA: No permita que el embrión se seque después de la eliminación de PBST. La solución de agarosa debe estar lo suficientemente caliente como para permanecer líquida cuando se añade al embrión. Añadir suficiente agarosa para cubrir el embrión, pero no demasiado, de lo contrario será difícil de imaginar. La profundidad de la imagen viene determinada en parte por la distancia de trabajo del objetivo.
5. Deshidratación y desparación de tejidos
NOTA: En esta sección, los embriones se deshidratan mediante la serie de metanol, luego se eliminan en el disolvente orgánico, BABB, y se montan entre dos cubiertas separadas por un espaciador de goma; en este protocolo se utilizan espaciadores de goma Fast Well. El parachoques Fast Well tiene una superficie adhesiva de doble cara. El espaciador es necesario para crear un pozo, en el que el embrión será colocado y mantenido entre dos cubreobjetos.
-
Deshidratación por metanol
- Etiquetar nuevos tubos de 2 ml, uno por embrión. Añadir 1 mL de 25% MeOH por tubo.
- Usando un bisturí limpio, corta suavemente la agarosa alrededor del embrión, dejando suficiente alrededor del embrión para que pueda ser recogido por fórceps. Utilice fórceps finos para agarrar suavemente la agarosa con el embrión incrustado y colóquela en el tubo etiquetado con 25% MeOH. No permita que los fórceps toquen el embrión.
- Incubar embriones a RT con agitación suave durante 1 hora en la oscuridad.
- Retire el 25% de MeOH del tubo, teniendo cuidado de no tocar el embrión. Añadir 1 mL de 50% MeOH por tubo. Incubar a RT con agitación suave durante 1 hora en la oscuridad.
- Retire el 50% de MeOH del tubo, teniendo cuidado de no tocar el embrión. Añadir 1 mL de 75% MeOH por tubo. Incubar a RT con agitación suave durante 1 hora en la oscuridad.
- Retire el 75% de MeOH del tubo, teniendo cuidado de no tocar el(los) embrión(s). Añadir 1 mL de 100% MeOH por tubo. Incubar a RT con agitación suave durante 1 hora en la oscuridad. Repita el lavado 100% MeOH dos veces.
-
Limpieza con BABB
- Retire 100% MeOH del tubo, teniendo cuidado de no tocar el embrión. Añadir 1 ml de 50% BABB por tubo. Incubar a RT con agitación suave durante 1 hora en la oscuridad.
- Retire el 50% babB del tubo, teniendo cuidado de no tocar el embrión. Añadir 1 mL de 100% BABB por tubo. Incubar a RT con agitación suave durante 1 hora en la oscuridad. Repita el lavado 100% BABB dos veces.
NOTA: (Punto de parada opcional) Los embriones pueden permanecer en 100% BABB en tubos durante aproximadamente una semana. Un almacenamiento más largo hará que BABB disuelva el plástico de los tubos.
-
Montaje de embriones para la toma de imágenes
- Coloque un parachoques Fast Well en un resbalón de la cubierta de vidrio de 24 mm x 60 mm #1,5, despegando el adhesivo de plástico de un lado. Asegúrese de que no haya burbujas de aire entre el cubreobjetos y el parachoques aplicando una presión suave sobre el adhesivo de plástico sobre el parachoques de goma. Etiquete el cubreobjetos según el número de embrión, el genotipo y los anticuerpos utilizados para la tinción.
NOTA: Cualquier espaciador se puede colocar entre los labios de las cubiertas siempre y cuando sea lo suficientemente grueso como para evitar aplastar o aplastar un embrión. Utilizamos espaciadores Fast Well debido a su grosor y conveniencia, que incluye superficies adhesivas a ambos lados del espaciador para asegurarlo a los cubreobjetos. - Retirar cuidadosamente y desechar el 100% BABB del tubo. Después de visualizar el embrión incrustado en agarosa en el tubo, utilice fórceps finos para recoger la agarosa y transfiera cuidadosamente el embrión al cubreobjetos dentro del Pozo Rápido - no permita que los fórceps toquen el embrión.
- Retire el segundo adhesivo de plástico del parachoques y coloque el segundo cubreobjetos en la parte superior. Retire las burbujas de aire presionando suavemente el cubreobjetos. Tenga cuidado de no romper el vidrio.
NOTA: Las muestras se pueden almacenar planas en un portaobjetos en la oscuridad en RT durante un año si el sello está apretado.
- Coloque un parachoques Fast Well en un resbalón de la cubierta de vidrio de 24 mm x 60 mm #1,5, despegando el adhesivo de plástico de un lado. Asegúrese de que no haya burbujas de aire entre el cubreobjetos y el parachoques aplicando una presión suave sobre el adhesivo de plástico sobre el parachoques de goma. Etiquete el cubreobjetos según el número de embrión, el genotipo y los anticuerpos utilizados para la tinción.
6. Adquisición de datos
NOTA: En los siguientes pasos, el endotelio de los arcos faríngeos 3, 4 y 6 se tomará una imagen mediante microscopía confocal.
-
Posicionamiento de diapositivas en el escenario del microscopio
- Para obtener imágenes de embriones, utilice un microscopio confocal equipado con un objetivo de inmersión en agua de 20x, apertura numérica 0,95, distancia de trabajo 0,95 mm y el software NIS-Elements AR 5.11.01 de 64 bits.
- Usando fluorescencia de campo ancho, localice visualmente los arcos faríngeos. Centrar la vista de campo alrededor de la4a PAA.
- Si el campo de visión del objetivo no captura toda el área del arco faríngeo, tome y suture un gran panel de imágenes con un 1% de superposición. Para evitar el movimiento de la muestra durante la adquisición de la imagen grande, fije suavemente el conjunto de deslizamiento de la cubierta al escenario utilizando arcilla de moldeo.
-
Configuración de parámetros de adquisición
- Establezca el tamaño del agujero en 1.0.
- En la pestaña Nd Acquisition, establezca los límites superior e inferior de la imagen mediante el ajuste grueso. Ajuste el tamaño del paso Z de acuerdo con las especificaciones del software. Determinar el grosor que se puede imaginar por la distancia de trabajo del objetivo y la claridad de la muestra.
- Debido al grosor del embrión, ajuste la ganancia en toda la pila Z. Establezca la intensidad del láser y la ganancia en el centro de la pila Z para cada canal (405, 488 y 555) y asigne valores en la pestaña Corrección de intensidad Z.
- Desplácese por el embrión hasta que las señales de fluorescencia comiencen a aparecer atoradas. Aumente la ganancia de cada canal hasta que la intensidad de la señal parezca similar al segmento anterior. Asigne el nuevo valor en la pestaña Corrección de intensidad Z. Repita hasta que se complete la pila z. Importar configuración de nuevo a Adquisición ND.
- Ejecute el análisis con la opción Ejecutar corrección Z.
7. Análisis utilizando el software Imaris
NOTA: En estos pasos, las imágenes confocales se analizarán utilizando el software de análisis de imágenes de microscopía, Imaris versión 9.2.0. Durante este análisis, primero seleccionaremos las regiones de interés que se analizarán mediante la creación de superficies. A continuación, usaremos la función Máscara para separar visualmente estas regiones. Por último, utilizaremos la función Spot para cuantificar el número de CE dentro de cada región de interés.
- Dependiendo del software de imágenes utilizado en el paso 6, convierta imágenes a .ims utilizando Imaris File Converter.
- Abra los archivos .ims. Establezca la imagen en Ortogonal en Cámara/Etiquetas de la imagen de la imagen de la imagen de la imagen de la imagen de la imagen de la imagen de la imagen de Panel Tipo de cámara.
- Localice los PAA y oriente la imagen para la superficie.
NOTA: Cuando los archivos se abran por primera vez, aparecerán como una compilación 3D de todos los sectores de la imagen. En este paso, los PAA se ubicarán convirtiendo la imagen 3D en una imagen 2D. La imagen 2D entonces permite que los PAA se orienten correctamente para el análisis.- En el panel Propiedades, desactive Volumen. En el panel Propiedades, haga clic en Agregar nueva segmentación ortogonal. Establezca Orientación de sectores en el plano XY. Utilice la posición de sector para desplazarse por la imagen hasta encontrar los PAA.
- Si los PAA no son paralelos a la parte superior e inferior de la imagen, gire libremente la imagen con el cursor del ratón para que los PAA se ejecuten de izquierda a derecha a través de la pantalla. En el menú desplegable Procesamiento de imágenes, seleccione Rotar libre y haga clic en Aceptar.
- Superficie del 3er Arco Faríngeo (Figura 2A, B - B")
NOTA: En estos pasos, los arcos faríngeos y PAA se trazarán utilizando la herramienta Superficie para generar una región de interés "superficie". Esto permitirá que cada región de interés se aísle visualmente del tejido circundante. Aquí describimos los pasos para salir a la superficie y analizar los componentes endoteliales del3er arco faríngeo. Los arcos faríngeos 4 y 6 se analizan de manera similar.- Para exponer el endotelio en todo el3er arco faríngeo, haga clic en el botón Agregar nueva superficie situado debajo del panel Propiedades. Haz doble clic en La Superficie 1 y cambia el nombre de la nueva superficie a "3er Arco Faríngeo".rd
- Seleccione Omitir creación automática, editar manualmente. Establezca Orientación de superficie en el plano YZ (orientación coronal). Utilice la Posición de corte para colocar el3er plano de superficie del arco faríngeo al que se conectan elPAA 3 y la Aorta dorsal.
- Gire la imagen de modo que elplano de superficie del arco faríngeo 3 rd esté a la vista. Desactive Ortho Slicer 1.
- Bajo el empate ? Contornodes ( Contour) Modo, seleccione la función Modo de dibujo a distancia. Ajuste la configuración de los parámetros si es necesario. Mantenga parámetros de superficie coherentes entre las muestras. Para este ejemplo, el espaciado de vértices es de 10 m.
- Para comenzar a surdar, presione la tecla Esc y luego haga clic en el botón Dibujar. Trace el perímetro del3er arco faríngeo con el cursor del ratón. Utilice la Posición de sector para mover entre 10 y 25 sectores. Traza el perímetro del arco faríngeo. Repita hasta que el arco faríngeo esté completamente trazado.
- Para generar la superficie de la región trazada, seleccione el botón Crear superficie en el panel Propiedades.
- Superficie de la 3a PAA (Figura 2C - C")
- Para exponer el endotelio de la3a PAA, primero apague la región de superficie del paso 7.4, deseleccionando la3a caja de superficie Arco Faríngeo. A continuación, vuelve a hacer clic en el botón Agregar nueva superficie. Haz doble clic en Surface 1 y cambia el nombre de la nueva superficie a"3rd PAA".
- Seleccione Omitir creación automática, editar manualmente. Establezca Orientación de superficie en el plano YZ (orientación coronal). Utilice la Posición de corte para colocar el plano de superficie3er PAA donde se conectan elPAA 3 y la Aorta dorsal y, a continuación, repita los pasos 7.4.
- Enmascaramiento de estructuras superficiales
NOTA: En los pasos siguientes, cada región de interés en superficie se enmascarará. El enmascaramiento permite que la región de interés sea visualmente distinta del resto del tejido de imagen y permite la cuantificación de estas distintas estructuras de interés. A continuación, describimos los pasos en Imaris para visualizar y analizar el endotelio PAA, así como el plexo - la vasculatura más pequeña que rodea los PAA dentro de los arcos faríngeos. En estos pasos, la3a superficie PAA se enmascarará para visualizar y realizar análisis solo en los PAA utilizando la función Spot descrita en la Sección 7.7.- Seleccione el volumen para visualizar todos los canales enmascarados. Pulse la tecla Esc para girar la imagen y colocarla en una posición XY.
- En la pestaña Editar, seleccione Selección de máscara para elPAA 3. Seleccione el canal DAPI y haga clic en Aceptar. Repita el proceso para los canales restantes.
- En el teclado, pulse Ctrl + D para ver el panel Ajustes de visualización. Seleccione cada canal nuevo y cámbiele el nombre para que quede claro lo que muestra cada canal. Por ejemplo, esto dará lugar a tres nuevos canales:"3rd PAA DAPI","3rd PAA ERG" y"3rd PAA VEGFR2".
NOTA: En los pasos 7.6.4-7.6.7 crearemos canales solo para el plexo del arco faríngeo. - Para visualizar el plexo endotelial por separado del PAA, primero seleccionaremos Selección de máscara para la3a superficie PAA. Seleccione el canal DAPI. Desmarque Seleccionar vóxeles fuera de la superficie y seleccione Seleccionar vóxeles dentro de la superficie a botones, establezca Seleccionar vóxeles dentrode la superficie a cero. Haga clic en Aceptar.
- Repita el proceso para los canales restantes. Esta operación excluirá la región que contiene el PAA de los nuevos canales enmascarados. Cambie el nombre de los canales para que quede claro lo que muestra cada canal. Por ejemplo, esto dará lugar a tres nuevos canales: "DAPI no PAA", "NO-PAA ERG" y "NO PAA VEGFR2".
- Para visualizar el plexo endotelial dentro del3er arco faríngeo, seleccione Selección de máscara en la pestaña Editar, para la superficie3er arco faríngeo. Seleccione el canal DAPI no PAA. Haga clic en Aceptar.
- Repita el proceso para los canales no PAA restantes. Cambie el nombre de los canales para que quede claro lo que muestra cada canal. Por ejemplo, esto dará lugar a tres nuevos canales: "Plexus DAPI", "Plexus ERG" y "Plexus VEGFR2".
- Cuantificación de números CE
NOTA: La expresión de las marcas ERG de núcleos endoteliales que facilitan la cuantificación de los números CE. En estos pasos, el número de CE se cuantificará utilizando la función Spot para generar un punto para cada EC marcada por la expresión ERG en el PAA y plexus. En la sección 7.7, se generarán puntos para cada célula ERG-positiva en el PAA enmascarado, seguido de la deselección de puntos en células ERG-positivas, VEGFR2-negativas.- En el teclado, pulse Ctrl + D para ver el panel Ajustes de visualización. Apague todos los canales excepto PAA ERG.
- En la pestaña de propiedades, haga clic en el botón Agregar nuevos puntos. Haga clic en Puntos 1 y cámbiele el nombre a "PAA Total Number of ECs". Haga clic en el botón de flecha azul. En Source Channel (Canal de origen),seleccione el canal PAA ERG. Ajuste el Diámetro XY Estimado a 4 m. Continúe con el siguiente panel haciendo clic en el botón de flecha azul.
- Ajuste el número de manchas vistas mediante la escala deslizante para asegurarse de que cada núcleo EC (marcado por la expresión ERG) esté representado por un punto. Haga clic en el botón verde de flecha doble.
- Desactive el canal PAA ERG en el Ajuste de pantalla. Encienda el canal PAA VEGFR2 para visualizar el endotelio PAA.
- Para cuantificar con precisión el número de CE, nos aseguramos de que cada punto exprese ambos marcadores CE, ERG y VEGFR2. Para ello, seleccione Superficie de objeto en la pestaña Editar . Añadir/Eliminar panel. Pulse la tecla Esc y elimine los puntos que no sean positivos veGFR2, manteniendo pulsada la tecla Mayús y seleccionando el punto.
NOTA: En el siguiente paso, se generarán puntos para cada celda ERG-positiva en el plexo enmascarado, seguido de la deselección de puntos en erg-positivas, células VEGFR2-negativas. - En la pestaña de propiedades, haga clic en el botón Agregar nuevos puntos. Seleccione Puntos 1 y cambie el nombre a "Número total de eCs de Plexus". Haga clic en el botón de flecha azul. En Source Channel (Canal de origen), seleccione el canal Plexus ERG. Ajuste el Diámetro XY Estimado a 4 m. Repita los pasos 7.7.1 - 7.7.5 para el número total de CE plexus.
- Haga clic en la pestaña Estadísticas de cada función de punto para determinar el número total de ECs en el3er PAA y el plexo del arco faríngeo.
- Repita los pasos 7.7.1 - 7.7.6 para los PAA restantes y el plexo del arco faríngeo.
Resultados
El protocolo de inmunofluorescencia de montaje completo presentado aquí produce resultados claros y limpios, permitiendo la reconstrucción 3D del endotelio del arco faríngeo, como se ve en la Figura 1A. Es importante incubar embriones durante una cantidad suficiente de tiempo en cada solución de anticuerpos para asegurar una penetración completa a través de la muestra, así como lavar a fondo los embriones después de la incubación de anticuerpos. En la Figura 1B,los puntos grandes y brillantes aparecen como resultado de partículas en las soluciones de anticuerpos o tampón de bloqueo. Hemos encontrado que centrifugar cada solución antes de su uso y períodos más largos de lavados PBST después de cada incubación de anticuerpos resuelve este problema.
La Figura 2 ilustra el proceso utilizado para exponer un arco faríngeo y un PAA para su análisis como se describe en la sección 7 del protocolo. Utilizando la función enmascarada, el software Imaris permite separar visualmente las regiones y analizarlas de forma independiente.
La Figura 3 muestra el enmascaramiento individual de diferentes compartimentos vasculares en los arcos faríngeos: el PAA(Figura 3A, B, C)y el plexo(Figura 3A', B', C'). El enmascaramiento permite el análisis y cuantificación de números CE en cada estructura por separado. En la Figura 3C-C',la función Spot se utiliza para cuantificar el número total de ECs tanto en el PAA como en el plexo, asignando un único punto para cada núcleo que expresa ERG. Es importante tener en cuenta que el algoritmo utilizado para la función Spot está diseñado para generar un punto para cualquier píxel de un tamaño especificado. ERG, que se utiliza aquí como un marcador de núcleos EC, también se expresa en las células de la cresta neural8; las células de la cresta neural no expresan VEGFR2. La Figura 3D ilustra un ejemplo de un punto ERG positivo (verde), VEGFR2 negativo (rosa) generado por la función de punto Imaris. Como resultado, es esencial verificar que cada punto representa una sola CE y está etiquetado con ERG y VEGFR2.
| Paso | hora | Temperatura | |
| 1 | Lavado/Permeabilización PBST | 24 h u O/N | 4 oC |
| 2 | Bloqueo de búfer | 25 h u O/N | 4 oC |
| 3 | Anticuerpo primario | 4-5 días | 4 oC |
| 4 | Lavado PBST | 4-5 veces al día durante 2 días | RT (o 4 oC si es O/N) |
| 5 | Anticuerpo secundario | 4-5 días | 4 oC |
| 6 | Lavado PBST | 4-5 veces al día durante 2 días | RT (o 4 oC si es O/N) |
| 7 | Insertar | N/A | Rt |
| 8 | Deshidratación de metanol y BABB | 1 hora por paso | Rt |
Tabla 1: Descripción general del protocolo de inmunofluorescencia de montaje completo. O/N - noche; RT - temperatura ambiente.
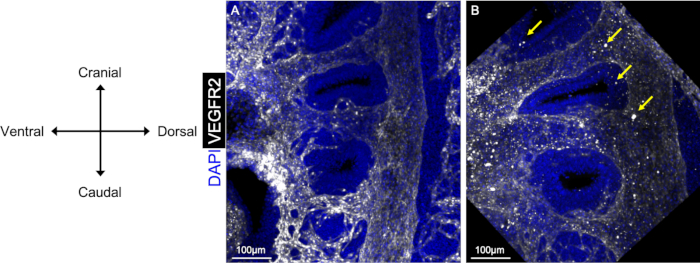
Figura 1: Comparación de imágenes limpias y sucias tras la inmunofluorescencia de montaje completo. Las vistas sagitales del embrión E10.5 muestran el uso de anticuerpos anti-VEGFR2 (blanco) para visualizar el endotelio PAA. Los embriones lavados a fondo con incubaciones post anticuerpos PBST (A) tienen una mayor relación señal-ruido y producen una imagen más limpia, en comparación con los embriones que no se lavan a fondo (B). Las flechas en B muestran áreas de ruido/suciedad que han aparecido en la imagen cuando un embrión no se lava a fondo o la solución de anticuerpos no se ha centrifugado. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Orientación del arco faríngeo (PA) y PAA. Se utiliza una vista sagital 2D (A) para identificar la ubicación de los PAA en la imagen confocal. Una ortocortadorcorona (A, línea amarilla) se coloca a través de los PAA. El arco faríngeo (B) y el PAA (C) se exponen en la orientación coronal mediante la herramienta Dibujo a distancia de Imaris. La herramienta Dibujo de distancia, establecida en 10 m, se utiliza para trazar el perímetro del3er arco faríngeo (B) o el PAA (C). Los contornos se dibujan cada 10-25 rodajas a través de todo el arco (B', C'). Los contornos se combinan para generar una superficie 3D del arco faríngeo (B") o el PAA (C"). Haga clic aquí para ver una versión más grande de esta figura.
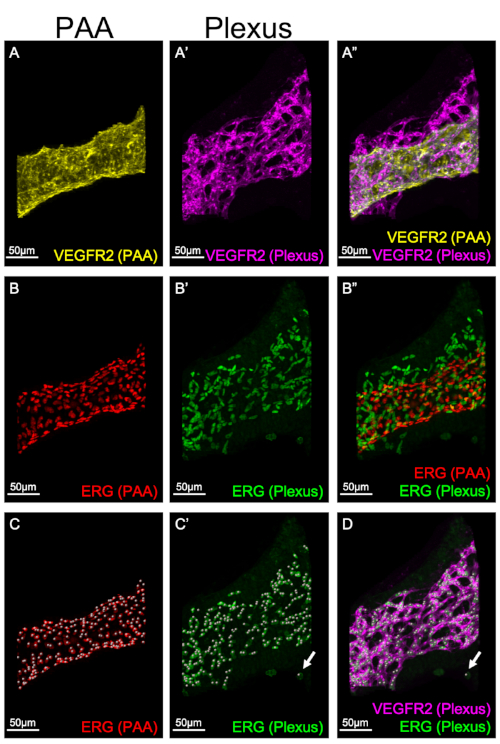
Figura 3: Cuantificación de los números CE en un PAA y un plexo. Las reconstrucciones 3D se utilizan para visualizar la estructura de los recipientes y la expresión de los marcadores CE en un PAA o en un plexo CE por separado. Los paneles A-A' muestran la expresión de VEGFR2 en el PAA (A, amarillo) y en el plexo (A', rosa). Panel A" ilustra una combinación de la expresión PAA y plexus VEGFR2. Los paneles B-B' muestran la expresión de ERG en el PAA (B, rojo) y en el plexo (B', verde). Panel B" ilustra la fusión del PAA y el plexo. C-C'. La función Spot en Imaris se utiliza para cuantificar el número de ECs en el PAA o plexo. A cada celda ERG positiva en el PAA (C, rojo) o plexo (C', verde) se les asigna un único punto para marcar una sola EC. La flecha en C'-D muestra un ejemplo ERG-positivo, VEGFR2-negativo en el plexo que ha sido generado por la función de punto Imaris. Este punto está excluido de la cuantificación. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La capacidad de visualizar el endotelio en embriones de ratón en 3D ha proporcionado nuevas perspectivas sobre su desarrollo3. Aquí presentamos un protocolo que permite la toma de imágenes 3D de alta resolución de embriones, la visualización de la conectividad vascular y los análisis cuantitativos de la formación de PAA. Este protocolo se puede emplear para ver cómo las alteraciones genéticas o los insultos ambientales afectan el desarrollo de la PAA. El procedimiento descrito aquí utiliza anticuerpos contra VEGFR2 y ERG para visualizar la formación de PAA y cuantificar el número DE EC; sin embargo, se pueden utilizar anticuerpos adicionales para visualizar y analizar otros aspectos del desarrollo de la arteria del arco, como el reclutamiento de crestas neurales o la diferenciación de células musculares lisas. Si este procedimiento se va a utilizar en etapas anteriores de la embriogénesis, es importante tener en cuenta que es posible que algunos antígenos (por ejemplo, ERG) detectados en este protocolo aún no se expresen. Otras manchas nucleares como DAPI o DRAQ5 o el etiquetado de linaje con trazadores con etiqueta nuclear se pueden utilizar para cuantificar el número CE.
Hay varios pasos críticos dentro del protocolo: asegurar que 1) los embriones no se desecienten entre los cambios de solución; 2) los embriones se lavan a fondo después de incubaciones de anticuerpos; y 3) que los embriones están completamente deshidratados con MeOH antes de limpiar el tejido con BABB.
Los lavados de metanol antes de la limpieza del tejido tienen dos propósitos: eliminar la fluorescencia debida a la expresión de proteínas fluorescentes (por ejemplo, la expresión de EGFP o tdTomato utilizado para el rastreo del linaje) en el embrión, y deshidratar el tejido. La eliminación de la fluorescencia de las proteínas fluorescentes permite el uso de cualquier combinación de fluoróforos para la toma de imágenes. Los anticuerpos contra EGFP y TdTomato (cereza) se pueden utilizar para visualizar la expresión de estas proteínas fluorescentes. Alternativamente, MeOH puede ser reemplazado por tetrahidrofurano para preservar la fluorescencia de las proteínas fluorescentes9.
Hemos descubierto que los embriones que no se han deshidratado adecuadamente antes de la limpieza de BABB son difíciles de imaginar debido a la dispersión de la luz. BABB es una solución hidrófoba que requiere deshidratación completa en un disolvente orgánico para limpiar el tejido opaco. La limpieza completa garantiza la capacidad de obtener imágenes en los niveles más profundos posibles dentro del embrión10,,11. En este protocolo, utilizamos un objetivo de inmersión en agua 20x, debido a su larga distancia de trabajo y disponibilidad en el momento de nuestros experimentos. Los objetivos de inmersión en aceite son más adecuados para este protocolo, ya que BABB y el aceite tienen índices de refracción más cercanos que el agua y babB. Sin embargo, a pesar de la diferencia en el índice de refracción, el objetivo de inmersión en agua utilizado en este protocolo proporcionaba una excelente calidad de imagen.
Hay algunas limitaciones de este protocolo. La limpieza de BABB utilizada aquí es tóxica y corrosiva11,12,13. BABB disuelve pegamento y plásticos. Si las muestras no se manipulan correctamente durante la toma de imágenes, babB puede dañar la lente objetivo del microscopio que puede escapar de la muestra a través de grietas en la cubierta o un sello roto entre el parachoques Fast Well y el cubreobjetos. Los métodos de compensación que no utilizan disolventes orgánicos, como CLARITY, se pueden utilizar como alternativas10,11,14. La solución de coincidencia de índices de refracción de CLARITY tiene un índice de refracción similar al del agua, lo que la convierte en un método de limpieza adecuado si se utiliza un objetivo de inmersión en agua. Una limitación adicional de este protocolo es que sólo se puede realizar en tejidos no vivos, evitando así su aplicación para imágenes en vivo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Brianna Alexander, Caolan O'Donnell y Michael Warkala por la lectura y edición cuidadosa de este manuscrito. Este trabajo fue apoyado por la financiación del Instituto Nacional del Corazón, Pulmón y Sangre del NIH R01 HL103920, R01 HL134935, R21 OD025323-01 a SA; AJR cuenta con el apoyo de NHLBI HL103920-08S1 y la beca T32052283-11 del Instituto Nacional de Artritis y Enfermedades Musculoesqueléticas y de La Piel.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x PBS | MP Biomedicals | PBS10X02 | |
| 20x water immersion objective | Nikon | MRD77200 | |
| Agarose | Bio-Rad Laboratories | 1613101 | |
| Alexa Fluor 488 anti-goat | Invitrogen | A-11055 | |
| Alexa Fluor 555 anti-mouse | Invitrogen | A-31570 | |
| Analysis Software | Imaris 9.2.0 | ||
| Benzyl Alcohol | Sigma-Aldrich | 305197 | |
| Benzyl Benzoate | Sigma-Aldrich | 8.18701.0100 | |
| Cover Slips | VWR | 16004-312 | |
| DAPI (5 mg/mL stock) | Fisher Scientific | D3571 | |
| Eppendorf Tubes (2.0 mL) | Fisher Scientific | 05-408-138 | |
| Ethanol | VWR | 89370-084 | |
| Falcon tubes (50 mL) | Corning | 352098 | |
| Fast wells | Grace Bio Labs | 664113 | |
| Forceps | Roboz | RS-5015 | |
| Goat anti-VEGFR2 | R&D Systems, Inc. | AF644 | |
| Methanol | VWR | BDH1135-4LP | |
| Microscope | Nikon | A1HD25 | |
| Mouse anti-ERG | Abcam | ab214341 | |
| Normal Donkey Serum | Sigma-Aldrich | D9663 | |
| Paraformaldehyde | Electron Microscopy Sciences | 15710 | |
| Pasteur pipets | Fisher Scientific | 13-678-20D | |
| Petri dishes (35 mm) | Genesee Scientific | 32-103 | |
| Petri dishes (60 mm) | Genesee Scientific | 32-105 | |
| Plastic Molds | VWR | 18000-128 | |
| Scapels | Exelint International Co. | 29552 | |
| Triton-X-100 | Fisher Scientific | BP 151-500 |
Referencias
- Hiruma, T., Nakajima, Y., Nakamura, H. Development of pharyngeal arch arteries in early mouse embryo. Journal of Anatomy. 201 (1), 15-29 (2002).
- Hutson, M. R., Kirby, M. L. Model systems for the study of heart development and disease Cardiac neural crest and conotruncal malformations. Seminars in Cell & Developmental Biology. 18 (1), 101-110 (2007).
- Wang, X., et al. Endothelium in the pharyngeal arches 3, 4 and 6 is derived from the second heart field. Developmental Biology. 421 (2), 108-117 (2017).
- Jerome, L. A., Papaioannou, V. E. DiGeorge syndrome phenotype in mice mutant for the T-box gene, Tbx1. Nature Genetics. 27 (3), 286-291 (2001).
- Lindsay, E. A., et al. Tbx1 haploinsufficieny in the DiGeorge syndrome region causes aortic arch defects in mice. Nature. 410 (6824), 97-101 (2001).
- Weninger, W., et al. Visualising the Cardiovascular System of Embryos of Biomedical Model Organisms with High Resolution Episcopic Microscopy (HREM). Journal of Cardiovascular Development and Disease. 5 (4), 58(2018).
- Phillips, H. M., et al. Pax9 is required for cardiovascular development and interacts with Tbx1 in the pharyngeal endoderm to control 4th pharyngeal arch artery morphogenesis. Development. 146 (18), (2019).
- Vlaeminck-Guillem, V., et al. The Ets family member Erg gene is expressed in mesodermal tissues and neural crests at fundamental steps during mouse embryogenesis. Mechanisms of Development. 91 (1-2), 331-335 (2000).
- Ertürk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-217 (2012).
- Azaripour, A., et al. A survey of clearing techniques for 3D imaging of tissues with special reference to connective tissue. Progress in Histochemistry and Cytochemistry. 51 (2), 9-23 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying Tissue Clearing. Cell. 162 (2), 246-257 (2015).
- Becker, K., Jährling, N., Saghafi, S., Weiler, R., Dodt, H. U. Chemical Clearing and Dehydration of GFP Expressing Mouse Brains. PLoS One. 7 (3), e33916(2012).
- Ertürk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados