Method Article
Visualisierung und Analyse von Pharyngeal Arch Arterien mittels ganzer Immunhistochemie und 3D-Rekonstruktion
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll zur Visualisierung und Analyse der Rachenbogenarterien 3, 4 und 6 von Mausembryonen mit ganzer Immunfluoreszenz, Gewebeklärung, konfokaler Mikroskopie und 3D-Rekonstruktion.
Zusammenfassung
Unsachgemäße Bildung oder Umgestaltung der Pharyngealbogenarterien (PAAs) 3, 4 und 6 tragen zu einigen der schwersten Formen der angeborenen Herzkrankheit bei. Um die Bildung von PAAs zu untersuchen, entwickelten wir ein Protokoll mit ganzer Immunfluoreszenz in Verbindung mit Benzylalkohol/Benzylbenzoat (BABB) Gewebeclearing und konfokaler Mikroskopie. Dies ermöglicht die Visualisierung des Pharyngealbogenendothel summieren bei feiner Zellauflösung sowie die 3D-Konnektivität der Vaskulatur. Mithilfe von Software haben wir ein Protokoll zur Quantifizierung der Anzahl der Endothelzellen (ECs) in PAAs sowie der Anzahl der ECs innerhalb des Gefäßplexus, der die PAAs in den Pharyngealbögen 3, 4 und 6 umgibt, erstellt. Wenn sie auf den gesamten Embryo angewendet wird, bietet diese Methode eine umfassende Visualisierung und quantitative Analyse der embryonalen Vaskulatur.
Einleitung
Während der Mausembryogenese entstehen Pharyngealbogenarterien (PAAs) als symmetrische, bilaterale Arterienpaare, die das Herz mit der dorsalen Aortae1verbinden. Während sich der Embryo entwickelt, gehen das erste und zweite PAAs-Paar zurück, während die3.,4.und6. PAAs eine Reihe asymmetrischer Umbauereignisse durchlaufen, um die Aortenbogenarterien zu bilden2.
Die PAAs 3, 4 und 6 entwickeln sich über Vaskulogenese, die die de novo Bildung von Blutgefäßen3ist. Defekte bei der Bildung oder Umgestaltung dieser Bogenarterien führen zu verschiedenen angeborenen Herzfehlern, wie sie bei Patienten mit DiGeorge-Syndrom4,5beobachtet werden. Daher kann das Verständnis von Mechanismen, die die Entwicklung von PAAs regulieren, zu einem besseren Verständnis der angeborenen Herzkrankheit (CHD) Ätiologie führen.
Aktuelle Ansätze zur Visualisierung und Analyse der PAA-Entwicklung umfassen die Immunfluoreszenz von Gewebeabschnitten, Gefäßabgüsse, Indien-Tinteninjektion, hochauflösende episkopische Mikroskopie und/oder Ganzleitimmunhistochemie1,4,5,6,7. Hierin beschreiben wir ein Protokoll, das Immunfluoreszenz, konfokale Mikroskopie und 3D-Bildwiedergabe kombiniert, um volumetrische Daten, vaskuläre Konnektivität und Zellidentität zu sammeln, zu analysieren und zu quantifizieren. Darüber hinaus beschreiben wir eine Methode zur Abschottung und Quantifizierung der Anzahl der ECs in jedem Rachenbogen als Mittel zur Untersuchung der Bildung des Pharyngealbogen-Gefäßplexus und dessen Umgestaltung in die PAAs. Während dieses Protokoll für die Analyse der PAA-Entwicklung entwickelt wurde, kann es verwendet werden, um andere sich entwickelnde Gefäßnetzwerke zu analysieren.
Protokoll
Die Verwendung und die Verfahren für Tiere wurden vom Institutional Animal Care and Use Committee der Rutgers University genehmigt.
1. Vorbereitung von Lösungen
- 1 L Phosphat gepufferte Salin mit 0,1% Triton-X-100 (PBST) und Filtersterilisieren vorbereiten. Diese Lösung kann mindestens ein Jahr lang bei Raumtemperatur (RT) gelagert werden.
- Bereiten Sie 600 l Blockierpuffer vor, der aus 10 % des normalen Eselsserums in PBST besteht. Machen Sie diese Lösung jedes Mal frisch.
- 50 ml der folgenden Methanolverdünnungen (MeOH) in einer Durchflusshaube vorbereiten: 25% MeOH in entionisiertem Wasser (dH2O), 50% MeOH in dH2O und 75% MeOH in dH2O. Vortex zum Mischen. Bei RT aufbewahren.
- 50 ml der folgenden Benzylalkohol-Benzylbenzoat-Lösungen (BABB) in 50 ml konischen Rohren vorbereiten.
- Für 100% BABB 32 ml Benzylbenzoat zu 16 ml Benzylalkohol hinzufügen (2:1 Volumen pro Volumenverhältnis).
- Für 50% BABB 16 ml Benzylbenzoat und 8 ml Benzylalkohol zu 24 ml MeOH hinzufügen.
- Abdeckung konische Rohre in Aluminiumfolie, um vor Licht zu schützen. Diese Lösungen können bis zu einem Jahr bei RT gelagert werden.
ACHTUNG: BABB ist giftig und ätzend. Es sollte gemäß MSDS behandelt und entsorgt werden.
2. Embryonizieren und Fixierung
HINWEIS: Dieses Protokoll eignet sich für E9.5- und E10.5-Mausembryonen (männlich oder weiblich), die von jedem Mausstamm isoliert sind. Bei jüngeren und älteren Embryonen sollten die Inkubationszeiten experimentell bestimmt werden, um das Signal-Rausch-Verhältnis des Fluoreszenzsignals zu maximieren.
- Füllen Sie eine 35 mm und eine 60 mm Petrischalen mit 1x PBS und legen Sie sie auf Eis, bis sie benötigt werden.
- Euthanisieren Sie eine2 schwangere Maus über CO2-Inhalation. Führen Sie zervikale Dislokation als sekundäres Maß für Euthanasie durch.
- Reinigen Sie den Bauchbereich des Damms mit 70% Ethanol. Kneifen Sie den Bauchbereich mit Zangen und machen Sie einen V-ähnlichen Schnitt mit chirurgischen Scheren beginnend von der Basis der Bauchwand an der Mittellinie; die Brusthöhle weiter zu öffnen. Heben Sie das Bauchgewebe und bewegen Sie den Darm zur Seite, um die Gebärmutterhörner zu belichten.
- Machen Sie einen Schnitt an der Basis des Vaginalkanals, und mit Zange, ziehen Sie die Gebärmutter weg vom Damm. Machen Sie einen zusätzlichen Schnitt an jedem Eierstock, um die Gebärmutter zu befreien. Die Gebärmutter in eines der 60 mm Petrischalen mit kalten 1x PBS geben.
- Schneiden Sie mit einer geraden Schere die Gebärmutterwand zwischen jeder Implantationsstelle. Nehmen Sie eine Decidua mit einer Glaspipette auf und geben Sie sie mit 1x PBS in die 35 mm Petrischale. Legen Sie unter einem Seziermikroskop eine gerade Schere in den Raum zwischen der Decidua und der Gebärmutterwand ein. Schneiden und entfernen Sie die Gebärmutterwand.
- Mit feinen Zangen entfernen Sie die Dezidua- und Reichert-Membranen aus dem Embryo, indem Sie vorsichtig Querschnitte entlang des Gewebes machen und das Gewebe vom Eigelbsack wegziehen. Entfernen Sie Dottersack und Fruchtwassersack, indem Sie das Gewebe vorsichtig vom Embryo wegziehen und Schnitte an der Allantois- und Nabelvene vornehmen.
HINWEIS: Yolk-Säcke können zur Genotypisierung von Embryonen verwendet werden. - Übertragen Sie jeden Embryo mit einer Glaspipette in einzelne 2 ml-Rohre, gefüllt mit 1 ml 1x PBS. Beschriften Sie jedes Rohr mit einem eindeutigen Bezeichner.
- Um Embryonen zu fixieren, entfernen Sie vorsichtig die 1x PBS und fügen Sie 4% Paraformaldehyd (PFA) Lösung in 1x PBS. Bei 4 °C mit sanfter Rührung über Nacht inkubieren.
HINWEIS: 4% PFA-Fixierung ist für die in diesem Protokoll genannten Antikörper geeignet. Fixierungsverfahren sollten jedoch für zusätzliche Antikörper optimiert werden.
3. Embryo-Färbung
HINWEIS: In diesem Abschnitt werden Embryonen durchmeabilisiert und mit primären und sekundären Antikörpern gefärbt. Da die PAA-Entwicklung rasch voranschreitet, werden Unterschiede im embryonalen Stadium die Analyse nachgelagert stark beeinflussen. Daher müssen Embryonen altersgerecht werden, indem sie vor weiteren Manipulationen sorgfältig auf Kontroll- und Mutantenpaare zählen.
- Um Embryonen zu waschen, entfernen Sie vorsichtig 4% PFA und fügen Sie 1x PBS hinzu. Umkehren Sie die Rohre vorsichtig mehrmals. Rohr(e) nach rechts nach oben stellen und den Embryo(en) sinken lassen. Waschen Sie 3 Mal. Legen Sie die Röhre mit Embryonen auf Eis.
ANMERKUNG: (Optionaler Haltepunkt) Nach den Wässen können Embryonen in einer abgestuften MeOH-Serie für 30 min pro Verdünnung, wie in Abschnitt 1.3, dehydriert und bei -20 °C in 100 % MeOH für die spätere Verwendung bis zu 6 Monate gelagert werden. - Für E10.5-Embryonen verwenden Sie eine Glaspipette, um einen Embryo auf eine 35 mm Petrischale zu übertragen, die mit gekühlten 1x PBS gefüllt ist. Kneifen Sie den Embryo vorsichtig mit feiner Zange über die Hinterbeinige und machen Sie einen Querschnitt, um die hintere Hälfte des Embryos zu entfernen. Dadurch kann der Embryo für Schritt 4.2 flach in einer sagittalen Position liegen. Legen Sie den Embryo mit frischen 1x PBS wieder in das 2 ml-Rohr.
HINWEIS: Ein Kontroll- und mutierter Embryo kann mit der gleichen Antikörperlösung in einem Rohr für die Schritte 3.3 bis 3.8 gepaart und gebeizt werden.- Wenn Sie zwei Embryonen zusammen färben, schneiden Sie den Kopf eines Embryos über dem ersten Rachenbogen ab, indem Sie mit feinen Zangen kneifen, um einen Querschnitt zu machen. Dadurch werden Embryonen von zwei verschiedenen Genotypen in jedem Rohr unterschieden.
- Um die Embryonen zu permeabilisieren, pfeifen Sie 1x PBS aus dem Rohr heraus, wobei Sie darauf achten, den Embryo(n) nicht zu berühren. Fügen Sie 1 ml PBST hinzu. Legen Sie das Rohr bei 4 °C mit sanfter Rührung über Nacht.
HINWEIS: (Optionaler Haltepunkt) Embryonen können mehrere Tage in PBST-Lösung bei 4 °C aufbewahrt werden. - Um eine unspezifische Bindung von Antikörpern zu verhindern, entfernen Sie zunächst PBST aus dem Rohr, wobei Sie darauf achten, die Embryonen nicht zu berühren. Fügen Sie dem Embryo(n) 600 L Blockierpufferlösung hinzu. Blockieren Sie den Embryo(n) bei 4 °C mit sanfter Erregung über Nacht.
HINWEIS: Die Blockierlösung muss unmittelbar vor dem Einsatz zur Beseitigung von Schmutz mit Höchstgeschwindigkeit auf einer Tischzentrifuge gesponnen werden. - Um ECs zu färben und zu quantifizieren, verwenden Sie Antikörper gegen VEGFR2 und ERG. Antikörperlösungen werden im Sperrpuffer hergestellt. Anti-VEGFR2 Antikörper wird 1:200 verdünnt und ERG Antikörper wird 1:1000 verdünnt.
HINWEIS: Antikörperlösungen müssen unmittelbar vor dem Einsatz zur Partikelentfernung mit Höchstgeschwindigkeit auf einer Tischzentrifuge gesponnen werden.- Um Embryonen mit primären Antikörpern zu inkubieren, entfernen Sie die Blockierende Pufferlösung aus dem Rohr, wobei Sie darauf achten, die Embryonen nicht zu berühren. Fügen Sie jedem Rohr 600 L Primärantikörperlösung hinzu. Embryo(n) bei 4 °C mit sanfter Erregung für 4-5 Tage inkubieren.
- Um die Embryonen der Antikörperlösung zu waschen, entfernen Sie zuerst die primäre Antikörperlösung aus dem Rohr. Waschen Sie Embryonen stündlich mit 1 ml PBST bei Raumtemperatur (RT) mit sanfter Rührung. Embryonen 4-5 Mal am Tag waschen und dann bei 4 °C mit sanfter Erregung über Nacht bebrüten. Wiederholte Säuerungen am nächsten Tag.
- Erstellen Sie sekundäre Antikörperlösungen, indem Sie Diegegen-Ziegenalexa Fluor 488 und Anti-Maus Alexa Fluor 555 1:300 im Blockierpuffer verdünnen. Verdünnen Sie den Lager-DAPI 1:1000 im Sperrpuffer.
HINWEIS: Antikörperlösungen müssen unmittelbar vor dem Einsatz zur Partikelentfernung auf einer Tischzentrifuge mit Höchstgeschwindigkeit gesponnen werden. Darüber hinaus können andere Alexa FluorFarbstoffe anstelle von 488 oder 555 verwendet werden.- Um die Embryonen mit sekundären Antikörpern zu inkubieren, entfernen Sie PBST aus dem Rohr. Fügen Sie jedem Rohr 600 L sekundäre Antikörperlösung hinzu. Embryo(n) bei 4 °C mit sanfter Erregung für 4-5 Tage inkubieren.
- Um die Embryonen der Antikörperlösung zu waschen, entfernen Sie zunächst die sekundäre Antikörperlösung aus dem Rohr. Waschen Sie die Embryonen stündlich mit 1 ml PBST bei RT mit sanfter Erregung. Embryonen 4-5 Mal am Tag waschen und dann bei 4 °C mit sanfter Erregung über Nacht bebrüten. Wiederholte Säuerungen am nächsten Tag.
4. Einbetten von Embryonen in Agarose
HINWEIS: In Abschnitt 4 werden die Embryonen in Agarose eingebettet. Dieser Einbettungsprozess dient zwei Zwecken: den Embryo vor der Bildgebung richtig auszurichten und bei der Lokalisierung des Embryos nach der Freigabe in BABB zu helfen (Schritte 5.2.2 - 5.3.2).
- Bereiten Sie 200 ml 1% Agaroselösung vor, indem Sie 2 g Agarose zu 200 ml dH2O. Mikrowelle hinzufügen, bis sich alle Agarose aufgelöst hat.
HINWEIS: Restagarose kann bei 4 °C gelagert und für spätere Verwendungen wieder erhitzt werden. - Mit einer Kunststoff-Paraffinform und Glaspipette, sanft übertragen einen Embryo auf die Form. Entfernen Sie PBST vorsichtig aus dem Embryo. Stellen Sie den Embryo in eine sagittale Position. Fügen Sie der Form schnell etwa 0,5 ml heiße Agarose hinzu - gerade genug, um den Embryo zu bedecken und die Form zu füllen. Stellen Sie sicher, dass keine Luftblasen den Embryo umgeben.
- Legen Sie die Form auf Eis und decken Sie sie mit Aluminiumfolie ab, bis die Agarose verfestigt ist.
HINWEIS: Lassen Sie den Embryo nach der Entfernung von PBST nicht trocknen. Die Agarose-Lösung muss warm genug sein, um flüssig zu bleiben, wenn sie dem Embryo zugesetzt wird. Fügen Sie gerade genug Agarose, um den Embryo zu bedecken, aber nicht zu viel, sonst wird es schwierig zu bilden sein. Die Bildtiefe wird zum Teil durch den Arbeitsabstand des Ziels bestimmt.
5. Dehydrierung und Geweberäumung
HINWEIS: In diesem Abschnitt werden Embryonen mit Methanol-Serie dehydriert, dann im organischen Lösungsmittel BABB gereinigt und zwischen zwei Durchdeckungslippen montiert, die durch einen Gummiabstandraum getrennt sind; in diesem Protokoll Werden Fast Well Gummi-Abstandshalter verwendet. Der Fast Well Stoßfänger hat eine doppelseitige Klebefläche. Der Abstandsraum wird benötigt, um einen Brunnen zu schaffen, in dem der Embryo zwischen zwei Abdeckungen platziert und gehalten wird.
-
Methanol-Dehydrierung
- Etikettieren Sie neue 2 ml-Rohre, eine pro Embryo. 1 ml 25% MeOH pro Tube hinzufügen.
- Schneiden Sie die Agarose mit einem sauberen Skalpell vorsichtig um den Embryo herum und lassen Sie genug um den Embryo herum, so dass er durch Zangen aufgenommen werden kann. Verwenden Sie feine Zangen, um die Agarose mit dem eingebetteten Embryo sanft zu greifen und mit 25% MeOH in das beschriftete Rohr zu legen. Lassen Sie die Zange den Embryo nicht berühren.
- Inkubieren Sie Embryonen bei RT mit sanfter Erregung für 1 Stunde im Dunkeln.
- Entfernen Sie 25% MeOH aus der Röhre, wobei Sie darauf achten, den Embryo nicht zu berühren. 1 ml 50% MeOH pro Tube hinzufügen. Inkubieren Sie bei RT mit sanfter Agitation für 1 Stunde im Dunkeln.
- Entfernen Sie 50% MeOH aus der Röhre, wobei Sie darauf achten, den Embryo nicht zu berühren. 1 ml 75% MeOH pro Röhre hinzufügen. Inkubieren Sie bei RT mit sanfter Agitation für 1 Stunde im Dunkeln.
- Entfernen Sie 75% MeOH aus der Röhre, wobei Sie darauf achten, den Embryo(n) nicht zu berühren. 1 ml 100% MeOH pro Tube hinzufügen. Inkubieren Sie bei RT mit sanfter Agitation für 1 Stunde im Dunkeln. 100% MeOH zweimal waschen.
-
Clearing mit BABB
- Entfernen Sie 100% MeOH aus der Röhre, wobei Sie darauf achten, den Embryo nicht zu berühren. 1 ml 50% BABB pro Röhre hinzufügen. Inkubieren Sie bei RT mit sanfter Agitation für 1 Stunde im Dunkeln.
- Entfernen Sie 50% BABB aus dem Rohr, wobei Sie darauf achten, den Embryo nicht zu berühren. 1 ml 100% BABB pro Röhre hinzufügen. Inkubieren Sie bei RT mit sanfter Agitation für 1 Stunde im Dunkeln. 100% BABB zweimal waschen.
HINWEIS: (Optionaler Haltepunkt) Embryonen können etwa eine Woche lang in 100% BABB in Röhren verbleiben. Eine längere Lagerung führt dazu, dass BABB den Kunststoff von Rohren auflöst.
-
Montage von Embryonen für die Bildgebung
- Legen Sie einen Fast Well Stoßfänger auf einen 24 mm x 60 mm #1,5 Glasabdeckungsschlupf, indem Sie den Kunststoffkleber von einer Seite abschälen. Stellen Sie sicher, dass sich keine Luftblasen zwischen Deckelrutsch und Stoßstange befinden, indem Sie sanften Druck auf den Kunststoffkleber auf der Gummistoßstange ausüben. Etikettieren Sie den Deckzettel nach Embryonummer, Genotyp und Antikörpern, die für die Färbung verwendet werden.
HINWEIS: Jeder Abstandser kann zwischen die Abdeckungen gelegt werden, solange er dick genug ist, um das Zerkleinern oder Zerquetschen eines Embryos zu verhindern. Wir verwenden Fast Well Abstandshalter aufgrund ihrer Dicke und Bequemlichkeit, die Klebeflächen auf beiden Seiten des Abstandshalters zur Sicherung zu Abdeckungen umfasst. - Die 100% BABB vorsichtig herausleiten und aus dem Rohr entsorgen. Nach der Visualisierung des agarose-eingebetteten Embryos in der Röhre, verwenden Sie feine Zangen, um die Agarose aufzunehmen und den Embryo vorsichtig auf den Deckelschlupf im Fast Well zu übertragen - lassen Sie die Zange den Embryo nicht berühren.
- Entfernen Sie den zweiten Kunststoffkleber aus der Stoßstange und legen Sie den zweiten Deckelrutsch darauf. Entfernen Sie Luftblasen, indem Sie sanft auf den Deckelbeschlag drücken. Achten Sie darauf, das Glas nicht zu brechen.
HINWEIS: Proben können bei einer Dichtung bis zu einem Jahr flach in einem Diahalter im Dunkeln bei RT gelagert werden.
- Legen Sie einen Fast Well Stoßfänger auf einen 24 mm x 60 mm #1,5 Glasabdeckungsschlupf, indem Sie den Kunststoffkleber von einer Seite abschälen. Stellen Sie sicher, dass sich keine Luftblasen zwischen Deckelrutsch und Stoßstange befinden, indem Sie sanften Druck auf den Kunststoffkleber auf der Gummistoßstange ausüben. Etikettieren Sie den Deckzettel nach Embryonummer, Genotyp und Antikörpern, die für die Färbung verwendet werden.
6. Erfassung von Daten
HINWEIS: In den folgenden Schritten wird das Endothel der Rachenbögen 3, 4 und 6 mittels konfokaler Mikroskopie abgebildet.
-
Positionierung von Dias auf der Mikroskopbühne
- Um Embryonen abzubilden, verwenden Sie ein konfokales Mikroskop, das mit einem 20-fachen Wasser-Eintauchobjektiv, einer numerischen Blende 0,95, einem Arbeitsabstand von 0,95 mm und der NIS-Elements AR 5.11.01 64-Bit-Software ausgestattet ist.
- Mit Weitfeldfluoreszenz, visuell lokalisieren sie die Pharyngealbögen. Zentrieren Sie die Feldansicht um die4. PAA.
- Wenn das Sichtfeld des Ziels nicht den gesamten Rachenbogenbereich erfasst, nehmen Sie eine große Fläche von Bildern mit 1% Überlappung auf und nähen Sie sie. Um die Bewegung der Probe während der Erfassung des großen Bildes zu verhindern, befestigen Sie vorsichtig die Deckelschlupf-Montage auf der Bühne mit Formton.
-
Einrichten von Erfassungsparametern
- Stellen Sie die Lochgröße auf 1,0 ein.
- Legen Sie unter der Registerkarte ND-Erfassung die oberen und unteren Grenzen der Bildgebung mithilfe der Grobeinstellung fest. Stellen Sie die Z-Schrittgröße gemäß den Softwarespezifikationen ein. Bestimmen Sie die Dicke, die durch den Arbeitsabstand des Ziels und die Klarheit der Probe abgebildet werden kann.
- Aufgrund der Dicke des Embryos, passen Sie die Verstärkung im gesamten Z-Stack an. Stellen Sie die Laserintensität und -verstärkung in der Mitte des Z-Stacks für jeden Kanal (405, 488 und 555) ein und weisen Sie Werte unter der Registerkarte Z-Intensitätskorrektur zu. Legen Sie die gleichen Werte für das untere Segment fest.
- Scrollen Sie durch den Embryo, bis Fluoreszenzsignale dimmer erscheinen. Erhöhen Sie die Verstärkung jedes Kanals, bis die Signalintensität ähnlich dem vorherigen Segment erscheint. Weisen Sie den neuen Wert unter der Registerkarte Z-Intensitätskorrektur zu. Wiederholen Sie dies, bis z-stack abgeschlossen ist. Importieren Sie die Einstellungen zurück in die ND-Erfassung.
- Führen Sie den Scan mit der Option Z-Korrektur ausführen aus.
7. Analyse mit der Imaris-Software
HINWEIS: In diesen Schritten werden konfokale Bilder mit der Mikrokopiebildanalyse-Software Imaris Version 9.2.0 analysiert. Während dieser Analyse werden zunächst Bereiche von Interesse ausgewählt, die durch Erstellen von Flächen analysiert werden sollen. Als Nächstes verwenden Mask wir die Mask-Funktion, um diese Bereiche visuell zu trennen. Schließlich werden wir die Spot-Funktion verwenden, um die Anzahl der ECs innerhalb jeder Region von Interesse zu quantifizieren.
- Je nach der in Schritt 6 verwendeten Bildverarbeitungssoftware, konvertieren Sie Bilder mit Imaris File Converterin .ims .
- Öffnen Sie die .ims-Dateien. Bild unter Kamera/Etiketten auf Orthogonal festlegen | Kamera-Typ-Bedienfeld.
- Suchen Sie die PAAs, und richten Sie das Bild für die Oberfläche aus.
HINWEIS: Wenn die Dateien zum ersten Mal geöffnet werden, werden sie als 3D-Kompilierung aller abgebildeten Slices angezeigt. In diesem Schritt werden die PAAs durch Erstellen des 3D-Bildes in ein 2D-Bild ermittelt. Das 2D-Bild ermöglicht dann, dass die PAAs für die Analyse richtig ausgerichtet werden.- Deaktivieren Sie im Bedienfeld Eigenschaften Volume. Klicken Sie im Eigenschaftenbedienfeld auf Neue Ortho-Slicer hinzufügen. Legen Sie die Slice-Ausrichtung auf die XY-Ebenefest. Verwenden Sie die Slice-Position, um durch das Bild zu scrollen, bis die PAAs gefunden werden.
- Wenn die PAAs nicht parallel zum oberen und unteren Rand des Bildes sind, drehen Sie das Bild mit dem Mauszeiger frei, sodass PAAs von links nach rechts über den Bildschirm laufen. Wählen Sie im Dropdown-Menü Bildverarbeitung Die Option Freies Drehen und klicken Sie auf OK.
- Oberfläche des 3. Pharyngealbogens (Abbildung 2A, B - B")
HINWEIS: In diesen Schritten werden die Pharyngealbögen und PAAs mit dem Werkzeug Oberfläche verfolgt, um einen "oberflächengebundenen" Bereich von Interesse zu generieren. Dadurch kann jede Region, die von Interesse ist, visuell vom umgebenden Gewebe isoliert werden. Hierin beschreiben wir die Schritte zur Oberfläche und analysieren die endotheliale Komponenten des3. Pharyngealbogens. Die Pharyngealbögen 4 und 6 werden ähnlich analysiert.- Um das Endothel im gesamten3. Pharyngealbogen anzuschaffen, klicken Sie auf die Schaltfläche Neue Oberfläche hinzufügen unter dem Eigenschaftenbedienfeld. Doppelklicken Sie auf Surface 1 und benennen Sie die neue Oberfläche in "3rd Pharyngeal Arch" um.
- Wählen Sie Automatische Erstellung überspringen, manuell bearbeiten. Legen Sie die Oberflächenausrichtung auf die YZ-Ebene fest (koronale Ausrichtung). Verwenden Sie die Slice-Position, um die3. Pharyngealbogen-Oberflächenebene an die Stelle zu platzieren, an der sich die3. PAA und Dorsal Aorta verbinden.
- Drehen Sie das Bild so, dass die3. Pharyngealbogen-Oberflächenebene sichtbar ist. Deaktivieren Sie Ortho Slicer 1.
- Unter der Auslosung | Kontur | Modus-Registerkarte, wählen Sie die Funktion "Entfernungszeichnungsmodus". Passen Sie bei Bedarf die Parametereinstellungen an. Konsistente Oberflächenparameter zwischen Denbändern beibehalten. In diesem Beispiel beträgt der Scheitelpunktabstand 10 m.
- Um mit der Oberfläche zu beginnen, drücken Sie die Esc-Taste und klicken Sie dann auf die Schaltfläche Zeichnen. Verfolgen Sie den Umfang des3. Pharyngealbogens mit dem Mauszeiger. Verwenden Sie die Slice-Position, um 10-25 Slices zu verschieben. Verfolgen Sie den Umfang des Rachenbogens. Wiederholen Sie dies, bis der Rachenbogen vollständig zurückverfolgt ist.
- Um die Oberfläche des verfolgten Bereichs zu generieren, wählen Sie im Eigenschaftenbedienfeld die Schaltfläche Fläche erstellen aus.
- Oberfläche der 3. PAA (Abbildung 2C - C")
- Um das Endothel der3. PAA an die Oberfläche zu stellen, schalten Sie zunächst den oberflächenbelagten Bereich aus Schritt 7.4 aus, indem Sie die Auswahl der3. Pharyngeal Arch-Oberflächenbox aufbrechen. Klicken Sie dann erneut auf die Schaltfläche Neues Surface hinzufügen. Doppelklicken Sie auf Surface 1 und benennen Sie die neue Oberfläche in "3rd PAA" um.
- Wählen Sie Automatische Erstellung überspringen, manuell bearbeiten. Legen Sie die Oberflächenausrichtung auf die YZ-Ebene fest (koronale Ausrichtung). Verwenden Sie die Slice-Position, um die3. PAA-Oberflächenebene an die Stelle zu platzieren, an der sich die3. PAA und Dorsal Aorta verbinden, und wiederholen Sie dann die Schritte 7.4.
- Maskierung von oberflächenbebauten Strukturen
HINWEIS: In den folgenden Schritten wird jeder angezeigte Bereich maskiert. Die Maskierung ermöglicht es, den Interessenbereich visuell vom Rest des abgebildeten Gewebes zu unterscheiden und ermöglicht die Quantifizierung dieser unterschiedlichen Interessenstrukturen. Im Folgenden beschreiben wir die Schritte in Imaris zur Visualisierung und Analyse des PAA-Endothels sowie des Plexus - der kleineren Vaskulatur, die die PAAs innerhalb der Pharyngealbögen umgibt. In diesen Schritten wird die3. PAA-Oberfläche maskiert, um die PAAs nur mit der in Abschnitt 7.7 beschriebenen Spot-Funktion zu visualisieren und analysen durchzuführen.- Wählen Sie Volume aus, um alle maskierten Kanäle zu visualisieren. Drücken Sie die Esc-Taste, um das Bild zu drehen und das Bild in eine XY-Position zu positionieren.
- Wählen Sie auf der Registerkarte Bearbeiten die Option Maskenauswahl für die3. PAA aus. Wählen Sie den DAPI-Kanal aus, und klicken Sie auf OK. Wiederholen Sie dies für die verbleibenden Kanäle.
- Drücken Sie auf der Tastatur Strg + D, um das Bedienfeld "Einstellungen anzeigen" anzuzeigen. Wählen Sie jeden neuen Kanal aus, und benennen Sie ihn um, um deutlich zu machen, was jeder Kanal anzeigt. Dies führt beispielsweise zu drei neuen Kanälen: "3rd PAA DAPI", "3rd PAA ERG" und "3rd PAA VEGFR2".
HINWEIS: In den Schritten 7.6.4-7.6.7 werden wir Kanäle nur für Denkbogenplexus erstellen. - Um den Endothelplexus getrennt von der PAA zu visualisieren, wählen wir zunächst Maskenauswahl für die3. PAA-Oberfläche aus. Wählen Sie den DAPI-Kanal aus. Deaktivieren Sie die Option Voxel außen auswählen zu und aktivieren Voxel innerhalb der Fläche zu Schaltflächen auswählen, setzen Sie Voxel innerhalb der Fläche auf Null auswählen. Klicken Sie auf OK.
- Wiederholen Sie dies für die verbleibenden Kanäle. Bei diesem Vorgang wird die Region, die die PAA enthält, von den neuen maskierten Kanälen ausgeschlossen. Benennen Sie Kanäle um, um deutlich zu machen, was jeder Kanal anzeigt. Dies führt beispielsweise zu drei neuen Kanälen: "Non-PAA DAPI", "Non-PAA ERG" und "Non-PAA VEGFR2".
- Um endotheliale Plexus innerhalb des3. Pharyngealbogens zu visualisieren, wählen Sie Maskenauswahl unter der Registerkarte Bearbeiten für die3. Pharyngeal Arch-Oberfläche aus. Wählen Sie den Nicht-PAA-DAPI-Kanal aus. Klicken Sie auf OK.
- Wiederholen Sie dies für die verbleibenden Nicht-PAA-Kanäle. Benennen Sie Kanäle um, um deutlich zu machen, was jeder Kanal anzeigt. Dies führt beispielsweise zu drei neuen Kanälen: "Plexus DAPI", "Plexus ERG" und "Plexus VEGFR2".
- Quantifizierung der EG-Nummern
HINWEIS: Der Ausdruck von ERG markiert endotheliale Kerne, die es bequem machen, EC-Zahlen zu quantifizieren. In diesen Schritten wird die Anzahl der ECs mithilfe der Spot-Funktion quantifiziert, um für jede EG, die durch ERG-Expression in PAA und Plexus gekennzeichnet ist, einen Spot zu generieren. In Abschnitt 7.7 werden für jede ERG-positive Zelle in der maskierten PAA Flecken erzeugt, gefolgt von der Abwahl von Spots in ERG-positiven, VEGFR2-negativen Zellen.- Drücken Sie auf der Tastatur Strg + D, um das Bedienfeld "Einstellungen anzeigen" anzuzeigen. Schalten Sie alle Kanäle außer PAA ERG aus.
- Klicken Sie unter der Registerkarte Eigenschaften auf die Schaltfläche Neue Flecken hinzufügen. Klicken Sie auf Spots 1 und benennen Sie es in "PAA Gesamtzahl der ECs" um. Klicken Sie auf den blauen Pfeil. Wählen Sie für Source Channelden PAA ERG-Kanal aus. Passen Sie den geschätzten XY-Durchmesser auf 4 m an. Fahren Sie mit dem nächsten Panel fort, indem Sie auf den blauen Pfeil klicken.
- Passen Sie die Anzahl der mit der Gleitskala sichtbaren Flecken an, um sicherzustellen, dass jeder EC-Kern (gekennzeichnet durch ERG-Ausdruck) durch einen Punkt dargestellt wird. Klicken Sie auf den grünen Doppelpfeil.
- Schalten Sie den PAA ERG-Kanal in der Display-Einstellung aus. Schalten Sie den PAA VEGFR2-Kanal ein, um PAA-Endothel zu visualisieren.
- Um die Anzahl der ECs genau zu quantifizieren, stellen wir sicher, dass jeder Punkt sowohl EC-Marker, ERG als auch VEGFR2 ausdrückt. Wählen Sie dazu Surface of Object unter der Registerkarte Bearbeiten | Bedienfeld Hinzufügen/Löschen. Drücken Sie die Esc-Taste und löschen Sie alle Punkte, die nicht VEGFR2 positiv sind, indem Sie die Verschiebung gedrückt halten und die Stelle auswählen.
ANMERKUNG: Im folgenden Schritt werden für jede ERG-positive Zelle im maskierten Plexus Flecken erzeugt, gefolgt von der Abwahl von Spots in ERG-positiven, VEGFR2-negativen Zellen. - Klicken Sie unter der Registerkarte Eigenschaften auf die Schaltfläche Neue Flecken hinzufügen. Wählen Sie Spots 1 aus und benennen Sie sie in "Plexus Gesamtzahl der ECs" um. Klicken Sie auf den blauen Pfeil. Wählen Sie für Source Channel den Plexus ERG-Kanal aus. Passen Sie den geschätzten XY-Durchmesser auf 4 m an. Wiederholen Sie die Schritte 7.7.1 - 7.7.5 für Plexus Gesamtanzahl der ECs.
- Klicken Sie auf die Registerkarte Statistik jeder Spotfunktion, um die Gesamtzahl der ECs im3. PAA und Pharyngealbogenplexus zu bestimmen.
- Wiederholen Sie die Schritte 7.7.1 - 7.7.6 für die verbleibenden PAAs und den Pharyngealbogenplexus.
Ergebnisse
Das hier vorgestellte Immunfluoreszenzprotokoll mit ganzer Montaterzeugtkeit liefert klare und saubere Ergebnisse, die eine 3D-Rekonstruktion des Pharyngealbogenendothel ermöglichen, wie in Abbildung 1Adargestellt. Es ist wichtig, Embryonen für eine ausreichende Zeit in jeder Antikörperlösung zu inkubieren, um eine vollständige Penetration durch die Probe zu gewährleisten, sowie, gründlich waschen Embryonen nach Derinkubation. In Abbildung 1Bwerden große, helle Punkte als Folge von Partikeln entweder im Antikörper oder in blockierenden Pufferlösungen angezeigt. Wir haben festgestellt, dass Zentrifugieren jeder Lösung vor der Verwendung und längere Zeiträume von PBST wäresst, nachdem jede Antikörperinkubation dieses Problem löst.
Abbildung 2 zeigt das Verfahren zur Oberfläche eines Pharyngealbogens und eines PAA zur Analyse, wie in Abschnitt 7 des Protokolls beschrieben. Mit der Maskierten-Funktion ermöglicht die Imaris-Software, flächensuchte Bereiche visuell voneinander zu trennen und unabhängig zu analysieren.
Abbildung 3 zeigt die individuelle Maskierung verschiedener Gefäßkompartimente in den Rachenbögen: die PAA (Abbildung 3A, B, C) und den Plexus ( Abbildung3A', B', C'). Die Maskierung ermöglicht die Analyse und Quantifizierung von EG-Nummern in jeder Struktur separat. In Abbildung 3C-C'wird das Spot-Feature verwendet, um die Gesamtzahl der ECs sowohl in der PAA als auch im Plexus zu quantifizieren, indem jedem Kern, der ERG ausdrückt, ein einziger Punkt zugewiesen wird. Es ist wichtig zu beachten, dass der Für die Spot-Funktion verwendete Algorithmus so konzipiert ist, dass ein Punkt für jedes Pixel einer angegebenen Größe generiert wird. ERG, das hier als Marker von EC-Kernen verwendet wird, wird auch in neuralen Kammzellen8exprimiert; neurale Kammzellen drücken NICHT VEGFR2 aus. Abbildung 3D zeigt ein Beispiel für einen ERG-positiven (grünen), VEGFR2-negativen (rosa) Spot, der von der Imaris Spot-Funktion erzeugt wurde. Daher ist es wichtig, zu überprüfen, ob jeder Punkt eine einzelne EG darstellt und sowohl mit ERG als auch mit VEGFR2 gekennzeichnet ist.
| Schritt | Zeit | Temperatur | |
| 1 | PBST Wash/Permeabilisierung | 24 h oder O/N | 4 °C |
| 2 | Blocking Buffer | 25 h oder O/N | 4 °C |
| 3 | Primärer Antikörper | 4-5 Tage | 4 °C |
| 4 | PBST Waschen | 4-5 mal täglich für 2 Tage | RT (oder 4 °C bei O/N) |
| 5 | Sekundärer Antikörper | 4-5 Tage | 4 °C |
| 6 | PBST Waschen | 4-5 mal täglich für 2 Tage | RT (oder 4 °C bei O/N) |
| 7 | Einbinden | N/A | Rt |
| 8 | Methanol-Dehydrierung und BABB | 1 Stunde pro Schritt | Rt |
Tabelle 1: Übersicht über das gesamte Mount-Immunfluoreszenzprotokoll. O/N - über Nacht; RT - Raumtemperatur.
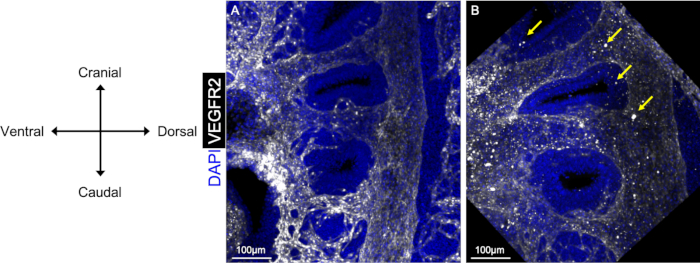
Abbildung 1: Vergleich von sauberen und schmutzigen Bildern nach ganzer Immunfluoreszenz. Sagittale Ansichten des E10.5-Embryos zeigen die Verwendung von Anti-VEGFR2-Antikörpern (weiß), um das PAA-Endothel zu visualisieren. Embryonen, die gründlich mit PBST-Post-Antikörper-Inkubationen gewaschen werden (A) haben ein höheres Signal-Rausch-Verhältnis und erzeugen ein saubereres Bild, im Vergleich zu Embryonen, die nicht gründlich gewaschen werden (B). Pfeile in B zeigen Bereiche von Lärm/Schmutz, die im Bild erschienen sind, wenn ein Embryo nicht gründlich gewaschen oder die Antikörperlösung nicht zentrifugiert wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Oberfläche des Rachenbogens (PA) und der PAA. Eine 2D-Sagittalansicht (A) wird verwendet, um die Position der PAAs im konfokalen Bild zu identifizieren. Ein koronaler Orthoschneider (A, gelbe Linie) wird durch die PAAs platziert. Der Pharyngealbogen (B) und PAA (C) werden dann in der koronalen Ausrichtung mit dem Werkzeug Entfernungszeichnung in Imaris angezeigt. Das Werkzeug "Entfernungszeichnung", das auf 10 m festgelegt ist, wird verwendet, um den Umfang des3. Pharyngealbogens (B) oder der PAA (C) nachzuverfolgen. Umrisse werden alle 10-25 Scheiben durch den gesamten Bogen gezeichnet (B', C'). Umrisse werden kombiniert, um eine 3D-Oberfläche des Rachenbogens (B") oder der PAA (C") zu erzeugen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
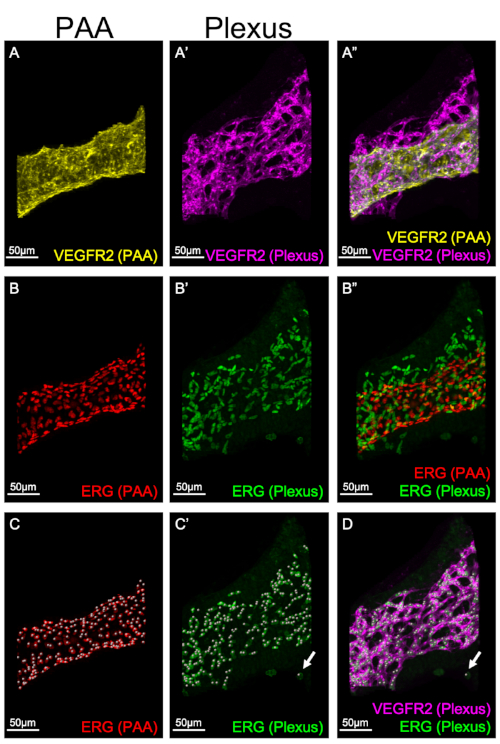
Abbildung 3: Quantifizierung der EG-Nummern in einer PAA und einem Plexus. 3D-Rekonstruktionen werden verwendet, um die Gefäßstruktur und den Ausdruck von EC-Markern in einer PAA oder in einem EC-Plexus separat zu visualisieren. Die Panels A-A' zeigen den Ausdruck von VEGFR2 in der PAA (A, gelb) und im Plexus (A', rosa). Panel A" veranschaulicht eine Zusammenführung des PAA- und Plexus-VEGFR2-Ausdrucks. Die Panels B-B' zeigen den Ausdruck von ERG in der PAA (B, rot) und im Plexus (B', grün). Panel B" veranschaulicht die Zusammenführung von PAA und Plexus. C-C'. Die Spot-Funktion in Imaris wird verwendet, um die Anzahl der ECs in der PAA oder im Plexus zu quantifizieren. Jeder ERG-positiven Zelle in der PAA (C, rot) oder plexus (C', grün) wird ein einzelner Punkt zugewiesen, um eine einzelne EG zu markieren. Der Pfeil in C'-D zeigt einen BEISPIEL ERG-positiven, VEGFR2-negativen Punkt im Plexus, der von der Imaris Spot-Funktion erzeugt wurde. Dieser Punkt ist von der Quantifizierung ausgeschlossen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Möglichkeit, das Endothel in Mausembryonen in 3D zu visualisieren, hat neue Einblicke in ihre Entwicklung 3gegeben. Hier stellen wir ein Protokoll vor, das eine hochauflösende 3D-Bildgebung von Embryonen, die Visualisierung der vaskulären Konnektivität und quantitative Analysen der PAA-Bildung ermöglicht. Dieses Protokoll kann verwendet werden, um zu sehen, wie genetische Veränderungen oder Umweltbeleidigungen die ENTWICKLUNG der PAA beeinflussen. Das hier beschriebene Verfahren verwendet Antikörper gegen VEGFR2 und ERG, um die PAA-Bildung zu visualisieren und die EC-Nummer zu quantifizieren; jedoch können zusätzliche Antikörper verwendet werden, um andere Aspekte der Bogenschlagaderentwicklung zu visualisieren und zu analysieren, wie z. B. die Rekrutierung von Neuronkflörzodern oder die Differenzierung von glatten Muskelzellen. Wenn dieses Verfahren in früheren Stadien der Embryogenese angewendet werden soll, ist es wichtig zu beachten, dass einige Antigene (z. B. ERG), die in diesem Protokoll nachgewiesen wurden, möglicherweise noch nicht exprimiert werden. Andere nukleare Flecken wie DAPI oder DRAQ5 oder die Linienkennzeichnung mit kernkraftmarkierten Tracern können zur Quantifizierung der EC-Nummer verwendet werden.
Es gibt mehrere kritische Schritte innerhalb des Protokolls: sicherstellen, dass 1) Embryonen nicht zwischen Lösungsänderungen ausgetrocknet werden; 2) Embryonen werden nach Antikörper-Inkubationen gründlich gewaschen; und 3) dass Embryonen mit MeOH vollständig dehydriert werden, bevor gewebeclearing mit BABB.
Methanolwäsche vor der Gewebeklärung dient zwei Zwecken: die Fluoreszenz durch die Expression fluoreszierender Proteine (z. B. die Expression von EGFP oder tdTomato, die für die Linienverfolgung verwendet wird) im Embryo zu beseitigen und das Gewebe zu dehydrieren. Die Eliminierung der Fluoreszenz aus fluoreszierenden Proteinen ermöglicht die Verwendung einer beliebigen Kombination von Fluorophoren für die Bildgebung. Antikörper gegen EGFP und TdTomato (Kirsche) können verwendet werden, um die Expression dieser fluoreszierenden Proteine zu visualisieren. Alternativ kann MeOH durch Tetrahydrofuran ersetzt werden, um die Fluoreszenz fluoreszierender Proteine zu erhalten9.
Wir haben festgestellt, dass Embryonen, die vor der BABB-Räumung nicht richtig dehydriert wurden, aufgrund der Lichtstreuung schwer abzubilden sind. BABB ist eine hydrophobe Lösung, die eine vollständige Austrocknung in einem organischen Lösungsmittel erfordert, um das undurchsichtige Gewebe zu entfernen. Vollständiges Clearing stellt die Fähigkeit sicher, Bilder auf den tiefsten möglichen Ebenen innerhalb des Embryos10,11zu erhalten. In diesem Protokoll haben wir ein 20-faches Wassertauchziel verwendet, aufgrund seiner langen Arbeitsdistanz und Verfügbarkeit zum Zeitpunkt unserer Experimente. Öl-Immersion-Ziele sind besser geeignet für dieses Protokoll, da BABB und Öl haben engere Brechungsindizes als Wasser und BABB. Trotz des Unterschieds im Brechungsindex bot das in diesem Protokoll verwendete Wasser-Immersion-Objektiv eine hervorragende Bildqualität.
Es gibt einige Einschränkungen dieses Protokolls. BABB Clearing hier verwendet wird, ist giftig und korrosiv11,12,13. BABB löst Kleber und Kunststoffe auf. Wenn Proben während der Bildgebung nicht richtig behandelt werden, kann die Mikroskopobjektivlinse durch BABB beschädigt werden, die durch Risse im Coverslip oder eine gebrochene Dichtung zwischen dem Fast Well Stoßfänger und dem Coverslip aus der Probe entweichen kann. Clearing-Methoden, die keine organischen Lösungsmittel wie CLARITY verwenden, können als Alternativen verwendet werden10,11,14. Die Brechungsindex-Matching-Lösung von CLARITY verfügt über einen Brechungsindex ähnlich dem von Wasser, was sie zu einer geeigneten Clearingmethode macht, wenn ein Wasser-Eintauchziel verwendet wird. Eine zusätzliche Einschränkung dieses Protokolls besteht darin, dass es nur an nicht lebenden Geweben durchgeführt werden kann, wodurch seine Anwendung für Live-Bildgebung verhindert wird.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Brianna Alexander, Caolan O'Donnell und Michael Warkala für die sorgfältige Lektüre und Bearbeitung dieses Manuskripts. Diese Arbeit wurde durch die Finanzierung durch das National Heart, Lung and Blood Institute des NIH R01 HL103920, R01 HL134935, R21 OD025323-01 bis SA unterstützt; AJR wird unterstützt von NHLBI HL103920-08S1 und dem National Institute of Arthritis and Musculoskeletal and Skin Diseases Training Grant T32052283-11.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x PBS | MP Biomedicals | PBS10X02 | |
| 20x water immersion objective | Nikon | MRD77200 | |
| Agarose | Bio-Rad Laboratories | 1613101 | |
| Alexa Fluor 488 anti-goat | Invitrogen | A-11055 | |
| Alexa Fluor 555 anti-mouse | Invitrogen | A-31570 | |
| Analysis Software | Imaris 9.2.0 | ||
| Benzyl Alcohol | Sigma-Aldrich | 305197 | |
| Benzyl Benzoate | Sigma-Aldrich | 8.18701.0100 | |
| Cover Slips | VWR | 16004-312 | |
| DAPI (5 mg/mL stock) | Fisher Scientific | D3571 | |
| Eppendorf Tubes (2.0 mL) | Fisher Scientific | 05-408-138 | |
| Ethanol | VWR | 89370-084 | |
| Falcon tubes (50 mL) | Corning | 352098 | |
| Fast wells | Grace Bio Labs | 664113 | |
| Forceps | Roboz | RS-5015 | |
| Goat anti-VEGFR2 | R&D Systems, Inc. | AF644 | |
| Methanol | VWR | BDH1135-4LP | |
| Microscope | Nikon | A1HD25 | |
| Mouse anti-ERG | Abcam | ab214341 | |
| Normal Donkey Serum | Sigma-Aldrich | D9663 | |
| Paraformaldehyde | Electron Microscopy Sciences | 15710 | |
| Pasteur pipets | Fisher Scientific | 13-678-20D | |
| Petri dishes (35 mm) | Genesee Scientific | 32-103 | |
| Petri dishes (60 mm) | Genesee Scientific | 32-105 | |
| Plastic Molds | VWR | 18000-128 | |
| Scapels | Exelint International Co. | 29552 | |
| Triton-X-100 | Fisher Scientific | BP 151-500 |
Referenzen
- Hiruma, T., Nakajima, Y., Nakamura, H. Development of pharyngeal arch arteries in early mouse embryo. Journal of Anatomy. 201 (1), 15-29 (2002).
- Hutson, M. R., Kirby, M. L. Model systems for the study of heart development and disease Cardiac neural crest and conotruncal malformations. Seminars in Cell & Developmental Biology. 18 (1), 101-110 (2007).
- Wang, X., et al. Endothelium in the pharyngeal arches 3, 4 and 6 is derived from the second heart field. Developmental Biology. 421 (2), 108-117 (2017).
- Jerome, L. A., Papaioannou, V. E. DiGeorge syndrome phenotype in mice mutant for the T-box gene, Tbx1. Nature Genetics. 27 (3), 286-291 (2001).
- Lindsay, E. A., et al. Tbx1 haploinsufficieny in the DiGeorge syndrome region causes aortic arch defects in mice. Nature. 410 (6824), 97-101 (2001).
- Weninger, W., et al. Visualising the Cardiovascular System of Embryos of Biomedical Model Organisms with High Resolution Episcopic Microscopy (HREM). Journal of Cardiovascular Development and Disease. 5 (4), 58(2018).
- Phillips, H. M., et al. Pax9 is required for cardiovascular development and interacts with Tbx1 in the pharyngeal endoderm to control 4th pharyngeal arch artery morphogenesis. Development. 146 (18), (2019).
- Vlaeminck-Guillem, V., et al. The Ets family member Erg gene is expressed in mesodermal tissues and neural crests at fundamental steps during mouse embryogenesis. Mechanisms of Development. 91 (1-2), 331-335 (2000).
- Ertürk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-217 (2012).
- Azaripour, A., et al. A survey of clearing techniques for 3D imaging of tissues with special reference to connective tissue. Progress in Histochemistry and Cytochemistry. 51 (2), 9-23 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying Tissue Clearing. Cell. 162 (2), 246-257 (2015).
- Becker, K., Jährling, N., Saghafi, S., Weiler, R., Dodt, H. U. Chemical Clearing and Dehydration of GFP Expressing Mouse Brains. PLoS One. 7 (3), e33916(2012).
- Ertürk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten