Method Article
Visualizzazione e analisi delle arterie dell'arco faringo utilizzando immunohistochimica a monte intero e ricostruzione 3D
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per visualizzare e analizzare le arterie dell'arco faringeo 3, 4 e 6 degli embrioni di topo usando immunofluorescenza a montaggio intero, radura dei tessuti, microscopia confocale e ricostruzione 3D.
Abstract
La formazione o il rimodellamento improprio delle arterie dell'arco faringeo (PAA) 3, 4 e 6 contribuiscono ad alcune delle forme più gravi di malattia cardiaca congenita. Per studiare la formazione di PAA, abbiamo sviluppato un protocollo che utilizza immunofluorescenza a montaggio intero accoppiato con la radura del tessuto alcool/benzyl benzoate (BABB) e la microscopia confocale. Ciò consente la visualizzazione dell'endotelio dell'arco faringeo ad una risoluzione cellulare fine e la connettività 3D della vascolatura. Utilizzando il software, abbiamo stabilito un protocollo per quantificare il numero di cellule endoteliali (EC) nelle PAA, così come il numero di EC all'interno del plesso vascolare che circonda i PAA all'interno degli archi pharyngeal 3, 4 e 6. Se applicata all'intero embrione, questa metodologia fornisce una visualizzazione completa e un'analisi quantitativa della vascolatura embrionale.
Introduzione
Durante l'embriogenesi del topo, le arterie dell'arco fantasma (PAA) sorgono come coppie simmetriche e bilaterali di arterie che collegano il cuore con le aortae dorsali1. Man mano che l'embrione si sviluppa, la prima e la seconda coppia di PAA regrediscono, mentre le 3rd,4the 6PAA subiscono una serie di eventi di rimodellamento asimmetrici per formare le arterie dell'arco aortico2.
I PAA 3, 4 e 6 si sviluppano attraverso la vasculogenesi, che è la formazione de novo dei vasi sanguigni3. I difetti nella formazione o nel rimodellamento di queste arterie ad arco danno origine a vari difetti cardiaci congeniti, come quelli osservati nei pazienti con sindrome di DiGeorge4,5. Pertanto, comprendere i meccanismi che regolano lo sviluppo dei PAA può portare a una migliore comprensione dell'eziologia delle malattie cardiache congenite (CHD).
Gli attuali approcci per la visualizzazione e l'analisi dello sviluppo PAA includono l'immunofluorescenza delle sezioni tissutali, i calchi vascolare, l'iniezione di inchiostro dell'India, la microscopia episcopica ad alta risoluzione e/o l'immunohistochimica a monte intero1,4,5,6,7. Qui, descriviamo un protocollo che combina l'immunofluorescenza a montaggio intero, la microscopia confocale e il rendering delle immagini 3D al fine di raccogliere, analizzare e quantificare i dati volumetrici, la connettività vascolare e l'identità cellulare. Inoltre, dettagliamo un metodo per compartimentare e quantificare il numero di EC in ogni arco faringeo come mezzo per studiare la formazione del plesso vascolare dell'arco faringeo e il suo rimodellamento nei PAA. Mentre questo protocollo è progettato per l'analisi dello sviluppo PAA, può essere utilizzato per analizzare altre reti vascolari in via di sviluppo.
Protocollo
L'uso e le procedure sugli animali sono stati approvati dal Comitato istituzionale per la cura e l'uso degli animali della Rutgers University.
1. Preparazione delle soluzioni
- Preparare 1 L di fosfato bufferizzato salina con 0.1% Triton-X-100 (PBST) e sterilizzare il filtro. Questa soluzione può essere conservata a temperatura ambiente (RT) per almeno un anno.
- Preparare 600 l di buffer di blocco costituito dal 10% del siero normale dell'asino in PBST. Rendere questa soluzione fresca ogni volta.
- Preparare 50 mL delle seguenti diluizioni di metanolo (MeOH) in una cappa a flusso: 25% MeOH in acqua deionizzata (dH2O), 50% MeOH in dH2O, e 75% MeOH in dH2O. Vortex per mescolare. Store presso RT.
- Preparare 50 mL delle seguenti soluzioni benzoate benzoate benzoate benzolo-benzyl (BABB) in tubi conici da 50 mL.
- Per 100% BABB, aggiungere 32 mL di benzyl benzoato a 16 mL di alcol benzile (2:1 volume per rapporto volume).
- Per il 50% BABB, aggiungere 16 mL di benzyl benzoato e 8 mL di alcool benzile a 24 mL di MeOH.
- Coprire i tubi conici in un foglio di alluminio per proteggersi dalla luce. Queste soluzioni possono essere archiviate in RT per un massimo di un anno.
ATTENZIONE: BabB è tossico e corrosivo. Deve essere maneggiato ed eliminato secondo MSDS.
2. Dissezione e fissazione dell'embrione
NOTA: Questo protocollo è adatto per gli embrioni di topo E9.5 ed E10.5 (maschio o femmina) isolati da qualsiasi ceppo di topo. Per gli embrioni più giovani e più anziani, i tempi di incubazione devono essere determinati sperimentalmente per massimizzare il rapporto segnale-rumore del segnale di fluorescenza.
- Riempire un piatto Petri da 35 mm e uno da 60 mm con 1x PBS e mettere sul ghiaccio fino a quando necessario.
- Eutanasia di un topo incinta tramite inalazione di CO2. Eseguire la lussazione cervicale come misura secondaria di eutanasia.
- Pulire l'area addominale della diga con il 70% di etanolo. Pizzicare l'area addominale usando pinze e fare un'incisione a V usando forbici chirurgiche a partire dalla base della parete addominale alla linea mediana; continuare ad aprire la cavità toracica. Sollevare il tessuto addominale e spostare l'intestino di lato per esporre le corna uterine.
- Fai un taglio alla base del canale vaginale e con le pinze, allontana l'utero dalla diga. Fare un taglio aggiuntivo ad ogni ovaia per liberare l'utero. Trasferire l'utero in uno dei piatti Petri da 60 mm contenenti 1x PBS freddi.
- Utilizzando forbici dritte, tagliare la parete uterina tra ogni sito di impianto. Prendere una decidua con un tubo di vetro e trasferirlo nel piatto Petri da 35 mm con 1x PBS. Sotto un microscopio di dissezione, inserire forbici dritte nello spazio tra la decidua e la parete uterina. Tagliare e rimuovere la parete uterina.
- Con le pinze sottili, rimuovere le membrane decidua e Reichert dall'embrione facendo con attenzione incisioni trasversali lungo il tessuto e tirando il tessuto lontano dal sacco del tuorlo. Rimuovere il sacco del tuorlo e il sacco amniotico tirando accuratamente il tessuto lontano dall'embrione e facendo tagli all'allantois e alla vena ombelicale.
NOTA: le sacche di tuorlo possono essere utilizzate per la genotipizzazione degli embrioni. - Trasferire ogni embrione con un tubo di vetro in singoli tubi da 2 mL riempiti con 1 mL di 1x PBS. Etichettare ogni tubo con un identificatore univoco.
- Per fissare gli embrioni, rimuovere con attenzione il 1x PBS e aggiungere la soluzione 4% paraformaldeide (PFA) in 1x PBS. Incubare a 4 gradi centigradi con agitazione delicata durante la notte.
NOTA: la fissazione PFA del 4% è adatta agli anticorpi menzionati in questo protocollo. Tuttavia, le procedure di fissazione dovrebbero essere ottimizzate per ulteriori anticorpi.
3. Colorazione embrionale
NOTA: In questa sezione, gli embrioni sono permeabilizzati e macchiati con anticorpi primari e secondari. Poiché lo sviluppo del PAA procede rapidamente, le differenze nello stadio embrionale influenzeranno notevolmente l'analisi a valle. Pertanto, gli embrioni devono essere abbinati all'età contando attentamente le somiti per abbinare coppie di controllo e mutanti prima di ulteriori manipolazioni.
- Per lavare gli embrioni, rimuovere con cura il 4% di PFA e aggiungere 1x PBS. Invertire delicatamente i tubi più volte. Posizionare i tubi a destra verso l'alto e lasciare che l'embrione(i) affondere. Ripetere il lavaggio 3 volte. Posizionare i tubi con embrioni sul ghiaccio.
NOTA: (punto di sosta opzionale) Dopo i fumi, gli embrioni possono essere disidratati in serie classificate di MeOH per 30 min per diluizione, come nella sezione 1.3, e conservati a -20 gradi centigradi nel 100% MeOH per un uso successivo per un massimo di 6 mesi. - Per gli embrioni E10.5, utilizzare un pipet di vetro per trasferire un embrione in un piatto Petri da 35 mm riempito con 1 x PBS refrigerato. Pizzicare con attenzione l'embrione appena sopra l'arto posteriore con pinze sottili e fare un taglio trasversale per rimuovere la metà posteriore dell'embrione. Questo permette all'embrione di giacere piatto in posizione sagittale per il passaggio 4.2. Riposizionare l'embrione nel tubo da 2 mL con 1x PBS fresco.
NOTA: Un embrione di controllo e mutante può essere accoppiato e macchiato con la stessa soluzione anticorpale in un tubo, per i passaggi da 3.3 a 3.8.- Se si macchiano due embrioni insieme, tagliare la testa di un embrione sopra il primo arco farangeale pizzicando con pinze fini per fare un taglio trasversale. Questo distinguerà gli embrioni di due genotipi diversi all'interno di ogni tubo.
- Per permeabilizzare l'embrione,s pipet fuori 1x PBS dal tubo, facendo attenzione a non toccare l'embrione(s). Aggiungere 1 mL di PBST. Mettere il tubo a 4 gradi centigradi con agitazione delicata durante la notte.
NOTA: (punto di arresto opzionale) Gli embrioni possono essere conservati in soluzione PBST a 4 gradi centigradi per diversi giorni. - Per evitare il legame non specifico degli anticorpi, rimuovere prima il PBST dal tubo, facendo attenzione a non toccare l'embrione(i). Aggiungere 600 l di soluzione di blocco del buffer agli embrioni. Bloccare l'embrione(i) a 4 gradi centigradi con agitazione delicata durante la notte.
NOTA: la soluzione di blocco deve essere filata alla massima velocità su una centrifuga di panchine immediatamente prima dell'uso per rimuovere i detriti. - Per colorare e quantificare gli EC, utilizzare anticorpi contro VEGFR2 ed ERG. Le soluzioni anticorpali sono fatte nel buffer di blocco. Anti-VEGFR2 anticorpo è diluito 1:200 e l'anticorpo ERG viene diluito 1:1000.
NOTA: Le soluzioni anticorpali devono essere filate alla massima velocità su una centrifuga panca immediatamente prima dell'uso per rimuovere il particolato.- Per incubare gli embrioni con anticorpi primari, rimuovere la soluzione di blocco tampone dal tubo, facendo attenzione a non toccare l'embrione(i). Aggiungere 600 l di soluzione anticorpale primaria ad ogni tubo. Incubare l'embrione(i) a 4 gradi centigradi con agitazione delicata per 4-5 giorni.
- Per lavare gli embrioni della soluzione anticorpale, rimuovere prima la soluzione anticorpale primaria dal tubo. Lavare gli embrioni ogni ora con 1 mL di PBST a temperatura ambiente (RT) con agitazione delicata. Lavare gli embrioni 4-5 volte durante il giorno e poi incubare a 4 gradi centigradi con agitazione delicata durante la notte. Il giorno seguente si lava ripetutamente.
- Crea soluzioni di anticorpi secondari diluindo Alexa Fluor 488 anti-goat e Alexa Fluor 555 1:300 anti-tono nel buffer di blocco. Diluire il titolo DAPI 1:1000 nel buffer di blocco.
NOTA: Le soluzioni anticorpali devono essere sfilate alla massima velocità su una centrifuga panca immediatamente prima dell'uso per rimuovere il particolato. Inoltre, altri coloranti Alexa Fluor possono essere utilizzati al posto di 488 o 555.- Per incubare l'embrione con anticorpi secondari, rimuovere il PBST dal tubo. Aggiungere 600 l di soluzione anticorpale secondaria ad ogni tubo. Incubare l'embrione(i) a 4 gradi centigradi con agitazione delicata per 4-5 giorni.
- Per lavare gli embrioni della soluzione anticorpale, rimuovere prima la soluzione anticorpale secondaria dal tubo. Lavare l'embrione ogni ora con 1 mL di PBST a RT con agitazione delicata. Lavare gli embrioni 4-5 volte durante il giorno e poi incubare a 4 gradi centigradi con agitazione delicata durante la notte. Il giorno seguente si lava ripetutamente.
4. Incorporare embrioni in agarose
NOTA: Nella sezione 4, l'embrione(i) sarà incorporato in agarose. Questo processo di incorporamento serve a due scopi: orientare correttamente l'embrione prima dell'imaging e aiutare a localizzare l'embrione dopo che è stato eliminato in BABB (passaggi 5.2.2 - 5.3.2).
- Preparare 200 mL di 1% soluzione di agarose aggiungendo 2 g di agarose a 200 mL di dH2O. Microwave fino a quando tutte le agarose sono sciolte.
NOTA: L'agarose residuo può essere conservato a 4 gradi centigradi e riscaldato per un utilizzo successivo. - Utilizzando uno stampo di paraffina di plastica e un tubo di vetro, trasferire delicatamente un embrione allo stampo. Rimuovere con attenzione il PBST dall'embrione. Posizionare l'embrione in posizione sagittale. Rapidamente, aggiungere circa 0,5 mL di agarose calde allo stampo - quanto basta per coprire l'embrione e riempire lo stampo. Assicurarsi che nessuna bolla d'aria circondano l'embrione.
- Posizionare lo stampo sul ghiaccio e coprire con un foglio di alluminio fino a quando l'agarose non si è solidificata.
NOTA: Non lasciare asciugare l'embrione dopo la rimozione del PBST. La soluzione agarose deve essere abbastanza calda da rimanere liquida quando viene aggiunta all'embrione. Aggiungere appena sufficiente agarose per coprire l'embrione, ma non troppo, altrimenti sarà difficile da immaginare. La profondità dell'immagine è determinata in parte dalla distanza di lavoro dell'obiettivo.
5. Disidratazione e sgombera dei tessuti
NOTA: In questa sezione, gli embrioni vengono disidratati utilizzando la serie di metanolo, quindi cancellati nel solvente organico, BABB, e montati tra due copricapi separati da un distanziale di gomma; in questo protocollo vengono utilizzati distanziali in gomma Fast Well. Il paraurti Fast Well ha una superficie adesiva a doppio lato. Il distanziale è necessario per creare un pozzo, in cui l'embrione sarà posizionato e tenuto tra due coprilabbra.
-
Disidratazione del metanolo
- Etichettare nuovi tubi da 2 mL, uno per embrione. Aggiungere 1 mL di 25% MeOH per tubo.
- Utilizzando un bisturi pulito, tagliare delicatamente l'agarose intorno all'embrione, lasciando abbastanza intorno all'embrione in modo che possa essere raccolto da pinze. Utilizzare pinze sottili per afferrare delicatamente l'agarose con l'embrione incorporato e posizionarlo nel tubo etichettato con 25% MeOH. Non permettere alle pinze di toccare l'embrione.
- Incubare gli embrioni a RT con agitazione delicata per 1 ora al buio.
- Rimuovere 25% MeOH dal tubo, facendo attenzione a non toccare l'embrione. Aggiungere 1 mL di 50% MeOH per tubo. Incubare a RT con agitazione delicata per 1 ora al buio.
- Rimuovere il 50% MeOH dal tubo, facendo attenzione a non toccare l'embrione. Aggiungere 1 mL di 75% MeOH per tubo. Incubare a RT con agitazione delicata per 1 ora al buio.
- Rimuovere 75% MeOH dal tubo, facendo attenzione a non toccare l'embrione(s). Aggiungere 1 mL di 100% MeOH per tubo. Incubare a RT con agitazione delicata per 1 ora al buio. Ripetere il lavaggio MeOH al 100% due volte.
-
Cancellazione con BABB
- Rimuovere 100% MeOH dal tubo, facendo attenzione a non toccare l'embrione. Aggiungere 1 mL del 50% BABB per tubo. Incubare a RT con agitazione delicata per 1 ora al buio.
- Rimuovere il 50% BABB dal tubo, facendo attenzione a non toccare l'embrione. Aggiungere 1 mL di 100% BABB per tubo. Incubare a RT con agitazione delicata per 1 ora al buio. Ripetere il lavaggio BABB al 100% due volte.
NOTA: (punto di arresto opzionale) Gli embrioni possono rimanere nel 100% BABB nei tubi per circa una settimana. Un deposito più lungo farà sì che BABB dissolva la plastica dei tubi.
-
Montaggio di embrioni per l'imaging
- Posizionare un paraurti Fast Well su uno scivolo di copertura di 24 mm x 60 mm #1,5, staccando l'adesivo di plastica da un lato. Assicurarsi che non ci siano bolle d'aria tra il coperchio e paraurti applicando una leggera pressione sull'adesivo di plastica in cima al paraurti di gomma. Etichettare la coverslip in base al numero di embrioni, al genotipo e agli anticorpi utilizzati per la colorazione.
NOTA: Qualsiasi distanziale può essere posizionato tra i coprilabbra, purché sia abbastanza spesso da evitare di schiacciare o schiacciare un embrione. Usiamo distanziali Fast Well a causa del loro spessore e convenienza, che include superfici adesive su entrambi i lati del distanziale per fissarlo ai telllips. - Convogliare con attenzione e scartare il 100% BABB dal tubo. Dopo aver visualizzate l'embrione incorporato ad agarose nel tubo, utilizzare pinze sottili per raccogliere l'agarose e trasferire con attenzione l'embrione sul coperchio all'interno del pozzo veloce - non consentire alle pinze di toccare l'embrione.
- Rimuovere il secondo adesivo di plastica dal paraurti e posizionare il secondo coperchio sulla parte superiore. Rimuovere le bolle d'aria premendo delicatamente sul coperchio. Fare attenzione a non rompere il vetro.
NOTA: I campioni possono essere conservati piatti in un supporto scorrevole al buio a RT per un massimo di un anno se la guarnire è stretta.
- Posizionare un paraurti Fast Well su uno scivolo di copertura di 24 mm x 60 mm #1,5, staccando l'adesivo di plastica da un lato. Assicurarsi che non ci siano bolle d'aria tra il coperchio e paraurti applicando una leggera pressione sull'adesivo di plastica in cima al paraurti di gomma. Etichettare la coverslip in base al numero di embrioni, al genotipo e agli anticorpi utilizzati per la colorazione.
6. Acquisizione di dati
NOTA: Nei passaggi seguenti, l'endotelio degli archi pharyngeal i 3, 4 e 6 verrà immaginato utilizzando la microscopia confocale.
-
Posizionamento dei vetrini sullo stadio del microscopio
- Per immaginare gli embrioni, utilizzare un microscopio confocale dotato di un obiettivo di immersione in acqua 20x, apertura numerica 0,95, distanza di lavoro 0,95 mm e il software NIS-Elements AR 5.11.01 a 64 bit.
- Utilizzando la fluorescenza a campo largo, individuare visivamente gli archi pharyngeal. Centra la vista del campo intorno al 4th PAA.
- Se il campo visivo dell'obiettivo non cattura l'intera area dell'arco pharyngeal, prendi e cuci un grande pannello di immagini con 1% di sovrapposizione. Per evitare il movimento del campione durante l'acquisizione dell'immagine grande, fissare delicatamente l'assemblaggio dello slittamento del coperchio sullo stadio utilizzando l'argilla di stampaggio.
-
Impostazione dei parametri di acquisizione
- Impostare la dimensione del foro stenopeico su 1.0.
- Nella scheda Acquisizione ND, impostare i limiti superiore e inferiore dell'imaging utilizzando la regolazione grossolana. Impostare la dimensione del passo z in base alle specifiche del software. Determinare lo spessore che può essere immagine dalla distanza di lavoro dell'obiettivo e dalla chiarezza del campione.
- A causa dello spessore dell'embrione, regolare il guadagno in tutto lo stack z. Impostare l'intensità e il guadagno del laser al centro della pila z per ogni canale (405, 488 e 555) e assegnare i valori nella scheda Correzione intensità z. Impostare gli stessi valori per la sezione inferiore.
- Scorrere l'embrione fino a quando i segnali di fluorescenza iniziano ad apparire dimmer. Aumentare il guadagno di ogni canale fino a quando l'intensità del segnale appare simile al segmento precedente. Assegnare il nuovo valore nella scheda Correzione intensità z Ripeti fino al completamento dello stack z. Importa le impostazioni in Acquisizione ND.
- Eseguire la scansione utilizzando l'opzione Esegui : Correzione.
7. Analisi con il software Imaris
NOTA: In questi passaggi, le immagini confocali verranno analizzate utilizzando il software di analisi delle immagini di microscopia, Imaris versione 9.2.0. Durante questa analisi, selezioneremo prima le regioni di interesse da analizzare creando superfici. Successivamente, useremo la funzione Maschera per separare visivamente queste aree. Infine, useremo la funzione Spot per quantificare il numero di EC all'interno di ogni regione di interesse.
- A seconda del software di imaging utilizzato nel passaggio 6, convertire le immagini in .ims utilizzando Imaris File Converter.
- Aprire i file .ims. Impostare l'immagine su Ortogonale in Fotocamera/Etichette nel pannello Tipo di fotocamera.
- Individuare i PAA e orientare l'immagine per la risalita.
NOTA: Quando i file vengono aperti per la prima volta, appariranno come una compilazione 3D di tutte le sezioni imaged. In questo passaggio, i PAA verranno individuati trasformando l'immagine 3D in un'immagine 2D. L'immagine 2D consente quindi di orientare correttamente i PAA per l'analisi.- Nel pannello Proprietà, disattivare Volume. Nel pannello Proprietà, fare clic su Aggiungi nuovo filtro orto. Impostare Orientamento sezione sul piano XY. Utilizzate posizione sezione per scorrere l'immagine fino a trovare gli APA.
- Se i PAA non sono paralleli alla parte superiore e inferiore dell'immagine, ruotare liberamente l'immagine utilizzando il cursore del mouse in modo che i PAA vengano eseguiti da sinistra a destra sullo schermo. Nel menu a discesa Elaborazione immagine, selezionare Rotazione libera e fare clic su OK.
- Superficie del 3 rd Arco Farangeale (Figura 2A, B - B")
NOTA: In questi passaggi, gli archi pharyngeal e i PAA verranno tracciati utilizzando lo strumento Superficie per generare una regione di interesse "superficie". Ciò consentirà a ogni regione di interesse di essere isolata visivamente dal tessuto circostante. Qui descriviamo i passi per la superficie e analizzare i componenti endoteliali dell'arco farangeale 3rd. Gli archi phariongeali 4 e 6 vengono analizzati in modo simile.- Per far emergere l'endotelio nell'intero arco pharyngeal 3rd, fate clic sul pulsante Aggiungi nuova superficie situato sotto il pannello Proprietà. Fare doppio clic su Superficie 1 e rinominare la nuova superficie in "3rd Pharyngeal Arch".
- Selezionare Ignora creazione automatica, modifica manualmente. Impostare Orientamento superficie sul piano Y' (orientamento coronale). Utilizzate la Posizione di taglio per posizionare il 3rd Pharyngeal piano di superficie dell'arco fino al punto in cui il 3rd PAA e Dorsal Aorta si connettono.
- Ruotare l'immagine in modo che il piano di superficie dell'arco pharyngeal 3rd sia in vista. Disattivare Ortho Slicer 1.
- Sotto l'estrazione Proprietà Contour . Mode (Modalità), selezionare la funzione Modalità disegno a distanza (Distance Drawing Mode). Se necessario, regolare le impostazioni dei parametri. Mantenere parametri di superficie coerenti tra i campioni. Per questo esempio, la spaziatura dei vertici è di 10 m.
- Per iniziare la risalita, premere il tasto Esc e fare clic sul pulsante Disegna. Tracciare il perimetro dell'arco pharyngeal 3rd con il cursore del mouse. Utilizzate Posizione sezione per spostare 10-25 sezioni. Traccia il perimetro dell'arco pharyngeal. Ripetere fino a quando l'arco fingetale è completamente tracciato.
- Per generare la superficie dell'area tracciata, selezionate il pulsante Crea superficie nel pannello Proprietà.
- Superficie del 3 rd PAA (Figura 2C- C")
- Per far emergere l'endotelio del 3rd PAA, prima spegnete la regione in superficie dal gradino 7.4, deselezionando la casella di superficie 3rd dell'arco Pharyngeal. Quindi, fare di nuovo clic sul pulsante Aggiungi nuova superficie. Fare doppio clic su Superficie 1 e rinominare la nuova superficie in "3rd PAA".
- Selezionare Ignora creazione automatica, modifica manualmente. Impostare Orientamento superficie sul piano Y' (orientamento coronale). Utilizzate la Posizione di taglio per posizionare il piano di superficie PAA a 3rd nel punto in cui si connettono PAA e Dorsal Aorta, quindi ripetete i passaggi 7.4.rd
- Mascheramento delle strutture in superficie
NOTA: Nei passaggi seguenti, ogni area di interesse emersa verrà mascherata. La mascheratura consente alla regione di interesse di essere visivamente distinta dal resto del tessuto immagine e consente la quantificazione di queste distinte strutture di interesse. Di seguito, descriviamo i passi in Imaris per visualizzare e analizzare l'endotelio PAA, così come il plesso - la vascolatura più piccola che circonda i PAA all'interno degli archi faaryngei. In questi passaggi, la superficie PAA di 3rd verrà mascherata per visualizzare ed eseguire analisi solo sui PAA utilizzando la funzione Spot descritta nella Sezione 7.7.- Selezionare Volume per visualizzare tutti i canali mascherati. Premere il tasto Esc per ruotare l'immagine e posizionare l'immagine in una posizione XY.
- Nella scheda Modifica, selezionare Selezione maschera per il 3rd PAA. Selezionare il canale DAPI e fare clic su OK. Ripetere l'operazione per i canali rimanenti.
- Sulla tastiera, premete Ctrl e D per visualizzare il pannello Regolazioni dello schermo. Seleziona ogni nuovo canale e rinominalo per rendere chiaro ciò che ogni canale mostra. Ad esempio, questo porterà a tre nuovi canali: "3rd PAA DAPI", "3rd PAA ERG" e "3rd PAA VEGFR2".
NOTA: Nei passaggi 7.6.4-7.6.7 creeremo canali solo per il plesso dell'arco fantasmangeale. - Per visualizzare il plesso endoteliale separatamente dal PAA, selezione della maschera per la superficie PAA di 3rd. Selezionare il canale DAPI. Deselezionare Seleziona voxel all'esterno della superficie e selezionare Seleziona voxel all'interno della superficie sui pulsanti, impostare Seleziona voxel all'internodella superficie su zero . Fare clic su OK.
- Ripetere l'operazione per i canali rimanenti. Questa operazione escluderà la regione contenente il PAA dai nuovi canali mascherati. Rinominare i canali per rendere chiaro ciò che ogni canale mostra. Ad esempio, questo porterà a tre nuovi canali: "Non-PAA DAPI", "Non-PAA ERG" e "Non-PAA VEGFR2".
- Per visualizzare il plesso endoteliale all'interno dell'arco pharyngeal 3rd, selezionare Selezione maschera nella scheda Modifica, per la superficie dell'arco faringeo 3rd. Selezionare il canale DAPI non PAA. Fare clic su OK.
- Ripetere l'operazione per i restanti canali Non-PAA. Rinominare i canali per rendere chiaro ciò che ogni canale mostra. Ad esempio, questo porterà a tre nuovi canali: "Plexus DAPI", "Plexus ERG" e "Plexus VEGFR2".
- Quantificazione dei numeri CE
NOTA: L'espressione dei nuclei endoteliali ERG che rende conveniente quantificare i numeri EC. In questi passaggi, il numero di EC sarà quantificato utilizzando la funzione Spot per generare un punto per ogni CE contrassegnato dall'espressione ERG nel PAA e nel plesso. Nella sezione 7.7, verranno generati punti per ogni cellula ERG-positiva nel PAA mascherato, seguita dalla deselezione dei punti nelle cellule ERG-positive e VEGFR2-negative.- Sulla tastiera, premete Ctrl e D per visualizzare il pannello Regolazioni dello schermo. Disattivare tutti i canali ad eccezione di PAA ERG.
- Nella scheda Proprietà, fare clic sul pulsante Aggiungi nuovi spot. Clicca su Spots 1 e rinominalo in "PAA Total Number of ECs". Fare clic sul pulsante freccia blu. Per Canale di origine, selezionare il canale PAA ERG. Regolare il diametro XY stimato a 4 m. Procedere al pannello successivo facendo clic sul pulsante freccia blu.
- Regolare il numero di punti visti utilizzando la scala scorrevole, per garantire che ogni nucleo CE (contrassegnato dall'espressione ERG) sia rappresentato da un punto. Fare clic sul pulsante a doppia freccia verde.
- Disattivare il canale PAA ERG nella Regolazione del display. Accendere il canale PAA VEGFR2 per visualizzare l'endotelio PAA.
- Per quantificare con precisione il numero di EC, ci assicuriamo che ogni posto esprima sia i marcatori EC, ERG e VEGFR2. A tale scopo, selezionare Superficie dell'oggetto nella scheda Modifica . gruppo Aggiungi/Elimina. Premere il tasto Esc ed eliminare tutti i punti che non sono POSITIVO a VEGFR2, tenendo premuto maiusc e selezionando il punto.
NOTA: Nella fase successiva, verranno generati punti per ogni cella ERG-positiva nel plesso mascherato, seguito dalla deselezione dei punti nelle celle ERG-positive e VEGFR2-negative. - Nella scheda Proprietà, fare clic sul pulsante Aggiungi nuovi spot. Selezionare Posizioni 1 e rinominare in "Plexus Total Number of ECs". Fare clic sul pulsante freccia blu. Per Canale di origine, selezionare il canale Plexus ERG. Regolare il diametro XY stimato a 4 m. Ripetere i passaggi da 7.7.1 a 7.7.5 per il numero totale di ED plexus.
- Fare clic sulla scheda Statistiche di ciascuna funzione spot per determinare il numero totale di EC nel plesso 3rd PAA e pharyngeal.
- Ripetere i passaggi da 7.7.1 a 7.7.6 per i restanti APA e il plesso dell'arco fingendo.
Risultati
L'intero protocollo di immunofluorescenza montato qui produce risultati chiari e puliti, consentendo la ricostruzione 3D dell'endotelio dell'arco fariano, come si vede nella Figura 1A. È importante incubare gli embrioni per una quantità sufficiente di tempo in ogni soluzione anticorpale per garantire una completa penetrazione attraverso il campione, nonché lavare accuratamente gli embrioni dopo l'incubazione dell'anticorpo. Nella figura 1B, i punti luminosi e grandi appaiono come risultato del particolato nelle soluzioni anticorpo o buffer di blocco. Abbiamo scoperto che centrifugare ogni soluzione prima dell'uso e lunghi periodi di pbST dopo ogni incubazione anticorpale risolve questo problema.
La figura 2 illustra il processo utilizzato per esporre un arco pharyngeal e un PAA per l'analisi, come descritto nella sezione 7 del protocollo. Utilizzando la funzione mascherata, il software Imaris consente alle regioni in superficie di essere visivamente separate e analizzate in modo indipendente.
La figura 3 illustra la mascheratura individuale di diversi compartimenti vascolari negli archi farangeali: il PAA (Figura 3A, B, C) e il plesso ( Figura3A', B', C'). La mascheratura consente l'analisi e la quantificazione dei numeri CE in ciascuna struttura separatamente. Nella figura 3C-C',la funzione Spot viene utilizzata per quantificare il numero totale di EC sia nel PAA che nel plesso, assegnando un unico punto per ogni nucleo che esprime ERG. È importante notare che l'algoritmo utilizzato per la funzione Spot è progettato per generare un punto per qualsiasi pixel di una dimensione specificata. ERG, che viene utilizzato qui come marcatore di nuclei CE, è espresso anche nelle cellule cresta neurale8; cellule creste neurali non esprimono VEGFR2. La figura 3D illustra un esempio di spot positivo (verde), VEGFR2-negativo (rosa) generato dalla funzione Imaris Spot. Di conseguenza, è essenziale verificare che ogni punto rappresenti una singola CE ed è etichettato sia con ERG che con VEGFR2.
| Passo | Tempo | Temperatura | |
| 1 | Lavaggio PBST/Permeabilizzazione | 24 h o O/N | 4 gradi centigradi |
| 2 | Buffer di blocco | 25 h o O/N | 4 gradi centigradi |
| 3 | Anticorpo primario | 4-5 giorni | 4 gradi centigradi |
| 4 | Lavaggio PBST | 4-5 volte al giorno per 2 giorni | RT (o 4 gradi centigradi se O/N) |
| 5 | Anticorpo secondario | 4-5 giorni | 4 gradi centigradi |
| 6 | Lavaggio PBST | 4-5 volte al giorno per 2 giorni | RT (o 4 gradi centigradi se O/N) |
| 7 | Incorpora | N/D | Rt |
| 8 | Disidratazione del metanolo e BABB | 1 ora per passo | Rt |
Tabella 1: Panoramica del protocollo di immunofluorescenza di montaggio completo. O/N - durante la notte; RT - temperatura ambiente.
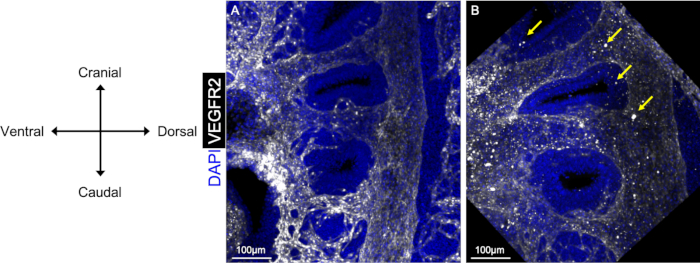
Figura 1: Confronto di immagini pulite e sporche dopo l'immunofluorescenza a montaggio completo. Le viste sagittali dell'embrione E10.5 mostrano l'uso di anticorpi anti-VEGFR2 (bianco) per visualizzare l'endotelio PAA. Gli embrioni accuratamente lavati con incubazioni di anticorpi PBST (A) hanno un rapporto segnale-rumore più elevato e producono un'immagine più pulita, rispetto agli embrioni che non vengono lavati completamente (B). Le frecce in B mostrano le aree di rumore/sporco che sono apparse nell'immagine quando un embrione non viene lavato completamente o la soluzione anticorpale non è stata centrifugata. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Rivestimento dell'arco pharyngeal (PA) e PAA. Una vista sagittale 2D (A) viene utilizzata per identificare la posizione dei PAA nell'immagine confocale. Un affettatrice ortogonale coronale (A, linea gialla) è posto attraverso i PAA. L'arco faringo (B) e il PAA (C) vengono poi visualizzati nell'orientamento coronale utilizzando lo strumento Disegno a distanza in Imaris. Lo strumento Disegno a distanza, impostato su 10 m, viene utilizzato per tracciare il perimetro dell'arco faaryngeale 3rd (B) o del PAA (C). I contorni sono disegnati ogni 10-25 fette attraverso l'intero arco (B', C'). I contorni vengono combinati per generare una superficie 3D dell'arco pharyingeale (B") o PAA (C"). Fare clic qui per visualizzare una versione più grande di questa figura.
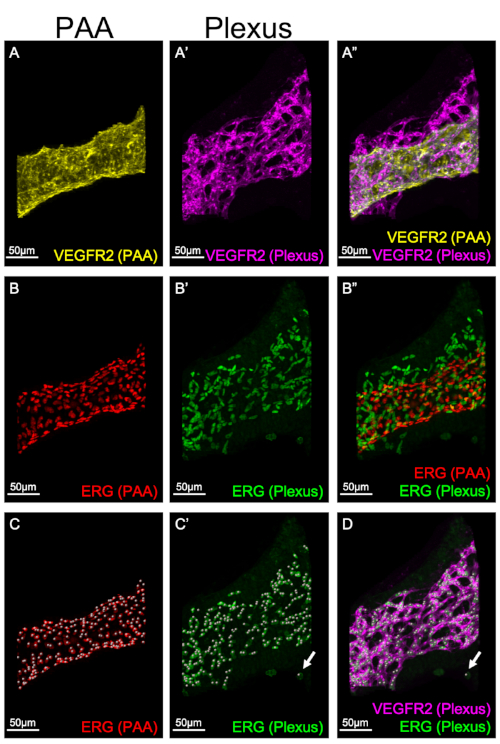
Figura 3: Quantificazione dei numeri CE in un PAA e in un plesso. Le ricostruzioni 3D sono utilizzate per visualizzare separatamente la struttura e l'espressione dei marcatori CE in un PAA o in un plesso CE. I pannelli A-A' mostrano l'espressione di VEGFR2 nel PAA (A, giallo) e nel plesso (A', rosa). Il Pannello A" illustra un'unione delle espressioni PAA e plexus VEGFR2. I pannelli B-B' mostrano l'espressione di ERG nel PAA (B, rosso) e nel plesso (B', verde). Il pannello B" illustra l'unione del PAA e del plesso. C-C'. La funzione Spot in Imaris viene utilizzata per quantificare il numero di EC nel PAA o nel plesso. A ogni cella positiva ERG nel PAA (C, rosso) o plexus (C', verde) viene assegnato un singolo punto per contrassegnare una singola CE. La freccia in C'-D mostra un esempio di punto positivo ERG, VEGFR2-negativo nel plesso che è stato generato dalla funzione Imaris Spot. Questo punto è escluso dalla quantificazione. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La capacità di visualizzare l'endotelio negli embrioni di topo in 3D ha fornito nuove intuizioni sul loro sviluppo3. Qui presentiamo un protocollo che consente l'imaging 3D ad alta risoluzione degli embrioni, la visualizzazione della connettività vascolare e analisi quantitative della formazione di PAA. Questo protocollo può essere impiegato per vedere come le alterazioni genetiche o gli insulti ambientali influiscono sullo sviluppo PAA. La procedura qui riportata utilizza anticorpi contro VEGFR2 ed ERG per visualizzare la formazione di PAA e quantificare il numero EC; tuttavia, gli anticorpi aggiuntivi possono essere utilizzati per visualizzare e analizzare altri aspetti dello sviluppo dell'arteria dell'arco, come il reclutamento di creste neurali o la differenziazione delle cellule muscolari lisce. Se questa procedura deve essere utilizzata nelle fasi precedenti dell'embriogenesi, è importante notare che alcuni antigeni (ad esempio, ERG) rilevati in questo protocollo potrebbero non essere ancora espressi. Altre macchie nucleari come DAPI o DRAQ5 o l'etichettatura di lignaggio con traccianti con tag nucleare possono essere utilizzate per quantificare il numero EC.
Ci sono diversi passaggi critici all'interno del protocollo: garantire che 1) gli embrioni non vengano desiccati tra i cambiamenti di soluzione; 2) gli embrioni vengono accuratamente lavati dopo le incubazioni di anticorpi; e 3) che gli embrioni siano completamente disidratati con MeOH prima della compensazione dei tessuti con BABB.
I fumi a metanolo prima della compensazione dei tessuti servono a due scopi: eliminare la fluorescenza dovuta all'espressione di proteine fluorescenti (ad esempio l'espressione di EGFP o tdTomato utilizzato per la tracciatura del lignaggio) nell'embrione e disidratare il tessuto. L'eliminazione della fluorescenza dalle proteine fluorescenti consente l'uso di qualsiasi combinazione di fluorofori per l'imaging. Gli anticorpi contro EGFP e TdTomato (ciliegia) possono essere utilizzati per visualizzare l'espressione di queste proteine fluorescenti. In alternativa, il MeOH può essere sostituito da tetraidrofuran per preservare la fluorescenza delle proteine fluorescenti9.
Abbiamo scoperto che gli embrioni che non sono stati adeguatamente disidratati prima dello sgomberaggio BABB sono difficili da immaginare a causa della diffusione della luce. BABB è una soluzione idrofobica che richiede una completa disidratazione in un solvente organico per eliminare il tessuto opaco. La compensazione completa garantisce la capacità di ottenere immagini ai livelli più profondi possibili all'interno dell'embrione10,,11. In questo protocollo, abbiamo usato un obiettivo di immersione in acqua 20x, a causa della sua lunga distanza di lavoro e disponibilità al momento dei nostri esperimenti. Gli obiettivi di immersione petrolifera sono più adatti a questo protocollo, poiché BABB e il petrolio hanno indici di rifrazione più stretti dell'acqua e del BABB. Tuttavia, nonostante la differenza nell'indice di rifrazione, l'obiettivo di immersione dell'acqua utilizzato in questo protocollo ha fornito un'eccellente qualità dell'immagine.
Ci sono alcune limitazioni di questo protocollo. La radura BABB utilizzata qui è tossica e corrosiva11,12,13. BABB scioglie colla e plastica. Se i campioni non vengono maneggiati correttamente durante l'imaging, l'obiettivo del microscopio può essere danneggiato da BABB che può fuoriuscire dal campione tramite crepe nel coperchio o una guarnici rotta tra il paraurti Fast Well e il coverslip. I metodi di compensazione che non utilizzano solventi organici, come CLARITY, possono essere utilizzati come alternative10,11,14. La soluzione di corrispondenza dell'indice di rifrazione di CLARITY ha un indice di rifrazione simile a quello dell'acqua, il che lo rende un metodo di compensazione adatto se si utilizza un obiettivo di immersione in acqua. Un'ulteriore limitazione di questo protocollo è che può essere eseguito solo su tessuti non viventi, impedendo così la sua applicazione per l'imaging dal vivo.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Brianna Alexander, Caolan O'Donnell e Michael Warkala per un'attenta lettura e modifica di questo manoscritto. Questo lavoro è stato sostenuto dai finanziamenti del National Heart, Lung and Blood Institute del NIH R01 HL103920, R01 HL134935, R21 OD025323-01 a SA; AJR è supportato da NHLBI HL103920-08S1 e dall'Istituto Nazionale di Artrite e Musculoscheletrie e Malattie della Pelle Sovvenzionia T32052283-11.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x PBS | MP Biomedicals | PBS10X02 | |
| 20x water immersion objective | Nikon | MRD77200 | |
| Agarose | Bio-Rad Laboratories | 1613101 | |
| Alexa Fluor 488 anti-goat | Invitrogen | A-11055 | |
| Alexa Fluor 555 anti-mouse | Invitrogen | A-31570 | |
| Analysis Software | Imaris 9.2.0 | ||
| Benzyl Alcohol | Sigma-Aldrich | 305197 | |
| Benzyl Benzoate | Sigma-Aldrich | 8.18701.0100 | |
| Cover Slips | VWR | 16004-312 | |
| DAPI (5 mg/mL stock) | Fisher Scientific | D3571 | |
| Eppendorf Tubes (2.0 mL) | Fisher Scientific | 05-408-138 | |
| Ethanol | VWR | 89370-084 | |
| Falcon tubes (50 mL) | Corning | 352098 | |
| Fast wells | Grace Bio Labs | 664113 | |
| Forceps | Roboz | RS-5015 | |
| Goat anti-VEGFR2 | R&D Systems, Inc. | AF644 | |
| Methanol | VWR | BDH1135-4LP | |
| Microscope | Nikon | A1HD25 | |
| Mouse anti-ERG | Abcam | ab214341 | |
| Normal Donkey Serum | Sigma-Aldrich | D9663 | |
| Paraformaldehyde | Electron Microscopy Sciences | 15710 | |
| Pasteur pipets | Fisher Scientific | 13-678-20D | |
| Petri dishes (35 mm) | Genesee Scientific | 32-103 | |
| Petri dishes (60 mm) | Genesee Scientific | 32-105 | |
| Plastic Molds | VWR | 18000-128 | |
| Scapels | Exelint International Co. | 29552 | |
| Triton-X-100 | Fisher Scientific | BP 151-500 |
Riferimenti
- Hiruma, T., Nakajima, Y., Nakamura, H. Development of pharyngeal arch arteries in early mouse embryo. Journal of Anatomy. 201 (1), 15-29 (2002).
- Hutson, M. R., Kirby, M. L. Model systems for the study of heart development and disease Cardiac neural crest and conotruncal malformations. Seminars in Cell & Developmental Biology. 18 (1), 101-110 (2007).
- Wang, X., et al. Endothelium in the pharyngeal arches 3, 4 and 6 is derived from the second heart field. Developmental Biology. 421 (2), 108-117 (2017).
- Jerome, L. A., Papaioannou, V. E. DiGeorge syndrome phenotype in mice mutant for the T-box gene, Tbx1. Nature Genetics. 27 (3), 286-291 (2001).
- Lindsay, E. A., et al. Tbx1 haploinsufficieny in the DiGeorge syndrome region causes aortic arch defects in mice. Nature. 410 (6824), 97-101 (2001).
- Weninger, W., et al. Visualising the Cardiovascular System of Embryos of Biomedical Model Organisms with High Resolution Episcopic Microscopy (HREM). Journal of Cardiovascular Development and Disease. 5 (4), 58(2018).
- Phillips, H. M., et al. Pax9 is required for cardiovascular development and interacts with Tbx1 in the pharyngeal endoderm to control 4th pharyngeal arch artery morphogenesis. Development. 146 (18), (2019).
- Vlaeminck-Guillem, V., et al. The Ets family member Erg gene is expressed in mesodermal tissues and neural crests at fundamental steps during mouse embryogenesis. Mechanisms of Development. 91 (1-2), 331-335 (2000).
- Ertürk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-217 (2012).
- Azaripour, A., et al. A survey of clearing techniques for 3D imaging of tissues with special reference to connective tissue. Progress in Histochemistry and Cytochemistry. 51 (2), 9-23 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying Tissue Clearing. Cell. 162 (2), 246-257 (2015).
- Becker, K., Jährling, N., Saghafi, S., Weiler, R., Dodt, H. U. Chemical Clearing and Dehydration of GFP Expressing Mouse Brains. PLoS One. 7 (3), e33916(2012).
- Ertürk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon