Method Article
Micromatrizes de biopartículas para análise quimiotática e molecular de swarming de nêutrons humanos in vitro
Neste Artigo
Resumo
Este protocolo gera micromatrizes de biopartículas que fornecem enxame de neutrófilos espacialmente controlados. Ele fornece fácil acesso aos mediadores que os neutrófilos liberam durante a migração e permite análise quantitativa de imagem.
Resumo
O enxame de neutrófilos é um processo cooperativo pelo qual os neutrófilos selam um local de infecção e promovem a reorganização tecidual. Swarming tem sido estudado clássicamente in vivo em modelos animais mostrando padrões característicos de migração celular. No entanto, nos modelos vivo possuem várias limitações, incluindo mediadores intercelulares que são de difícil acesso e análise, bem como a incapacidade de analisar diretamente os neutrófilos humanos. Devido a essas limitações, há a necessidade de uma plataforma in vitro que estude com neutrófilos humanos e ofereça fácil acesso aos sinais moleculares gerados durante o enxame. Aqui, um processo de microcarimbo múltiplo é usado para gerar uma micromatriz de biopartículas que estimula o enxame imitando uma infecção in vivo. A micromatriz de biopartículas induz os neutrófilos a se aglomerarem de forma controlada e estável. Na micromatriz, os neutrófilos aumentam a velocidade e formam enxames estáveis em torno de aglomerados de biopartículas. Além disso, o supernatante gerado pelos neutrófilos foi analisado e 16 proteínas foram descobertas como expressos diferencialmente ao longo do aquecimento. Esta plataforma de aquecimento in vitro facilita a análise direta da migração de neutrófilos e liberação de proteínas de forma reprodutível e controlada espacialmente.
Introdução
Os neutrófilos, os mais abundantes glóbulos brancos da corrente sanguínea1,estão ganhando atenção como potenciais metas diagnósticas e terapêuticas2,3, porque podem estar envolvidos em uma variedade de condições médicas, incluindo gota4,sepse3, trauma5,6,câncer1,7,8, e várias doenças autoimunes5,9. O enxame de neutrófilos é um processo multiestágio, fortementeregulado com uma complexidade que o torna um foco particularmente interessante do estudo5,10,11. Durante o enxame, os neutrófilos isolamum local de inflamação do tecido saudável circundante5,10,11. A regulação adequada do enxame de nêutrons é essencial para promover a cicatrização da ferida e, finalmente, a resolução de inflamação5,12. O enxame de neutrófilos tem sido estudado principalmente em roedores12,13,14,15 e zebrafish10,11,12,15 modelos. No entanto, a natureza desses modelos animais vivos dá origem a limitações5. Por exemplo, os mediadores liberados por neutrófilos durante o enxame não são facilmente acessíveis para análise5. Além disso, existem muitas fontes potenciais para um determinado mediador in vivo, por isso um experimento in vivo deve introduzir uma deficiência genética para inibir a produção celular e/ou interação, a fim de investigar o papel desse mediador em um determinado processo13. Um experimento in vitro contorna essa complicação permitindo a observação de neutrófilos sem o contexto de células adicionais. Além disso, pesquisas que descrevem a migração coordenada por neutrófilos humanos são limitadasem 16. Em uma plataforma de enxame in vitro, os neutrófilos humanos podem ser diretamente analisados. Uma plataforma de enxame in vitro poderia expandir o conhecimento adquirido a partir de estudos in vivo, proporcionando oportunidades para preencher as lacunas deixadas pelas limitações dos estudos in vivo.
Para atender à necessidade de uma plataforma in vitro que imita o aquecimento vivo de neutrófilos, desenvolvemos uma plataforma de microestampagem que nos permite padronizar micromatrizes de biopartículas que estimulam o aquecimento de neutrófilos de forma espacialmente controlada. Geramos micromatrizes de biopartículas em slides de vidro em um processo de duas etapas. Primeiro, usamos microestampagem para gerar uma micromatriz de manchas de polieletrolito cdoico (CP). Em segundo lugar, adicionamos uma solução de biopartículas que aderem às manchas CP via interação eletrostática. Ao padronizar primeiro a camada CP, podemos padronizar seletivamente partículas carregadas negativamente para gerar o padrão desejado de aquecimento de neutrófilos. A camada positivamente carregada contém as biopartículas carregadas negativamente através da etapa vigorosa de lavagem que remove as biopartículas das áreas no deslizamento de vidro que não têm o CP. Além disso, o CP usado aqui, um copolímero de acrilamida e momer cationic quaternizado, é biocompatível, por isso não induz uma resposta dos neutrófilos. Ele tem uma carga superficial muito alta que imobiliza as biopartículas do tamanho de mícrons para o deslizamento de vidro, inibindo assim os neutrófilos de remover as partículas da posição padronizada no deslizamento de vidro. Isso resulta em agrupamentos de biopartículas dispostos em uma micromatriz. Quando adicionamos neutrófilos à micromatriz, eles formaram enxames estáveis ao redor dos aglomerados de biopartículas. Através do rastreamento da migração de neutrófilos, descobrimos que os neutrófilos migram ativamente para os aglomerados de biopartículas. Além disso, usamos esta plataforma para analisar certos mediadores que os neutrófilos liberam durante o enxame. Encontramos 16 mediadores que são expressos diferencialmente durante o enxame. Suas concentrações seguem três tendências gerais ao longo do tempo: aumento, diminuição ou pico. Nossa plataforma de aquecimento de neutrófilos in vitro facilita a análise do enxame de neutrófilos humanos controladoespacialmente, bem como a coleta e análise de mediadores liberados pelo swarming de neutrófilos. Em uma publicação anterior, demonstramos que pacientes com determinadas condições médicas (trauma, doença autoimune e sepse), tinham neutrófilos que funcionavam de forma diferente daqueles de doadores saudáveis5. Em estudos futuros, nossa plataforma poderia ser usada para analisar a função neutrófilo entre uma variedade de populações de pacientes. Esta plataforma pode analisar quantitativamente a complexa coordenação envolvida no aquecimento de neutrófilos. Estudos adicionais podem ser feitos para fornecer informações sobre a função neutrófilo de uma população específica de pacientes ou resposta neutrófilo a um patógeno de interesse.
Protocolo
Os autores reconhecem os voluntários saudáveis que gentilmente doaram seu sangue. Os amostras de sangue foram obtidos após o consentimento voluntário informado de acordo com o protocolo do Conselho de Revisão Institucional (IRB) #2018H0268 revisado pelo Comitê de Ciências Biomédicas da Universidade Estadual de Ohio.
1. Microfabricação da micromatriz de biopartículas
- Utilizando procedimentos padrão de fotolitografia, gere o mestre wafer de silício.
- Gere uma prova do design desejado usando um software de design auxiliado por computador (CAD) e envie para um fabricante de fotomáscaras para produzir uma fotomáscara cromada. O design utilizado aqui é de 4 mm x 4 mm matrizes retangulares de 30 μm de diâmetro preenchidas em círculos com um espaçamento centro-a-centro de 150 μm. Este design pode ser modificado como desejado para diferentes aplicações.
- Spincoat uma camada de 40 μm de espessura de uma foto negativa resista a um wafer de silício. Asse wafer a 65 °C por 5 min e 95 °C por 10 min.
- Exponha wafer à luz UV através de uma fotomáscara cromada com 150-160 mJ/cm2 (Figura 1A).
- Asse wafer a 65 °C para 5 min e 95 °C por 10 min. Submersa em desenvolvedor de fotoresist por 10 min e enxágue com álcool isopropílico. Nesta fase, o padrão deve ser visível no wafer (Figura 1B).
- Misture completamente uma razão 10:1 de pré-polímero polidimetilexiano e seu agente de cura (ou seja, 20 g de pré-polímero e 2 g de agente de cura) e despeje a mistura de polidimetilsiloxano (PDMS) não curada sobre o wafer mestre em uma placa de Petri (Figura 1C).
- O vácuo trata a mistura de PDMS não curada até que não haja bolhas de ar sobre o wafer mestre. Cure a 65 °C durante a noite.
- Use um bisturi para cortar ao redor do exterior da seção padronizada do wafer, e remova lentamente a laje de PDMS curada. Coloque a laje do PDMS em uma tábua de corte limpa com o lado estampado voltado para cima (Figura 1D).
- Soque selos individuais da laje do PDMS com um soco de biópsia de 8 mm(Figura 1E). Para um slide de vidro, serão necessários oito selos.
- Coloque cada selo de bruçado na fita adesiva para remover quaisquer detritos.
- Com antecedência, prepare uma solução de 1,6 mg/mL de CP na água.
- Adicione a quantidade adequada do pó CP à água (por exemplo, de 0,8 g a 500 mL).
- Misture em uma placa de agitação à temperatura ambiente durante a noite, ou até que todo o sólido seja dissolvido na água. A solução CP pode ser armazenada em temperatura ambiente por 6 meses.
- Se desejar, torne a solução CP fluorescente adicionando poli-L-lysine rotulada com fluorescenina isothiocyanata (PLL-FITC).
- Aliquot cerca de 10 mL da solução CP. Adicione uma pequena quantidade de PLL-FITC (0,05 mg) ao volume alicitado. A quantidade pode ser alterada para ajustar o brilho da fluorescência conforme desejado.
- Vortex a solução CP rotulada com FITC para 20 s, ou até que a solução seja uma cor amarela uniforme e pálida. Proteja-se da luz e armazene a 4 °C por até 1 mês.
- Com os selos de face-up, prime cada selo com 100 μL de 1,6 mg/mL solução de CP, garantindo que não se forme bolhas de ar entre a solução CP e o carimbo (Figura 1F).
- Inverta os selos em uma camada de solução CP (Figura 1G).
- Remova os selos da solução CP após 1h.
- Dab cada carimbo molhado de frente para um slide de vidro limpo 6-8x para remover o excesso de líquido.
- O vácuo trata os selos por 1-2 min.
- Adere um espaçador de imagem de oito poços e 9 mm de diâmetro na parte superior de um slide de vidro limpo como guia para colocação de selos. Com este espaçador, cada escorregador de vidro pode ter oito micromatrizes.
- Coloque um carimbo de frente para baixo no escorregador de vidro no centro de cada poço do espaçador de imagem (ou seja, use oito selos no total).
- Coloque um peso equilibrado de 5,6 ± 0,1 g em cima de cada selo e deixe 10 min para estampagem(Figura 1H). Um peso equilibrado é necessário para garantir que o carimbo seja pressionado uniformemente para o slide de vidro e promover até mesmo a transferência do CP para o escorregador de vidro.
- Remova os pesos e selos do slide de vidro (Figura 1I). Deixe a camada CP secar à temperatura ambiente por 24 h antes de adicionar as biopartículas, conforme descrito na etapa 1.17. Se o CP for marcado com FITC, a eficácia da estampagem pode ser verificada neste momento com um microscópio fluorescente de 488 nm antes de prosseguir para a etapa 1.17 (Figura 1M).
- Corte uma placa de PDMS em branco para o tamanho do espaçador de imagem e use o soco de biópsia de 8 mm para criar poços no PDMS que se alinham com os poços de um espaçador de imagem. Adere a laje do PDMS ao slide de vidro com o espaçador de imagem(Figura 1J).
- Descongele uma solução de biopartículas (por exemplo, E. coli ou zymosan) e dilua até 500 μg/mL em água para injeção (WFI).
NOTA: As biopartículas não precisam ser opsonizadas. Receptores de superfície nutronéfilo reconhecem diretamente moléculas nessas partículas19,20,21. - Adicione 100 μL de solução de biopartículas a cada PDMS bem no slide de vidro(Figura 1K).
- Ame a lâmina de vidro por 30 min.
- Enxágüe os poços completamente com água. A micromatriz de biopartículas pode ser armazenada em um ambiente livre de poeira a 4 °C por até 3 meses. Neste ponto, o padrão deve ser verificado com um microscópio fluorescente de 594 nm antes de seguir para a etapa 2.1 (Figura 1N).
2. Preparação de amostras
- Colete pelo menos 2 mL de sangue fresco em tubos K2-EDTA do doador desejado. O rendimento esperado de neutrófilos é de 1-2 x 106 células/1 mL de sangue inteiro. O ensaio por imagem requer aproximadamente 1,5 x 105 neutrófilos, e a análise do supernatante requer 1 x 106 neutrófilos. Use o sangue dentro de 4 h.
- Separe glóbulos vermelhos (RBCs) adicionando um agente de agregação de eritrócitos em uma proporção de 1:5 para todo o sangue. Espere 45 min por uma camada translúcida (casaco buffy) para se separar da camada de RBCs.
- Retire o casaco de buffy e lave com salina tampão de fosfato (PBS) usando 1 mL buffy coat: 9 mL PBS.
- Centrífuga por 5 min a 190 x g e 20 °C.
- Aspirar o supernatante e resuspender a pelota em 5 x 107 células/mL.
- Use um kit de seleção imunomagnética negativa para isolar neutrófilos.
- Adicione 50 μL de coquetel de anticorpos/1 mL suspensão celular. Espere 10 min.
- Adicione 100 μL de suspensão de células magnéticas/1 mL. Espere 10 min.
- Adicione a suspensão da célula a um tubo de fundo redondo e coloque em um ímã cilíndrico. Espere 10 min.
- Despeje supernatant em um tubo de centrífuga. Adicione até 10 mL da PBS. Centrífuga por 5 min a 190 x g e 20 °C.
- Supernatant aspirado. Resuspender a pelota branca no IMDM com 0,4% de albumina de soro humano.
- Núcleos de manchas com 20 μg/mL Hoechst 33342 por 10 min a 37 °C.
- Adicione 5 mL de IMDM com 0,4% de albumina de soro humano para enxaguar. Centrífuga por 5 min a 190 x g e 20 °C.
- Resuspender as células a 7,5 x 105 células/mL no IMDM com 0,4% de albumina de soro humano.
- Adicione 100 μL da suspensão celular a um poço PDMS contendo uma micromatriz de biopartículas. Certifique-se de que a suspensão celular seja convexa sobre o topo do PDMS bem e não contenha bolhas.
- Veda com um deslizamento de cobertura de 12 mm de diâmetro. Cubra bem a abertura do PDMS com um deslizamento de cobertura de 12 mm de diâmetro. Pressione suavemente para o deslizamento de cobertura com pinças para que o excesso de suspensão celular escape até a borda do poço. Use um tecido para remover o excesso de suspensão celular.
3. Executando o ensaio e análise de imagem
- Carregar matriz de micropartículas com células na estação de imagens de células vivas com um microscópio equipado com uma incubadora de gaiola situada em 37 °C, 5% CO2e 90% umidade relativa.
- Use microscopia fluorescente e de campo brilhante para gravar imagens a cada 10x de ampliação a cada 10 s a 405 nm, 594 nm e brightfield. Em um experimento típico, as imagens são coletadas até 2 h.
- Use um software automatizado de rastreamento celular para rastrear a migração de neutrófilos individuais em direção ao cluster de biopartículas.
- Use o modo de autoregressão de um software de rastreamento de células de detecção de ponto. Defina o raio da mancha para 5 μm (o tamanho aproximado de um núcleo neutrófilo). Defina o comprimento mínimo da faixa para 120 s e um tamanho máximo de intervalo de um quadro.
- A partir dos dados gerados pelo software de rastreamento celular, extraios os arquivos que contêm a posição e a velocidade do nêutron. Esses arquivos podem ser usados com um software de grafografia para gerar faixas de migração de nêutrons(Figura2C)e um mapa de calor de velocidade versus tempo(Figura2D),respectivamente.
- Use as imagens fluorescentes de 405 nm para rastrear o tamanho do enxame ao longo do tempo em um software de análise de imagem de sua escolha.
- Defina regiões de interesse (ROIs) em torno de cada aglomerado de biopartículas onde os neutrófilos se aglomerarão. Mantenha o mesmo tamanho ROI para analisar cada aglomerado de biopartículas.
- Analise a intensidade fluorescente média das imagens de 405 nm dentro de cada ROI ao longo do tempo.
- Gere uma curva de calibração de intensidade fluorescente média ao tamanho do enxame, fazendo medições manuais em vários tamanhos de enxame de 0 μm2 ao tamanho máximo do enxame. Use esta calibração para calcular o tamanho do enxame ao longo do tempo.
4. Coleção supernatant e detecção de proteínas
- Incubar os neutrófilos nos poços contendo a micromatriz de biopartículas a 37 °C e 5% co2 por 3 h. Pegue amostras nos pontos de tempo desejados. Normalmente, as amostras serão colhidas às 0, 0,5, 1 e 3h. Para superar o limite de detecção do ensaio da matriz proteica, todo o volume de supernatant de um único poço (200 μL) foi usado para cada ponto de tempo. Cada ponto de tempo foi analisado em triplicado.
- Usando um microscópio de campo brilhante, verifique se os enxames são formados na micromatriz.
- Aspirar o supernatant com uma pipeta de 200 μL e carregar em um tubo de filtro de centrífuga de 0,45 μm.
- Centrífuga o supernatante em 190 x g e 20 °C por 5 min e coletar o volume filtrado.
- Armazene amostras a -80 °C até o horário de processamento.
- Use um kit de micromatriz que detecta uma gama de proteínas humanas para processar amostras.
- Adicione 200 μL de cada amostra a um tubo de diálise separado fornecido com o kit.
- Coloque os tubos de diálise em um béquer contendo pelo menos 500 mL de PBS (pH = 8,0). Mexa delicadamente em uma placa de agitação por pelo menos 3 h a 4 °C. Mude o PBS no béquer e repita este passo.
- Transfira cada amostra para um tubo de centrífuga limpa e centrífuga em 9.000 x g por 5 min para remover quaisquer precipitados. Transfira cada supernatante para um tubo limpo.
- Biotinylate cada amostra adicionando 36 μL de reagente de rotulagem de 1x do kit por 1 mg de proteína total na amostra dialisada a 180 μL de amostra dialisada. Incubar a 20 °C por 30 min. Misture suavemente a cada 5 min.
- Adicione 3 μL de solução de parada fornecida com o kit em cada tubo de amostra. Transfira cada amostra para um tubo de diálise fresco e repita as etapas 4.6.2-4.6.3. Nesta fase, a amostra pode ser armazenada a -20 °C ou -80 °C até que você esteja pronto para prosseguir.
- O escorregador de vidro fornecido com o kit é armazenado a -20 °C. Deixe-o chegar à temperatura ambiente. Coloque o escorregador de vidro montado em um capô de fluxo laminar por 1-2h à temperatura ambiente.
- Adicione 400 μL do buffer de bloqueio fornecido com o kit em cada poço do slide de vidro montado. Incubar a temperatura ambiente por 30 min.
- Centrífuga as amostras preparadas para 5 min a 9.000 x g para remover precipitados ou partículas. Diluir 5x com tampão de bloqueio.
- Remova o tampão de bloqueio de cada poço. Adicione 400 μL das amostras diluídas nos poços apropriados. Incubar por 2h à temperatura ambiente enquanto balança.
- Decante as amostras de cada poço. Lave 3x com 800 μL do tampão de lavagem de 1x que forneci com o kit em temperatura ambiente por 5 min cada enquanto balançava.
- Em um recipiente limpo, submerga o escorregador de vidro montado em 1x tampão de lavagem I. Lave 2x à temperatura ambiente por 5 min cada enquanto balança.
- Adicione 400 μL de 1x streptavidin conjugado cy3 a cada submatriz. Cubra com tiras de adesivo de plástico. Proteja-se da luz para o restante do protocolo.
- Incubar por 2h à temperatura ambiente enquanto balança.
- Decante a solução e desmonte o deslizamento de vidro das câmaras de amostra.
- No tubo centrífuga de 30 mL fornecido com o kit, adicione cuidadosamente o escorregador de vidro e o buffer de lavagem suficiente de 1x I para cobrir o escorregador de vidro. Lave 3x por 10 min cada à temperatura ambiente enquanto balança.
- No tubo centrífuga de 30 mL, lave 2x com 1x tampão de lavagem II por 5 min cada uma à temperatura ambiente enquanto balança.
- Lave o slide de vidro com 30 mL de ddH2O por 5 min. Remova o deslizamento de vidro do tubo de centrífuga e deixe secar por 20 min em capô de fluxo laminar. O slide de vidro preparado pode ser armazenado a -20 °C até estar pronto para escanear.
- Escaneie o deslizamento de vidro com um scanner de micromatriz em uma emissão de fluorescência de 555 nm.
Resultados
Quando os neutrófilos são adicionados à micromatriz de biopartículas, os neutrófilos que entram em contato com os clusters de biopartículas se ativam e iniciam a resposta ao enxame. A micromatriz de biopartículas foi validada usando microscopia fluorescente de lapso de tempo para rastrear a migração de neutrófilos para os clusters de biopartículas(Vídeo S1). A migração de núcleos de neutrófilos individuais é rastreada à medida que migram para o aglomerado de partículas. Quando os neutrófilos atingem o aglomerado de biopartículas, seus núcleos se sobrepõem a outros núcleos no aglomerado. Assim, não é possível rastrear com precisão um neutrófilo dentro do cluster usando este método. Os aglomerados de biopartículas zymosan e E. coli resultam na geração de enxames de neutrófilos. Para nossos resultados, a Figura 2B usa dados de enxames de neutrófilos gerados por partículas E. coli. A Figura 3 e os outros painéis da Figura 2 usam enxames de neutrófilos gerados por partículas de zimosan. Os resultados obtidos demonstram que os aglomerados de biopartículas estimulam a ativação de neutrófilos quando um neutrófilo entrou em contato com o cluster, o que acabou levando à formação de enxames de neutrófilos estáveis ao redor de cada aglomerado após 30-60 min(Figura 2A, topo). Em contrapartida, os neutrófilos não apresentaram migração coletiva na ausência de aglomerados de biopartículas (Figura 2A, fundo e Vídeo S2). Utilizando a intensidade fluorescente de núcleos de neutrófilo manchados a 405 nm, o tamanho médio do enxame de nêutrons em torno de 30 μm de diâmetro E. coli aglomerados de biopartículas foi encontrado como 1.490 ± 680 μm2 (média ± SD, Figura 2B, topo). A intensidade da fluorescência de uma determinada região de interesse onde não há aglomerados de biopartículas presentes foi aproximadamente constante ao longo do tempo, o que confirmou a ausência de migração coletiva nesse cenário (Figura 2B, fundo). Faixas de migração de neutrófilos mostram que os neutrófilos convergiram em um aglomerado de biopartículas quando um estava presente (Figura 2C, topo). Por outro lado, não foi observada convergência no sistema de controle (Figura 2C, inferior). A velocidade (distância percorrida/hora) dos neutrófilos swarming e não ativados foi medida e foi encontrada uma diferença estatisticamente significativa nas distribuições de velocidade (ANOVA, p < 0,0001), como mostrado na Figura 2D. A velocidade média para neutrófilos de enxame foi de 20,6 ± 13,0 μm/min (média ± SD), e a velocidade média para neutrófilos de controle foi de 2,0 ± 2,2 μm/min.
Além disso, foram analisadas a concentração de 16 proteínas liberadas durante o enxame (Figura 3). Calculou-se a concentração normalizada de cada proteína em diferentes pontos de tempo no processo de aquecimento (t = 0, 0,5, 1 e 3 h), onde a concentração normalizada é (C – Cmin) / (Cmax – Cmin). 10 proteínas aumentaram na concentração ao longo do aquecimento. Essas proteínas foram adipsina, galectin-3, GROa, IL-6R, MIP-1a, MMP-8, MMP-9, Nidogen-1, TIMP-1 e TLR2. Duas proteínas (pentraxiin 3 e RANK) diminuíram na concentração ao longo do enxame. As quatro proteínas restantes (clusterin, PF4, RANTES e Trappin-2) aumentaram durante a primeira hora de enxame, mas diminuíram depois disso. Em outras palavras, as concentrações dessas proteínas "aumentaram" durante o enxame. Das 16 proteínas identificadas, 12 proteínas foram identificadas em nossa publicação anterior5. Adipsina, galectin-3, nidogen-1, pentraxiin 3, TIMP-1 e TLR2 mostraram-se específicos do enxame, enquanto clusterin, IL-6R, MMP-8 e MMP-9, RANK e trappin-2 não eram5.

Figura 1: Produção de micromatriz de biopartículas. Um wafer de silício revestido com fotoresist negativa é exposto à luz UV através de uma fotomáscara cromada (A). Depois que o wafer de silício é assado e desenvolvido, um padrão de fotoresist permanece na superfície do wafer de silício. Este é o mestre wafer(B). Em uma placa de Petri, uma mistura PDMS é adicionada ao topo do wafer mestre. O PDMS é curado durante a noite a 65 °C para formar o molde PDMS(C). O molde PDMS é cortado do wafer mestre e um soco de biópsia é usado para socar selos PDMS individuais(D). Um selo PDMS(E)é revestido com solução CP (F). O carimbo é invertido em uma fina camada de solução CP (G). Depois de incubar na solução CP por 1h, o carimbo é manchado em um slide de vidro para remover o excesso de CP. Oito selos são então pressionados em um slide de vidro limpo com um peso de 5,6 ± 0,1 g, alinhado com um espaçador de imagem(H). Quando o carimbo é removido, permanece um padrão CP(I). Uma laje PDMS com poços precortados é aderida ao escorregador de vidro(J). Em seguida, uma solução de biopartículas é adicionada sobre o padrão CP (K). As biopartículas carregadas negativamente se ligam ao polieletrolito carregado positivamente através da interação eletrostática. A solução de biopartículas em excesso é lavada, deixando as biopartículas padronizadas em cima do padrão CP (L). (M) Imagem fluorescente da camada CP rotulada com FITC (barras de escala = 50 μm, imagem grande; Barra de escala = 25 μm, inset). (N) Imagem fluorescente de biopartículas de zimmosan padronizadas conjugadas com vermelho do Texas (barra de escala = 100 μm, imagem grande; Barra de escala = 50 μm, inset). Clique aqui para ver uma versão maior deste valor.

Figura 2: Crescimento de enxame swarm de nêutrons em torno de aglomerados de biopartículas. Na presença de aglomerados de biopartículas, os neutrófilos passam por migração coletiva em direção aos aglomerados de biopartículas (fundo,neutrófilos de controle). Quando não existem biopartículas presentes, os neutrófilos não realizam migração coletiva. (A)Na presença de aglomerados de biopartículas de zimosan, os neutrófilos formam enxames em 30 min. Sem clusters de biopartículas, sem formulário de enxames (barras de escala = 50 μm). (B) Enxames de nêutrons crescem a um tamanho médio de 1.490 ± 680 μm2 (média ± SD) em torno de 30 μm de diâmetro E. coli clusters de biopartículas. Os neutrófilos de controle exibem uma densidade constante que corresponde a nenhum crescimento enxame (ANOVA, p < 0,0001, n = 32 enxames de neutrófilos, N = 1 doador, barras de erro = desvio padrão). (C) As faixas de neutrófilos de enxame convergem sobre os aglomerados de biopartículas de zimosan, enquanto os neutrófilos de controle não mostram nenhum ponto convergente (barras de escala = 50 μm). (D) Os neutrófilos em direção a um alvo de zimmosan têm uma velocidade média de 20,6 ± 13,0 μm/min (média ± SD), enquanto os nêutrons de controle têm uma velocidade média de 2,0 ± 2,2 μm/min. Cada contagem no mapa de calor representa um neutrófilo com a velocidade instantânea dada no determinado ponto de tempo. Estes mapas de calor são representativos de um experimento (n = 1 enxame; N = 1 doador; ANOVA, 6.114 = neutrófilos, 32.116 = neutrófilos de controle, p < 0,0001). Clique aqui para ver uma versão maior deste valor.
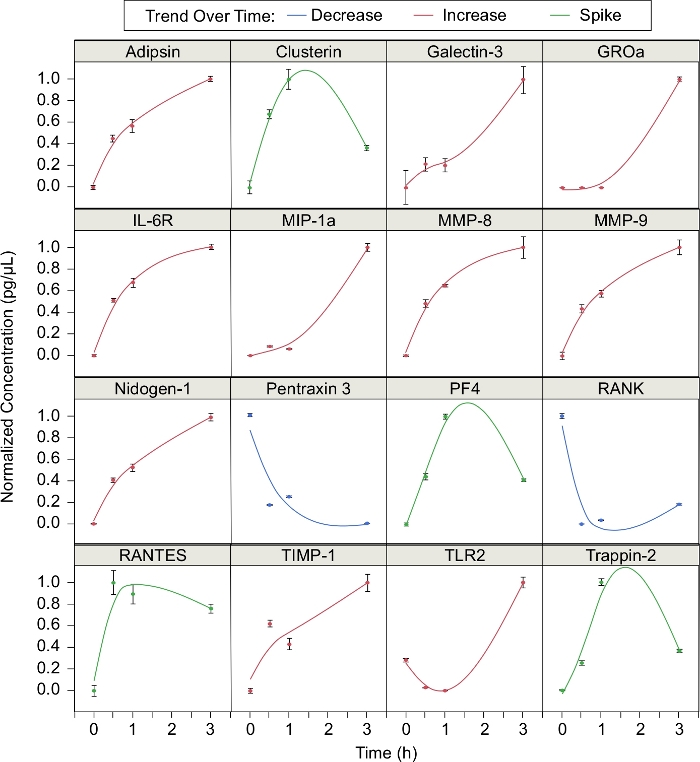
Figura 3: Mediadores livres liberados por neutrófilos. O enxame de neutrófilos afeta a produção de várias proteínas ao longo do tempo. A concentração de proteínas foi medida em 0, 0,5, 1 e 3h. Os pontos de dados foram equipados com splines de suavização (λ = 0,05). Essas proteínas tendem a seguir uma das três tendências características: diminuir com o tempo, aumentar ao longo do tempo ou espiar em torno de 1h e depois diminuir (barras de erro = desvio padrão; n = 3 réplicas; N = 1 doador). Clique aqui para ver uma versão maior deste valor.
Vídeo S1: Neutrófilo se aglomerando em direção a aglomerados de biopartículas zymosan. Núcleos nêutrons são mostrados em azul. Os alvos zymosanos são marcados com círculos vermelhos (Barra de escala = 50 μm; tempo original de aquisição = 60 min). Clique aqui para baixar este vídeo.
Vídeo S2: Migração aleatória de neutrófilo não ativado. Núcleos de nêutrons são mostrados em azul (Barra de escala = 50 μm; tempo original de aquisição = 60 min). Clique aqui para baixar este vídeo.
Discussão
Desenvolvemos uma plataforma de microestampagem para gerar matrizes uniformes de biopartículas para estimular o aquecimento in vitro de neutrófilos. A natureza in vitro de nossa plataforma nos permite contornar as complicações que surgem com experimentos in vivo, ou seja, a má capacidade de analisar mediadores liberados por neutrófilos5. Além disso, os modelos vivos são tipicamente realizados em roedores11,12,13,15,22,23, ou zebrafish11,12,15,23. Nossa plataforma utiliza neutrófilos humanos, o que nos permite interpretar mais diretamente nossos resultados no contexto da doença humana, embora certas semelhanças entre camundongos e neutrófilos humanos tenham sido observadas5,11. Além disso, mantemos um ambiente de enxame espacialmente controlado que distingue nossa plataforma de modelos in vivo, fornecendo um alto nível de reprodutibilidade que facilita a análise da migração coletiva de nêutrons humanos, bem como a coleta e análise de mediadores liberados por neutrófilos.
Durante o desenvolvimento do nosso protocolo de microestampagem, surgiram vários desafios que exigiram cuidadosa solução de problemas. Primeiro, o CP usado para o microcarimbo é altamente hidrofílico, e os selos PDMS são hidrofóbicos. Como o CP não tem uma alta afinidade com pdms, nosso procedimento foi cuidadosamente projetado para evitar a formação de bolhas e promover a maçaria. Ao primeiro preparar o selo com CP durante o confronto (etapa 1.8), minimizamos a formação de bolhas entre o CP e o carimbo. O carimbo é então invertido em uma camada de CP e incubado por 1h. Este longo tempo de incubação garante que cada seção do selo esteja molhada. Em segundo lugar, o processo de remoção do excesso de CP antes de estampar em um slide de vidro limpo (passo 1.11) pode ser inconsistente. Durante a realização da etapa 1.11, o selo deve ser cuidadosamente examinado. Quando o padrão começa a se tornar visível, o selo está pronto para a etapa 1.12. Além disso, o tempo de vácuo necessário para secar os selos (passo 1.12) pode variar. Isso depende principalmente do tempo. Em um dia quente e úmido, são necessários 2 min de tempo de vácuo. Em um dia frio e seco, 1 min de tempo de vácuo é suficiente.
Mostramos que neutrófilos de doadores saudáveis formam enxames estáveis em torno de aglomerados de partículas de partículas. Com microscopia fluorescente de lapso de tempo, podemos quantificar o tamanho do enxame e rastrear a migração de neutrófilos, o que nos permite analisar a quimiotaxe nutronphil quantitativamente5. Por exemplo, mostramos anteriormente que esta plataforma pode ser usada para calcular o índice quimiotático (IC, a cosseno do ângulo entre o vetor de velocidade de nêutrons e o vetor de posição entre o neutrófilo e o cluster de biopartículas mais próximo), velocidade (distância que o neutrófilo viaja dividido pelo tempo), velocidade radial (a velocidade multiplicada pelo IC) e a distância total percorrida (a diferença entre a posição inicial e última de neutrophil) de neutros migratórios individuais5 . Ao contrário da maioria dos estudos in vitro24,25,26, nossa plataforma não tem um gradiente quimiotático artificial, por isso a comunicação intercelular neutrófilo é a única força motriz da migração de neutrófilos. Além disso, o supernatante gerado por neutrófilos swarming é facilmente acessível. Podemos coletar e analisar o supernatante para mediadores intercelulares liberados por neutrófilos sem interferência de outros tipos de células que estão presentes no vivo. Observou-se 16 proteínas cuja expressão durante o enxame poderia ser descrita como uma das três tendências: aumento (10 proteínas), diminuição (duas proteínas) e pico (quatro proteínas). Seis dessas proteínas confirmaram tendências relatadas anteriormente durante o aquecimento ao longo do tempo5. Algumas das proteínas identificadas foram previamente mostradas como específicas do enxame, enquanto outras foram expressas diferencialmente por neutrófilos não-swarming ativados5. Proteínas que aumentam a concentração ao longo do tempo provavelmente estão associadas à resposta pró-inflamação. Algumas das proteínas que aumentam a concentração ao longo do tempo já são conhecidas por estarem envolvidas na resposta pró-inflamatória (por exemplo, galectin-3 e MMP-9)27,28. A relação entre outras proteínas e inflamação é menos bem compreendida. As proteínas que aumentam ou diminuem durante o enxame podem estar envolvidas na regulação da inflamação. No entanto, novas pesquisas são necessárias para entender o papel na inflamação de muitas das proteínas que são expressas diferencialmente durante o enxame. Analisar mediadores liberados juntamente com a migração de neutrófilos pode nos ajudar a entender melhor o quadro complexo de inflamação e como os neutrófilos impactam o tecido circundante durante o enxame.
Em estudos futuros, nossa plataforma pode ser usada para estudar minuciosamente a capacidade de neutrófilos insalubres para gerar enxames estáveis em torno de aglomerados de biopartículas padronizados. Várias condições médicas têm sido relacionadas a neutrófilos, incluindo sepse3,trauma6e câncer1,8,17,18, o que sugere que os neutrófilos nesses pacientes podem ter alterado a função. Esta plataforma pode ser usada para examinar diferenças entre os mediadores intercelulares liberados por neutrófilos saudáveis e insalubres. Além disso, esta plataforma poderia ser modificada para padronizar micróbios vivos e usada para analisar a resposta neutrófilo a micróbios vivos in vitro.
Em conclusão, desenvolvemos uma nova plataforma para analisar o enxame de neutrófiloin vitro. A natureza altamente controlada de nossa plataforma nos permite mitigar questões que surgem durante experimentos de aquecimento de nêutrons vivos. O enxame de neutrófilos em uma micromatriz de biopartículas é facilmente quantificável através de microscopia fluorescente de lapso de tempo. Além disso, podemos coletar os mediadores liberados pelos neutrófilos sem a interferência de outros tecidos que estão presentes no vivo. Esta plataforma pode ser usada em pesquisas futuras para quantificar diferenças entre os comportamentos migratórios de neutrófilos de doadores saudáveis e insalubres.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pelo financiamento do Departamento de Engenharia Química e Biomolecular William G. Lowrie e do Centro De Câncer Abrangente da Universidade Estadual de Ohio. Os dados apresentados neste relatório vieram de imagens processadas usando Imaris x64 (ver. 9.3.0 Bitplane) disponíveis no Campus Microscopy and Imaging Facility, The Ohio State University. Esta instalação é apoiada em parte pela bolsa P30 CA016058, Instituto Nacional de Câncer, Bethesda, MD.
Materiais
| Name | Company | Catalog Number | Comments |
| "The Big Easy" EasySep Magnet | STEMCELL Technologies | 18001 | Magnet to use with neutrophil isolation kit |
| Cell Incubator | Okolab | 777057437 / 77057343 | Okolab cage incubator for temperature and CO2 control |
| EasySep Human Neutrophil Isolation Kit | STEMCELL Technologies | 17957 | Kit for immunomagnetic negative selection of human neutrophils |
| Eclipse Ti2 | Nikon Instruments | MEA54010 / MEF55037 | Inverted research microscope |
| Escherichia coli (K-12 strain) BioParticles Texas Red conjugate | Invitrogen | E2863 | Bioparticle powder, dissolve in water prior to addition to Zetag® array |
| Harris Uni-Core 8-mm biopsy punch | Sigma Aldrich | Z708925 | To cut PDMS stamps |
| HetaSep | STEMCELL Technologies | 7906 | Erythrocyte aggregation agent for separating buffy coat from red blood cells in fresh human blood |
| Hoechst 33342 | Life Technologies | H3570 | Nucleus fluorescent stain |
| Human L1000 Array | Raybiotech Inc. | AAH-BLG-1000-4 | High density array to detect 1000 human proteins |
| Human Serum Albumin (HSA) | Sigma Aldrich | A5843 | Low endotoxin HSA, to prepare 2 % solutions in IMDM for isolated neutrophils |
| Iscove's Modified Dulbeccos' Medium (IMDM) | Thermo Fisher Scientific | 12440053 | To resuspend isolated neutrophils |
| K2-EDTA tubes | Thermo Fisher Scientific | 02-657-32 | Tubes for blood collection |
| Low Reflective Chrome Photomask | Front Range Photomask | N/A | Dimensions 5" x 5" x 0.09" (L x W x D) |
| Microarray Scanner | Perkin Elmer | ASCNGX00 | Fluorescence reader of protein patterned microdomains |
| Microscopy Image Analsysis Software - Imaris | Bitplane | 9.3.0 | Software for automatic cell tracking analysis |
| NiS Elements Advanced Research Software Package | Nikon Instruments | MQS31100 | Software for automatic live cell imaging and swarm size calculation |
| Poly-L-lysine fluorescein isothiocyanate (PLL-FITC) | Sigma Aldrich | P3069-10MG | 30,000 - 70,000 MW PLL labeled with FITC, used to fluorescently label CP solution |
| SecureSeal 8-well Imaging Spacer | Grace Bio-Labs | 654008 | 8-well, 9-mm diameter, adhesive imaging spacer |
| Silicon Wafer | University Wafer | 590 | Silicon 100 mm N/P (100) 0- 100 ohm-cm 500 μm SSP test |
| Spin Coater | Laurell | WS-650MZ-23NPPB | Used to spincoat a 40-µm layer of photoresist onto silicon wafer |
| SU-8 2025 | MicroChem | 2025 | Negative photoresist to make silicon master wafer |
| SU-8 Developer | MicroChem | Y020100 | Photoresist developer. Remove non-crosslinked SU-8 2025 from silicon wafer |
| Sylgard 184 (polydimethylsiloxane, PDMS) | Dow | 1673921 | 2-part silicone elastomer kit for making microstamps and PDMS wells |
| UV Exposure Masking System | Kloé | UV-KUB 2 | Used to crosslink photoresist on silicon wafer through chrome mask with UV light |
| Water | Thermo Fisher Scientific | A1287303 | High quality water to dilute bioparticles |
| Zetag 8185 | BASF | 8185 | Cationic polyelectrolyte (CP), powder, Copolymer of acrylamide and quaternized cationic monomer, forms "inking solution" for microstamping when dissolved in water |
| Zymosan A S. cerevisiae BioParticles Texas Red conjugate | Invitrogen | Z2843 | Bioparticle powder, dissolve in water prior to addition to Zetag array |
Referências
- Coffelt, S. B., Wellenstein, M. D., de Visser, K. E. Neutrophils in cancer: neutral no more. Nature Reviews Cancer. 16 (7), 431-446 (2016).
- Jones, C. N., et al. Microfluidic Platform for Measuring Neutrophil Chemotaxis from Unprocessed Whole Blood. Journal of Visualized Experiments. (88), 1-6 (2014).
- Ellett, F., et al. Diagnosis of sepsis from a drop of blood by measurement of spontaneous neutrophil motility in a microfluidic assay. Nature Biomedical Engineering. 2, 207-214 (2018).
- Malawista, S. E., Chevance de Boisfleury, A., Naccache, P. H. Inflammatory Gout: Observations over a Half-Century. The FASEB Journal. 25 (12), 4073-4078 (2011).
- Reátegui, E., et al. Microscale arrays for the profiling of start and stop signals coordinating human-neutrophil swarming. Nature Biomedical Engineering. 1 (7), 1-12 (2017).
- Morgan, A. S. Risk factors for infection in the trauma patient. Journal of the National Medical Association. 84 (12), 1019-1023 (1992).
- Szczerba, B. M., et al. Neutrophils escort circulating tumour cells to enable cell cycle progression. Nature. 566, 553-557 (2019).
- Treffers, L. W., Hiemstra, I. H., Kuijpers, T. W., van den Berg, T. K., Matlung, H. L. Neutrophils in cancer. Immunological Reviews. 273, 312-328 (2016).
- Grayson, P. C., Kaplan, M. J. At the Bench: Neutrophil extracellular traps (NETs) highlight novel aspects of innate immune system involvement in autoimmune diseases. Journal of Leukocyte Biology. 99 (2), 253-264 (2016).
- Lämmermann, T. In the eye of the neutrophil swarm--navigation signals that bring neutrophils together in inflamed and infected tissues. Journal of Leukocyte Biology. 100 (1), 55-63 (2016).
- Kienle, K., Lämmermann, T. Neutrophil swarming: an essential process of the neutrophil tissue response. Immunological Reviews. 273 (1), 76-93 (2016).
- de Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nature Reviews Immunology. 16 (6), 378-391 (2016).
- Lämmermann, T., et al. Neutrophil swarms require LTB4 and integrins at sites of cell death in vivo. Nature. 498 (7454), 371-375 (2013).
- Kreisel, D., et al. In vivo two-photon imaging reveals monocyte-dependent neutrophil extravasation during pulmonary inflammation. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18073-18078 (2010).
- Coombes, J. L., Robey, E. A. Dynamic imaging of host-pathogen interactions in vivo. Nature Reviews Immunology. 10 (5), 353-364 (2010).
- Lämmermann, T. Cell migration: Arraying neutrophils in swarms. Nature Biomedical Engineering. 1 (7), 1-2 (2017).
- Powell, D. R., Huttenlocher, A. Neutrophils in the Tumor Microenvironment. Trends in Immunology. 37 (1), 41-52 (2016).
- Uribe-querol, E., Rosales, C. Neutrophils in Cancer: Two Sides of the Same Coin. Journal of Immunology Research. 2015, (2015).
- Underhill, D. M., Ozinsky, A. Toll-like receptors: Key mediators of microbe detection. Current Opinion in Immunology. 14 (1), 103-110 (2002).
- Netea, M. G., Van Der Meer, J. W., Kullberg, B. J. Recognition of fungal pathogens by toll-like receptors. Immunology of Fungal Infections. , 259-272 (2007).
- Thomas, C. J., Schroder, K. Pattern recognition receptor function in neutrophils. Trends in Immunology. 34 (7), 317-328 (2013).
- Prince, L. R., Whyte, M. K., Sabroe, I., Parker, L. C. The role of TLRs in neutrophil activation. Current Opinion in Pharmacology. 11 (4), 397-403 (2011).
- Tan, S. Y., Weninger, W. Neutrophil migration in inflammation: intercellular signal relay and crosstalk. Current Opinion in Immunology. 44, 34-42 (2017).
- Menezes, G. B., Rezende, R. M., Pereira-Silva, P. E. M., Klein, A., Cara, D. C., Francischi, J. N. Differential involvement of cyclooxygenase isoforms in neutrophil migration in vivo and in vitro. European Journal of Pharmacology. 598 (1-3), 118-122 (2008).
- Afonso, P. V., et al. LTB4 Is a Signal-Relay Molecule during Neutrophil Chemotaxis. Developmental Cell. 22 (5), 1079-1091 (2012).
- Gounni, A. S., et al. In Vivo Regulates Neutrophil Migration In Vitro and Chemorepellent Semaphorin 3E Negatively Chemorepellent Semaphorin 3E Negatively Regulates Neutrophil Migration In Vitro and In Vivo. The Journal of Immunology. 198, 1023-1033 (2017).
- Dumic, J., Dabelic, S., Flögel, M. Galectin-3: An open-ended story. Biochimica et Biophysica Acta - General Subjects. 1760 (4), 616-635 (2006).
- Padmanabhan Iyer, R., et al. Matrix metalloproteinase-9-dependent mechanisms of reduced contractility and increased stiffness in the aging heart. Proteomics - Clinical Applications. 10 (1), 92-107 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados