Method Article
Microarrays de bioparticules pour l’analyse chimiotaxique et moléculaire de l’ésaimage humaine de neutrophile in vitro
Dans cet article
Résumé
Ce protocole génère des microréseaux de bioparticules qui fournissent un essaim de neutrophile contrôlé spatialement. Il permet un accès facile aux médiateurs qui neutrophiles libèrent pendant la migration et permet une analyse d’imagerie quantitative.
Résumé
L’essaimage de neutrophile est un processus coopératif par lequel les neutrophiles scellent un site d’infection et favorisent la réorganisation des tissus. L’essaimage a été classiquement étudiée in vivo dans des modèles animaux montrant des modèles caractéristiques de migration cellulaire. Cependant, les modèles in vivo ont plusieurs limites, y compris les médiateurs intercellulaires qui sont difficiles d’accès et d’analyse, ainsi que l’incapacité d’analyser directement les neutrophiles humains. En raison de ces limitations, il est nécessaire d’avoir une plate-forme in vitro qui étudie le grouillage de neutrophiles humains et facilite l’accès aux signaux moléculaires générés pendant l’essaimage. Ici, un processus de micro-amactinage en plusieurs étapes est utilisé pour générer un microréseau de bioparticules qui stimule l’essaimage en imitant une infection in vivo. Le microarray de bioparticules induit des neutrophiles à essaimer d’une manière contrôlée et stable. Sur le microarray, les neutrophiles augmentent en vitesse et forment des essaims stables autour des grappes de bioparticules. En outre, le supernatant généré par les neutrophiles a été analysé et 16 protéines ont été découvertes pour avoir été exprimées différemment au cours de l’essaimage. Cette plate-forme d’essaimage in vitro facilite l’analyse directe de la migration des neutrophiles et de la libération de protéines d’une manière reproductible et contrôlée spatialement.
Introduction
Neutrophiles, le globule blanc le plus abondant dans la circulation sanguine1, attirent l’attention en tant que cibles diagnostiques et thérapeutiques potentielles2,3 parce qu’ils peuvent être impliqués dans une variété de conditions médicales, y compris la goutte4, sepsis3, traumatisme5,6, cancer1,7,8, et diverses maladies auto-immunes5,9. Neutrophil essaimage est un processus en plusieurs étapes, étroitement réglementé avec une complexité qui en fait un centre particulièrement intéressant de l’étude5,10,11. Pendant l’essaimage, les neutrophiles isolent un site d’inflammation du tissu sain environnant5,10,11. Une réglementation appropriée de l’essaimage neutrophile est essentielle pour promouvoir la cicatrisation des plaies et, finalement, la résolution de l’inflammation5,12. Neutrophil essaimage a principalement été étudié in vivo chez les rongeurs12,13,14,15 et le poisson zèbre10,11,12,15 modèles. Cependant, la nature de ces modèles animaux in vivo donne lieu à des limitations5. Par exemple, les médiateurs libérés par les neutrophiles pendant l’essaimage ne sont pas facilement accessibles pour l’analyse5. En outre, il existe de nombreuses sources potentielles pour un médiateur donné in vivo, de sorte qu’une expérience in vivo doit introduire une déficience génétique pour inhiber la production cellulaire et / ou l’interaction afin d’étudier le rôle de ce médiateur dans un processus donné13. Une expérience in vitro contourne cette complication en permettant l’observation des neutrophiles sans le contexte de cellules supplémentaires. En outre, la recherche décrivant la migration coordonnée des neutrophiles humains est limitée16. Sur une plate-forme d’essaimage in vitro, les neutrophiles humains peuvent être analysés directement. Une plate-forme d’essaimage in vitro pourrait élargir les connaissances acquises à partir d’études in vivo en offrant des possibilités de combler les lacunes laissées par les limites des études in vivo.
Pour répondre à la nécessité d’une plate-forme in vitro qui imite l’essaimage in vivo neutrophile, nous avons développé une plate-forme de micro-estampillage qui nous permet de modeler des microréseaux de bioparticules qui stimulent l’essaimage de neutrophiles d’une manière contrôlée spatialement. Nous produisons des microréseaux de bioparticules sur des lames de verre en deux étapes. Tout d’abord, nous utilisons le micro-estampage pour générer un microarray de taches de polyélectrolyte cationique (CP). Deuxièmement, nous ajoutons une solution de bioparticules qui adhèrent aux taches de CP par l’interaction électrostatique. En modelant d’abord la couche de CP, nous pouvons modeler sélectivement les bioparticules chargées négativement pour produire le modèle d’essaimage désiré de neutrophile. La couche chargée positivement retient les bioparticules chargées négativement par l’étape de lavage vigoureuse qui enlève les bioparticules des secteurs sur la glissière de verre qui n’ont pas le CP. En outre, le CP utilisé ici, un copolymère d’acrylamide et de monomère cationique quatertisée, est biocompatible, de sorte qu’il n’induit pas une réponse des neutrophiles. Il a une charge de surface très élevée qui immobilise les bioparticules de la taille d’un micron à la glissière de verre, empêchant ainsi les neutrophiles d’enlever les particules de la position modelée sur la glissière de verre. Il en résulte des grappes de bioparticules disposées en microarray. Lorsque nous avons ajouté des neutrophiles au microréseau, ils ont formé des essaims stables autour des grappes de bioparticules. En suivant la migration des neutrophiles, nous avons constaté que les neutrophiles grouillants migrent activement vers les grappes de bioparticules. En outre, nous avons utilisé cette plate-forme pour analyser certains médiateurs qui neutrophiles libèrent pendant l’essaimage. Nous avons trouvé 16 médiateurs qui sont exprimés différemment pendant l’essaimage. Leurs concentrations suivent trois tendances générales au fil du temps : augmentation, diminution ou pic. Notre plate-forme d’essaimage in vitro neutrophile facilite l’analyse de l’essaimage de neutrophiles humains contrôlés spatialement, ainsi que la collecte et l’analyse des médiateurs libérés par l’essaimage neutrophile. Dans une publication précédente, nous avons démontré que les patients présentant certaines conditions médicales (traumatisme, maladie auto-immune, et sepsis), ont eu des neutrophiles qui ont fonctionné différemment de ceux des donateurs en bonne santé5. Dans de futures études de recherche, notre plate-forme pourrait être utilisée pour analyser la fonction neutrophile parmi une variété de populations de patients. Cette plate-forme peut analyser quantitativement la coordination complexe impliquée dans l’essaimage de neutrophiles. D’autres études peuvent être faites pour fournir un aperçu sur la fonction neutrophile d’une population spécifique de patients ou la réponse de neutrophile à un agent pathogène d’intérêt.
Protocole
Les auteurs reconnaissent les volontaires en bonne santé qui ont gentiment donné leur sang. Des échantillons de sang ont été obtenus après le consentement éclairé du volontaire selon le protocole de la commission d’examen institutionnel (CISR) #2018H0268 examiné par le Comité des sciences biomédicales de l’Université d’État de l’Ohio.
1. Microfabrication de microréseau de bioparticules
- À l’aide de procédures photolithographie standard, générer la plaquette de silicium maître.
- Générez une preuve de la conception souhaitée à l’aide d’un logiciel de conception assistée par ordinateur (CAD), puis envoyez à un fabricant de photomasques pour produire un photomask chromé. La conception utilisée ici est de 4 mm x 4 mm de tableaux rectangulaires de 30 m de diamètre remplis en cercles avec un espacement centre-centre de 150 m. Cette conception peut être modifiée comme souhaité pour différentes applications.
- Spincoat une couche de 40 m d’épaisseur d’un photoresist négatif sur une plaquette de silicium. Cuire la plaquette à 65 oC pendant 5 min et 95 oC pendant 10 min.
- Exposer la plaquette à la lumière UV à l’égard d’un photomasque chromé avec un masque photo de 150 à 160 mJ/cm2 (Figure 1A).
- Cuire la plaquette à 65 oC pendant 5 min et 95 oC pendant 10 min. Submergez la plaquette dans le développeur photorésistant pendant 10 min et rincez-la à l’alcool isopropyl. À ce stade, le motif doit être visible sur la plaquette (figure 1B).
- Mélanger soigneusement un rapport de 10:1 de polydiméthylsiloxane prépolymère et son agent de durcissement (c.-à-d., 20 g de prépolymère et 2 g d’agent de durcissement) et verser le mélange de polydiméthylsiloxane (PDMS) non durci sur la plaquette principale dans un plat Petri (Figure 1C).
- L’aspirateur traiter le mélange non durci de PDMS jusqu’à ce qu’aucune bulle d’air ne soit présente au-dessus de la plaquette principale. Cure à 65 oC pendant la nuit.
- Utilisez un scalpel pour couper autour de l’extérieur de la section à motifs de la plaquette, et enlever lentement la dalle PDMS durcie. Placez la dalle PDMS sur une planche à découper propre avec le côté à motifs orienté vers le haut (Figure 1D).
- Frappez les timbres individuels de la dalle PDMS avec un poinçon de biopsie de 8 mm (Figure 1E). Pour un toboggan en verre, huit timbres seront nécessaires.
- Placez chaque timbre face vers le bas sur le ruban adhésif pour enlever les débris.
- À l’avance, préparer une solution de CP de 1,6 mg/ml dans l’eau.
- Ajouter la bonne quantité de poudre de CP à l’eau (p. ex., 0,8 g à 500 ml).
- Mélanger sur une plaque à remuer à température ambiante pendant la nuit, ou jusqu’à ce que tout le solide soit dissous dans l’eau. La solution CP peut être stockée à température ambiante pendant 6 mois.
- Si désiré, rendre la solution CP fluorescente en ajoutant de la polylysine étiquetée avec de l’isothiocyanate de fluorescéine (PLL-FITC).
- Aliquot environ 10 ml de la solution CP. Ajoutez une petite quantité de PLL-FITC (0,05 mg) au volume aliquoted. La quantité peut être modifiée pour ajuster la luminosité de la fluorescence comme désiré.
- Vortex la solution CP étiqueté avec FITC pour 20 s, ou jusqu’à ce que la solution est d’un uniforme, couleur jaune pâle. Protégez-vous de la lumière et conservez-les à 4 oC jusqu’à 1 mois.
- Avec les timbres face haute, amorcez chaque timbre avec une solution de 1,6 mg/mL de CP, assurant qu’il n’y a pas de bulles d’air entre la solution CP et le timbre (Figure 1F).
- Inverser les timbres sur une couche de solution CP (Figure 1G).
- Retirez les timbres de la solution CP après 1 h.
- Tamponnez chaque tampon humide face vers le bas sur une glissière en verre propre 6-8x pour enlever l’excès de liquide.
- Traiter les timbres à l’aspirateur pendant 1 h à 2 min.
- Adhérez un espaceur d’imagerie de huit puits de 9 mm de diamètre sur le dessus d’une glissière en verre propre comme guide pour le placement des timbres. Avec cet espaceur, chaque lame de verre peut avoir huit microarrays.
- Placez un timbre face vers le bas sur la glissière de verre au centre de chaque puits de l’espaceur d’imagerie (c.-à-d., utilisez huit timbres au total).
- Placer un poids équilibré de 5,6 à 0,1 g sur chaque timbre et laisser 10 min pour l’estampage (figure 1H). Un poids équilibré est nécessaire pour s’assurer que le timbre est pressé uniformément sur la glissière de verre et favoriser le transfert même du CP à la glissière de verre.
- Retirez les poids et les timbres de la glissière en verre (Figure 1I). Laisser sécher la couche de CP à température ambiante pendant 24 h avant d’ajouter les bioparticules, telles que décrites à l’étape 1.17. Si le CP est étiqueté avec le FITC, l’efficacité de l’estampillage peut être vérifiée à ce stade avec un microscope fluorescent à 488 nm avant de passer à l’étape 1.17 (figure 1M).
- Couper une dalle Vierge PDMS à la taille de l’espaceur d’imagerie et utiliser le poinçon de biopsie de 8 mm pour créer des puits dans le PDMS qui s’alignent avec les puits d’un espaceur d’imagerie. Adhérer à la dalle PDMS à la glissière de verre avec l’espaceur d’imagerie (Figure 1J).
- Décongeler une solution de bioparticules (p. ex. E. coli ou zymosan) et diluer à 500 g/mL dans de l’eau pour injection (WFI).
REMARQUE : Les bioparticules n’ont pas besoin d’être opsonisées. Les récepteurs de surface de Neutrophile reconnaissent directement des molécules sur ces bioparticules19,20,21. - Ajouter 100 l de solution de bioparticules à chaque Puits de PDMS sur la glissière de verre (Figure 1K).
- Basculer la glissière en verre pendant 30 min.
- Rincer soigneusement les puits à l’eau. Le microréseau de bioparticules peut être stocké dans un environnement sans poussière à 4 oC pendant une durée pouvant aller jusqu’à 3 mois. À ce stade, le modèle doit être vérifié avec un microscope fluorescent à 594 nm avant de passer à l’étape 2.1 (Figure 1N).
2. Préparation de l’échantillon
- Recueillir au moins 2 ml de sang frais dans les tubes K2-EDTA du donneur désiré. Le rendement prévu des neutrophiles est de 1/2 x 106 cellules/1 mL de sang entier. L’analyse d’imagerie nécessite environ 1,5 x 105 neutrophiles, et l’analyse du supernatant nécessite 1 x 106 neutrophiles. Utilisez le sang dans les 4 h.
- Séparer les globules rouges (RBC) en ajoutant un agent d’agrégation d’érythrocytes dans un rapport de 1:5 au sang entier. Attendez 45 min pour qu’une couche translucide (manteau de buffy) se sépare de la couche de RBCs.
- Retirer le pelage poli et laver avec du phosphate tamponné salin (PBS) à l’aide d’une couche bouffie de 1 ml : 9 mL de PBS.
- Centrifugeuse de 5 min à 190 x g et 20 oC.
- Aspirer le supernatant et resuspendre la pastille à 5 x 107 cellules/mL.
- Utilisez un kit de sélection immunomagnétique négatif pour isoler les neutrophiles.
- Ajouter 50 ll de cocktail anticorps/1 mL de suspension cellulaire. Attendez 10 min.
- Ajouter 100 l de perles magnétiques/1 mL de suspension cellulaire. Attendez 10 min.
- Ajouter la suspension cellulaire à un tube à fond rond et placer dans un aimant cylindrique. Attendez 10 min.
- Verser le supernatant dans un tube de centrifugeuse. Ajouter jusqu’à 10 ml de PBS. Centrifugeuse de 5 min à 190 x g et 20 oC.
- Aspirate supernatant. Resuspendre la pastille blanche dans IMDM avec 0.4% d’albumine humaine de sérum.
- Tôle noyaux avec 20 g/mL Hoechst 33342 pendant 10 min à 37 oC.
- Ajouter 5 mL d’IMDM avec 0,4% d’albumine de sérum humain pour rincer. Centrifugeuse de 5 min à 190 x g et 20 oC.
- Resuspendre les cellules à 7,5 x 105 cellules/mL dans IMDM avec 0,4% d’albumine de sérum humain.
- Ajouter 100 l de la suspension cellulaire à un puits PDMS contenant un microréseau de bioparticules. Assurez-vous que la suspension cellulaire est convexe sur le dessus du PDMS bien et ne contient pas de bulles.
- Scellez avec un bordereau de 12 mm de diamètre. Couvrez bien l’ouverture du PDMS d’un bordereau de 12 mm de diamètre. Appuyez doucement sur la glissière de couverture avec une pince à épiler afin que l’excédent de suspension cellulaire s’échappe au bord du puits. Utilisez un tissu pour enlever l’excédent de suspension cellulaire.
3. Exécution de l’analyse d’analyse et d’image
- Chargez le réseau de microparticules avec des cellules sur la station d’imagerie cellulaire vivante avec un microscope équipé d’un incubateur de cage réglé à 37 oC, 5 % de CO2et 90 % d’humidité relative.
- Utilisez la microscopie fluorescente et brightfield en time-lapse pour enregistrer des images à 10x grossissement tous les 10 s à 405 nm, 594 nm, et brightfield. Dans une expérience typique, les images sont collectées jusqu’à 2 h.
- Utilisez un logiciel automatisé de suivi cellulaire pour suivre la migration des neutrophiles individuels vers le cluster de bioparticules.
- Utilisez le mode d’autorégression d’un logiciel de suivi des cellules de détection ponctuelle. Définir le rayon de la tache à 5 m (la taille approximative d’un noyau de neutrophiles). Définir la longueur minimale de la voie à 120 s et une taille d’écart maximale d’un cadre.
- À partir des données générées par le logiciel de suivi cellulaire, extraire les fichiers qui contiennent la position et la vitesse des neutrophiles. Ces fichiers peuvent être utilisés avec un logiciel de graphique pour générer des pistes de migration neutrophiles (figure 2C) et une carte thermique de la vitesse par rapport au temps (Figure 2D), respectivement.
- Utilisez les images fluorescentes de 405 nm pour suivre la taille des essaims au fil du temps sur un logiciel d’analyse d’images de votre choix.
- Définir les régions d’intérêt (ROI) autour de chaque grappe de bioparticules où les neutrophiles essaimeront. Gardez la même taille roi pour analyser chaque grappe de bioparticules.
- Analyser l’intensité fluorescente moyenne des images de 405 nm dans chaque retour sur investissement au fil du temps.
- Générez une courbe d’étalonnage d’intensité fluorescente moyenne à la taille de l’essaim en prenant des mesures manuelles à différentes tailles d’essaim s’établissant à partir de 0 m2 à la taille maximale de l’essaim. Utilisez cet étalonnage pour calculer la taille de l’essaim au fil du temps.
4. Collecte de supernatants et détection de protéines
- Incuber les neutrophiles dans les puits contenant le microréseau de bioparticules à 37 oC et 5 % de CO2 pour 3 h. Prélever des échantillons aux moments souhaités. En règle générale, les échantillons seront prélevés à 0, 0,5, 1 et 3 h. Pour dépasser la limite de détection de l’analyse du tableau de protéines, le volume entier de supernatant d’un seul puits (200 L) a été utilisé pour chaque point de temps. Chaque point de temps a été analysé en triplicate.
- À l’aide d’un microscope à champ lumineux, vérifiez que des essaims sont formés sur le microréseau.
- Aspirez le supernatant avec une pipette de 200 l et chargez dans un tube de filtre de centrifugeuse de 0,45 m.
- Centrifuger le supernatant à 190 x g et 20 oC pendant 5 min et recueillir le volume filtré.
- Conserver les échantillons à -80 oC jusqu’au moment de traitement.
- Utilisez un kit de microarray qui détecte une gamme de protéines humaines pour traiter les échantillons.
- Ajouter 200 l de chaque échantillon à un tube de dialyse séparé fourni avec le kit.
- Placer les tubes de dialyse dans un bécher contenant au moins 500 ml de PBS (pH à 8,0). Remuer délicatement sur une plaque à remuer pendant au moins 3 h à 4 oC. Changez le PBS dans le bécher et répétez cette étape.
- Transférer chaque échantillon dans un tube de centrifugeuse propre et une centrifugeuse à 9 000 x g pendant 5 min pour enlever les précipités. Transférer chaque supernatant dans un tube propre.
- Biotinylate chaque échantillon en ajoutant 36 L de réactif d’étiquetage 1x du kit par 1 mg de protéines totales dans l’échantillon dialysé à 180 l l d’échantillon dialysé. Incuber à 20 oC pendant 30 min. Mélanger délicatement toutes les 5 min.
- Ajouter 3 ll de solution d’arrêt fournie avec le kit dans chaque tube d’échantillon. Transférer chaque échantillon dans un tube de dialyse frais et répéter les étapes 4.6.2-4.6.3. À ce stade, l’échantillon peut être stocké à -20 oC ou -80 oC jusqu’à ce que vous soyez prêt à procéder.
- La lame de verre fournie avec le kit est stockée à -20 oC. Laissez-le venir à la température ambiante. Placez la glissière en verre assemblée dans un capuchon à débit laminaire pendant 1 h et 2 h à température ambiante.
- Ajouter 400 l l de la mémoire tampon de blocage fournie avec le kit dans chaque puits de la glissière en verre assemblée. Incuber à température ambiante pendant 30 min.
- Centrifuger les échantillons préparés pendant 5 min à 9 000 x g pour éliminer les précipités ou les particules. Diluer 5x avec le tampon de blocage.
- Retirez le tampon de blocage de chaque puits. Ajouter 400 l d’échantillons dilués dans les puits appropriés. Incuber pendant 2 h à température ambiante tout en se balançant.
- Décant les échantillons de chaque puits. Laver 3x avec 800 l de la mémoire tampon de lavage 1x j’ai fourni avec le kit à température ambiante pendant 5 min chacun tout en berçant.
- Dans un récipient propre, immerger la glissière en verre assemblée dans un tampon de lavage 1x I. Laver 2x à température ambiante pendant 5 min chacun tout en berçant.
- Ajouter 400 L de streptavidinie conjuguée au Cy3 1x à chaque sous-array. Couvrir de bandes adhésives en plastique. Protégez-vous de la lumière pour le reste du protocole.
- Incuber pendant 2 h à température ambiante tout en se balançant.
- Décant la solution et démontez la glissière en verre des chambres d’échantillon.
- Dans le tube de centrifugeuse de 30 ml fourni avec le kit, ajouter soigneusement la glissière en verre et assez de tampon de lavage 1x I pour couvrir la glissière en verre. Laver 3x pendant 10 min chacun à température ambiante tout en berçant.
- Dans le tube de centrifugeuse de 30 ml, laver 2x avec 1x tampon de lavage II pendant 5 min chacun à température ambiante tout en se balançant.
- Laver la lame de verre avec 30 ml de ddH2O pendant 5 min. Retirez la glissière en verre du tube de centrifugeuse et laissez sécher pendant 20 min dans une hotte à débit laminaire. La lame de verre préparée peut être entreposée à -20 oC jusqu’à ce qu’elle soit prête à être numérisée.
- Scanner la glissière en verre à l’eau à l’effet d’une émission de fluorescence de 555 nm.
Résultats
Lorsque des neutrophiles sont ajoutés au microréseau de bioparticules, les neutrophiles qui contactent les grappes de bioparticules s’activent et déclenchent la réponse de l’essaimage. Le microréseau de bioparticules a été validé à l’aide d’une microscopie fluorescente en accéléré pour suivre la migration des neutrophiles vers les grappes de bioparticules (Vidéo S1). La migration des noyaux neutrophiles individuels est suivie lorsqu’ils migrent vers le groupe de bioparticules. Lorsque les neutrophiles atteignent le groupe de bioparticules, leurs noyaux se chevauchent avec d’autres noyaux de l’amas. Ainsi, il n’est pas possible de suivre avec précision un neutrophile dans le cluster à l’aide de cette méthode. Les grappes de bioparticules zymosan et E. coli entraînent toutes deux la production d’essaims de neutrophiles. Pour nos résultats, la figure 2B utilise les données des essaims de neutrophiles générées par les particules E. coli. La figure 3 et les autres panneaux de la figure 2 utilisent des essaims de neutrophiles générés par des particules zymosan. Les résultats obtenus démontrent que les grappes de bioparticules stimulent l’activation des neutrophiles lorsqu’un neutrophile a contacté le cluster, ce qui a finalement conduit à la formation d’essaims stables de neutrophiles autour de chaque amas après 30 à 60 min (Figure 2A, en haut). En revanche, les neutrophiles n’ont pas montré de migration collective en l’absence de grappes de bioparticules(figure 2A, en bas et vidéo S2). En utilisant l’intensité fluorescente des noyaux neutrophiles tachés à 405 nm, la taille moyenne des essaims de neutrophiles d’environ 30 m de diamètre des grappes de bioparticules E. coli s’est avérée être de 1 490 à 680 m2 (moyenne - SD, figure 2B, en haut). L’intensité de fluorescence d’une région d’intérêt donnée où il n’y a pas de grappes de bioparticules n’était pas présente était à peu près constante au fil du temps, ce qui a confirmé l’absence de migration collective dans ce contexte (figure 2B, en bas). Les traces de migration des neutrophiles montrent que les neutrophiles ont convergé vers un amas de bioparticules en présence(figure 2C, en haut). Inversement, aucune convergence n’a été observée dans le système de contrôle(figure 2C, en bas). La vitesse (distance parcourue/temps) des neutrophiles essaimage et non activés a été mesurée et une différence statistiquement significative dans les distributions de vitesse a été constatée (ANOVA, p lt; 0,0001), comme le montre la figure 2D. La vitesse moyenne pour les neutrophiles grouillants était de 20,6 m/m/min (moyenne et SD), et la vitesse moyenne des neutrophiles témoins était de 2,0 à 2,2 m/min.
De plus, la concentration de 16 protéines libérées par les neutrophiles lors de l’essaimage a été analysée (Figure 3). La concentration normalisée de chaque protéine à des moments différents dans le processus d’essaimage (t - 0, 0,5, 1, et 3 h) ont été calculées, où la concentration normalisée est (C - Cmin) / (Cmax - Cmin). 10 protéines ont augmenté en concentration tout au long de l’essaimage. Ces protéines étaient adipsine, galectin-3, GROa, IL-6R, MIP-1a, MMP-8, MMP-9, Nidogen-1, TIMP-1, et TLR2. Deux protéines (pentraxin 3 et RANK) ont diminué dans la concentration tout au long de l’essaimage. Les quatre autres protéines (clusterin, PF4, RANTES et Trappin-2) ont augmenté au cours de la première heure d’essaimage, mais ont diminué par la suite. En d’autres termes, les concentrations de ces protéines "spiked" pendant l’essaimage. Sur les 16 protéines identifiées, 12 protéines ont été identifiées dans notre publication précédente5. Adipsin, galectin-3, nidogen-1, pentraxin 3, TIMP-1, et TLR2 ont été montrés pour être essaimage-spécifique, tandis que clusterin, IL-6R, MMP-8, et MMP-9, RANK, et trappin-2 n’étaient pas5.

Figure 1 : Production de microréseau de bioparticules. Une plaquette de silicium recouverte d’un photorésistant négatif est exposée à la lumière UV à l’intermédiaire d’un photomasque chromé (A). Une fois la plaquette de silicium cuite et développée, un motif photorésistant reste à la surface de la plaquette de silicium. C’est la plaquette maître (B). Dans un plat Petri, un mélange PDMS est ajouté au dessus de la plaquette principale. Le PDMS est guéri pendant la nuit à 65 oC pour former le moule PDMS (C). Le moule PDMS est coupé de la plaquette principale et un poinçon de biopsie est utilisé pour poinçonner les timbres PDMS individuels (D). Un timbre PDMS (E) est recouvert d’une solution CP (F). Le timbre est inversé sur une fine couche de solution CP (G). Après avoir incubé dans la solution CP pendant 1 h, le timbre est effacé sur une lame de verre pour enlever l’excès de CP. Huit timbres sont ensuite pressés sur une lame de verre propre avec un poids de 5,6 à 0,1 g, aligné saillage avec un espaceur d’imagerie (H). Lorsque le timbre est enlevé, un modèle CP reste (I). Une dalle PDMS avec des puits prédécoupés est adhérée à la glissière de verre (J). Ensuite, une solution de bioparticules est ajoutée sur le modèle CP (K). Les bioparticules chargées négativement se lient au polyélectrolyte chargé positivement par l’intermédiaire de l’interaction électrostatique. La solution de bioparticules excédentaire est emportée, laissant les bioparticules modelées sur le modèle CP (L). (M) Image fluorescente de la couche CP étiquetée avec FITC (barres d’échelle de 50 m, grande image; Barre d’échelle de 25 m, enset). (N) Image fluorescente de bioparticules zymosan à motifs conjuguées à Texas Red (barre d’échelle de 100 m, grande image; Barre d’échelle de 50 m, enset). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Croissance des essaims de neutrophiles autour des grappes de bioparticules. En présence de grappes de bioparticules, les neutrophiles subissent une migration collective vers les grappes de bioparticules (enbas, contrôlez les neutrophiles). Lorsqu’il n’y a pas de bioparticules, les neutrophiles n’effectuent pas de migration collective. (A) En présence de grappes de bioparticules zymosan, les neutrophiles forment des essaims en 30 min. Sans grappes de bioparticules, aucun essaim ne se forme (barres d’échelle de 50 m). (B) Les essaims de neutrophiles atteignent une taille moyenne de 1 490 à 680 m2 (moyenne et DD) autour de grappes de bioparticules E. coli de 30 m de diamètre. Les neutrophiles de contrôle présentent une densité constante qui ne correspond à aucune croissance d’essaim (ANOVA, p 'lt; 0,0001, n '32 essaims de neutrophiles, N '1 donateur, barres d’erreur ' déviation standard). (C) Les traces de neutrophiles grouillant convergent vers les grappes de bioparticules zymosan, tandis que les neutrophiles de contrôle ne montrent aucun point de convergence (barres d’échelle de 50 m). (D) Les neutrophiles qui essaiment vers une cible zymosan ont une vitesse moyenne de 20,6 à 13,0 m/min (moyenne et SD), tandis que les neutrophiles de contrôle ont une vitesse moyenne de 2,0 à 2,2 m/min. Chaque compte sur la carte thermique représente un neutrophile avec la vitesse instantanée donnée au point de temps donné. Ces cartes thermiques sont représentatives d’une expérience (n 1 essaim; N - 1 donneur; ANOVA, 6 114 neutrophiles essaimage, 32 116 neutrophiles de contrôle, p 'lt; 0,0001). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
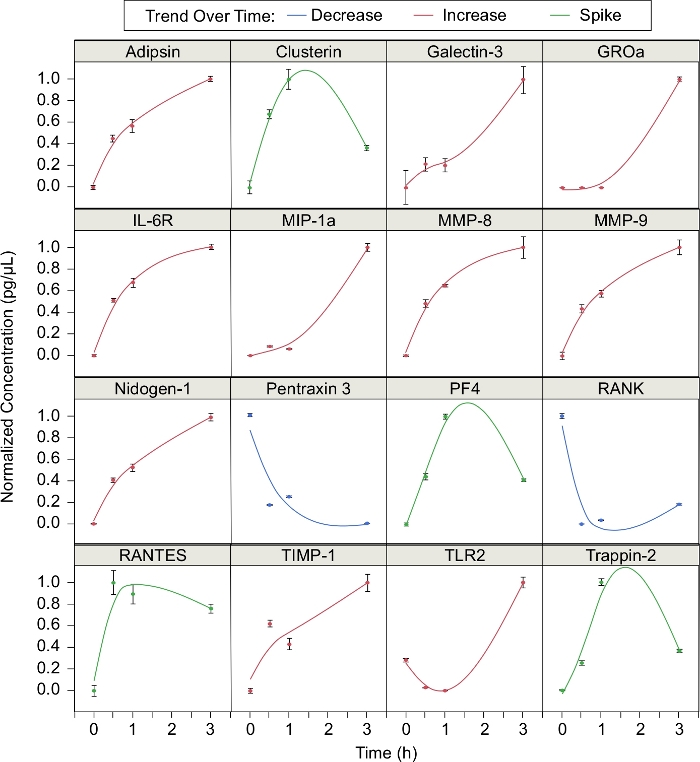
Figure 3 : Médiateurs libres libérés par des neutrophiles grouillants. L’essaimage de neutrophile affecte la production de diverses protéines au fil du temps. La concentration en protéines a été mesurée à 0, 0,5, 1 et 3 h. Les points de données ont été équipés de splines lissants (0,05 euros). Ces protéines ont tendance à suivre l’une des trois tendances caractéristiques : diminuer avec le temps, augmenter avec le temps, ou augmenter autour de 1 h, puis diminuer (barres d’erreur - déviation standard; n ' 3 répliques; N 1 donneur). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Vidéo S1 : Neutrophil grouillant vers les amas de bioparticules zymosan. Les noyaux neutrophiles sont représentés en bleu. Les cibles zymosan sont marquées de cercles rouges (barre d’échelle de 50 m; temps d’acquisition d’origine à 60 min). S’il vous plaît cliquez ici pour télécharger cette vidéo.
Vidéo S2 : Migration aléatoire de neutrophiles non activés. Les noyaux neutrophiles sont représentés en bleu (barre d’échelle de 50 m; temps d’acquisition d’origine à 60 min). S’il vous plaît cliquez ici pour télécharger cette vidéo.
Discussion
Nous avons développé une plate-forme de micro-ampillage pour générer des réseaux uniformes de bioparticules pour stimuler l’essaimage in vitro de neutrophiles. La nature in vitro de notre plate-forme nous permet de contourner les complications qui surgissent avec les expériences d’essaimage in vivo, à savoir la faible capacité d’analyser les médiateurs libérés par les neutrophiles grouillant s’agitent5. En outre, les modèles in vivo sont généralement effectués chez les rongeurs11,12,13,15,22,23, ou le poisson zèbre11,12,15,23. Notre plate-forme utilise des neutrophiles humains, ce qui nous permet d’interpréter plus directement nos résultats dans le contexte de la maladie humaine, bien que certaines similitudes entre la souris et les neutrophiles humains ont été observées5,11. En outre, nous maintenons un environnement d’essaimage contrôlé spatialement qui distingue notre plate-forme des modèles in vivo en fournissant un niveau élevé de reproductibilité qui facilite l’analyse de la migration collective neutrophile humaine ainsi que la collecte et l’analyse des médiateurs libérés par les neutrophiles grouillants.
Au cours de l’élaboration de notre protocole de micro-estampillage, plusieurs défis se sont posés qui ont nécessité un dépannage minutieux. Tout d’abord, le CP utilisé pour le micro-estampillage est très hydrophile, et les timbres PDMS sont hydrophobes. Parce que le CP n’a pas une forte affinité pour Le SPD, notre procédure a été soigneusement conçue pour éviter la formation de bulles et favoriser le mouillage. En amorçant d’abord le timbre avec le CP tout en faisant face-à-face (étape 1.8), nous minimisons la formation de bulles entre le CP et le timbre. Le timbre est ensuite inversé sur une couche de CP et incubé pendant 1 h. Ce long temps d’incubation garantit que chaque section du timbre est mouillée. Deuxièmement, le processus d’élimination de l’excès de CP avant l’estampillage sur une glissière en verre propre (étape 1.11) peut être incohérent. Lors de l’étape 1.11, le timbre doit être soigneusement examiné. Lorsque le motif commence à devenir visible, le timbre est prêt pour l’étape 1.12. En outre, le temps d’aspirateur requis pour sécher les timbres (étape 1.12) peut varier. Cela dépend principalement de la météo. Par une journée chaude et humide, 2 min de temps sous vide sont nécessaires. Par temps frais et sec, 1 min de temps sous vide suffit.
Nous avons montré que les neutrophiles des donneurs sains forment des essaims stables autour des grappes de bioparticules. Avec la microscopie fluorescente en time-lapse, nous pouvons quantifier la taille des essaims et suivre la migration des neutrophiles, ce qui nous permet d’analyser quantitativement la chimiotaxie neutrophile5. Par exemple, nous avons déjà montré que cette plate-forme peut être utilisée pour calculer l’indice chimiotaxique (CI, la cosine de l’angle entre le vecteur de vitesse neutrophile et le vecteur de position entre le neutrophile et le cluster de bioparticules le plus proche), la vitesse (distance du neutrophile se déplace divisé par le temps), la vitesse radiale (la vitesse multipliée par CI), et la distance totale parcourue (la différence entre la position initiale et finale des neutrophiles) des neutrophiles migrateurs individuels5. Contrairement à la plupart des études in vitro24,25,26, notre plate-forme n’a pas un gradient chimiothérapeutique artificiel, de sorte que la communication intercellulaire neutrophile est la seule force motrice de la migration neutrophile. En outre, le supernatant généré par les neutrophiles grouillant est facilement accessible. Nous pouvons recueillir et analyser le supernatant pour les médiateurs intercellulaires libérés par les neutrophiles sans interférence d’autres types de cellules qui sont présents in vivo. 16 protéines ont été observées dont l’expression pendant l’essaimage pourrait être décrite comme l’une des trois tendances : augmentation (10 protéines), diminution (deux protéines) et pointe (quatre protéines). Six de ces protéines ont confirmé des tendances précédemment rapportées pendant l’essaimage au fil du temps5. Certaines des protéines identifiées ont été précédemment montrées pour être essaimage-spécifiques, alors que d’autres ont été exprimées différentiellement par les neutrophiles non-essaimage activés5. Les protéines qui augmentent en concentration au fil du temps sont probablement associées à la réponse pro-inflammation. Certaines des protéines qui augmentent dans la concentration au fil du temps sont déjà connus pour être impliqués dans la réponse pro-inflammatoire (par exemple, galectin-3 et MMP-9)27,28. La relation entre les autres protéines et l’inflammation est moins bien comprise. Les protéines qui pic ou diminuent pendant l’essaimage peuvent être impliquées dans la régulation de l’inflammation. Cependant, d’autres recherches sont nécessaires pour comprendre le rôle dans l’inflammation de beaucoup de protéines qui sont exprimées différemment pendant l’essaimage. L’analyse des médiateurs libérés avec la migration de neutrophile peut nous aider à mieux comprendre l’image complexe de l’inflammation et comment les neutrophiles ont un impact sur les tissus environnants pendant l’essaimage.
Dans de futures études, notre plate-forme peut être utilisée pour étudier en profondeur la capacité des neutrophiles malsains à générer des essaims stables autour de grappes de bioparticules à motifs. Diverses conditions médicales ont été liées aux neutrophiles, y compris le sepsis3, trauma6, et le cancer1,8,17,18, qui suggère neutrophiles dans ces patients peuvent avoir modifié la fonction. Cette plate-forme peut être utilisée pour examiner les différences entre les médiateurs intercellulaires libérés par des neutrophiles sains et malsains. En outre, cette plate-forme pourrait être modifiée pour modeler des microbes vivants et utilisée pour analyser la réponse des neutrophiles aux microbes vivants in vitro.
En conclusion, nous avons développé une nouvelle plate-forme pour analyser l’essaimage de neutrophile in vitro. La nature hautement contrôlée de notre plate-forme nous permet d’atténuer les problèmes qui se posent lors d’expériences d’essaimage in vivo neutrophile. L’essaimage de neutrophile sur un microréseau de bioparticules est facilement quantifiable par microscopie fluorescente en accéléré. En outre, nous pouvons recueillir les médiateurs libérés par les neutrophiles sans l’interférence d’autres tissus qui sont présents in vivo. Cette plate-forme peut être utilisée dans des recherches futures pour quantifier les différences entre les comportements migratoires des neutrophiles des donneurs sains et malsains.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par le financement du Département William G. Lowrie de génie chimique et biomoléculaire et du Comprehensive Cancer Center de l’Ohio State University. Les données présentées dans ce rapport proviennent d’images traitées à l’aide d’Imaris x64 (ver. 9.3.0 Bitplane) disponibles à l’installation de microscopie et d’imagerie du campus de l’Université d’État de l’Ohio. Cette installation est soutenue en partie par la subvention P30 CA016058, National Cancer Institute, Bethesda, MD.
matériels
| Name | Company | Catalog Number | Comments |
| "The Big Easy" EasySep Magnet | STEMCELL Technologies | 18001 | Magnet to use with neutrophil isolation kit |
| Cell Incubator | Okolab | 777057437 / 77057343 | Okolab cage incubator for temperature and CO2 control |
| EasySep Human Neutrophil Isolation Kit | STEMCELL Technologies | 17957 | Kit for immunomagnetic negative selection of human neutrophils |
| Eclipse Ti2 | Nikon Instruments | MEA54010 / MEF55037 | Inverted research microscope |
| Escherichia coli (K-12 strain) BioParticles Texas Red conjugate | Invitrogen | E2863 | Bioparticle powder, dissolve in water prior to addition to Zetag® array |
| Harris Uni-Core 8-mm biopsy punch | Sigma Aldrich | Z708925 | To cut PDMS stamps |
| HetaSep | STEMCELL Technologies | 7906 | Erythrocyte aggregation agent for separating buffy coat from red blood cells in fresh human blood |
| Hoechst 33342 | Life Technologies | H3570 | Nucleus fluorescent stain |
| Human L1000 Array | Raybiotech Inc. | AAH-BLG-1000-4 | High density array to detect 1000 human proteins |
| Human Serum Albumin (HSA) | Sigma Aldrich | A5843 | Low endotoxin HSA, to prepare 2 % solutions in IMDM for isolated neutrophils |
| Iscove's Modified Dulbeccos' Medium (IMDM) | Thermo Fisher Scientific | 12440053 | To resuspend isolated neutrophils |
| K2-EDTA tubes | Thermo Fisher Scientific | 02-657-32 | Tubes for blood collection |
| Low Reflective Chrome Photomask | Front Range Photomask | N/A | Dimensions 5" x 5" x 0.09" (L x W x D) |
| Microarray Scanner | Perkin Elmer | ASCNGX00 | Fluorescence reader of protein patterned microdomains |
| Microscopy Image Analsysis Software - Imaris | Bitplane | 9.3.0 | Software for automatic cell tracking analysis |
| NiS Elements Advanced Research Software Package | Nikon Instruments | MQS31100 | Software for automatic live cell imaging and swarm size calculation |
| Poly-L-lysine fluorescein isothiocyanate (PLL-FITC) | Sigma Aldrich | P3069-10MG | 30,000 - 70,000 MW PLL labeled with FITC, used to fluorescently label CP solution |
| SecureSeal 8-well Imaging Spacer | Grace Bio-Labs | 654008 | 8-well, 9-mm diameter, adhesive imaging spacer |
| Silicon Wafer | University Wafer | 590 | Silicon 100 mm N/P (100) 0- 100 ohm-cm 500 μm SSP test |
| Spin Coater | Laurell | WS-650MZ-23NPPB | Used to spincoat a 40-µm layer of photoresist onto silicon wafer |
| SU-8 2025 | MicroChem | 2025 | Negative photoresist to make silicon master wafer |
| SU-8 Developer | MicroChem | Y020100 | Photoresist developer. Remove non-crosslinked SU-8 2025 from silicon wafer |
| Sylgard 184 (polydimethylsiloxane, PDMS) | Dow | 1673921 | 2-part silicone elastomer kit for making microstamps and PDMS wells |
| UV Exposure Masking System | Kloé | UV-KUB 2 | Used to crosslink photoresist on silicon wafer through chrome mask with UV light |
| Water | Thermo Fisher Scientific | A1287303 | High quality water to dilute bioparticles |
| Zetag 8185 | BASF | 8185 | Cationic polyelectrolyte (CP), powder, Copolymer of acrylamide and quaternized cationic monomer, forms "inking solution" for microstamping when dissolved in water |
| Zymosan A S. cerevisiae BioParticles Texas Red conjugate | Invitrogen | Z2843 | Bioparticle powder, dissolve in water prior to addition to Zetag array |
Références
- Coffelt, S. B., Wellenstein, M. D., de Visser, K. E. Neutrophils in cancer: neutral no more. Nature Reviews Cancer. 16 (7), 431-446 (2016).
- Jones, C. N., et al. Microfluidic Platform for Measuring Neutrophil Chemotaxis from Unprocessed Whole Blood. Journal of Visualized Experiments. (88), 1-6 (2014).
- Ellett, F., et al. Diagnosis of sepsis from a drop of blood by measurement of spontaneous neutrophil motility in a microfluidic assay. Nature Biomedical Engineering. 2, 207-214 (2018).
- Malawista, S. E., Chevance de Boisfleury, A., Naccache, P. H. Inflammatory Gout: Observations over a Half-Century. The FASEB Journal. 25 (12), 4073-4078 (2011).
- Reátegui, E., et al. Microscale arrays for the profiling of start and stop signals coordinating human-neutrophil swarming. Nature Biomedical Engineering. 1 (7), 1-12 (2017).
- Morgan, A. S. Risk factors for infection in the trauma patient. Journal of the National Medical Association. 84 (12), 1019-1023 (1992).
- Szczerba, B. M., et al. Neutrophils escort circulating tumour cells to enable cell cycle progression. Nature. 566, 553-557 (2019).
- Treffers, L. W., Hiemstra, I. H., Kuijpers, T. W., van den Berg, T. K., Matlung, H. L. Neutrophils in cancer. Immunological Reviews. 273, 312-328 (2016).
- Grayson, P. C., Kaplan, M. J. At the Bench: Neutrophil extracellular traps (NETs) highlight novel aspects of innate immune system involvement in autoimmune diseases. Journal of Leukocyte Biology. 99 (2), 253-264 (2016).
- Lämmermann, T. In the eye of the neutrophil swarm--navigation signals that bring neutrophils together in inflamed and infected tissues. Journal of Leukocyte Biology. 100 (1), 55-63 (2016).
- Kienle, K., Lämmermann, T. Neutrophil swarming: an essential process of the neutrophil tissue response. Immunological Reviews. 273 (1), 76-93 (2016).
- de Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nature Reviews Immunology. 16 (6), 378-391 (2016).
- Lämmermann, T., et al. Neutrophil swarms require LTB4 and integrins at sites of cell death in vivo. Nature. 498 (7454), 371-375 (2013).
- Kreisel, D., et al. In vivo two-photon imaging reveals monocyte-dependent neutrophil extravasation during pulmonary inflammation. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18073-18078 (2010).
- Coombes, J. L., Robey, E. A. Dynamic imaging of host-pathogen interactions in vivo. Nature Reviews Immunology. 10 (5), 353-364 (2010).
- Lämmermann, T. Cell migration: Arraying neutrophils in swarms. Nature Biomedical Engineering. 1 (7), 1-2 (2017).
- Powell, D. R., Huttenlocher, A. Neutrophils in the Tumor Microenvironment. Trends in Immunology. 37 (1), 41-52 (2016).
- Uribe-querol, E., Rosales, C. Neutrophils in Cancer: Two Sides of the Same Coin. Journal of Immunology Research. 2015, (2015).
- Underhill, D. M., Ozinsky, A. Toll-like receptors: Key mediators of microbe detection. Current Opinion in Immunology. 14 (1), 103-110 (2002).
- Netea, M. G., Van Der Meer, J. W., Kullberg, B. J. Recognition of fungal pathogens by toll-like receptors. Immunology of Fungal Infections. , 259-272 (2007).
- Thomas, C. J., Schroder, K. Pattern recognition receptor function in neutrophils. Trends in Immunology. 34 (7), 317-328 (2013).
- Prince, L. R., Whyte, M. K., Sabroe, I., Parker, L. C. The role of TLRs in neutrophil activation. Current Opinion in Pharmacology. 11 (4), 397-403 (2011).
- Tan, S. Y., Weninger, W. Neutrophil migration in inflammation: intercellular signal relay and crosstalk. Current Opinion in Immunology. 44, 34-42 (2017).
- Menezes, G. B., Rezende, R. M., Pereira-Silva, P. E. M., Klein, A., Cara, D. C., Francischi, J. N. Differential involvement of cyclooxygenase isoforms in neutrophil migration in vivo and in vitro. European Journal of Pharmacology. 598 (1-3), 118-122 (2008).
- Afonso, P. V., et al. LTB4 Is a Signal-Relay Molecule during Neutrophil Chemotaxis. Developmental Cell. 22 (5), 1079-1091 (2012).
- Gounni, A. S., et al. In Vivo Regulates Neutrophil Migration In Vitro and Chemorepellent Semaphorin 3E Negatively Chemorepellent Semaphorin 3E Negatively Regulates Neutrophil Migration In Vitro and In Vivo. The Journal of Immunology. 198, 1023-1033 (2017).
- Dumic, J., Dabelic, S., Flögel, M. Galectin-3: An open-ended story. Biochimica et Biophysica Acta - General Subjects. 1760 (4), 616-635 (2006).
- Padmanabhan Iyer, R., et al. Matrix metalloproteinase-9-dependent mechanisms of reduced contractility and increased stiffness in the aging heart. Proteomics - Clinical Applications. 10 (1), 92-107 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon