Method Article
ヒト好中球群れの化学化学および分子解析のための生体微粒子マイクロアレイ
要約
このプロトコルは、空間的に制御された好中球群を提供するバイオ粒子マイクロアレイを生成する。これは、好中球が移行中に放出するメディエーターに容易にアクセスでき、定量的な画像分析を可能にします。
要約
好中球の群れは、好中球が感染部位を封鎖し、組織の再編成を促進する協調プロセスである。群れの特徴を示す動物モデルにおいて、細胞移動の特徴的なパターンを示す動物モデルにおいて、古典的に生体内で研究されている。しかし、in vivoモデルには、アクセスと分析が困難な細胞間メディエーター、およびヒト好中球を直接分析することができないなど、いくつかの制限があります。これらの制限のため、ヒト好中球で群りを研究し、群れの間に生成される分子信号に容易にアクセスできるin vitroプラットフォームが必要です。ここでは、多段階マイクロスタンププロセスを使用して、生体内感染を模倣して群れを刺激するバイオ粒子マイクロアレイを生成します。バイオ粒子マイクロアレイは、制御された安定した方法で群れを作る好中球を誘導する。マイクロアレイでは、好中球は速度が上昇し、バイオ粒子クラスターの周りに安定した群れを形成します。さらに、好中球によって生成された上清が分析され、16個のタンパク質が群れの過程で異なって発現されていたことが発見された。このインビトロ・ストウォーミング・プラットフォームは、再現性のある空間的に制御された方法で好中球の移動およびタンパク質放出を直接分析します。
概要
好中球は、血流1で最も豊富な白血球であり、潜在的な診断および治療標的として注目を集めている2,3は、色あさ4、敗血症、外傷5、6、癌1、7、8、および様々な自己免疫疾患5,9を含む様々な病状に関与する可能性があるためである。好中球の群れは、多段階で厳しく調節されたプロセスであり、複雑さを伴う複雑性を有し、研究5、10、11の特に興味深い焦点となっている。群れの間、好中球は周囲の健康な組織5、10、11から炎症部位を単離する。好中球群れの適切な調節は創傷治癒を促進するために不可欠であり、最終的には炎症解決5、12。好中球の群れは、主にげっ歯類12、13、14、15およびゼブラフィッシュ10、11、12、15モデルで生体内で研究されている。しかし、これらのin vivo動物モデルの性質は、制限5を生じさせる。例えば、群れの間に好中球によって放出されるメディエーターは、分析5のために容易にアクセスできない。さらに、インビボで与えられたメディエーターには多くの潜在的な情報源があるので、インビボ実験は、所定のプロセス13におけるそのメディエーターの役割を調査するために細胞産生および/または相互作用を阻害する遺伝的欠乏を導入しなければならない。インビトロ実験は、追加の細胞のコンテキストなしで好中球観察を可能にすることによって、この合併症を回避する。さらに、ヒト好中球協調移動を記述する研究は限られた16である。インビトロの群れプラットフォームでは、ヒト好中球を直接分析することができます。インビトロの群れのプラットフォームは、in vivo研究の限界によって残されたギャップを埋める機会を提供することによって、インビボ研究から得られた知識を拡大する可能性がある。
インビボ好中球群れを模倣するインビトロプラットフォームの必要性に対処するために、好中球の群れを空間的に制御した方法でバイオ粒子マイクロアレイをパターン化できるマイクロスタンププラットフォームを開発しました。2段階のプロセスで、ガラススライド上にバイオ粒子マイクロアレイを生成します。まず、マイクロスタンプを使用して、カチオン性多電解質(CP)スポットのマイクロアレイを生成します。次に、静電気相互作用を介してCPスポットに付着するバイオ粒子の溶液を添加します。まずCP層をパターン化することにより、負に帯電したバイオ粒子を選択的にパターン化して、所望の好中球群れパターンを生成することができる。正に荷電した層は、CPを持たないガラススライド上の領域から生体粒子を取り除く精力的な洗浄ステップを経て負に荷電した生体粒子を保持する。これは、ガラススライドにミクロンサイズのバイオ粒子を固定化する非常に高い表面電荷を有し、したがって、ガラススライド上のパターン化された位置から粒子を除去することから好中球を阻害する。これは、マイクロアレイに配置されたバイオ粒子クラスターをもたらす。マイクロアレイに好中球を加えたところ、バイオ粒子クラスターの周りに安定した群れを形成しました。好中球の移動を追跡すると、群れの好中球が積極的にバイオ粒子クラスターに向かって移動することがわかりました。さらに、このプラットフォームを使用して、群れの間に好中球が放出する特定のメディエーターを分析しました。群れの間に差を出して表現される16のメディエーターを見つけました。彼らの濃度は、時間の経過とともに3つの一般的な傾向に従います: 増加, 減少, またはスパイク.私たちのインビトロ好中球群れプラットフォームは、空間的に制御されたヒト好中球群れの分析、ならびに好中球群れによって放出されるメディエーターの収集と分析を容易にする。以前の出版物では、特定の病状(外傷、自己免疫疾患、敗血症)を有する患者が、健康なドナーからの患者とは異なる機能を有する好中球を有することを実証した5。今後の研究では、当社のプラットフォームを使用して、さまざまな患者集団の中で好中球機能を分析することができます。このプラットフォームは、好中球群れの複雑な配位を定量的に分析することができる。特定の患者集団の好中球機能または目的の病原体に対する好中球応答に関する洞察を提供するために、追加の研究を行うことができる。
プロトコル
著者らは、親切に献血した健康なボランティアを認めている。血液検体は、オハイオ州立大学の生物医学委員会によって審査#2018H0268機関審査委員会(IRB)プロトコルに従って、インフォームド・ボランティアの同意の後に得られました。
1. バイオ粒子マイクロアレイの微細加工
- 標準的なフォトリソグラフィ手順を使用して、マスターシリコンウエハを生成します。
- コンピュータ支援設計(CAD)ソフトウェアを使用して目的の設計の証明を生成し、フォトマスクの製造者に送って、クロームフォトマスクを作ります。ここで使用される設計は、150 μmの中央から中央の間隔で円で満たされた直径30μmの4ミリメートルの長方形配列です。この設計は、さまざまなアプリケーションに合わせて変更できます。
- シリコンウェーハ上に40μmの厚い層の陰性フォトレジストをスピンコートします。ウェハを65°Cで5分間、95°Cで10分間焼きます。
- 150~160 mJ/cm2のクロムフォトマスクを通してウエハをUV光に露出させる(図1A)。
- 65°Cで5分間、95°Cで10分間焼き、フォトレジスト現像で10分間、イソプロピルアルコールでリンスします。この段階では、パターンがウエハ上に表示されているはずです(図1B)。
- ポリジメチルシロキサンプレポリマーと硬化剤の10:1の比率(すなわち、プレポリマー20gおよび硬化剤2g)を十分に混合し、未硬化ポリジメチルシロキサン(PDMS)混合物をペトリ皿のマスターウエハ上に注ぐ(図1C)。
- 真空は、マスターウェーハ上に気泡が存在しなくなるまで、未硬化のPDMS混合物を処理します。65°Cで一晩硬化する。
- メスを使用してウエハーのパターンセクションの外側をカットし、硬化したPDMSスラブをゆっくりと取り除きます。パターン化された側面を上に向けて、PDMSスラブを清潔なまな板に置きます(図1D)。
- PDMSスラブから8mmの生検パンチ(図1E)で個々のスタンプをパンチアウトします。1枚のガラススライドには、8枚の切手が必要です。
- 各スタンプを粘着テープの上に下に置き、破片を取り除きます。
- あらかじめ、水にCPの1.6mg/mL溶液を調製します。
- CP粉末の適正量を水に加えます(例:0.8g~500mL)。
- 室温で一晩かき混ぜ、またはすべての固体が水に溶解するまで混ぜる。CP溶液は、室温で6ヶ月間保存することができる。
- 必要に応じて、蛍光灯を発するポリL-リジンを蛍光(PLL-FITC)で標識した蛍光を発します。
- CP溶液のアリコート約10mL。少量のPLL-FITC(0.05 mg)を分量に加えます。量は、所望に応じて蛍光の明るさを調整するように変更することができる。
- 20 sのFITCで標識されたCP溶液をボルテックス、または溶液が均一な、淡い黄色になるまで。光から保護し、最大1ヶ月間4°Cで保管してください。
- スタンプを上にして、CPの1.6mg/mL溶液の100 μLで各スタンプをプライムし、CP溶液とスタンプの間に気泡が形成されないようにします(図1F)。
- CP溶液の層にスタンプを反転します(図1G)。
- CP溶液から1時間後にスタンプを取り外します。
- 濡れたスタンプを、きれいなガラスのスライド6~8xに下に下にして、余分な液体を取り除きます。
- 1~2分の切手を真空処理します。
- きれいなガラスのスライドの上に8つの井戸、9 mmの直径のイメージ投射スペーサーを押印の配置のためのガイドとして付着する。このスペーサーを使用すると、各ガラススライドは8つのマイクロアレイを持つことができます。
- イメージスペーサーの各ウェルの中央にあるスライドガラスの上にスタンプを下に置きます(合計で8枚のスタンプを使用します)。
- 各スタンプの上に5.6 ±0.1 gのバランスの取れた重さを置き、スタンプを10分にします(図1H)。バランスの取れた重量は、スタンプがガラススライドに均等に押し込まれ、CPをガラススライドに均等に移すために必要です。
- ガラススライドから重みとスタンプを取り外します(図1I)。CP層を、ステップ1.17で説明したように、バイオ粒子を加える前に24時間室温で乾燥させます。CPにFITCタグが付けられている場合、この時点で488nmの蛍光顕微鏡でスタンプの有効性を確認してから、ステップ1.17(図1M)に進むことができます。
- 空のPDMSスラブをイメージングスペーサーのサイズにカットし、8mmの生検パンチを使用して、イメージングスペーサーの井戸に合わせた井戸をPDMSに作成します。PDMSスラブをイメージングスペーサ(図1J)でガラススライドに付着させます。
- バイオ粒子(例えば、大腸菌またはzymosan)の溶液を解凍し、注射用水(WFI)で500μg/mLに希釈します。
注:バイオ粒子はオプソン化する必要はありません。好中球表面受容体は、これらの生体粒子19、20、21上の分子を直接認識する。 - 各PDMSウェルに100 μLのバイオ粒子溶液をグラススライドで加えます(図1K)。
- グラススライドを30分間揺らす。
- 井戸を水で十分に洗い流します。バイオ粒子マイクロアレイは、4°Cのダストフリー環境に最大3ヶ月間保存できます。この時点で、パターンは、ステップ2.1(図1N)に進む前に、594 nmの蛍光顕微鏡でチェックする必要があります。
2. サンプルの準備
- 目的のドナーからK2-EDTAチューブに少なくとも2 mLの新鮮な血液を集める。好中球の期待される収量は、1~2 x 106細胞/1 mL全血です。イメージングアッセイには約1.5 x 105の好中球が必要であり、上清の分析には1 x 106好中球が必要です。血液を4時間以内に使用します。
- 赤血球(RBCs)を分離し、全血に対して1:5の比率で赤血球凝集剤を添加する。RBCの層から分離するために、半透明の層(バフィーコート)が45分待ちます。
- バフィーコートを取り出し、1 mLバフィーコート(9 mL PBS)を使用してリン酸緩衝生理食塩水(PBS)で洗います。
- 遠心分離機は190×gおよび20°Cで5分間。
- 上清を吸引し、ペレットを5 x 107細胞/mLで再懸濁します。
- 負の免疫磁気選択キットを使用して好中球を分離します。
- 50 μLの抗体カクテル/1 mL細胞懸濁液を添加します。10分待ちます。
- 100 μLの磁気ビーズ/1 mLセルサスペンションを追加します。10分待ちます。
- 丸底チューブにセルサスペンションを追加し、円筒形の磁石に入れ.10分待ちます。
- 遠心分離管に上清を注ぎます。最大 10 mL の PBS を追加します。遠心分離機は190×gおよび20°Cで5分間。
- 吸海上清。0.4%のヒト血清アルブミンを用いてIMDM中の白いペレットを再懸濁する。
- 37 °Cで10分間、20 μg/mLのホーチスト33342を有する染色核。
- 0.4%のヒト血清アルブミンをリンスにIMDMの5 mLを加える。遠心分離機は190×gおよび20°Cで5分間。
- 0.4%のヒト血清アルブミンを用いてIMDM中の7.5 x 10 5細胞/mLで細胞を再懸濁する。
- バイオ粒子マイクロアレイを含むPDMSウェルに細胞懸濁液を100μL加える。セル懸濁液がPDMSの上部に凸で覆われ、泡が含まれていないことを確認します。
- 直径12mmのカバースリップでシール。PDMSの開口部を直径12mmのカバースリップでうまく覆います。余分な細胞懸濁液がウェルの端に逃げないように、ピンセットでカバースリップにそっと押し下げます。組織を使用して、余分な細胞懸濁液を除去する。
3. アッセイと画像解析の実行
- 37°C、5%CO2、および90%相対湿度に設定されたケージインキュベーターを備えた顕微鏡を備えた顕微鏡を用いて、生きた細胞イメージングステーションに細胞を搭載した微粒子アレイをロードします。
- タイムラプス蛍光および明視野顕微鏡を使用して、405 nm、594 nm、およびブライトフィールドで10sごとに10倍の倍率で画像を記録します。典型的な実験では、画像は最大2時間収集されます。
- 自動化された細胞追跡ソフトウェアを使用して、バイオ粒子クラスターに向かう個々の好中球の移動を追跡します。
- スポット検出セルトラッキングソフトウェアの自己回帰モードを使用します。スポット半径を 5 μm (好中球核のおおよそのサイズ) に設定します。最小トラック長を 120 s に設定し、最大ギャップサイズを 1 フレームに設定します。
- 細胞追跡ソフトウェアによって生成されたデータから、好中球の位置および速度を含むファイルを抽出する。これらのファイルは、好中球移動トラック(図2C)と速度対時間のヒートマップ(図2D)を生成するために、グラフ化ソフトウェアと共に使用することができます。
- 405 nm 蛍光画像を使用して、選択した画像解析ソフトウェアで時間の経過とともに群れのサイズを追跡します。
- 好中球が群がる各バイオ粒子クラスターの周辺の関心領域(ROI)を定義します。各バイオ粒子クラスターを分析するために同じサイズのROIを維持します。
- 各ROI内の405nm画像の平均蛍光強度を経時に分析します。
- 0 μm2から最大群サイズまでのさまざまな群れサイズで手動で測定を行い、平均蛍光強度の較正曲線を生成して群れサイズを測定します。このキャリブレーションを使用して、時間の経過に合った群れのサイズを計算します。
4. 上清の収集とタンパク質の検出
- 37°Cでのバイオ粒子マイクロアレイを含むウェル内の好中球をインキュベートし、3時間の5%CO2で。希望の時間にサンプルを取ります。通常、サンプルは 0、0.5、1、および 3 h で取得されます。タンパク質アレイアッセイの検出限界を克服するために、単一ウェルの上清の全容積(200μL)を各時点に用いた。各時点を三重で分析した。
- 明視野顕微鏡を使用して、マイクロアレイ上に群れが形成されていることを確認します。
- 上清を200μLピペットで吸引し、0.45μm遠心フィルターチューブに負荷を入れられます。
- 上清を190×g、20°Cで5分間遠心分離し、濾過容を収集します。
- サンプルは、処理時間まで-80°Cで保存します。
- サンプルを処理するために、さまざまなヒトタンパク質を検出するマイクロアレイキットを使用します。
- キットに付属の透析チューブに各サンプルの200 μLを加えます。
- 透析管を少なくとも500 mLのPBS(pH= 8.0)を含むビーカーに入れる。4°Cで少なくとも3時間、かき混ぜ皿の上で軽くかき混ぜます。ビーカーの PBS を変更し、この手順を繰り返します。
- 各サンプルをきれいな遠心分離管に移し、9,000 x gで5分間遠心分離し、沈殿物を除去します。各上清を清潔なチューブに移します。
- ビオチン化したサンプルは、透析サンプル中の全タンパク質1mg当てキットから36μLの1x標識試薬を180μLの透析試料に添加する。20°Cで30分間インキュベートし、5分ごとに穏やかに混ぜます。
- キットに付属の3μLの停止溶液を各サンプルチューブに加えます。各サンプルを新鮮な透析チューブに移し、ステップ4.6.2~4.6.3を繰り返します。この段階では、サンプルは、続行する準備ができるまで-20 °Cまたは -80 °Cで保存できます。
- キットに付属のガラススライドは-20°Cで保存されます。室温に戻すことを許可します。組み立てられたガラススライドを、室温で1~2時間の層流フードに入れます。
- 組み立てられたガラススライドの各ウェルに、キット付属のブロッキングバッファを400μLずつ加えます。室温で30分間インキュベートします。
- 調製したサンプルを9,000 x gで5分間遠心分離し、沈殿物または微粒子を除去する。ブロッキングバッファーで5倍希釈します。
- 各ウェルからブロッキングバッファを削除します。希釈したサンプルを400μLを適切なウェルに加えます。ロッキングしながら室温で2時間インキュベートします。
- 各井戸からサンプルをデカントします。1xウォッシュバッファの800μLで3xを洗浄し、ロッキングしながら、それぞれ5分間室温でキットを用意しました。
- きれいな容器に、組み立てられたガラスのスライドを1xウォッシュバッファI.5分間室温で洗い流し、揺らしながら2xを洗います。
- 各サブアレイに、1x Cy3-conjugated streptavidinの400 μLを追加します。プラスチック製の接着剤ストリップで覆います。プロトコルの残りの部分のために光から保護します。
- ロッキングしながら室温で2時間インキュベートします。
- 溶液をデカントし、サンプルチャンバーからガラススライドを分解します。
- キットに付属の30 mL遠心チューブに、ガラススライドと十分な1x洗浄バッファIを慎重に追加して、ガラススライドをカバーします。ロッキングしながら室温でそれぞれ10分間3倍洗います。
- 30 mL遠心管内で、1x洗浄バッファー II で 2x を洗浄し、ロッキングしながら室温でそれぞれ 5 分間洗浄します。
- 30 mLのddH2Oで5分間洗い流し、遠心管からガラススライドを取り出し、層流フードで20分間乾燥させます。準備したガラススライドは、スキャンの準備ができるまで-20°Cで保存することができる。
- 555 nmの蛍光発光でマイクロアレイスキャナーでガラススライドをスキャンします。
結果
好中球をバイオ粒子マイクロアレイに添加すると、バイオ粒子クラスターに接触する好中球が活性化し、群れ応答を開始します。バイオ粒子マイクロアレイは、タイムラプス蛍光顕微鏡法を用いて、バイオ粒子クラスターに向かう好中球の移動を追跡することを検証した(Video S1)。個々の好中球核の移動は、バイオ粒子クラスターに向かって移動する過程で追跡される。好中球がバイオ粒子クラスターに到達すると、その核はクラスター内の他の核と重なります。したがって、この方法を用いてクラスター内の好中球を正確に追跡することはできない。ザイモサンと大腸菌のバイオ粒子クラスターは、いずれも好中球群の生成をもたらす。我々の結果として、図2Bは大腸菌粒子によって生成された好中球群からのデータを使用する。図3および図2の他のパネルは、ジモサン粒子によって生成された好中球群を用いる。得られた結果は、好中球がクラスターに接触したときにバイオ粒子クラスターが好中球活性化を刺激し、最終的に30〜60分後に各クラスターの周りに安定した好中球群の形成につながったことを実証した(図2A、上)。対照的に、好中球は、バイオ粒子クラスターがない場合の集合的な移動を示さなかった(図2A、底部、およびビデオS2)。405nmで染色された好中球核の蛍光強度を用いて、平均好中球群の大きさは直径30μm前後の大腸菌バイオ粒子群が1,490±680μm2であることが判明した(平均±SD、図2B、上)。生物粒子クラスターが存在しない対象領域の蛍光強度は、時間の経過とともにほぼ一定であり、この設定では集合的な移動がないことが確認された(図2B、底)。好中球遊走の軌跡は、好中球が存在した際にバイオ粒子クラスター上に収束したことを示す(図2C、上)。逆に、制御系(図2C、底部)では収束は認められない。群れと非活性化された好中球の速度(移動距離/時間)を測定し、図2Dに示すように、速度分布の統計的有意差(ANOVA,p <0.0001)が見つかりました。 群れの好中球の平均速度は20.6±13.0μm/min(平均±SD)、制御好中球の平均速度は2.0±2.2μm/minであった。
さらに、群れの間に放出される好中球が放出する16個のタンパク質の濃度を分析した(図3)。群れプロセスの異なる時点における各タンパク質の正規化濃度(t = 0,0.5,1,3h)を計算し、ここで正規化濃度は(C -C分)/(Cmax - Cmin)である。10個のタンパク質が群れの間に濃度が上昇した。これらのタンパク質は、アジプシン、ガレクチン-3、GROa、IL-6R、MIP-1a、MMP-8、MMP-9、ニドゲン-1、TIMP-1、およびTLR2であった。2つのタンパク質(ペントラキシン3およびRANK)は群れの間、濃度が低下した。残りの4つのタンパク質(Clusterin、PF4、RANTES、およびトラッピン-2)は、群れの最初の1時間の間に増加したが、その後減少した。言い換えれば、それらのタンパク質の濃度は群れの間に「スパイク」される。同定された16個のタンパク質のうち、12個のタンパク質が以前の公報5で同定された。アジプシン、ガレクチン-3、ニドゲン-1、ペントラキシン3、TIMP-1、TLR2は群れ特異的であることが示されたが、クラスタリング、IL-6R、MMP-8、およびMMP-9、RANK、およびトラップピン-2は5ではなかった。

図1:バイオ粒子マイクロアレイの製造陰性フォトレジストでコーティングされたシリコンウェーハは、クロムフォトマスク(A)を通してUV光に曝露される。シリコンウエハが焼成して開発された後、シリコンウエハの表面にフォトレジストパターンが残ります。これがマスターウェハー(B)です。ペトリ皿では、PDMS混合物がマスターウェーハの上部に加えられます。PDMSを65°Cで一晩硬化し、PDMSモールド(C)を形成する。PDMS金型はマスターウエハから切断され、生検パンチを使用して個々のPDMSスタンプ(D)をパンチアウトします。PDMS スタンプ (E) は CP 溶液 (F) でコーティングされています。スタンプは、CP溶液(G)の薄い層に反転されます。CP溶液を1時間インキュベートした後、スタンプをグラススライドに吸い込んで余分なCPを除去します。スタンプが削除されると、CPパターンが残ります(I)。プレカットウェルを備えたPDMSスラブは、ガラススライド(J)に接着されます。次いで、CPパターン(K)の上にバイオ粒子溶液を添加する。負電荷の生物粒子は、静電相互作用を介して正電荷を帯びた高電解質に結合する。過剰なバイオ粒子溶液を洗い流し、CPパターンの上にパターン化されたバイオ粒子(L)を残す。(M) FITCでラベル付けされたCP層の蛍光画像(スケールバー= 50 μm、大きな画像;スケールバー = 25 μm, インセット).(N) テキサスレッドと結合したパターン化されたジモサンバイオ粒子の蛍光画像(スケールバー= 100 μm,大きな画像;スケールバー = 50 μm,インセット)。この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:バイオ粒子クラスター周辺の好中球群増殖バイオ粒子クラスターの存在下で、好中球は、バイオ粒子クラスター(下、制御好中球)に向かって一括移動を受ける。生物粒子が存在しない場合、好中球は集合的な移動を行わない。(A) ジモサンのバイオ粒子クラスターの存在下で、好中球は30分で群れを形成する。バイオ粒子クラスターがなければ、群れが形成されません(スケールバー = 50 μm)。(B)好中球群は、直径30μm前後の平均サイズ1,490±680μm2(平均±SD)に成長する。 コントロール好中球は群れ増殖に対応しない一定の密度を示す(ANOVA、p < 0.0001、n = 32好中球群、N = 1ドナー、誤差棒=標準偏差)。(C)群れの好中球のトラックは、zymosanのバイオ粒子クラスターに収束し、制御好中球は収束点を示さない(スケールバー= 50 μm)。(D) ジモサンターゲットに向かって群がる好中球の平均速度は20.6±13.0 μm/min(平均±SD)、制御好中球の平均速度は2.0±2.2μm/minです。ヒートマップ上の各カウントは、与えられた時点で与えられた瞬間速度を持つ好中球を表します。これらのヒートマップは、1つの実験(n = 1群れ;1群れ)を代表する。N = 1 ドナー;ANOVA, 6,114 = 群れの好中球, 32,116 = コントロール好中球, p < 0.0001).この図の大きなバージョンを表示するには、ここをクリックしてください。
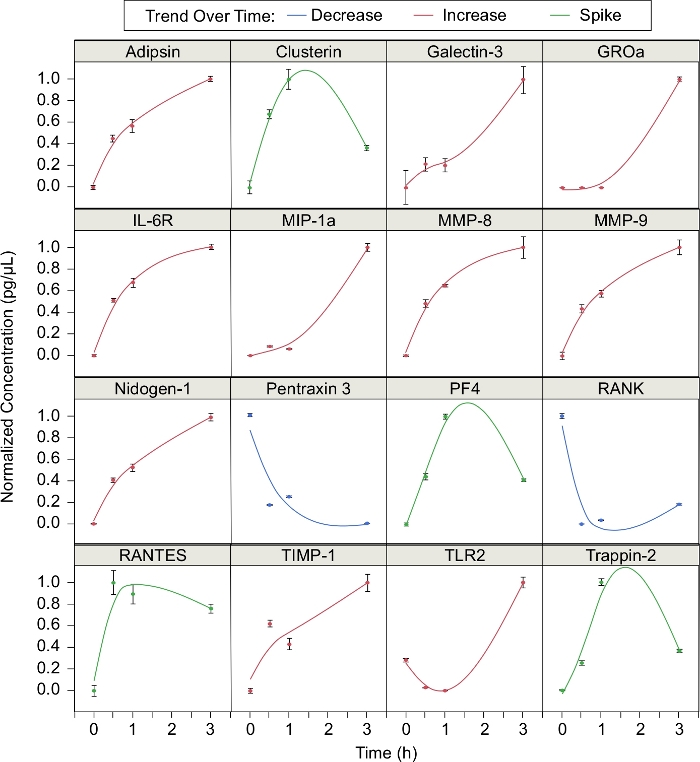
図3:好中球の群れによって放出されるフリーメディエーター。好中球の群れの影響は、時間の経過とともに様々なタンパク質の産生に影響を与えます。タンパク質濃度は0、0.5、1、および3hで測定した。データ点はスムージング スプライン(λ = 0.05)を取り付けました。これらのタンパク質は、時間の経過とともに減少、時間の経過とともに増加、または1時間の周りにスパイクし、減少する(誤差範囲= 標準偏差;n = 3反復;3反復;3;3;3の3つの特徴の傾向に従う傾向がある。N =1ドナー)。この図の大きなバージョンを表示するには、ここをクリックしてください。
ビデオS1:ジモサンバイオ粒子クラスターに向かって群がる好中球。好中球核は青色で示されている。Zymosanターゲットは赤い円でマークされています(スケールバー = 50 μm、元の取得時間 = 60 分)。このビデオをダウンロードするには、ここをクリックしてください。
ビデオS2:非活性化好中球ランダム移行。好中球核は青色で示されています(スケールバー = 50 μm、元の取得時間 = 60 分)。このビデオをダウンロードするには、ここをクリックしてください。
ディスカッション
インビトロ好中球群を刺激するバイオ粒子の均一な配列を生成するマイクロスタンププラットフォームを開発しました。私たちのプラットフォームのインビトロの性質は、私たちはin vivo群れの実験、すなわち群れの好中球5によって放出されるメディエーターを分析する能力が低い、で生じる合併症を回避することを可能にします。さらに、in vivoモデルは、通常、げっ歯類11、12、13、15、22、23、またはゼブラフィッシュ11、12、15、23で行われる。私たちのプラットフォームは、ヒト好中球を使用しており、マウスとヒト好中球の類似性は5,11で観察されていますが、ヒトの疾患の文脈で結果をより直接的に解釈することができます。さらに、ヒト好中球の集合移動の解析と、群れの好中球によって放出されるメディエーターの収集と分析を容易にする高いレベルの再現性を提供することにより、当社のプラットフォームとin vivoモデルを区別する空間制御された群れ環境を維持します。
マイクロスタンピングプロトコルの開発中に、慎重なトラブルシューティングが必要ないくつかの課題が発生しました。まず、マイクロスタンプに使用されるCPは親水性が高く、PDMSスタンプは疎水性です。CPはPDMSに対して高い親和性を持たないため、気泡の形成を避け、濡れを促進するように慎重に設計されました。まず、上向きの間にCPをプライミングすることで(ステップ1.8)、CPとスタンプの間の泡の形成を最小限に抑えます。スタンプは、CPの層上に反転され、1時間インキュベートされます。この長い潜伏時間は、スタンプのすべてのセクションが濡れていることを保証します。第二に、きれいなガラススライド(ステップ1.11)にスタンプする前に余分なCPを除去するプロセスは矛盾する可能性があります。ステップ1.11を実行する間、スタンプは慎重に調べなければなりません。パターンが表示され始めると、スタンプはステップ 1.12 に向けて準備が整います。さらに、切手を乾燥させるために必要な真空時間(ステップ1.12)は変化する可能性があります。これは主に天候に依存します。暖かく湿度の高い日には、真空時間の2分が必要です。涼しく乾燥した日には、真空時間の1分で十分です。
我々は、健康なドナーからの好中球がバイオ粒子クラスターの周りに安定した群れを形成することを示した。タイムラプス蛍光顕微鏡法により、群れの大きさを定量化し、好中球の移動を追跡することができ、好中球走動を定量的に5を分析することができます。例えば、このプラットフォームは、走力学的指数(CI、好中球速度ベクトルと好中球と最も近いバイオ粒子クラスターの間の位置ベクトル間の角度の余弦)、速度(好中球が時間で割った距離)、ラジアル速度(CIによって乗算された速度)、および総走行距離(CIによって乗算された速度)を計算するために使用できることを以前に示しました。ほとんどのインビトロ研究24、25、26とは異なり、私たちのプラットフォームは人工的な走性勾配を持たないので、好中球細胞間通信は好中球移行の唯一の原動力です。さらに、群れの好中球によって発生する上清は容易にアクセス可能である。我々は、生体内に存在する他の細胞タイプからの干渉なしに、好中球によって放出される細胞間メディエーターの上清を収集し、分析することができる。16個のタンパク質が群れの間に発現すると、増加(10タンパク質)、減少(2つのタンパク質)、スパイク(4つのタンパク質)の3つの傾向の1つとして説明できる。これらのタンパク質のうち6個は、時間の経過に従って群れの間に以前に報告された傾向を確認した5.同定されたタンパク質の一部は、以前に群れ特異的であることが示されたが、他のものは活性化された非群血好中球5によって異状に発現した。時間の経過とともに濃度が上昇するタンパク質は、炎症反応に関連している可能性が高い。時間の経過と同時に濃度が上昇するタンパク質の一部は、炎症促進反応(例えば、ガレクチン-3およびMMP-9)27、28に関与することが既に知られている。他のタンパク質と炎症との関係はあまりよく理解されていません。群れの間にスパイクまたは減少するタンパク質は、炎症の調節に関与する可能性があります。しかし、群れの間に異なって発現される多くのタンパク質の炎症における役割を理解するには、さらなる研究が必要である。放出されたメディエーターと好中球の移動を分析することは、炎症の複雑な画像と、好中球が群れの間に周囲の組織にどのような影響を与えるかについてをよりよく理解するのに役立ちます。
今後の研究では、私たちのプラットフォームは、パターン化されたバイオ粒子クラスターの周りに安定した群れを生成する不健康な好中球の能力を徹底的に研究するために使用することができます。敗血症3、外傷6、癌1、8、17、18を含む好中球に関連する様々な病状が、これらの患者の好中球が機能を変化させてきたことを示唆している。このプラットフォームは、健康な好中球と不健康な好中球によって放出される細胞間メディエーター間の違いを調べるために使用することができる。さらに、このプラットフォームは、生きている微生物をパターン化するように改変し、インビトロで生きている微生物に対する好中球応答を分析するために使用することができる。
結論として、我々は、インビトロでの好中球群れの分析のための新しいプラットフォームを開発した。当社のプラットフォームの高度に制御された性質により、インビボ好中球群れの実験中に発生する問題を軽減することができます。バイオ粒子マイクロアレイ上の好中球群れは、タイムラプス蛍光顕微鏡を介して容易に定量可能である。さらに、我々は、生体内に存在する他の組織の干渉なしに、好中球によって放出されるメディエーターを収集することができる。このプラットフォームは、健康なドナーと不健康なドナーからの好中球の移動行動の違いを定量化するために、将来の研究で使用することができます。
開示事項
著者らは利益相反を宣言していない。
謝辞
この研究は、ウィリアム・G・ロウリー化学・生体分子工学科とオハイオ州立大学総合がんセンターからの資金提供によって支えられた。このレポートで示されたデータは、オハイオ州立大学キャンパス顕微鏡イメージング施設で利用可能なイマリスx64(9.3.0ビットプレーン)を使用して処理された画像から来ました。この施設は、助成金P30 CA016058、国立がん研究所、ベセスダ、MDによって部分的にサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| "The Big Easy" EasySep Magnet | STEMCELL Technologies | 18001 | Magnet to use with neutrophil isolation kit |
| Cell Incubator | Okolab | 777057437 / 77057343 | Okolab cage incubator for temperature and CO2 control |
| EasySep Human Neutrophil Isolation Kit | STEMCELL Technologies | 17957 | Kit for immunomagnetic negative selection of human neutrophils |
| Eclipse Ti2 | Nikon Instruments | MEA54010 / MEF55037 | Inverted research microscope |
| Escherichia coli (K-12 strain) BioParticles Texas Red conjugate | Invitrogen | E2863 | Bioparticle powder, dissolve in water prior to addition to Zetag® array |
| Harris Uni-Core 8-mm biopsy punch | Sigma Aldrich | Z708925 | To cut PDMS stamps |
| HetaSep | STEMCELL Technologies | 7906 | Erythrocyte aggregation agent for separating buffy coat from red blood cells in fresh human blood |
| Hoechst 33342 | Life Technologies | H3570 | Nucleus fluorescent stain |
| Human L1000 Array | Raybiotech Inc. | AAH-BLG-1000-4 | High density array to detect 1000 human proteins |
| Human Serum Albumin (HSA) | Sigma Aldrich | A5843 | Low endotoxin HSA, to prepare 2 % solutions in IMDM for isolated neutrophils |
| Iscove's Modified Dulbeccos' Medium (IMDM) | Thermo Fisher Scientific | 12440053 | To resuspend isolated neutrophils |
| K2-EDTA tubes | Thermo Fisher Scientific | 02-657-32 | Tubes for blood collection |
| Low Reflective Chrome Photomask | Front Range Photomask | N/A | Dimensions 5" x 5" x 0.09" (L x W x D) |
| Microarray Scanner | Perkin Elmer | ASCNGX00 | Fluorescence reader of protein patterned microdomains |
| Microscopy Image Analsysis Software - Imaris | Bitplane | 9.3.0 | Software for automatic cell tracking analysis |
| NiS Elements Advanced Research Software Package | Nikon Instruments | MQS31100 | Software for automatic live cell imaging and swarm size calculation |
| Poly-L-lysine fluorescein isothiocyanate (PLL-FITC) | Sigma Aldrich | P3069-10MG | 30,000 - 70,000 MW PLL labeled with FITC, used to fluorescently label CP solution |
| SecureSeal 8-well Imaging Spacer | Grace Bio-Labs | 654008 | 8-well, 9-mm diameter, adhesive imaging spacer |
| Silicon Wafer | University Wafer | 590 | Silicon 100 mm N/P (100) 0- 100 ohm-cm 500 μm SSP test |
| Spin Coater | Laurell | WS-650MZ-23NPPB | Used to spincoat a 40-µm layer of photoresist onto silicon wafer |
| SU-8 2025 | MicroChem | 2025 | Negative photoresist to make silicon master wafer |
| SU-8 Developer | MicroChem | Y020100 | Photoresist developer. Remove non-crosslinked SU-8 2025 from silicon wafer |
| Sylgard 184 (polydimethylsiloxane, PDMS) | Dow | 1673921 | 2-part silicone elastomer kit for making microstamps and PDMS wells |
| UV Exposure Masking System | Kloé | UV-KUB 2 | Used to crosslink photoresist on silicon wafer through chrome mask with UV light |
| Water | Thermo Fisher Scientific | A1287303 | High quality water to dilute bioparticles |
| Zetag 8185 | BASF | 8185 | Cationic polyelectrolyte (CP), powder, Copolymer of acrylamide and quaternized cationic monomer, forms "inking solution" for microstamping when dissolved in water |
| Zymosan A S. cerevisiae BioParticles Texas Red conjugate | Invitrogen | Z2843 | Bioparticle powder, dissolve in water prior to addition to Zetag array |
参考文献
- Coffelt, S. B., Wellenstein, M. D., de Visser, K. E. Neutrophils in cancer: neutral no more. Nature Reviews Cancer. 16 (7), 431-446 (2016).
- Jones, C. N., et al. Microfluidic Platform for Measuring Neutrophil Chemotaxis from Unprocessed Whole Blood. Journal of Visualized Experiments. (88), 1-6 (2014).
- Ellett, F., et al. Diagnosis of sepsis from a drop of blood by measurement of spontaneous neutrophil motility in a microfluidic assay. Nature Biomedical Engineering. 2, 207-214 (2018).
- Malawista, S. E., Chevance de Boisfleury, A., Naccache, P. H. Inflammatory Gout: Observations over a Half-Century. The FASEB Journal. 25 (12), 4073-4078 (2011).
- Reátegui, E., et al. Microscale arrays for the profiling of start and stop signals coordinating human-neutrophil swarming. Nature Biomedical Engineering. 1 (7), 1-12 (2017).
- Morgan, A. S. Risk factors for infection in the trauma patient. Journal of the National Medical Association. 84 (12), 1019-1023 (1992).
- Szczerba, B. M., et al. Neutrophils escort circulating tumour cells to enable cell cycle progression. Nature. 566, 553-557 (2019).
- Treffers, L. W., Hiemstra, I. H., Kuijpers, T. W., van den Berg, T. K., Matlung, H. L. Neutrophils in cancer. Immunological Reviews. 273, 312-328 (2016).
- Grayson, P. C., Kaplan, M. J. At the Bench: Neutrophil extracellular traps (NETs) highlight novel aspects of innate immune system involvement in autoimmune diseases. Journal of Leukocyte Biology. 99 (2), 253-264 (2016).
- Lämmermann, T. In the eye of the neutrophil swarm--navigation signals that bring neutrophils together in inflamed and infected tissues. Journal of Leukocyte Biology. 100 (1), 55-63 (2016).
- Kienle, K., Lämmermann, T. Neutrophil swarming: an essential process of the neutrophil tissue response. Immunological Reviews. 273 (1), 76-93 (2016).
- de Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nature Reviews Immunology. 16 (6), 378-391 (2016).
- Lämmermann, T., et al. Neutrophil swarms require LTB4 and integrins at sites of cell death in vivo. Nature. 498 (7454), 371-375 (2013).
- Kreisel, D., et al. In vivo two-photon imaging reveals monocyte-dependent neutrophil extravasation during pulmonary inflammation. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18073-18078 (2010).
- Coombes, J. L., Robey, E. A. Dynamic imaging of host-pathogen interactions in vivo. Nature Reviews Immunology. 10 (5), 353-364 (2010).
- Lämmermann, T. Cell migration: Arraying neutrophils in swarms. Nature Biomedical Engineering. 1 (7), 1-2 (2017).
- Powell, D. R., Huttenlocher, A. Neutrophils in the Tumor Microenvironment. Trends in Immunology. 37 (1), 41-52 (2016).
- Uribe-querol, E., Rosales, C. Neutrophils in Cancer: Two Sides of the Same Coin. Journal of Immunology Research. 2015, (2015).
- Underhill, D. M., Ozinsky, A. Toll-like receptors: Key mediators of microbe detection. Current Opinion in Immunology. 14 (1), 103-110 (2002).
- Netea, M. G., Van Der Meer, J. W., Kullberg, B. J. Recognition of fungal pathogens by toll-like receptors. Immunology of Fungal Infections. , 259-272 (2007).
- Thomas, C. J., Schroder, K. Pattern recognition receptor function in neutrophils. Trends in Immunology. 34 (7), 317-328 (2013).
- Prince, L. R., Whyte, M. K., Sabroe, I., Parker, L. C. The role of TLRs in neutrophil activation. Current Opinion in Pharmacology. 11 (4), 397-403 (2011).
- Tan, S. Y., Weninger, W. Neutrophil migration in inflammation: intercellular signal relay and crosstalk. Current Opinion in Immunology. 44, 34-42 (2017).
- Menezes, G. B., Rezende, R. M., Pereira-Silva, P. E. M., Klein, A., Cara, D. C., Francischi, J. N. Differential involvement of cyclooxygenase isoforms in neutrophil migration in vivo and in vitro. European Journal of Pharmacology. 598 (1-3), 118-122 (2008).
- Afonso, P. V., et al. LTB4 Is a Signal-Relay Molecule during Neutrophil Chemotaxis. Developmental Cell. 22 (5), 1079-1091 (2012).
- Gounni, A. S., et al. In Vivo Regulates Neutrophil Migration In Vitro and Chemorepellent Semaphorin 3E Negatively Chemorepellent Semaphorin 3E Negatively Regulates Neutrophil Migration In Vitro and In Vivo. The Journal of Immunology. 198, 1023-1033 (2017).
- Dumic, J., Dabelic, S., Flögel, M. Galectin-3: An open-ended story. Biochimica et Biophysica Acta - General Subjects. 1760 (4), 616-635 (2006).
- Padmanabhan Iyer, R., et al. Matrix metalloproteinase-9-dependent mechanisms of reduced contractility and increased stiffness in the aging heart. Proteomics - Clinical Applications. 10 (1), 92-107 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved