Method Article
Biopartikel-Mikroarrays für chemotaktische und molekulare Analysen des menschlichen Neutrophilen-Schwärmens in vitro
In diesem Artikel
Zusammenfassung
Dieses Protokoll erzeugt Biopartikel-Mikroarrays, die räumlich gesteuerte Neutrophilenschwärme nerieren. Es bietet einfachen Zugriff auf die Mediatoren, die Neutrophile während der Migration freisetzen, und ermöglicht quantitative Bildgebungsanalysen.
Zusammenfassung
NeutrophileSchwärme ist ein kooperatives Verfahren, durch das Neutrophile eine Infektionsstelle abdichten und die Gewebereorganisation fördern. Swarming wurde klassischerweise in vivo in Tiermodellen untersucht, die charakteristische Muster der Zellmigration zeigen. In-vivo-Modelle weisen jedoch mehrere Einschränkungen auf, darunter interzelluläre Mediatoren, die schwer zugänglich sind und analysiert werden können, sowie die Unfähigkeit, menschliche Neutrophile direkt zu analysieren. Aufgrund dieser Einschränkungen ist eine In-vitro-Plattform erforderlich, die das Schwärmen mit menschlichen Neutrophilen untersucht und einen einfachen Zugang zu den molekularen Signalen bietet, die während des Schwärmens erzeugt werden. Hier wird ein mehrstufiger Mikrostamping-Prozess verwendet, um ein Biopartikel-Mikroarray zu erzeugen, das das Schwärmen stimuliert, indem es eine In-vivo-Infektion imitiert. Das Biopartikel-Mikroarray induziert Neutrophile, um kontrolliert und stabil zu schwärmen. Auf dem Mikroarray erhöhen Neutrophile die Geschwindigkeit und bilden stabile Schwärme um Biopartikelhaufen. Zusätzlich wurde überstand, überstanden von den Neutrophilen analysiert und 16 Proteine wurden entdeckt, dass sie im Laufe des Schwärmens differenziell exprimiert wurden. Diese In-vitro-Schwärmeplattform ermöglicht die direkte Analyse der Neutrophilenmigration und Proteinfreisetzung auf reproduzierbare, räumlich kontrollierte Weise.
Einleitung
Neutrophile, die am häufigsten vorkommende weiße Blutkörperchen im Blut1, gewinnen Aufmerksamkeit als potenzielle diagnostische und therapeutische Ziele2,3, weil sie in einer Vielzahl von Erkrankungen beteiligt sein können, einschließlich Gicht4, Sepsis3, Trauma5,6, Krebs 1,7,8, und verschiedene Autoimmunerkrankungen5,9. Neutrophiles Schwärmen ist ein mehrstufiger, streng regulierter Prozess mit einer Komplexität, die ihn zu einem besonders interessanten Schwerpunkt der Studie5,10,11macht. Während des Schwärmens isolieren Neutrophilen eine Entzündungsstelle aus dem umgebenden gesunden Gewebe5,10,11. Richtige Regulierung der neutrophilen Schwärmeist ist wichtig, um die Wundheilung und letztlich Entzündungsauflösung5,12zu fördern. Neutrophile Schwärmen wurde in erster Linie in vivo bei Nagetier12,13,14,15 und Zebrafisch10,11,12,15 Modelle untersucht. Die Art dieser in vivo Tiermodelle führt jedoch zu Einschränkungen5. Zum Beispiel sind die Von Neutrophilen während des Schwärmens freigesetzten Mediatoren für die Analyse5nicht leicht zugänglich. Darüber hinaus gibt es viele potenzielle Quellen für einen bestimmten Mediator in vivo, so dass ein In-vivo-Experiment einen genetischen Mangel einführen muss, um die zelluläre Produktion und/oder Interaktion zu hemmen, um die Rolle dieses Mediators in einem bestimmten Prozess zu untersuchen13. Ein In-vitro-Experiment umgeht diese Komplikation, indem es eine neutrophile Beobachtung ohne den Kontext zusätzlicher Zellen ermöglicht. Zusätzlich ist die Forschung zur Beschreibung der koordinierten Migration durch menschliche neutrophile begrenzt16. Auf einer In-vitro-Schwärmeplattform können menschliche Neutrophile direkt analysiert werden. Eine In-vitro-Schwärmeplattform könnte das Wissen aus In-vivo-Studien erweitern, indem sie Möglichkeiten bietet, die Lücken zu schließen, die die Grenzen von In-vivo-Studien hinterlassen haben.
Um der Notwendigkeit einer In-vitro-Plattform gerecht zu werden, die in vivo neutrophilen Schwärmen imitiert, haben wir eine Mikrostamping-Plattform entwickelt, die es uns ermöglicht, Biopartikel-Mikroarrays zu mustern, die das Neutrophilschwärmen in einer räumlich kontrollierten Weise stimulieren. Wir erzeugen Biopartikel-Mikroarrays auf Glasrutschen in einem zweistufigen Prozess. Zunächst verwenden wir Mikrostamping, um ein Mikroarray von kationischen Polyelektrolyten (CP)-Spots zu erzeugen. Zweitens fügen wir eine Lösung von Biopartikeln hinzu, die über eine elektrostatische Interaktion an den CP-Spots haften. Durch das erste Mustern der CP-Schicht können wir negativ geladene Biopartikel selektiv mustern, um das gewünschte neutrophile Schwarmmuster zu erzeugen. Die positiv geladene Schicht hält die negativ geladenen Biopartikel durch den kräftigen Waschschritt, der die Biopartikel aus den Bereichen auf dem Glasschlitten entfernt, die nicht über den CP verfügen. Zusätzlich ist der hier verwendete CP, ein Copolymer aus Acrylamid und quaternisiertem kationischen Monomer, biokompatibel, so dass es keine Reaktion der Neutrophilen induziert. Es hat eine sehr hohe Oberflächenladung, die die mikrongroßen Biopartikel zum Glasschlitten immobilisiert und so Neutrophilen daran hindert, die Partikel aus der gemusterten Position auf dem Glasschlitten zu entfernen. Dies führt zu Biopartikel-Clustern, die in einem Mikroarray angeordnet sind. Als wir dem Mikroarray Neutrophilen hinzufügten, bildeten sie stabile Schwärme um die Biopartikelhaufen. Durch die Verfolgung der Neutrophilenmigration fanden wir heraus, dass schwärmende Neutrophilen aktiv in richtungsumhergehende Biopartikel-Cluster. Darüber hinaus nutzten wir diese Plattform, um bestimmte Mediatoren zu analysieren, die Neutrophile während des Schwärmens freisetzen. Wir fanden 16 Mediatoren, die während des Schwärmens unterschiedlich ausgedrückt werden. Ihre Konzentrationen folgen drei allgemeinen Trends im Laufe der Zeit: Anstieg, Abnahme oder Spitze. Unsere in vitro neutrophile Schwärmeungsplattform erleichtert die Analyse des räumlich gesteuerten menschlichen Neutrophilenschwärmens sowie die Sammlung und Analyse von Mediatoren, die durch neutrophilesches Schwärmen freigesetzt werden. In einer früheren Publikation haben wir gezeigt, dass Patienten mit bestimmten Erkrankungen (Trauma, Autoimmunerkrankung und Sepsis) Neutrophile hatten, die anders funktionierten als Patienten von gesunden Spendern5. In zukünftigen Forschungsstudien könnte unsere Plattform verwendet werden, um die Neutrophilenfunktion bei einer Vielzahl von Patientenpopulationen zu analysieren. Diese Plattform kann die komplexe Koordination des Neutrophilenschwärmens quantitativ analysieren. Zusätzliche Studien können durchgeführt werden, um Einblicke in die Neutrophilenfunktion einer bestimmten Patientenpopulation oder neutrophilen Reaktionen auf einen Krankheitserreger von Interesse zu geben.
Protokoll
Die Autoren würdigen die gesunden Freiwilligen, die freundlicherweise ihr Blut gespendet haben. Blutproben wurden nach informierter freiwilliger Zustimmung gemäß dem Protokoll des Institutional Review Board (IRB) #2018H0268 vom Biomedical Sciences Committee der Ohio State University überprüft.
1. Mikrofertigung von Biopartikel-Mikroarray
- Erzeugen Sie mit Standard-Photolithographie-Verfahren den Master-Siliziumwafer.
- Generieren Sie einen Nachweis des gewünschten Designs mit einer CAD-Software (Computer-Aided Design), und senden Sie ihn dann an einen Fotomaskenhersteller, um eine Chrom-Fotomaske herzustellen. Hier wird 4 mm x 4 mm rechteckige Arrays mit einem Durchmesser von 30 mm in Kreisen mit einem Abstand von 150 m in der Mitte verwendet. Dieses Design kann für verschiedene Anwendungen nach Belieben geändert werden.
- Verfeinern Sie eine 40 m dicke Schicht eines negativen Photoresists auf einen Siliziumwafer. Wafer bei 65 °C für 5 min und 95 °C für 10 min backen.
- Stellen Sie den Wafer durch eine Chrom-Fotomaske mit 150–160 mJ/cm2 (Abbildung 1A) UV-Licht aus .
- Waffeln bei 65 °C für 5 min und 95 °C für 10 min backen. Wafer im Photoresist-Entwickler für 10 min untertauchen und mit Isopropylalkohol abspülen. In diesem Stadium sollte das Muster auf dem Wafer sichtbar sein (Abbildung 1B).
- Ein 10:1-Verhältnis von Polydimethylsiloxan-Prepolymer und dessen Härtungsmittel (d.h. 20 g Prepolymer und 2 g Aushärtungsmittel) gründlich mischen und das ungehärtete Polydimethylsiloxan -Gemisch (PDMS) über den Masterwafer in eine Petrischale gießen (Abbildung 1C).
- Vakuum behandeln Sie das ungehärtete PDMS-Gemisch, bis keine Luftblasen über dem Master-Wafer vorhanden sind. Bei 65 °C über Nacht aushärten.
- Verwenden Sie ein Skalpell, um das Äußere des gemusterten Teils des Wafers zu schneiden, und entfernen Sie langsam die ausgehärtete PDMS-Platte. Legen Sie die PDMS-Platte auf ein sauberes Schneidebrett mit der gemusterten Seite nach oben (Abbildung 1D).
- Stempel aus der PDMS-Platte mit einem 8 mm Biopsie-Stempel ausstechen (Abbildung 1E). Für eine Glasrutsche werden acht Stempel benötigt.
- Legen Sie jeden Stempel mit dem Gesicht nach unten auf das Klebeband, um Schmutz zu entfernen.
- Bereiten Sie im Voraus eine 1,6 mg/ml-Lösung von CP in Wasser vor.
- Fügen Sie die richtige Menge des CP-Pulvers zu Wasser hinzu (z. B. 0,8 g bis 500 ml).
- Mischen Sie auf einer Rührplatte bei Raumtemperatur über Nacht, oder bis der gesamte Feststoff in das Wasser gelöst ist. Die CP-Lösung kann 6 Monate bei Raumtemperatur gelagert werden.
- Auf Wunsch die CP-Lösung fluoreszierend machen, indem Sie Poly-L-Lysin hinzufügen, das mit Fluoresceinisothiocyanat (PLL-FITC) gekennzeichnet ist.
- Aliquot ca. 10 ml der CP-Lösung. Fügen Sie dem Aliquoted-Volumen eine kleine Menge PLL-FITC (0,05 mg) hinzu. Die Menge kann geändert werden, um die Helligkeit der Fluoreszenz wie gewünscht anzupassen.
- Vortex die CP-Lösung mit FITC für 20 s beschriftet, oder bis die Lösung eine einheitliche, blassgelbe Farbe ist. Vor Licht schützen und bis zu 1 Monat bei 4 °C lagern.
- Mit den Stempeln nach oben, Grund jeder Stempel mit 100 l von 1,6 mg/ml Lösung von CP, um sicherzustellen, dass keine Luftblasen zwischen der CP-Lösung und dem Stempel bilden (Abbildung 1F).
- Invertdiee der Stempel auf eine Schicht der CP-Lösung (Abbildung 1G).
- Entfernen Sie die Stempel aus der CP-Lösung nach 1 h.
- Dab jeder nasse Stempel nach unten auf eine saubere Glasrutsche 6–8x, um überschüssige Flüssigkeit zu entfernen.
- Vakuum behandeln Sie die Briefmarken für 1-2 min.
- Befestigen Sie einen acht Bohrer mit einem Durchmesser von 9 mm auf der Oberseite eines sauberen Glasschlittens als Leitfaden für die Stempelplatzierung. Mit diesem Abstandser kann jeder Glasschlitten acht Mikroarrays haben.
- Platzieren Sie einen Stempel mit dem Gesicht nach unten auf der Glasrutsche in der Mitte jeder Bohrung des Bildraums (d. h. verwenden Sie insgesamt acht Stempel).
- Legen Sie ein ausgewogenes Gewicht von 5,6 x 0,1 g auf jeden Stempel und lassen Sie 10 min zum Stanzen zu (Abbildung 1H). Ein ausgewogenes Gewicht ist erforderlich, um sicherzustellen, dass der Stempel gleichmäßig auf die Glasrutsche gedrückt wird und eine gleichmäßige Übertragung des CP auf die Glasrutsche gefördert wird.
- Entfernen Sie die Gewichte und Stempel von der Glasrutsche (Abbildung 1I). Lassen Sie die CP-Schicht bei Raumtemperatur für 24 h trocknen, bevor Sie die Biopartikel hinzufügen, wie in Schritt 1.17 beschrieben. Wenn der CP mit FITC markiert ist, kann die Wirksamkeit der Stanzung an dieser Stelle mit einem Fluoreszenzmikroskop bei 488 nm überprüft werden, bevor sie mit Schritt 1.17 fortfährt (Abbildung 1M).
- Schneiden Sie eine leere PDMS-Platte auf die Größe des bildgebenden Abstandsraums und verwenden Sie den 8 mm Biopsie-Punch, um Brunnen im PDMS zu erstellen, die sich an den Bohrungen eines bildgebenden Abstandsraums ausrichten. Mit dem Bildabstand(Abbildung 1J) kleben Sie die PDMS-Platte am Glasschlitten an .
- Eine Lösung aus Biopartikeln (z.B. E. coli oder Zymosan) auftauen und in Wasser zur Injektion (WFI) auf 500 g/ml verdünnen.
HINWEIS: Die Biopartikel müssen nicht opsonisiert werden. Neutrophile Oberflächenrezeptoren erkennen Moleküle auf diesen Biopartikeln direkt19,20,21. - Fügen Sie jedem PDMS-Brunnen auf dem Glasschlitten 100 l Biopartikellösung hinzu (Abbildung 1K).
- Schaukeln Sie die Glasrutsche für 30 min.
- Spülen Sie die Brunnen gründlich mit Wasser. Das Biopartikel-Mikroarray kann bis zu 3 Monate in einer staubfreien Umgebung bei 4 °C gelagert werden. An dieser Stelle sollte das Muster mit einem Fluoreszenzmikroskop bei 594 nm überprüft werden, bevor mit Schritt 2.1 (Abbildung 1N) fortfährt.
2. Probenvorbereitung
- Sammeln Sie mindestens 2 ml frisches Blut in K2-EDTA-Röhrchen vom gewünschten Spender. Die erwartete Ausbeute von Neutrophilen beträgt 1–2 x 106 Zellen/1 ml Vollblut. Der bildgebende Assay benötigt etwa 1,5 x 105 Neutrophile, und die Analyse des Überstandes erfordert 1 x 106 Neutrophile. Verwenden Sie das Blut innerhalb von 4 h.
- Trennen Sie rote Blutkörperchen (RBCs) durch Zugabe eines Erythrozyten-Aggregationsmittels im Verhältnis 1:5 zum Vollblut. Warten Sie 45 min auf eine transluzente Schicht (Buffy Coat), um sich von der Schicht der RBCs zu trennen.
- Entfernen Sie das buffy Mantel und waschen Sie mit Phosphat gepufferte Saline (PBS) mit 1 ml Buffy-Mantel: 9 ml PBS.
- Zentrifuge für 5 min bei 190 x g und 20 °C.
- Den Überstand ansaugen und das Pellet bei 5 x 107 Zellen/ml wieder aufhängen.
- Verwenden Sie ein negatives immunmagnetisches Selektionskit, um Neutrophile zu isolieren.
- Fügen Sie 50 l Antikörpercocktail/1 ml Zellsuspension hinzu. Warten Sie 10 min.
- Fügen Sie 100 l magnetische Perlen/1 ml Zellsuspension hinzu. Warten Sie 10 min.
- Fügen Sie eine Zellsuspension zu einem rund-untere Rohr hinzu und legen Sie sie in einen zylindrischen Magneten. Warten Sie 10 min.
- Überstand in ein Zentrifugenrohr gießen. Addieren Sie bis zu 10 ml PBS. Zentrifuge für 5 min bei 190 x g und 20 °C.
- Aspirat Überstand. Resuspend weißes Pellet in IMDM mit 0,4% humanem Serumalbumin.
- Fleckenkerne mit 20 g/ml Hoechst 33342 für 10 min bei 37 °C.
- Fügen Sie 5 ml IMDM mit 0,4% humanem Serumalbumin hinzu, um zu spülen. Zentrifuge für 5 min bei 190 x g und 20 °C.
- Resuspend Zellen bei 7,5 x 105 Zellen/ml in IMDM mit 0,4% menschlichem Serumalbumin.
- Fügen Sie 100 l der Zellsuspension in einen PDMS-Brunnen ein, das ein Biopartikel-Mikroarray enthält. Stellen Sie sicher, dass die Zellsuspension über dem PDMS-Brunnen konvex ist und keine Blasen enthält.
- Dichtung mit einem Abdeckschlupf von 12 mm Durchmesser. Bedecken Sie die Öffnung des PDMS-Brunnens mit einem Abdeckschlupf von 12 mm Durchmesser. Drücken Sie vorsichtig mit einer Pinzette auf den Deckelschlupf, damit die überschüssige Zellsuspension an den Rand des Brunnens entweicht. Verwenden Sie ein Gewebe, um die überschüssige Zellsuspension zu entfernen.
3. Ausführen der Assay- und Bildanalyse
- Laden Sie das Mikropartikelarray mit Zellen auf der Live-Zell-Bildgebungsstation mit einem Mikroskop, das mit einem Käfig-Inkubator ausgestattet ist, der auf 37 °C, 5 %CO2und 90 % relative Luftfeuchtigkeit eingestellt ist.
- Verwenden Sie Zeitraffer-Fluoreszenz- und Hellfeldmikroskopie, um Bilder mit 10-facher Vergrößerung alle 10 s bei 405 nm, 594 nm und hellfeldaufstellen zu lassen. In einem typischen Experiment werden Bilder bis zu 2 h gesammelt.
- Verwenden Sie eine automatisierte Zellverfolgungssoftware, um die Migration einzelner Neutrophilen in den Biopartikelcluster zu verfolgen.
- Verwenden Sie den Autoregressionsmodus einer Spot-Erkennungs-Zellverfolgungssoftware. Stellen Sie den Spotradius auf 5 m (die ungefähre Größe eines neutrophilen Kerns) ein. Legen Sie die minimale Spurlänge auf 120 s und eine maximale Spaltgröße von einem Frame fest.
- Extrahieren Sie aus den von der Zellverfolgungssoftware generierten Daten die Dateien, die die Position und Geschwindigkeit der Neutrophilen enthalten. Diese Dateien können mit einer Graphik-Software verwendet werden, um Neutrophilen-Migrationsspuren zu generieren (Abbildung 2C) und eine Heatmap von Geschwindigkeit vs. Zeit (Abbildung 2D), bzw. .
- Verwenden Sie die 405 nm fluoreszierenden Bilder, um die Schwarmgröße im Laufe der Zeit auf einer Bildanalysesoftware Ihrer Wahl zu verfolgen.
- Definieren Sie Regionen von Interesse (ROIs) um jeden Biopartikelhaufen, in dem Neutrophile schwärmen. Halten Sie den ROI in der gleichen Größe, um jeden Biopartikelcluster zu analysieren.
- Analysieren Sie die mittlere fluoreszierende Intensität der 405 nm-Bilder innerhalb jedes ROI im Laufe der Zeit.
- Generieren Sie eine Kalibrierkurve von mittlerer Fluoreszenzintensität bis zur Schwarmgröße, indem Sie manuelle Messungen in verschiedenen Schwarmgrößen von 0 bis2 bis zur maximalen Schwarmgröße durchführen. Verwenden Sie diese Kalibrierung, um die Schwarmgröße im Zeitverlauf zu berechnen.
4. Supernatant-Sammlung und Proteinnachweis
- Inkubieren Sie die Neutrophilen in den Brunnen, die das Biopartikel-Mikroarray bei 37 °C und 5%CO2 für 3 h enthalten. Nehmen Sie Proben zu den gewünschten Zeitpunkten. In der Regel werden die Proben bei 0, 0,5, 1 und 3 h entnommen. Um die Nachweisgrenze des Protein-Array-Assays zu überwinden, wurde für jeden Zeitpunkt das gesamte Überstandvolumen eines einzelnen Brunnens (200 l) verwendet. Jeder Zeitpunkt wurde in dreifacher Ausfertigung analysiert.
- Überprüfen Sie mit einem Hellfeldmikroskop, ob sich Schwärme auf dem Mikroarray bilden.
- Den Überstand mit einer 200-L-Pipette ansaugen und in ein 0,45-mm-Zentrifugenfilterrohr eintragen.
- Zentrifugieren Sie den Überstand bei 190 x g und 20 °C für 5 min und sammeln Sie das filtrierte Volumen.
- Proben bei -80 °C bis zur Bearbeitungszeit lagern.
- Verwenden Sie ein Microarray-Kit, das eine Reihe menschlicher Proteine erkennt, um Proben zu verarbeiten.
- Fügen Sie 200 l jeder Probe in ein separates Dialyserohr, das mit dem Kit geliefert wird, hinzu.
- Die Dialyseröhrchen in ein Becherglas mit mindestens 500 ml PBS (pH = 8,0) legen. Auf einer Rührplatte mindestens 3 h bei 4 °C vorsichtig umrühren. Ändern Sie die PBS im Becher, und wiederholen Sie diesen Schritt.
- Jede Probe in ein sauberes Zentrifugenrohr und eine Zentrifuge bei 9.000 x g für 5 min geben, um eventuelle Ausscheidungen zu entfernen. Übertragen Sie jeden Überstand auf ein sauberes Rohr.
- Biotinylat ieren Sie jede Probe, indem Sie 36 l 1x Etikettierungsreagenz aus dem Kit pro 1 mg Gesamtprotein in der dialysierten Probe zu 180 l Dialyseprobe hinzufügen. Bei 20 °C für 30 min inkubieren. Alle 5 min sanft mischen.
- Fügen Sie jedem Probenrohr 3 L Stop-Lösung mit dem Kit hinzu. Jede Probe in ein frisches Dialyserohr geben und die Schritte 4.6.2–4.6.3 wiederholen. In diesem Stadium kann die Probe bei -20 °C oder -80 °C gelagert werden, bis Sie bereit sind, fortzufahren.
- Die mit gelieferte Glasrutsche wird bei -20 °C gelagert. Lassen Sie es auf Raumtemperatur kommen. Legen Sie die montierte Glasrutsche für 1-2 h bei Raumtemperatur in eine laminare Durchflusshaube.
- Fügen Sie 400 L des mit dem Kit gelieferten Sperrpuffers in jeden Brunnen des montierten Glasschlittens ein. Bei Raumtemperatur 30 min inkubieren.
- Zentrifugieren Sie die vorbereiteten Proben für 5 min bei 9.000 x g, um Ausscheidungen oder Partikel zu entfernen. 5x mit Sperrpuffer verdünnen.
- Entfernen Sie den Blockierungspuffer aus jedem Brunnen. Fügen Sie 400 l der verdünnten Proben in die entsprechenden Bohrungen ein. 2 h bei Raumtemperatur beim Schaukeln inkubieren.
- Dekantdien Sie die Proben von jedem Brunnen. Waschen Sie 3x mit 800 l des 1x Waschpuffers, den ich beim Schaukeln bei Raumtemperatur für jeweils 5 min mit dem Kit zur Verfügung gestellt habe.
- In einem sauberen Behälter die montierte Glasrutsche in 1x Waschpuffer I untertauchen. 2x bei Raumtemperatur für jeweils 5 min beim Schaukeln waschen.
- Fügen Sie jedem Unterarray 400 L 1x Cy3-konjugiertes Streptavidin hinzu. Abdeckung mit Kunststoff-Klebestreifen. Schützen Sie sich für den Rest des Protokolls vor Licht.
- 2 h bei Raumtemperatur beim Schaukeln inkubieren.
- Dekantieren Sie die Lösung und zerlegen Sie die Glasrutsche aus den Probenkammern.
- In dem 30 ml Zentrifugenrohr, das mit dem Kit geliefert wird, fügen Sie vorsichtig den Glasschlitten und genügend 1x Waschpuffer I hinzu, um die Glasrutsche abzudecken. Waschen Sie 3x für jeweils 10 min bei Raumtemperatur beim Schaukeln.
- Im 30 ml Zentrifugenrohr 2x mit 1x Waschpuffer II für je 5 min bei Raumtemperatur beim Schaukeln waschen.
- Waschen Sie die Glasrutsche mit 30 ml ddH2O für 5 min. Entfernen Sie die Glasrutsche aus dem Zentrifugenrohr und lassen Sie 20 min in einer laminaren Durchflusshaube trocknen. Der vorbereitete Glasschlitten kann bei -20 °C gelagert werden, bis es zum Scannen bereit ist.
- Scannen Sie das Glasschlitten mit einem Mikroarray-Scanner bei einer Fluoreszenzemission von 555 nm.
Ergebnisse
Wenn Neutrophilen dem Biopartikel-Mikroarray zugesetzt werden, werden Neutrophile, die die Biopartikel-Cluster kontaktieren, aktiviert und initiieren die Schwarmreaktion. Das Biopartikel-Mikroarray wurde mittels Zeitraffer-Fluoreszenzmikroskopie validiert, um die Neutrophilenmigration zu den Biopartikelclustern zu verfolgen (Video S1). Die Migration einzelner neutrophiler Kerne wird verfolgt, während sie in Richtung des Biopartikelclusters wandern. Wenn Neutrophile den Biopartikelhaufen erreichen, überschneiden sich ihre Kerne mit anderen Kernen im Cluster. Daher ist es nicht möglich, einen Neutrophilen innerhalb des Clusters mit dieser Methode genau zu verfolgen. Zymosan- und E. coli-Biopartikel-Cluster führen beide zur Erzeugung von Neutrophilenschwärmen. Für unsere Ergebnisse verwendet Abbildung 2B Daten von neutrophilen Schwärmen, die von E. coli-Partikeln erzeugt werden. Abbildung 3 und die anderen Tafeln von Abbildung 2 verwenden neutrophile Schwärme, die von Zymosanpartikeln erzeugt werden. Die erzielten Ergebnisse zeigen, dass Biopartikelcluster die Neutrophilenaktivierung stimulieren, wenn ein Neutrophiler den Cluster kontaktiert, was schließlich zur Bildung stabiler Neutrophiler Schwärme um jeden Cluster nach 30–60 min führte(Abbildung 2A, oben). Im Gegensatz dazu zeigten Neutrophilen keine kollektive Migration ohne Biopartikelcluster (Abbildung 2A, unten und Video S2). Bei Verwendung der fluoreszierenden Intensität von gefärbten neutrophilen Kernen bei 405 nm wurde festgestellt, dass die durchschnittliche Neutrophilenschwarmgröße um 30 'm Durchmesser E. coli Biopartikelcluster 1.490 x 680 'm2 betrug (Mittelwert SD, Abbildung 2B, oben). Die Fluoreszenzintensität einer bestimmten Region von Interesse, in der keine Biopartikelcluster vorhanden sind, war im Laufe der Zeit annähernd konstant, was das Fehlen einer kollektiven Migration in dieser Umgebung bestätigte(Abbildung 2B, unten). Spuren der Neutrophilenmigration zeigen, dass Neutrophilen auf einem Biopartikelhaufen konvergierten, wenn man vorhanden war(Abbildung 2C, oben). Umgekehrt wurde keine Konvergenz im Kontrollsystem beobachtet(Abbildung 2C, unten). Die Geschwindigkeit (zurückgelegte Strecke/Zeit) von schwärmenden und nicht aktivierten Neutrophilen wurde gemessen und es wurde ein statistisch signifikanter Unterschied in den Geschwindigkeitsverteilungen gefunden (ANOVA, p < 0.0001), wie in Abbildung 2Ddargestellt. Die durchschnittliche Geschwindigkeit für schwärmende Neutrophile betrug 20,6 x 13,0 m/min (mittleres SD), und die Durchschnittsgeschwindigkeit für Kontrollneutrophile betrug 2,0 x 2,2 m/min.
Zusätzlich wurde die Konzentration von 16 Proteinen analysiert, die Neutrophile während des Schwärmens freisetzten (Abbildung 3). Die normalisierte Konzentration jedes Proteins zu unterschiedlichen Zeitpunkten im Schwarmprozess (t = 0, 0,5, 1 und 3 h) wurde berechnet, wobei die normalisierte Konzentration (C – Cmin)/ (Cmax – Cmin)beträgt. 10 Proteine erhöhten die Konzentration während des Schwarms. Diese Proteine waren Adipsin, Galectin-3, GROa, IL-6R, MIP-1a, MMP-8, MMP-9, Nidogen-1, TIMP-1 und TLR2. Zwei Proteine (Pentraxin 3 und RANK) verringerten sich während des Schwarms in der Konzentration. Die restlichen vier Proteine (Clusterin, PF4, RANTES und Trappin-2) nahmen in der ersten Stunde des Schwärmens zu, gingen danach aber zurück. Mit anderen Worten, die Konzentrationen dieser Proteine "spiked" während des Schwärmens. Von den 16 identifizierten Proteinen wurden 12 Proteine in unserer vorherigen Publikation5identifiziert. Adipsin, Galectin-3, Nidogen-1, Pentraxin 3, TIMP-1 und TLR2 wurden als schwärmend spezifisch gezeigt, während Clusterin, IL-6R, MMP-8 und MMP-9, RANK und Trappin-2 nicht5waren.

Abbildung 1: Herstellung von Biopartikel-Mikroarray. Ein mit negativem Photoresist beschichteter Siliziumwafer wird durch eine Chrom-Fotomaske (A )UV-Licht ausgesetzt. Nach dem Backen und Entwickeln des Siliziumwafers verbleibt ein Photoresist-Muster auf der Oberfläche des Siliziumwafers. Dies ist der Master-Wafer (B). In einer Petrischale wird eine PDMS-Mischung an der Oberseite des Master-Wafers hinzugefügt. Das PDMS wird über Nacht bei 65 °C zur PDMS-Form (C )ausgehärtet. Die PDMS-Form wird aus dem Master-Wafer geschnitten und ein Biopsie-Punch wird verwendet, um einzelne PDMS-Stempel auszustechen (D). Ein PDMS-Stempel (E) ist mit CP-Lösung beschichtet (F). Der Stempel wird auf eine dünne Schicht CP-Lösung (G) invertiert. Nach dem Inkubieren in der CP-Lösung für 1 h wird der Stempel auf eine Glasrutsche geschmiert, um überschüssiges CP zu entfernen. Acht Stempel werden dann auf einen sauberen Glasschlitten mit einem Gewicht von 5,6 x 0,1 g gedrückt, ausgerichtet auf einen bildgebenden Abstandsraum (H). Wenn der Stempel entfernt wird, bleibt ein CP-Muster erhalten (I). Eine PDMS-Platte mit vorgeschnittenen Bohrungen wird am Glasschlitten (J) verkrümt. Dann wird eine Biopartikellösung über das CP-Muster (K) hinzugefügt. Die negativ geladenen Biopartikel binden über elektrostatische Wechselwirkungen an den positiv geladenen Polyelektrolyten. Die überschüssige Biopartikellösung wird weggespült, so dass Biopartikel auf dem CP-Muster gemustert werden (L). (M) Fluoreszierendes Bild der CP-Schicht mit FITC beschriftet (Skalenbalken = 50 m, großes Bild; Skala bar = 25 'm, inset). (N) Fluoreszierendes Bild gemusterter Zymosan-Biopartikel, konjugiert mit Texas Red (Scale bar = 100 m, großes Bild; Skalenbalken = 50 m, Einschub). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Neutrophiler Schwarmwachstum um Biopartikel-Cluster. In Gegenwart von Biopartikelclustern durchlaufen Neutrophile eine kollektive Migration zu den Biopartikelclustern (Boden,Kontrolle von Neutrophilen). Wenn keine Biopartikel vorhanden sind, führen die Neutrophilen keine kollektive Migration durch. (A) In Gegenwart von zymosanischen Biopartikel-Clustern bilden Neutrophile in 30 min Schwärme. Ohne Biopartikel-Cluster bilden sich keine Schwärme (Scale bars = 50 'm). (B) Neutrophile Schwärme wachsen auf eine durchschnittliche Größe von 1.490 x 680 m2 (mittelwert sD) um 30 'm Durchmesser E. coli Biopartikel-Cluster. Kontrollneutrophile weisen eine konstante Dichte auf, die keinem Schwarmwachstum entspricht (ANOVA, p < 0,0001, n = 32 Neutrophilenschwärme, N = 1 Spender, Fehlerbalken = Standardabweichung). (C) Spuren von schwärmenden Neutrophilen konvergieren auf den zymosanischen Biopartikel-Clustern, während Kontrollneutrophile keinen konvergierenden Punkt zeigen (Scale bars = 50 m). (D) Neutrophile, die auf ein Zymosenziel zustürmen, haben eine mittlere Geschwindigkeit von 20,6 x 13,0 m/min (mittleres SD), während Die Kontrollneutrophile eine mittlere Geschwindigkeit von 2,0 x 2,2 m/min haben. Jeder Zähler auf der Heatmap stellt einen Neutrophilen mit der angegebenen Momentatsgeschwindigkeit zum angegebenen Zeitpunkt dar. Diese Heatmaps sind repräsentativ für ein Experiment (n = 1 Schwarm; N = 1 Spender; ANOVA, 6,114 = schwärmende Neutrophile, 32.116 = Kontrollneutrophile, p < 0,0001). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
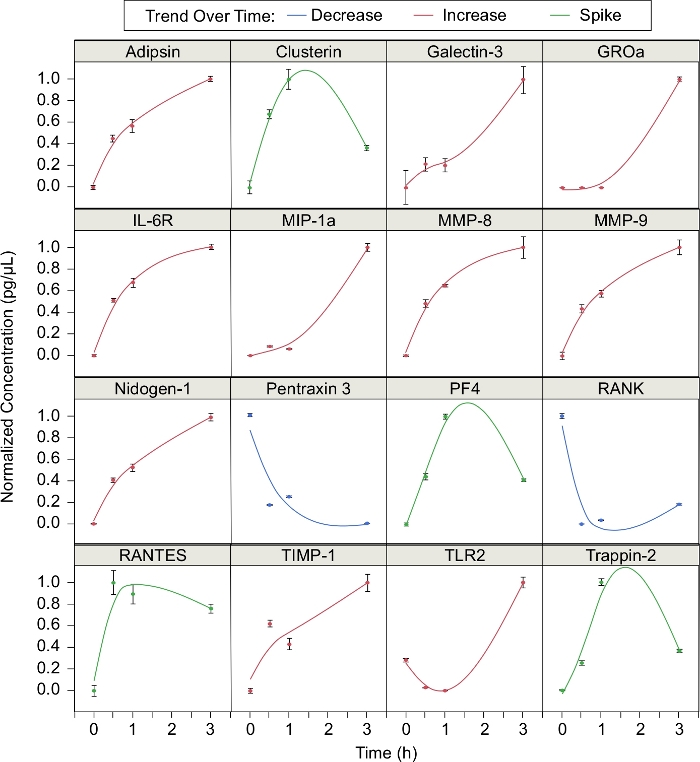
Abbildung 3: Freie Mediatoren, die von schwärmenden Neutrophilen freigesetzt werden. Neutrophiles Schwärmen beeinflusst die Produktion verschiedener Proteine im Laufe der Zeit. Die Proteinkonzentration wurde bei 0, 0,5, 1 und 3 h gemessen. Die Datenpunkte wurden mit glättungssplines ausgestattet ( n = 0,05). Diese Proteine folgen in der Regel einem von drei charakteristischen Trends: abnehmend im Laufe der Zeit, Zunahme im Laufe der Zeit, oder Spiking um 1 h und dann abnehmen (Fehlerbalken = Standardabweichung; n = 3 repliziert; N = 1 Spender). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Video S1: Neutrophile Schwärmen in Richtung zymosanbioser Biopartikel-Cluster. Neutrophile Kerne sind blau dargestellt. Zymosan-Targets sind mit roten Kreisen markiert (Skalenbalken = 50 m; ursprüngliche Erfassungszeit = 60 min). Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video S2: Nicht aktivierte neutrophile zuzufällige Migration. Neutrophile Kerne werden blau dargestellt (Scale bar = 50 m; ursprüngliche Erfassungszeit = 60 min). Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Wir entwickelten eine Mikrostamping-Plattform, um einheitliche Arrays von Biopartikeln zu erzeugen, um den in vitro neutrophilen Schwärmen zu stimulieren. Die In-vitro-Natur unserer Plattform ermöglicht es uns, die Komplikationen zu umgehen, die mit In-vivo-Schwärmeexperimenten entstehen, nämlich die schlechte Fähigkeit, Mediatoren zu analysieren, die von schwärmenden Neutrophilen freigesetzt werden5. Darüber hinaus werden In-vivo-Modelle in der Regel bei Nagetieren11,12,13,15,22,23oder Zebrafisch11,12,15,23. Unsere Plattform verwendet menschliche Neutrophile, die es uns ermöglichen, unsere Ergebnisse direkter im Kontext menschlicher Krankheiten zu interpretieren, obwohl bestimmte Ähnlichkeiten zwischen Maus und menschlichen Neutrophilen beobachtet wurden5,11. Darüber hinaus unterhalten wir eine räumlich gesteuerte Schwärmeumgebung, die unsere Plattform von In-vivo-Modellen unterscheidet, indem wir ein hohes Maß an Reproduzierbarkeit bieten, das die Analyse der menschlichen neutrophilen kollektiven Migration sowie die Sammlung und Analyse von Mediatoren erleichtert, die von schwärmenden Neutrophilen freigesetzt werden.
Bei der Entwicklung unseres Microstamping-Protokolls ergaben sich mehrere Herausforderungen, die eine sorgfältige Fehlerbehebung erforderten. Erstens ist der CP, der für die Mikroprägung verwendet wird, hochhydrophil, und die PDMS-Stempel sind hydrophob. Da der CP keine hohe Affinität zu PDMS hat, wurde unser Verfahren sorgfältig entwickelt, um die Bildung von Blasen zu vermeiden und die Benetzung zu fördern. Durch das erste Angrundieren der Marke mit CP während des Face-up (Schritt 1.8) minimieren wir die Bildung von Blasen zwischen dem CP und dem Stempel. Der Stempel wird dann auf eine Schicht von CP invertiert und für 1 h inkubiert. Diese lange Inkubationszeit sorgt dafür, dass jeder Abschnitt der Marke benetzt wird. Zweitens kann der Prozess des Entfernens überschüssiger CP vor dem Stempeln auf einem sauberen Glasschlitten (Schritt 1.11) inkonsistent sein. Bei der Durchführung von Schritt 1.11 muss der Stempel sorgfältig geprüft werden. Wenn das Muster sichtbar wird, ist der Stempel bereit für Schritt 1.12. Zusätzlich kann die erforderliche Vakuumzeit zum Trocknen der Stempel (Schritt 1.12) variieren. Das hängt in erster Linie vom Wetter ab. An einem warmen, feuchten Tag sind 2 min Vakuumzeit erforderlich. An einem kühlen, trockenen Tag reichen 1 min Vakuumzeit aus.
Wir haben gezeigt, dass Neutrophile von gesunden Spendern stabile Schwärme um Biopartikel-Cluster bilden. Mit der Zeitraffer-Fluoreszenzmikroskopie können wir die Größe des Schwarms quantifizieren und die Neutrophilenmigration verfolgen, was es uns ermöglicht, neutrophile Chemotaxis quantitativ zu analysieren5. Zum Beispiel haben wir zuvor gezeigt, dass diese Plattform verwendet werden kann, um den chemotaktischen Index (CI, den Kosinus des Winkels zwischen dem Neutrophilen-Geschwindigkeitsvektor und dem Positionsvektor zwischen dem Neutrophilen und dem nächsten Biopartikelcluster), Geschwindigkeit (Entfernung der Neutrophilenreisen geteilt durch die Zeit), radiale Geschwindigkeit (die Geschwindigkeit multipliziert mit CI) und die Gesamtentfernung (die Differenz zwischen der ursprünglichen und der endgültigen neutrophilen Position) der einzelnen Neutrophilenzu berechnen. Im Gegensatz zu den meisten In-vitro-Studien24,25,26, hat unsere Plattform keinen künstlichen chemotaktischen Gradienten, so dass neutrophile interzelluläre Kommunikation die einzige treibende Kraft der Neutrophilenmigration ist. Darüber hinaus ist der von schwärmenden Neutrophilen erzeugte Überstand leicht zugänglich. Wir können den Überstand für interzelluläre Mediatoren sammeln und analysieren, die von Neutrophilen freigesetzt werden, ohne Interferenzen von anderen Zelltypen, die in vivo vorhanden sind. Es wurden 16 Proteine beobachtet, deren Expression während des Schwärmens als einer von drei Trends beschrieben werden konnte: Zunahme (10 Proteine), Abnahme (zwei Proteine) und Spitze (vier Proteine). Sechs dieser Proteine bestätigten zuvor berichtete Trends während des Schwarms im Laufe der Zeit5. Einige der identifizierten Proteine wurden zuvor als schwärmend-spezifisch nachgewiesen, während andere differenziertdurch aktivierte nicht-schwärmende Neutrophile 5 exprimiert wurden. Proteine, die im Laufe der Zeit die Konzentration erhöhen, sind wahrscheinlich mit der Pro-Entzündungs-Reaktion verbunden. Einige der Proteine, die im Laufe der Zeit in der Konzentration zunehmen, sind bereits bekannt, dass sie an der entzündungshemmenden Reaktion beteiligt sind (z. B. Galectin-3 und MMP-9)27,28. Die Beziehung zwischen anderen Proteinen und Entzündungen ist weniger gut verstanden. Die Proteine, die während des Schwärmens spike oder abnehmen, können an der Regulierung von Entzündungen beteiligt sein. Jedoch, weitere Forschung ist notwendig, um die Rolle bei der Entzündung vieler Proteine zu verstehen, die während des Schwärmens differenziell exprimiert werden. Die Analyse freigesetzter Mediatoren zusammen mit der Neutrophilenmigration kann uns helfen, das komplexe Bild der Entzündung besser zu verstehen und wie Neutrophile das umgebende Gewebe während des Schwärmens beeinflussen.
In zukünftigen Studien kann unsere Plattform genutzt werden, um die Fähigkeit von ungesunden Neutrophilen gründlich zu untersuchen, stabile Schwärme um gemusterte Biopartikelcluster zu erzeugen. Verschiedene Erkrankungen wurden mit Neutrophilen in Verbindung gebracht, einschließlich Sepsis3, Trauma6, und Krebs1,8,17,18, was darauf hindeutet, dass Neutrophile bei diesen Patienten die Funktion verändert haben können. Diese Plattform kann verwendet werden, um Unterschiede zwischen den interzellulären Mediatoren zu untersuchen, die von gesunden und ungesunden Neutrophilen freigesetzt werden. Zusätzlich könnte diese Plattform modifiziert werden, um lebende Mikroben zu mustern und verwendet werden, um neutrophile Reaktion auf lebende Mikroben in vitro zu analysieren.
Abschließend haben wir eine neuartige Plattform zur Analyse des neutrophilen Schwärmens in vitro entwickelt. Die hochkontrollierte Natur unserer Plattform ermöglicht es uns, Probleme zu mildern, die während in vivo neutrophiler Schwärmeexperimente auftreten. Neutrophileschimten auf einem Biopartikel-Mikroarray ist durch Zeitraffer-Fluoreszenzmikroskopie leicht quantifizierbar. Zusätzlich können wir die von den Neutrophilen freigesetzten Mediatoren ohne die Einmischung anderer Gewebe, die in vivo vorhanden sind, sammeln. Diese Plattform kann in zukünftigen Forschungsarbeiten genutzt werden, um Unterschiede zwischen dem Migrationsverhalten von Neutrophilen von gesunden und ungesunden Spendern zu quantifizieren.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch Fördermittel des William G. Lowrie Department of Chemical and Biomolecular Engineering und des Comprehensive Cancer Center an der Ohio State University unterstützt. Die in diesem Bericht vorgestellten Daten stammen aus Bildern, die mit Imaris x64 (ver. 9.3.0 Bitplane) verarbeitet wurden, die auf der Campus Microscopy and Imaging Facility, The Ohio State University, verfügbar sind. Diese Einrichtung wird zum Teil durch das Stipendium P30 CA016058, National Cancer Institute, Bethesda, MD, unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| "The Big Easy" EasySep Magnet | STEMCELL Technologies | 18001 | Magnet to use with neutrophil isolation kit |
| Cell Incubator | Okolab | 777057437 / 77057343 | Okolab cage incubator for temperature and CO2 control |
| EasySep Human Neutrophil Isolation Kit | STEMCELL Technologies | 17957 | Kit for immunomagnetic negative selection of human neutrophils |
| Eclipse Ti2 | Nikon Instruments | MEA54010 / MEF55037 | Inverted research microscope |
| Escherichia coli (K-12 strain) BioParticles Texas Red conjugate | Invitrogen | E2863 | Bioparticle powder, dissolve in water prior to addition to Zetag® array |
| Harris Uni-Core 8-mm biopsy punch | Sigma Aldrich | Z708925 | To cut PDMS stamps |
| HetaSep | STEMCELL Technologies | 7906 | Erythrocyte aggregation agent for separating buffy coat from red blood cells in fresh human blood |
| Hoechst 33342 | Life Technologies | H3570 | Nucleus fluorescent stain |
| Human L1000 Array | Raybiotech Inc. | AAH-BLG-1000-4 | High density array to detect 1000 human proteins |
| Human Serum Albumin (HSA) | Sigma Aldrich | A5843 | Low endotoxin HSA, to prepare 2 % solutions in IMDM for isolated neutrophils |
| Iscove's Modified Dulbeccos' Medium (IMDM) | Thermo Fisher Scientific | 12440053 | To resuspend isolated neutrophils |
| K2-EDTA tubes | Thermo Fisher Scientific | 02-657-32 | Tubes for blood collection |
| Low Reflective Chrome Photomask | Front Range Photomask | N/A | Dimensions 5" x 5" x 0.09" (L x W x D) |
| Microarray Scanner | Perkin Elmer | ASCNGX00 | Fluorescence reader of protein patterned microdomains |
| Microscopy Image Analsysis Software - Imaris | Bitplane | 9.3.0 | Software for automatic cell tracking analysis |
| NiS Elements Advanced Research Software Package | Nikon Instruments | MQS31100 | Software for automatic live cell imaging and swarm size calculation |
| Poly-L-lysine fluorescein isothiocyanate (PLL-FITC) | Sigma Aldrich | P3069-10MG | 30,000 - 70,000 MW PLL labeled with FITC, used to fluorescently label CP solution |
| SecureSeal 8-well Imaging Spacer | Grace Bio-Labs | 654008 | 8-well, 9-mm diameter, adhesive imaging spacer |
| Silicon Wafer | University Wafer | 590 | Silicon 100 mm N/P (100) 0- 100 ohm-cm 500 μm SSP test |
| Spin Coater | Laurell | WS-650MZ-23NPPB | Used to spincoat a 40-µm layer of photoresist onto silicon wafer |
| SU-8 2025 | MicroChem | 2025 | Negative photoresist to make silicon master wafer |
| SU-8 Developer | MicroChem | Y020100 | Photoresist developer. Remove non-crosslinked SU-8 2025 from silicon wafer |
| Sylgard 184 (polydimethylsiloxane, PDMS) | Dow | 1673921 | 2-part silicone elastomer kit for making microstamps and PDMS wells |
| UV Exposure Masking System | Kloé | UV-KUB 2 | Used to crosslink photoresist on silicon wafer through chrome mask with UV light |
| Water | Thermo Fisher Scientific | A1287303 | High quality water to dilute bioparticles |
| Zetag 8185 | BASF | 8185 | Cationic polyelectrolyte (CP), powder, Copolymer of acrylamide and quaternized cationic monomer, forms "inking solution" for microstamping when dissolved in water |
| Zymosan A S. cerevisiae BioParticles Texas Red conjugate | Invitrogen | Z2843 | Bioparticle powder, dissolve in water prior to addition to Zetag array |
Referenzen
- Coffelt, S. B., Wellenstein, M. D., de Visser, K. E. Neutrophils in cancer: neutral no more. Nature Reviews Cancer. 16 (7), 431-446 (2016).
- Jones, C. N., et al. Microfluidic Platform for Measuring Neutrophil Chemotaxis from Unprocessed Whole Blood. Journal of Visualized Experiments. (88), 1-6 (2014).
- Ellett, F., et al. Diagnosis of sepsis from a drop of blood by measurement of spontaneous neutrophil motility in a microfluidic assay. Nature Biomedical Engineering. 2, 207-214 (2018).
- Malawista, S. E., Chevance de Boisfleury, A., Naccache, P. H. Inflammatory Gout: Observations over a Half-Century. The FASEB Journal. 25 (12), 4073-4078 (2011).
- Reátegui, E., et al. Microscale arrays for the profiling of start and stop signals coordinating human-neutrophil swarming. Nature Biomedical Engineering. 1 (7), 1-12 (2017).
- Morgan, A. S. Risk factors for infection in the trauma patient. Journal of the National Medical Association. 84 (12), 1019-1023 (1992).
- Szczerba, B. M., et al. Neutrophils escort circulating tumour cells to enable cell cycle progression. Nature. 566, 553-557 (2019).
- Treffers, L. W., Hiemstra, I. H., Kuijpers, T. W., van den Berg, T. K., Matlung, H. L. Neutrophils in cancer. Immunological Reviews. 273, 312-328 (2016).
- Grayson, P. C., Kaplan, M. J. At the Bench: Neutrophil extracellular traps (NETs) highlight novel aspects of innate immune system involvement in autoimmune diseases. Journal of Leukocyte Biology. 99 (2), 253-264 (2016).
- Lämmermann, T. In the eye of the neutrophil swarm--navigation signals that bring neutrophils together in inflamed and infected tissues. Journal of Leukocyte Biology. 100 (1), 55-63 (2016).
- Kienle, K., Lämmermann, T. Neutrophil swarming: an essential process of the neutrophil tissue response. Immunological Reviews. 273 (1), 76-93 (2016).
- de Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nature Reviews Immunology. 16 (6), 378-391 (2016).
- Lämmermann, T., et al. Neutrophil swarms require LTB4 and integrins at sites of cell death in vivo. Nature. 498 (7454), 371-375 (2013).
- Kreisel, D., et al. In vivo two-photon imaging reveals monocyte-dependent neutrophil extravasation during pulmonary inflammation. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18073-18078 (2010).
- Coombes, J. L., Robey, E. A. Dynamic imaging of host-pathogen interactions in vivo. Nature Reviews Immunology. 10 (5), 353-364 (2010).
- Lämmermann, T. Cell migration: Arraying neutrophils in swarms. Nature Biomedical Engineering. 1 (7), 1-2 (2017).
- Powell, D. R., Huttenlocher, A. Neutrophils in the Tumor Microenvironment. Trends in Immunology. 37 (1), 41-52 (2016).
- Uribe-querol, E., Rosales, C. Neutrophils in Cancer: Two Sides of the Same Coin. Journal of Immunology Research. 2015, (2015).
- Underhill, D. M., Ozinsky, A. Toll-like receptors: Key mediators of microbe detection. Current Opinion in Immunology. 14 (1), 103-110 (2002).
- Netea, M. G., Van Der Meer, J. W., Kullberg, B. J. Recognition of fungal pathogens by toll-like receptors. Immunology of Fungal Infections. , 259-272 (2007).
- Thomas, C. J., Schroder, K. Pattern recognition receptor function in neutrophils. Trends in Immunology. 34 (7), 317-328 (2013).
- Prince, L. R., Whyte, M. K., Sabroe, I., Parker, L. C. The role of TLRs in neutrophil activation. Current Opinion in Pharmacology. 11 (4), 397-403 (2011).
- Tan, S. Y., Weninger, W. Neutrophil migration in inflammation: intercellular signal relay and crosstalk. Current Opinion in Immunology. 44, 34-42 (2017).
- Menezes, G. B., Rezende, R. M., Pereira-Silva, P. E. M., Klein, A., Cara, D. C., Francischi, J. N. Differential involvement of cyclooxygenase isoforms in neutrophil migration in vivo and in vitro. European Journal of Pharmacology. 598 (1-3), 118-122 (2008).
- Afonso, P. V., et al. LTB4 Is a Signal-Relay Molecule during Neutrophil Chemotaxis. Developmental Cell. 22 (5), 1079-1091 (2012).
- Gounni, A. S., et al. In Vivo Regulates Neutrophil Migration In Vitro and Chemorepellent Semaphorin 3E Negatively Chemorepellent Semaphorin 3E Negatively Regulates Neutrophil Migration In Vitro and In Vivo. The Journal of Immunology. 198, 1023-1033 (2017).
- Dumic, J., Dabelic, S., Flögel, M. Galectin-3: An open-ended story. Biochimica et Biophysica Acta - General Subjects. 1760 (4), 616-635 (2006).
- Padmanabhan Iyer, R., et al. Matrix metalloproteinase-9-dependent mechanisms of reduced contractility and increased stiffness in the aging heart. Proteomics - Clinical Applications. 10 (1), 92-107 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten