Method Article
Polysome de criação de perfil em Leishmania, células humanas e testículo de rato
Neste Artigo
Resumo
O objectivo geral da técnica de criação de perfil de polysome é análise da actividade translacional de mRNAs individuais ou mRNAs transcriptome durante a síntese de proteína. O método é importante para estudos de regulamento de síntese de proteínas, ativação de tradução e repressão em saúde e várias doenças humanas.
Resumo
Expressão de proteínas adequada na hora certa e na quantidade certa é a base da função celular normal e sobrevivência em um ambiente de rápida mudança. Por um longo tempo, os estudos de expressão do gene foram dominados pela pesquisa sobre o nível transcricional. No entanto, os níveis de estado estacionário de mRNAs não se correlacionam bem com a produção de proteína, e a Traduzibilidade dos mRNAs próprios varia muito dependendo das condições. Em alguns organismos, como o parasita Leishmania, a expressão da proteína é regulada principalmente no nível de translação. Estudos recentes demonstraram que essa proteína tradução hipotalâmica é associada com câncer, metabólica, neurodegenerativas e outras doenças humanas. Polysome de criação de perfil é um poderoso método para estudar o Regulamento de tradução da proteína. Permite medir o status translacional de mRNAs individuais ou examinar tradução na escala do genoma. A base desta técnica é a separação de polissomos, ribossomos, suas subunidades e mRNAs gratuito durante a centrifugação de uma citoplasmática lisados através de um gradiente de sacarose. Aqui, apresentamos um polysome universal de perfil protocolo usado em três modelos diferentes - parasita Leishmania major, culturas de células humanas e tecidos animais. Células de Leishmania crescem livremente em suspensão e culturas de células humanas crescem em monocamada aderente, enquanto o testículo de rato representa uma amostra de tecido animal. Assim, a técnica é adaptada a todas estas fontes. O protocolo para a análise das frações polysomal inclui detecção de níveis de mRNA individuais por RT-qPCR, proteínas por Western blot e análise de RNAs ribossomal por eletroforese. O método pode ser novamente prorrogado por exame de mRNAs associação com o ribossoma num nível transcriptoma por RNA-seq profunda e análise de proteínas associadas Ribossoma por espectroscopia de massa das frações. O método pode ser facilmente ajustado para outros modelos biológicos.
Introdução
Regulação da expressão gênica em células é controlada por mecanismos transcriptional, posttranscriptional e pós-traducional. Avanços no sequenciamento de RNA profundo permitam o estudo dos níveis de mRNA de estado estacionário no genoma escala em um nível sem precedentes. No entanto, descobertas recentes revelaram que nível de mRNA de estado estacionário não sempre se correlaciona com a produção de proteína1,2. O destino de uma transcrição individual é muito complexo e depende de muitos fatores, como estímulos internos/externos, stress, etc. Regulação da expressão gênica durante a síntese de proteína fornece outra camada de controle de expressão necessária para uma resposta rápida na mudança de condições. Polysome (ou "polyribosome") de criação de perfil, a separação e visualização de ativamente traduzir ribossomas, é um poderoso método para estudar a regulação da síntese de proteínas. Embora, suas primeiras aplicações experimentais apareceram na década de 19603, polysome perfil é atualmente uma das técnicas mais importantes na proteína tradução estudos4. MRNAs único pode ser traduzidos por mais de um ribossoma, levando à formação de um polysome. As transcrições podem ser paralisadas em ribossomas com cicloheximida5 e mRNAs contendo números diferentes de polissomos podem ser separados no processo de fracionamento de polysome por ultracentrifugação gradiente de sacarose6,7 , 8 , 9. análise de RNA de frações polysomal então permite a medição de mudanças nos Estados translacionais de mRNAs individuais no genoma-larga escala e durante diferentes condições fisiológicas4,7, 10. o método também tem sido usado para revelar os papéis de 5' UTR e 3' UTR sequências no controle do mRNA Traduzibilidade próprios11, examinar o papel dos miRNAs em repressão traducional12, descobrir defeitos na biogênese Ribossoma13 e compreender o papel das proteínas Ribossoma-associados com doenças humanas14,15. Durante a última década, um crescente papel para regulação da expressão gênica durante a tradução emergiu que ilustra a sua importância em doenças humanas. A evidência para controle de translação em câncer, metabólica e doenças neurodegenerativas é esmagadora15,16,17,18. Por exemplo, a desregulação do controle traducional eIF4E-dependente contribui para autismo relacionados com os défices15 e FMRP está envolvido em empatar de ribossomos na mRNAs ligados ao autismo14. Assim, polysomal de criação de perfil é uma ferramenta muito importante para estudar os defeitos no Regulamento translacional em várias doenças humanas.
Análise de proteínas de frações polysomal sob diferentes condições fisiológicas disseca a função dos fatores associados a ribossomas durante a tradução. A técnica de polysome de perfil tem sido usada em muitas espécies, incluindo leveduras, células de mamíferos, plantas e protozoários10,19,20,21. Protozoários parasitas como o Trypanosoma e Leishmania exibem limitado controle transcricional da expressão do gene. Seus genomas são organizadas em clusters de gene policistrônico que falta a transcrição regulada promotor22. Em vez disso, a expressão gênica no desenvolvimento é predominantemente controlada ao nível da tradução de proteínas e estabilidade do mRNA em Trypanosomatidae espécie23,24. Portanto, a compreensão do controle translacional na ausência de regulamento transcriptional é particularmente importante para estes organismos. Perfil polysomal é uma ferramenta poderosa para estudar posttranscriptional regulação da expressão gênica em Leishmania25,26,,27,28.
Os recentes progressos na detecção de níveis de mRNAs individuais pelo real tempo PCR quantitativo (RT-qPCR) e transcriptoma completa por sequenciamento de próxima geração, bem como tecnologias de proteômica, traz a resolução e as vantagens de polysomal perfil a um novo nível. A utilização desses métodos pode ser estendida ainda mais através da análise das frações de polysomal individuais por sequenciamento de RNA profundo combinado com análise proteômica para monitorar o status de translação de células na escala do genoma. Isto permite a identificação de novos jogadores moleculares regulamenta tradução sob diferentes condições fisiológicas e patológicas. Aqui, apresentamos um polysome universal de perfil protocolo usado em três modelos diferentes: o parasita Leishmania major, culturas de células humanas e tecidos animais. Apresentamos o Conselho sobre a preparação de lisados celulares de organismos diferentes, otimização das condições de gradiente, escolha de inibidores de RNase e aplicação de RT-qPCR, Western blot e eletroforese de RNA para analisar frações de polysome neste estudo.
Protocolo
Todos os tratamentos de animais e manipulação de tecidos obtidos no estudo foram realizadas de acordo com protocolos aprovados pelo institucional Cuidado Animal e uso do Comité da Texas Tech University Health Science Center, em conformidade com os institutos nacionais de Diretrizes de saúde ao bem-estar dos animais, o número de protocolo 96005. Por favor, sacrificar animais vertebrados e preparar os tecidos de acordo com as orientações do Comité de uso e cuidado institucional do Animal. Se falta tal um comitê, por favor consulte as diretrizes de bem-estar animal do National Institutes of Health. Adulto (> 60 dias de idade) foram utilizados camundongos C57BL/6. Todos os animais e os tecidos foram obtidos de acordo com protocolos aprovados pelo Comité de uso no centro de Ciências de Texas Tech University saúde em conformidade com as diretrizes de bem-estar animal do National Institutes of Health e institucional Cuidado Animal. Por eutanásia, um único rato foi colocado em uma pequena câmara, e o ar foi deslocado gradualmente com cerca de 30% de dióxido de carbono para anestesiar e minimizar o sofrimento do animal. Após a cessação da respiração, usamos o deslocamento cervical para confirmar a morte do animal antes da colheita de tecidos.
Atenção: Todo trabalho com vivo Leishmania e culturas de células humanas foi feito no gabinete em BSL-2 certificado de laboratório de biossegurança.
1. preparação de Lysates citoplasmática de Leishmania Major , culturas de células humanas e tecidos de Mouse
Nota: Existem várias diferenças nas preparações lisadas dos materiais de fonte diferente. Outras etapas, incluindo a preparação de gradiente de sacarose e fracionamento polysomal são idênticas e não dependem de fonte de exemplo.
-
Preparação de lisado citoplasmática de Leishmania major
- Inocular a células de Leishmania major (FV1 estirpe) em 30 mL de 1 x M199 médio29 contendo a mistura de 10% de soro Fetal bovino (FBS) e penicilina/estreptomicina (100 unidades e 100 μg/mL correspondentemente)em densidade de 1 x 105 células/mL .
Nota: Todas as etapas que envolvem as células de Leishmania major devem efectuar-se em uma armário de biossegurança. - Coloque as células na incubadora e cultivá-las a 27 ° C até a fase logarítmica (meados log corresponde a 5 x 106 células/mL). Normalmente demora cerca de dois dias para crescer.
- Adicione cicloheximida à cultura da Leishmania major para uma concentração final de 100 μg/mL para prender os ribossomas na mRNAs traduzidos. Colocar as células em incubadora para 10 min a 27 ° C.
- Após tratamento de cicloheximida, transferir as células para um tubo cónico de 50 mL e girá-los em 1.800 x g e 4 ° C por 8 min. Discard sobrenadante.
- Lavam-se células com 30 mL de solução salina de tampão fosfato de Dulbecco (DPBS). Centrifugue a 1.800 x g e 4 ° C 8 min.
- Desprezar o sobrenadante. Ressuspender as células em 1 mL de DPBS.
- Tomar uma alíquota de células e misturá-lo com solução de formaldeído de 3,5%.
- Contar as células por hemocytometer e determinar a sua concentração. Transferi o número desejado de células no tubo de microcentrífuga. Lisado preparado a partir de 0.5x108-2 x 108 células/mL é suficiente para o carregamento de gradiente de um sacarose.
- Girar as células a 1.800 x g e 4 ° C por 8 min. descartar o sobrenadante.
- Ressuspender as células no gelo em 1 mL de tampão de Lise contendo inibidores de protease e o inibidor de RNase (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 2mm TDT, 1% NP-40, 1 x inibidor da protease cocktail (EDTA-free), inibidor de RNase 200 unidades/mL).
- Passe o lisado através de uma agulha de calibre 23 três vezes. O lisado deve tornar-se transparente após a passagem da agulha.
- Centrifugue a 11.200 x g e 4 ° C por 10 min esclarecer lisado. Transferir o esclareceu lisado para um novo tubo e mantê-lo no gelo até ultracentrifugação gradiente de sacarose.
- Colete 400-500 μL do lisado como entrada (para analisar mais tarde), congelá-lo imediatamente em nitrogênio líquido para análise futura da proteína ou adicionar o reagente de purificação de RNA antes de congelar para análise de RNA.
- Inocular a células de Leishmania major (FV1 estirpe) em 30 mL de 1 x M199 médio29 contendo a mistura de 10% de soro Fetal bovino (FBS) e penicilina/estreptomicina (100 unidades e 100 μg/mL correspondentemente)em densidade de 1 x 105 células/mL .
-
Preparação de lisado citoplasmática de culturas de células humanas de HeLa
- Dividir células HeLa e semente-los em 20 mL do meio DMEM contendo a mistura de 10% FBS e penicilina/estreptomicina (100 unidades e 100 μg/mL correspondentemente) com célula contar 2 x 105 células/mL em uma placa de 15 cm.
- Desenvolvem-se células HeLa a 37 ° C, 5% de CO2 para transfeccao de DNA de plasmídeo de executar de 20-24 h. de acordo com os protocolos do fabricante.
- Propaga as células para 24h depois do transfection a 37 ° C, 5% de CO2.
- Adicione cicloheximida células de HeLa crescidos para a concentração final de 100 μg/mL para prender os ribossomas na mRNAs traduzidos e incubar as células por 10 min a 37 ° C, 5% de CO2. Aspire médio. Lave as células duas vezes com frio DPBS no gelo.
- Adicionar 500 μL de tampão de lise (pH 7,4, 100 mM KCl, 5 mM MgCl2, 1 milímetro DTT, 0,5% NP-40, inibidor de protease 1x cocktail (EDTA-free), 200 unidades/mL de heparina de inibidor ou 1 mg/mL RNase de 20 mM HEPES-KOH) para a placa e raspe as células no gelo.
- Transferi as células lisadas para o tubo de microcentrífuga. Ajuste a concentração de NP-40 para 0,5% e MgCl2 a 5 mM de acordo com o aumento do volume da amostra.
- Passe o lisado através de uma agulha de calibre 23-3 - 6 vezes.
- Gire a 11.200 x g e 4 ° C, durante 8 min esclarecer lisado. Após a centrifugação, transferi o sobrenadante para um tubo novo. Use um espectrofotômetro para avaliar a eficiência de lise celular e para determinar a quantidade de amostra para o carregamento do gradiente. Adicionar 10 μL de amostra de 0,5 mL de 0,1% de sódio Dodecil sulfato de sódio (SDS). Em branco contra 0,1% SDS. Medir a absorvância a 260 nm. Valor de absorvância esperado é em torno de 15-20 unidades/mL.
- Dilua as amostras com tampão de Lise para o mesmo valor de absorvância antes de centrifugação gradiente de sacarose. Mantenha as amostras no gelo até centrifugação gradiente de sacarose.
-
Preparação de lisado citoplasmática de testículo de rato
- Disse o testículo de rato. Faça uma pequena incisão na túnica albugínea e coletar os túbulos seminíferos dos testículos e transferi-los em um tubo cônico de 15 mL contendo 5 mL de DPBS suplementado com fluoreto de phenylmethylsulfonyl de 0,1 mM (PMSF).
- Misture vigorosamente o tecido várias vezes por inversão. Permitir que o tecido pagar na gravidade da unidade no gelo por 5 min.
- Remova e descarte o nublado tampão contendo conjuntivo células e fragmentos de tecido. Repita o procedimento 2-3 vezes mais. A pelota branca restante é enriquecida por túbulos seminíferos e as células germinativas.
- Transfira a pelota do tubo seminífero para um tubo de microcentrifuga de 2ml e girar a 500 x g por 1 min. descartar o sobrenadante.
- Adicione 500 µ l de tampão de lise (20 mM Tris-HCl, pH 7,4, 100 mM KCl, 5 mM MgCl2, 1 milímetro DTT, 0,5% NP-40, inibidor de protease 1x cocktail (EDTA-livre), 1 mg/mL heparina ou 200 unidades/mL de inibidor de RNase) para os túbulos. Use uma pipeta para triture o tecido.
- A suspensão de transferência de um pequeno (0,5-1,0 mL) homogenizacao homogenizador. Perturbar o tecido com sete a oito cursos do pilão de vidro.
- Transfira o lisado para um tubo de microcentrifugadora de 1,5 mL.
- Centrifugar a amostra a 12.000 x g e 4 ° C, durante 8 min limpar o lisado. Transfira o sobrenadante para um tubo novo e a loja no gelo até o carregamento do gradiente de sacarose.
- Coletar 50 μL do lisado como exemplo de entrada, congelá-lo imediatamente a-80 ° C para análise futura da proteína; ou adicionar o reagente de purificação de RNA antes de congelar para análise de RNA.
2. ultracentrifugação e preparação de gradiente de sacarose
- Prepare duas gradiente soluções de sacarose (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 1 milímetro DTT, 1 x inibidor da protease cocktail), contendo 10% de sacarose ou 50% de sacarose. (Tris-HCl, pH 7,4, pode ser usado em vez de HEPES). Adicione 200 unidades/mL, heparina de inibidor ou 1 mg/mL RNase de acordo com o projeto experimental. Colocar um tubo de se para SW 41 rotor no bloco de marcador e desenhar a linha ao longo do nível superior do bloco. Transferi o tubo em um rack de estável.
- Pegue uma seringa de 10 mL com o dispositivo de estratificação anexado e encha a seringa com solução de sacarose a 10% (preparada como acima). Delicadamente, liberá-lo na parte inferior do tubo se até que ele atinja a marca no tubo.
- Encha outra seringa com 50% de solução de sacarose e insira cuidadosamente o seu dispositivo de camadas através da camada de sacarose 10% para o fundo do tubo. Solte suavemente solução de sacarose a partir do fundo, até que ele atinja a marca no tubo. Feche o tubo com a tampa fornecida.
- Para preparar o gradiente de sacarose, rode o dispositivo de fabricante gradiente ON. Nivele a placa usando a cima ou para baixo botões e pressione DONE. Nivelamento é importante para a linearidade do gradiente.
- Após o nivelamento da placa pressione GRAD para abrir o menu de gradiente. Ir para a lista no menu gradiente e selecione o rotor SW 41 Ti. Em seguida, escolha o gradiente de sacarose desejado da lista de menu usando os botões UP e DOWN . Usode imprensa.
- Coloque o suporte de tubo gradiente na placa do fabricante gradiente. Transferi o tubo no suporte. Até 6 gradientes podem ser preparados ao mesmo tempo. Pressione executar. O fazedor de gradiente gira os tubos na velocidade programada e ângulos formando um gradiente linear. Levará apenas alguns minutos para preparar o gradiente.
- Quando o processo estiver concluído, coloque os tubos em um rack. Tire as tampas. Retire o topo dos tubos se o mesmo volume que o volume da amostra.
- Cuidadosamente carrega 400-500 μL de lisado contendo 15-20260 unidades de polissomos na parte superior. Coloque os tubos em baldes do rotor e equilibrá-los.
- Centrifugue a 260.000 x g e 4 ° C por 2 h, usando o rotor SW 41.
3. polysome fracionamento e coleta de amostra
Nota: Enquanto lisadas preparações têm algumas diferenças dependendo da fonte, gradientes protocolos de fracionamento de preparação e polysome são as mesmas para todos os tipos de lisados.
- Após a conclusão da ultracentrifugação, coloque os baldes de rotor com os tubos em gelo. Vire o coletor de fração e sintonizei gradiente ON. Clique em SCAN no menu fracionadores. Colocar um rack com 24 tubos de colheita para o coletor de fração.
- Encha um reservatório de lavagem do lado o fracionador com água desionizada. Pressione a tecla enxágue durante 10 s para enxaguar a bomba no fracionador. Conecte um adaptador de enxaguar com a seringa cheia de água para o pistão para a calibração.
- Abra o software sintonizei no computador. Imprensa de calibrar. Use as configurações padrão e pressione Okey. Prepare-se injetar água da seringa.
- Pressione Okey para fazer a calibração. Iniciar imediatamente a injetar água para os próximos 5 s. Durante este tempo, água fluirá através da célula de fluxo do detector de UV e o instrumento pode ser calibrado. O sinal ZERO calibração concluída será exibida. O instrumento está pronto para fracionamento.
- Remova o adaptador de enxaguar com a seringa. Coloque uma ponta para o pistão do fracionador.
- Abra o ar da válvula e imprensa ar chave de bronze para 10 s para tubulação seca e célula de fluxo. Feche a válvula de ar.
- Delicadamente, retire o tubo gradiente do balde do rotor e colocá-lo no cesto. Aplicar a tampa do porta tubo até o topo do tubo e cuidadosamente mover o tubo no suporte do tubo e prenda-a na posição.
- Coloque o suporte sob o pistão do fracionador. Muitas vezes, bandas polysomal podem ser vistas a olho nu. Introduzir as configurações desejadas para números de fração e de volume (24 fracções em 500 μL/fração são geralmente suficientes). Nomeie o arquivo adequadamente. Pressione Okeye, em seguida, Vá para o gráfico de botão. Na janela seguinte, pressione Iniciar digitalização. Aparecerão as configurações, pressione Okey. O coletor se moverá da sarjeta para a primeira fração e o pistão se moverá dentro do tubo. Quando o pistão atinge o topo do gradiente reduzirá a velocidade selecionada e as frações serão coletadas. Quando concluído, o pistão move-se fora do tubo gradiente.
- Abra o ar da válvula e imprensa ar chave de bronze sobre o fracionador para recuperar a última fração.
- Mova os tubos de rack no gelo.
- Adicione 2 volumes de reagente de purificação de RNA de cada fração e flash congelam em nitrogênio líquido até a purificação do RNA. Como alternativa, se a proteína precisa ser analisado, adicione ácido tricloroacético para a concentração final de 10% para concentrá-las para a mancha ocidental (ver secção 8).
4. preparação do RNA sintético In Vitro para normalização dos mRNAs níveis durante a análise de dados do RT-qPCR
Nota: A e. coli OmpA mRNA é usada neste protocolo para normalização. Qualquer outro RNA que não tem identidade extensa com os mRNAs do organismo estudado (mamíferos ou Leishmania) pode ser usado.
- Prepare o fragmento de DNA OmpA contendo a sequência do promotor SP6 por uma reação de PCR padrão de um plasmídeo contendo o gene de OmpA30.
- Prepare-se 100 µ l da mistura: 80 mM HEPES-KOH, pH 7,5, 16 mM MgCl2, 2mm espermidina, 10 mM DTT, 3mm ATP, CTP, de 3 mM 3 mM UTP, 3mm GTP, inibidor de RNase U/μL 0.5, 1 μg do DNA de PCR OmpA, 3 μL SP6 RNA polimerase , Pyrophosphatase de U/μL 0.005.
- Incube a 40 ° C por 2 h.
- Purifica o RNA através de um kit de purificação de RNA.
- Medir a concentração por espectrofotômetro e examinar por eletroforese em gel de agarose.
5. RNA isolado do gradiente frações e cDNA preparação
Nota: Proceda diretamente com este protocolo para a purificação de RNA se um inibidor de RNase foi usado como um inibidor do ribonuclease. No entanto, quando usado como um inibidor do ribonuclease, heparina vai inibir a transcriptase reversa, utilizado na preparação de cDNA. Portanto, será necessário purificação adicional do RNA se heparina foi usada na Lise e gradiente. Consulte a seção 6 para preparar RNA para a síntese do cDNA, se heparina foi usada.
- Descongelar as amostras contendo o reagente de purificação de RNA, adicionar 20 ng do RNA sintético como controle interno para a normalização dos resultados de RT-qPCR. Prosseguir com a preparação do RNA de acordo com o protocolo do fabricante exceto uma modificação. Adicione 1 µ l de precipitação de isopropanol prévia do RNA da classe glicogênio (20 µ g). Dissolva pelotas de RNA em 20-25 µ l de água livre de RNase.
Nota: Glicogênio serve como um portador e ajuda a evitar perdas e visualizar a pelota do RNA durante a purificação. OmpA mRNA é usada para normalização ainda mais nas reações de RT-qPCR. - Medir a concentração de RNA usando espectrofotômetro para garantir o rendimento adequado. Combinam-se volumes iguais de frações de RNA contendo 40, 60 e monosomes como prepolysomes. Frações contendo 2-4 ribossomas combinam como polissomos luz e frações com 5-8 ribossomos combinam como polissomos pesados.
- Use 5-10 µ l de RNA das fracções combinadas para preparar os cDNAs usando um kit e seguindo as recomendações do fabricante.
- Adicione 80 µ l de água livre de nuclease a 20 µ l de cDNA. Congelar as amostras de cDNA a-20 ° C.
6. RNA purificação da contaminação de heparina
Nota: Heparina inibe enzimas como a transcriptase reversa de processamento do ácido nucleico. Portanto, use este protocolo de purificação adicional quando a heparina é usada a Lise e/ou gradiente.
- Adicione o LiCl para uma concentração final de 1 M, para as amostras de RNA purificadas.
- Misturar as amostras e incubar no gelo por 1h.
- Gire as amostras a 16.000 x g e 4 ° C por 15 min.
- Remova o sobrenadante tão completo quanto possível, usando uma pipeta.
- Secar ao ar livre as pelotas por cerca de 5 min.
- Re-suspenda as pelotas do volume inicial de água livre de RNase.
- Executar a medição espectrofotométrica em 260 nm para determinar a concentração do RNA. Geralmente, a perda da amostra é mínima.
7. RT-qPCR e análise de dados de distribuição de mRNA
- Combine 10.2 μL de água, 20 μL de SYBR Green, 4.8 μL de primers específicos do gene (conjunto de 2,5 μM cada), 5 μL de cDNA, misture bem e carregar 10 μL por poço no triplica em placa de 384.
- Cubra o prato com película adesiva firmemente e centrifugar a placa a 1.800 x g por 5 min.
- Usar um instrumento de PCR em tempo real configurar qPCR reação sob condições mostrada na tabela 1.
- Usando o limite do ciclo (CT) valores e CT (ΔΔCT) método comparativo31 calcular o percentual (%) de distribuição de mRNA em prepolysomes, luz e polissomos pesados como descrito32 com uma modificação. Use o RNA sintético (OmpA aqui) para a normalização de dados na análise de dados de RT-qPCR. O RNA sintético fornece um controle de normalização que permite calcular os níveis relativos de mRNAs e compará-los em diferentes frações de um gradiente.
8. análise de proteínas em frações Polysomal pela mancha ocidental
- De um estoque de 100% (p/v), adicionar o ácido tricloroacético (TCA) para as frações selecionadas (500 µ l) a uma concentração final de 10%, manter no gelo pelo menos 15 min, centrífuga em uma microcentrífuga por 5 min, descartar o sobrenadante, lave duas vezes com acetona gelada e dissolver em 25 μL de S Amortecedor de carregamento da amostra do DS-página para a electroforese.
- Carregar sobre o SDS-PAGE e conduta electroforese padrão com transferência seguinte à membrana de PVDF. Proceda à mancha33ocidental.
Resultados
Neste estudo, descrevemos a aplicação da técnica de criação de perfil polysomal de três fontes diferentes: o parasita Leishmania major, culturas de células humanas e testículo de rato. Células de Leishmania crescem livremente nos meios líquidos em suspensão, culturas de células humanas crescem em monocamada a aderente em placas, e o testículo de rato representa uma amostra de tecido. O método pode ser facilmente ajustado para outros tipos de células cultivadas livremente em suspensão, diferentes tipos de tecidos, ou de outro organismo e diferentes tipos de células cultivadas. A abordagem consiste em quatro etapas principais: preparação lisada, uma preparação de gradiente de sacarose e ultracentrifugação passo, fracionamento de polysome e coleta de amostra, seguido de análise das frações. Células de diferentes fontes são coletadas, lavadas e lysed em tampão de Lise por passagem através de uma agulha ou homogenizacao homogenizador. Centrifugação é usada para remover os resíduos de célula, esclarecendo o lisado. O esquema de fracionamento gradiente é mostrado na figura 1A. Um gradiente de sacarose contínuo é formado pela mistura de soluções de sacarose 10% e 50% em uma máquina de gradiente. O lisado é carregado no topo do gradiente. Ultracentrifugação separa mRNAs associados com um número diferente de ribossomos, que é monitorado por um detector de UV durante o fracionamento, formando um espectro de absorbância distintas. Frações coletadas são usadas para análise do RNA e proteína (figura 1B). RNA pode ser analisado por eletroforese seguido por Northern blot ou usado para a produção de cDNA, seguida por uma reação de RT-qPCR para analisar a Associação dos mRNAs individuais com polissomos. Próxima geração sequenciamento pode ser usado para analisar o status de translação dos mRNAs em um genoma-larga escala7. Para análise de proteínas de polysomal frações, proteínas são precipitadas com ácido tricloroacético para concentrá-las. As proteínas são então analisadas por Western mancha ou por espectroscopia de massa no nível do proteoma.
Um típico perfil polysomal gerado a partir de Leishmania major crescendo ativamente a cultura é mostrado na Figura 2A. O gráfico de absorbância do fracionamento tem uma forma distinta com picos típicos para o ribossoma subunidades (anos 40 e 60), único ribossomas (80 ou monosomes) e polissomos.
RT-PCR quantitativo (RT-qPCR) foi empregado para detectar Associação dos mRNAs individuais de Leishmania com ribossomas e polissomos. O método comparativo de31 CT (ΔΔCT) é uma abordagem simples e adequada para estudar os mRNAs relativo níveis nas células. Este método requer um controle interno (um mRNA estável, que não muda de expressão durante tratamentos ou condições da experiência) para cálculos. No entanto, não há nenhum controle interno em frações polysomal porque os níveis de mRNA ou RNA ribossómico variará em frações, dependendo de sua associação com ribossomos, polissomos, etc. Para resolver o problema de um controle interno, usamos bacteriana sintética OmpA mRNA para normalização dos níveis do mRNA relativos Leishmania individuais nas fracções. OmpA mRNA foi sintetizado em vitro e adicionados em quantidades iguais de cada fração antes das extrações de RNA. Adição do RNA sintético é importante porque faz cálculos de RT-qPCR dados mais precisos, servindo como controle interno para cálculos pelo método comparativo de CT (ΔΔCT).
Alíquotas das frações gradientes misturavam-se em três grupos: prepolysomes (subunidades e monosomes), luz polissomos (consistindo de ribossomas de 2-4) e pesados polissomos (consistindo de ribossomas de 5-8). RT-qPCR foi a RNA de frações combinadas para analisar a distribuição de mRNA entre essas frações combinadas (Figura 2B). 18s que RNA ribosomal foi usado como um controle. Seus níveis relativas determinadas por RT-qPCR correlacionaram com a distribuição estimada das pequenas subunidades ribossomais (subunidade livre e como parte de monosomes e polissomos) no espectro. Análise de RT-qPCR revelou que os mRNAs individuais testados tem um grau diferente de engajamento na tradução durante a fase logarítmica de crescimento de Leishmania . Tubulina mRNA é preferencialmente associado polissomos pesados, sugerindo a tradução eficiente. Em contraste, Sherp mRNA é encontrado principalmente com prepolysomes e luz polissomos apoiando tradução menos ativa em comparação com a tubulina mRNA.
Expressão de proteínas recombinantes em células cultivadas é uma abordagem experimental importante em diversas categorias de estudos. Aqui, apresentamos um exemplo de criação de perfil polysomal de proteína recombinante mRNAs em outra fonte de exemplo, culturas de células humanas. Células HeLa transitoriamente foram transfectadas com plasmídeo expressando recombinante fibrose cística condutância transmembrana regulador (CFTR)34 ou proteína de doença de Norrie (NDP). Espectros de absorvância de fracionamento de polysome dos dois exames independentes eram muito semelhantes e contidos distintos picos correspondentes (80), monosomes de (anos 40 e 60), subunidades ribossomais e polissomos (Figura 3A). Similaridade no espectro destas experiências ilustra a reprodutibilidade de fracionamento do gradiente. Como nos estudos de Leishmania , a distribuição dos mRNAs foi determinada por RT-qPCR nas fracções que representam prepolysomes, polissomos leves e pesados polissomos (Figura 3B). Detecção de pequena subunidade ribossomal 18S RNA correlacionada com sua distribuição estimada no espectro. NDP mRNAs eram principalmente associados com frações polysomal leves e pesadas, enquanto CFTR mRNAs foram encontrados principalmente em frações prepolysome, sugerindo que o NDP é traduzido mais eficientemente. Enquanto o NDP é uma proteína relativamente pequena, CFTR é uma proteína muito grande (resíduos de aminoácidos de 1480), consistindo de vários domínios, que dobra de forma independente durante a tradução de35. Compromissos mais baixos de CFTR mRNA com polissomos podem refletir a tradução mais lenta que é necessária para cotranslational de dobramento de seus domínios distintos.
Frações de polysome também podem ser usadas para a deteção de proteínas. Deteção de proteínas nas fracções gradientes realizou-se no exemplo de proteínas ribossomais em células HeLa (Figura 4). As proteínas foram concentradas por precipitação com 10% TCA das fracções e borrão ocidental foi usado para detectar a subunidade pequena ribosomal proteína RPS6 e proteína de grande subunidade ribossomal RPL11 (Figura 4, painel superior). Sua distribuição correlacionados com os picos distintos sobre o espectro de absorção. Estes experimentos demonstram claramente que as frações de polysome podem ser usadas para analisar as proteínas neles.
Foram utilizados muitos sacarose de diferentes concentrações gradientes (por exemplo, 7-47%36, 5-50%7, 7-50%6 , 10-50%37, 15-50%8e outros) para fracionamento de polissomos. Aqui, nós comparamos dois gradientes 10-50% e 17-51% (Figura 5). Embora, 17-51% produziu resultados aceitáveis, a separação no gradiente de 10-50% melhor foi geral.
Está bem documentado que os agentes quelantes, como EDTA, perturbem ribossomas e polissomos8,9. Como é mostrado na Figura 6, tratamento de EDTA de lisado antes de carregamento do gradiente leva ao desaparecimento dos picos, correspondentes a monosomes e polissomos, e aumento significativo em subunidades do Ribossoma picos a HeLa. Esta experiência serviu como um controle e demonstrou que os picos observados sem tratamento de EDTA são polissomos e monosomes na verdade ribosomal.
A Figura 7 mostra os resultados do fracionamento do polysome de testículos de rato. O espectro de absorbância tem semelhanças com aqueles de Leishmania de células HeLa: picos distintos de subunidades ribossomais, monosomes e polissomos. Sua forma e distribuição produzem uma aparência de assinatura, que tornam mais fácil identificá-las em diferentes espectros de polysomal. RNAs totais foram purificados de frações e RNAs de frações selecionadas foram analisados por eletroforese em gel de agarose (Figura 7, painel superior). A eletroforese mostra uma distribuição típica de 18S e 28S RNAs ribossomal. Suas bandas nítidas indicam a integridade das amostras. O gel pode ser usado para a deteção de mRNAs individuais por um borrão do Norte seguir ou ele pode ser usado para avaliar a qualidade das amostras antes de novas experiências na análise do RNA ou proteína - os RNAs ribossomal difusa bandas indicam degradação de RNA nas amostras.
Durante nossos estudos, nós usamos o inibidor de RNase e heparina como inibidores de RNase nos gradientes lysates e sacarose. Enquanto ambos proporcionou resultados satisfatórios, o uso de inibidor de RNase era preferível para análise do RNA, porque ele não inibe o cDNA e RT-qPCR reações. Assim, não requerem passos adicionais de purificação de RNA. No entanto, se pesquisadores decidem usar heparina durante a preparação de polysome, esteja ciente que a heparina inibe a jusante aplicações tais como RT-qPCR e etapa de purificação de RNA adicional é necessário (Veja o protocolo seção 6).
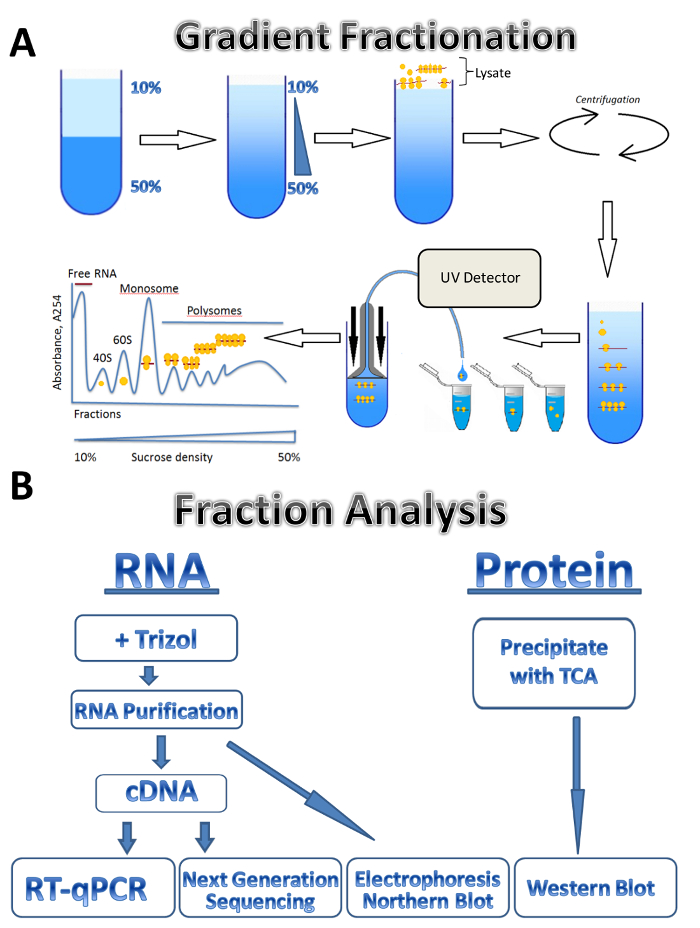
Figura 1 . Criação de perfil polysome. (A) esquema de gradiente preparação, perfil de fracionamento e absorção de polysome. (B) esquema de análise de fração. Clique aqui para ver uma versão maior desta figura.
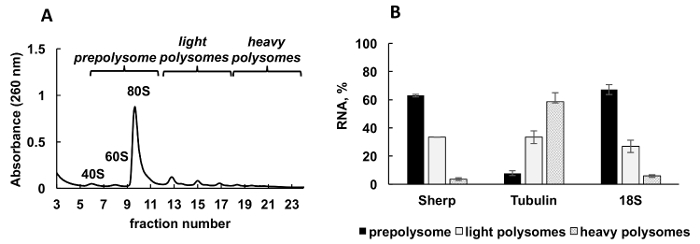
Figura 2 . Análise do perfil de polysome de de Leishmania major a cultura em fase logarítmica de crescimento. (A) citoplasmáticos lisado foi fracionado em gradiente de sacarose 10-50%. (B) distribuição relativa de RNA 18S, tubulina e Sherp mRNAs (%) em prepolysomes, leves e pesados polissomos de log células analisadas por RT-qPCR. Frações que contêm 40, 60 e monosomes foram combinados como prepolysomes. Fracções com ribossomas de 2-4 foram combinadas como polissomos luz, enquanto fracções com 5-8 ribossomos formaram polissomos pesados. Sintético e. coli OmpA mRNA adicionado para extração de RNA frações prévia servida como um controle de normalização em RT-qPCR. Método de31 comparativo CT (ΔΔCT) foi usado para cálculo dos níveis de mRNA. Barras de erro representam erros-padrão. Clique aqui para ver uma versão maior desta figura.
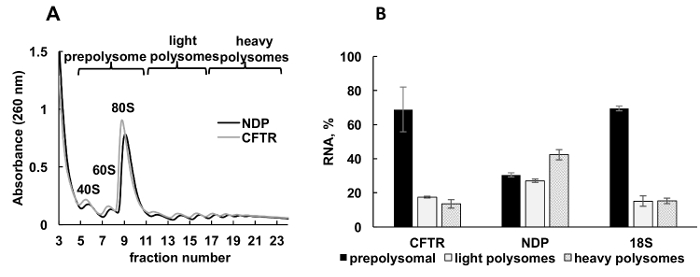
Figura 3 . Fracionamento de polysome e análise de associação de mRNAs CFTR e NDP recombinante com ribossomos nas células HeLa transfected com plasmídeo DNAs. (A) Polysomal perfil em células HeLa transfected com plasmídeos CFTR e NDP. gradiente de sacarose 10% - 50% foi aplicado para obter a separação de polissomos. Os picos para pequeno (40 anos) e grande (60) subunidades, bem como monosome (80) são indicados. Frações foram combinadas como mostrado no painel A e usadas para posterior análise. (B) distribuição de mRNAs de CFTR e NDP em diferentes frações. Deteção de 18S por RT-qPCR foi usada como um controle para fracionamento de polysome. Níveis de RNA foram avaliados por análise de RT-qPCR. Dados foram normalizados usando mRNA sintético. Método de31 comparativo CT (ΔΔCT) foi usado para cálculo dos níveis de mRNA. Barras de erro representam erros-padrão. Clique aqui para ver uma versão maior desta figura.
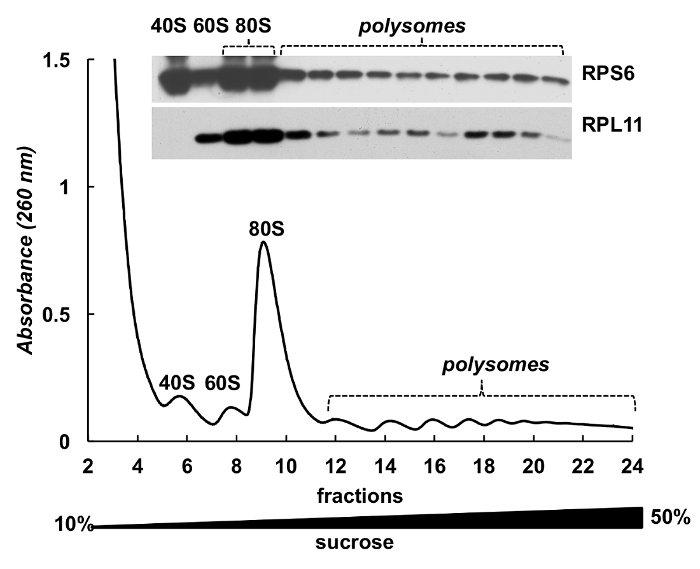
Figura 4 . Deteção de proteínas ribossomais em frações polysomal HeLa. Lisado celular de Hela foi submetido por centrifugação gradiente de 10% - 50% de sacarose. As proteínas em frações selecionadas foram precipitadas com TCA e analisadas por eletroforese em 12% SDS-PAGE com a seguinte mancha ocidental usando mouse monoclonal RPS6 e Anticorpos policlonais de RPL11 coelho como os anticorpos primários e cabra Peroxidase-Conjugated anti-mouse ou anticoelho anticorpos secundários. Visualização de sinais foi feita por SuperSignal West Pico PLUS substrato quimioluminescente. Clique aqui para ver uma versão maior desta figura.
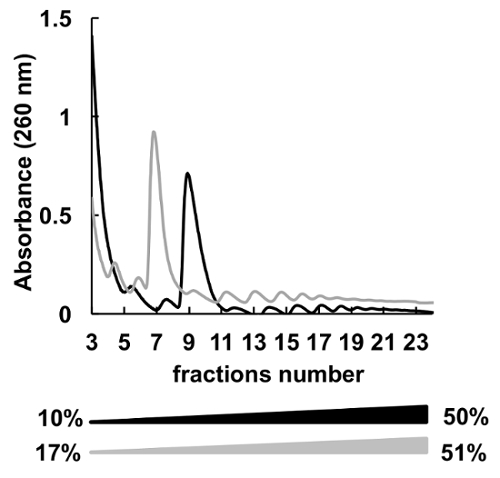
Figura 5 . Comparação de HeLa polysomal de criação de perfil em 10% - 50% (preto) ou gradientes de sacarose 17% - 51% (cinza). Clique aqui para ver uma versão maior desta figura.
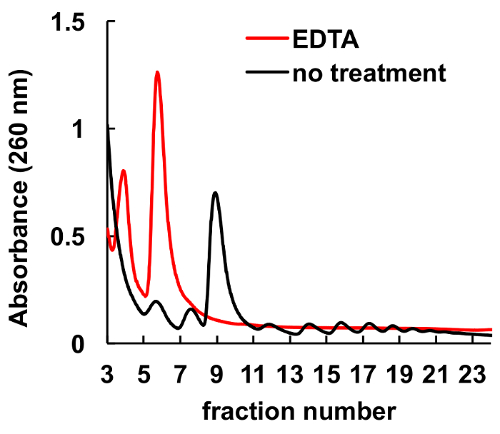
Figura 6 . Efeito do tratamento com EDTA em perfil polysomal em células HeLa. Lisado celular de Hela foi tratado com 10 mM de EDTA no gelo por 10 minutos imediatamente antes da centrifugação gradiente de sacarose. MgCl2 foi substituído por 5 mM EDTA, em soluções de gradiente de sacarose. Clique aqui para ver uma versão maior desta figura.
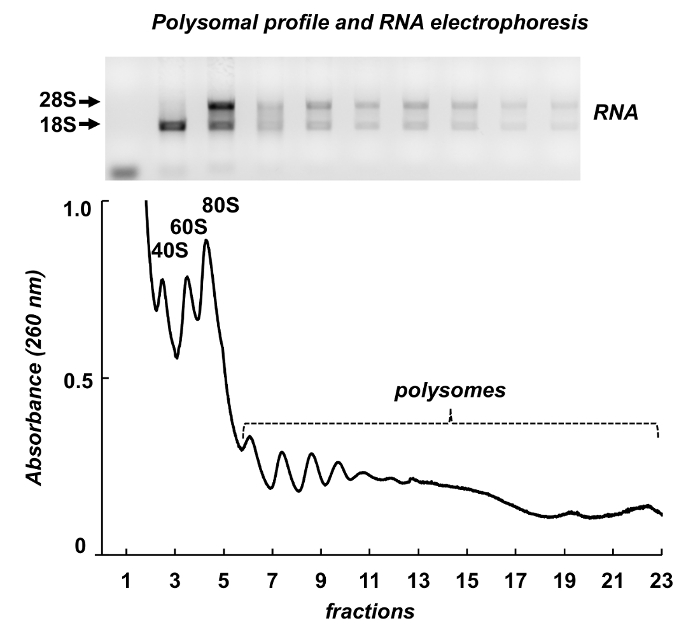
Figura 7 . Perfil polysomal de tecido de testículo de rato lisado. Frações foram submetidas a extração de RNA com um reagente de purificação de RNA e analisadas por eletroforese em gel de agarose 1%. Clique aqui para ver uma versão maior desta figura.
| Estágio | Passo | Condição |
| Segure | Etapa 1 | Aumentar a temperatura de 25 a 50° C, com 1,6 ° C/s |
| Incubar a 50° C para 02:00 min | ||
| Passo 2 | Aumentar a temperatura de 50 a 95° C, com 1,6 ° C/s | |
| Incubar a 95° C para 10:00 min | ||
| PCR | Etapa 1 | Incubar a 95° C por 15:00 min |
| Passo 2 | Diminuir a temperatura de 95 a 60° C, com 1,6 ° C / | |
| Incubar a 60° C para 01:00 min | ||
| Número de ciclos de 40 | ||
| Derreter a curva | Step1 | Aumentar a temperatura de 60 a 95° C, com 1,6 ° C/s |
| Passo 2 | Diminuir a temperatura de 95 a 60° C, com 1,6 ° C/s | |
| Incubar a 60° C para 01:00 min | ||
| Etapa 3 (dissociação) | Aumentar a temperatura de 60 a 95° C, com 0,05 ° C/s | |
| Incubar a 95° C por 15:00 min | ||
Tabela 1. Condições para RT-qPCR
Discussão
Fracionamento de polysome por gradiente de sacarose combinado com RNA e proteína análise de frações é um poderoso método para analisar o status translacional de mRNAs individuais ou a translatome toda, bem como funções de fatores de proteína regulação traducional máquinas durante o estado fisiológico ou doença normal. Perfil polysomal é uma técnica especial apropriada para estudar Regulamento translacional em organismos como tripanossomatídeos incluindo Leishmania onde controle transcricional é em grande parte ausente e regulação da expressão gênica ocorre principalmente durante a tradução.
Aqui, descrevemos um protocolo de fracionamento de polysome usado em três modelos: parasitas Leishmania , culturas de células humanas e tecidos de rato. A etapa de fracionamento de polysome é essencialmente o mesmo para diferentes organismos utilizados neste estudo; no entanto, a preparação de lisado tem algumas diferenças. Células de Leishmania crescem em cultura líquida e é coletado por centrifugação e células são contadas antes da lise para garantir iguais a carregar do gradiente. As células humanas podem ser lavadas e lysed diretamente na placa. O carregamento igual é controlado pela densidade óptica. Tecidos de mouse exigem um homogenizador homogenizacao para lise eficiente enquanto no caso da Leishmania e células humanas, é suficiente para passá-las através da agulha de calibre 23.
Todos os reagentes utilizados devem ser RNase e protease livre. Comparamos o heparina e inibidor de RNase como inibidores da atividade da RNase nos lysates citoplasmáticas. Achamos que ambos os reagentes podem efetivamente bloquear RNase. No entanto, heparina afeta a jusante aplicações tais como a preparação do cDNA e RT-qPCR. Como resultado, a preparação do RNA requer uma etapa de purificação adicional quando a heparina é usada. Em nossa opinião, o inibidor de RNase é a escolha mais conveniente e pode ser usado eficazmente em polysome protocolo de criação de perfil.
Polysomal de criação de perfil é de trabalho intensivo, que é uma grande limitação do método. Até seis gradientes podem ser preparados ao mesmo tempo. O gradiente fracionadores gera 144 fracções que precisam ser processadas em um curto período de tempo. Análise de frações individuais pode ser demorado e caro também. Portanto, combinando frações individuais em pre-polissomos, polissomos leves e pesados fornece uma maneira rápida e menos trabalhosa para estimar a atividade translacional de mRNAs individuais. Nossos resultados de RT-qPCR em frações combinadas nos permitiram identificar diferenças na Traduzibilidade próprios dos mRNAs diferentes tanto em Leishmania e células HeLa (figuras 2, 3). No entanto, se houver necessidade de resolução mais fina, análise de frações individuais pode ser executada.
Ribossoma profiling é outro método para estudar o status translacional de mRNA e baseia-se na medição da produção de proteína através do sequenciamento de fragmentos do mRNA protegido pelo Ribossoma38. Essa tecnologia fornece informações quantitativas associar sequências de mRNA específicas frações polysomal ser traduzidas em uma amostra e pode fornecer informações precisas sobre o status de translação dos mRNAs em resolução de códon em comparação com polysome tecnologia de criação de perfil. No entanto, polysome de criação de perfil pode ser usado para análise tanto do RNA e proteína, assim fornecendo informações adicionais sobre proteome de polissomos e identificar os fatores que contribuem para a regulação da tradução.
Portanto, polysomal de criação de perfil é uma técnica versátil que pode ser usada para analisar o estado translacional de mRNAs individuais, examinar as proteínas associadas a Ribossoma e estudar Regulamento translacional em organismos modelo diferente sob diferentes experimentais condições.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores agradecer Ching Lee ajuda com gravação de áudio. A pesquisa foi apoiada pelos fundos de start-up da Texas Tech University Centro de Ciências da saúde e pelo centro de excelência de neurociência translacional e terapêutica (Miocardite) concede PN-Miocardite 2017-05 AKHRJDHW a A.L.K.; em parte, pela concessão de NIH R01AI099380 K.Z. James C. Huffman e Kristen R. Baca foram estudiosos CISER (centro para a integração da haste Educação & pesquisa) e foram apoiadas pelo programa.
Materiais
| Name | Company | Catalog Number | Comments |
| Instruments: | |||
| Gradient master | Biocomp Instruments Inc. | 108 | |
| Piston Gradient Fractionator | Biocomp Instruments Inc. | 152 | |
| Fraction collector | Gilson, Inc. | FC203B | |
| NanoDrop One | Thermo Scientific | NanoDrop One | |
| Nikon inverted microscope | Nikon | ECLIPSE Ts2-FL/Ts2 | |
| 2720 Thermal Cycler | Applied Biosystems by Life Technologies | 4359659 | |
| CO2 incubator | Panasonic Healthcare Co. | MCO-170A1CUV | |
| HERATHERM incubator | Thermo Scientific | 51028063 | |
| Biological Safety Cabinet, class II, type A2 | NuAire Inc. | NU-543-400 | |
| Revco freezer | Revco Technologies | ULT1386-5-D35 | |
| Beckman L8-M Ultracentifuge | Beckman Coulter | L8M-70 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424 | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Swing-bucket rotor | Eppendorf | A-4-62 | |
| Fixed angle rotor | Eppendorf | F-45-30-11 | |
| Quant Studio 12K Flex Real-Time PCR machine 285880228 | Applied Biosystems by life technologies | 4470661 | |
| TC20 Automated cell counter | Bio-Rad | 145-0102 | |
| Hemacytometer | Hausser Scientific | 02-671-51B | |
| Software | |||
| Triax software | Biocomp Instruments Inc. | ||
| Materials: | |||
| Counting slides, dual chamber for cell counter | Bio-Rad | 145-0011 | |
| 1.5 mL microcentrifuge tube | USA Scientific | 1615-5500 | |
| Open-top polyclear centrifuge tubes, (14 mm x 89 mm) | Seton Scientific | 7030 | |
| Syringe, 5 mL | BD | 309646 | |
| BD Syringe 3 mL23 Gauge 1 Inch Needle | BD | 10020439 | |
| Nunclon Delta Surface plate, 14 cm | Thermo Scientific | 168381 | |
| Nunclon Delta Surface plate, 9 cm | Thermo Scientific | 172931 | |
| Nalgene rapid-flow 90mm filter unit, 500 mL, 0.2 aPES | Thermo Scientific | 569-0020 | |
| BioLite 75 cm3 flasks | Thermo Scientific | 130193 | |
| Nunc 50 mL conical centrifuge tubes | Thermo Scientific | 339653 | |
| Chemicals: | |||
| Trizol LS | Ambion by Life Technologies | 10296028 | |
| HEPES | Fisher Scientific | BP310-500 | |
| Trizma base | Sigma | T1378-5KG | |
| Dulbecco's Modified Eagle's Medium-high glucose (DMEM) | Sigma | D6429-500ML | |
| Fetal Bovine Serum (FBS) | Sigma | F0926-50ML | |
| Penicillin-Streptomycin (P/S) | Sigma | P0781-100ML | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | |
| Dulbecco's phosphate buffered saline (DPBS) | Sigma | D8537-500ML | |
| Magnesium chloride hexahydrate (MgCl2x6H2O) | Acros Organics | AC413415000 | |
| Potassium Chloride (KCl) | Sigma | P9541-500G | |
| Nonidet P 40 (NP-40) | Fluka (Sigma-Aldrich) | 74385 | |
| Recombinant Rnasin Ribonuclease Inhibitor | Promega | N2511 | |
| Heparin sodium salt | Sigma | H3993-1MU | |
| cOmplete Mini EDTA-free protease inhibitors | Roche Diagnostics | 11836170001 | |
| Glycogen | Thermo Scientific | R0551 | |
| Water | Sigma | W4502-1L | |
| Cycloheximide | Sigma | C7698-1G | |
| Chloroform | Fisher Scientific | 194002 | |
| Dithiotreitol (DTT) | Fisher Scientific | BP172-5 | |
| Ethidium Bromide | Fisher Scientific | BP-1302-10 | |
| Ethylenediaminetetraacetic acid disodium dehydrate (EDTA) | Fisher Scientific | S316-212 | |
| Optimem | Life Technologies | 22600050 | |
| Puromycin dihydrochloride | Sigma | P8833-100MG | |
| Sucrose | Fisher Scientific | S5-3KG | |
| Trypsin-EDTA solution | Sigma | T4049-100ML | |
| Hgh Capacity cDNA Reverse Transcriptase Kit | Applied Biosystems by life technologies | 4368814 | |
| Power SYBR Green PCR Master Mix | Applied Biosystems by life technologies | 4367659 | |
| HCl | Fisher Scientific | A144SI-212 | |
| Isopropanol | Fisher Scientific | BP26324 | |
| Potassium Hydroxide (KOH) | Sigma | 221473-500G | |
| Anti-RPL11 antibody | Abcam | ab79352 | |
| Ribosomal protein S6 (C-8) antibody | Santa Cruz Biotechnology Inc. | sc-74459 | |
| 1xM199 | Sigma | M0393-10X1L | |
| Lithium cloride | Sigma | L-9650 | |
| Dimethyl sulfoxide (DMSO) | Fisher Scientific | D128-500 | |
| Gel Loading Buffer II | Thermo Scientific | AM8546G | |
| UltraPure Agarose | Thermo Scientific | 16500-100 | |
| Trichloracetic acid (TCA) | Fisher Scientific | A322-100 | |
| SuperSignal West Pico PLUS chemiluminescent substrate | Thermo Scientific | 34580 | |
| Formaldehyde | Fisher Scientific | BP531-500 | |
| Sodium Dodecyl Sulfate (SDS) | Sigma | L5750-1KG | |
| Phenylmethylsulfonyl fluoride (PMSF) | Sigma | P7626-5G | |
| RNeasy Mini kit | Qiagen | 74104 | |
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A1852-1VL | |
| Cytosine 5'-triphosphate disodium salt hydrate (CTP) | Sigma | C1506-250MG | |
| Uridine 5'-triphosphate trisodium salt hydrate (UTP) | Sigma | U6625-100MG | |
| Guanosine 5'-triphosphate sodium salt hydrate (GTP) | Sigma | G8877-250MG | |
| SP6 RNA Polymerase | NEB | M0207S | |
| Pyrophoshatase | Sigma | I1643-500UN | |
| Spermidine | Sigma | S0266-1G |
Referências
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Capewell, P., et al. Regulation of Trypanosoma brucei Total and Polysomal mRNA during Development within Its Mammalian Host. PLoS One. 8 (6), e67069 (2013).
- Warner, J. R., Knopf, P. M., Rich, A. A multiple ribosomal structure in protein synthesis. Proceedings of the National Academy of Science, USA. 49, 122-129 (1963).
- Piccirillo, C. A., Bjur, E., Topisirovic, I., Sonenberg, N., Larsson, O. Translational control of immune responses: from transcripts to translatomes. Nature Immunology. 15 (6), 503-511 (2014).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of Inhibition of Protein Synthesis in Mammalian Cells. Science. 146 (3650), 1474-1476 (1964).
- Masek, T., Valasek, L., Pospisek, M. Polysome analysis and RNA purification from sucrose gradients. Methods in Molecular Biology. 703, 293-309 (2011).
- Gandin, V., et al. Polysome fractionation and analysis of mammalian translatomes on a genome-wide scale. Journal of Visualized Experiments. (87), (2014).
- Zuccotti, P., Modelska, A. Studying the Translatome with Polysome Profiling. Methods in Molecular Biology. 1358, 59-69 (2016).
- Chasse, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), e15 (2017).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Science, USA. 100 (7), 3889-3894 (2003).
- Gandin, V., et al. nanoCAGE reveals 5' UTR features that define specific modes of translation of functionally related MTOR-sensitive mRNAs. Genome Research. 26 (5), 636-648 (2016).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Zanchin, N. I., Goldfarb, D. S. Nip7p interacts with Nop8p, an essential nucleolar protein required for 60S ribosome biogenesis, and the exosome subunit Rrp43p. Molecular Cell Biology. 19 (2), 1518-1525 (1999).
- Darnell, J. C., et al. FMRP stalls ribosomal translocation on mRNAs linked to synaptic function and autism. Cell. 146 (2), 247-261 (2011).
- Gkogkas, C. G., et al. Autism-related deficits via dysregulated eIF4E-dependent translational control. Nature. 493 (7432), 371-377 (2013).
- Robichaud, N., Sonenberg, N. Translational control and the cancer cell response to stress. Curr Opin Cell Biol. 45, 102-109 (2017).
- Gordon, B. S., Kelleher, A. R., Kimball, S. R. Regulation of muscle protein synthesis and the effects of catabolic states. International Journal of Biochemistry and Cell Biology. 45 (10), 2147-2157 (2013).
- Ishimura, R., et al. RNA function. Ribosome stalling induced by mutation of a CNS-specific tRNA causes neurodegeneration. Science. 345 (6195), 455-459 (2014).
- Petersen, C. P., Bordeleau, M. E., Pelletier, J., Sharp, P. A. Short RNAs repress translation after initiation in mammalian cells. Molecular Cell. 21 (4), 533-542 (2006).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. Proceedings of the National Academy of Science, USA. 111 (1), E203-E212 (2014).
- Bunnik, E. M., et al. Polysome profiling reveals translational control of gene expression in the human malaria parasite Plasmodium falciparum. Genome Biology. 14 (11), R128 (2013).
- De Gaudenzi, J. G., Noe, G., Campo, V. A., Frasch, A. C., Cassola, A. Gene expression regulation in trypanosomatids. Essays in Biochemistry. 51, 31-46 (2011).
- Alves, L. R., Goldenberg, S. RNA-binding proteins related to stress response and differentiation in protozoa. World Journal of Biological Chemistry. 7 (1), 78-87 (2016).
- De Pablos, L. M., Ferreira, T. R., Walrad, P. B. Developmental differentiation in Leishmania lifecycle progression: post-transcriptional control conducts the orchestra. Current Opinions in Microbiology. 34, 82-89 (2016).
- Soto, M., et al. Cell-cycle-dependent translation of histone mRNAs is the key control point for regulation of histone biosynthesis in Leishmania infantum. Biochemical Journal. 379, 617-625 (2004).
- McNicoll, F., et al. Distinct 3 '-untranslated region elements regulate stage-specific mRNA accumulation and translation in Leishmania. Journal of Biological Chemistry. 280 (42), 35238-35246 (2005).
- Folgueira, C., et al. The translational efficiencies of the two Leishmania infantum HSP70 mRNAs, differing in their 3 '-untranslated regions, are affected by shifts in the temperature of growth through different mechanisms. Journal of Biological Chemistry. 280 (42), 35172-35183 (2005).
- Dumas, C., Chow, C., Muller, M., Papadopoulou, B. A novel class of developmentally regulated noncoding RNAs in Leishmania. Eukaryotic Cell. 5 (12), 2033-2046 (2006).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular Cell Biology. 10 (3), 1084-1094 (1990).
- Karamyshev, A. L., Johnson, A. E. Selective SecA association with signal sequences in ribosome-bound nascent chains: a potential role for SecA in ribosome targeting to the bacterial membrane. Journal of Biological Chemistry. 280 (45), 37930-37940 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Panda, A. C., Martindale, J. L., Gorospe, M. Polysome Fractionation to Analyze mRNA Distribution Profiles. Bio Protocols. 7 (3), (2017).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning. A Laboratory Manual. , (1989).
- Patrick, A. E., Karamyshev, A. L., Millen, L., Thomas, P. J. Alteration of CFTR transmembrane span integration by disease-causing mutations. Molecular Biology of the Cell. 22 (23), 4461-4471 (2011).
- Kleizen, B., van Vlijmen, T., de Jonge, H. R., Braakman, I. Folding of CFTR is predominantly cotranslational. Molecular Cell. 20 (2), 277-287 (2005).
- van den Elzen, A. M., Schuller, A., Green, R., Seraphin, B. Dom34-Hbs1 mediated dissociation of inactive 80S ribosomes promotes restart of translation after stress. EMBO Journal. 33 (3), 265-276 (2014).
- Morita, M., et al. mTOR Controls Mitochondrial Dynamics and Cell Survival via MTFP1. Molecular Cell. 67 (6), 922-935 (2017).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados