Method Article
Polysome profilage en Leishmania, les cellules humaines et des testicules de souris
Dans cet article
Résumé
L’objectif global de la technique de profilage polysome est analyse de l’activité traductionnelle de différents mRNAs ou transcriptome ARNm au cours de la synthèse des protéines. La méthode est importante pour les études de règlement de la synthèse de protéine, l’activation de la traduction et répression en santé et les maladies humaines multiples.
Résumé
Expression de la protéine appropriée au bon moment et dans les bonnes quantités est la base de la fonction des cellules normales et de survie dans un environnement en évolution rapide. Pendant longtemps, les études d’expression de gène étaient dominées par la recherche au niveau transcriptionnel. Cependant, les niveaux d’équilibre des ARNm ne concordent pas bien avec la production de protéines et la traductibilité du mRNA varie grandement selon les conditions. Chez certains organismes, comme le parasite Leishmania, l’expression de la protéine est réglementée principalement au niveau traductionnel. Des études récentes ont démontré que le dérèglement de cette protéine traduction est associé avec le cancer, métabolique, neurodégénératives et autres maladies humaines. Polysome profilage est une méthode puissante pour étudier la régulation traduction protéique. Il permet de mesurer l’État translationnelle des ARNm individuels ou examiner la traduction sur une échelle de tout le génome. La base de cette technique est la séparation des polysomes, ribosomes, leurs sous-unités et l’ARNm libre lors de la centrifugation d’un cytoplasme lysat par un gradient de saccharose. Nous présentons ici un polysome universel profilage protocole utilisé sur trois modèles différents - parasite Leishmania majeur, des cellules humaines cultivées et tissus d’origine animales. Leishmania cellules poussent librement en suspension et des cellules humaines cultivées croissent en monocouche adhérente, tandis que des testicules de souris représentent un échantillon de tissus d’origine animale. Ainsi, la technique est adaptée à l’ensemble de ces sources. Le protocole pour l’analyse des fractions polysomique permet la détection des ARNm individuels par RT-qPCR, protéines par Western blot et l’analyse des ARN ribosomiques par électrophorèse. La méthode peut être étendue par examen des mRNAs association avec le ribosome au niveau du transcriptome par profonde RNA-seq et analyse des protéines associées aux ribosomes par spectrométrie de masse des fractions. La méthode peut être facilement ajustée aux autres modèles biologiques.
Introduction
Régulation de l’expression de gène dans les cellules est contrôlée par des mécanismes transcriptionnels, post-transcriptionnel et post-traductionnelle. Les progrès dans le séquençage en profondeur RNA permettent l’étude des niveaux d’ARNm stationnaire sur une échelle de tout le génome à un niveau sans précédent. Cependant, les découvertes récentes ont révélé que d’ARNm stationnaire n’est pas toujours corrélée avec la production de protéine1,2. Le sort d’un relevé de notes individuel est très complex et dépend de nombreux facteurs comme des stimuli internes/externes, stress, etc.. Régulation de l’expression génique au cours de la synthèse des protéines fournit une autre couche de contrôle d’expression nécessaire pour une réponse rapide dans des conditions changeantes. Polysome (ou « polyribosomique »), le profilage, la séparation et la visualisation de traduire activement des ribosomes, est une méthode puissante pour étudier la régulation de la synthèse protéique. Bien que, ses premières applications expérimentales est apparu dans les années 19603, polysome profilage est actuellement une des techniques plus importants en protéines translation studies4. Seul ARNm peut se traduire par plus d’un ribosome conduisant à la formation d’un polysome. Relevés de notes peuvent être calés sur les ribosomes avec cycloheximide5 et ARNm contenant des nombres différents de polysomes se distingue dans le processus de fractionnement polysome de saccharose ultracentrifugation dégradé6,7 , 8 , 9. l’analyse des fractions polysomique RNA puis permet de mesurer des changements dans les États translationnelles de différents mRNAs à l’échelle du génome et au cours de différentes conditions physiologiques4,7, 10. la méthode a été également utilisée pour révéler les rôles de 5' UTR et 3' UTR des séquences dans le contrôle de l’ARNm traduisibilité11, examiner le rôle des miARN dans la répression traductionnelle12, découvrir des défauts dans la biogenèse des ribosomes13 et comprendre le rôle des protéines associées aux ribosomes avec des maladies humaines14,15. Au cours de la dernière décennie, un rôle croissant pour la régulation de l’expression génique au cours de la traduction a émergé qui illustre son importance dans les maladies humaines. Les éléments de preuve pour le contrôle de la traduction dans le cancer, métabolique et maladies neurodégénératives est écrasante15,16,17,18. Par exemple, le dérèglement du contrôle de la traduction eIF4E dépendant contribue à autisme associés déficits15 et FMRP participe au blocage des ribosomes sur mRNA liés à l’autisme,14. Ainsi, polysomique de profilage est un outil très important pour l’étude des défauts de régulation traductionnelle dans plusieurs maladies humaines.
Analyse des protéines des fractions polysomique dans différentes conditions physiologiques dissèque la fonction des facteurs associés aux ribosomes pendant la traduction. La technique de profilage polysome a été utilisée dans de nombreuses espèces, y compris les levures, les cellules de mammifères, plantes et protozoaires10,19,20,21. Parasites protozoaires comme Trypanosoma et Leishmania pièce contrôle transcriptionnel limité de l’expression génique. Leurs génomes sont organisés en groupes de gènes polycistronique qui n’ont pas réglementés promoteur de transcription22. Au lieu de cela, l’expression des gènes du développement est surtout contrôlée au niveau de la traduction des protéines et à la stabilité des ARNm dans trypanosomatide espèce23,24. Compréhension du contrôle de la traduction en l’absence de régulation de la transcription est donc particulièrement importante pour ces organismes. Polysomique de profilage est un outil puissant pour étudier la régulation post-transcriptionnelle de l’expression génique à Leishmania25,26,27,28.
Les récents progrès dans la détection des niveaux d’ARNm individuels par le real time PCR quantitative (RT-qPCR) et pleine transcriptome par séquençage de nouvelle génération, ainsi que technologies de protéomique, apporte la résolution et les avantages du profilage polysomique à un nouveau niveau. L’utilisation de ces méthodes peut être étendue par l’analyse des différentes fractions polysomique par séquençage en RNA profondeur combiné à l’analyse protéomique pour surveiller l’État translationnelle de cellules sur une échelle de tout le génome. Cela permet l’identification de nouveaux acteurs moléculaires régissant la traduction dans différentes conditions physiologiques et pathologiques. Nous présentons ici un polysome universel profilage protocole utilisé sur trois modèles différents : le parasite Leishmania majeur, des cellules humaines cultivées et tissus d’origine animales. Nous présentons des conseils sur la préparation des lysats de cellules de différents organismes, l’optimisation des conditions du gradient, choix des inhibiteurs de la RNase et l’application de la RT-qPCR, Western blot et électrophorèse RNA pour analyser les fractions de polysome dans cette étude.
Protocole
Tous les traitements animales et la manipulation des tissus obtenus dans l’étude ont été réalisés selon les protocoles approuvés par le Comité de l’urbanisme à la Texas Tech University Health Science Center, selon le National Institutes of et d’institutionnels animalier Lignes directrices de santé bien-être animal, numéro de protocole 96005. S’il vous plaît sacrifier des animaux vertébrés et préparer les tissus selon les lignes directrices animalier institutionnel et du Comité de l’emploi. Si la faute d’un tel comité, veuillez consulter les directives de protection des animaux des National Institutes of Health. Adultes (> 60 jours ans) souris C57BL/6 ont été utilisés. Tous les animaux et les tissus ont été obtenus selon les protocoles approuvés par le Comité de l’urbanisme à la Texas Tech University Health Sciences Center selon les directives de protection des animaux des National Institutes of Health et d’institutionnels animalier. Pour l’euthanasie, une seule souris a été placée dans une petite chambre, et l’air a été déplacée graduellement avec environ 30 % du dioxyde de carbone pour anesthésier et minimiser la souffrance de l’animal. Après l’arrêt de la respiration, nous avons utilisé la dislocation cervicale pour confirmer la mort de l’animal avant la récolte de tissus.
ATTENTION : Tous les travaux avec direct Leishmania et des cellules humaines cultivées a été fait en biosécurité armoire BSL-2 laboratoire certifié.
1. préparation des lysats cytoplasmiques de Leishmania grandes , des cellules humaines cultivées et les tissus de souris
Remarque : Il y a plusieurs différences dans les préparations de lysats avec les matériaux de source différente. Autres mesures, y compris la préparation de gradients de sucrose et de fractionnement polysomique sont identiques et ne dépendent pas de la source de l’échantillon.
-
Préparation de lysat cytoplasmique Leishmania majeur
- Ensemencer les cellules Leishmania majeur (souche FV1) dans 30 mL de 1 x M199 moyen29 contenant 10 % de sérum bovin fœtal (SVF) et pénicilline/streptomycine de mélange (100 unités et 100 μg/mL proportionnellement)à une densité de 1 x 105 cellules/mL .
Remarque : Toutes les étapes impliquant des cellules de Leishmania majeur doivent être effectués dans une armoire de sécurité biologique. - Placer les cellules dans un incubateur et cultivez-les à 27 ° C jusqu'à ce que la phase logarithmique (mi journal correspond à 5 x 106 cellules/mL). Il prend habituellement environ deux jours à se développer.
- Ajouter cycloheximide à la culture de Leishmania majeur à une concentration finale de 100 μg/mL d’arrêter les ribosomes sur traduction ARNm. Replacez les cellules dans l’incubateur pendant 10 min à 27 ° C.
- Fin du traitement de cycloheximide, transférer des cellules dans un tube conique de 50 mL et eux tournent à 1 800 x g et 4 ° C pendant 8 min. jetez surnageant.
- Laver les cellules avec 30 mL de solution saline tamponnée au phosphate de Dulbecco (SPD). Centrifuger à 1 800 x g et 4 ° C pendant 8 min.
- Jeter le surnageant. Remettre en suspension les cellules dans 1 mL de SPD.
- Prélever une partie aliquote de cellules et mélangez-le avec la solution de formaldéhyde de 3,5 %.
- Compter les cellules par hémocytomètre et déterminer leur concentration. Transvaser le nombre désiré de cellules dans le tube à centrifuger. Lysate préparé à partir de 0.5x108-2 x 108 cellules/mL est suffisante pour le chargement de gradient d’un saccharose.
- Faire tourner les cellules à 1 800 x g et 4 ° C pendant 8 min. éliminer le surnageant.
- Resuspendre le culot cellulaire sur glace dans 1 mL de tampon de lyse contenant des inhibiteurs de la protéase et inhibiteur de RNase (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 2 mM TNT, 1 % NP-40, 1 x inhibiteur de protéase cocktail (exempt d’EDTA), inhibiteur de RNase 200 unités/mL).
- Passer le lysat par une aiguille de calibre 23 trois fois. Le lysat doit devenir transparent après passage de l’aiguille.
- Centrifugeuse à 11 200 x g et 4 ° C pendant 10 min afin de clarifier le lysat. Transférer le clarifié lysat pour un nouveau tube et le garder sur la glace jusqu’au saccharose gradient ultracentrifugation.
- Frais virés 400-500 μL du lysat en entrée (pour analyser plus tard), congelez-la immédiatement dans l’azote liquide pour analyse des protéines futures ou ajouter réactif de purification d’ARN avant de les congeler pour l’analyse de la RNA.
- Ensemencer les cellules Leishmania majeur (souche FV1) dans 30 mL de 1 x M199 moyen29 contenant 10 % de sérum bovin fœtal (SVF) et pénicilline/streptomycine de mélange (100 unités et 100 μg/mL proportionnellement)à une densité de 1 x 105 cellules/mL .
-
Préparation lysate cytoplasmique des cellules humaines cultivées de HeLa
- Fractionner les cellules HeLa et leur semence dans 20 mL de milieu DMEM contenant 10 % FBS et pénicilline/streptomycine mélange (100 unités et 100 μg/mL proportionnellement) avec cellule compter 2 x 105 cellules/mL dans une plaque de 15 cm.
- La croissance de cellules HeLa à 37 ° C, 5 % de CO2 pour la transfection d’ADN de plasmide 20 à 24 h. effectuer conformément aux protocoles du fabricant.
- Propagez les cellules pendant 24 h après la transfection à 37 ° C, 5 % de CO2.
- Le cycloheximide s’ajoute les cellules HeLa cultivées à la concentration finale de 100 μg/mL d’arrêter les ribosomes sur mRNA traduits et incuber les cellules pendant 10 min à 37 ° C, 5 % de CO2. Aspirez le milieu. Laver les cellules deux fois avec SPD froid sur la glace.
- Ajouter 500 μl de tampon de lyse (20 mM HEPES-KOH pH 7,4, 100 mM KCl, 5 mM MgCl2, 1 mM DTT, 0,5 % NP-40, 1 x inhibiteur de protéase cocktail (EDTA-libre), 200 unités/mL d’héparine de RNase inhibiteur ou 1 mg/mL) à la plaque et raclez les cellules sur la glace.
- Transférer les cellules lysées vers le tube à centrifuger. Ajuster la concentration de NP-40 à 0,5 % et MgCl2 à 5 mM selon l’augmentation du volume de l’échantillon.
- Passer le lysat par une aiguille de calibre 23 3 à 6 fois.
- Tourner à 11 200 x g et 4 ° C pendant 8 min à clarifier lysat. Après centrifugation, transférer le surnageant dans un nouveau tube. Utilisez un spectrophotomètre pour évaluer l’efficacité de la lyse des cellules et pour déterminer la quantité d’échantillon pour le chargement sur le gradient. Ajouter 10 μL d’échantillon à 0,5 mL de 0,1 % Sodium Dodecyl Sulfate (SDS). Vide contre SDS 0,1 %. Mesurer l’absorbance à 260 nm. Valeur d’absorbance attendu est d’environ 15-20 unités/mL.
- Diluer les échantillons avec tampon de lyse de la même valeur d’absorbance avant la centrifugation en gradient de saccharose. Conservez les échantillons sur la glace jusqu'à la centrifugation en gradient de saccharose.
-
Cytoplasmique préparation lysate de testicule de souris
- Disséquer les testicules de souris. Faites une petite incision à l’albuginée et recueillir les tubules séminifères des testicules et de les transférer dans un tube conique 15 mL contenant 5 mL de SPD additionné le fluorure phénylméthylsulfonyle (PMSF) de 0,1 mM.
- Mélanger vigoureusement le tissu en retournant plusieurs fois. Laisser le tissu pour s’établir à la gravité de l’appareil sur la glace pendant 5 min.
- Retirer et jeter le trouble tampon contenant les cellules conjonctives et fragments tissulaires. Répétez l’opération 2 à 3 fois plus. Le granule blanc restant s’enrichit des tubules séminifères et des cellules germinales.
- Transférer le culot tubules séminifères dans un tube de microcentrifuge de 2 mL et essorage à 500 g pendant 1 min. éliminer le surnageant.
- Ajouter 500 µL de tampon de lyse (20 mM Tris-HCl, pH 7,4, 100 mM de KCl, 5 mM MgCl2, 1 mM DTT, 0,5 % NP-40, 1 x inhibiteur de protéase cocktail (exempt d’EDTA), héparine de 1 mg/mL ou 200 unités/mL d’inhibiteur de RNase) pour les tubules. Utiliser une pipette pour triturer les tissus.
- Transférer la suspension dans un petit (0,5 à 1,0 mL) Dounce homogénéisateur. Perturber le tissu avec sept ou huit coups du pilon de verre.
- Transférer le lysat dans un tube de microtubes de 1,5 mL.
- Centrifuger l’échantillon à 12 000 x g et 4 ° C pendant 8 min effacer le lysate. Transférer le surnageant dans un nouveau tube et le magasin sur la glace jusqu’au chargement le long du gradient de saccharose.
- Collecter 50 μL du lysat comme échantillon d’entrée, tout de suite le congeler à-80 ° C pour l’analyse de la protéine future ; ou ajouter le réactif de purification d’ARN avant de les congeler pour l’analyse des RNA.
2. ultracentrifugation et préparation gradients de sucrose
- Préparer deux gradients solutions de saccharose (20 mM HEPES-KOH, pH 7,4, KCl, 10 mM MgCl2, 1 mM TNT, 1 inhibiteur de protéase x cocktail 100 mM), contenant 10 % de saccharose ou sucrose à 50 %. (Tris-HCl, pH 7,4, peut servir au lieu de HEPES). Ajouter 200 unités/mL héparine inhibiteur ou 1 mg/mL de RNase selon le protocole expérimental. Placer un tube d’ultracentrifugeuse pour rotor SW 41 dans le bloc marqueur et tracer la ligne le long de l’étage supérieur du bloc. Transférer le tube dans un support stable.
- Prenez une seringue de 10 mL avec l’appareil de superposition fixée et remplir la seringue avec la solution de 10 % de saccharose (préparée comme indiqué ci-dessus). Doucement Libérez-le en bas du tube ultracentrifugeuse jusqu'à ce qu’il atteigne le repère sur le tube.
- Remplir une autre seringue avec solution de sucrose à 50 % et introduire son dispositif de superposition à travers la couche de saccharose de 10 % jusqu’au fond du tube. Relâchez doucement la solution de saccharose à partir du bas jusqu'à atteindre le repère sur le tube. Fermer le tube avec le capot de protection fourni.
- Pour préparer le gradient de saccharose, éteignez l’appareil de gradient maker ON. Niveau de la plaque à l’aide de la vers le haut ou vers le bas les touches et appuyez sur DONE. Mise à niveau est important pour la linéarité du dégradé.
- Après la mise à niveau la presse plaque GRAD pour ouvrir le menu dégradé. Aller à la liste dans le menu dégradé et sélectionnez le rotor SW 41 Ti. Puis choisissez le gradient de saccharose désiré dans la liste du menu à l’aide des boutons haut et bas . Appuyez sur utilisation.
- Placez le porte-tube dégradé sur la plaque de gradient maker. Transférer le tube dans le support. Jusqu'à 6 dégradés peuvent être préparés en même temps. Appuyez sur exécuter. Le gradient maker fait pivoter les tubes à la vitesse programmée et les angles formant un dégradé linéaire. Il faudra que quelques minutes pour préparer le gradient.
- Lorsque le processus est terminé, placer les tubes dans un rack. Enlever les bouchons. Retirer le même volume que le volume de l’échantillon sur le dessus des tubes ultracentrifugeuse.
- Soigneusement charger 400-500 μL de lysat contenant 15-20260 unités de polysomes sur le dessus. Placer les tubes dans les seaux de rotor et leur équilibre.
- Centrifugeuse à 260 000 x g et 4 ° C pendant 2 h à l’aide de rotor SW 41.
3. polysome fractionnement et prélèvement d’échantillons
NOTE : Tandis que les préparations lysats présentent quelques différences selon les sources, gradient préparation et polysome fractionnement protocoles sont les mêmes pour tous les types de lysats.
- A l’issue de l’ultracentrifugation, placer les seaux du rotor avec les tubes sur la glace. Tourner le collecteur de fraction et gradient fractionnateur ON. Cliquez sur Rechercher dans le menu de fractionnement. Mettre un rack avec 24 tubes de prélèvement dans le collecteur de fraction.
- Remplir un réservoir de rinçage sur le côté le fractionnement à l’eau désionisée. Appuyez sur la touche RINSE pendant 10 s pour rincer la pompe sur le fractionnement. Fixer un adaptateur de rinçage avec la seringue remplie d’eau au piston pour l’étalonnage.
- Ouvrez le logiciel de fractionnement sur l’ordinateur. Appuyez sur calibrer. Utilisez les paramètres par défaut et appuyez sur OK. Être prêt à injecter de l’eau de la seringue.
- Appuyez sur OK pour faire le calibrage. Immédiatement commencer à injecter de l’eau pour le prochain 5 s. Pendant ce temps, l’eau s’écoule à travers la cellule de flux détecteur UV et l’instrument sera calibré. Le symbole Zéro étalonnage terminé s’affiche. L’instrument est prêt pour le fractionnement.
- Retirez l’adaptateur de rinçage avec la seringue. Fixer un embout sur le piston de la fractionnateur.
- Ouvrez la clé en laiton d’air valve et presse AIR pour 10 s à tuyauterie sèche et cellule d’écoulement. Fermez la valve d’air.
- Doucement, retirer le tube dégradé le seau de rotor et placez-le dans le panier. Appliquer le capuchon du tube vers le haut du tube et soigneusement déplacer le tube dans le porte-tube et bloquer en position.
- Placez le support sous le piston de la fractionnateur. Souvent, polysomique bandes peuvent être vu par les yeux. Introduire les paramètres souhaités pour les numéros de fraction et de volume (24 fractions à 500 μL/fraction sont généralement suffisantes). Nommez le fichier de façon appropriée. Appuyez sur OK, puis sur le bouton Aller à graphique . Dans la fenêtre suivante, appuyez sur Démarrer l’analyse. Les paramètres seront affiche, appuyez sur OK. Le collecteur se déplacera de la gouttière à la première fraction et le piston se déplace dans le tube. Lorsque le piston atteint le sommet de la pente, il va ralentir à la vitesse sélectionnée et les fractions seront recueillies. Une fois terminé, le piston se déplace hors du tube de gradient.
- Ouvrez la clé en laiton d’air valve et presse AIR sur le fractionnement pour récupérer la dernière fraction.
- Déplacer les tubes de la crémaillère sur la glace.
- Ajouter 2 volumes de réactif de purification d’ARN à chaque fraction et flash congélation dans l’azote liquide jusqu'à purification RNA. Sinon, si la protéine doit être analysée, ajouter l’acide trichloroacétique à la concentration finale de 10 % pour les concentrer pour Western blotting (voir Section 8).
4. préparation d’ARN synthétique In Vitro pour la normalisation des ARNm niveaux pendant l’analyse de données de RT-qPCR
Remarque : L’ARNm OmpA d’e. coli est utilisée dans le présent protocole pour la normalisation. N’importe quel autre ARN qui n’a pas d’identité vaste avec l’ARNm de l’organisme étudié (mammifères ou Leishmania) peut être utilisé.
- Préparer le fragment d’ADN OmpA contenant la séquence du promoteur SP6 par une réaction de PCR standard d’un plasmide contenant le gène de OmpA30.
- Préparer 100 µL du mélange : 80 mM HEPES-KOH, pH 7.5, 16 mM MgCl2, 2 mM Spermidine, 10 mM DTT, ATP, 3 mM CTP, 3 mM 3 mM UTP, 3 mM GTP, inhibiteur de RNase U/μl 0,5, 1 μg de OmpA PCR ADN, 3 μL SP6 ARN polymérase , 0,005 pyrophosphatase U/μl.
- Incuber à 40 ° C pendant 2 h.
- Purifier les RNA par un kit de purification d’ARN.
- Mesurer la concentration de spectrophotomètre et examiner par électrophorèse sur gel d’agarose.
5. isolement de Fractions dégradé et préparation de cDNA
Remarque : Passez directement avec ce protocole pour la purification de RNA si un inhibiteur de RNase a été utilisé comme un inhibiteur de la ribonucléase. Toutefois, lorsqu’il est utilisé comme un inhibiteur de la ribonucléase, héparine nuira à la transcriptase inverse, utilisée dans la préparation de l’ADNc. Par conséquent, purification supplémentaire de l’ARN est nécessaires si l’héparine a été utilisé dans le tampon de lyse et dégradé. Voir la Section 6 pour préparer l’ARN si l’héparine a été utilisé pour la synthèse de cDNA.
- Décongeler les échantillons contenant le réactif de purification d’ARN, ajouter 20 ng d’ARN synthétique comme contrôle interne pour la normalisation des résultats de la RT-qPCR. Procéder à la préparation d’ARN selon le protocole du fabricant sauf une modification. Ajouter 1 µL de précipitation d’isopropanol préalable ARN grade glycogène (20 µg). Dissoudre les granules de RNA dans 20 à 25 µL d’eau exempte de RNase.
NOTE : Glycogène sert comme support et aide à éviter les pertes et visualiser des granulés de RNA pendant la purification. OmpA ARNm est utilisé pour plus de normalisation dans les réactions de la RT-qPCR. - Mesurer la concentration d’ARN à l’aide du spectrophotomètre pour assurer le rendement adéquat. Mélanger des quantités égales de fractions de RNA contenant 40 s, 60 s et monosomes comme prepolysomes. Fractions contenant des ribosomes 2-4 combinent comme lumière polysomes et fractions de 5-8 ribosomes combinent comme lourds polysomes.
- 5 à 10 µL d’ARN de fractions combinées permet de préparer des ADNc à l’aide d’un kit et suite aux recommandations du fabricant.
- Ajouter 80 µL d’eau libre nucléase à 20 µL de cDNA. Congeler les échantillons de cDNA à-20 ° C.
6. RNA Purification de la Contamination de l’héparine
Remarque : L’héparine inhibe enzymes telles que la transcriptase inverse de la transformation des acides nucléiques. Par conséquent, utiliser ce protocole de purification supplémentaire lorsque l’héparine est utilisée dans le tampon de lyse et/ou dans le gradient.
- Ajouter LiCl à une concentration finale de 1 M pour les échantillons d’ARN purifiés.
- Mélanger les échantillons et incuber sur la glace pendant 1 h.
- Faites tourner les échantillons à 16 000 x g et 4 ° C pendant 15 minutes.
- Retirez le surnageant aussi complet que possible à l’aide d’une pipette.
- Sécher les boulettes pendant environ 5 min.
- Remettre en suspension les boulettes dans le volume initial d’eau exempte de RNase.
- Effectuer une mesure spectrophotométrique à 260 nm pour déterminer la concentration de l’ARN. Habituellement, la perte de l’échantillon est minime.
7. RT-qPCR et analyse de données de Distribution de l’ARNm
- Mélanger 10,2 μL d’eau, 20 μL de SYBR Green, 4.8 μL d’amorces spécifiques de gènes (ensemble de 2,5 μM de chaque), 5 μL d’ADNc, bien mélanger et charger 10 μL / puits dans réanalysés dans plaque 384 puits.
- Couvrez le plat avec film adhésif et centrifuger la plaque à 1 800 g pendant 5 min.
- À l’aide d’un appareil de PCR en temps réel mis en place qPCR réaction selon les modalités indiquées dans le tableau 1.
- En utilisant le cycle seuil (CT) les valeurs et la méthode comparative de la CT (ΔΔCT)31 calculent le pourcentage (%) de répartition des ARNm dans prepolysomes, la lumière et les polysomes lourds comme décrit32 avec une modification. Utiliser l’ARN synthétique (OmpA ici) pour la normalisation des données en analyse de données RT-qPCR. L’ARN synthétique fournit un contrôle de normalisation qui permet de calculer les niveaux de mRNA relative et de les comparer dans les différentes fractions d’un dégradé.
8. l’analyse des protéines dans les Fractions polysomique Western Blot
- Provenant d’un stock de 100 % (p/v), ajouter l’acide trichloracétique (TCA) pour les fractions sélectionnées (500 μL) à une concentration finale de 10 %, rester sur la glace pendant au moins 15 min, centrifuger dans une microcentrifugeuse pendant 5 min, jeter le surnageant, laver deux fois avec de l’acétone glacée et dissoudre dans 25 μL de S DS-PAGE chargement tampon d’électrophorèse.
- Chargez sur le SDS-PAGE et mener l’électrophorèse standard avec transfert suivant à la membrane PVDF. Passez à la Western blotting33.
Résultats
Dans cette étude, nous décrivons l’application de la technique de profilage polysomique à trois sources différentes : parasite Leishmania majeurdes cellules humaines cultivées et des testicules de souris. Leishmania cellules poussent librement dans les milieux liquides en suspension, des cellules humaines cultivées se développent dans la monocouche adhérente sur les plaques et les testicules de souris représentent un échantillon de tissu. La méthode peut être facilement ajustée à d’autres types de cellules librement cultivés en suspension, différents types de tissus ou d’un autre organisme et les différents types de cellules en culture. L’approche se compose de quatre étapes principales : lysate préparation préparation gradients de sucrose et ultracentrifugation étape, fractionnement de polysome et un prélèvement suivi d’analyse des fractions. Cellules provenant de différentes sources sont récoltés, lavés et lysées dans le tampon de lyse par passage à travers une aiguille ou un homogénéisateur Dounce. Centrifugation est utilisée pour supprimer les débris cellulaires, clarifiant le lysate. Le schéma de fractionnement de gradient est montré dans la Figure 1 a. Un gradient continu de saccharose est formé par le mélange des solutions de saccharose de 10 % et 50 % dans un gradient maker. Le lysat est chargé sur le haut de la pente. Ultracentrifugation sépare ARNm associé à un nombre différent de ribosomes qui est surveillé par un détecteur d’UV durant le fractionnement, formant un spectre d’absorbance distinctes. Fractions collectées sont utilisées pour l’analyse de l’ARN et des protéines (Figure 1 b). RNA peut être analysé par électrophorèse suivie par Northern blot ou cDNA production suivie d’une réaction de la RT-qPCR pour analyser l’association entre différents mRNAs et les polysomes. Séquençage de prochaine génération peut être utilisé pour analyser l’État translationnelle des mRNAs sur un génome-large échelle7. Pour l’analyse des protéines des fractions polysomique, les protéines sont précipitées avec l’acide trichloroacétique pour les concentrer. Les protéines sont ensuite analysées par Western blot ou par spectrométrie de masse au niveau du protéome.
Un profil polysomique typique généré de Leishmania majeur en croissance active de la culture est illustré à la Figure 2 a. Le graphique de l’absorbance de fractionnement a une forme distincte avec des pics typiques pour ribosome sous-unités (40 s et 60 s), seul les ribosomes (80 ou monosomes) et les polysomes.
RT-PCR quantitative (RT-qPCR) a été employé pour détecter la liaison des ARNm de Leishmania individuels avec les ribosomes et les polysomes. La méthode comparative de31 CT (ΔΔCT) est une approche simple et appropriée pour étudier les niveaux de mRNA relative dans les cellules. Cette méthode nécessite un contrôle interne (un ARNm stable, qui ne change pas d’expression pendant les traitements ou les conditions de l’expérience) pour les calculs. Cependant, il n’y a aucun contrôle interne dans les fractions polysomique parce que les niveaux de mRNA ou ARN ribosomique variera en fractions, selon leur association aux ribosomes, polysomes, etc.. Pour résoudre le problème d’un contrôle interne, nous avons utilisé la synthèse bactérienne OmpA ARNm à la normalisation de l’ARNm Leishmania individuelle relative dans les fractions. OmpA ARNm a été synthétisée in vitro et ajouté en parts égales à chaque fraction avant les extractions de RNA. Ajout de l’ARN synthétique est important car il effectue des calculs de la RT-qPCR données plus précises, servant de contrôle interne pour les calculs par la méthode comparative de CT (ΔΔCT).
Aliquotes des fractions dégradées étaient partagés en trois groupes : prepolysomes (sous-unités et monosomes), lumière polysomes (composé des ribosomes 2-4) et les polysomes lourds (comprenant les ribosomes 5-8). RT-qPCR déroulée sur RNA de fractions combinées pour analyser la distribution des ARNm entre ces fractions combinées (Figure 2 b). ARN ribosomique 18 s a été utilisé comme témoin. Ses niveaux relatifs, déterminé par la RT-qPCR corrélation avec la répartition estimative de la petite sous-unité ribosomique (sous-unité libre et dans le cadre de monosomes et les polysomes) sur le spectre. RT-qPCR analyse a révélé que différents mRNAs testés par le degré d’engagement dans la traduction au cours de la phase logarithmique de la croissance de Leishmania . Tubuline ARNm est préférentiellement associé à lourdes polysomes, suggérant de traduction efficace. En revanche, personne ARNm est trouvée principalement prepolysomes et lumière polysomes soutenant une traduction moins actif en comparaison avec la tubuline ARNm.
Expression de protéines recombinantes dans les cellules cultivées est une approche expérimentale importante dans diverses catégories d’études. Nous présentons ici un exemple de profilage polysomique des ARNm de la protéine recombinante dans un autre exemple de source, des cellules humaines cultivées. Les cellules HeLa ont été transfectées de manière transitoire avec des plasmides exprimant la mucoviscidose recombinant transmembrane conductance regulator (CFTR)34 ou protéine de la maladie de Norrie (NPD). Spectre d’absorbance de fractionnement polysome de ces deux cultures indépendantes ont été très similaires et confinées pics distincts correspondant sous-unités ribosomiques (40 et 60), monosomes (80) et les polysomes (Figure 3 a). Similitude dans les spectres de ces expériences illustre reproductibilité de fractionnement dégradé. Comme dans les études de la leishmaniose , la distribution des ARNm a été déterminée par RT-qPCR dans les fractions représentant prepolysomes, polysomes légers et lourds polysomes (Figure 3 b). Détection de l’ARN de la petite sous-unité ribosomique 18 s en corrélation avec leur répartition estimée dans les spectres. NPD mRNAs étaient pour la plupart associées à des fractions polysomique légers et lourdes, alors que l’ARNm CFTR était surtout présents dans les fractions prepolysome, ce qui suggère que le NPD est traduit plus efficacement. Tandis que le NPD est une relativement petite protéine, CFTR est une très grosse protéine (résidus d’acides aminés 1480) composé de plusieurs domaines, qui se replient indépendamment au cours de la traduction,35. Des engagements inférieurs de l’ARNm CFTR avec polysomes peuvent refléter la traduction plus lente qui est nécessaire pour plier ses domaines distincts du présent.
Fractions de polysome peuvent être également utilisées pour la détection des protéines. Détection de protéines dans les fractions dégradées a été menée sur l’exemple des protéines ribosomiques dans les cellules HeLa (Figure 4). Les protéines ont été concentrées par précipitation avec 10 % a été utilisé le TCA des fractions et Western blot pour détecter la protéine ribosomique petite sous-unité RPS6 et la protéine ribosomique grande sous-unité RPL11 (Figure 4, panneau supérieur). Leur distribution en corrélation avec les pics distincts sur le spectre d’absorbance. Ces expériences démontrent clairement que les fractions de polysome peuvent être utilisées pour analyser les protéines en eux.
Gradients de concentrations saccharose différents beaucoup (par exemple, 7-47 %36, 5-50 %,7, 7-50 %6 , 10-50 %37, 15-50 %8et autres) pour le fractionnement des polysomes ont été utilisés. Ici, nous avons comparé deux gradients 10 à 50 % et 17 à 51 % (Figure 5). Bien que, 17-51 % produit des résultats acceptables, la séparation dans le gradient de 10 à 50 % a été globalement mieux.
Il est bien établi que des agents de chélation, comme l’EDTA, perturbent les ribosomes et les polysomes8,9. Comme il est indiqué dans la Figure 6, traitement de la HeLa lysat avant chargement le long du gradient conduit à la disparition des pics correspondant aux monosomes et les polysomes, et une augmentation significative dans les sous-unités du ribosome culmine à l’EDTA. Cette expérience a été un contrôle et ont démontré que les pics observés sans traitement à l’EDTA sont polysomes et effectivement ribosomiques monosomes.
La figure 7 illustre les résultats de fractionnement polysome des testicules de souris. Le spectre d’absorbance présente des similitudes avec ceux des cellules Leishmania et HeLa : pics distincts des sous-unités ribosomiques, les monosomes et les polysomes. Leur forme et leur distribution produisent un aspect de signature, qui le rendent facile de les identifier sur différents spectres polysomique. ARN total ont été purifiés de fractions et ARN de fractions sélectionnées ont été analysés par électrophorèse en gel d’agarose (Figure 7, panneau supérieur). L’électrophorèse montre une distribution typique des ARN ribosomique 18 s et 28. Leurs forte bandes indiquent l’intégrité des échantillons. Le gel peut être utilisé pour différents mRNAs détection par un suivant par Northern blot ou il peut être utilisé pour évaluer la qualité des échantillons avant d’autres expériences sur l’ARN ou protéine analyse - l’ARN ribosomal diffuse bandes indiquent la dégradation de l’ARN dans les échantillons.
Au cours de nos études, nous avons utilisé inhibiteur de RNase et héparine comme inhibiteurs de la RNase dans les gradients de lysats et saccharose. Tandis que deux d'entre eux a fourni des résultats satisfaisants, l’utilisation de l’inhibiteur de RNase était préférable pour l’analyse de RNA, car il n’inhibe pas les ADNc et les réactions de la RT-qPCR. Ainsi, il n’exige pas des étapes de purification RNA supplémentaires. Toutefois, si les chercheurs décident d’utiliser l’héparine pendant la préparation de polysome, sachez que l’héparine inhibe en aval des applications comme la RT-qPCR et étape de purification RNA supplémentaire est nécessaire (voir le protocole section 6).
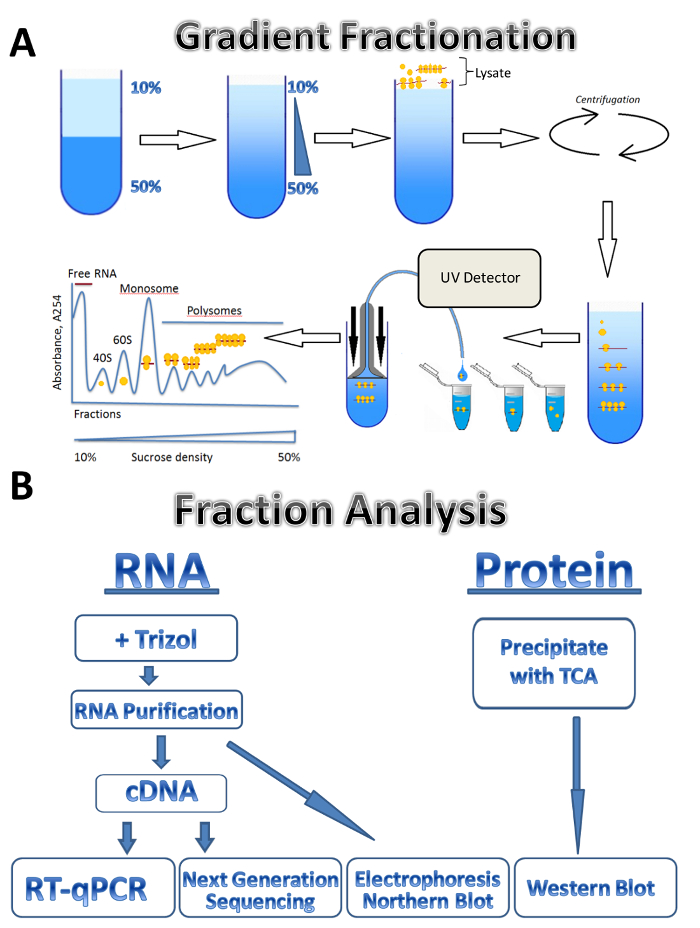
Figure 1 . Profil de polysome. (A) schéma de préparation dégradée, profil de fractionnement et l’absorbance polysome. (B) régime d’analyse de la fraction. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
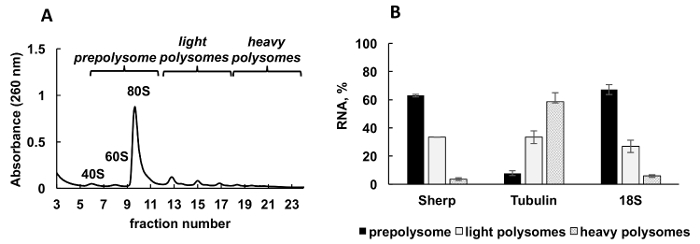
Figure 2 . Analyse du profil de polysome Leishmania majeur culture en phase logarithmique de croissance. (A) cytoplasmiques lysat a été fractionné en gradient de saccharose de 10 à 50 %. (B) la distribution Relative du 18 s RNA, de tubuline et de personne d’ARNm (%) à prepolysomes, polysomes légers et lourds du journal des cellules analysées par RT-qPCR. Fractions contenant 40 s, 60 s et monosomes ont été réunis pour former prepolysomes. Fractions avec 2-4 les ribosomes ont été combinées sous forme de lumière polysomes, alors que fractions avec ribosomes 5-8 forment des polysomes lourds. Synthétique d’e. coli OmpA ARNm ajouté au préalable extraire l’ARN fractions servi de témoin de normalisation dans la RT-qPCR. Comparative CT (ΔΔCT)31 méthode a été utilisée pour le calcul des niveaux d’ARNm. Barres d’erreur représentent des écarts-types. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
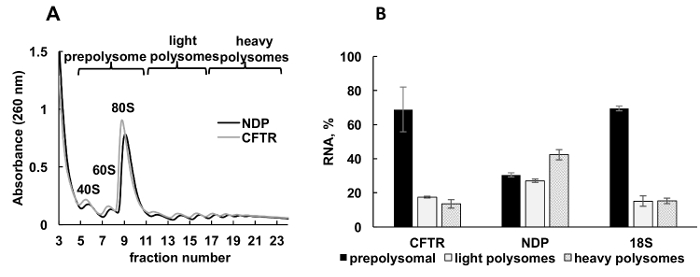
Figure 3 . Polysome fractionnement et l’analyse des recombinante association d’ARNm CFTR et NPD avec ribosomes dans les cellules HeLa transfectées avec plasmide DNAs. (A) profil polysomique dans les cellules HeLa transfectées avec les plasmides CFTR et le NPD. gradient de saccharose de 10 à 50 % a été appliqué pour réaliser la séparation des polysomes. Les pics pour petit (40 ans) et grande (60 s) sous-unités, ainsi que monosome (années 80) sont indiqués. Fractions ont été combinées comme indiqué sur le panneau A et utilisées pour une analyse ultérieure. (B) Distribution des ARNm CFTR et NPD dans différentes fractions. Détection de 18 par RT-qPCR a été utilisée comme contrôle pour fractionnement polysome. Niveaux d’ARN ont été évalués par l’analyse de la RT-qPCR. Données ont été normalisées à l’aide d’ARNm synthétique. Comparative CT (ΔΔCT)31 méthode a été utilisée pour le calcul des niveaux d’ARNm. Barres d’erreur représentent des écarts-types. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
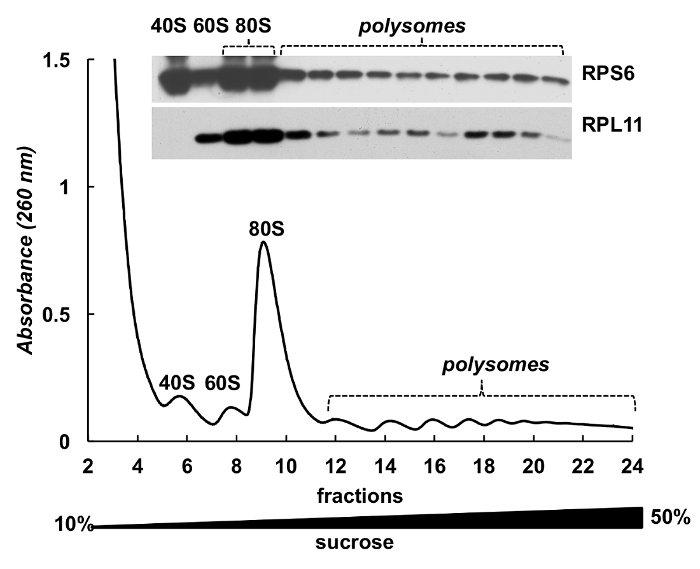
Figure 4 . Détection de protéines ribosomales HeLa fractions polysomique. Hela lysat cellulaire a été soumis par centrifugation en gradient de 10 à 50 % de sucrose. Protéines dans les fractions sélectionnées ont été précipités par TCA et analysés par électrophorèse dans 12 % SDS-PAGE avec la suite Éponger occidental à l’aide de la souris RPS6 monoclonaux et polyclonaux RPL11 anticorps de lapin comme anticorps primaires et Peroxidase-Conjugated chèvre Anticorps secondaires anti-souris ou anti-lapin. Visualisation des signaux a été faite par substrat chimioluminescent SuperSignal West Pico PLUS. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
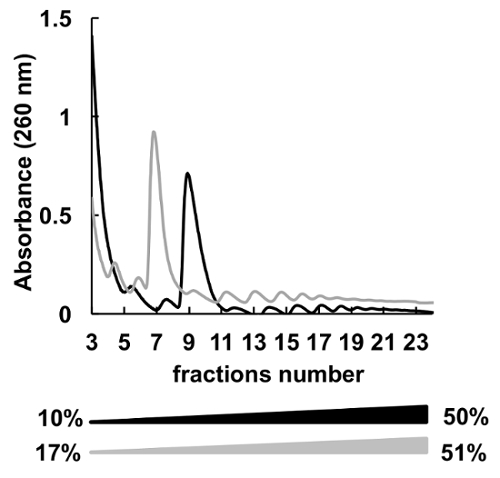
Figure 5 . Comparaison de HeLa polysomique profilage dans 10 à 50 % (noir) ou des gradients de sucrose de 17 à 51 % (gris). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
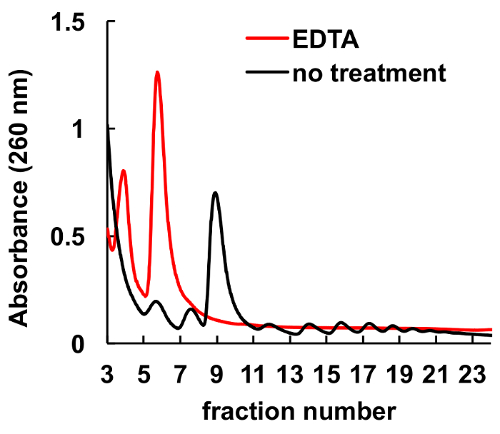
Figure 6 . Effet du traitement à l’EDTA sur Profil polysomique dans les cellules HeLa. Hela lysat cellulaire a été traité avec 10 mM EDTA sur glace pendant 10 minutes juste avant la centrifugation en gradient de saccharose. MgCl2 a été remplacé par 5 mM EDTA dans les solutions de gradient de saccharose. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
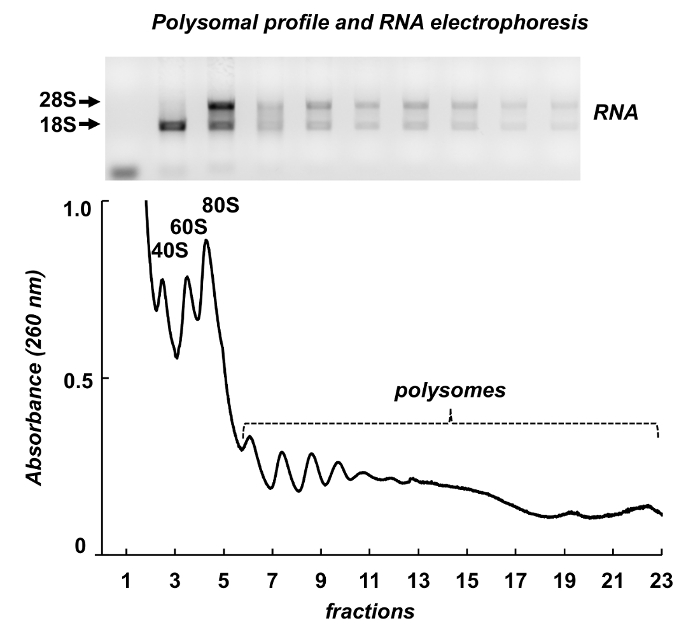
Figure 7 . Profil polysomique de tissu de testicule de souris lysat. Fractions ont été soumises à l’extraction de l’ARN avec un réactif de purification d’ARN et analysées par électrophorèse en gel d’agarose à 1 %. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Stade | Étape | Condition |
| Maintenez | Étape 1 | Augmenter la température de 25 à 50° C avec 1,6 ° C/s |
| Incuber à 50° C, à 02:00 min | ||
| Étape 2 | Augmenter la température de 50 à 95° C, avec 1,6 ° C/s | |
| Incuber à 95° C pour 10:00 min | ||
| PCR | Étape 1 | Incuber à 95° C à 00:15 min |
| Étape 2 | Diminuer la température de 95 à 60° C avec 1,6 ° C / | |
| Incuber à 60° C à 01:00 min | ||
| Nombre de cycles 40 | ||
| Faire fondre la courbe | Etape 1 | Augmenter la température de 60 à 95° C, avec 1,6 ° C/s |
| Étape 2 | Diminuer la température de 95 à 60° C avec 1,6 ° C/s | |
| Incuber à 60° C à 01:00 min | ||
| Étape 3 (dissociation) | Augmenter la température de 60 à 95° C avec 0,05 ° C/s | |
| Incuber à 95° C à 00:15 min | ||
Le tableau 1. Conditions pour la RT-qPCR
Discussion
Fractionnement de polysome par gradient de saccharose combinés avec de l’ARN et analyse des protéines des fractions est une méthode puissante pour analyser translationnelle statut des différents mRNAs ou le translatome entier ainsi que des rôles de facteurs protéiques régulant translationnelle machines au cours de l’état normal, physiologique ou une maladie. Polysomique de profilage est une technique particulièrement adaptée pour étudier la régulation traductionnelle organismes tels que les trypanosomatidés y compris Leishmania où contrôle transcriptionnel est largement absent et régulation de l’expression génique se produit principalement au cours de la traduction.
Nous décrivons ici un protocole de fractionnement polysome utilisé sur trois modèles : parasites Leishmania , des cellules humaines cultivées et des tissus de souris. L’étape de fractionnement polysome est essentiellement la même pour différents organismes utilisés dans la présente étude ; Cependant, la préparation du lysate a quelques différences. Leishmania cellules croissent en milieu liquide et sont recueillis par centrifugation et les cellules sont comptées avant lyse pour égales chargement le long du gradient. Les cellules humaines peuvent être lavés et lysées directement sur la plaque. Le chargement égal est contrôlé par densité optique. Tissus de souris requièrent un homogénéisateur Dounce pour lyse efficace alors que dans le cas de leishmaniose et de cellules humaines, il suffit de les transmettre par l’intermédiaire de l’aiguille de calibre 23.
Tous les réactifs utilisés doivent être RNase et protéase libre. Nous avons comparé l’héparine et l’inhibiteur de RNase en tant qu’inhibiteurs de l’activité de la RNase dans les lysats cytoplasmiques. Nous avons constaté que les deux réactifs peuvent bloquer efficacement la RNase. Toutefois, l’héparine affecte les applications en aval telles que la préparation de l’ARNC et RT-qPCR. En conséquence, préparation d’ARN nécessite une étape de purification supplémentaire lorsque l’héparine est utilisée. À notre avis, l’inhibiteur de RNase est choix plus pratique et peut être utilisé efficacement dans polysome protocole de profilage.
Polysomique de profilage est demande beaucoup de travail, qui est une limitation majeure de la méthode. Jusqu'à six dégradés peuvent être préparés en même temps. Le gradient fractionnateur génère des 144 fractions qui doivent être traités dans un court laps de temps. Analyse des différentes fractions peut être trop fastidieux et coûteux. Par conséquent, combinant différentes fractions dans les polysomes, polysomes légers et lourds fournit une façon rapide et moins laborieuse pour estimer l’activité traductionnelle de différents mRNAs. Nos résultats de la RT-qPCR sur les fractions combinées a permis d’identifier des différences de traduisibilité des différents ARNm en Leishmania et les cellules HeLa (Figures 2, 3). Toutefois, si une résolution plus fine est nécessaire, puis l’analyse des différentes fractions est possible.
Profilage de ribosome est une autre méthode pour étudier l’État translationnelle des ARNm et repose sur la mesure de la production de protéines par séquençage de fragments d’ARNm protégée par ribosome38. Cette technologie fournit des renseignements quantitatifs associant des séquences d’ADN messagère des fractions polysomique spécifique en cours de traduction dans un échantillon et peut fournir des informations précises sur l’État translationnelle des ARNm à résolution de codon en comparaison avec polysome technologie de profilage. Cependant, le polysome profilage peut être utilisé pour l’analyse de l’ARN et de protéine, fournissant ainsi des informations supplémentaires sur le protéome de polysomes et identifier les facteurs qui contribuent à la régulation de la traduction.
Polysomique de profilage est donc une technique polyvalente qui peut être utilisée pour analyser un État translationnel des ARNm individuels, examiner les protéines associées aux ribosomes et d’étudier la régulation traductionnelle chez des organismes différents modèle sous différentes expérimentales conditions.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient Ching Lee pour aide à l’enregistrement audio. La recherche a été soutenue par les fonds de démarrage de la Texas Tech University Health Sciences Center et par le centre d’Excellence pour la Neuroscience translationnelle et thérapeutique (CTNT) accorde le PN-CTNT 2017-05 AKHRJDHW à A.L.K. ; en partie par des subventions des NIH R01AI099380 K.Z. James C. Huffman et Kristen R. Baca étaient des érudits CISER (Centre pour l’intégration des souches éducation et recherche) et appuyés par le programme.
matériels
| Name | Company | Catalog Number | Comments |
| Instruments: | |||
| Gradient master | Biocomp Instruments Inc. | 108 | |
| Piston Gradient Fractionator | Biocomp Instruments Inc. | 152 | |
| Fraction collector | Gilson, Inc. | FC203B | |
| NanoDrop One | Thermo Scientific | NanoDrop One | |
| Nikon inverted microscope | Nikon | ECLIPSE Ts2-FL/Ts2 | |
| 2720 Thermal Cycler | Applied Biosystems by Life Technologies | 4359659 | |
| CO2 incubator | Panasonic Healthcare Co. | MCO-170A1CUV | |
| HERATHERM incubator | Thermo Scientific | 51028063 | |
| Biological Safety Cabinet, class II, type A2 | NuAire Inc. | NU-543-400 | |
| Revco freezer | Revco Technologies | ULT1386-5-D35 | |
| Beckman L8-M Ultracentifuge | Beckman Coulter | L8M-70 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424 | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Swing-bucket rotor | Eppendorf | A-4-62 | |
| Fixed angle rotor | Eppendorf | F-45-30-11 | |
| Quant Studio 12K Flex Real-Time PCR machine 285880228 | Applied Biosystems by life technologies | 4470661 | |
| TC20 Automated cell counter | Bio-Rad | 145-0102 | |
| Hemacytometer | Hausser Scientific | 02-671-51B | |
| Software | |||
| Triax software | Biocomp Instruments Inc. | ||
| Materials: | |||
| Counting slides, dual chamber for cell counter | Bio-Rad | 145-0011 | |
| 1.5 mL microcentrifuge tube | USA Scientific | 1615-5500 | |
| Open-top polyclear centrifuge tubes, (14 mm x 89 mm) | Seton Scientific | 7030 | |
| Syringe, 5 mL | BD | 309646 | |
| BD Syringe 3 mL23 Gauge 1 Inch Needle | BD | 10020439 | |
| Nunclon Delta Surface plate, 14 cm | Thermo Scientific | 168381 | |
| Nunclon Delta Surface plate, 9 cm | Thermo Scientific | 172931 | |
| Nalgene rapid-flow 90mm filter unit, 500 mL, 0.2 aPES | Thermo Scientific | 569-0020 | |
| BioLite 75 cm3 flasks | Thermo Scientific | 130193 | |
| Nunc 50 mL conical centrifuge tubes | Thermo Scientific | 339653 | |
| Chemicals: | |||
| Trizol LS | Ambion by Life Technologies | 10296028 | |
| HEPES | Fisher Scientific | BP310-500 | |
| Trizma base | Sigma | T1378-5KG | |
| Dulbecco's Modified Eagle's Medium-high glucose (DMEM) | Sigma | D6429-500ML | |
| Fetal Bovine Serum (FBS) | Sigma | F0926-50ML | |
| Penicillin-Streptomycin (P/S) | Sigma | P0781-100ML | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | |
| Dulbecco's phosphate buffered saline (DPBS) | Sigma | D8537-500ML | |
| Magnesium chloride hexahydrate (MgCl2x6H2O) | Acros Organics | AC413415000 | |
| Potassium Chloride (KCl) | Sigma | P9541-500G | |
| Nonidet P 40 (NP-40) | Fluka (Sigma-Aldrich) | 74385 | |
| Recombinant Rnasin Ribonuclease Inhibitor | Promega | N2511 | |
| Heparin sodium salt | Sigma | H3993-1MU | |
| cOmplete Mini EDTA-free protease inhibitors | Roche Diagnostics | 11836170001 | |
| Glycogen | Thermo Scientific | R0551 | |
| Water | Sigma | W4502-1L | |
| Cycloheximide | Sigma | C7698-1G | |
| Chloroform | Fisher Scientific | 194002 | |
| Dithiotreitol (DTT) | Fisher Scientific | BP172-5 | |
| Ethidium Bromide | Fisher Scientific | BP-1302-10 | |
| Ethylenediaminetetraacetic acid disodium dehydrate (EDTA) | Fisher Scientific | S316-212 | |
| Optimem | Life Technologies | 22600050 | |
| Puromycin dihydrochloride | Sigma | P8833-100MG | |
| Sucrose | Fisher Scientific | S5-3KG | |
| Trypsin-EDTA solution | Sigma | T4049-100ML | |
| Hgh Capacity cDNA Reverse Transcriptase Kit | Applied Biosystems by life technologies | 4368814 | |
| Power SYBR Green PCR Master Mix | Applied Biosystems by life technologies | 4367659 | |
| HCl | Fisher Scientific | A144SI-212 | |
| Isopropanol | Fisher Scientific | BP26324 | |
| Potassium Hydroxide (KOH) | Sigma | 221473-500G | |
| Anti-RPL11 antibody | Abcam | ab79352 | |
| Ribosomal protein S6 (C-8) antibody | Santa Cruz Biotechnology Inc. | sc-74459 | |
| 1xM199 | Sigma | M0393-10X1L | |
| Lithium cloride | Sigma | L-9650 | |
| Dimethyl sulfoxide (DMSO) | Fisher Scientific | D128-500 | |
| Gel Loading Buffer II | Thermo Scientific | AM8546G | |
| UltraPure Agarose | Thermo Scientific | 16500-100 | |
| Trichloracetic acid (TCA) | Fisher Scientific | A322-100 | |
| SuperSignal West Pico PLUS chemiluminescent substrate | Thermo Scientific | 34580 | |
| Formaldehyde | Fisher Scientific | BP531-500 | |
| Sodium Dodecyl Sulfate (SDS) | Sigma | L5750-1KG | |
| Phenylmethylsulfonyl fluoride (PMSF) | Sigma | P7626-5G | |
| RNeasy Mini kit | Qiagen | 74104 | |
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A1852-1VL | |
| Cytosine 5'-triphosphate disodium salt hydrate (CTP) | Sigma | C1506-250MG | |
| Uridine 5'-triphosphate trisodium salt hydrate (UTP) | Sigma | U6625-100MG | |
| Guanosine 5'-triphosphate sodium salt hydrate (GTP) | Sigma | G8877-250MG | |
| SP6 RNA Polymerase | NEB | M0207S | |
| Pyrophoshatase | Sigma | I1643-500UN | |
| Spermidine | Sigma | S0266-1G |
Références
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Capewell, P., et al. Regulation of Trypanosoma brucei Total and Polysomal mRNA during Development within Its Mammalian Host. PLoS One. 8 (6), e67069 (2013).
- Warner, J. R., Knopf, P. M., Rich, A. A multiple ribosomal structure in protein synthesis. Proceedings of the National Academy of Science, USA. 49, 122-129 (1963).
- Piccirillo, C. A., Bjur, E., Topisirovic, I., Sonenberg, N., Larsson, O. Translational control of immune responses: from transcripts to translatomes. Nature Immunology. 15 (6), 503-511 (2014).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of Inhibition of Protein Synthesis in Mammalian Cells. Science. 146 (3650), 1474-1476 (1964).
- Masek, T., Valasek, L., Pospisek, M. Polysome analysis and RNA purification from sucrose gradients. Methods in Molecular Biology. 703, 293-309 (2011).
- Gandin, V., et al. Polysome fractionation and analysis of mammalian translatomes on a genome-wide scale. Journal of Visualized Experiments. (87), (2014).
- Zuccotti, P., Modelska, A. Studying the Translatome with Polysome Profiling. Methods in Molecular Biology. 1358, 59-69 (2016).
- Chasse, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), e15 (2017).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Science, USA. 100 (7), 3889-3894 (2003).
- Gandin, V., et al. nanoCAGE reveals 5' UTR features that define specific modes of translation of functionally related MTOR-sensitive mRNAs. Genome Research. 26 (5), 636-648 (2016).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Zanchin, N. I., Goldfarb, D. S. Nip7p interacts with Nop8p, an essential nucleolar protein required for 60S ribosome biogenesis, and the exosome subunit Rrp43p. Molecular Cell Biology. 19 (2), 1518-1525 (1999).
- Darnell, J. C., et al. FMRP stalls ribosomal translocation on mRNAs linked to synaptic function and autism. Cell. 146 (2), 247-261 (2011).
- Gkogkas, C. G., et al. Autism-related deficits via dysregulated eIF4E-dependent translational control. Nature. 493 (7432), 371-377 (2013).
- Robichaud, N., Sonenberg, N. Translational control and the cancer cell response to stress. Curr Opin Cell Biol. 45, 102-109 (2017).
- Gordon, B. S., Kelleher, A. R., Kimball, S. R. Regulation of muscle protein synthesis and the effects of catabolic states. International Journal of Biochemistry and Cell Biology. 45 (10), 2147-2157 (2013).
- Ishimura, R., et al. RNA function. Ribosome stalling induced by mutation of a CNS-specific tRNA causes neurodegeneration. Science. 345 (6195), 455-459 (2014).
- Petersen, C. P., Bordeleau, M. E., Pelletier, J., Sharp, P. A. Short RNAs repress translation after initiation in mammalian cells. Molecular Cell. 21 (4), 533-542 (2006).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. Proceedings of the National Academy of Science, USA. 111 (1), E203-E212 (2014).
- Bunnik, E. M., et al. Polysome profiling reveals translational control of gene expression in the human malaria parasite Plasmodium falciparum. Genome Biology. 14 (11), R128 (2013).
- De Gaudenzi, J. G., Noe, G., Campo, V. A., Frasch, A. C., Cassola, A. Gene expression regulation in trypanosomatids. Essays in Biochemistry. 51, 31-46 (2011).
- Alves, L. R., Goldenberg, S. RNA-binding proteins related to stress response and differentiation in protozoa. World Journal of Biological Chemistry. 7 (1), 78-87 (2016).
- De Pablos, L. M., Ferreira, T. R., Walrad, P. B. Developmental differentiation in Leishmania lifecycle progression: post-transcriptional control conducts the orchestra. Current Opinions in Microbiology. 34, 82-89 (2016).
- Soto, M., et al. Cell-cycle-dependent translation of histone mRNAs is the key control point for regulation of histone biosynthesis in Leishmania infantum. Biochemical Journal. 379, 617-625 (2004).
- McNicoll, F., et al. Distinct 3 '-untranslated region elements regulate stage-specific mRNA accumulation and translation in Leishmania. Journal of Biological Chemistry. 280 (42), 35238-35246 (2005).
- Folgueira, C., et al. The translational efficiencies of the two Leishmania infantum HSP70 mRNAs, differing in their 3 '-untranslated regions, are affected by shifts in the temperature of growth through different mechanisms. Journal of Biological Chemistry. 280 (42), 35172-35183 (2005).
- Dumas, C., Chow, C., Muller, M., Papadopoulou, B. A novel class of developmentally regulated noncoding RNAs in Leishmania. Eukaryotic Cell. 5 (12), 2033-2046 (2006).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular Cell Biology. 10 (3), 1084-1094 (1990).
- Karamyshev, A. L., Johnson, A. E. Selective SecA association with signal sequences in ribosome-bound nascent chains: a potential role for SecA in ribosome targeting to the bacterial membrane. Journal of Biological Chemistry. 280 (45), 37930-37940 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Panda, A. C., Martindale, J. L., Gorospe, M. Polysome Fractionation to Analyze mRNA Distribution Profiles. Bio Protocols. 7 (3), (2017).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning. A Laboratory Manual. , (1989).
- Patrick, A. E., Karamyshev, A. L., Millen, L., Thomas, P. J. Alteration of CFTR transmembrane span integration by disease-causing mutations. Molecular Biology of the Cell. 22 (23), 4461-4471 (2011).
- Kleizen, B., van Vlijmen, T., de Jonge, H. R., Braakman, I. Folding of CFTR is predominantly cotranslational. Molecular Cell. 20 (2), 277-287 (2005).
- van den Elzen, A. M., Schuller, A., Green, R., Seraphin, B. Dom34-Hbs1 mediated dissociation of inactive 80S ribosomes promotes restart of translation after stress. EMBO Journal. 33 (3), 265-276 (2014).
- Morita, M., et al. mTOR Controls Mitochondrial Dynamics and Cell Survival via MTFP1. Molecular Cell. 67 (6), 922-935 (2017).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon