Method Article
Polysome perfilado en Leishmania, las células humanas y testículo de ratón
En este artículo
Resumen
El objetivo general de la técnica de generación de perfiles polysome es análisis de actividad traslacional de mRNAs individuales o transcriptoma mRNAs durante la síntesis de proteínas. El método es importante para los estudios de regulación de la síntesis de proteínas, activación de la traducción y represión en salud y múltiples enfermedades humanas.
Resumen
Expresión de la proteína adecuada en el momento adecuado y en las cantidades adecuadas es la base de la función normal de la célula y la supervivencia en un entorno rápidamente cambiante. Durante mucho tiempo, los estudios de expresión génica fueron dominados por la investigación a nivel transcripcional. Sin embargo, los niveles de estado estacionario de mRNAs no se correlacionan bien con la producción de proteínas, y la traducción de los mRNAs varía grandemente dependiendo de las condiciones. En algunos organismos, como el parásito Leishmania, la expresión de proteínas está regulada principalmente a nivel traslacional. Estudios recientes demostraron que dysregulation de traducción de proteínas se asocia con cáncer, metabólico, neurodegenerativas y otras enfermedades humanas. Polysome perfilado es un poderoso método para estudiar la regulación de la traducción de proteínas. Permite medir el estado traslacional de mRNAs individuales o estudiar traducción en una escala genoma-ancha. La base de esta técnica es la separación de polisomas, ribosomas, sus subunidades y mRNAs gratis durante la centrifugación de un citoplásmico lisado a través de un gradiente de sacarosa. Aquí, presentamos un universal polysome perfiles protocolo utilizado en tres modelos diferentes - parásito Leishmania major, células humanas cultivadas y tejidos animales. Las células de Leishmania crecen libremente en suspensión y células humanas cultivadas crecen en monocapa adherente, mientras que el testículo del ratón representa una muestra de tejido animal. Así, la técnica se adapta a todas estas fuentes. El protocolo para el análisis de fracciones polysomal incluye la detección de los niveles de mRNA por RT-qPCR, proteínas por Western blot y análisis de ARN ribosómico mediante electroforesis. El método puede ampliarse aún más por la examinación de mRNAs de asociación con el ribosoma a nivel de transcriptoma por RNA-seq profunda y análisis de proteínas ribosoma asociados por espectroscopía de masas de las fracciones. El método se puede ajustar fácilmente a otros modelos biológicos.
Introducción
Regulación de la expresión génica en las células es controlada por mecanismos transcripcionales y postranscripcional y postraduccionales. Avances en la secuenciación del RNA profunda permiten el estudio de los niveles de mRNA de estado estacionario en una escala genoma-ancha en un nivel sin precedentes. Sin embargo, hallazgos recientes revelaron que nivel de mRNA de estado estacionario no se correlaciona siempre con proteína producción1,2. El destino de una transcripción individual es muy complejo y depende de muchos factores como estímulos internos/externos, estrés, etcetera. Regulación de la expresión génica durante la síntesis de proteína proporciona otra capa de control de expresión necesaria para una respuesta rápida en condiciones cambiantes. Polysome (o "polyribosome") de perfiles, la separación y visualización de la traducción activa de ribosomas, es un poderoso método para estudiar la regulación de la síntesis de proteínas. Aunque sus primeras aplicaciones experimentales aparecieron en la década de 19603, polysome perfilado es actualmente una de las técnicas más importantes en la proteína traducción estudios4. MRNAs individuales puede ser traducida en más de un ribosoma a la formación de un polysome. Las transcripciones pueden ser estancadas en los ribosomas con cicloheximida5 y mRNAs que contienen diferentes cantidades de polisomas pueden ser separados en el proceso de fraccionamiento polysome por ultracentrifugación gradiente de sacarosa6,7 , 8 , 9. Análisis de ARN de fracciones polysomal entonces permite la medición de cambios en los Estados traslacionales de mRNAs individuales a escala de todo el genoma y en diferentes condiciones fisiológicas4,7, 10. el método se ha utilizado también para revelar el papel de la 5' UTR y 3' UTR secuencias en el control del ARNm traducción11, examinar el papel de miRNAs en represión traduccional12, descubrir defectos en biogénesis del ribosome13 y entender el papel de las proteínas del ribosoma asociados con enfermedades humanas14,15. Durante la última década, ha surgido un creciente papel para la regulación de la expresión génica durante la traducción que ilustra su importancia en enfermedades humanas. Las pruebas de control traduccional en cáncer, metabólico y enfermedades neurodegenerativas son abrumador15,16,17,18. Por ejemplo, dysregulation de eIF4E-dependiente control traduccional contribuye al autismo relacionadas con déficits15 y FMRP participa en estancamiento de los ribosomas en mRNAs vinculados al autismo14. Así, polysomal de perfiles es una herramienta muy importante para el estudio de defectos en la regulación traduccional en múltiples enfermedades humanas.
Análisis de la proteína de las fracciones polysomal bajo diferentes condiciones fisiológicas analiza la función de los factores asociados a los ribosomas durante la traducción. La técnica de generación de perfiles polysome se ha utilizado en muchas especies incluyendo levaduras, células de mamíferos, plantas y protozoos10,19,20,21. Parásitos protozoos como Trypanosoma y Leishmania presentan limitado control transcripcional de la expresión génica. Sus genomas se organizan en grupos de gen policistrónico que carecen de transcripción regulada por el promotor22. En cambio, expresión génica desarrollo predominante está controlada a nivel de la proteína traducción y estabilidad del mRNA en tripanosomatídeos especies23,24. Por lo tanto, la comprensión de control traduccional en la ausencia de regulación transcripcional es particularmente importante para estos organismos. Perfiles de polysomal es una poderosa herramienta para estudiar la regulación postranscripcional de la expresión génica en Leishmania25,26,27,28.
Los recientes avances en la detección de niveles de los mRNAs por real tiempo cuantitativo PCR (RT-qPCR) y transcriptoma completo de secuenciación de próxima generación, así como tecnologías de proteómica, trae ventajas de perfiles polysomal a un nuevo nivel y resolución. El uso de estos métodos puede ampliarse aún más por el análisis de fracciones individuales de polysomal por la secuencia de RNA profunda combinada con análisis proteómico para supervisar el estado traslacional de las células en una escala genoma-ancha. Esto permite la identificación de nuevos actores moleculares regular traducción bajo diferentes condiciones fisiológicas y patológicas. Aquí, presentamos un universal polysome perfiles de protocolo que se utiliza en tres modelos diferentes: el parásito Leishmania major, células humanas cultivadas y tejidos animales. Presentamos consejos sobre la preparación de lisados de células de diferentes organismos, optimización de condiciones de gradiente, de inhibidores de la Rnasa y aplicación de RT-qPCR, Western blot y electroforesis de RNA para analizar fracciones polysome en este estudio.
Protocolo
Todos los tratamientos de animales y manejo de los tejidos obtenidos en el estudio fueron realizados según protocolos de actuación aprobados por el cuidado de Animal institucional y Comité de uso en la Texas Tech University Health Science Center, según los institutos nacionales de Pautas de bienestar animal salud, número de protocolo 96005. Por favor, sacrificar animales vertebrados y preparar los tejidos según las directrices del Comité de uso y cuidado de Animal institucional. Si carece de dicho Comité, por favor consulte las pautas de bienestar animal de institutos nacionales de salud. Adultos (> 60 días de edad) fueron utilizados ratones C57BL/6. Todos los animales y los tejidos fueron obtenidos según protocolos aprobados por el cuidado institucional de animales y uso en la Texas Tech University Health Sciences Center según las pautas de bienestar animal de institutos nacionales de salud. Eutanasia, un solo ratón fue colocado en un compartimiento pequeño, y el aire fue desplazado poco a poco con cerca de 30% dióxido de carbono para anestesiar y minimizar el sufrimiento del animal. Tras el cese de la respiración, utilizamos la dislocación cervical para confirmar la muerte del animal antes de la cosecha de los tejidos.
PRECAUCIÓN: Todo el trabajo con células humanas cultivadas y vivo Leishmania se realizó en gabinete en BSL-2 laboratorio certificado de bioseguridad.
1. preparación de lisados citoplásmicos de Leishmania principales , las células humanas cultivadas y tejidos de ratón
Nota: Hay varias diferencias en los preparativos del lisado de los diferentes materiales. Otras medidas, incluyendo la preparación de gradiente de sacarosa y fraccionamiento polysomal son idénticos y no dependen de la fuente de la muestra.
-
Preparación de lisado citoplásmico comandante de Leishmania
- Inocular de Leishmania major (FV1 cepa) las células en 30 mL de 1 x M199 media29 que contiene la mezcla 10% de suero bovino Fetal (FBS) y penicilina/estreptomicina (100 unidades y 100 μg/mL respectivamente)a densidad de 1 x 105 células/mL .
Nota: Se realizarán todos los pasos que involucran células comandante de Leishmania en un gabinete de bioseguridad. - Colocar las células en la incubadora y cultivarlas a 27 ° C hasta la fase logarítmica (mediados de registro corresponde a 5 x 106 células/mL). Toma generalmente cerca de dos días para crecer.
- Añadir cicloheximida al cultivo de Leishmania major a una concentración final de 100 μg/mL para arrestar a los ribosomas en mRNAs traducido. Vuelva a colocar las células en la incubadora durante 10 minutos a 27 ° C.
- Después de completa el tratamiento de cicloheximida, transferir las células a un tubo cónico de 50 mL y spin en 1.800 x g y 4 ° C durante 8 min descartar sobrenadante.
- Lavar las células con 30 mL de solución salina amortiguada de fosfatos de Dulbecco (DPBS). Centrífuga a 1.800 x g y 4 ° C durante 8 minutos.
- Deseche el sobrenadante. Resuspender las células en 1 mL de DPBS.
- Tomar una alícuota de células y mezclar con una solución de formaldehído de 3.5%.
- Contar celdas por hemocitómetro y determinar su concentración. Transferir el número de células en el tubo de microcentrífuga. Lisado de 0.5x108-2 x 108 células/mL es suficiente para la carga de gradiente de un sacarosa.
- Girar las células a 1.800 x g y 4 ° C durante 8 minutos descarte el sobrenadante.
- Resuspender el precipitado de células en hielo en 1 mL de tampón de lisis que contienen inhibidores de la proteasa e inhibidor de Rnasa (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 2 mM DTT, 1% NP-40, 1 x inhibidor de la proteasa cóctel (EDTA-libre), inhibidor de Rnasa de 200 unidades/mL).
- Pasar el lisado a través de una aguja de calibre 23 tres veces. El lisado debería ser transparente después de paso a través de la aguja.
- Centrífuga a 11.200 x g y 4 ° C por 10 minutos aclarar lisado. Transfiera las aclarado lisado a un nuevo tubo y mantenerlo en hielo hasta ultracentrifugación gradiente de sacarosa.
- Recoge 400-500 μL del lisado como entrada (para analizar más adelante), congelar inmediatamente en nitrógeno líquido para análisis de la futura proteína o añadir Reactivo de purificación de RNA antes de congelar para análisis de ARN.
- Inocular de Leishmania major (FV1 cepa) las células en 30 mL de 1 x M199 media29 que contiene la mezcla 10% de suero bovino Fetal (FBS) y penicilina/estreptomicina (100 unidades y 100 μg/mL respectivamente)a densidad de 1 x 105 células/mL .
-
Preparación lisado citoplásmica de las células humanas cultivadas del HeLa
- Las células HeLa y semilla en 20 mL de medio DMEM que contiene la mezcla 10% de FBS y penicilina/estreptomicina (100 unidades y 100 μg/mL respectivamente) con célula cuenta 2 x 105 células/mL en una placa de 15 cm.
- Crecen las células HeLa a 37 ° C, 5% CO2 para la transfección de DNA de plásmido 20-24 h. realizar según los protocolos del fabricante.
- Se propagan las células durante 24 h después de transfección a 37 ° C, 5% CO2.
- Añadir cicloheximida cultiva células HeLa a la concentración final de 100 μg/mL a arrestar a los ribosomas en mRNAs traducido e incubar las células durante 10 min a 37 ° C, 5% CO2. Aspire el medio. Lavan las células dos veces con DPBS frío en hielo.
- Añadir 500 μL de tampón de lisis (20 mM HEPES-KOH pH 7.4, 100 mM KCl, 5 mM MgCl2, 1 mM TDT, 0,5% NP-40, 1 x inhibidor de la proteasa cóctel (EDTA-libre), 200 unidades/mL de heparina de Rnasa inhibidor o 1 mg/mL) a la placa y raspe las células en hielo.
- Transferencia de las células sometidas a lisis para el tubo de microcentrífuga. Ajustar la concentración de NP-40 para 0,5% y MgCl2 a 5 mM según el aumento del volumen de la muestra.
- Pasar el lisado a través de una aguja de calibre 23 3 - 6 veces.
- La vuelta a 11.200 x g y 4 ° C durante 8 minutos aclarar lisado. Después de la centrifugación, transferir el sobrenadante a un tubo nuevo. Use un espectrofotómetro para evaluar la eficiencia de la lisis de la célula y para determinar la cantidad de muestra para el cargamento en el gradiente. Añadir 10 μL de muestra a 0,5 mL de 0.1% sodio dodecil sulfato (SDS). En blanco contra 0,1% SDS. Medir la absorbancia a 260 nm. Valor de la absorbancia esperada es alrededor de 15-20 unidades/mL.
- Diluir todas las muestras con tampón de lisis en el mismo valor de la absorbancia antes de centrifugación gradiente de sacarosa. Mantener las muestras en hielo hasta centrifugación gradiente de sacarosa.
-
Preparación de lisado citoplásmico del testículo de ratón
- Diseccionar el testículo de ratón. Hacer una pequeña incisión en la túnica albugínea y recoger los túbulos seminíferos de los testículos y transferirlos en un tubo cónico de 15 mL que contiene 5 mL de DPBS complementado con fluoruro de phenylmethylsulfonyl de 0,1 mM (PMSF).
- Mezcle vigorosamente el tejido por inversión varias veces. Permitir que el tejido para instalarse en la gravedad de la unidad en hielo durante 5 minutos.
- Retire y deseche el nublado tampón que contiene células conectivas y fragmentos de tejido. Repita el procedimiento 2-3 veces más. La pelotilla blanca restante se enriquece de los túbulos seminíferos y células de germen.
- Transfiera el sedimento de túbulos seminíferos a un tubo de microcentrífuga de 2 mL y centrifugado a 500 x g durante 1 minuto descarte el sobrenadante.
- Añadir 500 μl de tampón de lisis (20 mM Tris-HCl, pH 7,4, 100 mM KCl, 5 mM MgCl2, 1 mM TDT, 0,5% NP-40, 1 x inhibidor de la proteasa cóctel (EDTA-libre), heparina 1 mg/mL o 200 unidades/mL de inhibidor de la Rnasa) a los túbulos. Utilice una pipeta para triturate el tejido.
- Transferir la suspensión a una pequeña (0.5-1.0 mL) homogeneizadores Dounce. Alteran el tejido con siete a ocho golpes de la mano del mortero de vidrio.
- Transferir el lisado a un tubo de microcentrífuga de 1,5 mL.
- Centrifugar la muestra a 12.000 x g y 4 ° C durante 8 min a claro el lisado. Transferir el sobrenadante a un tubo nuevo y de tienda en hielo hasta la carga en el gradiente de sacarosa.
- Recoger 50 μL del lisado como muestra de entrada, congelar inmediatamente a-80 ° C para el análisis de la futura proteína; o añadir Reactivo de purificación de RNA antes de congelar para análisis de ARN.
2. ultracentrifugación y preparación de gradiente de sacarosa
- Preparar dos sacarosa gradiente soluciones (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM de MgCl2, 1 mM TDT, 1 inhibidor de proteasa x cóctel), que contienen sacarosa al 10% o 50% de sacarosa. (Tris-HCl, pH 7.4, puede utilizarse en lugar de HEPES). Agregar 200 unidades/mL heparina inhibidor o 1 mg/mL Rnasa según el diseño experimental. Coloque un tubo de ultracentrífuga para rotor SW 41 en el bloque de marcador y dibujar la línea en el nivel superior del bloque. Transferir el tubo a un bastidor estable.
- Tomar una jeringa de 10 mL con el dispositivo y conectado y llene la jeringa con solución de sacarosa al 10% (preparado como el anterior). Suelte lentamente lo en la parte inferior del tubo de ultracentrífuga hasta llegar a la marca en el tubo.
- Llene otra jeringa con solución de sacarosa al 50% y con cuidado Inserte su dispositivo y a través de la capa de sacarosa del 10% en la parte inferior del tubo. Suelte lentamente la solución de sacarosa a partir de la parte inferior hasta llegar a la marca en el tubo. Sellar el tubo con la tapa provista.
- Para preparar el gradiente de sacarosa, gire el gradiente maker ON. Nivel de la placa con el encima de o abajo botones y presiona DONE. Nivelación es importante para la linealidad de la pendiente.
- Después de nivelar la placa de prensa GRAD para abrir el menú degradado. Ir a la lista en el menú degradado y seleccione el rotor SW 41 Ti. Decídase por el gradiente de sacarosa deseado de la lista del menú utilizando los botones arriba y abajo . Prensa uso.
- Coloque el soporte del tubo de gradiente en la placa del fabricante de la gradiente. Transferir el tubo en el soporte. Hasta 6 gradientes se pueden preparar al mismo tiempo. Pulse Ejecutar. La gradiente eléctrica gira los tubos a la velocidad programada y los ángulos formando un degradado lineal. Se tarda sólo unos minutos para preparar el gradiente.
- Cuando haya completado el proceso, coloque los tubos en una parrilla. Sacar los tapones. Retire el mismo volumen que el volumen de muestra de la parte superior de los tubos de la ultracentrífuga.
- Cuidadosamente la carga 400-500 μL lisado contiene 15-20260 unidades de polisomas en la parte superior. Colocar los tubos en los cubos de rotor y equilibrarlos.
- Centrífuga a 260.000 x g y 4 ° C por 2 h con rotor SW 41.
3. polysome fraccionamiento y recogida de muestras
Nota: Mientras que preparaciones lisadas tienen algunas diferencias dependiendo de la fuente, gradiente preparación polysome fraccionamiento los protocolos y son las mismas para todos los tipos de lisados.
- Después de la terminación de la ultracentrifugación, coloque los cubos de rotor con los tubos en hielo. Girar a colector de fracciones y gradiente fraccionador ON. Haga clic en explorar en el menú del fraccionador. Poner un estante con 24 tubos en el colector de fracciones.
- Llenar un recipiente de enjuague en el fraccionador con agua desionizada. Pulsar la tecla de enjuague durante 10 s para enjuagar la bomba en fraccionador. Fíjele un adaptador de enjuague con la jeringa llenada de agua a los pistones para la calibración.
- Abra el software del fraccionador en el equipo. Prensa de calibrar. Utilizar la configuración por defecto y pulse OK. Esté listo para inyectar el agua de la jeringa.
- Pulse OK para hacer la calibración. Iniciar inmediatamente la inyección de agua para los próximos 5 s. Durante este tiempo, el agua fluirá a través de la célula de flujo UV detector y el instrumento se calibra. El signo Cero calibración completada aparecerá. El instrumento está listo para el fraccionamiento.
- Quite el adaptador de enjuague con la jeringa. Coloque una punta al pistón el fraccionador.
- Abrir el aire válvula y presione aire llave de cobre amarillo para 10 s para tubería seca y celda de flujo. Cierre la válvula de aire.
- Suavemente Retire el tubo de gradiente de la cubeta de rotor y coloque en el canal. Aplicar la tapa del soporte del tubo a la parte superior del tubo y cuidadosamente mueva el tubo en el soporte de tubo y bloquearlo en posición.
- Coloque el soporte bajo el pistón el fraccionador. A menudo, se observan bandas polysomal por el ojo. Introducir los valores deseados para los números de fracción y volumen (24 fracciones a 500 μL/fracción son generalmente suficientes). Nombre del archivo correctamente. Pulse OKy, a continuación, Ir a gráfico de botón. En la siguiente ventana, pulse Iniciar escaneo. Configuración aparecerá, pulse OK. El colector se moverá de la cuneta a la primera fracción y el pistón se mueve en el tubo. Cuando el pistón alcanza la parte superior del gradiente se reducirá a la velocidad seleccionada y se recogerán las fracciones. Cuando haya completado, el pistón se desplaza el tubo de gradiente.
- Abrir el aire válvula y presione aire llave de cobre amarillo en el fraccionador para recuperar la última fracción.
- Pasar los tubos de la parrilla en hielo.
- Añadir 2 volúmenes de reactivo de purificación de RNA para cada fracción y flash congelación en nitrógeno líquido hasta la purificación del ARN. Alternativamente, si la proteína tiene que analizarse, Añadir ácido tricloroacético a la concentración final de 10% a concentrarse para Western Blot (ver sección 8).
4. preparación de ARN sintético In Vitro para la normalización de los mRNAs niveles durante el análisis de datos de RT-qPCR
Nota: El mRNA de la OmpA de e. coli se utiliza en el presente Protocolo para la normalización. Otros ARN, que no tiene identidad extensa con los mRNAs del organismo estudiado (mamífero o Leishmania) puede ser utilizado.
- Preparar el fragmento de ADN de OmpA que contiene SP6 promotor secuencia por una reacción de polimerización en cadena estándar de un plásmido que contiene OmpA gen30.
- Preparar 100 μl de la mezcla: 80 mM HEPES-KOH, pH 7,5, 16 mM MgCl2, espermidina, 10 mM DTT, ATP, CTP, de 3 mM de 3 mM 3 mM UTP, GTP de 3 mM de 2 mM, 0.5 inhibidor de Rnasa U/μL, 1 μg de ADN PCR OmpA 3 μL SP6 RNA polimerasa , 0.005 pyrophosphatase U/μL.
- Incubar a 40 ° C por 2 h.
- Purificar RNA mediante un kit de purificación de RNA.
- Medir la concentración de espectrofotómetro y examinar por electroforesis en gel de agarosa.
5. RNA aislamiento de fracciones degradado y preparación del cDNA
Nota: Proceder directamente con este protocolo para la purificación de RNA si un inhibidor de la Rnasa fue utilizado como un inhibidor de la ribonucleasa. Sin embargo, cuando se usa como un inhibidor de la ribonucleasa, heparina inhiben la transcriptasa inversa utilizada en la preparación de ADNc. Por lo tanto, se necesitará adicional purificación de ARN si la heparina se utilizó en el tampón de lisis y gradiente. Véase el capítulo 6 para preparar síntesis de cDNA RNA si se usó heparina.
- Descongelar las muestras que contiene RNA purificación reactivo, añadir 20 ng de ARN sintético como control interno para la normalización de los resultados de la RT-qPCR. Continuar con la preparación de RNA según protocolo del fabricante excepto una modificación. Añadir 1 μl de precipitación de isopropanol previo de RNA grado del glicógeno (20 μg). Disolver el pellet de RNA en 20-25 μl de agua libre de ARNasa.
Nota: Glucógeno sirve como soporte y ayuda a evitar pérdidas y visualizar el pellet de RNA durante la purificación. OmpA mRNA se utiliza para más de normalización en las reacciones de RT-qPCR. - Medir la concentración de RNA utilizando espectrofotómetro para asegurar una producción adecuada. Combinar volúmenes iguales de las fracciones de RNA que contiene 40S, 60S y monosomes como prepolysomes. Fracciones que contienen ribosomas de 2-4 combinan como polisomas luz y combinan fracciones con 5-8 ribosomas como polisomas pesados.
- Usar 5-10 μl de ARN de fracciones combinadas para preparar cDNAs usando un kit y siguiendo las recomendaciones del fabricante.
- Añadir 80 μl de agua libre de nucleasa a 20 μl de cDNA. Congelar las muestras de cDNA a-20 ° C.
6. RNA purificación de la contaminación de la heparina
Nota: Heparina inhibe el ácido nucleico procesamiento de enzimas como la transcriptasa inversa. Por lo tanto, utilizar este protocolo de purificación adicional cuando se utiliza heparina en el buffer de lisis o en el gradiente.
- Añadir LiCl a una concentración final de 1 M a las muestras de RNA purificadas.
- Mezclar las muestras e incubar en hielo durante 1 hora.
- Girar las muestras a 16.000 x g y 4 ° C por 15 minutos.
- Quite el sobrenadante tan completo como sea posible utilizando una pipeta.
- Deje secar al aire los pellets durante unos 5 minutos.
- Vuelva a suspender los pellets en el volumen inicial de agua libre de ARNasa.
- Realizar la medida espectrofotométrica a 260 nm para determinar la concentración del RNA. Generalmente, la pérdida de la muestra es mínima.
7. RT-qPCR y análisis de datos de distribución de mRNA
- Combinar 10.2 μL de agua, 20 μL de SYBR Green, 4.8 μL de primers específicos gene (juego de 2,5 μM), 5 μL de cDNA, mezcla bien y cargar 10 μL por pocillo en triplicado en la placa de 384 pozos.
- Cubra la placa con la película adhesiva firmemente y centrifugar la placa a 1.800 x g durante 5 minutos.
- Utilizando un instrumento de PCR en tiempo real creado qPCR reacción bajo condiciones que se muestra en la tabla 1.
- El ciclo umbral (CT) valores y CT (ΔΔCT) método comparativo31 calculan el porcentaje (%) de distribución de mRNA en prepolysomes, la luz y polisomas pesados descrito32 con una modificación. Utilizar el RNA sintético (OmpA aquí) para la normalización de los datos en análisis de datos de RT-qPCR. El RNA sintético proporciona un control de normalización que permite calcular los niveles relativos de mRNAs y comparar en diferentes fracciones de un degradado.
8. Análisis de proteínas en las fracciones Polysomal por Western Blot
- De un stock de 100% (w/v), Añadir ácido tricloroacético (TCA) a las fracciones seleccionadas (500 μL) a una concentración final de 10%, mantener en hielo durante al menos 15 min, centrifugar en una microcentrífuga durante 5 minutos, descartar el sobrenadante, lavar dos veces con acetona helada y disolver en 25 μL de S DS-página carga tampón de electroforesis.
- La carga en el SDS-PAGE y realizar electroforesis estándar con la siguiente transferencia a la membrana PVDF. Proceder a Western Blot33.
Resultados
En este estudio, describimos la aplicación de la técnica de generación de perfiles polysomal en tres fuentes diferentes: parásito Leishmania major, células humanas cultivadas y testículo de ratón. Las células de Leishmania crecen libremente en el medio líquido en suspensión células humanas cultivadas crecen en monocapa adherente en las placas y el testículo de ratón representa una muestra de tejido. El método se puede ajustar fácilmente a otros tipos de células cultivadas libremente en suspensión diferentes tipos de tejidos, o de otro organismo y diferentes tipos de células cultivadas. El método consta de cuatro pasos principales: preparación lisado, preparación de gradiente de sacarosa y paso de ultracentrifugación, fraccionamiento polysome y recogida de la muestra seguido por el análisis de las fracciones. Las células de diferentes fuentes son recogidas, lavadas y lisis en tampón de lisis por paso a través de una aguja u homogeneizadores Dounce. Centrifugación se utiliza para eliminar restos celulares, clarificar el lisado. El esquema de fraccionamiento degradado se muestra en la figura 1A. Un gradiente de sacarosa continuo está formado por la mezcla de soluciones de sacarosa 10% y 50% en un fabricante de gradiente. El lisado se carga en la parte superior del gradiente. Ultracentrifugación separa mRNAs asociados con un número diferente de los ribosomas que es monitoreado por un detector de UV durante el fraccionamiento, formando un espectro de absorbancia distinta. Fracciones recogidas se utilizan para el análisis de ARN y proteína (figura 1B). RNA puede ser analizada por electroforesis seguida de Northern blot o utilizado para la producción de cDNA seguido de una reacción de RT-qPCR para analizar la Asociación de mRNAs individuales con polisomas. Secuenciación de próxima generación puede utilizarse para analizar el estado traslacional de mRNAs en una escala genoma-ancha7. Para el análisis de la proteína de las fracciones polysomal, proteínas son precipitadas con ácido tricloroacético para concentrarlos. Luego se analizan proteínas por Western Blot o por espectrometría de masas en el nivel del proteoma.
Un perfil típico de polysomal generado a partir de comandante de Leishmania activamente creciente cultura se muestra en la figura 2A. La gráfica de absorbancia del fraccionamiento tiene una forma distinta con picos típicos de ribosome subunidades 40S y 60S, solo ribosomas (80 o monosomes) y polisomas.
RT-PCR cuantitativa (RT-qPCR) fue empleado para detectar la Asociación de los mRNAs de la Leishmania con ribosomas y polisomas. El método comparativo de31 CT (ΔΔCT) es un enfoque simple y apropiado para el estudio de niveles de mRNAs relativa en las células. Este método requiere un control interno (un mRNA estable, que no cambia de expresión durante los tratamientos o las condiciones del experimento) para los cálculos. Sin embargo, hay ningún control interno en fracciones polysomal porque los niveles de mRNA o ARN ribosómico variará en fracciones, dependiendo de su asociación con los ribosomas, polisomas, etcetera. Para resolver el problema de control interno, hemos utilizado sintética bacteriana OmpA mRNA para la normalización de los niveles de mRNA individuales relativa de Leishmania en las fracciones. OmpA mRNA sintetizado en vitro y agregado en cantidades iguales a cada fracción antes de extracciones de RNA. Además del RNA sintético es importante porque hace cálculos de RT-qPCR datos más precisos, que sirve como control interno para el cálculo por el método comparativo de CT (ΔΔCT).
Alícuotas de las fracciones de gradiente se mezclaron en tres grupos: prepolysomes (subunidades y monosomes), luz polisomas (consistiendo en los ribosomas de 2-4) y pesados polisomas (consistiendo en los ribosomas de 5-8). RT-qPCR fue realizada en RNA de fracciones combinadas para analizar distribución de mRNA entre estas fracciones combinadas (figura 2B). ARN ribosómico 18S fue utilizado como control. Sus niveles relativos determinados por RT-qPCR correlacionaron bien con la distribución estimada de las subunidades ribosomales pequeña (subunidad libre y como parte de monosomes y polisomas) en el espectro. El análisis de RT-qPCR reveló que mRNAs individuales probados tienen diferente grado de participación en la traducción durante la fase logarítmica de crecimiento de Leishmania . MRNA de tubulina se asocia preferentemente pesados polisomas, sugiriendo traducción eficaz. Por el contrario, Sherp mRNA se encuentra principalmente con prepolysomes y luz polisomas apoyo traducción menos activa en comparación con tubulina mRNA.
Expresión de proteínas recombinantes en células cultivadas es un importante enfoque experimental en diversas categorías de estudios. Aquí, presentamos un ejemplo de perfiles polysomal de los mRNAs de la proteína recombinante en otra fuente de la muestra, las células humanas cultivadas. Las células HeLa fueron transfectadas transitoriamente con plásmidos de expresión recombinante fibrosis quística conductancia transmembrana (CFTR) del regulador34 o proteína de la enfermedad de Norrie (NDP). Espectros de la absorbencia del fraccionamiento polysome de estas dos culturas independientes fueron muy similares y contenidos distintos picos correspondientes monosomes (40S y 60S) de subunidades ribosomal (80) y polisomas (Figura 3A). Similitud en los espectros de estos experimentos muestra reproducibilidad del fraccionamiento degradado. Como en los estudios de Leishmania , la distribución de los mRNAs se determinó por RT-qPCR en las fracciones que representan prepolysomes, polisomas ligeros y pesados polisomas (figura 3B). Detección de ARN de la subunidad pequeña ribosomal 18S correlacionada con su distribución estimada en los espectros. NDP mRNAs eran sobre todo asociados con fracciones polysomal livianos y pesados, mientras que los mRNAs CFTR se encuentra principalmente en fracciones prepolysome, sugiriendo que el NDP se traduce más eficientemente. Mientras que el NDP es una proteína relativamente pequeña, CFTR es una proteína muy grande (1480 residuos de aminoácidos) que consta de varios dominios, que se pliegan independientemente durante traducción35. Contratos menor de mRNA CFTR con polisomas pueden reflejar traducción más lento que se requiere para plegable cotranslacional de sus dominios distintos.
Fracciones polysome también pueden utilizarse para la detección de proteínas. Detección de proteínas en las fracciones de gradiente se realizó en el ejemplo de proteínas ribosomales en células HeLa (figura 4). Se concentraron las proteínas por precipitación con 10% TCA de las fracciones y el Western blot se utilizó para detectar la proteína ribosomal de la subunidad pequeña RPS6 y proteína ribosomal de la subunidad grande RPL11 (figura 4, panel superior). Su distribución se correlacionaron bien con los distintos picos en el espectro de absorbancia. Estos experimentos demuestran claramente que las fracciones polysome pueden utilizarse para analizar proteínas en ellos.
Se utilizaron los gradientes de muchos sacarosa diferentes concentraciones (por ejemplo, 7-47%36, 5-50%7, 7-50%6 , 10-50%37, 15-50%8y otros) para el fraccionamiento de polisomas. Aquí comparamos dos gradientes 10-50% y 17-51% (figura 5). Aunque, 17-51% producido resultados aceptables, la separación del gradiente de 10-50% era general mejor.
Está bien documentado que agentes quelantes como el EDTA, interrumpen los ribosomas y polisomas8,9. Como se muestra en la figura 6, tratamiento de EDTA de la HeLa lisado antes de cargar en el gradiente conduce a la desaparición de los picos, correspondientes a monosomes y polisomas, y picos de aumento significativo de las subunidades del ribosoma. Este experimento sirvió como control y había demostrado que los picos observados sin tratamiento de EDTA son polisomas y monosomes realmente ribosomales.
La figura 7 muestra resultados del fraccionamiento polysome de testículos de ratón. El espectro de absorbancia tiene similitudes con los de las células Leishmania y HeLa: picos distintos de subunidades ribosomales, monosomes y polisomas. Su forma y distribución producen un aspecto de la firma, que lo hacen fácil de identificar en diferentes espectros de polysomal. ARN total se purificaron de las fracciones y RNAs de las fracciones se analizaron por electroforesis en gel de agarosa (figura 7, panel superior). La electroforesis muestra una distribución típica de RNAs ribosomal 18S y 28S. Sus bandas agudas indican integridad de las muestras. El gel puede utilizarse para la detección de mRNAs individuales por una mancha blanca /negra norteña siguiente o puede ser utilizado para evaluar la calidad de las muestras antes de otros experimentos en ARN o proteína análisis - el ARN ribosómico difusa bandas indican la degradación del RNA en las muestras.
Durante nuestros estudios, hemos utilizado inhibidor de RNasa y heparina como inhibidores de la Rnasa en los gradientes de lisados y sacarosa. Mientras que ambos proporcionan resultados satisfactorios, el uso de inhibidor de Rnasa era preferible para el análisis de ARN porque no inhibe el cDNA y las reacciones de RT-qPCR. Por lo tanto, no requería pasos adicionales de purificación de RNA. Sin embargo, si los investigadores deciden utilizar heparina durante la preparación de polysome, ten en cuenta que la heparina inhibe la últimas aplicaciones como RT-qPCR y paso adicional de purificación de RNA es necesario (ver protocolo de sección 6).
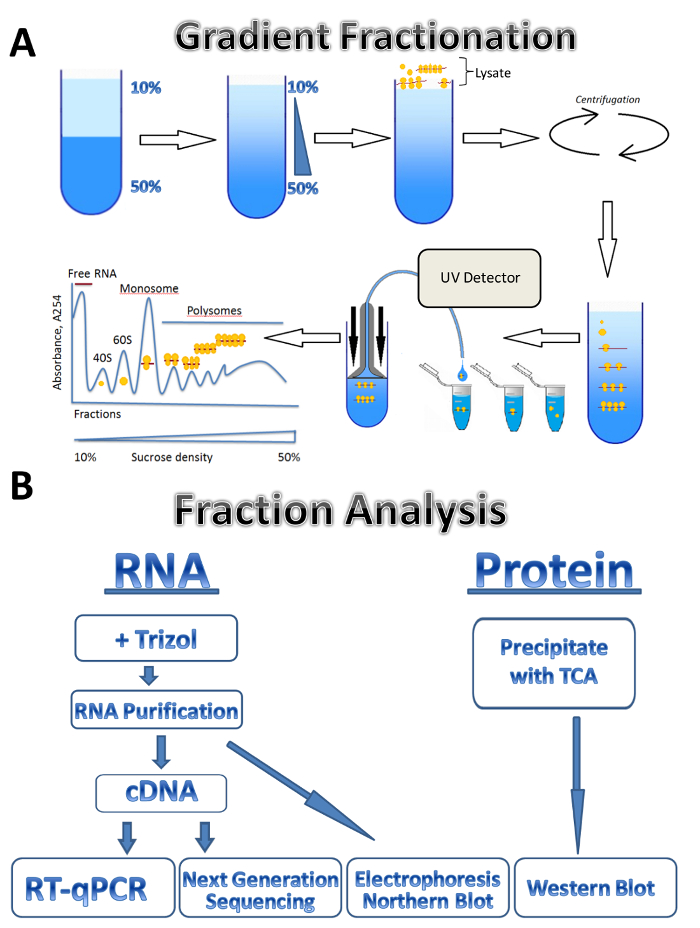
Figura 1 . Perfiles de polysome. (A) esquema de gradiente preparación, perfil de fraccionamiento y absorbancia polysome. (B) esquema del análisis de la fracción. Haga clic aquí para ver una versión más grande de esta figura.
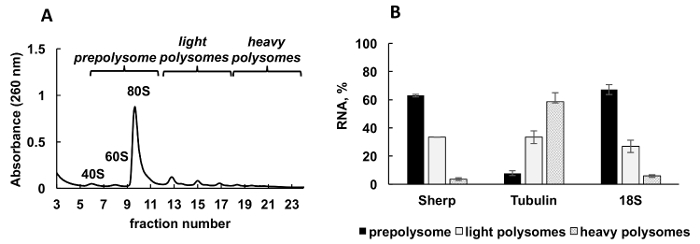
Figura 2 . Análisis del perfil de polysome de comandante de Leishmania cultivo en fase logarítmica de crecimiento. (A) citoplásmicos lisado fue fraccionado en gradiente de sacarosa 10-50%. (B) distribución relativa de ARN 18S, tubulina y Sherp mRNAs (%) en prepolysomes, polisomas ligeros y pesados de las células de registro analizadas por RT-qPCR. Fracciones que contienen 40S, 60S y monosomes fueron combinados como prepolysomes. Fracciones con 2-4 ribosomas se combinaron como polisomas luz, mientras que fracciones con 5-8 ribosomas forman polisomas pesados. Sintético e. coli OmpA mRNA añadido a la extracción de RNA previo fracciones sirve como un control de normalización en RT-qPCR. Comparativa de CT (ΔΔCT)31 método fue utilizado para el cálculo de niveles de mRNA. Barras de error representan el error estándar. Haga clic aquí para ver una versión más grande de esta figura.
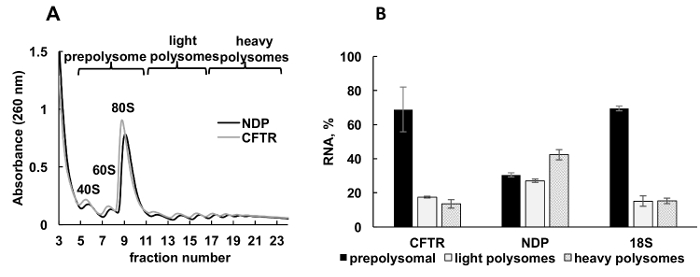
Figura 3 . Polysome fraccionamiento y análisis de Asociación de mRNAs recombinante de la CFTR y NDP con ribosomas en células HeLa transfectadas con el plásmido DNAs. (A) perfil Polysomal en células HeLa transfectadas con plásmidos CFTR y NDP. gradiente de sacarosa 10% - 50% se aplica para lograr separación de polisomas. Se indican los picos para pequeño (40) y grande (60S) subunidades, así como monosome (80). Fracciones fueron combinados como se muestra en el panel A y utilizados para su posterior análisis. (B) distribución de mRNAs de CFTR y NDP en diferentes fracciones. Detección de 18 años por RT-qPCR fue utilizada como un control para fraccionamiento polysome. Niveles de ARN fueron evaluados por análisis de RT-qPCR. Los datos fueron normalizados utilizando ARNm sintéticos. Comparativa de CT (ΔΔCT)31 método fue utilizado para el cálculo de niveles de mRNA. Barras de error representan el error estándar. Haga clic aquí para ver una versión más grande de esta figura.
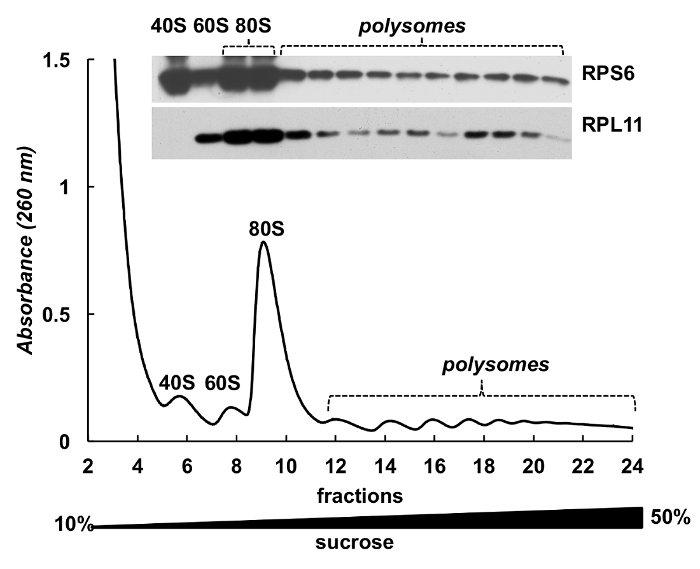
Figura 4 . Detección de proteínas ribosomales en fracciones polysomal HeLa. Lisado de células de Hela fue sometido por centrifugación gradiente de sacarosa 10% - 50%. Las proteínas en las fracciones fueron precipitadas con ácido Tricloroacético y analizadas por electroforesis en 12% SDS-PAGE con la siguiente Western Blot usando ratón RPS6 monoclonal y policlonal RPL11 anticuerpos como anticuerpos primarios y cabra Peroxidase-Conjugated el ratón o Anti-conejos anticuerpos secundarios. Visualización de las señales fue realizada por sustrato quimioluminiscencia SuperSignal West Pico PLUS. Haga clic aquí para ver una versión más grande de esta figura.
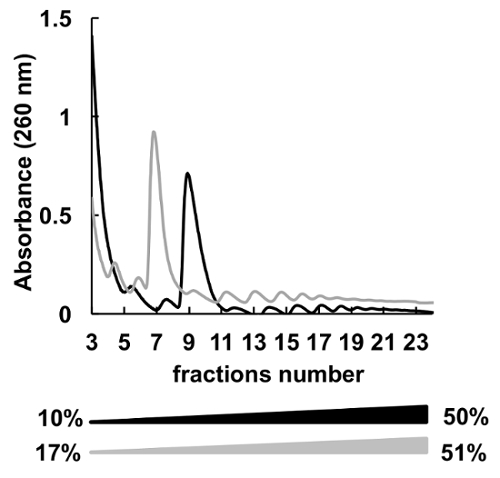
Figura 5 . Comparación de HeLa polysomal perfiles en 10% - 50% (negro) o gradientes de sacarosa 17% - 51% (gris). Haga clic aquí para ver una versión más grande de esta figura.
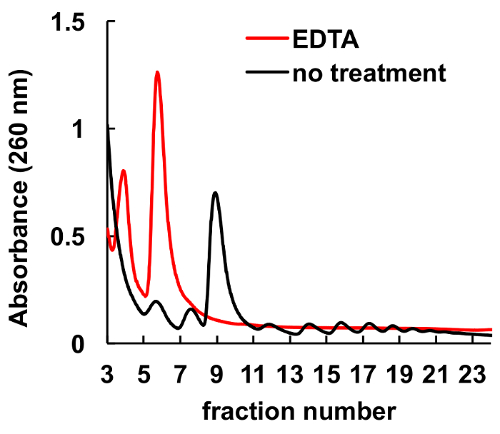
Figura 6 . Efecto del tratamiento con EDTA en perfil polysomal en células HeLa. Lisado de células de Hela fue tratada con 10 mM EDTA en hielo durante 10 minutos inmediatamente antes de la centrifugación de gradiente de sacarosa. MgCl2 fue sustituido por 5 mM de EDTA en soluciones gradiente de sacarosa. Haga clic aquí para ver una versión más grande de esta figura.
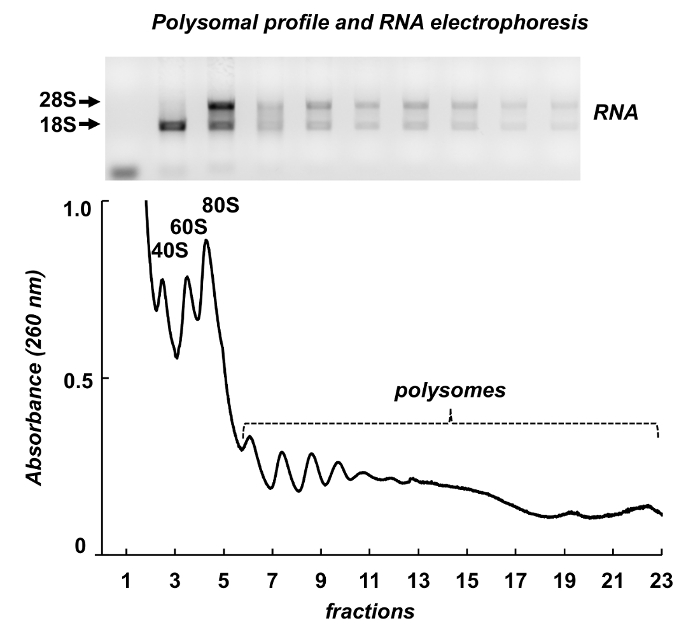
Figura 7 . Perfil polysomal del tejido de testículo de ratón lisado. Fracciones fueron sometidas a extracción de RNA con un reactivo de purificación de RNA y analizadas por electroforesis en gel de agarosa al 1%. Haga clic aquí para ver una versión más grande de esta figura.
| Etapa | Paso | Condición |
| Mantenga | Paso 1 | Aumentar la temperatura de 25 a 50° C con 1,6 ° C/s |
| Incubar a 50° C para 2:00 min | ||
| Paso 2 | Aumentar la temperatura de 50 a 95° C con 1,6 ° C/s | |
| Incubar a 95° C por 10:00 min | ||
| POLIMERIZACIÓN EN CADENA | Paso 1 | Incubar a 95° C para 00:15 min |
| Paso 2 | Disminuir la temperatura de 95 a 60° C con 1,6 ° C / | |
| Incubar a 60° C de 1:00 min | ||
| Número de ciclos de 40 | ||
| Curva del derretimiento | Paso 1 | Aumentar la temperatura de 60 a 95° C con 1,6 ° C/s |
| Paso 2 | Disminuir la temperatura de 95 a 60° C con 1,6 ° C/s | |
| Incubar a 60° C de 1:00 min | ||
| Paso 3 (disociación) | Aumentar la temperatura de 60 a 95° C con 0,05 ° C/s | |
| Incubar a 95° C para 00:15 min | ||
Tabla 1. Condiciones para RT-qPCR
Discusión
Fraccionamiento polysome por gradiente de sacarosa combinada con ARN y análisis de la proteína de las fracciones es un método poderoso para analizar estado traslacional de mRNAs individuales o el conjunto translatome, así como roles de factores de la proteína de regulación traduccional maquinaria durante el normal estado fisiológico o enfermedad. Polysomal perfilado es una técnica especialmente adecuada para el estudio de regulación traduccional en organismos como trypanosomátidos incluyendo Leishmania donde control transcripcional es ausente y regulación de la expresión génica ocurre sobre todo durante la traducción.
Aquí, describimos un protocolo de fraccionamiento polysome utilizado en tres modelos: Leishmania parásitos, células humanas cultivadas y tejidos de ratón. El paso de fraccionamiento polysome es esencialmente el mismo para los diferentes organismos utilizados en este estudio; sin embargo, la preparación lisada tiene algunas diferencias. Células de Leishmania crecen en cultivo líquido y se recoge por centrifugación y las células se cuentan antes de la lisis para iguales carga en el gradiente. Las células humanas se pueden lavar y lisis directamente en la placa. La carga equivalente es controlada por la densidad óptica. Tejidos de ratón requieren un homogeneizador Dounce para lisis eficiente mientras que en el caso de la Leishmania y las células humanas, es suficiente para pasar a través de la aguja de calibre 23.
Todos los reactivos utilizados deben ser RNasa y proteasa libre. Se comparan la heparina y un inhibidor de la Rnasa como inhibidores de la actividad de Rnasa en los lisados citoplásmicos. Encontramos que ambos reactivos pueden bloquear con eficacia la Rnasa. Sin embargo, la heparina afecta aplicaciones posteriores tales como preparación de ADNc y RT-qPCR. Como resultado, preparación de ARN requiere un paso de purificación adicional cuando se utiliza heparina. En nuestra opinión, el inhibidor de Rnasa es la opción más conveniente y puede ser utilizado con eficacia en polysome perfiles de protocolo.
Perfil polysomal es mano de obra, que es una limitación importante del método. Hasta seis gradientes se pueden preparar al mismo tiempo. El fraccionador gradiente genera 144 fracciones que deben ser procesados en un período corto de tiempo. Análisis de fracciones individuales pueden ser desperdiciador de tiempo y costoso. Por lo tanto, combinar fracciones individuales en los polisomas, polisomas ligeros y pesados proporciona una manera rápida y menos laboriosa para estimar actividad traslacional de mRNAs individuales. Nuestros resultados de RT-qPCR sobre las fracciones combinadas nos permitieron identificar diferencias en la traducción de los mRNAs diferentes en Leishmania y en células HeLa (figuras 2, 3). Sin embargo, si se necesita resolución más fina, luego análisis de fracciones individuales se pueden realizar.
Ribosome profiling es otro método para estudiar el estado traslacional de mRNA y se basa en la medición de la producción de proteínas a través de la secuencia de ARNm fragmentos protegidos por ribosoma38. Esta tecnología proporciona información cuantitativa de asociar secuencias de mRNA con fracciones polysomal específicas se traduce en una muestra y puede proporcionar información precisa sobre el estado traslacional de mRNAs en la resolución del codón en comparación con polysome perfiles de tecnología. Sin embargo, polysome perfiles puede utilizarse para el análisis de ARN y proteínas, proporcionando información adicional en el proteoma de polisomas e identificar factores que contribuyen a la regulación de la traducción.
Por lo tanto, polysomal perfilado es una técnica versátil que puede utilizarse para analizar estado traslacional de mRNAs individuales, examinar las proteínas ribosoma asociados y estudio de regulación traduccional en organismos modelo diferente bajo diferentes experimentales condiciones.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen Ching Lee ayuda con grabación de audio. La investigación fue apoyada por los fondos de puesta en marcha de la Texas Tech University Health Sciences Center y por el centro de excelencia de Neurociencia traslacional y terapéutica (CTNT) concede reposo PN 2017-05 AKHRJDHW a A.L.K.; en parte por subvenciones de NIH R01AI099380 K.Z. James C. Huffman y Kristen R. Baca eran eruditos CISER (centro para la integración de la madre educación e investigación) y fueron apoyados por el programa.
Materiales
| Name | Company | Catalog Number | Comments |
| Instruments: | |||
| Gradient master | Biocomp Instruments Inc. | 108 | |
| Piston Gradient Fractionator | Biocomp Instruments Inc. | 152 | |
| Fraction collector | Gilson, Inc. | FC203B | |
| NanoDrop One | Thermo Scientific | NanoDrop One | |
| Nikon inverted microscope | Nikon | ECLIPSE Ts2-FL/Ts2 | |
| 2720 Thermal Cycler | Applied Biosystems by Life Technologies | 4359659 | |
| CO2 incubator | Panasonic Healthcare Co. | MCO-170A1CUV | |
| HERATHERM incubator | Thermo Scientific | 51028063 | |
| Biological Safety Cabinet, class II, type A2 | NuAire Inc. | NU-543-400 | |
| Revco freezer | Revco Technologies | ULT1386-5-D35 | |
| Beckman L8-M Ultracentifuge | Beckman Coulter | L8M-70 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424 | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Swing-bucket rotor | Eppendorf | A-4-62 | |
| Fixed angle rotor | Eppendorf | F-45-30-11 | |
| Quant Studio 12K Flex Real-Time PCR machine 285880228 | Applied Biosystems by life technologies | 4470661 | |
| TC20 Automated cell counter | Bio-Rad | 145-0102 | |
| Hemacytometer | Hausser Scientific | 02-671-51B | |
| Software | |||
| Triax software | Biocomp Instruments Inc. | ||
| Materials: | |||
| Counting slides, dual chamber for cell counter | Bio-Rad | 145-0011 | |
| 1.5 mL microcentrifuge tube | USA Scientific | 1615-5500 | |
| Open-top polyclear centrifuge tubes, (14 mm x 89 mm) | Seton Scientific | 7030 | |
| Syringe, 5 mL | BD | 309646 | |
| BD Syringe 3 mL23 Gauge 1 Inch Needle | BD | 10020439 | |
| Nunclon Delta Surface plate, 14 cm | Thermo Scientific | 168381 | |
| Nunclon Delta Surface plate, 9 cm | Thermo Scientific | 172931 | |
| Nalgene rapid-flow 90mm filter unit, 500 mL, 0.2 aPES | Thermo Scientific | 569-0020 | |
| BioLite 75 cm3 flasks | Thermo Scientific | 130193 | |
| Nunc 50 mL conical centrifuge tubes | Thermo Scientific | 339653 | |
| Chemicals: | |||
| Trizol LS | Ambion by Life Technologies | 10296028 | |
| HEPES | Fisher Scientific | BP310-500 | |
| Trizma base | Sigma | T1378-5KG | |
| Dulbecco's Modified Eagle's Medium-high glucose (DMEM) | Sigma | D6429-500ML | |
| Fetal Bovine Serum (FBS) | Sigma | F0926-50ML | |
| Penicillin-Streptomycin (P/S) | Sigma | P0781-100ML | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | |
| Dulbecco's phosphate buffered saline (DPBS) | Sigma | D8537-500ML | |
| Magnesium chloride hexahydrate (MgCl2x6H2O) | Acros Organics | AC413415000 | |
| Potassium Chloride (KCl) | Sigma | P9541-500G | |
| Nonidet P 40 (NP-40) | Fluka (Sigma-Aldrich) | 74385 | |
| Recombinant Rnasin Ribonuclease Inhibitor | Promega | N2511 | |
| Heparin sodium salt | Sigma | H3993-1MU | |
| cOmplete Mini EDTA-free protease inhibitors | Roche Diagnostics | 11836170001 | |
| Glycogen | Thermo Scientific | R0551 | |
| Water | Sigma | W4502-1L | |
| Cycloheximide | Sigma | C7698-1G | |
| Chloroform | Fisher Scientific | 194002 | |
| Dithiotreitol (DTT) | Fisher Scientific | BP172-5 | |
| Ethidium Bromide | Fisher Scientific | BP-1302-10 | |
| Ethylenediaminetetraacetic acid disodium dehydrate (EDTA) | Fisher Scientific | S316-212 | |
| Optimem | Life Technologies | 22600050 | |
| Puromycin dihydrochloride | Sigma | P8833-100MG | |
| Sucrose | Fisher Scientific | S5-3KG | |
| Trypsin-EDTA solution | Sigma | T4049-100ML | |
| Hgh Capacity cDNA Reverse Transcriptase Kit | Applied Biosystems by life technologies | 4368814 | |
| Power SYBR Green PCR Master Mix | Applied Biosystems by life technologies | 4367659 | |
| HCl | Fisher Scientific | A144SI-212 | |
| Isopropanol | Fisher Scientific | BP26324 | |
| Potassium Hydroxide (KOH) | Sigma | 221473-500G | |
| Anti-RPL11 antibody | Abcam | ab79352 | |
| Ribosomal protein S6 (C-8) antibody | Santa Cruz Biotechnology Inc. | sc-74459 | |
| 1xM199 | Sigma | M0393-10X1L | |
| Lithium cloride | Sigma | L-9650 | |
| Dimethyl sulfoxide (DMSO) | Fisher Scientific | D128-500 | |
| Gel Loading Buffer II | Thermo Scientific | AM8546G | |
| UltraPure Agarose | Thermo Scientific | 16500-100 | |
| Trichloracetic acid (TCA) | Fisher Scientific | A322-100 | |
| SuperSignal West Pico PLUS chemiluminescent substrate | Thermo Scientific | 34580 | |
| Formaldehyde | Fisher Scientific | BP531-500 | |
| Sodium Dodecyl Sulfate (SDS) | Sigma | L5750-1KG | |
| Phenylmethylsulfonyl fluoride (PMSF) | Sigma | P7626-5G | |
| RNeasy Mini kit | Qiagen | 74104 | |
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A1852-1VL | |
| Cytosine 5'-triphosphate disodium salt hydrate (CTP) | Sigma | C1506-250MG | |
| Uridine 5'-triphosphate trisodium salt hydrate (UTP) | Sigma | U6625-100MG | |
| Guanosine 5'-triphosphate sodium salt hydrate (GTP) | Sigma | G8877-250MG | |
| SP6 RNA Polymerase | NEB | M0207S | |
| Pyrophoshatase | Sigma | I1643-500UN | |
| Spermidine | Sigma | S0266-1G |
Referencias
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Capewell, P., et al. Regulation of Trypanosoma brucei Total and Polysomal mRNA during Development within Its Mammalian Host. PLoS One. 8 (6), e67069 (2013).
- Warner, J. R., Knopf, P. M., Rich, A. A multiple ribosomal structure in protein synthesis. Proceedings of the National Academy of Science, USA. 49, 122-129 (1963).
- Piccirillo, C. A., Bjur, E., Topisirovic, I., Sonenberg, N., Larsson, O. Translational control of immune responses: from transcripts to translatomes. Nature Immunology. 15 (6), 503-511 (2014).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of Inhibition of Protein Synthesis in Mammalian Cells. Science. 146 (3650), 1474-1476 (1964).
- Masek, T., Valasek, L., Pospisek, M. Polysome analysis and RNA purification from sucrose gradients. Methods in Molecular Biology. 703, 293-309 (2011).
- Gandin, V., et al. Polysome fractionation and analysis of mammalian translatomes on a genome-wide scale. Journal of Visualized Experiments. (87), (2014).
- Zuccotti, P., Modelska, A. Studying the Translatome with Polysome Profiling. Methods in Molecular Biology. 1358, 59-69 (2016).
- Chasse, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), e15 (2017).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Science, USA. 100 (7), 3889-3894 (2003).
- Gandin, V., et al. nanoCAGE reveals 5' UTR features that define specific modes of translation of functionally related MTOR-sensitive mRNAs. Genome Research. 26 (5), 636-648 (2016).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Zanchin, N. I., Goldfarb, D. S. Nip7p interacts with Nop8p, an essential nucleolar protein required for 60S ribosome biogenesis, and the exosome subunit Rrp43p. Molecular Cell Biology. 19 (2), 1518-1525 (1999).
- Darnell, J. C., et al. FMRP stalls ribosomal translocation on mRNAs linked to synaptic function and autism. Cell. 146 (2), 247-261 (2011).
- Gkogkas, C. G., et al. Autism-related deficits via dysregulated eIF4E-dependent translational control. Nature. 493 (7432), 371-377 (2013).
- Robichaud, N., Sonenberg, N. Translational control and the cancer cell response to stress. Curr Opin Cell Biol. 45, 102-109 (2017).
- Gordon, B. S., Kelleher, A. R., Kimball, S. R. Regulation of muscle protein synthesis and the effects of catabolic states. International Journal of Biochemistry and Cell Biology. 45 (10), 2147-2157 (2013).
- Ishimura, R., et al. RNA function. Ribosome stalling induced by mutation of a CNS-specific tRNA causes neurodegeneration. Science. 345 (6195), 455-459 (2014).
- Petersen, C. P., Bordeleau, M. E., Pelletier, J., Sharp, P. A. Short RNAs repress translation after initiation in mammalian cells. Molecular Cell. 21 (4), 533-542 (2006).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. Proceedings of the National Academy of Science, USA. 111 (1), E203-E212 (2014).
- Bunnik, E. M., et al. Polysome profiling reveals translational control of gene expression in the human malaria parasite Plasmodium falciparum. Genome Biology. 14 (11), R128 (2013).
- De Gaudenzi, J. G., Noe, G., Campo, V. A., Frasch, A. C., Cassola, A. Gene expression regulation in trypanosomatids. Essays in Biochemistry. 51, 31-46 (2011).
- Alves, L. R., Goldenberg, S. RNA-binding proteins related to stress response and differentiation in protozoa. World Journal of Biological Chemistry. 7 (1), 78-87 (2016).
- De Pablos, L. M., Ferreira, T. R., Walrad, P. B. Developmental differentiation in Leishmania lifecycle progression: post-transcriptional control conducts the orchestra. Current Opinions in Microbiology. 34, 82-89 (2016).
- Soto, M., et al. Cell-cycle-dependent translation of histone mRNAs is the key control point for regulation of histone biosynthesis in Leishmania infantum. Biochemical Journal. 379, 617-625 (2004).
- McNicoll, F., et al. Distinct 3 '-untranslated region elements regulate stage-specific mRNA accumulation and translation in Leishmania. Journal of Biological Chemistry. 280 (42), 35238-35246 (2005).
- Folgueira, C., et al. The translational efficiencies of the two Leishmania infantum HSP70 mRNAs, differing in their 3 '-untranslated regions, are affected by shifts in the temperature of growth through different mechanisms. Journal of Biological Chemistry. 280 (42), 35172-35183 (2005).
- Dumas, C., Chow, C., Muller, M., Papadopoulou, B. A novel class of developmentally regulated noncoding RNAs in Leishmania. Eukaryotic Cell. 5 (12), 2033-2046 (2006).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular Cell Biology. 10 (3), 1084-1094 (1990).
- Karamyshev, A. L., Johnson, A. E. Selective SecA association with signal sequences in ribosome-bound nascent chains: a potential role for SecA in ribosome targeting to the bacterial membrane. Journal of Biological Chemistry. 280 (45), 37930-37940 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Panda, A. C., Martindale, J. L., Gorospe, M. Polysome Fractionation to Analyze mRNA Distribution Profiles. Bio Protocols. 7 (3), (2017).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning. A Laboratory Manual. , (1989).
- Patrick, A. E., Karamyshev, A. L., Millen, L., Thomas, P. J. Alteration of CFTR transmembrane span integration by disease-causing mutations. Molecular Biology of the Cell. 22 (23), 4461-4471 (2011).
- Kleizen, B., van Vlijmen, T., de Jonge, H. R., Braakman, I. Folding of CFTR is predominantly cotranslational. Molecular Cell. 20 (2), 277-287 (2005).
- van den Elzen, A. M., Schuller, A., Green, R., Seraphin, B. Dom34-Hbs1 mediated dissociation of inactive 80S ribosomes promotes restart of translation after stress. EMBO Journal. 33 (3), 265-276 (2014).
- Morita, M., et al. mTOR Controls Mitochondrial Dynamics and Cell Survival via MTFP1. Molecular Cell. 67 (6), 922-935 (2017).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados