Method Article
Polysome Profilerstellung in Leishmaniose, menschliche Zellen und Maus Hoden
In diesem Artikel
Zusammenfassung
Das übergeordnete Ziel der Polysome Profilerstellung Technik ist Analyse der übersetzerischen Tätigkeit der einzelnen mRNAs oder Transkriptom mRNAs während der Proteinsynthese. Die Methode ist für Studien von Protein-Synthese-Verordnung, Übersetzung Aktivierung und Repression in Gesundheit und mehrere menschliche Krankheiten wichtig.
Zusammenfassung
Richtige Protein-Expression zum richtigen Zeitpunkt und in der richtigen Menge ist die Grundlage der normalen Zellfunktion und Überleben in einer sich schnell verändernden Umgebung. Für eine lange Zeit dominierten die genexpressionsstudien Forschung auf transkriptioneller Ebene. Jedoch die Steady-State Ebenen der mRNAs nicht korrelieren gut mit Protein-Produktion und die Übersetzbarkeit von mRNAs variiert stark je nach Bedingungen. In einigen Organismen, wie den Parasiten Leishmaniaist die Proteinexpression vor allem auf der translationalen Ebene geregelt. Jüngste Studien gezeigt, dass dieses Protein Übersetzung Dysregulation mit Krebs, Stoffwechselstörungen, neurodegenerativen und anderen Krankheiten des Menschen verknüpft ist. Polysome Profilerstellung ist eine leistungsfähige Methode, Protein Übersetzung Verordnung zu studieren. Es ermöglicht die translationalen Status der einzelnen mRNAs zu messen oder Übersetzung auf einer genomweiten Skala zu untersuchen. Die Grundlage dieser Technik ist die Trennung von Polysomes, Ribosomen, deren Untereinheiten und kostenlose mRNAs bei der Zentrifugation von einer zytoplasmatischen lysate durch einen Farbverlauf von Saccharose. Hier präsentieren wir Ihnen eine universelle Polysome Profilerstellung verwendete Protokoll auf drei verschiedenen Modellen - Parasiten Leishmania großen, kultivierten Zellen der menschlichen und tierischen Geweben. Leishmaniose Zellen wachsen frei schwebend und kultivierte menschlichen Zellen wachsen in adhärenten Monolage während Maus Hoden eine tierisches Gewebeprobe darstellt. Somit ist die Technik an all diese Quellen angepasst. Das Protokoll für die Analyse der polysomal Brüche beinhaltet Erkennung von einzelnen mRNA-Niveaus von RT-qPCR Proteine durch Western-Blot und Analyse der ribosomalen RNAs durch Elektrophorese. Die Methode kann durch Untersuchung der mRNAs Association mit dem Ribosom auf Transkriptom Ebene durch tiefe RNA-Seq und Analyse des Ribosom-assoziierte Proteine durch Massenspektroskopie der Fraktionen erweitert werden. Die Methode ist leicht zu anderen biologischen Modellen einstellbar.
Einleitung
Regulation der Genexpression in den Zellen wird durch transkriptionelle, posttranskritionelle und posttranslationale Mechanismen gesteuert. Fortschritte in der Tiefe RNA Sequenzierung ermöglichen die Untersuchung der stationären mRNA-Niveaus auf einer genomweiten Skala auf einem noch nie da gewesenen Niveau. Neuere Erkenntnisse ergaben jedoch, dass stationäre mRNA Ebene nicht immer mit Protein Produktion1,2 korreliert. Das Schicksal eines einzelnen Niederschrift ist sehr komplex und hängt von vielen Faktoren wie interne/externe Reize, Stress, etc.. Regulation der Genexpression während der Proteinsynthese bietet eine weitere Schicht von Expressions notwendig für eine schnelle Reaktion in wechselnden Bedingungen. Polysome (oder "Polyribosome"), die Profilierung, die Trennung und Visualisierung von Ribosomen, aktiv zu übersetzen ist eine leistungsfähige Methode, die Regulierung der Proteinsynthese zu studieren. Obwohl die ersten experimentellen Anwendungen in den 1960er Jahren3erschien, ist Polysome Profilerstellung derzeit eine der wichtigsten Techniken in Protein Übersetzung Studien4. Einzelnen mRNAs kann durch mehrere Ribosomen führt zur Bildung von einem Polysome übersetzt werden. Protokolle können auf Ribosomen mit Cycloheximide5 ins Stocken geraten und mRNAs mit unterschiedlichen Anzahlen von Polysomes getrennt werden können, in den Prozess der Polysome Fraktionierung von Saccharose gradient Ultrazentrifugation6,7 , 8 , 9. RNA Analyse der polysomal Brüche dann erlaubt Messung von Veränderungen in der translationalen Staaten der einzelnen mRNAs auf Genom-weite Skala und bei verschiedenen physiologischen Bedingungen4,7, 10. die Methode wurde auch verwendet, um offenbaren die Rollen von 5' UTR und 3' UTR-Sequenzen in der Steuerung von mRNA Übersetzbarkeit11, untersuchen die Rolle der MiRNAs in translational Repression12, entdecken Mängel im Ribosom Biogenese13 , und die Rolle des Ribosom-assoziierte Proteine mit menschlichen Krankheiten14,15verstehen. Während des letzten Jahrzehnts entstanden eine immer größere Rolle für die Regulation der Genexpression während der Übersetzung, die ihre Bedeutung im menschlichen Krankheiten zeigt. Der Beweis für translational Control in Krebs, Stoffwechselstörungen und neurodegenerativen Erkrankungen ist überwältigend15,16,17,18. Z. B. Dysregulation eIF4E-abhängige translational Steuerung trägt zum Autismus im Zusammenhang mit Defiziten15 und FMRP engagiert sich in der Ribosomen im Zusammenhang mit Autismus14mRNAs abwürgen. Polysomal Profilierung ist somit ein sehr wichtiges Instrument, um Mängel in translational Regelung in mehrere menschliche Krankheiten zu studieren.
Protein-Analyse der polysomal Brüche unter verschiedenen physiologischen Bedingungen seziert die Funktion der Faktoren im Zusammenhang mit Ribosomen während der Übersetzung. Die Polysome Profilerstellungs-Technik ist in vielen Arten wie Hefe, Säugetier-Zellen, Pflanzen und Protozoen10,19,20,21verwendet worden. Protozoen-Parasiten wie Trypanosomen und Leishmanien begrenzte transkriptionelle Kontrolle der Genexpression aufweisen. Ihre Genome sind in Polycistronic Gen-Cluster organisiert, die Promoter-regulierte Transkription22fehlt. Developmental Genexpression wird hingegen überwiegend auf der Ebene der Protein-Übersetzung und mRNA Stabilität in Trypanosomatid Arten23,24gesteuert. Deshalb Verständnis für translationale Kontrolle bei fehlender transcriptional Regelung für diese Organismen besonders wichtig. Polysomal die Profilerstellung ist ein leistungsfähiges Werkzeug zur posttranskritionelle Regulation der Genexpression in Leishmaniose25,26,27,28zu studieren.
Die jüngste Fortschritte in der Erkennung der einzelnen mRNAs Ebenen von Real time quantitative PCR (RT-qPCR) und volle Transkriptom Sequenzierung der nächsten Generation sowie Proteomics Technologien, Auflösung und Vorteile des polysomal profiling auf ein neues Niveau bringt. Die Verwendung dieser Methoden kann durch Analyse der einzelnen polysomal Fraktionen durch tiefe RNA-Sequenzierung zusammen mit Proteomic Analyse der translationalen Statusüberwachung von Zellen in einer genomweiten Skala erweitert werden. Dies ermöglicht die Identifizierung neuer molekularer Spieler Regulierung Übersetzung unter verschiedenen physiologischen und pathologischen Bedingungen. Hier präsentieren wir eine universelle Polysome Profilierung auf drei verschiedenen Modellen verwendete Protokoll: die Parasiten Leishmania großen, kultivierten menschlichen Zellen und tierischen Geweben. Beratung präsentieren bei der Vorbereitung der Zelle Lysates wir aus verschiedenen Organismen, Optimierung der Gradientenbedingungen, Auswahl von RNase-Inhibitoren und Anwendung von RT-qPCR, Western-Blot und RNA-Elektrophorese Polysome Brüche in dieser Studie zu analysieren.
Protokoll
Alle tierischen Behandlungen und Handhabung von Geweben, die in der Studie gewonnen wurden nach Protokolle durch die institutionelle Animal Care and Use Committee an der Texas Tech University Health Science Center in Übereinstimmung mit den National Institutes of genehmigt durchgeführt. Gesundheit Tierschutzrichtlinien, Protokollnummer 96005. Bitte Wirbeltieren zu Opfern und Gewebe nach den Richtlinien von der institutionellen Animal Care und Nutzung vorbereiten. Fehlt einen solcher Ausschuss entnehmen Sie bitte der National Institutes of Health Tierschutzrichtlinien. Erwachsene (> 60 Tage alten) C57BL/6 Mäusen verwendet wurden. Alle Tiere und Geweben wurden nach Protokollen durch die institutionelle Animal Care and Use Committee an der Texas Tech University Health Sciences Center in Übereinstimmung mit den National Institutes of Health Tierschutzrichtlinien genehmigt erhalten. Für Euthanasie, wurde ein einziger Mausklick in eine kleine Kammer platziert, und die Luft wurde allmählich verdrängt, mit ca. 30 % Kohlendioxid zu betäuben und die Not des Tieres zu minimieren. Nach Beendigung der Atmung verwendeten wir zervikale Dislokation um den Tod des Tieres vor der Ernte Gewebe zu bestätigen.
Achtung: Alle Arbeiten mit live Leishmaniose und kultivierten menschlichen Zellen erfolgte im Biosafety Schrank in zertifizierten Labors BSL-2.
1. Vorbereitung der zytoplasmatischen Lysates aus Leishmania große , kultivierten menschlichen Zellen und Geweben Maus
Hinweis: Es gibt einige Unterschiede in der lysate Zubereitungen aus verschiedenen Ausgangsstoffen. Weitere Schritte einschließlich Saccharose gradient Vorbereitung und polysomal Fraktionierung sind identisch und hängen nicht Aufnahmequelle.
-
Leishmaniose großen zytoplasmatischen lysate Vorbereitung
- Impfen Leishmania große (FV1-Stamm) Zellen in 1 x M199 mittlere29 , 10 % fetalen Bovine Serum (FBS) und Penicillin/Streptomycin Mischung enthält 30 mL (100 Einheiten und 100 μg/mL entsprechend)in Dichte von 1 x 105 Zellen/mL .
Hinweis: Alle Schritte mit Leishmania große Zellen müssen in ein Kabinett Biosafety durchgeführt werden. - Zellen in den Inkubator und wachsen sie bei 27 ° C bis zur logarithmischen Phase (Mitte Log entspricht 5 x 106 Zellen/mL). Es dauert in der Regel etwa zwei Tage um zu wachsen.
- Fügen Sie Cycloheximide Leishmania große Kultur, eine Endkonzentration von 100 μg/mL, die Ribosomen auf übersetzten mRNAs zu verhaften. Setzen Sie Zellen wieder in den Inkubator für 10 min bei 27 ° C.
- Nachdem Cycloheximide Behandlung abgeschlossen ist, Zellen auf eine 50 mL konische Rohr übertragen und mit 1.800 x g und 4 ° C für 8 min. verwerfen überstand drehen.
- Zellen mit 30 mL Dulbeccos Phosphat gepufferte Kochsalzlösung (DPBS) zu waschen. Zentrifuge bei 1.800 x g und 4 ° C für 8 Minuten.
- Verwerfen Sie den überstand. Zellen in 1 mL des DPBS aufzuwirbeln.
- Nehmen Sie eine Aliquote von Zellen und mischen Sie es mit 3,5 % Formaldehyd-Lösung.
- Zählen von Zellen durch Hemocytometer und ihre Konzentration zu bestimmen. Übertragen Sie die gewünschte Anzahl von Zellen in Microfuge Rohr. Lysate zubereitet aus 0.5x108-2 x 108 Zellen/mL ist ausreichend für eine Saccharose gradient laden.
- Drehen Sie die Zellen bei 1.800 x g und 4 ° C für 8 min. Überstands verwerfen.
- Aufschwemmen der Zelle Pellet auf Eis in 1 mL Lyse-Puffer mit Protease-Inhibitoren und RNase-Inhibitor (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 2 mM DVB-t, 1 % NP-40, 1 X Proteaseinhibitor cocktail (EDTA-frei), 200 Einheiten/mL RNase-Inhibitor).
- Durchlaufen der lysate eine 23-Gauge-Nadel dreimal. Die lysate sollte nach dem Durchtritt durch die Nadel durchsichtig.
- Zentrifuge bei 11.200 x g und 4 ° C für 10 min, lysate zu klären. Übertragen die geklärte lysate, eine neue tube und lassen Sie ihn auf Eis bis Saccharose gradient Ultrazentrifugation.
- Sammeln 400-500 μL der lysate als Eingabe (für später zu analysieren), fixieren Sie sie sofort in flüssigem Stickstoff für zukünftige Proteinanalytik oder fügen Sie RNA Reinigung Reagenz vor dem Einfrieren für RNA-Analyse.
- Impfen Leishmania große (FV1-Stamm) Zellen in 1 x M199 mittlere29 , 10 % fetalen Bovine Serum (FBS) und Penicillin/Streptomycin Mischung enthält 30 mL (100 Einheiten und 100 μg/mL entsprechend)in Dichte von 1 x 105 Zellen/mL .
-
Zytoplasmatischen lysate Vorbereitung von kultivierten menschlichen HeLa-Zellen
- HeLa-Zellen teilen und Säen sie in 20 mL des Mediums DMEM mit 10 % FBS und Penicillin/Streptomycin Mischung (100 Einheiten und 100 μg/mL entsprechend) mit Zelle zählen 2 x 105 Zellen/mL in einer 15 cm-Platte.
- HeLa-Zellen bei 37 ° C, 5 % CO2 für 20-24 h. Perform Plasmid DNA Transfektion laut des Herstellers Protokolle zu wachsen.
- Zellen für 24 h nach Transfektion bei 37 ° C, 5 % CO2zu propagieren.
- Gewachsenen HeLa-Zellen auf die Endkonzentration von 100 μg/mL, die Ribosomen auf übersetzten mRNAs zu verhaften und inkubieren Sie Zellen für 10 min bei 37 ° C, 5 % CO2fügen Sie Cycloheximide hinzu. Aspirieren Sie Medium. Waschen Sie die Zellen zweimal mit kalten DPBS auf Eis.
- Die Platte 500 μL der Lyse Puffer (20 mM HEPES-KOH pH 7.4, 100 mM KCl, MgCl2, 1 mM DTT, 0,5 % NP-40, 1 X Proteaseinhibitor cocktail (EDTA-frei), 5 mM 200 Einheiten/mL von RNase-Inhibitor oder 1 mg/mL Heparin) hinzu und kratzen Sie die Zellen auf dem Eis zu.
- Übertragen Sie die lysierten Zellen auf das Microfuge Rohr. Passen Sie die Konzentration der NP-40 um 0,5 % und MgCl2 bis 5 mM je nach das erhöhte Volumen der Probe.
- Durchlaufen der lysate eine 23-Gauge-Nadel 3 - 6 Mal.
- Drehen Sie bei 11.200 x g und 4 ° C für 8 min, lysate zu klären. Übertragen Sie nach Zentrifugation überstand auf einen neuen Schlauch. Verwenden Sie ein Spektralphotometer auszuwertende Zelleffizienz Lyse und Probenmenge für die Verladung auf die Steigung bestimmen. 0,5 mL 0,1 % 10 μl Probe hinzufügen Sodium Dodecyl Sulfat (SDS). Leere gegen 0,1 % SDS. Messung der Absorption bei 260 nm. Erwartete Absorption Wert liegt um 15-20 Einheiten/mL.
- Verdünnen Sie alle Proben mit Lyse Puffer auf den gleichen Wert der Extinktion vor Saccharose-Gradienten-Zentrifugierung. Bewahren Sie die Proben auf Eis bis Saccharose-Gradienten-Zentrifugierung.
-
Zytoplasmatischen lysate Vorbereitung von Maus Hoden
- Sezieren Sie die Maus-Hoden. Machen Sie einen kleinen Schnitt in der Tunica Albuginea und sammeln Sie die seminiferous Röhrchen der Hoden und übertragen Sie sie in einem 15 mL konische Röhrchen mit 5 mL des DPBS mit 0,1 mM Phenylmethylsulfonyl Fluorid (PMSF) ergänzt.
- Mischen Sie das Gewebe durch invertieren mehrmals kräftig. Lassen Sie das Gewebe auf Einheit Schwerkraft auf Eis für 5 min zu begleichen.
- Entfernen Sie und entsorgen Sie den bewölkten Puffer mit verbindenden Zellen und gewebefragmente. Wiederholen Sie den Vorgang 2-3 mal mehr. Die verbleibenden weißen Pellet wird für seminiferous Röhrchen und Keimzellen bereichert.
- Übertragen von seminiferous Röhrchen Pellet zu einem 2 mL Microcentrifuge Schlauch und Spin bei 500 X g für 1 min. Überstands verwerfen.
- Die Tubuli 500 µL Puffer Lyse (20 mM Tris-HCl, pH 7,4, 100 mM KCl, 5 mM MgCl2, 1 mM DTT, 0,5 % NP-40, 1 X Proteaseinhibitor cocktail (EDTA-frei), 1 mg/mL Heparin oder 200 Einheiten/mL RNase-Inhibitor) hinzufügen. Verwenden Sie eine Pipette, um das Gewebe genannte.
- Übertragen Sie die Aussetzung zu einem kleinen (0,5-1,0 mL) Dounce-Homogenisator. Das Gewebe mit sieben bis acht Striche von der Glas-Stößel zu stören.
- Übertragen Sie die lysate auf einen 1,5 mL Microcentrifuge Schlauch.
- Zentrifugieren Sie Probe bei 12.000 x g und 4 ° C für 8 min, der lysate zu löschen. Übertragen Sie den überstand auf eine neue Röhre und speichern auf Eis bis Laden auf der Saccharose-Gradient.
- Sammeln Sie 50 μL der lysate als eingabesample, fixieren Sie sie sofort bei-80 ° C für zukünftige Protein-Analyse; oder fügen Sie RNA Reinigung Reagenz vor dem Einfrieren für RNA-Analyse.
(2) Saccharose Gradient Vorbereitung und Ultrazentrifugation
- Bereiten Sie zwei Saccharose gradient Lösungen (20 mM HEPES-KOH, pH 7,4, 100 mM KCl, 10 mM MgCl2, 1 mM DVB-t, 1 X Proteaseinhibitor cocktail), mit 10 % Saccharose oder 50 % Saccharose. (Tris-HCl, pH 7,4, kann anstelle von HEPES verwendet werden). Fügen Sie 200 Einheiten/mL RNase-Inhibitor oder 1 mg/mL Heparin nach dem experimentellen Design. Legen Sie eine Ultrazentrifuge Rohr für SW 41 Rotor in der Marker-Block und zeichnen Sie die Linie entlang der oberen Ebene des Blocks. Übertragen Sie das Rohr in einem stabilen Gestell.
- Nehmen Sie eine 10-mL-Spritze mit der Schichtung Gerät angeschlossen und füllen Sie die Spritze mit 10 % Saccharoselösung (wie oben beschrieben vorbereitet). Lassen Sie ihn sanft an der Unterseite des Rohres Ultrazentrifugen, bis die Markierung auf das Rohr erreicht.
- Füllen Sie eine weitere Spritze mit 50 % Saccharoselösung und legen Sie seine Schichtung Gerät durch die 10 % Saccharose Schicht auf dem Boden des Röhrchens. Lösen Sie sanft Saccharoselösung von unten beginnend, bis die Markierung auf das Rohr erreicht. Verschließen Sie das Röhrchen mit der mitgelieferten Kappe.
- Schalten Sie zur Vorbereitung der Saccharose-Gradient der Farbverlauf Maker Gerät ON. Ebene der Platte mit UP oder DOWN Tasten und drücken Sie Fertig. Nivellierung ist wichtig für die Linearität des Farbverlaufs.
- Nach dem Abgleich der plattenpresse GRAD um den Farbverlauf Menü zu öffnen. Zur Liste im Farbverlauf Menü und wählen Sie den Rotor SW 41 Ti. Wählen Sie aus der Liste im Menü mit UP und DOWN Tasten die gewünschte Saccharose Steigung. Presse Verwendung.
- Ort der gradient Schlauchhalter auf den Farbverlauf Maker-Platte. Übertragen Sie das Röhrchen in den Halter. Bis zu 6 Steigungen können gleichzeitig zubereitet werden. Drücken Sie auf Ausführen. Die Farbverlauf Maker dreht die Rohre mit der programmierten Geschwindigkeit und Winkel bilden einen linearen Farbverlauf. Es dauert nur ein paar Minuten um den Farbverlauf vorzubereiten.
- Wenn der Prozess abgeschlossen ist, legen Sie die Rohre in einem Rack. Nehmen Sie die Kapseln aus. Entfernen Sie das gleiche Volumen wie das Probenvolumen von der Oberseite der Ultrazentrifuge Röhren.
- 400-500 μL der lysate mit 15-20260 Einheiten des Polysomes auf der Oberseite sorgfältig zu laden. Legen Sie die Rohre in den Rotor-Eimern und auszugleichen.
- Zentrifuge bei 260.000 x g und 4 ° C für 2 h mit SW 41 Rotor.
3. Polysome Fraktionierung und Probenentnahme
Hinweis: Während lysate Vorbereitungen einige Unterschiede je nach Quelle haben, gradient Vorbereitung und Polysome Fraktionierung Protokolle sind die gleichen für alle Arten von Lysaten.
- Legen Sie nach Abschluss der Ultrazentrifugation die Rotor-Eimer mit den Rohren auf Eis. Schalten Sie Fraktionssammler und gradient plasmafraktionierer ON. Klicken Sie im Menü plasmafraktionierer Scannen . Genommen Sie eine Rack mit 24 Röhrchen in den Fraktionssammler.
- Füllen Sie auf der Seite der plasmafraktionierer mit entionisiertem Wasser spülen Reservoir. Drücken Sie die Taste spülen für 10 s, die Pumpe auf plasmafraktionierer zu spülen. Legen Sie einen spülen-Adapter mit der Spritze mit Wasser gefüllt um die Kolben für die Kalibrierung.
- Öffnen Sie die plasmafraktionierer-Software auf dem Computer. Presse zu kalibrieren. Verwenden Sie die Standard- Einstellungen und drücken Sie "OK". Bereit sein, Wasser aus Spritze injizieren.
- Drücken Sie "OK" , um die Kalibrierung zu tun. Sofort starten Einspritzen von Wasser für die nächsten 5 s. Während dieser Zeit Wasser fließt durch die UV-Detektor-Durchflusszelle und das Gerät kalibriert werden. Wird erscheinen das Zeichen Null Kalibrierung abgeschlossen . Das Gerät ist bereit für die Fraktionierung.
- Entfernen Sie die spülen-Adapter mit der Spritze. Legen Sie einen Tipp auf den Kolben des plasmafraktionierer.
- Öffnen Sie die Messing Luft Ventil und Presse AIR -Taste für 10 s zu trockene Schläuche und Messzelle. Schließen Sie das Luftventil.
- Schonend Entfernen der Gradienten Röhre aus dem Rotor-Eimer und legen Sie sie in das Rack. Gelten Sie die Rohr-Halter GAP an die Spitze des Rohres und vorsichtig bewegen Sie das Rohr im Rohr-Halter und Position einrasten.
- Bringen Sie die Halterung unter dem Kolben der plasmafraktionierer. Oft, können polysomal Bänder mit Auge gesehen werden. Stellen Sie die gewünschten Einstellungen für Bruchteil Zahlen und Volumen (24 Fraktionen zu 500 μL/Bruchteil sind in der Regel ausreichend). Benennen Sie die Datei entsprechend. Drücken Sie "OK"und Gehen zum Diagramm -Taste. Drücken Sie im nächsten Fenster START SCAN. Einstellungen angezeigt werden, drücken Sie "OK". Kollektor wird nach der ersten Fraktion aus der Gosse und der Kolben bewegt sich in die Röhre. Wenn der Kolben die Spitze des Farbverlaufs erreicht wird es langsam zu der gewählten Geschwindigkeit und die Fraktionen werden gesammelt. Nach Fertigstellung, bewegt den Kolben aus dem Farbverlauf Rohr.
- Öffnen Sie den Messing Luft Ventil und Presse Luft Schlüssel auf der plasmafraktionierer der letzten Bruchteil abrufen.
- Verschieben Sie die Rohre aus dem Rack auf Eis.
- Fügen Sie 2 Bände von RNA Reinigung Reagenz zu jedem Bruchteil und Blitz einfrieren in flüssigem Stickstoff bis RNA Reinigung. Alternativ, wenn Protein werden analysiert muss, fügen Sie Trichloressigsäure Säure die Endkonzentration von 10 % für Western blotting (siehe Abschnitt 8) zu konzentrieren hinzu.
4. Vorbereitung der synthetische RNA In vitro- Normalisierung der mRNAs Ebenen bei RT-qPCR Datenanalyse
Hinweis: Die E. Coli OmpA mRNA dient in diesem Protokoll zur Normalisierung. Anderen RNA, die keinen umfassenden Identität mit der mRNAs des untersuchten Organismus (Säugetier oder Leishmaniose) verwendet werden.
- Bereiten Sie das OmpA DNA-Fragment mit SP6 promotorsequenz durch eine standard-PCR-Reaktion aus einem Plasmid enthält OmpA gen30.
- 100 µL der Mischung vorbereiten: 80 mM HEPES-KOH, pH 7.5, 16 mM MgCl2, 2 mM Spermidine, 10 mM DTT, 3 mM ATP, CTP, 3 mM 3 mM UTP, 3 mM GTP, 0,5 U/μl RNase-Inhibitor, 1 μg OmpA PCR DNA, 3 μL SP6 RNA-Polymerase , 0,005 U/μl Pyrophosphatase.
- Bei 40 ° C für 2 h inkubieren.
- Reinigen Sie RNA durch ein RNA-Reinigung-Kit.
- Konzentration von Spektralphotometer Messen und prüfen von Agarose-Gelelektrophorese.
(5) RNA isoliert von Gradient Brüche und cDNA Vorbereitung
Hinweis: Fahren Sie direkt mit diesem Protokoll zur Reinigung von RNA, wenn ein RNase-Inhibitor als Ribonuklease-Inhibitor verwendet wurde. Jedoch beim Einsatz als eine Ribonuklease-Inhibitor wird Heparin hemmen Reverse Transkriptase in cDNA Zubereitung verwendet. Zusätzliche Reinigung von RNA wird daher erforderlich sein, wenn Heparin in der Lyse Puffer und Farbverlauf verwendet wurde. Siehe Abschnitt 6, RNA für die cDNA-Synthese vorzubereiten, wenn Heparin verwendet wurde.
- Tauen Sie die Proben, die RNA-Reinigung-Reagenz, fügen Sie 20 ng synthetische RNA als interne Kontrolle für die Normalisierung der RT-qPCR Ergebnisse. Gehen Sie mit RNA Zubereitung nach Protokoll des Herstellers mit Ausnahme einer Änderung. Fügen Sie 1 µL RNA Grade Glykogen (20 µg) vorherige Isopropanol Niederschlag. RNA-Pellets in 20-25 µL RNase-freies Wasser auflösen.
Hinweis: Glykogen als Träger dient und hilft, um Verluste zu vermeiden und RNA Pellet während der Reinigung zu visualisieren. OmpA mRNA dient zur weiteren Normalisierung in RT-qPCR Reaktionen. - Messen Sie RNA-Konzentration mit Spektralphotometer zur angemessenen Ertrag zu gewährleisten. Gleiche Volumina der RNA Brüche mit 40er, 60er und Monosomes als Prepolysomes zu kombinieren. Brüche mit 2-4 Ribosomen zu kombinieren, als leichte Polysomes und Brüche mit 5-8 Ribosomen als schwere Polysomes zu kombinieren.
- Verwenden Sie 5-10 µL RNA aus kombinierten Fraktionen cDNAs mithilfe eines Kits und nach Empfehlungen des Herstellers vorbereiten.
- 20 µL cDNA 80 µL Nuklease freies Wasser hinzufügen. Einfrieren der cDNA-Proben bei-20 ° C.
(6) RNA-Reinigung von Heparin Kontamination
Hinweis: Heparin hemmt Nukleinsäure Verarbeitung Enzyme wie Reverse Transkriptase. Daher verwenden Sie dieses zusätzliche Reinigung-Protokoll, wenn Heparin im Puffer Lyse und/oder im Farbverlauf verwendet wird.
- Hinzu kommt eine Endkonzentration von 1 M zu den gereinigten RNA-Proben LICI.
- Mischen Sie die Proben und inkubieren Sie für 1 Stunde auf Eis.
- Drehen Sie die Proben bei 16.000 x g und 4 ° C für 15 Minuten.
- Den überstand so vollständig wie möglich mit einer Pipette zu entfernen.
- Trocknen Sie die Pellets für ca. 5 min an der Luft.
- Wieder aussetzen Sie, die Pellets in das Ausgangsvolumen der RNase-freies Wasser.
- Führen Sie photometrische Messung bei 260 nm zur Bestimmung der Konzentration der RNA. Der Verlust der Probe ist in der Regel minimal.
(7) RT-qPCR und Datenanalyse der mRNA Distribution
- 10.2 μL des Wassers, 20 μL der SYBR Green, 4.8 μL gen spezifische Primer (2,5 μM eingestellt), 5 μl cDNA zu kombinieren, gut mischen und 384-Well-Platte 10 μL pro Well in Triplicates bestücken.
- Abdeckplatte mit Klebefilm fest und Platte bei 1.800 x g für 5 min zentrifugieren.
- Mit einem Real-Time PCR Instrument einrichten qPCR Reaktion unter Bedingungen, die in Tabelle 1dargestellt.
- Mit der Zyklus-Schwelle (C-T) berechnen Werte und die vergleichende CT (ΔΔCT) Methode31 Prozentsatz (%) der mRNA Verteilung in Prepolysomes, Licht und schwere Polysomes wie beschrieben32 mit einer Modifikation. Verwenden Sie synthetische RNA (hier OmpA) für Datennormalisierung in RT-qPCR Datenanalyse. Die synthetische RNA bietet eine Normalisierung-Steuerelement, das relative mRNAs Ebenen berechnen und vergleichen sie in verschiedene Fraktionen eines Verlaufs ermöglicht.
8. Analyse von Proteinen in Polysomal Bruchteilen von Western Blotting
- Aus einem 100 % (w/V) bestand hinzu Trichloressigsäure Säure (TCA) ausgewählte Brüche (500 μL), eine Endkonzentration von 10 % halten Sie für mindestens 15 min, Zentrifuge in einer Microfuge für 5 min auf Eis, überstand verwerfen, waschen zweimal mit eiskaltem Aceton und lösen sich in 25 μL der S DS-Seite laden Probenpuffer für Elektrophorese.
- Laden auf der SDS-PAGE und standard Elektrophorese mit folgenden Übertragung der PVDF-Membran führen. Fahren Sie mit Western Blot-33.
Ergebnisse
In dieser Studie beschreiben wir die Anwendung der polysomal Profilerstellung Technik zu drei verschiedenen Quellen: parasitäre Leishmaniose großen, kultivierten menschlichen Zellen und Maus Hoden. Leishmaniose Zellen frei wachsen in den flüssigen Medien in der Schwebe, kultivierte menschlichen Zellen wachsen in die anhaftende Monolage auf Tellern und Maus Hoden stellt eine Gewebeprobe. Die Methode kann leicht auf andere Arten von frei gewachsene Zellen in Suspension, verschiedene Arten von Gewebe oder aus einem anderen Organismus und verschiedene Arten von kultivierten Zellen angepasst werden. Der Ansatz besteht aus vier Hauptschritte: lysate Vorbereitung, Saccharose gradient Vorbereitung und Ultrazentrifugation Schritt, Polysome Fraktionierung und Probenentnahme gefolgt von Analyse der Fraktionen. Zellen aus verschiedenen Quellen gesammelt, gewaschen und in Lyse Puffer durch Durchgang durch eine Nadel oder Dounce-Homogenisator lysiert. Zentrifugation wird verwendet, um die zellenrückstand, Klärung der lysate zu entfernen. Das Schema der gradient Fraktionierung ist in Figur 1Agezeigt. Ein kontinuierlichen Saccharose Farbverlauf entsteht durch das Mischen von 10 % und 50 % Saccharose-Lösungen in einem Farbverlauf Maker. Die lysate ist auf der Oberseite der Gradient geladen. Ultrazentrifugation trennt mRNAs, verbunden mit einer unterschiedlichen Anzahl von Ribosomen, die durch einen UV-Detektor während der Fraktionierung, überwacht wird, bildet eine ausgeprägte Absorptionsspektrum. Gesammelte Fraktionen dienen zur Analyse der RNA und Protein (Abbildung 1 b). RNA kann durch Elektrophorese gefolgt von Northern Blot oder für cDNA-Produktion, gefolgt von einer RT-qPCR-Reaktion verwendet, um die Verbindung der einzelnen mRNAs mit Polysomes analysieren analysiert werden. Next Generation Sequencing kann verwendet werden, um die translationale Status der mRNAs auf einem Genom-weite Skala7zu analysieren. Für Protein-Analyse der polysomal Brüche ausgefällt werden Proteine mit Trichloressigsäure Säure auf sie zu konzentrieren. Proteine werden dann durch Western Blot oder Massenspektroskopie Proteom Ebene analysiert.
Abbildung 2Azeigt ein typisches polysomal Profil von Leishmania große aktiv wachsende Kultur erzeugt. Die Fraktionierung der Extinktion Graph hat eine klare Form mit typischen Spitzen für Ribosomen-Untereinheiten (40er und 60er Jahren), einzelne Ribosomen (80er oder Monosomes) und Polysomes.
Quantitative RT-PCR (RT-qPCR) wurde eingesetzt, um die Verbindung der einzelnen Leishmania mRNAs mit Ribosomen und Polysomes zu erkennen. Die vergleichende CT (ΔΔCT)31 Methode ist ein einfacher und geeignete Ansatz, relative mRNAs Ebenen in den Zellen zu studieren. Diese Methode erfordert eine interne Kontrolle (eine stabile mRNA, das Ausdruck während Behandlungen oder Bedingungen des Experiments nicht ändert) für Berechnungen. Allerdings gibt es keine interne Kontrolle in polysomal Fraktionen, weil Ebenen von mRNA oder ribosomaler RNA in Sekundenbruchteilen, abhängig von ihrer Verbindung mit Ribosomen, Polysomes, etc.variieren. Zur Lösung des Problems der interne Kontrolle haben wir synthetische bakterielle OmpA mRNA für die Normalisierung der relativen einzelne Leishmaniose -mRNA-Niveaus in den Fraktionen verwendet. OmpA mRNA wurde synthetisiert in-vitro- und in gleicher Höhe für jede Fraktion vor RNA-Extraktion hinzugefügt. Zusatz von synthetischen RNA ist wichtig, weil es Berechnungen der RT-qPCR Daten genauer gesagt, dient als interne Kontrolle für Berechnungen durch vergleichende CT (ΔΔCT) Methode macht.
Aliquote der Gradienten Fraktionen wurden in drei Gruppen gemischt: Prepolysomes (Untereinheiten und Monosomes), leichte Polysomes (bestehend aus 2-4 Ribosomen) und schwere Polysomes (bestehend aus 5-8 Ribosomen). RT-qPCR erfolgte auf RNA aus kombinierten Brüche, mRNA-Verteilung zwischen diesen kombinierten Brüchen (Abb. 2 b) zu analysieren. 18 s ribosomalen RNA diente als Kontrolle. Die relativen Pegel bestimmt durch RT-qPCR korreliert gut mit der geschätzte Verteilung der kleinen ribosomalen Untereinheiten (freie Untereinheit und als Teil der Monosomes und Polysomes) auf dem Spektrum. RT-qPCR-Analyse ergab, dass die einzelnen mRNAs getestet haben einen unterschiedlichen Grad an Engagement in der Übersetzung während der logarithmischen Wachstumsphase Leishmaniose . Tubulin mRNA ist bevorzugt mit schweren Polysomes, effiziente Übersetzung vorschlagen. Im Gegensatz dazu findet sich in erster Linie mit Prepolysomes und leichten Polysomes, die Unterstützung weniger aktiv Übersetzung im Vergleich zu Tubulin Sherp mRNA mRNA.
Rekombinante Proteinexpression in kultivierten Zellen ist ein wichtiger experimenteller Ansatz in verschiedenen Kategorien von Studien. Hier präsentieren wir Ihnen ein Beispiel für polysomal Profilierung des rekombinanten Proteins mRNAs in eine weitere Aufnahmequelle, kultivierten menschlichen Zellen. HeLa-Zellen wurden vorübergehend mit Plasmiden rekombinante Mukoviszidose transmembranen Leitwert Regulator (CFTR)34 oder Norrie Krankheit Protein (NDP) auszudrücken transfiziert. Extinktion-Spektren von Polysome Fraktionierung aus diesen zwei unabhängige Kulturen waren sehr ähnlich sind und enthalten verschiedene Gipfel entsprechende ribosomalen Untereinheiten (40er und 60er Jahren), Monosomes (80er Jahre) und Polysomes (Abb. 3A). Ähnlichkeit in den Spektren aus diesen Experimenten veranschaulicht Reproduzierbarkeit der gradient Fraktionierung. Wie wurde in den Leishmaniose -Studien, die Verteilung der mRNAs durch RT-qPCR in den Fraktionen vertreten, Prepolysomes, leichte Polysomes und schwere Polysomes (Abb. 3 b) bestimmt. Erkennung der kleinen ribosomalen Untereinheit 18 s RNA korreliert mit ihrer geschätzten Verteilung in den Spektren. NDP mRNAs waren meist verbunden mit leichten und schweren polysomal Brüche, während CFTR-mRNAs wurden vorwiegend in prepolysome Brüche, was darauf hindeutet, dass NDP effizienter umgesetzt wird. NDP ist ein relativ kleines Protein, zwar CFTR ein sehr großes Protein (1480 Aminosäurereste) bestehend aus mehreren Domänen, die unabhängig voneinander während der Übersetzung35Falten. Unteren Engagements von CFTR-mRNA mit Polysomes möglicherweise langsamer Übersetzung widerspiegeln, die für cotranslational Faltung der unterschiedlichen Domänen erforderlich ist.
Polysome Brüche können auch zur Detektion von Proteinen verwendet werden. Nachweis von Proteinen in den Farbverlauf Fraktionen wurde am Beispiel der ribosomale Proteine in HeLa-Zellen (Abbildung 4) durchgeführt. Konzentrierten sich die Proteine durch Fällung mit 10 % TCA von Brüchen und Western-Blot verwendet wurde, um die kleine Untereinheit ribosomale Protein RPS6 und große Untereinheit ribosomale Protein RPL11 erkennen (Abb. 4, oben). Ihre Verteilung korreliert gut mit den verschiedenen Gipfeln auf das Absorptionsspektrum. Diese Experimente zeigen deutlich, dass die Polysome Brüche verwendet werden können, um Proteine in ihnen zu analysieren.
Viele verschiedene Saccharose Konzentrationen Gradienten (z. B. 7-47 %36, 5-50 %7, 7-50 %6 , 10-50 %37, 15-50 %8, usw.) für Polysomes Fraktionierung verwendet wurden. Hier haben wir zwei Steigungen 10-50 % und 17-51 % (Abbildung 5) verglichen. Obwohl 17-51 % akzeptable Ergebnisse produziert, war die Trennung in der 10-50 % Steigung insgesamt besser.
Es ist gut dokumentiert, dass Komplexbildnern wie EDTA, Ribosomen und Polysomes8,9stören. Wie es in der Abbildung 6, EDTA Behandlung von der HeLa lysate gezeigt vor Belastung auf der Steigung zum Verschwinden der Zinnen führt, entsprechend Monosomes und Polysomes, und signifikante Erhöhung der Ribosomen-Untereinheiten erreicht seinen Höhepunkt. Dieses Experiment als Kontrolle diente und gezeigt, dass die beobachteten Gipfel ohne EDTA Behandlung tatsächlich ribosomale Monosomes und Polysomes.
Abbildung 7 zeigt die Ergebnisse der Polysome Fraktionierung von Maus Hoden. Das Absorptionsspektrum hat Ähnlichkeiten mit denen von Leishmaniose und HeLa-Zellen: verschiedene Gipfel der ribosomalen Untereinheiten, Monosomes und Polysomes. Ihre Form und Verteilung produzieren eine Signaturdarstellung, die es einfach, sie auf verschiedene polysomal Spektren zu identifizieren. Insgesamt RNAs wurden von den Fraktionen gereinigt und RNAs aus ausgewählten Fraktionen wurden durch Elektrophorese im Agarosegel (Abbildung 7, Oberboden) analysiert. Die Elektrophorese zeigt typische Verteilung der 18 s und 28 s ribosomalen RNAs. Ihre scharfen Bands zeigen Unversehrtheit der Proben. Das Gel für die einzelnen mRNAs Erkennung von folgenden Nordfleck verwendet werden oder es kann zur Bewertung der Qualität der Proben vor weiteren Experimenten auf RNS oder Protein-Analyse - die diffuse ribosomalen RNAs, dass RNA-Abbau in den Proben zeigen.
Während des Studiums verwendet RNase-Inhibitor und Heparin als RNase-Inhibitoren in der Lysates und Saccharose Steigungen. Während beide zufriedenstellende Ergebnisse erbracht, war der Einsatz von RNase-Inhibitor für RNA Analyse vorzuziehen, weil es nicht die cDNA und RT-qPCR Reaktionen zu hemmen. So war es nicht weitere RNA Reinigungsschritte erforderlich. Jedoch wenn Forscher Heparin während Polysome Vorbereitung verwenden möchten, beachten Sie, dass Heparin Downstream-Anwendungen wie RT-qPCR hemmt und zusätzliche Reinigungsstufe der RNA erforderlich ist (siehe Protokoll Abschnitt 6).
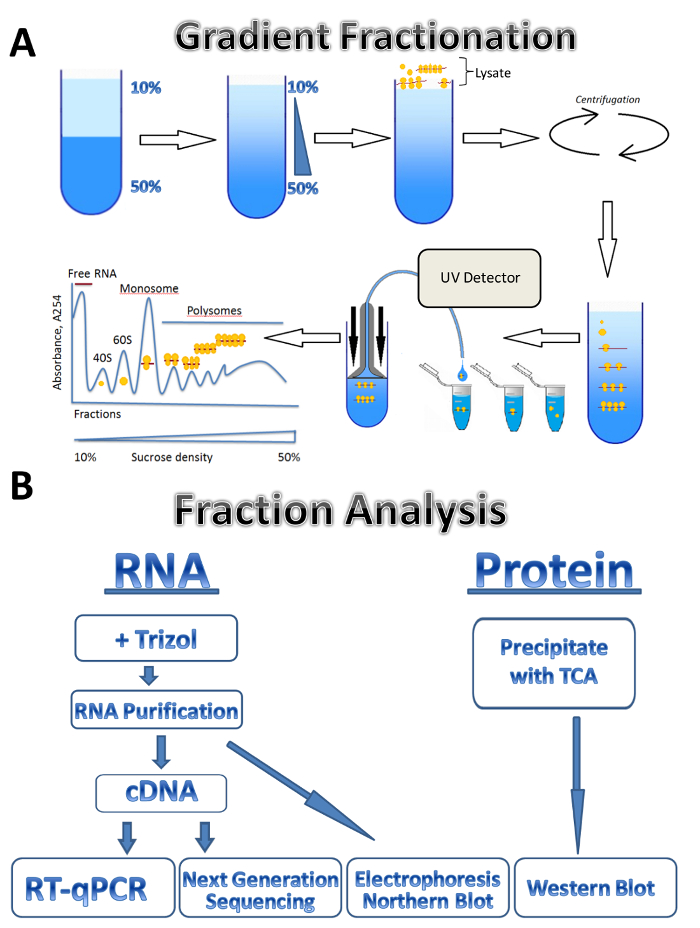
Abbildung 1 . Polysome Profilierung. (A) Regelung der gradient Vorbereitung Polysome Fraktionierung und Extinktion Profil. (B) Regelung der Bruchteil Analyse. Bitte klicken Sie hier für eine größere Version dieser Figur.
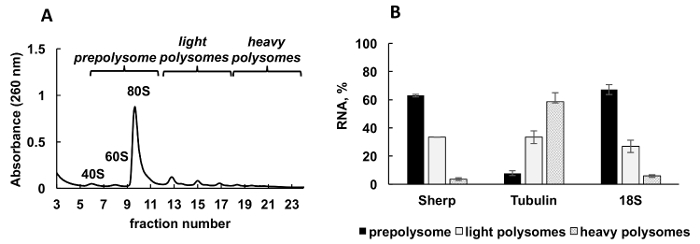
Abbildung 2 . Polysome Profilanalyse der Leishmania große Kultur in der logarithmischen Phase des Wachstums. (A) Cytoplasmic lysate war in 10-50 % Saccharose Steigung fraktioniert. (B) Relative Verteilung der 18 s RNA, Tubulin und Sherp mRNAs (%) in Prepolysomes, leichte und schwere Polysomes Log Zellen von RT-qPCR analysiert. Brüche mit 40er, verbanden 60er und Monosomes als Prepolysomes. Brüche mit 2-4 Ribosomen wurden als leichte Polysomes kombiniert, während Brüche mit 5-8 Ribosomen schweren Polysomes gebildet. Synthetische E. Coli OmpA mRNA hinzugefügt, um Brüche vor RNA-Extraktion als Normalisierung Kontrolle in RT-qPCR diente. Vergleichende CT (ΔΔCT)31 -Methode wurde für die Berechnung der mRNA-Niveaus verwendet. Fehlerbalken darzustellen Standardfehler. Bitte klicken Sie hier für eine größere Version dieser Figur.
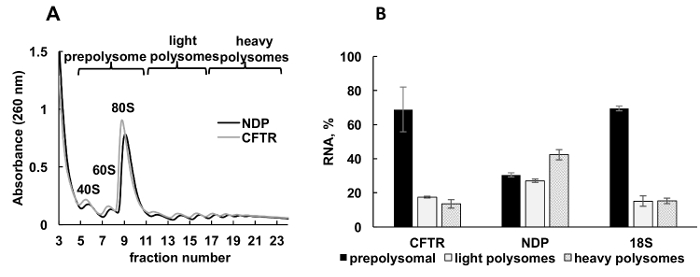
Abbildung 3 . Polysome Fraktionierung und Analyse von rekombinanten CFTR und NDP mRNAs Verband mit Ribosomen in HeLa-Zellen mit Plasmid DNA transfiziert. (A) Polysomal Profil in HeLa-Zellen mit CFTR und NDP Plasmide transfiziert. 10 % - 50 % Saccharose Steigung wurde angewandt, um die Trennung von Polysomes zu erreichen. Der Gipfel für kleine (40er) und große (60er Jahre) Untereinheiten sowie Monosome (80er Jahre) sind angegeben. Brüche wurden kombiniert, siehe Panel A und zur weiteren Analyse verwendet. (B) Vertrieb von mRNAs von CFTR und NDP in verschiedene Fraktionen. Erkennung von 18 von RT-qPCR diente als Kontrolle Polysome Fraktionierung. RNA-Ebenen wurden durch RT-qPCR Analyse ausgewertet. Daten wurden mit synthetischen mRNA normalisiert. Vergleichende CT (ΔΔCT)31 -Methode wurde für die Berechnung der mRNA-Niveaus verwendet. Fehlerbalken darzustellen Standardfehler. Bitte klicken Sie hier für eine größere Version dieser Figur.
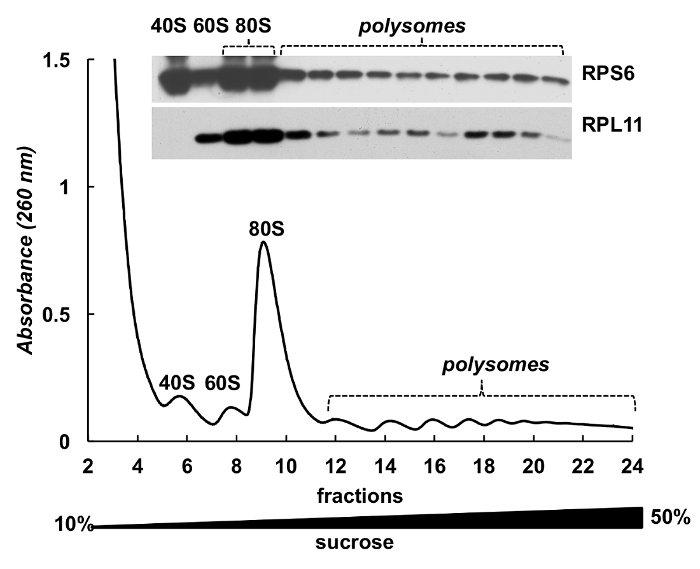
Abbildung 4 . Erkennung von ribosomale Proteine in HeLa polysomal Brüche. Hela-Zelle lysate wurde von 10 % - 50 % Saccharose Steigung Zentrifugierung unterzogen. Proteine in ausgewählten Brüche wurden mit TCA ausgefällt und analysiert durch Elektrophorese in 12 % SDS-PAGE mit folgenden Western blotting mit Maus monoklonalen RPS6 und Rabbit polyklonalen RPL11 Antikörper als primären Antikörper und Peroxidase-Conjugated Ziege Anti-Maus oder Anti-Kaninchen Sekundärantikörper. Visualisierung der Signale erfolgte durch SuperSignal West Pico PLUS chemiluminescent Substrat. Bitte klicken Sie hier für eine größere Version dieser Figur.
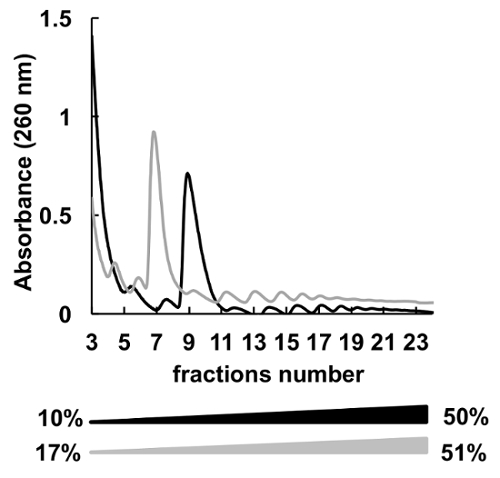
Abbildung 5 . Vergleich von HeLa polysomal Profilerstellung in 10 % - 50 % (schwarz) oder 17-51 % (grau) Saccharose Steigungen. Bitte klicken Sie hier für eine größere Version dieser Figur.
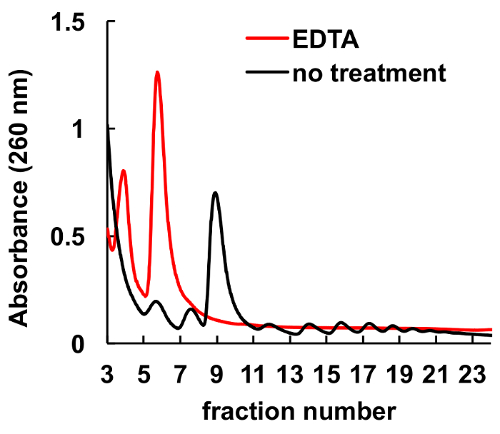
Abbildung 6 . Wirkung von EDTA Behandlung auf polysomal Profil in HeLa-Zellen. Hela-Zelle lysate wurde mit 10 mM EDTA für 10 min unmittelbar vor der Saccharose-Gradienten-Zentrifugierung auf Eis behandelt. MgCl2 wurde durch 5 mM EDTA in Saccharose gradient Lösungen ersetzt. Bitte klicken Sie hier für eine größere Version dieser Figur.
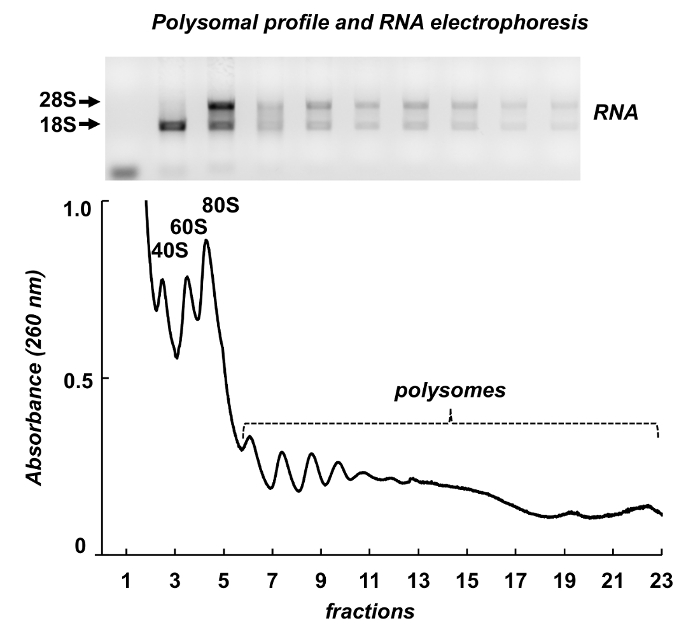
Abbildung 7 . Polysomal Profil aus Maus Hoden Gewebe lysate. Brüche wurden ausgesetzt, RNA-Extraktion mit einem RNA-Reinigung-Reagenz und analysiert durch Elektrophorese in 1 % Agarosegel. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Bühne | Schritt | Zustand |
| Halten | Schritt 1 | Erhöhen Sie die Temperatur von 25 bis 50° C mit 1,6 ° C/s |
| Bei 50° C 02:00 min inkubieren | ||
| Schritt 2 | Erhöhen Sie die Temperatur von 50 bis 95° C mit 1,6 ° C/s | |
| Bei 95° C für 10:00 min inkubieren | ||
| PCR | Schritt 1 | Bei 95° C inkubieren 00:15 min |
| Schritt 2 | Verringern Sie die Temperatur von 95 bis 60° C mit 1,6 ° C / | |
| Bei 60° C für 01:00 min inkubieren | ||
| Anzahl der Zyklen 40 | ||
| Kurve zu schmelzen | Schritt 1 | Erhöhen Sie die Temperatur von 60 bis 95° C mit 1,6 ° C/s |
| Schritt 2 | Verringern Sie die Temperatur von 95 bis 60° C mit 1,6 ° C/s | |
| Bei 60° C für 01:00 min inkubieren | ||
| Schritt 3 (Dissoziation) | Erhöhen Sie die Temperatur von 60 bis 95° C mit 0,05 ° C/s | |
| Bei 95° C inkubieren 00:15 min | ||
Tabelle 1. Bedingungen für RT-qPCR
Diskussion
Polysome Fraktionierung von Saccharose Farbverlauf in Kombination mit RNA und Protein-Analyse der Fraktionen ist eine leistungsfähige Methode, translationale Status der einzelnen mRNAs oder die ganze Translatome sowie Rollen der Protein Faktoren translational Regelung zu analysieren Maschinen während der normalen physiologischen oder Krankheit Zustand. Polysomal die Profilerstellung ist eine besonders geeignete Technik studieren translational Regelung in Organismen wie Trypanosomatids einschließlich Leishmaniose wo transcriptional Control ist weitgehend abwesend und Genregulation Ausdruck tritt meist während der Übersetzung.
Hier beschreiben wir ein Polysome Fraktionierung Protokoll auf drei Modelle: Leishmaniose Parasiten, kultivierten menschlichen Zellen und Geweben der Maus. Die Polysome Fraktionierung Schritt entspricht im Wesentlichen dem für verschiedene Organismen, die in dieser Studie verwendeten; die lysate Vorbereitung hat jedoch einige Unterschiede. Leishmaniose Zellen wachsen in der flüssigen Kultur und wird durch Zentrifugieren gesammelt und Zellen vor der Zerstörung zu gewährleisten gleich laden auf den Farbverlauf gezählt. Menschliche Zellen können gewaschen und lysiert direkt auf den Teller. Die gleiche Beladung wird durch optische Dichte gesteuert. Maus-Gewebe erfordern eine Dounce-Homogenisator für effiziente Lyse bei Leishmaniose und menschlichen Zellen, es ist ausreichend, sie passieren die 23-Gauge-Nadel.
Alle verwendete Reagenzien sollten RNase und Protease frei sein. Wir verglichen die Heparin und RNase-Inhibitor als Inhibitoren der RNase Aktivität in den zytoplasmatischen Lysates. Wir fanden, dass beide Reagenzien RNase effektiv blockieren können. Heparin wirkt sich jedoch auf downstream-Anwendungen wie cDNA Vorbereitung und RT-qPCR. Infolgedessen erfordert Vorbereitung der RNA eine zusätzliche Reinigungsstufe, wenn Heparin verwendet wird. Unserer Meinung nach die RNase-Inhibitor ist bequemer Wahl und Polysome Profilierung Protokoll effektiv einsetzbar.
Polysomal die Profilerstellung ist arbeitsintensiv, die eine erhebliche Einschränkung der Methode ist. Bis zu sechs Gradienten können gleichzeitig zubereitet werden. Der gradient plasmafraktionierer erzeugt 144 Brüche, die in kurzer Zeit verarbeitet werden müssen. Analyse der einzelnen Fraktionen kann zeitraubend und kostspielig zu sein. Daher Kombination von einzelnen Fraktionen in Pre-Polysomes, leichte und schwere Polysomes bietet einen schnellen und weniger mühsamen Weg, übersetzerische Tätigkeit der einzelnen mRNAs zu schätzen. Unsere RT-qPCR-Ergebnisse auf die kombinierte Brüche konnten wir Unterschiede in der Übersetzbarkeit von verschiedenen mRNAs in Leishmaniose und HeLa-Zellen (Abbildungen 2, 3) zu identifizieren. Bei Bedarf feinere Auflösung kann, Analyse der einzelnen Fraktionen erfolgen.
Ribosom profiling ist eine weitere Methode um die translationale Status der mRNA zu studieren und basiert auf der Messung der Proteinproduktion durch Sequenzierung von mRNA Fragmenten durch Ribosom38geschützt. Diese Technologie liefert quantitative Informationen verknüpfen mRNA-Sequenzen mit bestimmten polysomal Fraktionen in einer Probe übersetzt wird, und kann genaue Auskunft über die translationale Status der mRNAs Codon Auflösung im Vergleich zu Polysome profiling-Technologie. Jedoch Polysome profiling für RNA und Protein-Analyse, also Bereitstellung zusätzlicher Informationen über Proteom von Polysomes einsetzbar und Faktoren, die bei der Regulierung von Übersetzung zu identifizieren.
Deshalb polysomal profiling eine vielseitige Technik, die zum Analysieren der translationalen Zustand der einzelnen mRNAs Ribosom-assoziierte Proteine zu untersuchen und translational Regelung in verschiedenen Modellorganismen unter verschiedenen experimentellen Studie Bedingungen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken für die Hilfe mit Audioaufnahme Ching Lee. Die Forschung wurde durch das Startkapital von Texas Tech University Health Sciences Center unterstützt und durch das Center of Excellence für translationale Neurowissenschaften und Therapeutika (CTNT) gewähren, PN-CTNT 2017-05 AKHRJDHW A.L.K.; Teil von NIH Grant R01AI099380 K.Z James C. Huffman und Kristen R. Baca waren CISER (Zentrum für die Integration von Mint-Ausbildung & Forschung) Gelehrten und wurden vom Programm unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Instruments: | |||
| Gradient master | Biocomp Instruments Inc. | 108 | |
| Piston Gradient Fractionator | Biocomp Instruments Inc. | 152 | |
| Fraction collector | Gilson, Inc. | FC203B | |
| NanoDrop One | Thermo Scientific | NanoDrop One | |
| Nikon inverted microscope | Nikon | ECLIPSE Ts2-FL/Ts2 | |
| 2720 Thermal Cycler | Applied Biosystems by Life Technologies | 4359659 | |
| CO2 incubator | Panasonic Healthcare Co. | MCO-170A1CUV | |
| HERATHERM incubator | Thermo Scientific | 51028063 | |
| Biological Safety Cabinet, class II, type A2 | NuAire Inc. | NU-543-400 | |
| Revco freezer | Revco Technologies | ULT1386-5-D35 | |
| Beckman L8-M Ultracentifuge | Beckman Coulter | L8M-70 | |
| Centrifuge | Eppendorf | 5810R | |
| Centrifuge | Eppendorf | 5424 | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Swing-bucket rotor | Eppendorf | A-4-62 | |
| Fixed angle rotor | Eppendorf | F-45-30-11 | |
| Quant Studio 12K Flex Real-Time PCR machine 285880228 | Applied Biosystems by life technologies | 4470661 | |
| TC20 Automated cell counter | Bio-Rad | 145-0102 | |
| Hemacytometer | Hausser Scientific | 02-671-51B | |
| Software | |||
| Triax software | Biocomp Instruments Inc. | ||
| Materials: | |||
| Counting slides, dual chamber for cell counter | Bio-Rad | 145-0011 | |
| 1.5 mL microcentrifuge tube | USA Scientific | 1615-5500 | |
| Open-top polyclear centrifuge tubes, (14 mm x 89 mm) | Seton Scientific | 7030 | |
| Syringe, 5 mL | BD | 309646 | |
| BD Syringe 3 mL23 Gauge 1 Inch Needle | BD | 10020439 | |
| Nunclon Delta Surface plate, 14 cm | Thermo Scientific | 168381 | |
| Nunclon Delta Surface plate, 9 cm | Thermo Scientific | 172931 | |
| Nalgene rapid-flow 90mm filter unit, 500 mL, 0.2 aPES | Thermo Scientific | 569-0020 | |
| BioLite 75 cm3 flasks | Thermo Scientific | 130193 | |
| Nunc 50 mL conical centrifuge tubes | Thermo Scientific | 339653 | |
| Chemicals: | |||
| Trizol LS | Ambion by Life Technologies | 10296028 | |
| HEPES | Fisher Scientific | BP310-500 | |
| Trizma base | Sigma | T1378-5KG | |
| Dulbecco's Modified Eagle's Medium-high glucose (DMEM) | Sigma | D6429-500ML | |
| Fetal Bovine Serum (FBS) | Sigma | F0926-50ML | |
| Penicillin-Streptomycin (P/S) | Sigma | P0781-100ML | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | |
| Dulbecco's phosphate buffered saline (DPBS) | Sigma | D8537-500ML | |
| Magnesium chloride hexahydrate (MgCl2x6H2O) | Acros Organics | AC413415000 | |
| Potassium Chloride (KCl) | Sigma | P9541-500G | |
| Nonidet P 40 (NP-40) | Fluka (Sigma-Aldrich) | 74385 | |
| Recombinant Rnasin Ribonuclease Inhibitor | Promega | N2511 | |
| Heparin sodium salt | Sigma | H3993-1MU | |
| cOmplete Mini EDTA-free protease inhibitors | Roche Diagnostics | 11836170001 | |
| Glycogen | Thermo Scientific | R0551 | |
| Water | Sigma | W4502-1L | |
| Cycloheximide | Sigma | C7698-1G | |
| Chloroform | Fisher Scientific | 194002 | |
| Dithiotreitol (DTT) | Fisher Scientific | BP172-5 | |
| Ethidium Bromide | Fisher Scientific | BP-1302-10 | |
| Ethylenediaminetetraacetic acid disodium dehydrate (EDTA) | Fisher Scientific | S316-212 | |
| Optimem | Life Technologies | 22600050 | |
| Puromycin dihydrochloride | Sigma | P8833-100MG | |
| Sucrose | Fisher Scientific | S5-3KG | |
| Trypsin-EDTA solution | Sigma | T4049-100ML | |
| Hgh Capacity cDNA Reverse Transcriptase Kit | Applied Biosystems by life technologies | 4368814 | |
| Power SYBR Green PCR Master Mix | Applied Biosystems by life technologies | 4367659 | |
| HCl | Fisher Scientific | A144SI-212 | |
| Isopropanol | Fisher Scientific | BP26324 | |
| Potassium Hydroxide (KOH) | Sigma | 221473-500G | |
| Anti-RPL11 antibody | Abcam | ab79352 | |
| Ribosomal protein S6 (C-8) antibody | Santa Cruz Biotechnology Inc. | sc-74459 | |
| 1xM199 | Sigma | M0393-10X1L | |
| Lithium cloride | Sigma | L-9650 | |
| Dimethyl sulfoxide (DMSO) | Fisher Scientific | D128-500 | |
| Gel Loading Buffer II | Thermo Scientific | AM8546G | |
| UltraPure Agarose | Thermo Scientific | 16500-100 | |
| Trichloracetic acid (TCA) | Fisher Scientific | A322-100 | |
| SuperSignal West Pico PLUS chemiluminescent substrate | Thermo Scientific | 34580 | |
| Formaldehyde | Fisher Scientific | BP531-500 | |
| Sodium Dodecyl Sulfate (SDS) | Sigma | L5750-1KG | |
| Phenylmethylsulfonyl fluoride (PMSF) | Sigma | P7626-5G | |
| RNeasy Mini kit | Qiagen | 74104 | |
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A1852-1VL | |
| Cytosine 5'-triphosphate disodium salt hydrate (CTP) | Sigma | C1506-250MG | |
| Uridine 5'-triphosphate trisodium salt hydrate (UTP) | Sigma | U6625-100MG | |
| Guanosine 5'-triphosphate sodium salt hydrate (GTP) | Sigma | G8877-250MG | |
| SP6 RNA Polymerase | NEB | M0207S | |
| Pyrophoshatase | Sigma | I1643-500UN | |
| Spermidine | Sigma | S0266-1G |
Referenzen
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Capewell, P., et al. Regulation of Trypanosoma brucei Total and Polysomal mRNA during Development within Its Mammalian Host. PLoS One. 8 (6), e67069(2013).
- Warner, J. R., Knopf, P. M., Rich, A. A multiple ribosomal structure in protein synthesis. Proceedings of the National Academy of Science, USA. 49, 122-129 (1963).
- Piccirillo, C. A., Bjur, E., Topisirovic, I., Sonenberg, N., Larsson, O. Translational control of immune responses: from transcripts to translatomes. Nature Immunology. 15 (6), 503-511 (2014).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of Inhibition of Protein Synthesis in Mammalian Cells. Science. 146 (3650), 1474-1476 (1964).
- Masek, T., Valasek, L., Pospisek, M. Polysome analysis and RNA purification from sucrose gradients. Methods in Molecular Biology. 703, 293-309 (2011).
- Gandin, V., et al. Polysome fractionation and analysis of mammalian translatomes on a genome-wide scale. Journal of Visualized Experiments. (87), (2014).
- Zuccotti, P., Modelska, A. Studying the Translatome with Polysome Profiling. Methods in Molecular Biology. 1358, 59-69 (2016).
- Chasse, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), e15(2017).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Science, USA. 100 (7), 3889-3894 (2003).
- Gandin, V., et al. nanoCAGE reveals 5' UTR features that define specific modes of translation of functionally related MTOR-sensitive mRNAs. Genome Research. 26 (5), 636-648 (2016).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Zanchin, N. I., Goldfarb, D. S. Nip7p interacts with Nop8p, an essential nucleolar protein required for 60S ribosome biogenesis, and the exosome subunit Rrp43p. Molecular Cell Biology. 19 (2), 1518-1525 (1999).
- Darnell, J. C., et al. FMRP stalls ribosomal translocation on mRNAs linked to synaptic function and autism. Cell. 146 (2), 247-261 (2011).
- Gkogkas, C. G., et al. Autism-related deficits via dysregulated eIF4E-dependent translational control. Nature. 493 (7432), 371-377 (2013).
- Robichaud, N., Sonenberg, N. Translational control and the cancer cell response to stress. Curr Opin Cell Biol. 45, 102-109 (2017).
- Gordon, B. S., Kelleher, A. R., Kimball, S. R. Regulation of muscle protein synthesis and the effects of catabolic states. International Journal of Biochemistry and Cell Biology. 45 (10), 2147-2157 (2013).
- Ishimura, R., et al. RNA function. Ribosome stalling induced by mutation of a CNS-specific tRNA causes neurodegeneration. Science. 345 (6195), 455-459 (2014).
- Petersen, C. P., Bordeleau, M. E., Pelletier, J., Sharp, P. A. Short RNAs repress translation after initiation in mammalian cells. Molecular Cell. 21 (4), 533-542 (2006).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. Proceedings of the National Academy of Science, USA. 111 (1), E203-E212 (2014).
- Bunnik, E. M., et al. Polysome profiling reveals translational control of gene expression in the human malaria parasite Plasmodium falciparum. Genome Biology. 14 (11), R128(2013).
- De Gaudenzi, J. G., Noe, G., Campo, V. A., Frasch, A. C., Cassola, A. Gene expression regulation in trypanosomatids. Essays in Biochemistry. 51, 31-46 (2011).
- Alves, L. R., Goldenberg, S. RNA-binding proteins related to stress response and differentiation in protozoa. World Journal of Biological Chemistry. 7 (1), 78-87 (2016).
- De Pablos, L. M., Ferreira, T. R., Walrad, P. B. Developmental differentiation in Leishmania lifecycle progression: post-transcriptional control conducts the orchestra. Current Opinions in Microbiology. 34, 82-89 (2016).
- Soto, M., et al. Cell-cycle-dependent translation of histone mRNAs is the key control point for regulation of histone biosynthesis in Leishmania infantum. Biochemical Journal. 379, 617-625 (2004).
- McNicoll, F., et al. Distinct 3 '-untranslated region elements regulate stage-specific mRNA accumulation and translation in Leishmania. Journal of Biological Chemistry. 280 (42), 35238-35246 (2005).
- Folgueira, C., et al. The translational efficiencies of the two Leishmania infantum HSP70 mRNAs, differing in their 3 '-untranslated regions, are affected by shifts in the temperature of growth through different mechanisms. Journal of Biological Chemistry. 280 (42), 35172-35183 (2005).
- Dumas, C., Chow, C., Muller, M., Papadopoulou, B. A novel class of developmentally regulated noncoding RNAs in Leishmania. Eukaryotic Cell. 5 (12), 2033-2046 (2006).
- Kapler, G. M., Coburn, C. M., Beverley, S. M. Stable transfection of the human parasite Leishmania major delineates a 30-kilobase region sufficient for extrachromosomal replication and expression. Molecular Cell Biology. 10 (3), 1084-1094 (1990).
- Karamyshev, A. L., Johnson, A. E. Selective SecA association with signal sequences in ribosome-bound nascent chains: a potential role for SecA in ribosome targeting to the bacterial membrane. Journal of Biological Chemistry. 280 (45), 37930-37940 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Panda, A. C., Martindale, J. L., Gorospe, M. Polysome Fractionation to Analyze mRNA Distribution Profiles. Bio Protocols. 7 (3), (2017).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Molecular Cloning. A Laboratory Manual. , Cold Spring Harbor Laboratory Press. (1989).
- Patrick, A. E., Karamyshev, A. L., Millen, L., Thomas, P. J. Alteration of CFTR transmembrane span integration by disease-causing mutations. Molecular Biology of the Cell. 22 (23), 4461-4471 (2011).
- Kleizen, B., van Vlijmen, T., de Jonge, H. R., Braakman, I. Folding of CFTR is predominantly cotranslational. Molecular Cell. 20 (2), 277-287 (2005).
- van den Elzen, A. M., Schuller, A., Green, R., Seraphin, B. Dom34-Hbs1 mediated dissociation of inactive 80S ribosomes promotes restart of translation after stress. EMBO Journal. 33 (3), 265-276 (2014).
- Morita, M., et al. mTOR Controls Mitochondrial Dynamics and Cell Survival via MTFP1. Molecular Cell. 67 (6), 922-935 (2017).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten