Method Article
Direcionamento de tióis cisteína para in Vitro site-specific glicosilação de proteínas recombinantes
Neste Artigo
Resumo
Análises bioquímicas e estruturais das proteínas glicosilados requerem relativamente grandes quantidades de amostras homogêneas. Aqui, apresentamos um método químico eficiente para site-specific glicosilação de proteínas recombinantes purificado a partir de bactérias pela segmentação reativa Cys tióis.
Resumo
Do estroma interação molécula-1 (STIM1) é um tipo-eu proteína transmembrana localizado no retículo endoplasmático (ER) e membranas de plasma (PM). ER-residente STIM1 regula a atividade de PM Orai1 canais em um processo conhecido como armazenar entrada operado de cálcio (Ca2 +), que é o principal Ca2 + sinalização processo que orienta a resposta imune. STIM1 sofre N- glicosilação pós-traducional em dois locais de Asn luminal dentro do Ca2 + sensoriamento domínio da molécula. No entanto, a bioquímica, biofísica, e efeitos biológicos de estrutura de N- glicosilados STIM1 foram mal compreendidos até recentemente devido a uma incapacidade para obter facilmente altos níveis de proteína de - glicosilados homogênea N. Aqui, descrevemos a implementação de uma em vitro abordagem química que une partes de glicose para sites de proteína específica aplicáveis para entender os efeitos subjacentes do N- glicosilação na estrutura da proteína e mecanismo. Usando a espectroscopia de ressonância magnética nuclear de solução avaliamos tanto eficiência da modificação, bem como as consequências estruturais do acessório glicose com uma única amostra. Esta abordagem pode ser adaptada facilmente para estudar as proteínas miríade glicosilados encontradas na natureza.
Introdução
Armazene operado de cálcio (Ca2 +) entrada (SOCE) é a principal via pela qual as células imunes ocupam Ca2 + do espaço extracelular para o citoplasma. Em linfócitos T, células T receptores situados na membrana plasmática (PM) ligam antígenos que ativam quinases de proteína tirosina (revistos em 1,2,3). Uma cascata de fosforilação leva à ativação da fosfolipase-γ (PLCγ), que posteriormente, Medeia a hidrólise da membrana fosfatidilinositol 4,5-bisfosfato (PIP2) em diacilglicerol e inositol 1, 4,5-triphosphate (IP3 ). IP3 é um pequeno mensageiro diffusible que liga ao IP3 receptores (IP3R) sobre o retículo endoplasmático (ER), assim, abrindo este canal receptor e permitindo Ca2 + para fluir para baixo o gradiente de concentração da emergência lúmen para o citosol (revisto em 4). Receptor de sinalização da proteína G acoplada e receptores de tirosina quinase em uma variedade de outra células não-excitáveis e excitável tipos chumbo para a mesma produção de IP3 e a ativação do IP3Rs.
Devido a Ca2 + armazenamento capacidade finita do PS, o IP3-liberação mediada e resultante aumento citosólico Ca2 + é apenas transitório; no entanto, esta depleção do ER luminal Ca2 + profundamente efeitos do estroma interação molécula-1 (STIM1), um tipo-eu transmembrana (TM) proteína encontrada principalmente no ER membrana 5,6,7. STIM1 contém um lúmen-orientado Ca2 + sensoriamento domínio composto por um par de EF-mão e estéril α-motif (EFSAM). Três domínios coiled-coil citosólica orientado são separados da EFSAM pelo domínio do TM único (revisto em 8). Em cima de ER luminal Ca2 + da prostração, EFSAM sofre uma desestabilização acoplados oligomerização 7,9 que faz com que rearranjos estruturais do TM e domínios coiled-coil 10. Estas mudanças estruturais culminaram em uma captura de STIM1 ER-PM entroncamentos 11,12,13,14 através de interações com remodelamento PM 15, 16 e de Orai1 subunidades 17,18. Orai1 proteínas são as subunidades de PM que montam para formar Ca2 + canais de de21, 19,20,22. As interações de STIM1-Orai1 nos cruzamentos de ER-PM facilitam uma aberto Ca2 + lançamento ativado Ca2 + (CRAC) canal conformação que permite a circulação de Ca2 + para o citosol das altas concentrações do no espaço extracelular. Em células do sistema imunológico, as sustentado citosólico Ca2 + elevações através de canais de CRAC induzem a Ca2 +- calmodulina/calcineurina dependente desfosforilação do fator nuclear das células T ativadas que posteriormente entra no núcleo e começa o Regulamento transcricional de genes, promovendo a ativação de células T 1,3. O processo de ativação de canal CRAC por STIM1 23,24 via induzida pelo agonista ER luminal Ca2 + depleção e a resultante sustentado citosólico Ca2 + elevação é coletivamente denominado SOCE 25. O papel vital da SOCE em células T é evidente por estudos demonstrando que hereditários mutações em ambos os STIM1 e Orai1 podem causar imunodeficiência combinada grave síndromes 3,19,26, 27. EFSAM inicia SOCE após detecção ER-luminal Ca2 + depleção através da perda de Ca2 + coordenação no canônico EF-lado, finalmente levando à desestabilização acoplados auto associação 7, 28,29.
Glycosylation é a ligação covalente e processamento de estruturas de oligossacarídeo, também conhecido como os glicanos, através de várias etapas biossintéticas no ER e Golgi (revisto em 30,32,33). Existem dois tipos predominantes de glicosilação em eucariontes: N-vinculado e O-ligada, dependendo do aminoácido específico e átomo colmatar o enlace. Na N- glicosilação, os glicanos são anexados a amida de cadeia lateral do Asn, e na maioria dos casos, a etapa de iniciação ocorre no PS como o polipeptídeo cadeia move-se para o lúmen 34. O primeiro passo do N- glicosilação é a transferência de uma núcleo de quatorze-açúcar estrutura composta de glicose (Glc), manose (homem) e N- acetilglicosamina (GlcNAc) (ou seja, Glc3homem9GlcNAc2) da emergência lipídios de membrana por uma oligosaccharyltransferase 35,36. Outras medidas, tais como a clivagem ou transferência de resíduos de glicose, são catalisadas nas urgências por glycosyltransferases e glicosidases específico. Algumas proteínas que deixe o ER e mover para o Golgi podem ser ainda mais processados 37. O- glicosilação refere-se à adição dos glicanos, geralmente para o grupo hidroxila de cadeia lateral de resíduos Ser ou Thr, e essa modificação ocorre inteiramente no complexo de Golgi 33,34. Existem várias estruturas de glicano - Oque podem ser feitas de N- acetilglicosamina, fucose, galactose e ácido siálico com cada monossacarídeo adicionado sequencialmente 33.
Enquanto nenhuma sequência específica foi identificada como pré-requisito para muitos tipos de O- glicosilação, uma sequência de consenso comum tem sido associada com a N-ligados a modificação: Asn-X-Ser/Thr/Cys, onde X pode ser qualquer aminoácido exceto Pro 33. STIM1 EFSAM contém dois desses sites - glicosilação do consenso N: Asn131-Trp132-Thr133 e Asn171-Thr172-Thr173. De fato, estudos anteriores mostraram que EFSAM pode ser N- glicosilados em células de mamíferos em Asn131 e Asn171 38,39,40,41. No entanto, estudos anteriores sobre as consequências da N- glicosilação na SOCE tem sido incongruentes, sugerindo suprimida, potencializado ou nenhum efeito por esta modificação borne-translational na SOCE ativação 38,= "xref" > 39 de40,,41. Assim, a investigação sobre as consequências de biofísicas, bioquímicas e estruturais subjacentes de EFSAM N- glicosilação é vital para compreender os efeitos regulatórios da modificação. Devido à exigência de altos níveis de proteínas homogêneas nesses experimentos em vitro , foi aplicada uma abordagem local-seletiva covalentemente anexar partes de glicose para EFSAM. Curiosamente, Asn131 e Asn171 de glicosilação causou mudanças estruturais que convergem dentro do núcleo EFSAM e aumentar as propriedades biofísicas que promovem SOCE mediada por STIM1 42.
O sistema químico de glycosyl grupos de tióis Cys tem sido bem estabelecido por uma obra seminal que primeiro demonstrou a utilidade desta abordagem de enzima livre para entender os efeitos específicos do site da glicosilação na proteína função 43 , 44. mais recentemente e com respeito a STIM1, os resíduos de Asn131 e Asn171 foram uma mutação de Cys e glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] foi usado para ligar covalentemente glicose para a enciclopédia tióis 42. Aqui, descrevemos esta abordagem que não só usa o mutagenesis de incorporar resíduos de Cys local específicos para a modificação, mas também se aplica a espectroscopia de ressonância magnética nuclear (NMR) solução para avaliar rapidamente a eficiência de modificação e estrutural perturbações como resultado da glicosilação. Notavelmente, esta metodologia geral é facilmente adaptável para estudar os efeitos de ambos O- ou N- glicosilação de qualquer recombinantes produzidos a proteína.
Protocolo
1. reação em cadeia da polimerase (PCR)-mediada mutagenesis local-dirigido para a incorporação de CIS em um vetor de expressão de animal de estimação-28a bacteriana.
- Determinar a concentração do animal de estimação-28a vector (ou seja, ADN encalhado dobro) usando um coeficiente de extinção ultravioleta (UV) de 0,020 (μg/mL) cm -1 em 260 nm.
- Sintetizar um par de primers mutagénicas complementares para cada mutação Cys tais que eu) há um mínimo de 15 nucleotídeos complementares ao modelo antes a incompatibilidade de primeira base e 15 nucleotídeos complementares ao modelo após a incompatibilidade de base final, ii) comprimento total da primeira demão não exceda 45 nucleotídeos e iii) uma guanina ou citosina está localizada na posição do primeiro e o último nucleotídeo de cada primer (tabela 1). Certifique-se de síntese da primeira demão é executada usando uma purificação de escala e cartucho de 0.025 μmol.
- Usando uma alta fidelidade DNA polimerase, configurar misturas de reação de PCR de dois 20 µ l: uma contendo o primer para a frente e o segundo contendo o primer reverso. Prepare cada mistura que contêm concentrações finais de 1 buffer de x PCR com 1,5 mM de MgCl 2, dNTPs de 0,2 mM, 0.5 cartilha μM, 0,4 μL DMSO, modelo de 1,25 ng/μL DNA, 0,02 U/μL alta fidelidade DNA polymerase.
- Ciclo termicamente as misturas separadas usando um protocolo de três etapas: 98 ° C por 30 s (desnaturação), 53-56 ° C por 30 s (recozimento), 72 ° C por 30 s kilobase(kb) -1 (extensão) do modelo de DNA. Repetir o programa de temperatura por 5 ciclos e adicionar uma etapa de extensão final a 72 ° C para 7,5 min.
- Após o inicial do PCR com primers para diante e reversos em tubos separados, combinar os produtos em um único tubo (ou seja, 40 μL de total) e continuar a reação de PCR para um adicional de 20 ciclos usando os mesmos parâmetros de ciclismo, conforme descrito na etapa 1.4.
- 15 electrophorese μL da mistura de reação de PCR em uma agarose a 1% (p/v) gel usando 0,5 x Tris, ácido acético, etileno diamina tetra acético (EDTA) executando o tampão (TAE). Como controles, electrophorese uma quantidade equivalente de modelo de DNA que não tem sido amplificado por PCR e uma alíquota de escada de DNA de referência que contém bandas de marcador tanto maiores e menor que o tamanho esperado do produto do PCR.
- Após eletroforese em 120 V por 40 min, mergulhe o gel em água contendo 0,5 brometo de etídio μg/mL e agitar durante 30 min à temperatura ambiente. Confirmar o modelo full-length tem sido amplificado pelas primeiras demão mutagénicas como um aumento da intensidade fluorescente brometo de ethidium relativo da banda amplificada em comparação com a banda de modelo de controle sob luz UV (302 nm).
- Se nenhuma amplificação é aparente, repetir a PCR depois de ajustar a temperatura do recozimento em incrementos de 0,5 ° C entre a faixa de temperatura de 53-56 ° C.
- Mediante confirmação de amplificação do modelo pelas primeiras demão mutagénicas, tratar o restante ~ 25 μL da mistura de reação de PCR com a enzima de restrição DpnI para digerir o modelo metilado DNA. Use DpnI 0,5 µ l (10 unidades) por mistura de reacção 25 μL do PCR e uma concentração final de 1 × DpnI amortecedor da reação. Incubar durante 2,5 h a 37 ° C.
- Digestão do modelo, a seguir adicionar ~ 5-10 μL da mistura digerida para 100 µ l de calor choque competentes células DH5α Esherichia coli em um tubo de microcentrifugadora 1,75 mL. Incubar a mistura de DNA de células no gelo por 60 min.
- Choque térmico da mistura de celular-DNA no tubo microcentrifuga a 42 ° C, durante 45 s em um bloco de calor seco. Após a incubação a mistura no gelo por 3 min, adicionar 900 μL de caldo temperatura Luria-Bertani (LB) para as células e transferir a suspensão total de células em um tubo de fundo redondo de estéril 14 mL.
- Incube a suspensão de eritrócitos a 37 ° C por 90 min com agitação constante a 190 rpm.
- Transferir posteriormente, a suspensão de células em um tubo de microcentrifugadora 1,75 mL e centrifugar 10.000 x g por 5 min à temperatura ambiente.
- Após a centrifugação, remover 900 μL do sobrenadante e ressuspender as células bacterianas pipetando gentil nas restantes 100 μL de lb.
- Transferir a suspensão concentrada de células resultante em uma placa LB-ágar contendo antibiótico que é seletivo para o vetor de expressão (ou seja, 60 μg/mLKanamycin). Assepticamente espalhar a suspensão uniformemente na placa de ágar e incubar durante 16 ~ h a 37 ° C.
- No dia seguinte, inocular uma única colônia da placa em 5 mL de líquido LB contendo a pressão de seleção de antiobiotic (ou seja, 60 μg/mL canamicina). Crescer a cultura líquida durante a noite a 37 ° C com agitação constante a 37 ° C.
- Isolar e purificar o plasmídeo propagado partir de células de Escherichia coli usando um kit disponível comercialmente baseado o processo de Lise alcalina 45.
- Confirmar a mutação de interesse está presente e no quadro de leitura por sequenciamento de DNA de Sanger da plasmídeo 46.
2. Uniforme 15 expressão N-etiquetou a proteína em BL21 ΔE3 Escherichia coli.
Nota: proteínas recombinantes diferentes exigem condições de expressão diferente. O seguinte é o procedimento otimizado para a expressão da proteína humana STIM1 EFSAM.
- Transformar o vetor de expressão abrigando as mutações de Cys (ou seja, animal de estimação-28a-EFSAM) em BL21 ΔE3 codão (+) calor choque pilhas competentes e placa em placas LB-ágar contendo a pressão de seleção antibiótico conforme descrito nas etapas 1.9-1.14) com o as modificações a seguir: placa diretamente uma alíquota de 150 μL da suspensão de célula total μL ~ 1.000 na placa de ágar LB sem a necessidade de concentrar as células em um tubo de microcentrifugadora por centrifugação.
- No dia seguinte, transferir assepticamente uma única colônia para um Erlenmeyer contendo 20 mL de LB suplementado com o antibiótico apropriado (ou seja, 60 canamicina para μg/mL para o animal de estimação-28a-EFSAM) de 200 mL. Crescer este líquido cultura durante a noite (ou seja, ~ 16 h) a 37 ° C, com agitação constante no ~ 190 rpm.
- No mesmo dia como passo 2.2, preparar M9 médio por 15 a expressão da proteína N-rotulado por autoclavagem 1L de M9 buffer sais (ou seja, 42 mM Na 2 HPO 4, 22mm KH 2 PO 4, 8,6 mM NaCl, pH 7,4) em um frasco de Erlenmeyer de 4 L. Legal, uma vez que filtrar uma mistura de 20% (p/v) D-glicose, 1m CaCl 2, tiamina 1 M, 1 M MgSO 4, biotina 1 mg/mL e 0,2 g/mL 15 N-NH 4 Cl através de um filtro de seringa estéril 0,2 μm para a solução salina estéril de 1 L M9 para que o concentrações finais desses componentes são 0,2% (p/v) D-glicose, 100 μM CaCl 2, 50 tiamina μM, 1 mM de MgSO 4, 1 μg/mL biotina e 1 mg/mL 15 N-NH 4 CL.
- No dia seguinte, assepticamente, transferir a cultura starter líquido durante a noite de 20 mL para um tubo cônico estéril 50 mL e centrifugar a 2.400 × g durante 15 min para as células de Pelotas.
- Após a decantação o meio LB, resuspenda o pellet de células resultante em 10 mL de meio mínimo M9 e transferir a mistura ressuspenso em 1 L de meio mínimo M9 com antibiótico (ou seja, 60 canamicina de μg/mL).
- Crescer a 1 L de M9 meio mínimo contendo a cultura bacteriana a 37 ° C e constante ~ 190 rpm tremer até a densidade óptica em 600 nm (OD600) atinge ~0.6-0.8.
- Quando é atingido o intervalo de specfified OD600, adicionar 200 μM de isopropil β-D-1-thiogalactopyranoside (IPTG) para induzir a expressão da proteína.
- Após a adição de IPTG, continuar incubando as pilhas para a expressão da proteína à temperatura ambiente com agitação constante a ~ 190 rpm para ~ 16 h (ou seja, durante a noite).
- No dia seguinte, colher as bactérias por centrifugação a 10.000 ~ × g, 4 ° C por 30 min.
- Decantar o LB e transferir o sedimento celular em um tubo cónico de 50 mL. Armazenar a pelota a-80 ° C até purificação.
3. Purificação de proteínas recombinantes de e. coli.
Nota: proteínas recombinantes diferentes exigem procedimentos de purificação distintas. O seguinte é o protocolo para 6 & #215; Purificação de EFSAM com sua Tag de corpos de inclusão expressado de construção de animal de estimação-28a.
- Manualmente homogeneizar a pelota de célula bacteriana congelado em 6m guanidina-HCl, 20 mM Tris-HCl (pH 8) e 5 mM β-Mercaptoetanol usando uma pipeta de transferência motorizado 10 mL. Adicionar aproximadamente 40 mL de HCl-guanidina por 5 mL de wet centrifugado para esta etapa.
- Seguir um 90 min incubação à temperatura ambiente com rotação constante em um forno de hibridização, centrifugar a mistura no ~ 15.000 × g, 8 ° C por 40 min separar os detritos celulares insolúveis (ou seja, sedimento) da proteína solúvel mistura (ou seja, sobrenadante).
- Adicionar 750 µ l de uma 50% (v/v) Ni 2 +-agarose ácido nitrilotriacético talão da pasta para o esclareceu lisada e incube por outro 90 min à temperatura ambiente com inversão em um forno de hibridização.
- , Posteriormente, capturar a proteína com sua tag 6 × vinculada a Ni 2 +, a recolher os grânulos de agarose em uma coluna de purificação de proteínas de fluxo de gravidade. Permitir o lisado a fluir através da coluna completamente antes de mover-se para a etapa 3.5.
- Lave os grânulos coletados três vezes com 10ml de ureia de 6 M, 20 mM Tris-HCl pH 8 e 5 mM β-Mercaptoetanol. Certifique-se de que o todo 10ml passa através da coluna antes da cada 10ml subsequente Wash.
- Eluir as proteínas em uma série de frações de 2 mL, usando ureia de 6 M, pH 20 mM Tris-HCl 8, imidazol 300 mM e 5 mM β-Mercaptoetanol com um tempo de incubação s 90 entre frações. Certifique-se de que o todo 2ml passa através da coluna antes de cada etapa de eluição subsequentes.
- Nesta fase, confirmar a proteína de interesse está presente nas fracções eluted por Coomassie azul manchada de sódio Dodecil sulfato do gel de polyacrylamide eletroforese (SDS-PAGE) usando o método de Laemmli 47. Avaliar o tamanho da proteína, a quantidade e a pureza por comparação contra bandas de marcador de peso molecular padrão que são ambos menor que e maior do que o esperado peso molecular da proteína de interesse.
- As frações da proteína eluted da piscina em uma membrana de diálise com 3.500 peso molecular Da corte e incubar em buffer redobramento 1L (20 mM Tris, 300 mM de NaCl, 1 milímetro DTT, 5 mM CaCl 2, pH 8) a 4 ° C durante a noite enquanto o buffer está sendo agitado por um magneti agitador de c.
- Depois de ~ 16 h de tempo redobramento, adicionar ~ 1 U de trombina por mg de proteína directamente para o saco de diálise e incubar a 4 ° C para um adicional h ~ 24.
- Verificar a extensão da × 6 sua clivagem marca pela coloração de azul de Coomassie de ~ 15 μL proteína alíquotas retiradas do saco de diálise antes e após a incubação com trombina que são electrophoresed em gel de poliacrilamida (SDS-PAGE) usando de desnaturação o método de Laemmli 47. Se uma mudança de ~ 2 kDa na migração é observada correspondente ao peso molecular da clivada 6 × sua marca, continuar a etapa 3.11; se uma fração de restos de proteínas não digeridas que é detectável pela coloração de azul de Coomassie, adicione ~0.2 U de trombina por mg de proteína directamente para o saco de diálise e incubar a 4 ° C para um adicional h ~ 24.
- Uso tamanho exclusão ou troca iônica da cromatografia mais purificar a proteína. Por cromatografia de troca de ânion de EFSAM, remover a solução da proteína do saco de diálise e concentrado ~ 10 vezes usando um concentrador de ultrafiltração centrífuga com 10.000 peso molecular Da corte. Posteriormente, re-diluir a solução ~ 20-fold em um buffer livre de NaCl (20 mM Tris, 5 mM CaCl 2, 1 milímetro DTT, pH 8).
- Equilibrar uma coluna de troca de ânion pré-embalados com 10 volumes de coluna do buffer livre de NaCl descrito na etapa 3.11. Equilibrar usando buffer carregado em uma seringa luer-lock, não contendo nenhuma bolha de ar empurrando a solução através da coluna em forma de gota a gota e evitando pressões de seringa que causam fluxos constantes da solução de saída da coluna. Use um trocador de ânion forte (por exemplo, quitosana agarose com grupos funcionais de quaternário de amônio).
- Carregar a proteína solução diluída em tampão isento de NaCl (etapa 3.11) na coluna, conforme descrito na etapa 3.12.
- Eluir as proteínas em um gradiente [ou seja, 0 - 60% (v/v)] do aumento de NaCl de buffer (20 mM Tris, 1 M de NaCl, 5 mM CaCl 2, 1 milímetro DTT, pH 8) usando um sistema de cromatografia líquida (FPLC) proteína rápida de duas bombas. Definir o sistema FPLC coletar frações mL de ~1-1.5 e monitorar o perfil de eluição de proteínas usando a absorvância de UV 280 nm e um fluxo taxa de 0,5 mL/min.
- Identificar os picos de eluição e frações que contêm a proteína de interesse, bem como a pureza da proteína por manchadas de azul de Coomassie SDS-PAGE gel usando o método de Laemmli 47.
- Frações de piscina mostrando > 95% (ou seja, tomadas como frações que mostram apenas uma banda única proteína no azul de Coomassie manchado géis) em um saco de diálise e troca em buffer experimental de interesse pela diálise conforme descrito na etapa 3.8.
4. Fixação química de glicose-5-MTS a proteína pela diálise.
- Prepare um 55mm solução stock de N-(β-D-glucopyranosyl)-N '-[2-methanethiosulfonyl) etil] ureia (glicose-5-MTS), dissolvendo-se 10 mg do composto em 500 μl de 100% (v/v) DMSO. Armazenar não utilizada glicose-5-MTS solubilizado em DMSO em -20 ° C. Buffer de
- preparar a amostra de proteína para a modificação por dializando 1,5 mL de proteína de ~ 60 μM em 1 L de modificação composta de MOPS 20 mM, 150 mM de NaCl, 5 mM CaCl 2 e 0,1 mM TCEP, pH 8.3. Use um corte de peso molecular de membrana de diálise que é menor que o tamanho da proteína seja modificada (por exemplo, usar um 3.500 Da corte para o ~ 17.500 Da EFSAM).
- Após 24 h a 4 ° C, transferir a amostra proveniente do saco de diálise em um tubo de microcentrifugadora. Adicionar o DMSO-solubilizado glicose-5-MTS para uma concentração final de 2 mM.
- Incubar a amostra no escuro por 1h à temperatura ambiente. Durante o período de incubação de 1 h, misturar a solução tocando suave do tubo de cada 10 min.
- Posteriormente, re-trocar a proteína para o buffer experimental final não contendo nenhum agente redutor em diálise a 4 ° C, conforme descrito na etapa 4.2 ou por ultrafiltração centrífuga. Para o procedimento de ultrafiltração, concentrar a amostra de proteína ~1.5 mL de < mL 0,5 e diluem-se posteriormente com o concentrador mesmo com o buffer de experimental. Repita essa etapa de diluição concentração duas vezes adicionais para que a troca total é um mínimo de 30 × 30 × 30 = 27,000-fold. Para EFSAM, use 20 mM Tris, 150 mM de NaCl, 5 mM CaCl 2, pH 7,5, como o experimental buffer.
- Preparar a amostra para espectrometria de massa de ionização electrospray por diálise ou ultrafiltração troca conforme descrito nas etapas 4.2 e 4.5, respectivamente, em 25mm bicarbonato de amônio ou acetato de amónio de 25 mM. Se utilizar a diálise, certifique-se de você troca pelo menos três vezes para remover quaisquer sais residuais de 2 NaCl e CaCl.
- Determinar a massa exata (ou seja, ± 1 Da) da proteína de interesse usando ionização electrospray mass espectrometria 48 , 49. Espera que cada adição covalente de glicose para um tiol Cys através do produto químico de methanethiosulfonate para adicionar a proteína 281.3 Da massa (ou seja, adicionar 360.4 para a glicose-5-MTS e subtrair Da 79.1 para o CH 3, então 2 saindo do grupo durante a ligação covalente o acessório).
5. Avaliação de solução NMR de modifperturbações de eficiência e estrutural autent.
Proteína- certifique-se a concentração do modificado é > 100 μM após a penhora de glicose e a troca de buffer final. Para EFSAM, estimar a concentração de proteína usando um coeficiente de extinção UV em 280 nm de 1,54 (mg mL – 1) cm – 1.
- a solução de proteína com 60 μM de suplemento de ácido 4,4-dimethyl-4-silapentane-1-sulfonic (DSS) para calibração de shims e pulso e 10% (v/v) D 2 O para bloquear o sinal. Para uso de sinal-ruído elevado 600 amostras μL em tubos de frequência-combinadas 5mm NMR, inserido em um espectrômetro de 600 MHz mínimo equipado com uma sonda criogênica de HCN ressonância tripla.
- Coletar espectros de HSQC N padrão 1 H - 15 como anteriormente detalhada 50 , 51 em temperatura, 1 H e 15 N larguras de varredura, transiente e incremento ajustes apropriados para a amostra particular. Para espectros EFSAM, uso de 20 ° C, 256 transientes de 1 H, 64 incrementos de cota de 15 N e 1 H e 15 N varrer larguras definidas para 1.800 e 8.000 Hz, respectivamente.
- Após a aquisição do espectro de proteína glicosilada, adicionar ditiotreitol (DTT) para a amostra NMR de um estoque de 1 M para uma concentração final de 15 mM. A DTT remove a fracção de glicose a proteína por redução do acessório mediada por dissulfeto.
- Adquirir um segundo 1 H - 15 N HSQC nestas condições reduzidas/sem modificações, fornecendo um espectro de referência para avaliar as perturbações de eficiência e estrutural de modificação causadas pelo acessório glicose.
- Processar dados NMR usando NMRPipe como detalhado anteriormente 52. Certifique-se de que o processamento minimamente inclui conversão de dados, eliminação, supressão de solvente, transformada de Fourier e visualização inicial dos espectros.
- Avaliar eficiência de modificação, medindo os valores de deslocamento químico no espectro modificado e reduzido usando o plugin NEASY na CARA 53 e intensidades de pico de Amida. Certifique-se para avaliar a intensidade do pico de Amida Cys ambos na glicose anexado e reduziram de espectros. Se a amida Cys não pode confiantemente ser localizada em ambos os espectros, use as intensidades de resíduos adjacentes a Cys como uma leitura.
- Calcular a eficiência como a intensidade de amida do espectro Cys-modificado dividida pela intensidade de amida de Cys-reduzido (isto é, DTT-tratada) espectro, multiplicado por 100:
 , onde m é a intensidade de Amida no espectro Cys-modificado e que R é a intensidade de Amida no espectro Cys-reduzido. Alternativamente, avaliar a eficiência média em vários picos de Amida:
, onde m é a intensidade de Amida no espectro Cys-modificado e que R é a intensidade de Amida no espectro Cys-reduzido. Alternativamente, avaliar a eficiência média em vários picos de Amida:
 , onde a eficiência i é a separadamente determinada eficiência calculada para cada um resíduo, eu e n é o número total de resíduos utilizados no cálculo.
, onde a eficiência i é a separadamente determinada eficiência calculada para cada um resíduo, eu e n é o número total de resíduos utilizados no cálculo. - Calcular perturbações de deslocamento químico (CSP) de diferenças entre ambos os espectros observados no 15 N e dimensões de 1 H de cada pico e normalizando para o maior 15 N gama shift química usando shift química a seguinte equação:
 , onde ΔH é a mudança de ppm na dimensão de prótons e ΔN é a mudança de ppm na dimensão nitrogênio.
, onde ΔH é a mudança de ppm na dimensão de prótons e ΔN é a mudança de ppm na dimensão nitrogênio.
Resultados
A primeira etapa desta abordagem requer a mutagênese dos resíduos de glicosilação candidato a Cys resíduos, que podem ser modificados usando a glicose-5-MTS. EFSAM tem sem resíduos de Cys endógenos, então não há considerações especiais que precisam ser feitas antes do mutagênese. No entanto, nativo Cys de resíduos devem ser uma mutação de resíduos não-modificável antes de realizar a química descrita. Ao efeito minimamente a estrutura nativa, sugerimos realizar um alinhamento global da sequência da proteína de interesse e determinação que outros resíduos são encontrados mais frequentemente na Cys endógena ões. Cys mutação para estes outros resíduos que ocorrem naturalmente em outros organismos pode ter o menor impacto na estrutura da proteína. Se o resíduo Cys endógeno é estritamente conservado, sugerimos que mudando para Ser o mais estruturalmente semelhante ao CIS. A Figura 1 mostra um típico gel de mutagénese PCR avaliando o sucesso da reação de PCR, com a amostra de DNA amplificada demonstrando several-fold intensidade mais elevada do que uma quantidade de controle de peças modelo animal de estimação-28a DNA que foi usado para o PCR. Os próximos passos incluem modelo DNA digestão e transformação em Escherichia coli para reparação do plasmídeo. Depois da propagação do plasmídeo em cultura líquida, isolamento do plasmídeo e confirmação da mutation(s) de sequenciação, o vetor mutante pode ser utilizado para a expressão da proteína. Figura 2A mostra um perfil de eluição típica de EFSAM da coluna de troca de ânion em relação ao aumentar as concentrações de NaCl. Figura 2B mostra a pureza da EFSAM em geles de SDS-PAGE Coomassie azul manchado.
Depois de adquirir a proteína pura, uma série de etapas de diálise é usada para anexar a parte activa de glicose via a reação de MTS com o tiol livre. Figura 3A mostra uma imagem da instalação típica de um volume de pequenas proteínas seladas em diálise membrana por membrana clipes e contido em um grande copo de 1 L contendo o buffer de interesse. Uma verificação inicial do sucesso da modificação pode ser realizada por espectrometria de massa. Figura 3B mostra um espectro de massa electrospray representativa de EFSAM modificada em um único Cys tiol. Após o estabelecimento do protocolo de uma proteína específica, perturbações de eficiência e estrutural de modificação podem ser avaliadas de um único uniformemente 15N-rotulado de amostra. O espectro de15N-HSQC 1H - é adquirido antes e após a adição do agente redutor DTT (Figura 4A). Cálculos de eficiência a modificação podem ser feitos através de uma comparação entre as intensidades de pico de Amida no espectro reduzido e modificada conforme detalhado na etapa de protocolo 5.8 (Figura 4B). Finalmente, quando as atribuições shift química são conhecidas por uma proteína, a DEP que correlacionam com as mudanças estruturais pode ser calculada como detalhado no passo 5.9 (Figura 4).
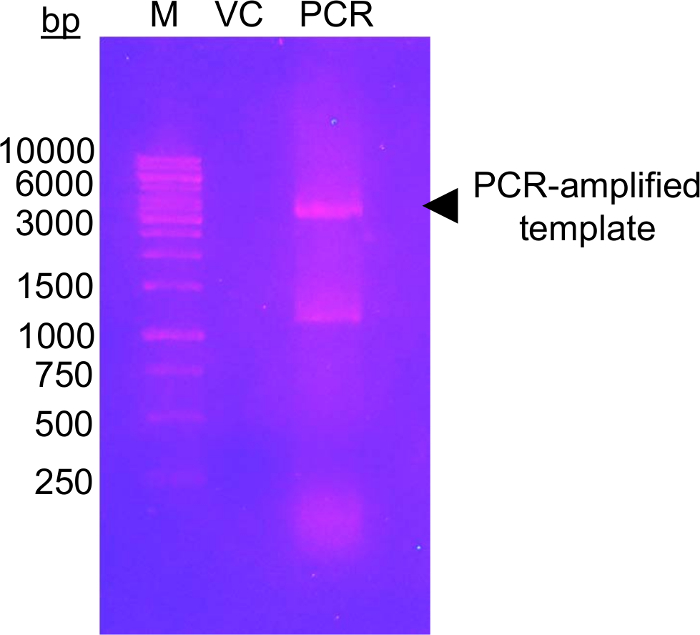
Figura 1: Gel de agarose de DNA mostrando seleção de amplificação do vetor modelo com primers mutagénicas.
A imagem mostra um gel de agarose 1,0% (p/v) com marcador de DNA (M), vetor de controle (VC) e modelo amplificado por PCR (PCR). O DNA foi separado por eletroforese em 120 V por 45 min em 0,5 x TAE buffer. Um total de 0,5 ng de VC foi carregado, equivalente ao montante de modelo carregado na faixa da PCR. O gel foi manchado usando brometo de etídio (~0.5 μg/mL) por 20 min antes da visualização sob luz UV (302 nm). O gel mostra um alto nível de DNA amplificado perto o tamanho esperado do vetor (seta preta). A segunda banda na pista do PCR correndo entre 1.000 e 1.500 bp marcador bandas provavelmente representa um produto PCR não especìfica amplificado. O nível de intensidade de DNA amplificado deve ser superior ao nível de intensidade de VC para ser considerada um sucesso. Vários outros corantes de DNA podem ser usados como alternativas menos mutagênicos, mais seguras de brometo de etídio coloração (ver, por exemplo, 57). Clique aqui para ver uma versão maior desta figura.
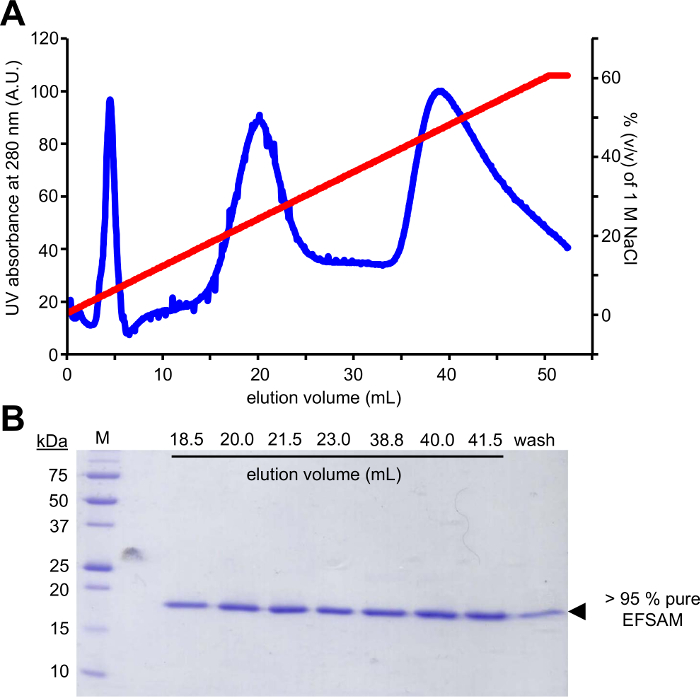
Figura 2: Pureza e purificação cromatográfica típica verifiquem para STIM1 EF-SAM.
(A) ânion troca cromatografia eluição perfil de STIM1 EF-SAM. Após a ligação manual EF-SAM para a coluna de troca de ânion (Q FF) em pH básico e baixa concentração de NaCl com uma seringa e AKTA FPLC (GE Healthcare) é usada para eluir a proteína com um gradiente de NaCl. A eluição é monitorada pela AKTA usando o sinal de nm 280 UV ao longo de um gradiente de 0-60% (v/v) de 1 M de NaCl. (B) Coomassie azul manchado SDS-PAGE gel de fracções de eluição do (A). O gel de proteína a desnaturação revela que EF-SAM elutes em dois picos principais no ~ 250mm e ~ 450 mM de NaCl. O protocolo de purificação produz > 95% puro EF-SAM, como é evidenciado pela ausência de qualquer contaminante banda aparecendo no gel Coomassie azul manchado. Clique aqui para ver uma versão maior desta figura.

Figura 3: Instalação de diálise e confirmação de em vitro proteína glycosylation.
(A) instalação de diálise típico utilizada para fixação em vitro de glicose para o tiol Cys através da reatividade MTS. A imagem mostra ~1.5 mL de proteína contida dentro da tubulação da diálise tamponada contra ~ 1 L de tampão experimental. É importante que o buffer é constantemente agitado para garantir a troca completa. A imagem mostra um tubo microcentrifuga recortado para a tubulação da diálise em excesso para evitar o afundamento do saco de diálise e danos pela barra de agitação rotativa. (B) Electrospray ionização massa espectro da proteína modificada Asn171Cys EF-SAM. Espectrometria de massa é uma abordagem conveniente e precisa para avaliar se o procedimento de modificação foi bem sucedido. Um cromatograma típico de massa é mostrado com as massas teóricas e medidas de não modificado e modificado Asn171Cys EF SAM indicado. A maioria da amostra massa corresponde a uma macromolécula que está dentro ~1.3 da massa teórica esperada de glicose-conjugados Asn171Cys EF-SAM. Os dados em (B) é replotted e modificado de 42. Clique aqui para ver uma versão maior desta figura.

Figura 4: Avaliação de solução NMR de perturbações de eficiência e estrutural de modificação de uma única amostra NMR.
(A) 1 H -15N-HSQC espectral sobreposição de glicose-conjugados Asn131Cys EFSAM antes (crosspeaks vermelha) e depois (crosspeaks preta) a adição de 15 mM DTT. A sobreposição mostra claramente várias mudanças de deslocamento químico amida de resíduo específico indicativo de ambos modificação de perturbações da proteína e estrutural. A caixa vermelha mostra a localização de Amida Asn131Cys. (B) Zoomed vista da região 1H -15N-HSQC, contendo a amida Asn131Cys. A intensidade do pico no espectro modificado (m) amida Asn131Cys é dividida pela intensidade do espectro (sem modificações) reduzida (euR) para o cálculo da eficiência (mostrado). Um cálculo da eficiência média dos vários resíduos efetuadas fornece uma melhor estimativa de eficiência, incluindo uma estimativa de erro. A eficiência média é mostrada para EFSAM de Asn131Cys com base em 5 resíduos (ou seja, 129-133). (C) Normalized shift química as perturbações causadas por uma conjugação de glicose para a proteína Asn131Cys EFSAM. O conjunto de experimentos HSQC coletados em uma única amostra antes e após suplementação com agente redutor não só fornece uma conveniente estimativa da eficiência de modificação por análise de intensidade do pico [mostrado em (B)], mas também fornece dados para o avaliação das mudanças estruturais associadas com a modificação. Conjugação de glicose faz com que as maiores perturbações localizadas perto da posição 131; no entanto, esta análise revela as perturbações que são inesperadas unicamente com base na proximidade de sequência, indicando o valor nesta análise. Os dados em (C) são replotted e modificados de 42. Clique aqui para ver uma versão maior desta figura.
| STIM1 mutaçãouma | direçãob | Sequência de DNAc | |
| Asn131Cys | para a frente | 5'-GTCATCAGAAGTATACTGTTGGACCGTGGATGAGG-3' | |
| Asn131Cys | reversa | 5'-CCTCATCCACGGTCCAACAGTATACTTCTGATGAC-3' | |
| Asn171Cys | para a frente | 5'-CCAAGGCTGGCTGTCACCTGCACCACCATGACAGGG-3' | |
| Asn171Cys | reversa | 5'-CCCTGTCATGGTGGTGCAGGTGACAGCCAGCCTTGG-3' | |
| um STIM1 ácido aminado numeração baseado na adesão de NCBI AFZ76986.1. | |||
| b O primer 'reverso' corresponde a sequência inversa complemento do primer 'Avançar'. | |||
| c O triplet sublinhado codão corresponde a mutação de Cys. | |||
Tabela 1. Constroem sequências de exemplo do oligonucleotide (primeira demão) para Asn para mutagénese Cys dentro o EFSAM de STIM1 do animal de estimação-28a.
Discussão
Proteína glycosylation é uma modificação pós-traducional onde açúcares estão covalentemente ligados a polipeptídeos principalmente através de ligações com cadeias laterais de aminoácidos. Até 50% de proteínas de mamíferos são glicosilados 54, onde as proteínas glicosilados posteriormente podem ter uma variada gama de efeitos de alterando a afinidade de ligação biomolecular, influenciando a proteína dobráveis, alterando a atividade do canal, visando moléculas para celular tráfico, para citar alguns (revistos em33) e degradação. O importante papel da glicosilação na fisiologia dos mamíferos é evidente por várias centenas de proteínas evoluídas para construir a completa diversidade de mamíferos glicano estruturas 33. Alterou o N- e O- glicosilação padrões foram associados com doença numerosos Estados incluindo próstata (aumentada e diminuição), mama (aumento e diminuição), fígado (aumentado), ovário (aumentado), pancreática (aumentado) e câncer gástrico (aumentado) 55. Além disso, glicosilação de Tau, huntingtina, α-synuclein foi encontrada para regular a toxicidade destas proteínas associadas com doenças de Alzheimer, doença de Huntington e de Parkinson 56, e ter sido um grupo de doenças congénitas da glicosilação identificadas resultantes de defeitos hereditários de enzimas que medeiam glycosylation 54. Assim, entender os efeitos biofísicos, bioquímicos e estruturais precisos de glicosilação tem o potencial de impacto tremendamente a nossa compreensão da regulação de proteína e função na saúde e na doença.
Os blocos de construção de dez açúcar que originar a diversidade de estruturas de glicano encontrados no glycome mamíferos incluem a fucose, galactose, glicose, N- acetilgalactosamina, N- acetilglicosamina, ácido glucurônico, ácido iduronic, manose, ácido siálico e xilose 33. Enquanto N- glicosilação invariavelmente links um N- acetilglicosamina açúcar diretamente para a proteína, O- glicosilação pode resultar de qualquer um dos acetylgalacotose - N, N- acetilglicosamina, xilose, fucose, glicose ou galactose covalentemente ligados ao polipeptídeo. Para começar a entender como esses açúcares imediatamente adjacentes à superfície da proteína afetam as propriedades estruturais e biofísicas, descrevemos aqui uma abordagem para anexar seletivamente de site açúcares Cys resíduos através de tióis projetados para a proteína sequência. Aqui, os resíduos que são endogenamente glicosilados são substituídos por Cys e modificado em vitro através de uma simples abordagem química. Desta forma, única e múltipla glycosylation sites poderão ser avaliados para destrinchar o contributo de cada local específico, bem como as modificações cumulativas para a dobradura e estabilidade bem como a total estrutura e função da proteína.
Recentemente, esta abordagem foi utilizada com sucesso com EFSAM individualmente e cumulativamente avaliar o papel da Asn131 e Asn171 N- glicosilação sites 42. Mutação de Cys e ligação covalente de glicose para os sites Asn131 ou Asn171 revelaram uma diminuição da Ca2 + afinidade de ligação e suprimiram a estabilidade. Quando os dois locais foram modificados simultaneamente com o acessório de glicose, a diminuição na afinidade de ligação e estabilidade foram potencializadas levando a reforçada oligomerização propensão em vitro. Estruturalmente, a abordagem aqui descrita mostrou que as modificações de Asn131 ou Asn171 mutuamente no caminho da hélice α8 núcleo localizada no domínio do SAM, imediatamente adjacente ao par EF-mão. Esta análise estrutural expõe como modificações de glicose na superfície da proteína levam a uma mudança estrutural convergente e potentiated dentro o EF-interface de mão: SAM que, finalmente, desestabiliza a proteína e aumenta a SOCE 42.
Enquanto a aplicação desta abordagem selectiva local ajudou galpão luz sobre como um monossacarídeo perto da superfície do EFSAM efeitos de dobramento, estabilidade e estrutura, este procedimento pode facilmente ser modificado para anexar mais carboidratos específicos para ER, Golgi e PM localização (ou seja, Estados de glicosilação de maturidade diferente), desde que lá é uma fonte confiável para esses carboidratos contendo grupos funcionais que pode vincular a tióis como MTS MTS. é preferível, uma vez que a modificação de tiol é reversível usando um agente redutor e um espectro de referência podem ser facilmente adquiridos. Esta abordagem também pode ser adaptada para vincular outras partes borne-translational da proteína tais como lipídios. Ao mesmo tempo, existem várias limitações dessa abordagem que devem ser considerados. Primeiro, o método baseia-se na mutação dos sítios de glicosilação que Cys que pode afetar a estrutura, a estabilidade e a dobradura mesmo na ausência de qualquer alteração de glicose. Da mesma forma, resíduos de Cys nativos na proteína devem também ser uma mutação para evitar penhora de glicose em locais não-glicosilados. Além disso, a adição de resíduos de Cys muitas vezes promove a formação de corpo de inclusão em bactérias devido a reticulação de Cys e enrolamento, fazendo a purificação mais desafiador. No entanto, esta abordagem local-seletiva de Cys-reticulação aqui descrita fornece um meio controlado para destrinchar os efeitos estruturais, bioquímicos e biofísicos dos sítios de glicosilação específico em experimentos que exigem altos níveis de proteína homogênea. Os efeitos dos resíduos de Cys não-nativas na estrutura, estabilidade e dobramento podem ser simplesmente verificados na ausência de quaisquer modificações em comparação ao selvagem-tipo proteína atribui 42. Tomados em conjunto com dados funcionais obtidos em células eucarióticas, que expressam o bloqueio de modificação versões mutantes da proteína (por exemplo, Asn-para-Ala), a abordagem descrita neste momento irá produzir novos insights sobre os mecanismos estruturais da proteína Regulamento por modificações borne-translational.
Agradecimentos
Esta pesquisa foi apoiada pela ciências naturais e engenharia Conselho de pesquisa do Canadá (05239 de P.B.S), Fundação Canadense para a inovação/Ontario Research Fund (de P.B.S), Fundação de lutar próstata câncer - Telus Ride para o pai (PBS) e Ontário Bolsa de pós-graduação (para Y.J.C. e N.S.).
Materiais
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
Referências
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L., Agostinis, P., Afshin, S. . Endoplasmic Reticulum Stress in Health and Disease. , 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados