Method Article
재조합 형 단백질의 생체 외에서 사이트별 Glycosylation에 대 한 대상 시스테인 Thiols
요약
당화 단백질의 생화학 및 구조 분석 균질 샘플 상대적으로 많은 양의 필요합니다. 여기, 우리는 효율적인 화학 반응 Cys thiols를 대상으로 박테리아에서 정화 하는 재조합 형 단백질의 사이트별 glycosylation 방법 제시.
초록
Stromal 상호 작용 분자-1 (STIM1)은-내가 막 횡단 단백질 바인딩과 그물 (ER) 및 플라즈마 막 (오후)에 위치 해 있습니다. ER-상주 STIM1 오후 Orai1 채널 알려져 주 캘리포니아2 + 신호 과정 면역 응답을 구동 하는 운영된 칼슘 (캘리포니아2 +) 항목을 저장 하는 과정에서의 활동을 통제 한다. STIM1 Ca2 + 감지의 도메인 내에서 분자 두 luminal Asn 사이트에 닢 Nglycosylation를 겪 습. 그러나,는 생화학, 생물, 그리고 N-당화 STIM1의 구조 생물학적 효과 쉽게 균질 N-당화 단백질의 높은 수준을 얻을 수 없음 인해 최근까지 제대로 이해 했다. 여기, 우리가 시험관에 화학 접근의 단백질 구조와 메커니즘에 Nglycosylation의 기본 효과 이해 하는 데 적용 하는 특정 단백질 사이트로 포도 moieties 부착 구현을 설명 합니다. 우리 모두 효율성의 수정 뿐만 아니라 단일 샘플 포도 당 첨부 파일의 구조 결과 평가 솔루션 핵 자기 공명 분광학을 사용 하 여. 이 이렇게 자연에서 발견 하는 무수 한 당화 단백질 연구를 쉽게 적응 수 있습니다.
서문
운영된 칼슘 (캘리포니아2 +) 저장 항목 (SOCE)는 면역 세포는 cytosol로 세포 외 공간에서 캘리포니아2 + 을 차지 하는 주요 통로 이다. T 세포, T 세포 수용 체 (오후) 원형질 막에 있는 항 원 단백질 tyrosine kinases ( 1,2,3검토)을 활성화 하는 바인딩합니다. 인 산화 폭포 phospholipase-γ (PLCγ)는 이후 diacylglycerol 및 이노 시 톨 1,4,5-trisphosphate (IP3에 막 phosphatidylinositol 4, 5 bisphosphate (핍2)의 가수분해를 중재의 활성화로 연결 ). IP3 은 응급실에서 농도 기온 변화도 아래로 흐름 IP3 수용 체 (IP3R) 바인딩과 그물 (ER)에 그로 인하여이 수용 체 수로 열고 허용 캘리포니아2 + 바인딩하는 작은 diffusible 메신저 cytosol ( 4에서검토)에 루멘. 수용 체 G 단백질 결합 및 다양 한 기타 고르기 및 비 고르기 셀 유형 리드에에서 티로신 키 니 아 제 수용 체에서 IP3 의 동일한 생산 및 IP3Rs의 활성화 신호.
유한 캘리포니아2 + 의 저장 용량은 응급실, IP3인-중재 릴리스 및 cytosolic 캘리포니아2 + 에서 결과 증가 일시적;만 그러나, 응급실 luminal 캘리포니아2 + 이 고갈 뿌리깊은 stromal 상호 작용 분자-1 (STIM1), 유형 효과-난 막 횡단 (TM) 단백질은 주로 응급실 막 5,,67에 발견. STIM1 루멘 지향 Ca2 + 감지 도메인을 EF-손 쌍과 마른 α-주제 (EFSAM) 구성 포함 되어 있습니다. Cytosolic 지향 코일 코일 도메인 3 개 ( 8에서 검토) 단일 TM 도메인에서 EFSAM에서 분리 된다. ER luminal 캘리포니아2 + 고갈, 시 EFSAM TM 및 코일 코일 도메인 10의 구조상 재배열을 일으키는 원인이 되는 불안정 결합 oligomerization 7,9 을 겪 습. 이러한 구조 변화 응급실 오후 접속점 11,12,13,14 오후 phosphoinositides 15, 와 상호 작용을 통해 STIM1의 트랩에 달 하다 16 및 Orai1 소 단위 17,18. Orai1 단백질은 조립 형태로 캘리포니아2 + 채널 19,20,,2122는 오후 소 단위. ER-오후 접합에서 STIM1 Orai1 상호 작용 촉진는 오픈 캘리포니아2 + 릴리스 활성화 캘리포니아2 + (CRAC) 채널 구조의 높은 농도에서 cytosol에 캘리포니아2 + 의 움직임을 가능 하 게 합니다 세포 외 공간입니다. 면역 세포에는 지속적인된 cytosolic 캘리포니아2 + 고도 CRAC 채널을 통해 유도 캘리포니아2 +-calmodulin/calcineurin 종속 dephosphorylation 이후 핵을 입력 하면 활성화 된 T-세포의 핵 요인의 고 T 세포 활성화 1,3를 홍보 하는 유전자의 transcriptional 규칙을 시작 합니다. STIM1 23,24 주 작동 근 유도 응급실 luminal 캘리포니아2 + 고갈 통해 결과 지속적인된 cytosolic 캘리포니아2 + 고도로 CRAC 채널 활성화의 과정은 공동으로 SOCE 25불린다. T-세포에 SOCE의 중요 한 역할은 STIM1와 Orai1에서 상속 변이 심한 결합 된 면역 결핍 증후군 3,,1926, 를 발생할 수 있습니다 입증 하는 연구에 의해 분명 하다 27. 정식 EF-손에서 궁극적으로 자체 협회 불안 결합 7, 로 이어지는 Ca2 + 조정의 손실을 통해 응급실 luminal 캘리포니아2 + 고갈 감지 후 SOCE를 시작 하는 EFSAM 28,29.
Glycosylation 공유 첨부 파일 올리고 당 구조, 일컬어 glycans, ER 및 Golgi ( 30,,3233검토) 다양 한 생 합성 단계를 통해의 처리입니다. 진핵생물에서 glycosylation의 두 가지 주된 유형이 있다: N-연결 된 및 O-연결, 특정 아미노산 및 atom 가교 결합에 따라. N-glycosylation에서 glycans, Asn의 사이드 체인 아 미드에 연결 그리고 개시 단계 응급실에서 발생 하는 대부분의 경우에는 polypeptide 사슬 루멘 34로 이동. N-glycosylation의 첫 번째 단계는 포도 당 (Glc)만 노 오 스 (남자), 및 N-acetylglucosamine (GlcNAc) (예: Glc3남자9GlcNAc2)는 ER에서 만들어진 14 설탕 코어 구조 이전 oligosaccharyltransferase 35,36여 막 지질. 분열 또는 포도 당 잔류물의 양도 등의 더 단계는 특정 glycosidases와 glycosyltransferases 응급실에서 촉매. 응급실을 두고는 골으로 이동 하는 일부 단백질은 처리 37추가 될 수 있습니다. Oglycosylation Ser 또는 Thr 잔류물의 사이드 체인 수 산 기 그룹에 일반적으로 glycans의 추가를 참조 하 고이 수정 골 복잡 한 33,34에서 전적으로 발생 합니다. Nacetylglucosamine, fucose, 갈 락 토스, 구성 될 수 있는 여러 오-glycan 구조 이며 각 단 당 류와 sialic acid 33순차적으로 추가.
아니 특정 순서 Oglycosylation의 많은 종류에 대 한 사전 요구 사항 확인 되었습니다 동안 일반적인 합의 순서 N와 연결 되었습니다-수정 연결: Asn-X-Ser/Thr/Cys, X 어떤 아미노산을 될 수 있는 프로 33제외 하 고. STIM1 EFSAM 두 이러한 합의 N-glycosylation 사이트 포함: Asn131-Trp132-Thr133와 Asn171-Thr172-Thr173. 실제로, 이전 연구 EFSAM Asn131 및 Asn171 38,39,,4041에서 포유류 세포에서 N-당화 수 나타났습니다. 그러나, 이전 연구의 SOCE에서 Nglycosylation의 결과 되었다 합동, 제안 억제, potentiated 또는 SOCE 활성화 38, 에이 포스트 번역 상 수정에 의해 영향을 주지 않습니다"외부 참조" = > 39,,4041. 따라서, EFSAM Nglycosylation의 기본, 생화학, 생물 및 구조 결과 대 한 연구는이 수정의 규제 효과 이해 하는 중요 한. 이러한 생체 외에서 실험에 균질 단백질의 높은 수준에 대 한 요구 사항으로 인해 포도 당 moieties EFSAM covalently 연결할 사이트 선택적 접근 방식을 적용 했다. 흥미롭게도, Asn131 및 Asn171 glycosylation EFSAM 코어 내 수렴을 홍보 STIM1이 중재 SOCE 42생물 속성 강화 구조 변경 발생 합니다.
Cys thiols glycosyl 그룹의 화학 첨부 파일 정액 작품 처음 시연 단백질 기능 43 , glycosylation의 사이트 효과 이해 하 고이 효소 무료로 접근의 유틸리티에 의해 잘 설립 되었습니다. , 44. 최근에 STIM1에 관하여 Asn131과 Asn171 잔류물 했다 Cys을 돌연변이 glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] covalently 무료 thiols 42에 포도 당을 연결 하는 데 사용 되었다. 여기, 우리 뿐만 아니라 mutagenesis 통합 수정에 대 한 사이트 특정 Cys의 잔류물을 사용 하지만 빠르게 수정 효율성 및 구조적 평가 솔루션 핵 자기 공명 (NMR) 분광학은 또한 적용 됩니다이 이렇게 설명 섭은 glycosylation 결과로입니다. 특히,이 일반적인 방법론 중 O-의 효과 연구에 쉽게 적응 또는 N-glycosylation의 recombinantly 단백질을 생산.
프로토콜
1. 중 합 효소 연쇄 반응 (PCR)-세균 애완 동물-28a 식 벡터에 Cys의 설립에 대 한 사이트 이동 mutagenesis 중재.
- 260에서 0.020 (μ g/mL) cm -1의 자외선 (UV) 소멸 계수를 사용 하 여 애완 동물-28a 벡터 (즉, 두 배 좌초 된 DNA)의 농도 결정 nm.
- 합성 각 Cys 돌연변이 대 한 보완 돌연변이 뇌관의 쌍 난) 이전에 1 루 불일치 및 15 뉴클레오티드 보완 후 서식 파일에 서식 파일을 보완 15 뉴클레오티드의 최소 있다는 마지막 기본 불일치, ii) 45 뉴클레오티드, 및 iii 총 뇌관 길이 초과 하지 않는) 구 아닌 또는 시 토 신은 각 뇌관 (표 1)의 첫 번째 및 마지막 뉴클레오티드 위치에 위치. 뇌관 종합 0.025 μmol 규모 및 카트리지 정화를 사용 하 여 수행 됩니다 확인 하십시오.
- 고 충실도 DNA 중 합 효소를 사용 하 여 두 개의 20 µ L PCR 반응 혼합물을 설정: 앞으로 뇌관과 두 번째가지고 있는 역방향 뇌관을 포함. 1.5 m m MgCl 2, 0.2 m m dNTPs, 0.5 μ M 뇌관, 0.4 μ DMSO, 1 x PCR 버퍼의 최종 농도 포함 하는 각 혼합물 준비 1.25 ng/μ 템플릿 DNA, 0.02 U/μ 고 충실도 DNA 중 합 효소.
- 열 3 단계 프로토콜을 사용 하 여 별도 혼합물을 주기: 98 ° C 30에 대 한 s (변성 시키기), 53-56 ° C 30에 대 한 s (어 닐 링), 30 s kilobase(kb) -1 (확장) 서식 파일 DNA의 72 ° C. 5 주기 위한 온도 프로그램을 반복 하 고 추가 7.5 분에 대 한 최종 72 ° C 확장 단계
- 별도 튜브에 앞으로 역 뇌관으로 초기 PCR 후 단일 튜브 (즉 40 μ 총)으로 제품을 결합 하 고 계속 1.4 단계에서 설명한 대로 동일한 자전거 매개 변수를 사용 하 여 추가 20 사이클에 대 한 PCR 반응.
- 1% (w/v) agarose에 PCR 반응 혼합물의 electrophorese 15 μ 젤 0.5 x Tris, 아세트산, 에틸렌 diamine tetra 아세트산 (EDTA) 실행 버퍼 (태)을 사용 하 여. 컨트롤 템플릿 하지 PCR에 의해 증폭 된 DNA의 상응 하는 금액 및 마커 밴드 모두 큰 고 예상된 PCR 제품의 크기 보다는 더 적은 포함 하는 참조 DNA 사다리의 약 수를 electrophorese.
- 40 분 120 V에서 전기 이동 법, 후 잠수함 물 0.5 μ g/mL ethidium 평범한 사람 및 실 온에서 30 분 동안 동요에에서 젤. 전체 길이 서식 파일은 증폭 되었다 돌연변이 뇌관에 의해 증가으로 UV 빛에서 컨트롤 템플릿 밴드에 비해 증폭된 밴드의 상대 ethidium 평범한 사람 형광 강도 확인 (302 nm).
- 아무 증폭 명백한 경우 0.5 ° C 증가 53-56 ° C 온도 범위에서 어 닐 링 온도 조정 후 PCR을 반복.
- 돌연변이 뇌관에 의해 템플릿의 증폭의 확인, 치료 DpnI 제한 효소 소화 methylated 템플릿 DNA PCR 반응 혼합물의 나머지 ~ 25 μ. DpnI를 사용 하 여 25 μ PCR 반응 혼합물 및 1 × DpnI 반응 버퍼의 마지막 농도 0.5 µ L (10 단위)에서. 2.5 h 37 ° c.에 대 한 품 어
- 열 100 µ L 소화 혼합물의 5 ~ 10 μ 충격 1.75 mL microcentrifuge 튜브에 유능한 DH5α Esherichia 대장균 세포 추가 템플릿 소화, 다음. 60 분에 대 한 얼음에 세포 DNA 혼합물을 품 어
- 열 충격 45 42 ° C에서 microcentrifuge 튜브에 세포 DNA 혼합물 건조 열 블록에 s. 3 분 동안 얼음에 혼합물, 잠복기 후 실내 온도 Luria Bertani 국물 (파운드)의 900 μ 셀 추가 하 고 총 세포 현 탁 액 살 균 14 mL 라운드-하단 튜브로 전송.
- 190 rpm에서 일정 한 동요와 90 분 동안 37 ° C에 세포 현 탁 액을 품 어.
- 이후, 세포 현 탁 액 1.75 mL microcentrifuge 튜브 및 실 온에서 5 분 동안 10000 x g에서 원심 분리기로 다시 전송.
- 원심, 후는 상쾌한의 900 μ를 제거 하 고 부드러운 pipetting 파운드의 나머지 100 μ에 의해 세균성 세포 resuspend
- 전송 식 벡터 (즉 60 μ g/mLKanamycin)에 대 한 선택은 항생제를 포함 하는 파운드-한 천 격판덮개에 결과 집중된 세포 현 탁 액. Aseptically 한 천 격판덮개에 균등 하 게 정지를 확산 하 고 37 ~ 16 h에 대 한 품 어 ° c.
- 다음 날, antiobiotic 선택 압력 (예: 60 μ g/mL 대)를 포함 하는 액체 LB의 5 mL에 접시에서 단일 식민지를 예방. 성장 37에 지속적인 동요를 37 ° C에서 하룻밤 액체 문화 ° c.
- 격리 및 45의 알칼리 성 세포의 용 해 절차 따라 상업적으로 사용할 수 있는 키트를 사용 하 여 대장균 세포에서 전파 된 플라스 미드 정화.
- 관심의 돌연변이 존재 하 고 적절 한 독서 프레임 46 플라스 미드 생어 DNA 연속 확인.
2. 15 BL21 ΔE3 대장균에 N 표시 된 단백질 식 통일.
참고: 다른 재조합 단백질 필요 다른 식을 조건. 다음은 인간 STIM1 EFSAM 단백질의 표현에 대 한 최적화 절차.
- BL21 ΔE3 codon (+) 열 충격 유능한 세포에 Cys 변이 (즉: 애완 동물-28a-EFSAM)을 품고 식 벡터를 변환 하 고 1.9-1.14 단계에 설명 된 대로 항생제 선택의 압력을 포함 하는 파운드-한 접시에 접시)와 다음 수정: 직접 원심 분리에 의해 microcentrifuge 튜브에 세포를 집중 하지 않아도 파운드-한 접시에 ~ 1000 μ 총 세포 현 탁 액에서 약 150 μ 수 접시.
- 다음 날, aseptically 200 mL 삼각 플라스 크를 포함 하는 적절 한 항생제 (예: 60 μ g/mL 대 애완 동물-28a-EFSAM에 대 한) 보충 파운드의 20 mL에 단일 식민지 전송. 이 액체 시 동기 문화 하룻밤 (즉 ~ 16 h) 37 ° C에서 일정 한 동요에 성장 ~ 190 rpm.
- M9 준비 단계 2.2로 같은 날 15 압력가 마로 소독 하 여 N 표시 된 단백질 표정 매체 1 L m 9의 4 L 삼각 플라스 크에 소금 (42 m m 나 2 HPO 4, 22 mM KH 2 포 4, 8.6 m m NaCl, pH 7.4)를 버퍼. 일단, 20% (w/v) D-포도 당, 1 M CaCl 2, 1 M 티 아민, 1 M MgSO 4, 1 mg/mL biotin, 0.2 g/mL 15 N-NH 4 Cl의 혼합물 1 L 살 균 M9 소금 솔루션으로 0.2 μ m의 무 균 주사기 필터를 통해 필터링 되도록는 이러한 구성의 최종 농도 0.2% (w/v) D-포도 당, 100 μ M CaCl 2, 50 μ M 티 아민, 1mm MgSO 4, 1 μ g/mL biotin과 1 mg/mL 15 N-NH 4 cl.
- 다음 날, aseptically 50 mL 살 균 원뿔 튜브와 작은 셀을 15 분 동안 2400 × g에서 원심 분리기로 20 mL 하룻밤 액체 시 동기 문화를 전송.
- 파운드 매체 decanting, 후 resuspend M9 최소 매체의 10 mL에 결과 셀 펠 릿 및 항생제 (예: 60 μ g/mL 대)와 M9 최소 매체의 1 L에 resuspended 펠 릿 혼합 전송.
- 성장 포함 하는 37 ° C에서 600 nm (OD600)에 도달에서 광학 밀도까지 떨고 ~ 190 rpm 상수 세균 스타터 문화 M9 최소 매체의 1 L ~0.6-0.8.
- Specfified OD600 범위에 도달 하면 추가 이소프로필 β-D-1-thiogalactopyranoside 단백질 표정 유도 (IPTG)의 200 μ M.
- IPTG 추가 후 ~ 190 rpm ~ 16 h (즉, 하룻밤)에 대 한 지속적인 동요와 주위 온도에 단백질 식 세포를 배양 하는 것을 계속.
- 다음 날, 수확 ~ 10000 × g, 30 분에 4 ° C에서 원심 분리 하 여 박테리아
- LB를 가만히 따르다와 50 mL 원뿔 튜브로 셀 펠 릿을 전송. 정화까지-80 ° C에서 펠 릿 저장.
3. 대장균에서 재조합 형 단백질의 정화.
참고: 다른 재조합 단백질 별개 정화 절차 필요. 다음은 6 & # 215에 대 한 프로토콜; 애완 동물-28a 구문에서 표현 된 포함 시체에서 그의 태그 EFSAM 정화.
- 수동으로 6 M guanidine-HCl, 20 mM Tris HCl (pH 8)에 냉동된 세균성 세포 pellet 균질 및 5mm β-mercaptoethanol 동력된 10 mL 전송 피 펫을 사용 하 여. Guanidine-HCl이이 단계에 대 한 젖은 셀 펠 릿의 5 mL 당 약 40 mL를 추가.
- 다음는 90 분 부 화 교 잡 오븐에 일정 회전 주위 온도에 원심 ~ 15000 × g, 수용 성 단백질에서 불용 성 세포 파편 (즉 펠 릿)을 40 분 동안 8 ° C에 혼합물 혼합 (즉, 표면에 뜨는).
- 50% (v/v) Ni 2 +의 추가 750 µ L-nitrilotriacetic 산 agarose 구슬 슬러리는 명확히 lysate와 교 잡 오븐에 반전으로 실 온에서 또 다른 90 분 동안 품 어.
- 이후, 6 × 태그 그의 단백질을 중력 흐름 단백질 정화 열에서 agarose 구슬 수집 하 여 Ni 2 +에 바인딩된 캡처합니다. Lysate 3.5 단계로 이동 하기 전에 완전히 열을 통해 흐름을 허용.
- 수집된 구슬 6 M 요소, 20 mM Tris HCl pH 8과 5 m m β-mercaptoethanol의 10 mL로 세 번 세척. 전체 10 mL 각 후속 10 mL 이전 열 통과 확인 워시
- 6 M 요소, 20 mM Tris HCl pH 8를 사용 하 여 2 mL 분수의 시리즈에 있는 단백질을 elute 300mm 이미, 그리고 5mm β-mercaptoethanol 분수 사이 90 s 보육 시간. 전체 2 mL 각 후속 차입 단계 전에 열 통과 확인 하십시오.
- 가이 단계에서 관심사의 단백질은 Coomassie 파란 얼룩이 나트륨 라우릴 황산 polyacrylamide 젤 전기 이동 법 (SDS-PAGE) 47 Laemmli 방법을 사용 하 여 의해 eluted 분수에 확인. 둘 다 표준 분자량 마커 밴드에 대 한 단백질 크기, 양 및 순수성을 비교 하 여 평가 및 보다 적게 보다는 예상된 분자량 보다 큰 관심사의 단백질의.
- 컷오프 3500 다 분자 무게와 투 석 막에 eluted 단백질 분수 수영장 및 4 ° C에서 1 L refolding 버퍼 (트리 스, 300 m m NaCl, 1 mM DTT, 5mm CaCl 2, pH 8 m 20 m)에서 품 어 하룻밤 동안 버퍼는 자석과 의해 자극 되는 c 활동가.
- ~ 16 h refolding 시간 후 ~ 1 투 석 가방에 직접 단백질의 밀리 그램 당 트 롬 빈의 U를 추가 하 고 추가 ~ 24 h. 4 ° C에서 품 어
- 는 Coomassie 파란 얼룩이 ~ 15 μ 단백질 aliquots 트 롬 빈과 보육 전후 투 석 가방에서 가져온 polyacrylamide 젤 (SDS 페이지)를 사용 하 여 변성 시키기에 electrophoresed는의 지에 의해 그의 태그 분열 6 ×의 범위를 확인 Laemmli 47의 방법입니다. 마이그레이션에 ~ 2 kDa 변화 그의 태그 쪼개진된 6 ×의 분자량에 해당 관찰, 3.11; 단계로 계속 경우 일부 소화 되지 않은 단백질 남아 있다 Coomassie 파란 얼룩에 의해 감지 되, 투 석 가방에 직접 단백질의 밀리 그램 당 트 롬 빈의 ~0.2 U를 추가 하 고 추가 ~ 24 h. 4 ° C에서 품 어
- 더 사용 크기 또는 이온 교환 크로마토그래피는 단백질 정화. EFSAM의 음이온 교환 크로마토그래피, 투 석 가방과 집중에서 단백질 솔루션 제거 ~는 10000 Da 분자량 컷오프로 한 원심 집중 장치를 사용 하 여 사진. 그 후, 다시 희석 솔루션 ~ 20-fold NaCl 무료 버퍼 (20 mM Tris, 5mm CaCl 2, 1mm DTT, pH 8)에.
- Prepacked 음이온 교환 열 단계 3.11에서에서 설명한 NaCl 무료 버퍼의 10 열 볼륨을 equilibrate. Equilibrate 로드 luer 잠금 주사기 dropwise 방식과 열 종료 솔루션의 꾸준한 흐름을 일으키는 원인이 되는 피하 주사기 압력 열을 통해 솔루션을 추진 하 여 아무 공기 방울을 포함 하는 버퍼를 사용 하 여. (예: 4 급 암모늄 기능 그룹으로 가교 된 agarose) 강한 음이온 교환기를 사용 하 여.
- NaCl 무료 (단계 3.11) 버퍼 열에 희석 단계 3.12에서에서 설명한 단백질 솔루션 로드.
- 그라데이션에 단백질을 elute [즉, 0-60% (v/v)] NaCl 증가의 버퍼 (20 mM Tris, 1 M NaCl, 5mm CaCl 2, 1mm DTT, pH 8) 2-펌프 고속 단백질 액체 착 색 인쇄기 (FPLC) 시스템을 사용 하 여. FPLC 시스템 ~1-1.5 mL 분수를 수집 하 고 모니터링 UV 280 nm 흡 광도 사용 하 여 단백질 차입 프로필을 설정 하 고 흐름 속도 0.5 mL/분
- 차입 봉우리와 분수 뿐만 아니라 단백질 순도 Coomassie 파란 얼룩이 SDS 페이지 젤 Laemmli 47의 방법을 사용 하 여 관심사의 단백질에 포함 된 식별.
- 풀 분수 보여주는 > 95% (즉 분수 Coomassie 파란 얼룩이 젤에만 단일 단백질 밴드를 표시로 찍은) 투 석 가방 및 투 석 단계 3.8에서에서 설명한 대로 의해 관심의 실험적인 버퍼 교환으로.
4. 투 석 하 여 단백질을 포도 당-5-MTS 화학 부착.
- 준비 55 m m a N-(β-D-glucopyranosyl)-N의 재고 솔루션 '-[2-methanethiosulfonyl) 에틸] 10 mg의 100% (v/v) DMSO의 500 μ에 화합물을 용 해 하 여 우 레 아 (포도 당-5-MTS). 사용 하지 않는 포도 당-5-MTS-20에서 DMSO에서 solubilized 저장 ° c.
- 준비 수정 1 리터에 ~ 60 μ M 단백질의 dialyzing 1.5 mL 수정에 대 한 단백질 견본 버퍼 20mm MOPS, 150 mM NaCl, 5mm CaCl 2와 0.1 m m TCEP, pH 8.3의 구성. 사용 하는 투 석 막 분자량 컷오프는 수정 되 고 단백질의 크기 보다 작은 (예: 17500 ~ 다 EFSAM에 대 한 3500 다 구분 사용).
- 후 24 h 4 ° C에서 microcentrifuge 튜브로 투 석 가방에서 샘플을 전송. 2mm의 최종 농도 DMSO solubilized 포도 당-5-MTS 추가.
- 주위 온도에서 1 h에 대 한 어둠 속에서 샘플을 품 어. 1 시간 잠복기 동안 혼합 솔루션 튜브의 부드러운 드려서 10 분 마다
- 이후에, 다시 4.2 또는 원심 한 단계에서 설명한 대로 4 ° C에서 투 석 하 여 아무 환 원제를 포함 하는 최종 실험 버퍼에 단백질 교환. 적용 절차에 대 한 집중 ~1.5 mL 단백질 샘플을 < 0.5 mL 이후에 실험 버퍼와 같은 집중에 희석. 반복이 농도 희석 단계 2 추가 번 총 exchange는 최소 30 × 30 × 30 = 27,000-fold. EFSAM, 20 mM Tris, 150 mM NaCl, 5mm CaCl 2, 실험적인 버퍼 pH 7.5 사용.
- 투 석 또는 한 exchange 25 m m 염화 중 탄산염 또는 25 mM 암모늄 아세테이트로 각각 4.2와 4.5, 단계에서 설명한 대로 의해 분무 이온화 질량 분석에 대 한 샘플을 준비 합니다. 투를 사용 하 여, 모든 잔여 NaCl와 CaCl 2 소금 제거를 적어도 세 번을 교환 확인.
- 정확한 질량을 결정 (예: ± 1 다) 분무 이온화를 사용 하 여 관심사의 단백질의 대량 분석 48 , 49. Methanethiosulfonate 화학 281.3 다 단백질에 추가를 통해 Cys thiol 각 공유 포도 당 뿐만 아니라 기대 질량 (360.4 다 포도 당 5 MTS에 대 한 추가 및 공유 하는 동안 그룹 2 떠나 그래서 채널 3 79.1 다 빼기 첨부 파일).
5. Modif 솔루션 NMR 평가ication 효율성과 구조 섭.
- 단백질은 > 포도 당 첨부 파일 및 최종 버퍼 교환 후 100 μ M. EFSAM에 대 한 추정 단백질 농도 UV 소멸 계수를 사용 하 여 280에서 1.54 (mg mL - 1) cm - 1 nm.
- 메우는 및 펄스 교정 및 10% (v/v) D 4,4-dimethyl-4-silapentane-1-sulfonic 산 (DSS)의 보충 60 μ M와 단백질 해결책 2 O 신호 잠금에 대 한. 높은 신호-잡음 사용 주파수 일치 5mm NMR 튜브에 600 μ 샘플, 트리플 공명 HCN 극저온 프로브를 갖춘 최소 600mhz 분석기에 삽입.
- 이전 상세한 50 , 51 온도, 1 H와 15 N 청소 폭, 변이 및 증가에서 표준 1 H-15 N HSQC 스펙트럼 수집 특정 샘플에 대 한 적합 한 설정입니다. EFSAM 스펙트럼, 사용 20 ° C, 256 1 H 과도, 64 15 N 차원 증가 1 H 15 N 그런 후 폭 8, 000와 1800 헤르쯔, 각각 설정.
- 당화 단백질 스펙트럼의 인수에 따라 추가 dithiothreitol (DTT) NMR 샘플 15 m m의 최종 농도를 1 M 주식에서. DTT 이황화 중재 첨부의 감소에 의해 단백질에서 포도 당 moiety를 제거 합니다.
- 두 번째 1 H-15 N HSQC 수정 효율성 및 구조적 섭 포도 당 첨부 파일에 의해 발생을 평가 하기 위해 참조 스펙트럼 제공이 감소/수정 되지 않은 조건 하에서 취득.
- 이전 52 자세한 NMRPipe를 사용 하 여 NMR 데이터 처리. 최소한 처리 데이터 변환, 단계적으로, 용 매 억제, 푸리에 변환 및 스펙트럼의 초기 시각화 포함 되어 있는지 확인 하십시오.
- 아 미드 피크 농도 및 카라 53에 NEASY 플러그인을 사용 하 여 수정 및 감소 된 스펙트럼에 화학 변화 값을 측정 하 여 평가 수정 효율성. 모두는 포도 당에 연결 하 고 스펙트럼을 감소 Cys 아 미드의 피크 강도 평가를 확인 합니다. Cys 아 미드 두 스펙트럼에 안정적으로 있을 수 없습니다, 경우는 Cys에 인접 한 잔류물의 농도 사용 하 여 한 판독으로.
- Cys 감소 (즉, DTT 취급) 스펙트럼에서 아 미드의 강도 나눈 Cys 수정 스펙트럼에서 아 미드의 농도로 효율 계산 100 곱한:
 , 어디 내가 M Cys 수정 스펙트럼에 아 미드의 농도 이며 나 R Cys 감소 스펙트럼에 아 미드의 농도. 또는, 여러 아 미드 봉우리 위에 평균 효율 평가:
, 어디 내가 M Cys 수정 스펙트럼에 아 미드의 농도 이며 나 R Cys 감소 스펙트럼에 아 미드의 농도. 또는, 여러 아 미드 봉우리 위에 평균 효율 평가:
 효율성 i가 별도로 결정된 효율성 각 계산, 잔류물, 나, 그리고 n 계산에 사용 하는 잔류물의 총 수입니다.
효율성 i가 별도로 결정된 효율성 각 계산, 잔류물, 나, 그리고 n 계산에 사용 하는 잔류물의 총 수입니다. - 계산 화학 변화 차이 두 스펙트럼 15 N과 각 피크의 1 H 치수에서 관찰 하 고 더 큰 15 N 화학 변화 범위 사용에 대 한 정상화에서 화학 이동 섭 (CSP)는 다음 방정식:
 , 어디 ΔH 양성자 차원에 ppm 변화 이며 ΔN 질소 차원에서 ppm 변화.
, 어디 ΔH 양성자 차원에 ppm 변화 이며 ΔN 질소 차원에서 ppm 변화.
결과
이 방법의 첫 번째 단계 하려면 포도 당-5-MTS. EFSAM를 사용 하 여 수정할 수 있는 Cys의 잔류물을 후보 glycosylation 잔류물의 mutagenesis 아무 특별 한 고려 사항 이전에 만들어질 필요가 없는 생 Cys 잔류물에는 mutagenesis입니다. 그러나, 네이티브 Cys 잔류물 설명된 화학을 수행 하기 전에 수정 불가능 잔류물을 변경 해야 합니다. 최소한 기본 구조 효과, 우리 관심사의 단백질의 글로벌 시퀀스 정렬 수행 제안 하 고 가장 자주 생 Cys에서 position(s) 발견 다른 잔류물 확인. 다른 유기 체에서 자연스럽 게 발생 하는 이러한 다른 잔류물에 Cys 돌연변이 단백질 구조에 최소 영향을 할 수 있습니다. 생 Cys의 잔류물이 엄격 하 게 보존 하는 경우는 가장 구조적으로 유사한 Cys Ser을 변경 하는 것이 좋습니다. 그림 1 에서는 일반적인 PCR mutagenesis 젤 unamplified 템플릿 애완 동물-28a DNA는 PCR를 위해 사용 되었다의 제어 금액 보다 몇 높은 강도 보여 주는 증폭 된 DNA 샘플을 PCR 반응의 성공 평가. 다음 단계는 서식 파일 DNA 소화 및 플라스 미드 수리에 대 한 대장균 으로 변환 포함 됩니다. 액체 문화, 플라스 미드 분리 및 연속 mutation(s)의 확인에 플라스 미드 전파, 후 돌연변이 벡터 단백질 식에 사용할 수 있습니다. 그림 2A 는 NaCl 농도 증가 상대적인 음이온 교환 열에서 EFSAM의 전형적인 차입 프로필을 보여 줍니다. 그림 2B Coomassie 파란 얼룩이 SDS 페이지 젤에 EFSAM의 순수성을 보여줍니다.
순수 단백질, 취득 후 일련의 투 석 단계 무료 thiol와 MTS 반응을 통해 포도 당 moiety를 연결 하는 데 사용 됩니다. 그림 3A 작은 단백질 볼륨의 전형적인 설정의 이미지는 투 석 막으로 막 클립 봉인 관심의 버퍼를 포함 하는 큰 1 리터 비 커에 포함 된 보여줍니다. 질량 분석에 의해 수정 성공의 초기 검사를 수행할 수 있습니다. 그림 3B 단일 Cys thiol에 EFSAM의 대표 분무 질량 스펙트럼을 보여준다. 특정 단백질에 대 한 프로토콜의 설립, 다음 수정 효율성 및 구조적 섭 평가 될 수 있다 단일에서 균일 15N 표시 된 샘플. 1H-15N HSQC 스펙트럼 전과 원제 DTT (그림 4A)의 추가 후에 취득 된다. 아 미드 피크 농도 프로토콜 단계 5.8 (그림 4B)에서 설명한 대로 수정 및 감소 된 스펙트럼의 비교를 통해 수정 효율의 계산을 만들 수 있습니다. 마지막으로 때 화학 변화 과제는 단백질 구조 변경과 상관에서 상세한 단계 5.9 (그림 4C)로 계산할 수 있다 Csp에 대 한 알려져 있다.
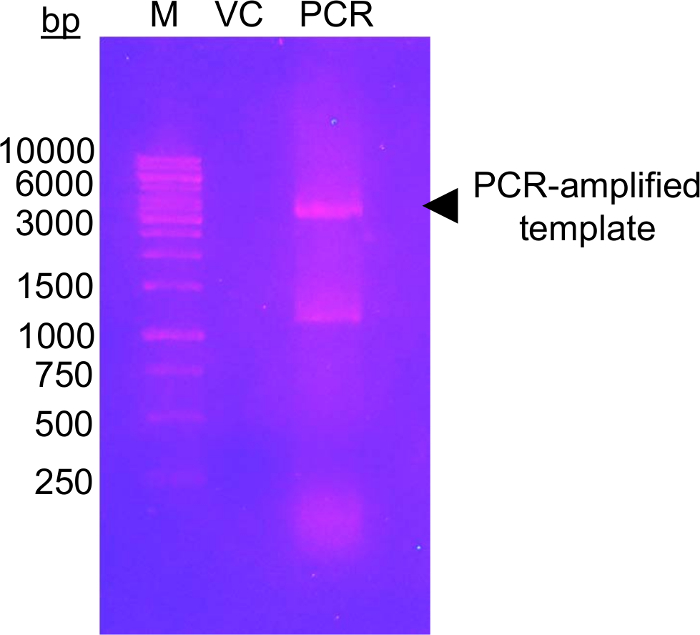
그림 1: DNA agarose 젤 돌연변이 뇌관 서식 파일 벡터의 증폭 검사를 보여주는.
이미지 표시 1.0% (w/v) agarose 젤 DNA 마커 (M), 벡터 제어 (VC) 및 템플릿 PCR 증폭 (PCR). DNA는 0.5 x TAE 버퍼에서 45 분 120 V에서 전기 이동 법으로 분리 되었다. 총 0.5 ng VC의 로드, 서식 파일의 금액에 해당 PCR 레인에 로드. 젤은 UV 빛에서 시각화 이전 20 분 ethidium 평범한 사람 (~0.5 μ g/mL)을 사용 하 여 스테인드 (302 nm). 젤 (검은 화살표) 벡터의 예상된 크기에 가까운 증폭 된 DNA의 높은 수준을 보여 줍니다. 두 번째 밴드는 1, 000와 1500 bp 마커 밴드 사이의 가능성이 실행 PCR 레인에서 비 특히 증폭 된 PCR 제품을 나타냅니다. 증폭 된 DNA의 강도 성공적인 것으로 간주 될 VC 강도 수준 보다 높은 해야 합니다. 다른 여러 가지 DNA 염료 ethidium 평범한 사람 (예를 들어 참조 57) 얼룩에 더 적은 변이 원 성, 안전 대신 사용할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
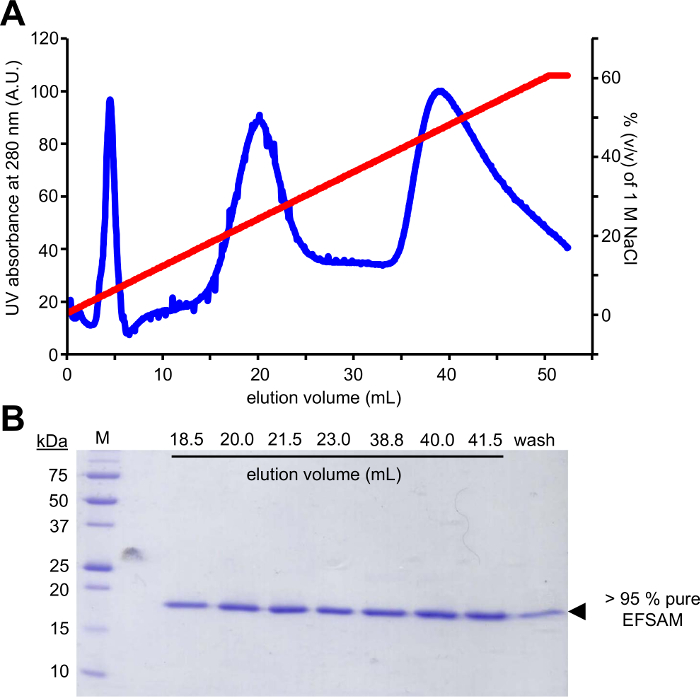
그림 2: 전형적인 컬럼에 정화 고 순도 STIM1 EF-샘에 대 한 확인.
(A) 음이온 교환 크로마토그래피 차입 프로필 STIM1 EF-샘의. 수동 바인딩 후 기본 pH와 주사기, 낮은 NaCl 농도에서 음이온 교환 열 (Q FF)에 EF-샘과 AKTA FPLC (GE 헬스케어) NaCl 그라데이션으로 단백질을 elute 데 사용 됩니다. 차입 AKTA UV 280 nm 신호를 사용 하 여 1 M NaCl의 0-60% (v/v) 기온 변화도에 의해 모니터링 됩니다. (B) Coomassie 파란 얼룩이 SDS 페이지 젤 (A)에서 차입 분수의. 변성 단백질 젤 EF-샘 두 주요 봉우리 ~ 250 m m와 ~ 450 m m NaCl elutes 보여준다. 정화 프로토콜 수익률 > 95% 순수 EF-어떤 오염 물질의 부족에 의해 입증으로 샘 밴드 Coomassie 파란 얼룩이 젤에 표시. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 투 석 설치 및 단백질 glycosylation 생체 외에서 의 확인.
(A) 전형적인 투 석 설치 생체 외에서 포도 당을 부착 Cys thiol MTS 반응성을 통해 사용 합니다. 화면은 투 석 배관에 대 한 버퍼에 포함 된 단백질의 ~1.5 mL ~ 1 L 실험 버퍼의. 버퍼 전체 교환 되도록 흔들 지속적으로 중요 하다. 이미지는 microcentrifuge 튜브를 투 석 백의 침 몰을 방지 하기 위해 회전 저 어 바에 의해 손상 초과 투 석 튜브까지 보여줍니다. (B) 분무 이온화 질량 스펙트럼 수정된 Asn171Cys EF-샘 단백질의. 질량 분석은 수정 절차 성공 여부를 평가 하기 위해 편리 하 고 정확한 방법입니다. 전형적인 질량 크로마 이론 및 측정 된 질량의 수정 되지 않은 및 수정 Asn171Cys EF-샘 표시와 함께 표시 됩니다. 대부분 샘플의 질량 ~1.3 예상된 이론 질량의 포도 당 활용 Asn171Cys EF-샘의 다 내 고분자에 해당. (B 에 데이터) 합니다 고 42에서 수정. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4: 단일 NMR 샘플에서 수정 효율성 및 구조적 섭의 솔루션 NMR 평가.
(A) 1 H15N HSQC 스펙트럼 오버레이 (레드 crosspeaks) 전에 Asn131Cys EFSAM 포도 당 활용의 전후 (블랙 crosspeaks) 15mm DTT의 추가. 오버레이 명확 하 게 몇 가지 잔류물 관련 아 미드 화학 변화 변화 단백질 및 구조 섭의 두 수정 나타내는 보여줍니다. 빨간색 상자는 Asn131Cys 아 미드의 위치를 보여줍니다. (B) Asn131Cys 아 미드를 포함 하는 1H-15N HSQC 영역의 Zoomed 보기. 수정 된 스펙트럼 (IM)에 Asn131Cys 아 미드 피크의 강도 (IR) 감소 (수정된) 스펙트럼의 강도 의해 효율 (그림 참조)의 계산에 대 한 나누어져 있습니다. 여러 영향된 잔류물의 평균 효율의 계산 오류 견적을 포함 하 여, 효율성의 더 나은 견적을 제공 합니다. 평균 효율 Asn131Cys EFSAM 5 잔류물 (즉, 129-133)에 따라 표시 됩니다. (C) 정규화 화학 변화 섭 Asn131Cys EFSAM 단백질을 포도 당 활용으로 인 한. HSQC 실험 세트 수집 하기 전에 단일 샘플에서 환 원제로 보충 뿐만 아니라 피크 강도 분석 [ (B)에 표시 된]에 의해 수정 효율의 편리한 예상 제공 하지만 대 한 데이터도 제공 합니다. 구조 변경 관련 수정 된 평가. 포도 당 활용 하면 위치 131; 근처 지역화 최대 섭 그러나,이 분석이 보여준다 섭을이 분석 값을 나타내는 시퀀스 근접에 전적으로 기반으로 예상 되지 않습니다. (C) 데이터 합니다 있으며 42에서 수정. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| STIM1 돌연변이는 | 방향b | DNA 시퀀스c | |
| Asn131Cys | 앞으로 | 5'-GTCATCAGAAGTATACTGTTGGACCGTGGATGAGG-3' | |
| Asn131Cys | 역 | 5'-CCTCATCCACGGTCCAACAGTATACTTCTGATGAC-3' | |
| Asn171Cys | 앞으로 | 5'-CCAAGGCTGGCTGTCACCTGCACCACCATGACAGGG-3' | |
| Asn171Cys | 역 | 5'-CCCTGTCATGGTGGTGCAGGTGACAGCCAGCCTTGG-3' | |
| 는 STIM1 아미노산 번호 기반으로 NCBI 가입 AFZ76986.1. | |||
| b '역방향' 뇌관 '앞 으로' 뇌관의 역방향 보완 시퀀스에 해당합니다. | |||
| c 밑줄이 그어진된 codon triplet Cys 돌연변이에 해당합니다. | |||
표 1입니다. 애완 동물-28a STIM1 EFSAM 내의 Cys mutagenesis에 Asn 예 oligonucleotide (뇌관) 시퀀스를 생성합니다.
토론
단백질 glycosylation 설탕 covalently 주로 아미노산 사이드 체인 관계를 통해 polypeptides에 연결 된 포스트 번역 상 수정입니다. 많은 포유류 단백질의 50%는 당화 54, 당화 단백질 수 이후에 있는 다양 한 바이오 바인딩 선호도 변경에서 효과, 단백질 폴딩, 채널 활동을 변경, 대상에 영향을 미치는 저하 및 셀룰러 (33에서 검토 한 결과) 몇 가지 이름을, 인신 매매에 대 한 분자. 포유류의 생리학에서 glycosylation의 중요 한 역할 구축 포유류 glycan 구조 33의 전체 다양성을 진화 하는 단백질의 몇몇 수백에 의해 분명 하다. 변경 N-및 O-glycosylation 패턴 연관 수많은 질병 전립선 (증가 감소), 유 방 (증가 감소)를 포함 하 여 간 (증가), 난소 (증가), 췌 장 (증가) 그리고 위 암 (증가) 55. 또한, 타우, huntingtin, glycosylation α-synuclein 발견 되었습니다. 규제 Alzheimer, 헌팅턴의와 파 킨 슨 병의 질병 56와 관련 된 이러한 단백질의 독성 그리고 glycosylation의 선 천 성 장애의 그룹 되었습니다. 효소를 중재 glycosylation 54에 상속 결함이 발생 확인. 따라서, glycosylation의 정확한 생물, 생화학 및 구조 효과 이해 대단히 단백질 규제 및 건강과 질병에서 함수에 대 한 우리의 이해에 영향을 줄 가능성이 있다.
Glycan 구조 포유류 glycome에서의 다양성으로 이어질 10 설탕 빌딩 블록 포함 fucose, 갈 락 토스, 포도 당, N-acetylgalactosamine, N-acetylglucosamine, glucuronic 산, iduronic 산만 노 오 스, sialic 산 및 xylose 33. Oglycosylation Nacetylgalacotose, N-acetylglucosamine, xylose, fucose의 어떤에서 발생할 수 있습니다 동안 Nglycosylation 변함없이 링크 하지는 N-acetylglucosamine 설탕 단백질에 직접, 포도 당 또는 갈 락 토스는 polypeptide에 연결 covalently. 이러한 설탕 단백질 표면에 인접 한 생물 및 구조 특성에 미치는 영향을 이해 하기 시작, 우리가 설명 여기 설탕 Cys의 잔류물 단백질으로 설계 하는 thiols 통해 사이트에 선택적으로 연결할 접근 시퀀스입니다. 여기, 당화 Cys로 대체 하 고 체 외에 는 간단한 화학 방법을 통해 수정 endogenously는 잔류물. 이러한 방식으로, 단일 및 다중 glycosylation 사이트 애타게 폴딩 및 안정성 뿐만 아니라 전체 구조에 누적 수정 및 각 특정 사이트의 기여와 단백질의 기능을 부과 수 있습니다.
최근,이 접근을 개별적으로 그리고 누적 평가는 Asn131 및 Asn171 N-glycosylation 사이트 42의 역할 EFSAM와 함께 사용 성공적으로 되었습니다. Cys 돌연변이 포도의 공유 첨부 파일을 Asn131 또는 Asn171 사이트에 감소 Ca2 + 바인딩 선호도 공개 하 고 안정성을 억제. 때 두 사이트 동시에 포도 당 첨부 파일, 바인딩 선호도 감소와 수정 및 안정성으로 이끌어 potentiated 했다 oligomerization 성향 시험관을 향상. 구조적으로, 여기에 설명 된 방법은 Asn131 또는 Asn171 수정 상호 SAM 도메인, EF 손 쌍에 인접 한에 있는 코어 α8 나선 교란 보여주었다. 이 구조 분석 단백질의 표면에 포도 당 수정 과정 안에서 수렴 하 고 potentiated 구조 변화를 리드 하는 어떻게 expounds-궁극적으로 단백질 destabilizes 그리고 SOCE 42향상 손: 샘 인터페이스.
더 이상 탄수화물 응급실에 연결할 어떻게 EFSAM의 표면 가까이 단 당 류 효과 폴딩, 안정성 및 구조,이 절차에 도움이 헛간 빛 수 쉽게이 사이트 선택 방법의 응용 프로그램을 수정 하는 동안 골 및 오후 지역화 (즉, glycosylation의 상태 다른 성숙), 거기에 제공 되는 thiols와 같은 MTS. MTS 바람직 thiol 수정 이기 때문에 되돌릴 수 사용 하 여 연결할 수 있는 기능 그룹에 포함 된 이러한 탄수화물에 대 한 신뢰할 수 있는 소스는 원제와 참조 스펙트럼 쉽게 취득 될 수 있다. 이 방법은 또한 다른 닢 moieties 지질 같은 단백질에 연결할 적용할 수 있습니다. 동시에 고려 한다이 방법 몇 가지 제한이 있습니다. 먼저, 메서드 glycosylation 사이트 구조, 안정성 및 접는 포도 당 수정의 부재에도 영향을 미칠 수 있는 Cys의 돌연변이에 의존 합니다. 마찬가지로, 단백질에서 네이티브 Cys의 잔류물도 포도 당화가 아닌 사이트에서 첨부 파일을 방지 하기 위해 변경 해야 합니다. 또한, 종종 Cys의 잔류물의 추가 더 도전 정화를 만드는 Cys 가교 및 misfolding, 박테리아에 포함 체 형성을 촉진 합니다. 그럼에도 불구 하 고,이 사이트 선택 Cys 가교 방법은 여기에 설명 된 특정 glycosylation 사이트의 높은 수준의 요구 하는 실험에서의 구조적, 생화학 및 생물 효과 애타게 제어 수단을 제공 합니다. 균질 단백질입니다. 효과의 구조에 기본이 아닌 Cys 잔류물, 안정성과 접는 수 수 단순히 ascertained 수정 비교 하 여에 야생-타입의 부재에 단백질 특성 42. 기능 데이터 표현 (예: Asn-하-Ala) 단백질의 돌연변이 체 버전 수정 차단 하는 진 핵 세포에서 얻은 함께 찍은, 현재 기술된 접근 단백질의 구조적 메커니즘에 새로운 통찰력을 얻을 것입니다. 포스트 번역 상 수정에 의해 규정입니다.
감사의 말
이 연구는 자연과학 및 공학 연구 위원회 캐나다의 (P.B.S.에 05239), 캐나다 재단에 의해 지원 되었다 혁신/온타리오 연구 기금 (P.B.S.), 전립선 암 싸 워 재단-Telus 타고 아빠 (P.B.S.) 하 고 온타리오에 대 한 (Y.J.C. N.S.)를 대학원 장학입니다.
자료
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
참고문헌
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L., Agostinis, P., Afshin, S. . Endoplasmic Reticulum Stress in Health and Disease. , 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유