Method Article
Dirigida a tioles de la cisteína para in Vitro específica glicosilación de proteínas recombinantes
En este artículo
Resumen
Análisis bioquímicos y estructurales de proteínas glicosiladas requieren cantidades relativamente grandes de muestras homogéneas. Aquí, presentamos un método químico eficiente para el glycosylation sitio-específico de proteínas recombinantes purificados de bacterias dirigiéndose a tioles reactivos de Cys.
Resumen
Interacción stromal molecule-1 (STIM1) es un tipo-yo proteína transmembrana localizado en el retículo endoplásmico (ER) y membranas del plasma (PM). ER-residente STIM1 regula la actividad de PM Orai1 canales en un proceso conocido como tienda de entrada operado de calcio (Ca2 +) que es el principal Ca2 + proceso de señalización que conduce a la respuesta inmune. STIM1 se somete poste-de translación N- glicosilación en dos sitios de Asn luminales en el Ca2 + detección del dominio de la molécula. Sin embargo, los bioquímicos, biofísicos, y efectos biológicos de la estructura de N- glicosilada STIM1 fueron mal entendidos hasta recientemente debido a una incapacidad para obtener rápidamente altos niveles de homogénea N- glicosilada proteína. Aquí, describimos la implantación de un vitro química enfoque que une moléculas de glucosa a los sitios de proteína específica aplicables para entender los efectos subyacentes del N- glycosylation en mecanismo y estructura de la proteína. Usando la espectroscopia de resonancia magnética nuclear solución evaluamos tanto eficacia de la modificación, así como las consecuencias estructurales de la fijación de glucosa con una sola muestra. Este enfoque puede ser adaptado fácilmente para estudiar las proteínas glycosylated múltiples encontradas en la naturaleza.
Introducción
Almacenar calcio operado (Ca2 +) (SOCE) es la vía principal por la que las células inmunes toman Ca2 + desde el espacio extracelular en el citosol. En los linfocitos T, receptores de células T localizados en la membrana plasmática (PM) unen los antígenos que activan proteínas quinasas de tirosina (revisadas en 1,2,3). Una cascada de fosforilación conduce a la activación de fosfolipasa-γ (PLCγ) que posteriormente interviene en la hidrólisis de la membrana fosfatidilinositol 4, 5-bifosfato (PIP2) en diacilglicerol e inositol 1,4,5-trifosfato (IP3 ). IP3 es un pequeño mensajero difusible que se une a la IP3 receptores (IP3R) en el retículo endoplásmico (ER) tal modo abriendo este canal del receptor y permitiendo Ca2 + fluyan abajo del gradiente de la concentración de la ER Lumen en el citosol (revisado en 4). Receptores acoplados a proteínas G y receptores de tirosina quinasa en una variedad de otros plomo de los tipos de células excitables y no excitables de señalización para la misma producción de IP3 y activación del IP3Rs.
Debido al finito Ca2 + capacidad de almacenamiento de la ER, la IP3-liberación mediada y el resultante aumento citosólico Ca2 + es sólo transitorio; sin embargo, este agotamiento del ER luminal Ca2 + efectos profundamente interacción stromal molecule-1 (STIM1), un tipo-yo transmembrana (TM) proteína que se encuentra sobre todo en el ER membrana 5,6,7. STIM1 contiene un lumen-orientado Ca2 + detección dominio formado por un par de EF-mano y estéril α-adorno (EFSAM). Tres dominios de en espiral-arrolle citosólica orientado se separan del EFSAM por el dominio TM (revisado en 8). Sobre ER luminal Ca2 + agotamiento, EFSAM sufre una Oligomerización de desestabilización junto 7,9 que causa los cambios estructurales de la TM y dominios coiled-coil 10. Estos cambios estructurales culminan en una captura de STIM1 en ER-PM las ensambladuras 11,12,13,14 a través de interacciones con PM fosfoinosítidos 15, 16 y Orai1 subunidades 17,18. Proteínas de Orai1 son las subunidades PM que montan en forma de Ca2 + canales21,de 19,20,22. Las interacciones STIM1 Orai1 en ER-PM ensambladuras facilitan una abierto Ca2 + desbloqueo activado Ca2 + (CRAC) canal conformación que permite el movimiento de Ca2 + en el citosol de las altas concentraciones de la espacio extracelular. En las células inmunes, el sostenido citosólica Ca2 + elevaciones vía CRAC inducen el Ca2 +- calmodulina/calcineurina dependiente desfosforilación del factor nuclear de células T activadas que posteriormente entra en el núcleo y comienza la regulación transcripcional de genes promoviendo la activación de células T 1,3. El proceso de activación de canal CRAC por STIM1 23,24 vía inducida por el agonista ER luminal Ca2 + agotamiento y la Ca2 + elevación citosólica sostenida resultante se denomina colectivamente SOCE 25. El papel vital de la SOCE en células de T es evidente por estudios que demuestran que las mutaciones heredables en STIM1 y Orai1 pueden causar inmunodeficiencia combinada severa síndromes 3,19,26, 27. EFSAM inicia SOCE después de detección luminal ER Ca2 + agotamiento de la vía la pérdida de Ca2 + coordinación de la mano de EF canónica, en última instancia conduciendo a la desestabilización junto autoasociación 7, 28,29.
Glicosilación es el accesorio covalente y procesamiento de estructuras de oligosacáridos, también conocido como glycans, a través de varios pasos biosintéticos en el ER y Golgi (revisado en 30,32,33). Hay dos tipos predominantes de glicosilación en eucariotas: N-ligados y O-ligado, según el aminoácido específico y el átomo tiende un puente sobre el acoplamiento. En la N- glicosilación, glicanos se unen a la amida de la cadena de lado de Asn, y en la mayoría de los casos, el paso de iniciación ocurre en el retículo endoplasmático como el polipéptido cadena se mueve en la luz 34. El primer paso de la N- glicosilación es la transferencia de una estructura de base 14-azúcar compuesta por glucosa (Glc), manosa (hombre) y N- acetilglucosamina (GlcNAc) (es decir, Glc3hombre9GlcNAc2) de una ER lípidos de membrana por un oligosaccharyltransferase 35,36. Otras medidas, como el escote o la transferencia de residuos de glucosa, son catalizadas en ER por glicosiltransferasas y glicosidasas específicas. Algunas proteínas que las ER y en el Golgi pueden ser otros procesados 37. O- glicosilación se refiere a la adición de glycans, generalmente para el grupo de hidroxilo de cadena lateral de residuos de Ser o Thr, y esta modificación ocurre enteramente en el complejo de Golgi 33,34. Hay varios O- glicanos las estructuras que pueden hacerse de N- acetilglucosamina fucosa, galactosa y ácido siálico con cada monosacárido añadido secuencialmente 33.
Mientras que ninguna secuencia específica se ha identificado como prerrequisito para muchos tipos de O- glicosilación, una secuencia consenso común se ha asociado con la N-ligado modificación: Asn-X-Ser/Thr/Cys, donde X puede ser cualquier aminoácido excepto Pro 33. EFSAM STIM1 contiene dos de estos sitios de consenso de N- glicosilación: Asn131-Trp132-Thr133 y Asn171-Thr172-Thr173. De hecho, estudios previos han demostrado que la EFSAM puede ser N- glicosilada en células de mamífero en Asn131 y Asn171 38,39,40,41. Sin embargo, estudios previos de las consecuencias de la N- glicosilación en SOCE han sido incongruentes, sugiriendo suprimido, potenciada o no efecto por esta modificación poste-de translación en SOCE activación 38,= "xref" > 39,40,41. Así, la investigación sobre las consecuencias biofísicas, bioquímicas y estructurales subyacentes de EFSAM N- glicosilación es vital para comprender los efectos regulatorios de esta modificación. Debido a la exigencia de altos niveles de proteínas homogéneas en estos experimentos en vitro , se aplicó un enfoque selectivo de sitio para unir covalentemente moléculas de glucosa a EFSAM. Curiosamente, glicosilación Asn131 y Asn171 había causado cambios estructurales que convergen dentro de la base EFSAM y mejoran las propiedades biofísicas que promueven la SOCE STIM1 mediada por 42.
La fijación química de glycosyl grupos al Cys tioles ha sido bien establecida por un trabajo que demostró por primera vez la utilidad de este enfoque de enzima libre para entender los efectos específicos del sitio de glicosilación en función de la proteína 43 , 44. más recientemente y con respecto a STIM1, los residuos Asn131 y Asn171 fueron transformados a Cys y glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] fue utilizado a covalente enlace glucosa a los tioles libres 42. Aquí, describimos este enfoque que no sólo utiliza la mutagénesis para incorporar residuos de Cys específicos del sitio para la modificación, pero también se aplica la espectroscopia de resonancia magnética nuclear (RMN) de solución para rápidamente evaluar eficacia de modificación y estructural perturbaciones como consecuencia de la glicosilación. En particular, esta metodología general es fácilmente adaptable para estudiar los efectos de cualquiera de los dos O- o N- glicosilación de cualquiera producido por vía recombinante proteína.
Protocolo
1. reacción en cadena de polimerasa (polimerización en cadena)-mediada mutagénesis sitio-dirigida de la incorporación de Cys en un vector de expresión bacteriano pET-28a.
- Determinar la concentración del vector pET-28a (es decir, doble trenzado DNA) utilizando un coeficiente de extinción (UV) ULTRAVIOLETA de 0.020 (μg/mL) cm -1 a 260 nm.
- Sintetizar un par de primers mutagénicos complementarios para cada mutación de Cys tal que yo) hay un mínimo de 15 nucleótidos complementarios a la plantilla antes de la coincidencia de la primera base y 15 nucleótidos complementarios a la plantilla después de la discrepancia de base final, ii) primer total longitud no supere los 45 nucleótidos y iii) una guanina o citosina se encuentra en el la primera y la última posición de cada primer (tabla 1). Asegurar la síntesis de la cartilla se realizaron mediante una purificación de la escala y el cartucho de 0.025 μmol.
- Usando una ADN polimerasa, de alta fidelidad creado mezclas de reacción de PCR de dos 20 μl: uno que contiene la cartilla hacia adelante y el segundo que contiene la cartilla reversa. Preparar cada mezcla contener concentraciones finales de 1 buffer de x PCR con 1,5 mM de MgCl 2, 0.2 mM dNTPs, cartilla de 0.5 μM, 0,4 de μL de DMSO, 1.25 plantilla ng/μL ADN, 0.02 U/μL alta fidelidad ADN polimerasa.
- Ciclo térmicamente las mezclas separadas mediante un protocolo de tres pasos: 98 ° C por 30 s (desnaturalización), 53-56 ° C durante 30 s (recocido), 72 ° C por 30 s kilobase(kb) -1 (extensión) de la plantilla de ADN. Repita el programa de temperatura para 5 ciclos y añadir un paso de extensión final de 72 ° C por min 7,5
- Después de la PCR inicial con las cartillas de avance y retroceso en tubos separados, combinar los productos en un solo tubo (es decir, 40 μL de total) y continuar la reacción de PCR para un adicional 20 ciclos con los mismos parámetros de ciclismo como se describe en el paso 1.4. Gel
- electrophorese 15 μL de la mezcla de reacción de PCR en una agarosa 1% (p/v) con 0,5 x Tris, ácido acético, etileno diamina tetra acético (EDTA) funcionamiento tampón (TAE). Como controles, electrophorese una cantidad equivalente de la plantilla de ADN que no ha sido amplificada por PCR y una alícuota de la escalera de ADN de referencia que contiene bandas marcadoras tanto mayor como menor que el tamaño del producto PCR esperado.
- Después de la electroforesis a 120 V durante 40 minutos, sumergir el gel en agua que contenga 0,5 bromuro de etidio μg/mL y agitar durante 30 minutos a temperatura ambiente. Confirmar la plantilla completa se ha ampliado por los cebadores mutagénicos como un aumento en la intensidad fluorescente bromuro de etidio relativa de la banda amplificada en comparación con la banda de la plantilla de control bajo luz UV (302 nm).
- Si no hay amplificación es evidente, repita la PCR después de ajustar la temperatura de recocido en incrementos de 0.5 ° C entre la temperatura de 53-56 ° C.
- Sobre la confirmación de amplificación de la plantilla por los cebadores mutagénicos, tratar los restantes ~ 25 μL de la mezcla de reacción de PCR con la enzima de restricción de DpnI digerir la plantilla metilada ADN. Utilizar DpnI 0,5 μl (10 unidades) por la mezcla de reacción de PCR de 25 μL y una concentración final de 1 × DpnI tampón de reacción. Incubar durante 2,5 h a 37 ° C.
- Digestión de la plantilla, añadir células competentes DH5α Esherichia coli en un tubo de microcentrífuga de 1,75 mL de choque ~ 5-10 μL de la mezcla digerida a 100 μl de calor. Incubar la mezcla del ADN de la célula en el hielo durante 60 minutos
- Choque del calor la mezcla de ADN de la célula en el tubo de microcentrífuga a 42 ° C por 45 s en un bloque de calor seco. Después de incubar la mezcla en hielo durante 3 min., agregar 900 μL de caldo de temperatura Luria-Bertani (LB) a las células y transferir la suspensión total de células en un tubo de fondo redondo de estéril 14 mL.
- Incubar la suspensión de células a 37 ° C durante 90 minutos con agitación constante a 190 rpm.
- Posteriormente, transferir la suspensión de células en un tubo de microcentrífuga de 1,75 mL y centrifugar a 10.000 x g durante 5 min a temperatura ambiente.
- Después de la centrifugación, retirar 900 μL del sobrenadante y resuspender las células bacterianas mediante pipeteo suave en el resto 100 μL de lb
- Transferir la suspensión concentrada de células resultante en una placa de LB-agar que contiene antibiótico que es selectiva para el vector de expresión (es decir, 60 μg/mLKanamycin). Asépticamente extendió la suspensión uniformemente en la placa de agar e incubar por ~ 16 h a 37 ° C.
- Al día siguiente, inocular una sola Colonia de la placa en 5 mL de LB líquido con la presión de selección de ejemplo (es decir, 60 μg/mL de kanamicina). Crece el cultivo líquido durante la noche a 37 ° C con agitación constante a 37 ° C.
- Aislar y purificar el plásmido reproducido de las células de e. coli utilizando un kit comercial basado en el procedimiento de lisis alcalina 45.
- Confirmar la mutación de interés está presente y en el marco de la correcta lectura por secuenciación del ADN Sanger de los plásmidos 46.
2. Uniforme 15 expresión de la proteína N-etiquetados en BL21 ΔE3 Escherichia coli.
Nota: diferentes proteínas recombinantes requieren condiciones diferentes de expresión. El siguiente es el procedimiento optimizado para la expresión de la proteína humana STIM1 EFSAM.
- Transformar el vector de expresión que las mutaciones de Cys (es decir, del animal doméstico-28a-EFSAM) en células competentes BL21 ΔE3 codón (+) calor choque y planchas en placas de LB-agar que contiene la presión de selección antibiótica como se describe en pasos 1.9-1.14) con la siguientes modificaciones: directamente de la placa una alícuota de 150 μL de la suspensión celular total de ~ 1.000 μL en la placa de LB-agar sin necesidad de concentración por centrifugación de las células en un tubo de microcentrífuga.
- Al día siguiente, transferir asépticamente una sola Colonia en un erlenmeyer que contenga 20 mL de LB suplementado con antibiótico apropiado (es decir, 60 μg/mL de kanamicina para pET-28a-EFSAM) de 200 mL. Crecer esta cultura arrancador líquido durante la noche (es decir, ~ 16 h) a 37 ° C con agitación constante a ~ 190 rpm.
- En el mismo día como paso 2.2 preparar M9 media 15 expresión de la proteína N-etiquetados en autoclave 1 L de M9 buffer sales (es decir, 42 m m de Na 2 HPO 4, 22 m m KH 2 PO 4, 8,6 mM NaCl, pH 7,4) en un erlenmeyer de 4 L. Una vez frío, filtrar una mezcla de 20% (w/v) D-glucosa 1 M CaCl 2, tiamina de 1 M, 1 M MgSO 4, biotina 1 mg/mL y 0.2 g/mL 15 N-NH 4 Cl con un 0.2 μm filtro de jeringa estéril en la solución salina M9 estéril 1 L para que la concentraciones finales de estos componentes están de 0.2% (w/v) D-glucosa, 100 μM de CaCl 2, 50 tiamina μM, 1 mM MgSO 4, 1 biotina μg/mL y 1 mg/mL 15 N-NH 4 cl.
- Al día siguiente, transferir asépticamente la cultura de arrancador líquido durante la noche de 20 mL a un tubo cónico estéril de 50 mL y centrifugar a 2.400 × g durante 15 minutos para que sedimenten las células.
- Después de decantar el medio LB, Resuspender el precipitado resultante de células en 10 mL de medio mínimo M9 y transferir la mezcla resuspendido en 1 L de medio mínimo M9 con antibiótico (es decir, 60 kanamicina de μg/mL).
- Crecer el 1 L de medio mínimo M9 que contiene la cultura de arrancador bacteriana a 37 ° C y constante ~ 190 rpm agitación hasta que la densidad óptica a 600 nm (OD600) alcanza ~0.6-0.8.
- Cuando se alcanza la gama specfified OD600, añadir 200 μM de isopropilo (IPTG) para inducir la expresión de la proteína β-D-1-tiogalactopiranósido.
- Después de la adición de IPTG, continuar incubando las células para la expresión de la proteína a temperatura ambiente con agitación constante a ~ 190 rpm ~ 16 h (es decir, durante la noche).
- Al día siguiente, la cosecha las bacterias por centrifugación a 10.000 ~ × g, 4 ° C por 30 min.
- Decantar los LB y transferir el precipitado de células en un tubo cónico de 50 mL. Guardar el sedimento a-80 ° C hasta purificación.
3. Purificación de proteínas recombinantes de e. coli.
Nota: diferentes proteínas recombinantes requieren procedimientos diferenciados de purificación. El siguiente es el protocolo de 6 & #215; Su etiqueta de purificación de EFSAM de cuerpos de inclusión expresada desde el constructo de pET-28a.
- Homogeneizar manualmente el precipitado de células bacterianas congelados en guanidina-HCl de 6 M, 20 mM Tris-HCl (pH 8) y 5 mM β-mercaptoetanol utilizando una pipeta de 10 mL motorizado. Añadir aproximadamente 40 mL de guanidina-HCl por 5 mL de líquido de la pelotilla para este paso.
- Siguientes a 90 min de incubación a temperatura ambiente con rotación constante en un horno de hibridación, centrifugar la mezcla a ~ 15.000 × g, 8 ° C durante 40 minutos separar los residuos insolubles de la célula (es decir, pellets) de la proteína soluble mezcla (es decir, sobrenadante).
- Añadir 750 μl de un 50% (v/v) Ni 2 +-agarosa ácido nitrilotriacético del grano de la mezcla para el clarificado lisada e incube durante otros 90 minutos a temperatura ambiente con la inversión en un horno de hibridación.
- Posteriormente, capturar a la proteína de su etiquetado 6 × enlazada al Ni 2 + recogiendo las perlas de agarosa en una columna de purificación de proteínas de flujo de gravedad. Permitir que el lisado a fluir a través de la columna completamente antes de mover a paso 3.5.
- Lave los granos recogidos tres veces con 10 mL de urea de 6 M, 20 mM Tris-HCl pH 8 y 5 mM β-mercaptoetanol. Asegúrese de que el entero 10 mL pasa a través de la columna antes de cada posterior 10 mL Washington
- Eluir las proteínas en una serie de fracciones de 2 mL con 6 M urea, 20 mM Tris-HCl de pH 8, imidazol de 300 mM y 5 mM de β-mercaptoetanol con un tiempo de incubación s 90 entre fracciones. Asegúrese de que el entero 2 mL pasa a través de la columna antes de cada paso posterior elución.
- En esta etapa, confirmar la proteína del interés está presente en las fracciones eluídas por Coomassie sodio teñido de azul dodecil sulfato poliacrilamida gel electroforesis (SDS-PAGE) utilizando el método de Laemmli 47. Evaluar el tamaño de la proteína, la cantidad y la pureza en comparación contra las bandas del marcador de peso molecular estándar que son menores y mayores que el peso molecular esperado de la proteína de interés.
- Piscina las fracciones eluídas de la proteína en una membrana de diálisis con un peso molecular de la 3.500 Da corte e incubar en 1 L de tampón repliegue (20 mM Tris, 300 mM NaCl, 1 mM TDT, 5 mM CaCl 2, pH 8) a 4 ° C durante la noche mientras el buffer se se agita por un magneti agitador c.
- Después de ~ 16 h de tiempo de repliegue, añadir ~ 1 U de la trombina por mg de proteína directamente a la bolsa de diálisis e incubar a 4 ° C para un adicional h. ~ 24
- Verificar la medida de 6 × su escote de etiqueta por la coloración de azul de Coomassie de ~ 15 μL proteína alícuotas tomadas de la bolsa de diálisis antes y después de la incubación con trombina que se en geles de poliacrilamida (SDS-PAGE) con la desnaturalización el método de Laemmli 47. Si se observa un cambio de ~ 2 kDa en la migración correspondiente al peso molecular de la hendida 6 × su tag, continuar al paso 3.11; Si una fracción de restos de proteína sin digerir que es detectable por la coloración de azul de Coomassie, Añadir ~0.2 U de la trombina por mg de proteína directamente a la bolsa de diálisis e incubar a 4 ° C para un adicional h. ~ 24
- Uso exclusión o intercambio iónico cromatografía por tamaño más purificar la proteína. Para la cromatografía de intercambio de aniones de EFSAM, eliminar la solución de la proteína de la bolsa de diálisis y concentrados ~ 10 veces usando un concentrador centrífugo de ultrafiltración con un peso molecular de la 10.000 Da corte. Posteriormente, volver a diluir la solución de ~ 20-fold en un tampón libre de NaCl (20 mM Tris, 5 mM CaCl 2, 1 mM TDT, pH 8).
- Equilibrar una columna de intercambio aniónico preenvasados con 10 volúmenes de columna de la solución de NaCl-free descrito en el paso 3.11. Equilibrar el uso de tampón de carga en una jeringa de luer-lock que contiene sin burbujas de aire empujando la solución a través de la columna en forma de gota a gota y evitando presiones de jeringa que causan corrientes constantes de solución de salir de la columna. Utilizar un intercambiador de aniones fuertes (e.g. reticulado agarosa con grupos funcionales de amonio cuaternario).
- Cargar la solución de proteína diluida en tampón libre de NaCl (paso 3.11) en la columna tal como se describe en el paso 3.12.
- Eluir las proteínas en un gradiente [es decir, 0 - 60% (v/v)] de aumento de NaCl buffer (20 mM Tris, 1 M NaCl, 5 mM CaCl 2, 1 mM TDT, pH 8) usando un sistema de cromatografía líquida (FPLC) proteína rápida dos bombas. El sistema FPLC para recoger fracciones de mL ~1-1.5 y monitorear el perfil de elución de la proteína usando absorbencia de UV 280 nm y un caudal de 0,5 mL/min
- Identificar los picos de elución y fracciones que contiene la proteína de interés, así como la pureza de proteínas por SDS-PAGE teñido de azul de Coomassie geles utilizando el método de Laemmli 47.
- Fracciones piscina mostrando > 95% (es decir, tomado como fracciones que presentan sólo una banda de proteína única tinción de geles de azul de Coomassie) en una bolsa de diálisis y el intercambio en búfer experimental de interés por diálisis como se describe en el paso 3.8.
4. Fijación química de glucosa-5-MTS a proteína por diálisis.
- Prepare un 55 mM solución de N-(β-D-glucopyranosyl)-N '-[2-methanethiosulfonyl) etil] urea (glucosa-5-MTS) disolviendo 10 mg del compuesto en 500 μl del 100% (v/v) DMSO. Almacenar glucosa-5-MTS solubilizado en DMSO a -20 ° C.
- Prepare la muestra de proteína para modificación por diálisis de 1.5 mL de proteína de ~ 60 μM en 1 L de modificación buffer formado por MOPS 20 mM, NaCl 150 mM, 5 mM CaCl 2 y 0,1 mM TCEP, pH 8.3. Use un corte de peso molecular de la membrana de diálisis que es más pequeño que el tamaño de la proteína modificando (por ejemplo usar un atajo de 3.500 para la EFSAM de Da ~ 17.500).
- Después de 24 h a 4 ° C, transferir la muestra de la bolsa de diálisis en un tubo de microcentrífuga. Añadir la glucosa-5-MTS solubilizado de DMSO para una concentración final de 2 mM.
- Incubar la muestra en la oscuridad durante 1 h a temperatura ambiente. Durante el período de incubación de 1 h, mezclar la solución golpeando suave del tubo cada 10 minutos Posteriormente,
- volver a cambio de la proteína en el búfer experimental final con ningún agente de reducción en diálisis a 4 ° C como se describe en el paso 4.2 o por ultrafiltración centrífuga. Para el procedimiento de la ultrafiltración, concentrar la muestra de proteína mL de ~1.5 < 0,5 mL y diluir posteriormente en el mismo concentrador con el tampón de experimental. Repita este paso de concentración dilución dos veces más para que el cambio total es de un mínimo de 30 × 30 × 30 = 27,000-fold. Para EFSAM, utilice 20 mM Tris, 150 mM NaCl, CaCl 2, de 5 mM, pH 7.5 como el búfer experimental.
- Preparar la muestra para espectrometría de masas de ionización por electrospray por diálisis o ultrafiltración intercambio como se describe en los pasos 4.2 y 4.5, respectivamente, en 25 mM bicarbonato de amonio o el acetato de amonio 25 mM. Si usa diálisis, asegúrese de intercambio por lo menos tres veces para eliminar cualquier residual NaCl y CaCl 2 sales.
- Determinar la masa exacta (es decir, ± 1 Da) de la proteína de interés mediante la ionización por electrospray espectrometría 48 , 49 en masa. Esperar que cada adición covalente de la glucosa a un tiol de Cys por el químico methanethiosulfonate a añadir 281.3 Da a la proteína de masa (es decir, sumar 360.4 Da para glucosa-5-MTS y restar Da 79,1 para CH 3 para dejar 2 grupo durante covalente archivo adjunto).
5. Evaluación de la solución NMR de modiflas perturbaciones estructurales y de eficiencia y.
- Asegúrese de que la concentración de la modificada de la proteína es > 100 μM después de la fijación de la glucosa y el intercambio de búfer final. Para EFSAM, estimar la concentración de proteína usando un coeficiente de extinción de la UV a 280 nm de 1.54 cm –-(mg mL – 1) 1.
- Suplemento la solución de proteína con 60 μM de ácido 4,4-dimethyl-4-silapentane-1-sulfonic (DSS) para la calibración equilibrados y pulso y 10% (v/v) D 2 O para el bloqueo de la señal. Para uso de señal a ruido alto 600 μL las muestras en tubos emparejados en frecuencia 5 m m NMR, inserta en un espectrómetro mínimo de 600 MHz equipado con una sonda criogénica del HCN de triple resonancia.
- Recoger espectros de estándar 1 H - 15 N HSQC anteriormente detallada 50 , 51 en temperatura, 1 H y anchos de barrido de 15 N, transitorio y el incremento ajustes adecuados para la muestra determinada. Para espectros EFSAM, uso de 20 ° C, transitorios 1 H 256, 64 15 N dimensión incrementos y 1 H y 15 N barren ancho establecido en 1.800 y 8.000 Hz, respectivamente.
- Tras la adquisición del espectro de proteínas glicosiladas, añada dithiothreitol (DTT) al NMR de un stock de 1 M a una concentración final de 15 mM. La TDT quita la molécula de glucosa de la proteína por la reducción mediada por el disulfuro del accesorio de la.
- Adquirir una segunda 1 H - 15 N HSQC bajo estas condiciones de reducción/sin modificar, proporcionando un espectro de referencia para evaluar modificación estructural y eficiencia las perturbaciones causadas por la fijación de la glucosa. Datos
- proceso NMR NMRPipe como detallado anteriormente 52. Asegúrese de que mínimamente procesado conversión de datos, eliminación, supresión de disolvente, transformada de Fourier y visualización inicial de los espectros.
- Evaluar eficacia de modificación mediante la medición de la intensidad de pico de amida y valores de cambio químico en los espectros reducidos y modificados usando el plugin NEASY CARA 53. Asegúrese de evaluar la intensidad de pico de la amida de Cys en la glucosa Unido y había reducido espectro. Si la amida Cys no puede ser confiablemente situada en ambos espectros, utilizar las intensidades de residuos adyacentes a la Cys como una lectura.
- Calcular la eficiencia como la intensidad de la amida del espectro modificado Cys dividida por la intensidad de la amida del espectro reducido de Cys (es decir, tratados con TDT), multiplicado por 100:
 , donde es la intensidad de la amida en el espectro modificado de Cys y R es la intensidad de la amida en el espectro reducido de Cys. Por otra parte, evaluar la eficiencia media sobre varios picos de amida:
, donde es la intensidad de la amida en el espectro modificado de Cys y R es la intensidad de la amida en el espectro reducido de Cys. Por otra parte, evaluar la eficiencia media sobre varios picos de amida:
 , donde eficiencia i es la eficacia por separado determinada calculada para cada uno residuo, i y n es el número total de los residuos utilizados en el cálculo de.
, donde eficiencia i es la eficacia por separado determinada calculada para cada uno residuo, i y n es el número total de los residuos utilizados en el cálculo de. - Calcular las perturbaciones de la cambio química (CSP) de las diferencias de cambio química entre los dos espectros observados en los 15 N y dimensiones de 1 H de cada pico y normalización para la más grande 15 N cambio química gama usando la siguiente ecuación:
 , donde ΔH es el cambio de ppm en la dimensión de protones y ΔN es el cambio de ppm en la dimensión de nitrógeno.
, donde ΔH es el cambio de ppm en la dimensión de protones y ΔN es el cambio de ppm en la dimensión de nitrógeno.
Resultados
El primer paso de este enfoque requiere la mutagénesis de los residuos de glicosilación de candidato a residuos de Cys, que pueden ser modificables mediante la EFSAM de glucosa-5-MTS. tiene ningunos residuos Cys endógenos, por lo que no hay consideraciones especiales deben hacerse antes del mutagénesis. Sin embargo, residuos Cys nativos deben ser transformados a residuos no modificables antes de realizar la química describe. Para mínimamente la estructura nativa, sugerimos realizar una alineación global de la secuencia de la proteína de interés y determinar que otros residuos son encontrados más frecuentemente en la Cys endógeno puesto (s). Mutación de Cys para estos los residuos que se producen naturalmente en otros organismos tenga el menor impacto en la estructura de la proteína. Si el residuo de Cys endógeno se conserva estrictamente, sugerimos mutando al Ser que es el más estructural similar a Cys. La figura 1 muestra un típico gel de mutagénesis PCR evaluar el éxito de la reacción de PCR, con la muestra de ADN amplificada, demostrando un mayor intensidad de una cantidad de control de ADN de pET-28a amplificados de la plantilla que se utilizó para PCR. Los próximos pasos incluyen plantilla ADN digestión y transformación en e. coli para la reparación de plásmido. Después de la propagación de plásmido en cultivos líquidos, aislamiento de plásmidos y confirmación de la mutación de la secuencia, el vector transformado puede utilizarse para la expresión de la proteína. Figura 2A muestra un perfil típico de la elución de EFSAM de la columna de intercambio aniónico en relación a aumento de concentraciones de NaCl. Figura 2B muestra la pureza de EFSAM en geles de SDS-PAGE de teñido de azul de Coomassie.
Después de adquirir proteína pura, una serie de pasos de la diálisis se utiliza para fijar la molécula de glucosa mediante la reacción de MTS con el tiol libre. Figura 3A muestra una imagen de la configuración típica de un volumen de proteína sellado en la diálisis la membrana por la membrana clips y contenido en un gran vaso de precipitados de 1 L que contiene el tampón de interés. Una comprobación inicial del éxito de la modificación puede realizarse por espectrometría de masas. Figura 3B muestra un espectro de masa electrospray representante de EFSAM modificado en un tiol Cys solo. Tras el establecimiento del protocolo para una proteína específica, perturbaciones de modificación estructural y eficacia pueden evaluarse desde una sola uniforme muestra marcado con N 15. El espectro de 1H -15N-HSQC se adquiere antes y después de la adición del agente reductor DTT (Figura 4A). Cálculos de la eficacia de la modificación pueden realizarse por una comparación de las intensidades de pico de amida en los espectros modificadas y reducidas como se indica en el paso de protocolo 5.8 (Figura 4B). Finalmente, cuando las tareas de cambio químico son conocidas por una proteína, el CSP que se correlacionan con los cambios estructurales puede calcularse como paso detallado en 5.9 (figura 4).
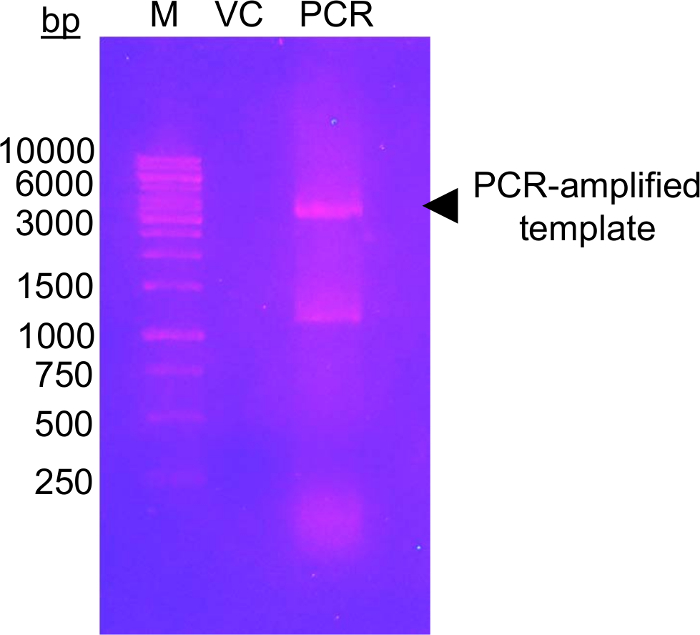
Figura 1: La gel de la agarosa de la DNA muestra control de amplificación del vector plantilla con primers mutagénicos.
La imagen muestra un gel de agarosa al 1.0% (w/v) con marcadores de ADN (M), vector control (VC) y plantilla amplificado por la polimerización en cadena (PCR). El ADN fue separado por electroforesis en 120 V de 45 minutos en tampón x TAE 0,5. Un total de 0.5 ng de VC se cargó, equivalente a la cantidad de plantilla cargada en el carril de la polimerización en cadena. El gel se tiñó con bromuro de etidio (~0.5 μg/mL) durante 20 min antes de la visualización bajo luz UV (302 nm). El gel muestra un alto nivel de DNA amplificada cerca el tamaño esperado del vector (flecha negra). La segunda banda en el carril PCR entre las 1.000 y 1.500 bp bandas del marcador probablemente representa un producto PCR amplificado no específica. El nivel de intensidad del ADN amplificado debe ser mayor que el nivel de intensidad de VC se considere exitosa. Varios otros tintes del ADN pueden utilizarse como menos mutágenos y seguras alternativas al bromuro de etidio tinción (ver por ejemplo 57). Haga clic aquí para ver una versión más grande de esta figura.
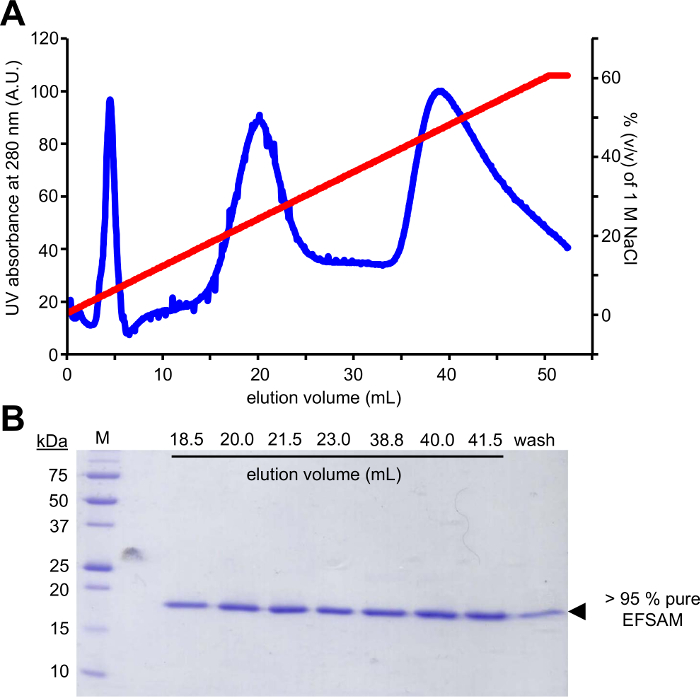
Figura 2: Pureza y purificación cromatográfica típica comprueben para STIM1 EF-SAM.
(A) anión intercambio cromatografía elución Perfil de STIM1 EF-SAM. Después manual EF-SAM a la columna de intercambio aniónico (Q FF) en pH básico y baja concentración de NaCl con una jeringa y AKTA FPLC (GE Healthcare) se utilizan para eluir la proteína con un gradiente de NaCl. La elución es supervisada por el AKTA usando la señal de UV 280 nm una pendiente de 0-60% (v/v) de 1 M de NaCl. (B) Coomassie azul manchado SDS-PAGE gel de fracciones elución de (A). El gel de proteínas desnaturalización revela que EF-SAM elutes en dos picos importantes en 250 m m y ~ 450 mM NaCl. El protocolo de purificación produce > 95% pura EF-SAM evidenciada por la ausencia de cualquier contaminante banda aparecen en el gel teñido de azul de Coomassie. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Configuración de la diálisis y la confirmación de en vitro el glycosylation de la proteína.
(A) instalación de diálisis típico utilizado para en vitro la fijación de glucosa a la Cys tiol mediante la reactividad MTS. La imagen muestra ~1.5 mL de proteína contenida dentro de tubos de diálisis intermedia contra ~ 1 L de tampón experimental. Es importante que el búfer se revuelve constantemente para intercambio completo. La imagen muestra un tubo de microcentrífuga recortado a la tubería de la diálisis exceso para prevenir el hundimiento de la bolsa de diálisis y dañar la barra de agitación rotatorio. Espectro de (B) Electrospray ionización total de la proteína modificada de Asn171Cys EF-SAM. Espectrometría de masas es un método conveniente y exacto para evaluar si el procedimiento de la modificación fue exitoso. Se muestra un cromatograma típico de masa con las masas teóricas y medidas de modificado y sin modificar Asn171Cys EF-SAM indicada. La mayoría de la muestra de masa corresponde a una macromolécula que se encuentra ~1.3 Da de la masa teórica esperada de glucosa conjugada Asn171Cys EF-SAM. Los datos de (B) es volver y modificado de 42. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Evaluación de la solución NMR de perturbaciones estructurales y de eficiencia de la modificación de una sola muestra de NMR.
(A) 1 H -15N-HSQC superposición espectral de glucosa conjugada Asn131Cys EFSAM antes (crosspeaks rojo) y después (crosspeaks negro) la adición de 15 mM DTT. La superposición muestra claramente varios cambios de cambio química de la amida de residuos específicos indicativos de ambos modificación de las perturbaciones estructurales y proteína. El cuadro rojo muestra la ubicación de la amida Asn131Cys. (B) vista de Zoomed de la 1H -15N-HSQC región que contiene el Asn131Cys amida. La intensidad del pico Asn131Cys amida en el espectro modificado () es dividida por la intensidad en el espectro (no modificado) reducida (IR) para el cálculo de la eficiencia (se muestra). Un cálculo de la eficiencia media de varios residuos efectuadas proporciona una mejor estimación de la eficacia, incluyendo una estimación del error. La eficacia promedio se muestra Asn131Cys EFSAM basado en 5 residuos (es decir, 129-133). (C) normalizado cambio químico las perturbaciones causadas por la conjugación de la glucosa a la proteína de EFSAM de Asn131Cys. El conjunto de experimentos HSQC recogidos en una sola muestra antes y después de suplementar con reductor no sólo proporciona una estimación conveniente de la eficacia de la modificación por el análisis de la intensidad de pico [se muestra en (B)], pero también proporciona datos para evaluación de los cambios estructurales asociados con la modificación. Verbal de glucosa provoca las perturbaciones más grande localizadas cerca de la posición 131; sin embargo, este análisis revela las perturbaciones que son inesperadas basado únicamente en la proximidad de la secuencia, indicando el valor en este análisis. Los datos en (C) volver y modificados a partir de 42. Haga clic aquí para ver una versión más grande de esta figura.
| STIM1 mutaciónun | Direcciónb | DNA secuenciac | |
| Asn131Cys | hacia adelante | TGTde 5'-GTCATCAGAAGTATAC TGGACCGTGGATGAGG-3' | |
| Asn131Cys | inversa | ACAde 5'-CCTCATCCACGGTCCA GTATACTTCTGATGAC-3' | |
| Asn171Cys | hacia adelante | 5'-CCAAGGCTGGCTGTCACCTGCACCACCATGACAGGG-3' | |
| Asn171Cys | inversa | ACGde 5'-CCCTGTCATGGTGGT GGTGACAGCCAGCCTTGG-3' | |
| un STIM1 aminoácido numeración basada en la adhesión de NCBI AFZ76986.1. | |||
| b La cartilla 'inversa' corresponde a la secuencia del complemento reverso de la cartilla 'adelante'. | |||
| c La tripleta codón subrayado corresponde a la mutación de Cys. | |||
Tabla 1. Construyen secuencias de oligonucleótidos (cartilla) ejemplo de Asn a mutagénesis Cys dentro del animal doméstico-28a STIM1 EFSAM.
Discusión
Glycosylation de la proteína es una modificación post-translacional donde azúcares se unen covalentemente a polipéptidos principalmente a través de vínculos con las cadenas laterales del aminoácido. 50% de proteínas mamíferos son glicosiladas 54, donde las proteínas glicosiladas posteriormente pueden tener una amplia gama de efectos de alterar la afinidad biomolecular, que influyen en la proteína plegable, alterando la actividad del canal, dirigido a moléculas de degradación y tráfico, por nombrar algunos (revisados en33) celular. La importancia de la glicosilación en mamífero fisiología es evidente por los cientos de proteínas evolucionadas hasta para construir la diversidad completa de mamíferos glycan estructuras 33. Modificado N- y O- glicosilación patrones han sido asociados con enfermedad numerosos Estados incluyendo de próstata (aumento y disminución), mama (aumento y disminución), hígado (aumentado), ovárico (aumentado), pancreático (aumentado) y cáncer gástrico (mayor) 55. Además, glicosilación de Tau, la huntingtina, α-sinucleína se ha encontrado para regular la toxicidad de estas proteínas asociadas con enfermedades de Alzheimer, Huntington y Parkinson 56, y han sido un grupo de trastornos congénitos de la glicosilación identifica resultantes de defectos hereditarios en las enzimas que median la glicosilación 54. Por lo tanto, comprender los efectos biofísicos, bioquímicos y estructurales precisos de glicosilación tiene el potencial de afectar enormemente nuestra comprensión de la regulación de la proteína y función en salud y enfermedad.
Los bloques de diez azúcar que conducen a la diversidad de estructuras de glicanos en la glycome mamífero incluyen fucosa, galactosa, glucosa, N- acetilgalactosamina, N- acetilglucosamina, ácido glucurónico, ácido idurónico, manosa, ácido siálico y xilosa 33. Mientras que la N- glicosilación une invariablemente un N- acetilglucosamina azúcar directamente a la proteína, O- glicosilación puede resultar de cualquiera de los N- acetylgalacotose, N- acetilglucosamina, xilosa, fucosa, glucosa o galactosa covalente el polipéptido. Para comenzar a comprender cómo estos azúcares inmediatamente adyacentes a la superficie de la proteína afectan las propiedades biofísicas y estructurales, en este documento describimos un acercamiento para fijar sitio selectivamente azúcares a residuos de Cys por tioles diseñadas en la proteína secuencia. Aquí, los residuos que son endógenamente glucosilada son substituidos por Cys y modificado en vitro vía un acercamiento simple química. De esta manera, sitios de glicosilación simple y múltiple pueden evaluarse para embromar hacia fuera la contribución de cada sitio específico, así como las modificaciones acumuladas en el plegamiento y estabilidad así como la total estructura y función de la proteína.
Recientemente, este enfoque fue utilizado con éxito con EFSAM individualmente y en conjunto evaluar el papel de la Asn131 y Asn171 N- glicosilación sitios 42. Mutación de Cys y accesorio covalente de la glucosa a los sitios Asn131 o Asn171 revelaron una disminución Ca2 + afinidad y suprimieron la estabilidad. Cuando los dos sitios al mismo tiempo fueron modificados con el accesorio de la glucosa, los descensos en la afinidad de enlace y estabilidad fueron potenciados a mejoran Oligomerización propensión in vitro. Estructuralmente, el procedimiento descrito en este documento mostraron que las modificaciones de Asn131 o Asn171 mutuamente perturban la hélice de α8 núcleo situada en el dominio SAM, inmediatamente adyacente al par de EF-mano. Este análisis estructural expone cómo modificaciones de glucosa en la superficie de la proteína conducen a un cambio estructural convergente y potenciado en la EF-interfaz de mano: SAM que en última instancia desestabiliza la proteína y mejora la SOCE 42.
Mientras que la aplicación de este enfoque selectivo sitio ayudó a arrojar luz sobre cómo un monosacárido cerca de la superficie de la EFSAM efectos de plegamiento, estabilidad y estructura, este procedimiento puede modificarse para fijar más hidratos de carbono específicos de ER, Golgi y PM localización (es decir, Estados de glicosilación de madurez diferentes), siempre que haya es una fuente confiable de estos hidratos de carbono que contienen grupos funcionales que puede enlazar con tioles como MTS MTS., es preferible ya que la modificación de tiol es reversible utilizando un agente de reducción y un espectro de referencia pueden ser fácilmente adquiridas. Este enfoque también puede ser adaptado para vincular otros moieties poste-de translación de la proteína tales como lípidos. Al mismo tiempo, hay varias limitaciones a este enfoque que debe considerar. En primer lugar, el método se basa en la mutación de sitios de glicosilación a Cys que afecte la estructura, estabilidad y doblar incluso en ausencia de cualquier modificación de la glucosa. Del mismo modo, nativos residuos de Cys en la proteína también deben ser transformados para evitar la fijación de la glucosa en sitios no-glycosylated. Además, la adición de residuos de Cys a menudo promueve la formación de cuerpo de inclusión en las bacterias debido a la reticulación de Cys y mal plegamiento, hacer purificación más desafiante. Sin embargo, este enfoque selectivo sitio de Cys-reticulación descrito proporciona un medio controlado para embromar hacia fuera los efectos estructurales, bioquímicos y biofísicos de sitios de glicosilación específica en experimentos que requieren altos niveles de proteína homogénea. Los efectos de residuos de Cys no nativos en la estructura, estabilidad y plegamiento pueden ser simplemente comprobados en ausencia de modificaciones en comparación al salvaje-tipo proteína atribuye 42. Tomado junto con los datos funcionales obtenidos en células eucariotas que expresan bloqueo de modificación de versiones mutantes de la proteína (e.g. Asn a Ala), el enfoque actualmente descrito dará nuevas perspectivas sobre los mecanismos estructurales de la proteína regulación por modificaciones post-traduccionales.
Agradecimientos
Esta investigación fue apoyada por las ciencias naturales y Consejo de investigación de ingeniería de Canadá (05239 a P.B.S.), Fundación Canadiense para la investigación del fondo de innovación/Ontario (a P.B.S.), Fundación lucha contra el de próstata cáncer - Telus Ride para papá (P.B.S.) y Ontario Beca de postgrado (para Y.J.C. y N.S.).
Materiales
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
Referencias
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L., Agostinis, P., Afshin, S. . Endoplasmic Reticulum Stress in Health and Disease. , 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados