Method Article
Targeting Cystein Thiole für in-vitro- standortspezifische Glykosylierung von rekombinanten Proteinen
In diesem Artikel
Zusammenfassung
Biochemische und strukturelle Analysen des glykosylierten Proteinen erfordern relativ große Mengen an homogenen Proben. Hier präsentieren wir Ihnen eine effiziente chemische Methode für standortspezifische Glykosylierung von rekombinanten Proteinen durch gezielte reaktive Cys Thiole von Bakterien gereinigt.
Zusammenfassung
Stromale Interaktion Molekül-1 (STIM1) ist eine Art-ich transmembranen Protein im endoplasmatischen Retikulum (ER) und die Plasmamembranen (PM) gelegen. ER-Resident STIM1 reguliert die Aktivität der PM Orai1 Kanäle in einem Prozeß bekannt als betriebenen Kalzium (Ca2 +) Eintrag zu speichern, die die wichtigsten Ca2 + Signalisierung erfolgt, die die Immunantwort antreibt. STIM1 erfährt post-translationalen N- Glykosylierung an zwei luminalen Asn-Standorten innerhalb der Ca2 + Fernerkundung Domäne des Moleküls. Aber die biochemische, biophysikalische, und Struktur biologischen Wirkungen von N- glykosylierten STIM1 waren schlecht verstanden, bis vor kurzem aufgrund einer Unfähigkeit, ohne weiteres hohes Maß an homogenen N- glykosyliertes Protein zu erhalten. Hier beschreiben wir die Umsetzung eines in-vitro- chemische Ansatzes, das Glukose Moieties spezifisches Protein Websites anwendbar zum Verständnis der zugrunde liegenden Auswirkungen der N- Glykosylierung auf Protein-Struktur und Mechanismus beimisst. Mit magnetischen Kernresonanz-Spektroskopie Lösung bewerten wir sowohl Effizienz der Änderung sowie die strukturellen Konsequenzen der Glukose-Anlage mit einer einzigen Probe. Dieser Ansatz kann leicht angepasst werden, um die unzähligen glycosylierten Proteine in der Natur zu studieren.
Einleitung
Speichern betriebenen Kalzium (Ca2 +) Eintrag (SOCE) ist der große Weg durch die Immunzellen Ca2 + aus dem extrazellulären Raum in das Zytosol nehmen. In T-Lymphozyten binden T-Zell-Rezeptoren auf der Plasmamembran (PM) Antigene die Protein-Tyrosin-Kinasen (rezensiert in 1,2,3) aktivieren. Eine Phosphorylierung Kaskade führt zur Aktivierung der Phospholipase-γ (PLCγ), die anschließend die Hydrolyse von Membran Phosphatidylinositol-4,5-Bisphosphate (PIP2) in Diacylglycerol und Inositol-1,4,5-Trisphosphate (IP-3 vermittelt ). IP-3 ist ein kleines diffusiblem Bote die IP3 an Rezeptoren (IP-3R) auf dem endoplasmatischen Retikulum (ER bindet) damit dieser Rezeptor-Kanal öffnen und schönem Ca2 + nach unten das Konzentrationsgefälle aus der Notaufnahme fortgesetzt Lumen zu dem Zytosol (rezensiert in 4). Rezeptor Signalisierung von G-Protein gekoppelt und Tyrosin-Kinase-Rezeptoren in einer Vielzahl von anderen erregbaren und nicht erregbaren Zelle Arten führen die gleiche Produktion von IP-3 und Aktivierung von IP3Rs.
Aufgrund der endlichen Ca2 + Speicherkapazität von der Notaufnahme, die IP-3-vermittelte Freisetzung und daraus resultierende Erhöhung der cytosolischen Ca2 + ist nur vorübergehend; diese Erschöpfung des ER luminalen Ca2 + Effekte jedoch zutiefst Stromazellen Interaktion Molekül-1 (STIM1), eine Art-ich transmembranen (TM) Protein meist zu finden, auf die ER Membran 5,6,7. STIM1 enthält eine Lumen-orientierte Ca2 + Fernerkundung Domäne bestehend aus ein paar EF-Hand und sterile α-Motiv (EFSAM). Drei cytosolische orientierten coiled-Coil-Domänen sind von EFSAM durch die TM-Einzeldomäne (rezensiert in 8) getrennt. Auf ER luminalen Ca2 + Erschöpfung erfährt EFSAM eine Destabilisierung gekoppelt Oligomerisierung 7,9 wodurch strukturelle Umstellungen der TM und coiled-Coil-Domänen 10. Diese strukturellen Veränderungen münden in ein Überfüllen des STIM1 bei ER-PM Kreuzungen 11,12,13,14 durch Interaktionen mit PM Phosphoinositides 15, 16 und Orai1 Untereinheiten 17,18. Orai1 Proteine sind die PM-Untereinheiten die Form Ca2 + Kanäle 19,20,21,22montieren. Die STIM1-Orai1-Interaktionen an ER-PM Kreuzungen erleichtern eine offene Ca2 + Freigabe aktiviert Ca2 + (CRAC) Kanal Konformation ermöglicht die Bewegung von Ca2 + in der Zellflüssigkeit aus den hohen Konzentrationen von der Extrazellulärraum. In Immunzellen, die nachhaltige cytosolischen Ca2 + Höhen CRAC Dienstweg induzieren die Ca2 +- Calmodulin/Calcineurin abhängigen Dephosphorylation des nuklearen Faktors der aktivierten T-Zellen die Folge den Kern tritt und transkriptionelle Regulierung der Gene, die Förderung der T-Zell-Aktivierung 1,3beginnt. Der Prozess der CRAC Kanal Aktivierung durch STIM1 23,24 über Agonist-induzierte ER luminalen Ca2 + Erschöpfung und die daraus resultierende nachhaltige cytosolischen Ca2 + Höhe wird Kollektiv SOCE 25bezeichnet. Die entscheidende Rolle der SOCE in T-Zellen ist offensichtlich durch Studien, die zeigen, dass vererbbare Mutationen in STIM1 und Orai1 dazu führen, Severe Combined Immunodeficiency Syndrome 3,19,26, dass können 27. EFSAM initiiert SOCE nach sensing ER luminalen Ca2 + Erschöpfung über den Verlust von Ca2 + Koordinierung auf die kanonische EF-Hand, letztlich zu der Destabilisierung gekoppelt Selbstassoziation 7, 28,29.
Glykosylierung ist die kovalente Bindung und Verarbeitung von Oligosaccharid Strukturen, auch bekannt als Glykane, durch verschiedene biosynthetischen Schritte in der ER und Golgi (rezensiert in 30,32,33). Es gibt zwei vorherrschende Arten der Glykosylierung in Eukaryoten: N-verknüpften und O-verbunden, abhängig von der spezifischen Aminosäure und Atom überbrücken die Verknüpfung. In N- Glykosylierung Glykane Seite Kette Amid von Asn befestigt sind und in den meisten Fällen die Einleitung Schritt erfolgt in der Notaufnahme als das Polypeptid Kette bewegt sich in das Lumen- 34. Der erste Schritt der N- Glykosylierung ist die Übertragung einer vierzehn-Zucker Kern-Struktur, bestehend aus Glukose (Glc), Mannose (Mann) und N- Acetylglucosamin (GlcNAc) (d. h. Glc3Man9GlcNAc-2) aus einem ER Membran-Lipid durch eine Oligosaccharyltransferase 35,36. Weitere Schritte, wie Dekolleté oder Übertragung von Glukose Rückstände sind in der Notaufnahme von spezifischen Glycosidases und Glycosyltransferases katalysiert. Einige Proteine, die ER verlassen und bewegen in die Golgi können weiter verarbeiteten 37sein. O- Glykosylierung bezieht sich auf die Zugabe von Glykoproteinen, in der Regel auf der Seite Kette Hydroxylgruppe des Ser oder Thr Rückstände, und diese Änderung tritt ganz in die Golgi-Komplex 33,34. Gibt es mehrere O- Glykan-Strukturen, die von N- Acetylglucosamin, Fucose, Galaktose gebildet werden können, und Sialinsäure Säure mit jeder Monosaccharid hinzugefügt sequenziell 33.
Während keine bestimmte Reihenfolge als Voraussetzung für viele Arten von O- Glykosylierung identifiziert wurde eine gemeinsame Konsensussequenz Nzugeordnet wurde-Änderung verbunden: Asn-X-Ser/Thr/Cys, wo X kann jede Aminosäure sein mit Ausnahme von Pro- 33. STIM1 EFSAM enthält zwei dieser Konsens N- Glykosylierung Seiten: Asn131-Trp132-Thr133 und Asn171-Thr172-Thr173. In der Tat haben frühere Studien gezeigt, dass EFSAM N- glykosylierten in Säugerzellen bei Asn131 und Asn171 38,39,40,41sein kann. Jedoch frühere Studien der Folgen der N- Glykosylierung auf SOCE inkongruent gewesen, unterdrückt was darauf hindeutet, potenzierte oder keinen Einfluss durch diese Post-translationale Modifikation am SOCE Aktivierung 38,= "Xref" > 39,40,41. Forschung über die zugrunde liegenden biophysikalischen, biochemischen und strukturelle Folgen der EFSAM N- Glykosylierung unbedingt so verstehen die rechtlichen Auswirkungen dieser Änderung. Aufgrund der Voraussetzung für hohe homogene Proteine in diesen in-vitro- Experimenten wurde eine Website selektiv kovalent Glukose Moieties EFSAM beimessen angewendet. Interessant ist, verursacht Asn131 und Asn171 Glycosylation Strukturveränderungen, die innerhalb des EFSAM Kerns konvergieren und verbessern die biophysikalischen Eigenschaften, die STIM1-vermittelten SOCE 42zu fördern.
Die chemische Befestigung der a1 Gruppen mit Cys Thiole hat durch eine Samen-Arbeit etabliert, die die Nützlichkeit dieser enzymfreien Ansatz zum Verständnis der standortspezifischen Auswirkungen der Glykosylierung auf Protein-Funktion 43 erstmals nachgewiesen , 44. jüngerer Zeit und in Bezug auf STIM1, die Asn131 und Asn171 Rückstände waren, Cys mutiert und glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] wurde verwendet, um Glukose kovalent mit kostenlosen Thiole 42zu verknüpfen. Hier beschreiben wir dieses Konzept bei dem nicht nur Mutagenese benutzt Website bestimmte Cys Rückstände für Änderung zu integrieren, sondern gilt auch Lösung Kernspinresonanz (NMR) Spektroskopie schnell Änderung Effizienz und strukturelle bewerten Störungen durch die Glykosylierung. Insbesondere diese allgemeine Methodik ist leicht anpassbar Studie zu den Auswirkungen der beiden O- oder N- Glykosylierung eines rekombinant Protein produziert.
Protokoll
1. Polymerase-Kettenreaktion (PCR)-vermittelte Site-verwiesene Mutagenese für die Einbeziehung der Cys in eine bakterielle Haustier-28a Expressionsvektor.
- Bestimmen die Konzentration des pET-28a Vektors (d.h. doppelte stranded DNA) mit einem ultravioletten (UV) Aussterben Koeffizienten von 0.020 (μg/mL) cm -1 bei 260 nm.
- Synthetisieren ein paar ergänzende mutagene Zündkapseln für jede Cys Mutation die ich) gibt es ein Minimum von 15 Nukleotide komplementär zu der Vorlage, bevor das erste Base Missverhältnis und 15 Nukleotide komplementär zu der Vorlage nach der endgültige Basis Missverhältnis, Ii) total Grundierung Länge nicht mehr als 45 Nukleotiden und Iii) ein Guanin und Cytosin befindet sich an der ersten und letzten Nukleotid Position jedes Primers (Tabelle 1). Primer-Synthese erfolgt mit Hilfe einer 0,025 μmol Skala und Patrone Reinigung zu gewährleisten.
- Mit einem High-Fidelity DNA-Polymerase, zwei 20 µL PCR Reaktionsgemischen eingerichtet: eine mit der forward Primer und die zweite mit der rückwärts-Primer. Bereiten Sie jede Mischung Endkonzentrationen von 1 X PCR-Puffer mit 1,5 mM MgCl 2, 0,2 mM dNTPs, 0,5 μM Grundierung, 0.4 μl DMSO enthalten 1.25 ng/μl Schablone DNA, 0,02 U/μl High Fidelity DNA Polymerase.
- Zyklus thermisch getrennte Mischungen mit einem drei-Stufen-Protokoll: 98 ° C für 30 s (Denaturierung), 53-56 ° C für 30 s (Tempern), 72 ° C für 30 s kilobase(kb) -1 (Erweiterung) der DNA-Vorlage. Wiederholen Sie das Temperaturprogramm für 5 Zyklen und fügen Sie eine endgültige 72 ° C Erweiterung Schritt für 7,5 min.
- Nach der ersten PCR mit vorwärts- und Grundierungen in getrennten Röhren, kombinieren Sie die Produkte in einer einzigen Röhre (d. h. insgesamt 40 μl) und die PCR-Reaktion für eine zusätzliche 20 Zyklen mit den gleichen Radsport Parametern, wie unter Punkt 1.4 beschrieben.
- Electrophorese 15 μl des PCR Reaktionsgemisches auf einem 1 % (w/V) Agarose-Gel mit 0,5 X Tris, Essigsäure, Ethylen Diamin Tetra Acetic Acid (EDTA) Puffer (TAE) ausgeführt. Als Steuerelemente electrophorese eine äquivalente Menge an Schablone DNA, die nicht durch PCR verstärkt worden und eine aliquote Referenz DNA-Leiter enthält Marker Bands sowohl größer und kleiner als die erwartete PCR Produktgröße.
- Nach Elektrophorese bei 120 V 40 min, Tauchen Sie das Gel in Wasser mit 0,5 μg/mL Interkalation Bromid und schütteln für 30 min bei Raumtemperatur. Bestätigen Sie die Full-length Vorlage hat wurde verstärkt durch die mutagenen Primer als Erhöhung in der relativen Interkalation Bromid Fluoreszenz Intensität der verstärkten Band im Vergleich zur Vorlage Kontrollbande unter UV-Licht (302 nm).
- Wenn keine Verstärkung ersichtlich ist, wiederholen Sie die PCR bereinigt Anlasstemperatur in Schritten von 0,5 ° C zwischen 53-56 ° C Temperaturbereich.
- Nach der Bestätigung der Verstärkung der Vorlage durch die mutagenen Primer, behandeln die restlichen ~ 25 μL des Reaktionsgemisches PCR mit dem Restriktionsenzym DpnI methylierte Schablone DNA zu verdauen. Verwenden Sie DpnI 0,5 µL (10 Einheiten) pro 25 μL PCR Reaktionsmischung und eine Endkonzentration von 1 × DpnI Reaktion Puffer. 2,5 h bei 37 ° c inkubieren
- Nach Vorlage Verdauung, ~ 5-10 μL der verdauten Mischung zu 100 µL der Hitze Schock kompetente DH5α Esherichia coli Zellen in einem 1,75 mL Microcentrifuge Schlauch hinzufügen. Inkubieren Sie die Zelle-DNA-Mischung auf Eis für 60 min.
- Hitzeschock Zelle-DNA-Gemisch in den Microcentrifuge Schlauch bei 42 ° C für 45 s auf einen Block, trockene Hitze. Nach Inkubation der Mischung für 3 min auf Eis, 900 μl Raumtemperatur Luria-Bertani Brühe (LB) zu den Zellen und übertragen die gesamte Zellsuspension in ein steriles 14 mL Rundboden Röhrchen.
- Die Zellsuspension bei 37 ° C für 90 min mit konstanter Schütteln bei 190 u/min inkubieren.
- Anschließend die Zellsuspension zurück auf ein 1,75 mL Microcentrifuge Schlauch und Zentrifuge bei 10.000 x g für 5 min bei Raumtemperatur überweisen.
- Nach der Zentrifugation 900 μl des Überstands zu entfernen und erneut die Bakterienzellen durch sanfte Pipettieren in die restlichen 100 μL lb
- Übertragen die resultierende konzentrierte Zellsuspension auf eine LB-Agar-Platte mit Antibiotikum, das selektiv für den Ausdruck Vektor (d. h. 60 μg/mLKanamycin). Aseptisch verbreiten die Suspension gleichmäßig auf der Agarplatte und inkubieren ~ 16 h bei 37 ° c
- Am Folgetag impfen eine einzige Kolonie von der Platte in 5 mL liquid LB mit dem Antibiotikum Selektionsdruck (d. h. 60 μg/mL Kanamycin). Wachsen die Flüssigkultur über Nacht bei 37 ° C mit konstanter Schütteln bei 37 ° c
- Isolieren und reinigen das propagierte Plasmid aus den E. Coli-Zellen mit einem kommerziell erhältlichen Kit basiert auf der alkalischen Lyse Verfahren 45.
- Bestätigen die Mutation von Interesse ist vorhanden und in den richtigen Leseraster durch Sanger-Sequenzierung von Plasmid 46.
2. Einheitliche 15 N-markierten Protein-Expression in BL21 ΔE3 Escherichia coli.
Hinweis: verschiedene rekombinante Proteine erfordern unterschiedliche Ausdruck Bedingungen. Im folgenden finden Sie das optimierte Verfahren zur Expression des menschlichen Proteins STIM1 EFSAM.
- Dem Expressionsvektor beherbergen die Cys-Mutationen (d. h. Tier-28a-EFSAM) in BL21 ΔE3 Codon (+) Hitze Schock kompetente Zellen verwandeln und Platte auf LB-Agar-Platten mit der antibiotische Selektionsdruck wie 1,9-1.14 beschrieben) mit der folgende Änderungen: Platte direkt eine 150 μL aliquoten aus insgesamt ~ 1.000 μl Zellsuspension auf der LB-Agar-Platte ohne die Notwendigkeit für die Konzentration der Zellen in einem Microcentrifuge Schlauch durch Zentrifugation.
- Am Folgetag aseptisch übertragen eine einzige Kolonie in einen 200-mL-Erlenmeyerkolben mit 20 mL LB ergänzt mit dem entsprechenden Antibiotikum (d. h. 60 μg/mL Kanamycin für Haustier-28a-EFSAM). Wachsen diese flüssige Starterkultur über Nacht (d.h. ~ 16 h) bei 37 ° C mit konstanter Schütteln bei ~ 190 u/min.
- Am selben Tag als Schritt 2.2 vorbereiten M9 Medium für 15 N beschriftet Proteinexpression durch Autoklavieren Puffer 1 L M9 Salze (42 mM Na 2 HPO 4, 22 mM KH 2 PO 4, 8,6 mM NaCl, pH 7,4) in einem 4 L-Erlenmeyer-Kolben. Nach dem Abkühlen Filtern eine Mischung von 20 % (w/V) D-Glucose, 1 M CaCl 2, 1 M Thiamin, 1 M MgSO 4, 1 mg/mL Biotin und 0,2 g/mL 15 N-NH 4 Cl durch einen 0,2 μm sterile Spritze Filter in der 1 L sterile M9 Salzlösung so dass die Endkonzentrationen dieser Komponenten sind 0,2 % (w/V) D-Glucose, 100 μM CaCl 2, 50 μm Thiamin, 1 mM MgSO 4, 1 μg/mL Biotin und 1 mg/mL 15 N-NH 4 Kl.
- Am nächsten Tag, aseptisch übertragen die 20 mL über Nacht flüssige Starterkultur in ein 50 mL sterile konischem Rohr und Zentrifuge bei 2.400 × g für 15 min auf die Zellen pellet.
- Nach der LB-Medium dekantieren, Aufschwemmen der daraus resultierenden Zelle Pellet in 10 mL M9 minimaler Medium und übertragen Sie die resuspendierte Pellet-Mischung in 1 L des M9 minimaler Medium mit Antibiotikum (d. h. 60 μg/mL Kanamycin).
- Wachsen die 1 L des M9 minimal die bakterielle Starterkultur bei 37 ° C und ~ 190 u/min konstant schütteln bis die Extinktion bei 600 nm (OD600) erreicht-haltigem Medium ~0.6-0.8.
- Wenn der Specfified OD600-Bereich erreicht ist, fügen Sie 200 μM Isopropyl β-D-1-Thiogalactopyranoside (IPTG) induzieren Proteinexpression.
- Nach IPTG hinaus weiter Inkubation der Zellen für die Proteinexpression bei Raumtemperatur mit konstanter Schütteln bei ~ 190 u/min für ~ 16 h (d.h. über Nacht).
- Am nächsten Tag ernten die Bakterien durch Zentrifugation bei ~ 10.000 × g, 4 ° C für 30 min.
- Dekantieren die LB und die Zelle Pellet in ein 50 mL konische Röhrchen zu übertragen. Speichern Sie die Pellets bei-80 ° C bis Reinigung.
3. Reinigung von rekombinanten Proteinen aus E. Coli.
Hinweis: verschiedene rekombinante Proteine erfordern unterschiedliche Reinigung Verfahren. Im folgenden finden Sie das Protokoll für 6 & #215; Sein-tagged EFSAM Reinigung von Einschlusskörperchen von pET-28a Konstrukt ausgedrückt.
- Homogenisieren manuell das gefrorene Bakterienzelle Pellet in 6 M Guanidin-HCl, 20 mM Tris-HCl (pH 8) und 5 mM β-Mercaptoethanol mit einer motorisierten 10 mL-Transfer-Pipette. Fügen Sie ca. 40 mL Guanidin-HCl pro 5 mL Nasszelle Pellet für diesen Schritt.
- Folgenden ein 90 min Inkubation bei Raumtemperatur mit konstanter Rotation in einem Ofen Hybridisierung Zentrifugieren die Mischung ~ 15.000 × g, 8 ° C für 40 min. das lösliche Protein die unlöslichen Zellenrückstand (d. h. Pellet) getrennt Mischung (d.h. überstand).
- Fügen Sie 750 µL von 50 % (V/V) Ni 2 +-Nitrilotriacetic Säure Agarose bead Gülle, die geklärte lysate und weitere 90 min bei Raumtemperatur mit Inversion in einem Ofen Hybridisierung inkubieren.
- Erfassen Sie anschließend 6 × sein-tagged Proteins gebunden an die Ni 2 + durch das Sammeln der Agarose-Perlen in einer Schwerkraft fließen Protein Reinigung Spalte. Ermöglichen die lysate durchfließen der Spalte vollständig vor seinem Umzug nach Schritt 3.5.
- Waschen die gesammelten Perlen dreimal mit 10 mL 6 M Harnstoff, 20 mM Tris-HCl pH 8 und 5 mM β-Mercaptoethanol. Sicherzustellen, dass die gesamte 10 mL die Spalte vor jeder nachfolgenden 10 mL durchläuft Wash
- Eluieren die Proteine in einer Reihe von 2 mL Fraktionen mit 6 M Harnstoff, 20 mM Tris-HCl pH 8, 300 mM Imidazol und 5 mM β-Mercaptoethanol mit einer 90 s Inkubationszeit zwischen Brüchen. Sicherzustellen, dass die gesamte 2 mL der Spalte vor jedem nachfolgenden Elution Schritt durchläuft.
- Zu diesem Zeitpunkt bestätigen das Protein des Interesses in der eluierten Bruchteile von Coomassie Blau gefärbten Sodium Dodecyl Sulfat Polyacrylamid Gelelektrophorese (SDS-PAGE) mit der Methode von Laemmli 47 vorliegt. Bewerten der Proteingröße, Menge und Reinheit im Vergleich gegen standard Molekulargewicht Marker-Bands, die beide sind kleiner als und größer als die erwarteten Molekulargewicht des Proteins des Interesses.
- Der eluierten Proteinfraktionen in eine Dialysemembran mit einem 3.500 Da Molekulargewicht cutoff zu bündeln und in 1 L Umfaltung Puffer (20 mM Tris, 300 mM NaCl, 1 mM DVB-t, 5 mM CaCl 2, pH 8) bei 4 ° C inkubieren Übernachtung während der Puffer ist durch eine Magneti gerührt wird C Rührer.
- Nach ~ 16 h Umfaltung Zeit ~ 1 U Thrombin pro mg Protein direkt an die Dialyse-Tasche und inkubieren Sie bei 4 ° C für eine zusätzliche ~ 24 h.
- Überprüfen Sie dem Ausmaß der 6 × seiner Tag Spaltung von Coomassie blaue Färbung von ~ 15 μl Protein Aliquote entnommen die Dialyse-Tasche vor und nach der Inkubation mit Thrombin die auf denaturierenden Polyacrylamid-Gele (SDS-PAGE) mit electrophoresed sind die Methode von Laemmli 47. Wenn eine ~ 2 kDa-Verschiebung in der Migration beobachtet wird, entspricht das Molekulargewicht des gespalten 6 × seinen Tag fahren Sie mit Schritt 3.11; Wenn ein Bruchteil der unverdaute Eiweiß bleibt, die nachweisbar durch Coomassie blaue Färbung ist, fügen ~0.2 U Thrombin pro mg Protein direkt um die Dialyse-Tasche und inkubieren Sie bei 4 ° C für eine zusätzliche ~ 24 h
- Einsatz Größe Ausgrenzung oder Ionenaustausch-Chromatographie, das Protein weiter zu reinigen. Anion-Austausch-Chromatographie von EFSAM, entfernen Sie die Proteinlösung aus der Dialyse-Tasche und Konzentrat ~ 10-divisibel mit einer Ultrafiltration zentrifugale Konzentrator mit einem 10.000 Da Molekulargewicht cutoff. Anschließend wieder Verdünnen der Lösung ~ 20-fold in einem NaCl-freie Puffer (20 mM Tris, 5 mM CaCl 2, 1 mM DTT, pH 8).
- Equilibrate eine abgepackte Anion Austausch Spalte mit 10 Spalte Bände der NaCl-freie Puffer im Schritt 3.11 beschrieben. Equilibrate mit Puffer geladen in einen Luer-Lock-Spritze enthält keine Luftblasen durch Drücken der Lösung durch die Säule in gewissem Sinne tropfenweise und Vermeidung von Spritze Drücke die stetige Ströme von Lösung verlassen die Spalte verursachen. Verwenden Sie ein starkes Anion Exchanger (z.B. vernetzte Agarose mit quaternären Ammonium Funktionsgruppen).
- Laden die Proteinlösung verdünnt in NaCl-freie Puffer (Schritt 3.11) auf die Säule, wie unter Schritt 3.12.
- Der Proteine in einem Farbverlauf eluieren [d.h. 0 - 60 % (V/V)] der Erhöhung der NaCl-Puffer (20 mM Tris, 1 M NaCl 5 mM CaCl 2, 1 mM DTT, pH 8) mit einem zwei-Pumpen schnell Protein Flüssigkeitschromatographie (FPLC) System. Legen Sie das FPLC System ~1-1.5 mL Fraktionen zu sammeln und das Protein Elution Monitorprofil mit UV-280 nm Absorption und ein Durchfluss 0,5 mL/min
- Identifizieren die Elution Spitzen und Brüche, enthält das Protein des Interesses sowie Protein Reinheit von Coomassie Blau gefärbten SDS-PAGE Gelen mit der Methode von Laemmli 47.
- Pool Brüche zeigen > 95 % (d. h. als Brüche, die nur eine einziges Protein Band auf Coomassie-Blau gefärbten Gele zeigen genommen) in eine Dialyse Tasche und Austausch in experimentellen Puffer des Interesses durch Dialyse wie unter Schritt 3.8.
4. Chemische Befestigung des Glukose-5-MTS an Protein durch Dialyse.
- Prepare a 55 mM Stammlösung der N-(β-D-glucopyranosyl)-N '-[2-methanethiosulfonyl) Ethyl] Harnstoff (Glukose-5-MTS) von 10 mg der Verbindung im 500 μl von 100 % (V/V) DMSO auflösen. Speichern Sie nicht verwendete Glucose-5-MTS solubilisiert in DMSO bei-20 ° c
- Bereiten die Protein-Probe für die Modifikation von dialyzing 1,5 mL ~ 60 μM Protein in 1 L der Änderung Puffer bestehend aus 20 mM MOPS, 150 mM NaCl, 5 mM CaCl 2 und 0,1 mM DÄMMUND, pH 8,3. Verwenden Sie eine Dialyse Membran Molekulargewicht Cutoff, die kleiner ist als die Größe des Proteins geändert wird (z.B. Nutzung einem 3.500 Da Cutoff für ~ 17.500 Da EFSAM).
- Nach 24 h bei 4 ° C, die Probe von der Dialyse-Tasche zu einem Microcentrifuge Schlauch übertragen. Fügen Sie das DMSO solubilisiert Glukose-5-MTS, eine Endkonzentration von 2 mM.
- Inkubieren Sie die Probe in der Dunkelheit für 1 h bei Raumtemperatur. Während der Inkubationszeit 1 h, mischen Sie die Lösung durch sanftes Antippen des Rohres alle 10 min.
- Anschließend wieder tauschen das Protein in den endgültigen experimentelle Puffer enthält keine Reduktionsmittel durch Dialyse bei 4 ° C, wie in Schritt 4.2 oder durch zentrifugale Ultrafiltration beschrieben. Für die Ultrafiltration Verfahren konzentrieren die ~1.5 mL Protein Probe < 0,5 mL und anschließend in der gleichen Konzentrator mit dem experimentellen Puffer verdünnen. Wiederholen Sie diesen Schritt Konzentration-Verdünnung zwei weitere Male, so dass der gesamte Austausch ein Minimum von 30 × 30 × 30 ist = 27,000-fold. Verwenden Sie für EFSAM, 20 mM Tris, 150 mM NaCl, 5 mM CaCl 2, pH 7.5 als experimentelle Puffer.
- Die Probe vorbereiten Electrospray Ionisierung Massenspektrometrie durch Dialyse oder Ultrafiltration Austausch wie in 4.2 und 4.5, bzw. in Ammonium Bicarbonat 25 mM oder 25 mM Ammoniumacetat beschrieben. Wenn Dialyse verwenden, stellen Sie sicher, Sie tauschen mindestens dreimal um jede verbleibende NaCl und CaCl 2 Salze zu entfernen.
- Bestimmen die genaue Masse (d. h. ± 1 Da) des Proteins des Interesses mit Electrospray Ionisierung mass Spectrometry 48 , 49. Erwarten, dass jeder kovalenten Traubenzucker zusätzlich zu einem Cys Thiol über die Methanethiosulfonate chemische 281.3 Da das Protein hinzu Masse (d.h. 360.4 Da für die Glukose-5-MTS addieren und Subtrahieren von 79,1 Da für die CH 3, also 2 verlassen während kovalente Gruppe Attachement).
5. Lösung NMR Bewertung der modifEffizienz und strukturelle Störungen Ication.
- Stellen Sie sicher die Konzentration der geänderten Protein ist > 100 μM nach dem Glukose-Anlage und der letzte Buffer Tausch. Für EFSAM, schätzen die Proteinkonzentration mit einem UV-Aussterben-Koeffizienten bei 280 nm von 1,54 (mg mL – 1) cm – 1.
- Ergänzung der Proteinlösung mit 60 μM 4,4-dimethyl-4-silapentane-1-sulfonic Säure (DSS) für Shims und Puls Kalibrierung und 10 % (V/V) D 2 O für die Signal-Sperre. Für hohen Signal-Rausch-Einsatz 600 μL Proben in Frequenz abgestimmt 5 mm NMR Röhrchen, eingefügt in eine mindestens 600 MHz-Spektrometer mit einer dreifachen Resonanz HCN Kryo-Sonde ausgestattet.
- Standard 1 H - 15 N HSQC-Spektren wie zuvor detaillierte 50 , 51 auf Temperatur, 1 H und 15 N Sweep breiten, Transient und Inkrement zu sammeln Einstellungen für die jeweiligen Probe. Für EFSAM Spektren, Verwendung 20 ° C, 256 1 H Transienten, 64 15 N Dimension Schritten und 1 H und 15 N fegen breiten bis 8.000 und 1.800 Hz "bzw." festgelegt.
- Nach der Übernahme des glykosylierten Proteinen Spektrums hinzufügen Dithiothreitol (DTT) NMR-Probe aus einem 1 M bestand, eine Endkonzentration von 15 mM. Die DVB-t-Glukose glyko-entzieht das Protein durch Reduktion der Disulfid-vermittelten Anlage.
- Erwerben eine zweite 1 H - 15 N HSQC-unter diesen reduziert/unverändert bietet ein Referenzspektrum um Änderung Effizienz und strukturelle Störungen verursacht durch die Glukose-Anlage zu bewerten.
- Mit NMRPipe wie bisher 52 detaillierte NMR Daten verarbeiten. Sicherzustellen, dass die Verarbeitung minimal Datenkonvertierung, Ausstieg, solvent Unterdrückung, Fourier-Transformation und erste Visualisierung der Spektren umfasst.
- Assess Modifikation Effizienz durch Messen der Amid Peak Intensitäten und chemischen Verschiebungswerte in den veränderten und reduzierte Spektren mit dem NEASY-Plugin auf CARA 53. Sicherstellen Sie, dass die Peak-Intensität der Cys Amid bewerten sowohl in Glukose befestigt und reduziert Spektren. Wenn der Cys Amid in beiden Spektren nicht zuverlässig gefunden werden kann, verwenden die Intensitäten der Rückstände, die neben der Cys als ein auslesen.
- Berechnen die Effizienz als die Intensität der Amid aus dem Cys-modifizierte Spektrum durch die Intensität der Amid aus dem Cys reduziert (d. h. DTT-behandelt) Spektrum geteilt, multipliziert mit 100:
 , wo ich bin die Intensität der Amid im Cys-modifizierte Spektrum ist und I R die Intensität der Amid im Cys verringert Spektrum ist. Alternativ, bewerten die mittlere Effizienz über mehrere Amid-Gipfel:
, wo ich bin die Intensität der Amid im Cys-modifizierte Spektrum ist und I R die Intensität der Amid im Cys verringert Spektrum ist. Alternativ, bewerten die mittlere Effizienz über mehrere Amid-Gipfel:
 , wo Effizienz i der getrennt ermittelten Effizienz berechnet für jede Rückstände, i, und n ist die Gesamtzahl der Rückstände in der Berechnung verwendet.
, wo Effizienz i der getrennt ermittelten Effizienz berechnet für jede Rückstände, i, und n ist die Gesamtzahl der Rückstände in der Berechnung verwendet. - Chemische Verschiebung Störungen (CSP) durch die chemische Verschiebung-Unterschiede zwischen den beiden Spektren in 15 N und 1 H Abmessungen der einzelnen Gipfel beobachtet und Normalisierung für die größeren 15 N chemische Verschiebung mit unterschiedlichen berechnen die folgende Gleichung:
 , wo ΔH die ppm-Änderung in der Proton-Dimension ist und ΔN die ppm-Änderung in der Stickstoff-Dimension ist.
, wo ΔH die ppm-Änderung in der Proton-Dimension ist und ΔN die ppm-Änderung in der Stickstoff-Dimension ist.
Ergebnisse
Der erste Schritt dieses Ansatzes erfordert die Mutagenese der Kandidat Glykosylierung Rückstände Cys Rückstände die modifizierbar mit Glukose-5-m EFSAM sein können hat keine endogenen Cys-Rückstände, also keine besonderen Überlegungen vor dem vorgenommen werden müssen die Mutagenese. Allerdings müssen native Cys Rückstände nicht veränderbaren Rückstände vor der Durchführung der beschriebenen Chemie mutiert werden. Um die native Struktur minimal zu bewirken, empfiehlt sich die Durchführung einer globalen Sequenzalignment des Proteins des Interesses und bestimmen, welche andere Rückstände sind die meisten häufig bei der endogenen Cys Position(en) gefunden. Cys Mutation auf diese andere Rückstände, die natürlich in anderen Organismen vorkommen haben die geringsten Auswirkungen auf Protein-Struktur. Wenn der endogene Cys Rückstand streng konserviert ist, empfiehlt es sich, an Ser die meisten strukturell ähnlich Cys ist mutiert. Abbildung 1 zeigt eine typische PCR Mutagenese Gel Bewertung des Erfolgs der PCR-Reaktion, mit der amplifizierten DNA-Probe Nachweis besteht höheren Intensität als eine Kontrolle Menge an unverstärkten Vorlage Haustier-28a DNA, die für die PCR verwendet wurde. Die nächsten Schritte umfassen Vorlage DNA Verdauung und Umwandlung in E. Coli Plasmid Reparatur. Nach Plasmid Vermehrung in Flüssigkultur, Plasmid-Isolierung und Bestätigung für das Auge durch Sequenzierung kann der mutierte Vektor für Protein-Expression verwendet werden. Abbildung 2A zeigt eine typische Elution Profil des EFSAM aus der Anion Austausch Spalte relativ zur Erhöhung der NaCl-Konzentration. Abbildung 2 b zeigt die Reinheit des EFSAM auf Coomassie Blau gefärbten SDS-PAGE Gelen.
Nach dem Erwerb reinen Proteins, wird eine Reihe von Dialyse-Schritte verwendet, um Glukose glyko-über die MTS-Reaktion mit freien Thiol befestigen. Abbildung 3A zeigt ein Bild des typischen Aufbaus der ein kleines Protein Volumen versiegelt in der Dialyse Membran durch Membran Clips und in eine große 1 L-Becher mit den Puffer des Interesses enthalten. Eine erste Überprüfung des Erfolgs der Änderung kann durch Massenspektrometrie erfolgen. Abbildung 3 b zeigt eine repräsentative Elektrospray-Massenspektrum von EFSAM zu einer einzigen Cys Thiol geändert. Nach Einrichtung des Protokolls für ein bestimmtes Protein, Änderung Effizienz und strukturelle Störungen beurteilt werden können aus einem einzigen einheitlich 15N beschriftet Probe. 1H -15N-HSQC-Spektrum wird vor und nach der Zugabe des Reduktionsmittels DVB-t (Abb. 4A) erworben. Berechnungen der Modifikation Effizienz können über einen Vergleich der Amid Peak Intensitäten in den veränderten und reduzierte Spektren wie im Protokoll Schritt 5.8 (Abbildung 4 b) erfolgen. Schließlich, wenn chemische Verschiebung Zuordnungen für ein Protein, das LSP bekannt sind welche korrelieren mit den strukturellen Veränderungen im Schritt 5.9 (Abbildung 4) berechnet werden kann.
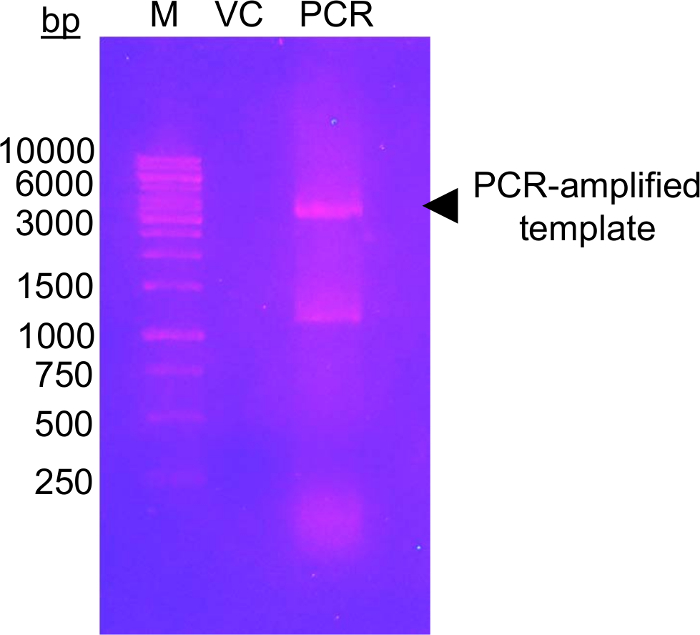
Abbildung 1: DNA-Agarosegel mit Verstärkung Check Vorlage Vektors mit mutagenen Primer.
Das Bild zeigt eine 1,0 % (w/V) Agarose-Gel mit DNA-Marker (M), Vektor-Kontrolle (VC) und PCR verstärkt Vorlage (PCR). Die DNA wurde durch Elektrophorese bei 120 V für 45 min in 0,5 X TAE Puffer getrennt. Insgesamt 0,5 ng von VC geladen wurde, entspricht die Höhe der Vorlage in der PCR-Spur geladen. Das Gel war fleckig mit Interkalation Bromid (~0.5 μg/mL) für 20 min vor Visualisierung unter UV-Licht (302 nm). Das Gel zeigt ein hohes Maß an amplifizierten DNA in der Nähe der erwarteten Größe des Vektors (schwarze Pfeilspitze). Das zweite Band in der PCR-Gasse zwischen 1.000 und 1.500 bp Marker Bänder wahrscheinlich stellt ein unspezifisch amplifizierten PCR-Produkt. Die Intensität der amplifizierten DNA muss höher als die VC Intensitätsstufe erfolgreich anzusehen sein. Mehrere andere DNA-Farbstoffe können als weniger mutagenen, sichereren Alternativen zu Interkalation Bromid Färbung (siehe z. B. 57) verwendet werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
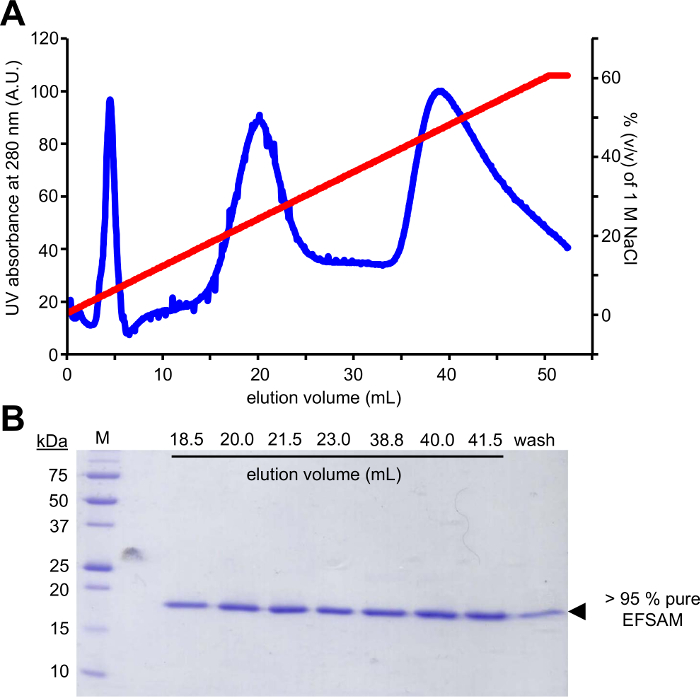
Abbildung 2: Typische chromatographische Reinigung und Reinheit überprüfen für STIM1 EF-SAM.
(A) Anion Austausch-Chromatographie Elution Profil des STIM1 EF-SAM. Nach der manuellen Bindung wird EF-SAM, Anion Austausch Spalte (Q FF) auf dem grundlegenden pH-Wert und geringer NaCl-Konzentration mit einer Spritze und AKTA FPLC (GE Healthcare) verwendet, um das Protein mit einem NaCl-Gradienten zu eluieren. Die Elution wird durch die AKTA mit UV-280 nm Signal über ein 0-60 % (V/V) Gefälle von 1 M NaCl überwacht. (B) Coomassie Blau gefärbten SDS-PAGE Gel der Elution Bruchteile von (A). Das denaturierenden Protein Gel zeigt, dass EF-SAM in zwei großen Gipfel bei ~ 250 mM und ~ 450 mM NaCl elutes. Die Reinigung Protokoll ergibt > 95 % reines EF-SAM als belegt durch das Fehlen von jeder Verunreinigung band zeigt sich in der Coomassie Blau gefärbten Gel. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Dialyse-Einrichtung und Bestätigung der in Vitro Protein Glycosylation.
(A) typische Dialyse Setup verwendet zur in-vitro- Befestigung von Glukose an der Cys Thiol über die MTS-Reaktivität. Das Bild zeigt ~1.5 mL Protein enthaltenen Dialyse Schlauch gegen gepuffert ~ 1 L des experimentellen Puffer. Es ist wichtig, dass der Puffer ständig gerührt ist, um komplette Austausch. Das Bild zeigt einen Microcentrifuge Schlauch abgeschnitten, um die überschüssige Dialyse-Schläuche zu verhindern, Untergang der Dialyse-Tasche und Beschädigungen durch die rotierenden Stir Bar. (B) Electrospray Ionisierung Massenspektrum des modifizierten Asn171Cys EF-SAM-Proteins. Massenspektrometrie ist eine bequeme und genaue Ansatz zu beurteilen, ob die Änderung erfolgreich war. Eine typische Masse Chromatogramm zeigt sich mit den theoretischen und gemessenen Massen von unveränderten und veränderten Asn171Cys EF-SAM angegeben. Die Mehrheit der Probe entspricht Masse ein Makromolekül, die innerhalb von ~1.3 Da der erwarteten theoretischen Masse von Glukose konjugiert Asn171Cys EF-SAM ist. Die Daten in (B) ist replotted und von 42geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Lösung NMR Bewertung der Änderung Effizienz und strukturelle Störungen von einer einzigen NMR-Probe.
(A) 1 H -15N-HSQC-spektrale Überlagerung der Glukose-konjugiert Asn131Cys EFSAM vor (rote Crosspeaks) und nach (schwarzer Crosspeaks) die Zugabe von 15 mM DTT. Das Overlay zeigt mehrere chemische Rückstände-spezifische Amid Schichtwechsel bezeichnend für beide Modifikation von Protein und strukturelle Störungen. Das rote Feld zeigt die Lage der Asn131Cys Amid. (B) Zoomed Blick auf der 1H -15N-HSQC--Region, enthält die Asn131Cys-Amid. Die Intensität des Asn131Cys Amid Peak im modifizierten Spektrum (ichM) dividiert durch die Intensität in der reduzierten (unveränderten) Spektrum (ir) für die Berechnung der Effizienz (dargestellt). Eine Berechnung der mittlere Wirkungsgrad von mehreren betroffenen Rückstände bietet eine bessere Einschätzung der Effizienz, einschließlich einer Schätzung der Fehler. Der mittlere Wirkungsgrad wird für Asn131Cys EFSAM basierend auf 5 Reststoffen (d. h. 129-133) angezeigt. (C) normalisierte chemische Verschiebung Störungen verursacht durch Glukose Konjugation mit dem Asn131Cys EFSAM Protein. Die Reihe von HSQC-Experimenten gesammelt auf einer einzigen Probe vor und nach Ergänzung mit Reduktionsmittel nicht nur ermöglicht eine bequeme Schätzung Modifikation Effizienz durch Peak Intensität Analyse [angezeigt (b)], sondern auch Daten für liefert Bewertung der strukturelle Veränderungen im Zusammenhang mit der Änderung. Glukose-Konjugation verursacht die größten Störungen lokalisiert in der Nähe von Stellung 131; Diese Analyse zeigt jedoch Störungen die unerwartete ausschließlich basierend auf Sequenz Nähe sind, die den Wert in dieser Analyse. Die Daten in (C) replotted und von 42geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
| STIM1 Mutationeine | Richtungb | DNA-Sequenzc | |
| Asn131Cys | nach vorn | 5'-GTCATCAGAAGTATACTGTTGGACCGTGGATGAGG-3 " | |
| Asn131Cys | umgekehrte | 5'-CCTCATCCACGGTCCAACAGTATACTTCTGATGAC-3 " | |
| Asn171Cys | nach vorn | 5'-CCAAGGCTGGCTGTCACCTGCACCACCATGACAGGG-3 " | |
| Asn171Cys | umgekehrte | 5'-CCCTGTCATGGTGGTGCAGGTGACAGCCAGCCTTGG-3 " | |
| ein STIM1 Aminosäure Nummerierung anhand von NCBI Beitritt AFZ76986.1. | |||
| b Die "rückwärts"-Primer entspricht der Reihenfolge der umgekehrte Ergänzung des Primers "vorwärts". | |||
| c Die unterstrichenen Codon Triplett entspricht der Cys-Mutation. | |||
Tabelle 1. Beispiel-Oligonukleotid (Primer) Sequenzen für Asn Cys Mutagenese innerhalb der Haustier-28a STIM1 EFSAM zu konstruieren.
Diskussion
Protein Glycosylation ist eine Post-translationale Modifikation wo Zucker kovalent an Polypeptide in erster Linie durch Verbindungen zu den Aminosäure-Seitenketten befestigt sind. Mehr als 50 % der Säugetiere Proteine sind glykosylierten 54, wo der glycosylierten Proteine anschließend haben können vielfältige Effekte von biomolekularen Bindungsaffinität zu verändern, Protein Falten, Channel-Aktivität verändern, gezielt zu beeinflussen Moleküle für Abbau und zellulären Menschenhandel, to name a Few (rezensiert in33). Die wichtige Rolle der Glykosylierung in Säugetieren Physiologie ist offensichtlich durch mehrere hundert Proteine entwickelt, um die ganze Vielfalt von Säugetieren Glycan Strukturen 33bauen. N- verändert und O- Glykosylierung Muster zugeordnet wurden zahlreiche Krankheit Staaten einschließlich Prostata (erhöhte und verminderte), Brust (erhöht und verringert), Leber (erhöht), Eierstockkrebs (erhöht), Pankreas (erhöht) und Magen-Krebs (erhöht) 55. Darüber hinaus Glykosylierung von Tau, Huntingtin, α-Synuclein gefunden wurde, die Toxizität dieser Zusammenhang mit Parkinson und Alzheimer, Chorea Huntington Erkrankungen 56Proteine regulieren, und eine Gruppe von angeborenen Störungen der Glykosylierung gewesen ermittelt aus erblichen Defekte Enzyme die Glykosylierung 54zu vermitteln. Verständnis der genauen biophysikalischen, biochemischen und strukturelle Auswirkungen der Glykosylierung hat somit das Potenzial enorm beeinflussen unser Verständnis von Protein-Verordnung und Funktion in Gesundheit und Krankheit.
Die zehn Zucker-Bausteine, die zu der Vielfalt der Glycan Strukturen in den Säugetieren Glycome führen gehören Fucose, Galaktose, Glukose, N- Acetylgalactosamine, N- Acetylglucosamin, Glucuron-Säure, Iduronic Säure, Mannose, Sialinsäure Säure und Xylose 33. N- Glykosylierung stets eine N- Acetylglucosamin Zucker direkt an das Protein verbindet, kann O- Glykosylierung von jedem der N- Acetylgalacotose, N- Acetylglucosamin, Xylose Fucose führen, Glukose oder Galaktose kovalent verknüpft, das Polypeptid. Um beginnen zu verstehen, wie diese Zucker direkt an der Oberfläche Protein die biophysikalischen und strukturellen Eigenschaften beeinflussen, beschreiben wir hier einen Ansatz zur Website selektiv Zucker Cys Rückstände über die Thiole entwickelt, in das Protein zuordnen Sequenz. Hier werden die Rückstände, die endogen glykosylierten werden ersetzt durch Cys und in-vitro- über einen einfachen chemischen Ansatz geändert. Auf diese Weise können einzelne und mehrere Glykosylierung Websites bewertet werden, um den Beitrag der jede spezifische Baustelle sowie die kumulierten Änderungen an der Faltung und Stabilität sowie die allgemeine Struktur und Funktion des Proteins herauskitzeln.
Vor kurzem wurde dieser Ansatz erfolgreich mit EFSAM verwendet, um individuell und kumulativ die Rolle der Asn131 und Asn171 N- Glykosylierung Seiten 42zu beurteilen. Mutation, Cys und kovalente Bindung von Glukose an der Asn131 oder Asn171 Seiten offenbart eine verminderte Ca2 + Bindungsaffinität und Stabilität unterdrückt. Wenn die beiden Standorte wurden gleichzeitig mit der Glukose-Anlage, die Rückgänge in verbindliche Affinität geändert und Stabilität wurden potenzierte führt zu verstärkt Oligomerisierung Neigung in Vitro. Strukturell, zeigte der hierin beschriebene Ansatz, dass die Asn131 oder Asn171 Änderungen gegenseitig die Kern-α8-Helix befindet sich auf der SAM-Domäne, unmittelbar angrenzend an das EF-Hand paar durcheinanderbringen. Diese strukturelle Analyse erklärt wie Glukose-Änderungen an der Oberfläche des Proteins führen zu einem konvergierenden und potenzierten Strukturwandel innerhalb der EF-Hand: SAM-Schnittstelle, die letztlich das Protein destabilisiert und SOCE 42erhöht.
Während die Anwendung von dieser Website-selektiven Ansatz half beleuchten auf wie ein Monosaccharid nah an der Oberfläche des EFSAM, Falten, Stabilität und Struktur, dieses Verfahren bewirkt leicht kann geändert werden, um speziell für ER, mehr Kohlenhydrate zu befestigen Golgi und PM Lokalisierung (d.h. Glykosylierung Staaten unterschiedlichen Reifegrad), vorausgesetzt, es ist eine zuverlässige Quelle für diese Kohlenhydrate mit funktionellen Gruppen, die mit Thiole verknüpfen können, wie z. B. MTS MTS vorzuziehen ist, da die Thiol-Modifikation ist reversibel mit einem Reduktionsmittel und ein Referenzspektrum können leicht erworben werden. Dieser Ansatz kann auch zu anderen post-translationalen Moieties anbinden das Protein wie Lipide angepasst werden. Zur gleichen Zeit gibt es mehrere Einschränkungen dieses Ansatzes, die berücksichtigt werden sollten. Zuerst setzt die Methode auf Mutation der Glykosylierung Sites, Cys, die Struktur, Stabilität und Faltung auch in Abwesenheit von Glukose Änderungen beeinträchtigen können. Ebenso müssen auch native Cys-Rückstände im Protein mutiert werden, um Glukose Befestigung an nicht-glykosylierten Standorten zu verhindern. Darüber hinaus fördert die Zugabe von Cys Rückstände oft Einbeziehung Körper Bildung in Bakterien durch Cys Vernetzung und Fehlfaltung, wodurch Reinigung schwieriger. Dennoch bietet diese Website-Cys-Vernetzung selektiv hierin beschriebenen kontrollierten Mittel um die strukturellen, biochemischen und biophysikalischen Auswirkungen von bestimmten Glykosylierung Websites in Experimenten zu necken erfordern hohe homogene Protein. Die Auswirkungen von nicht-Native Cys Rückstände auf die Struktur, Stabilität und Faltung können einfach festgestellt werden keine Änderungen im Vergleich zu Wildtyp-Protein Attribute 42. Zusammen mit funktionellen Daten in eukaryontischen Zellen die Änderung blockieren mutierte Versionen des Proteins (z.B. Asn-zu-Ala) zum Ausdruck bringen, wird die derzeit beschriebene Vorgehensweise neue Einblicke in die strukturelle Mechanismen des Proteins Ausbeute. Verordnung durch die post-translationalen Modifikationen.
Danksagungen
Diese Forschung wurde unterstützt durch die Naturwissenschaften und Engineering Research Council of Canada (05239, B.P.S), kanadische Stiftung für Innovation/Ontario Research Fund (zu B.P.S), Prostata Krebs kämpfen Foundation - Telus Ride für Papa (zu B.P.S) und Ontario Graduate Stipendium (für Y.J.C. und N.S.).
Materialien
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
Referenzen
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), Epub 2005 May 2002 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L. Endoplasmic Reticulum Stress in Health and Disease. Agostinis, P., Afshin, S. , Bristol-Myers Squibb. Syracuse, NY. 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten