Method Article
Sistema Induzível de Expressão Multigênica mediada por polimerização T7 de T7, pMGX
Neste Artigo
Resumo
Este estudo descreve métodos para a co-expressão mediada por T7 de múltiplos genes de um único plasmídeo em Escherichia coli usando o sistema de plasmídeo pMGX.
Resumo
A co-expressão de múltiplas proteínas é cada vez mais essencial para a biologia sintética, estudando complexos proteína-proteína e caracterizando e aproveitando os caminhos biossintéticos. Neste manuscrito, descreve-se o uso de um sistema altamente eficaz para a construção de operões sintéticos multigenes sob o controle de uma polimerase de ARN T7 induzível. Este sistema permite que muitos genes sejam expressos simultaneamente a partir de um plasmídeo. Aqui, um conjunto de quatro vetores relacionados, pMGX-A, pMGX-hisA, pMGX-K e pMGX-hisK, com o marcador selecionável por resistência à ampicilina ou kanamicina (A e K) e possuindo ou não uma hexahistidina N-terminal A tag (his) é divulgada. São fornecidos protocolos detalhados para a construção de operões sintéticos usando este sistema de vetores juntamente com os dados correspondentes, mostrando que um sistema baseado em pMGX contendo cinco genes pode ser facilmente construído e usado para produzir todas as cinco proteínas codificadas em Escherichia coli . Este sistemaEm e protocolo permite que os pesquisadores rotineiramente expressam módulos complexos de múltiplos componentes e caminhos em E. coli .
Introdução
A co-expressão de múltiplas proteínas é cada vez mais essencial, particularmente em aplicações de biologia sintética, onde vários módulos funcionais devem ser expressos 1 ; No estudo dos complexos proteína-proteína, onde a expressão e função muitas vezes requerem co-expressão 2 , 3 ; E na caracterização e aproveitamento de caminhos biossintéticos, onde cada gene na via deve ser expresso 4 , 5 , 6 , 7 , 8 . Vários sistemas foram desenvolvidos para coexpressão, particularmente no organismo hospedeiro Escherichia coli , o cavalo de trabalho para a expressão de proteína recombinante em laboratório 9 . Por exemplo, múltiplos plasmídeos com diferentes marcadores selecionáveis podem ser usados para expressar proteínas individuais usando uma riqueza de diferentes ex Vetores de pressão 10 , 11 . Os sistemas plasmídicos únicos para expressão múltipla de proteínas utilizaram múltiplos promotores para controlar a expressão de cada gene 10 , 12 ; Operões sintéticos, onde múltiplos genes são codificados em uma única transcrição 2 , 13 ; Ou, em alguns casos, um único gene que codifica um polipéptido que é processado de forma proteolítica, produzindo as proteínas desejadas de interesse 14 .

Figura 1: fluxo de trabalho pMGX que mostra a construção de um vetor policristão. O sistema pMGX fornece uma estratégia flexível e fácil de usar para a construção de operões sintéticos sob o controle de um promotor T7 indutível.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Clique aqui para ver uma versão maior deste número.
Neste manuscrito, é descrito o uso de um sistema altamente eficaz para a construção de operões sintéticos multigene sob o controle de uma ARN polimerase T7 indutível ( Figura 1 ). Este sistema permite que muitos genes sejam expressos simultaneamente a partir de um plasmídeo. É baseado em um sistema de plasmídeo, originalmente chamado pKH22, que foi utilizado com sucesso para várias aplicações diferentes 6 , 7 , 8 . Aqui, este conjunto de plasmídeo é expandido para incluir quatro vetores relacionados: pMGX-A, um vetor de expressão que não possui quaisquer marcas C ou N-terminal e com o marcador de resistência à ampicilina; PMGX-hisA, um vetor de expressão que codifica uma etiqueta de hexahistidina N-terminal e com o marcador de resistência à ampicilina; PMGX-K, um vetor de expressão lObtendo quaisquer tags C ou N-terminal e com o marcador de resistência à kanamicina; E pMGX-hisK, um vetor de expressão que codifica uma etiqueta de hexahistidina N-terminal e com o marcador de resistência à canamicina. Neste estudo, o método para a geração de um vetor policristão contendo cinco genes usando o sistema pMGX, especificamente pMGX-A, é demonstrado juntamente com a produção bem-sucedida de cada proteína individual em Escherichia coli .
Protocolo
1. Obtendo Genes de Interesse
- Desenhe genes sintéticos.
- Otimize uma sequência de genes para a expressão de E. coli .
- Remova todos os sites de restrição problemáticos da sequência (AvrII, NdeI, EcoRI e XbaI).
- Incorporar sites de restrição para clonagem; É recomendado um site 5 '-NdeI e um site 3'-EcoRI. Outros locais podem ser utilizados, se necessário; Referem-se à região de multiclonagem do plasmídeo selecionado ( Figura S1- S4 ). Se desejado, inclua uma etiqueta de codificação 5 'ou 3' para a detecção Western Blot.
- Comercialmente, ordene o gene projetado.
- Ou o clone clone o gene em um vetor contundente usando um kit de clonagem contundente, de acordo com as instruções do fabricante; Projete iniciadores para amplificar o gene (então, avance para o passo 1.2.2); Ou adicionar extremidades 5 'e 3' adicionais para a clonagem direta em um plasmídeo pMGX e passar para o Passo 2. Aqui, thOs resultados representativos são da clonagem brusca dos genes de interesse.
- Amplificar o gene desejado (a partir de um gene sintético projetado e otimizado ou de DNA modelo contendo o gene desejado) 15 .
- Desenhe primers com sites de restrição para clonagem; É recomendado um site 5 '-NdeI e um site 3'-EcoRI. Outros locais podem ser utilizados, se necessário; Referem-se à região de multiclonagem do plasmídeo selecionado ( Figura S1- S4 ). Se desejado, inclua uma etiqueta de codificação de 5 'ou 3' para a detecção de Western Blot.
- PCR amplifica o gene desejado 15 .
- Analisar o PCR por eletroforese em gel de agarose 16 .
- Se houver amplificação inespecífica, limpe o gene amplificado por extração de gel. Caso contrário, use um kit de limpeza de enzimas.
- Quantifique o DNA usando um espectrofotômetro, verificando o absorventeE a 260 nm; Em branco com tampão de eluição 17 .
2. Clonagem de Genes de Interesse em um Vector de Sistema de Expressão Multigênica, pMGX 18
- Restrição digerir o gene obtido de interesse e o vetor desejado, pMGX, com NdeI e EcoRI.
NOTA: Se uma grande quantidade de plasmídeos recircularizados forem obtidos, permita que a reação de NdeI prossiga por 1 h antes de adicionar EcoRI.- Use uma reação de digestão de 40 μL contendo 0,5-1,5 μg de DNA e 1 μL de cada enzima com 4 μL do tampão 10x apropriado.
- Para um resumo NdeI e EcoRI, use o tampão EcoRI e primeiro adicione a endonuclease NdeI. Digerir por 1 h a 37 ° C. Em seguida, adicione a endonuclease EcoRI e permita que o resumo continue por uma hora adicional. NdeI é sensível às clivagens próximas ao fim do DNA, então a digestão inicial por EcoRI pode prevenir a digestão efetiva pelo NdeI.
- ElectrOfora a digestão de restrição em gel de agarose (para um gene de 1 kb, use um gel de agarose a 0,7% a 110 V durante 55 min, para selecionar a porcentagem de gel de agarose para diferentes tamanhos de genes, consulte a referência 16 .
- Inserção de impostos e bandas de vetores usando um bisturi limpo ou uma lâmina de barbear e coloque o segmento de gel excisado em um tubo de 1,5 mL.
- Extrair o DNA do gel de agarose usando um kit de extração de gel de acordo com o protocolo do fabricante.
- Ligar o gene de interesse para o vector pMGX utilizando uma proporção de inserção para vector de 3: 1; Configurar uma ligação negativa de pMGX digerido sem a inserção.
- Configure uma reação de ligação de 20 μL contendo 1 μL de T4 DNA ligase, 0,15-0,5 μg de DNA de vetor (~ 5 μL) e uma quantidade apropriada de inserção com base em uma proporção de inserção a vetor de 3: 1 e do tamanho de O gene que está sendo inserido; Os backbones pMGX estão entre 5,312 e 5,504 pb de tamanho. Inclua uma reação de controle negativo que contenha tudoT o gene que está sendo inserido. Certifique-se de que a quantidade de DNA de vetor é equivalente na ligação de controle negativo e as reações de ligação de inserção vetorial mais.
- Reagir as reacções de ligação em células de E. coli quimicamente competentes XL1-Blue e plasmídeos ligados pMGX- yfg1 (contendo o gene do interesse 1), pMGX- yfg2 (contendo o gene do interesse 2) ... pMGX- yfgn (contendo o gene do interesse n). Use técnica asséptica (dentro de um gabinete de biossegurança ou sob uma chama).
- Destilar alíquotas de 100 μL de XL1-Blue E. coli quimicamente competente em gelo durante 5 min e depois adicionar 5 μL das reações de ligação. Incube durante 30 minutos no gelo.
- Choque de calor das células por 45 s em um banho de água a 42 ° C e depois adicione 200 μL de LB frio. Incubar em gelo durante 2 min.
- Agite as células a 37 ° C, 220 rpm durante 1 h e, em seguida, aplique a placa 100 μL sobre uma placa de LB-agar contendo uma aprovaçãoMarcador selecionável adequado (ampicilina ou kanamicina).
- Teste os clones para transformantes positivos.
- Compare as contagens de colônias nas placas de controle negativo e ligadura. É desejada uma relação de contagem de colônias maior que 1: 2. Se houver um grande número de colônias na placa de controle negativo, volte para o Passo 2.1 e reveja a nota.
- Selecione 4-8 colônias individuais (dependendo do controle negativo: relação de ligação) de cada reação de ligação dos diferentes genes inseridos para rastreio (1 n) e inocule um LB de 4 mL e uma cultura durante a noite de antibiótico apropriada / colônia individual. Cultive culturas durante a noite com agitação a 37 ° C e 220 rpm.
- Isolar o DNA do plasmídeo usando um kit de isolamento do DNA do plasmídeo de acordo com o protocolo do fabricante.
- Configure uma reação de 20 μL NdeI + EcoRI digest contendo 150-500 ng de DNA e 1 μL de cada enzima com 2 μL do tampão 10x apropriado. NaoÉ caso, adicione NdeI e EcoRI ao mesmo tempo em que o gene de interesse estará entre os sites de restrição. É recomendado um controle negativo com o vetor pMGX vazio. Digerir durante 2 h a 37 ° C em banho-maria.
3. Inserindo o gene 2 no vetor pMGX contendo o gene 1, pMGX- yfg1
- Restrição digerir pMGX- yfg1 com AvrII e tratar com fosfatase intestinal de bezerro (CIP).
- Use uma reação de digestão de 40 μL contendo 0,5-1,5 μg de DNA vetorial (~ 5-10 μL de DNA isolado) e 1 μL de AvrII com 4 μL do tampão 10x apropriado. Digerir por 1,5 h a 37 ° C e depois adicionar 1,5 μL de CIP. Deixe-o a 37 ° C durante 30 minutos adicionais.
- Restriction digest pMGX- yfg2 com AvrII e XbaI para libertar o gene de interesse.
- Use uma reação de digestão de 40 μL contendo 0,5-1,5 μg de DNA e 1 μ; L de cada enzima com 4 μL do tampão 10x apropriado. Digerir por 2 h a 37 ° C.
- A restrição de eletroforese digere em um percentual apropriado (0,7%) de gel de agarose e adiciona as inserções e bandas de vetores usando um bisturi limpo / lâmina de barbear (consulte as Etapas 2.2-2.3).
- Extraia o DNA usando um kit de extração de gel de acordo com o protocolo do fabricante e quantifica o DNA 17 .
- Ligate o gene 2 em pMGX- yfg1 usando uma proporção de inserção / vetor 3: 1. Configure uma ligação negativa de pMGX- yfg1 digerido sem a inserção adicional. Configure como acima no Passo 2.5.
- Transforme 5 μL das reações de ligação em células XL1-Blue quimicamente competentes de E. coli , plasmídeos ligados pMGX- yfg1,2 (contendo o gene de interesse 1 e 2) e controle negativo de pMGX- yfg1 , como visto no Passo 2.6.
- Compare a contagem de colônias nas placas de controle e ligadura negativas. Uma razão de contagem de colônias gÉ desejável reater do que 1: 2. Se houver um grande número de colônias na placa de controle negativo, volte para o Passo 3.1 e reveja o tratamento com CIP.
- Selecione 4-8 colônias individuais (dependendo do controle negativo: relação de ligação) da reação de ligação e inocule 4 mL de LB mais o antibiótico apropriado por colônia individual e cresça durante a noite a 37 ° C e 220 rpm.
- Isolar DNA de plasmídeo usando um kit de isolamento de DNA plasmídico de acordo com o protocolo do fabricante e quantificar o DNA 17 .
- Tira a inserção efetiva do segundo gene, realizando uma digestão de restrição de pMGX- yfg1,2 com EcoRI.
- Use uma reação de 20 μL de digestão contendo 150-500 ng de DNA e 1 μL de enzima EcoRI com 2 μL de tampão EcoRI 10x. Digerir por 2 h a 37 ° C.
- Eletroforese a síntese de restrição em uma porcentagem adequada de gel de agarose; Procure uma banda que correspondaS ao tamanho do gene 2 (ver passo 2.2). Um gene pode inserir no vetor na orientação indesejada e reversa.
4. Adicionando um terceiro gene ao vetor pMGX contendo os genes 1 e 2, pMGX- yfg1,2
- Restrição digerir pMGX- yfg1,2 com AvrII e tratar com CIP, como visto no Passo 3.1.
- Restriction digest pMGX- yfg3 com AvrII e XbaI, conforme observado na Etapa 3.2.
- Eletroforese as digestões de restrição em uma porcentagem apropriada de gel de agarose e inserção de impostos especiais de consumo e bandas de vetores usando uma bisturí limpa / lâmina de barbear; Consulte os Passos 2.2-2.3.
- Extraia o DNA do gel de agarose usando um kit de extração de gel e quantifica o DNA 17 .
- O gene de ligato 3 em pMGX- yfg1,2 utilizando uma relação de inserção a vector de 3: 1 e estabeleceu uma ligação livre de pMGX- yfg1,2 digerido sem inserção adicional, como se observa no Passo 3.5.
- Transforme 5 μL das reações de ligação em XLCélulas de E. coli quimicamente competentes de 1-azul, plasmídeo ligado pMGX- yfg1,2,3 (contendo genes de interesse 1, 2 e 3) e controle negativo de pMGX- yfg1,2 , como visto na Etapa 2.6.
- Compare a contagem de colônias nas placas de controle e ligadura negativas. Se houver um grande número de colônias na placa de controle negativo, volte ao Passo 4.1 e reveja o tratamento com CIP.
- Selecione 4-8 colônias individuais (dependendo do controle negativo: relação de ligação) da reação de ligação e inocule 4 mL de LB mais o antibiótico apropriado por colônia individual; Crescer durante a noite a 37 ° C e 220 rpm.
- Isolar o DNA plasmídico usando um kit de isolamento de DNA plasmídico de acordo com o protocolo do fabricante.
- Tira a inserção efetiva do terceiro gene executando uma síntese de restrição de pMGX- yfg1,2,3 com EcoRI, conforme observado na Etapa 3.10.
- Eletroforese a síntese de restrição em uma porcentagem adequada de gel de agarose; VejaPara bandas que correspondem aos tamanhos do gene 2 e do gene 3 (ver passo 2.2). Nota: o gene 3 pode inserir no vetor na orientação não desejada, invertida. Se os genes 2 e 3 forem do mesmo tamanho, outro local de restrição de restrição apropriado deve ser selecionado para triagem.
NOTA: Repita conforme necessário para cada novo gene.
5. Produção de proteínas de interesse usando um sistema de expressão multigênica e avaliação da produção por Western Blot
- Transforme o clone positivo contendo todos os genes de interesse em quimicamente competente, produção de proteína E. coli , como BL21- (λDE3).
- Descongelar alíquotas de 100 μL de E. coli BL21- (λDE3) químicamente competente em gelo durante 5 min e depois adicionar 1 μL do DNA plasmídico clonado positivo; Incubar durante 30 min no gelo.
- Choque térmico das células durante 45 s em banho-maria mantido a 42 ° C e depois adicione 200 μL de LB frio. Incubar no gelo durante 2 min.
- Agite as células a 37 ° C e 220 rpm durante 1 h e depois aplique a placa 100 μL sobre uma placa de LB-agar contendo o marcador selecionável apropriado (ampicilina ou kanamicina).
- Expresse a proteína pela indução de isopropil-p-D-1-tiogalactopiranósido (IPTG).
- Selecione uma colônia isolada da placa de transformação B21- (λDE3) e inocule 4 mL de LB mais o antibiótico apropriado; Cresça durante a noite, agitando a 37 ° C e 220 rpm.
- Inocular 100 mL de LB mais a cultura de antibiótico apropriada utilizando 1 mL de cultura durante a noite.
- Crescer a 37 ° C com agitação a 220 rpm para uma DO 600 de 0,6.
- Induzir a cultura com 100 μM de IPTG e crescer durante 15 h a 25 ° C e 220 rpm.
- Remova 1 mL da cultura e centrifugá-la a 13.000 xg durante 1 min; Descarte o sobrenadante.
- Lyse as células usando solução de lise de acordo com as instruções do fabricanteE preforme uma mancha de Western do lisado celular solúvel para determinar se todas as proteínas foram produzidas com sucesso 19 .
Resultados
Neste estudo, o objetivo era co-expressar cinco proteínas de um único plasmídeo. Os fragmentos de gene sintético otimizados com cinco codões que codificam as marcas de hexahistidina N- ou C-terminal foram adquiridos comercialmente. Os genes sintéticos foram amplificados por PCR e clonados individualmente em um vetor PCR-blunt e sequenciados. Para gerar o plasmídeo policórónico, os cinco genes de interesse foram primeiro clonados num plasmídeo pMGX adequado, pMGX-A. A Figura 2 mostra PCRBlunt- yfg1 e PCRBlunt- yfg2 além de pMGX-A digerido com NdeI + EcoRI (ver Etapa 2). Para clonar os dois primeiros genes em pMGX-A, o plasmídeo foi digerido com NdeI + EcoRI ( Figura 2 ). Após a clonagem dos genes de interesse em pMGX-A, os plasmídeos recém construídos, designados como pMGX- yfg1 e pMGX- yfg2 , foram digeridos com AvrII + XbaI para confirmar que foram obtidos clones bem-sucedidos, comoMostrado nas bandas de 1,1 kb e 1,3 kb obtidas na Figura 3 .
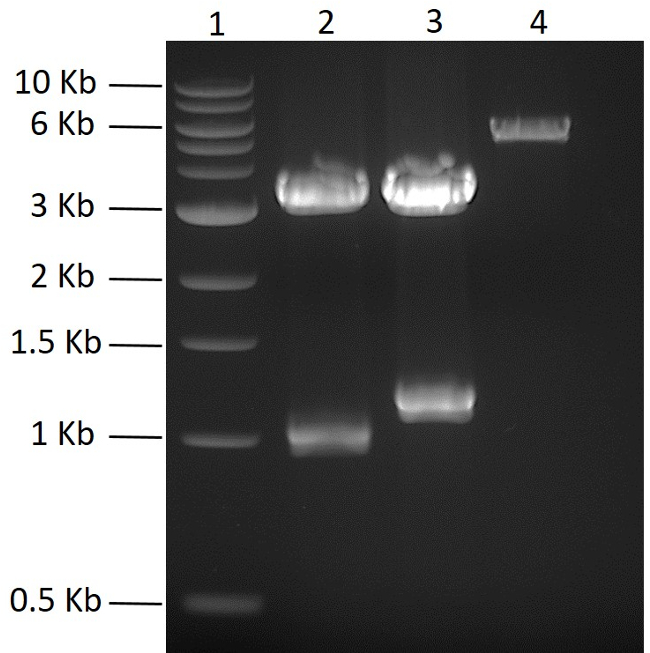
Figura 2: Clonagem de plasmídeos pMGX. Os resultados esperados de PCR plasmídeos contundentes contendo yfg1 e yfg2 após digestão com NdeI + EcoRI são mostrados em um gel de agarose 0,7% (110 V, 55 min). Pista 1, escala de DNA de 1 kb; Pista 2, PCRBlunt- yfg1 ; Pista 3, PCRBlunt- yfg2 ; Lane 4, controle pMGX-A. Clique aqui para ver uma versão maior dessa figura.
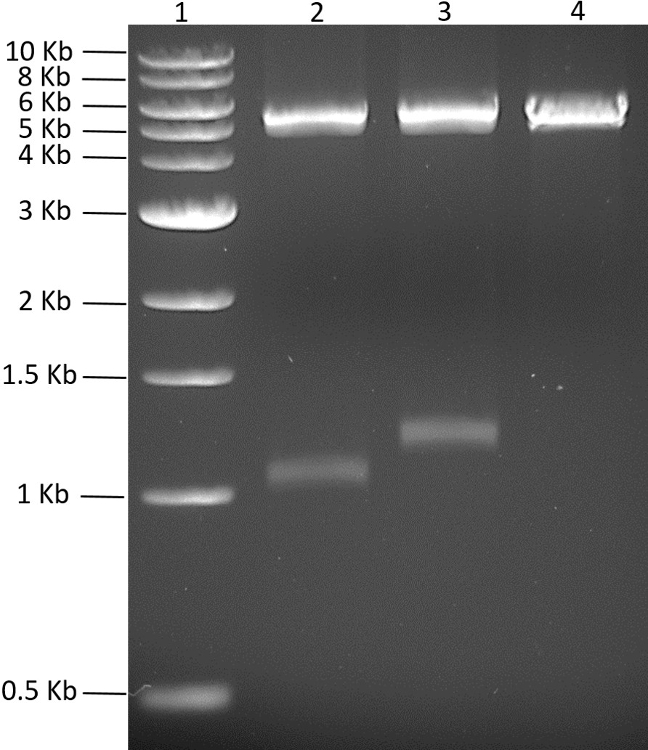
Figura 3: rastreio de pMGX- yfg1 e pMGX- yfg2. O gel de agarose a 0,7% (110 V, 55 min) mostra As duas bandas que são geradas por pMGX- yfg1 e pMGX- yfg2 após a digestão com AvrII + XbaI. Lane 1, 1-kb DNA ladder; Pista 2, pMGX- yfg1 ; Pista 3, pMGX- yfg2 ; Lane 4, controle pMGX-A. Clique aqui para ver uma versão maior dessa figura.
Com ambos os genes no vector pMGX apropriado, construiu-se o plasmídeo policistrônico. Para gerar o plasmídeo pMGX- yfg1,2 , pMGX- yfg1 foi digerido com AvrII, como mostrado na Figura 4 . PMGX- yfg2 foi digerido com AvrII + XbaI e o gene de 1,3 kb que contém o fragmento foi ligado ao sítio AvrII de pMGX- yfg1 linearizado, gerando pMGX- yfg1,2 . A digestão de pMGX- yfg1,2 com EcoRI confirmou a clonagem bem sucedida do plasmídeo desejado e é mostrada em> Figura 5.

Figura 4: Geração do plasmídeo bicistrônico pMGX-yfg1,2. Os resultados de eletroforese em gel de agarose a 0,7% (110 V, 55 min) de pMGX- yfg1 digeridos com AvrII e pMGX- yfg2 digeridos com AvrII + XbaI. Lane 1, 1-kb DNA ladder; Pista 2, pMGX- yfg1 digerido com AvrII; Pista 3, pMGX- yfg2 digerido com AvrII + XbaI; Lane 4, controle pMGX-A. Clique aqui para ver uma versão maior dessa figura.
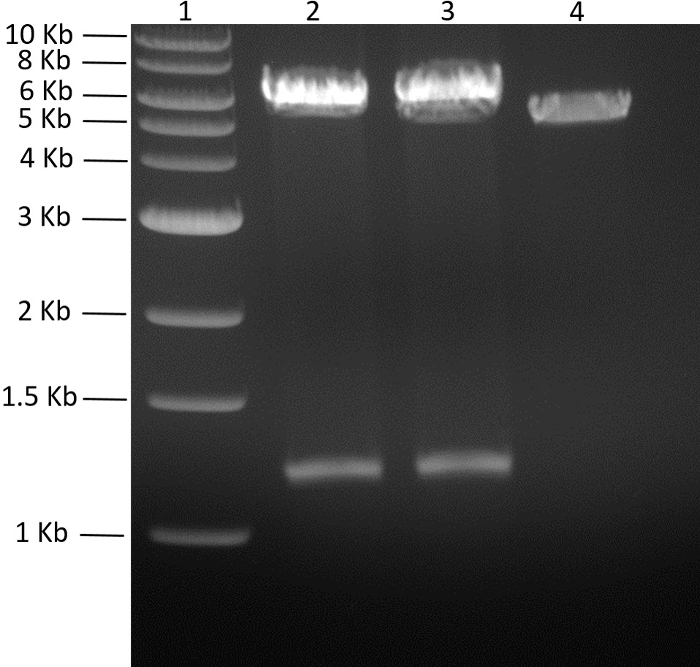
Figura 5: rastreio de clones pMGX- yfg1,2 . O 0,7% de gel de agarose resultante (110 V, 55 min) de pMGX- yfSão mostrados clones de g1,2 digeridos com EcoRI. Os clones bem sucedidos gerarão duas bandas, uma para o gene efg2 inserido e outra correspondente ao backbone + yfg1 ( pMGX-yfg1 ). Pista 1, escala de DNA de 1 kb; Pista 2, pMGX- yfg1,2 digerido com EcoRI, clone 1; Pista 3, pMGX- yfg1,2 digerido com EcoRI, clone 2; Lane 4, controle pMGX-A. Clique aqui para ver uma versão maior dessa figura.
Ao gerar um plasmídeo policistrônico contendo dois ou mais genes, pode-se encontrar uma inserção reversa indesejável do gene clonado no site AvrII. A Figura 6 é um gel que destaca a diferença entre os resultados de um plasmídeo digerido com um gene inserido na orientação correta em oposição a um gene inserido na orientação invertida. Se o gene forLigada e clonada na orientação indesejada, o sítio EcoRI no final do gene será inserido ao lado do site EcoRI do gene anterior. Isso produziria um tamanho de fragmento que é praticamente indetectável por eletroforese em gel de DNA (menos de 50 pb). Além disso, o último gene inserido apareceria na espinha dorsal, o que é facilmente identificável devido ao tamanho da espinha dorsal ser maior do que o esperado. A diferença no tamanho da espinha dorsal do tamanho esperado indicará o tamanho do último gene inserido.

Figura 6: rastreio de clones pMGX- yfg1-5 . É mostrado um gel de agarose a 1,3% (110 V, 65 min) que representa os resultados de pMGX- yfg1-5 digerido com EcoRI. Os clones bem-sucedidos (pista 2) gerarão uma faixa correspondente ao último gene inserido ( yfg neste caso, 1,1 kb). LanE 1, escada de DNA de 1 kb; Pista 2, clone positivo de pMGX- yfg1-5, digerido com EcoRI; Pista 3, clone negativo de pMGX- yfg1-5, digerido com EcoRI; Lane 4, controle pMGX- yfg1-4 . Clique aqui para ver uma versão maior dessa figura.
Para completar o vector de expressão policristonal de cinco genes, os três genes restantes foram clonados um após o outro em pMGX- yfg1,2 para gerar pMGX- yfg1-5 (ver Etapa 5). Este plasmídeo foi então transformado em BL21- (λDE3), e a expressão das proteínas foi induzida numa cultura de fase de log intermediário pela adição de IPTG. A Figura 7 é uma transferência Western de lisados celulares, confirmando que todas as cinco proteínas de um único plasmídeo contendo vários genes num único operão podem ser expressas. A expressão de pMGX- yfg1 (contendo 1 gene),PMGX- yfg1,2 (contendo 2 genes), e pMGX- yfg1-5 (contendo 5 genes) são mostrados.
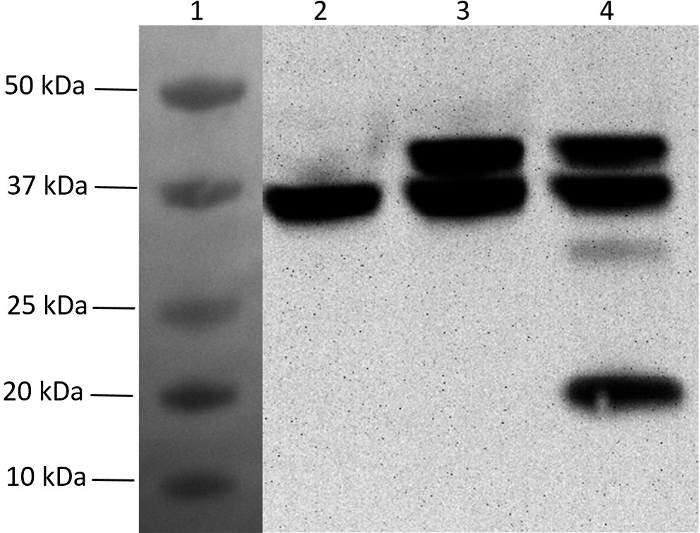
Figura 7: Análise de transferência de Western da expressão multi-gene usando o sistema pMGX. A incorporação de marcas de hexahistidina N- ou C-terminal permitiu a detecção de Western blot da expressão da proteína. As amostras foram separadas em géis de acrilamida sem deformação a 4-20% pré-moldados (200 V, 35 min, 10 cm x 8,5 cm), e depois uma transferência (40 V, 90 min) sobre uma membrana de nitrocelulose (0,45 μm, 10 cm x 7 cm). O anticorpo monoclonal anti-His conjugado com HRP, que não requer um anticorpo secundário, foi utilizado. Os tampões de bloqueio, transferência e diluição de anticorpos foram preparados conforme recomendado pelo fabricante do anticorpo. A detecção foi realizada com o substrato Western Chemiluminescent HRP seguindo o ins da fabricanteTrucções. A membrana resultante foi visualizada usando uma imagem sem filtro. Pista 1, padrão de duas cores transferido para a membrana (não quimioluminiscente); Pista 2, His6pMGX- yfg1 ; Pista 3, His6pMGX- yfg1,2 ; Lane 4, His6pMGX- yfg1-5. Nota: A proteína produzida por yfg5 é do mesmo tamanho que a proteína produzida por yfg1 (ambos são 36 kDa) e não podem ser separados no gel. Clique aqui para ver uma versão maior dessa figura.
Figuras suplementares: clique aqui para baixar este arquivo.
Discussão
A co-expressão de múltiplos genes é cada vez mais essencial, particularmente na caracterização e reconstituição das vias metabólicas complexas multigênicas 3 , 4 , 5 . O sistema pMGX faz co-expressão multigene na rotina 6 , 7 , 8 de E. coli e acessível a diversos pesquisadores. Neste estudo, mostraram-se cinco proteínas de interesse que foram produzidas simultaneamente a partir de um único sistema plasmídico usando pMGX-A. Muitos sistemas de coexpressão atualmente disponíveis apenas permitem a inserção de dois genes, gerando plasmídeos bicistrônicos, como os vetores 12 do pET Duet. Se alguém requer a adição de mais genes, são necessários múltiplos vetores bicistrônicos com diferentes marcadores selecionáveis. Em contraste com outros sistemas de expressão multigene, o sistema pMGX permite a clonagem fácilDe múltiplos genes em um plasmídeo, com a capacidade de reutilizar os locais de restrição sem a necessidade de diferentes plasmídeos doadores ou para a remoção de múltiplos locais de enzimas de restrição dos genes 13 , 14 . Os passos críticos neste protocolo incluem o desenho do gene, a prevenção da recircularização de fundo de vetores pMGX digeridos individualmente e as telas dos vetores policistrônicos para a orientação correta do gene inserido.
Ao planejar a estratégia de clonagem para construir um sistema policistrônico, é essencial garantir que os genes de interesse não contenham sites de restrição necessários para a clonagem a jusante. Em particular, estes incluem XbaI e AvrII, que são necessários para a inserção do segundo e subsequente genes no vector pMGX contendo o primeiro gene de interesse (Passos 3 e 4). Além disso, é essencial eliminar locais de restrição que serão utilizados para a clonagem inicial do gInteresse em pMGX (Passo 2). Tipicamente, estes incluem NdeI ou NheI na extremidade 5 'do gene e BamHI ou EcoRI na extremidade 3' do gene, embora outros locais de restrição também possam ser utilizados para esta etapa, se necessário. A eliminação de locais de restrição extra e indesejáveis pode ser realizada facilmente no estágio de síntese de genes ou, no caso de genes clonados de outras fontes, por substituições sinônimas de nucleotídeos via mutagênese dirigida ao local 20 .
A adição de subsequentes genes de interesse em pMGX contendo o primeiro gene de interesse requer a linearização de pMGX- yfg1 com AvrII (Passo 3.1, note que também pode ser linearizado com XbaI). Este vetor linearizado se religará rapidamente durante a inserção do segundo gene de interesse (Passo 3.5), levando a um número extremamente elevado de clones de fundo. Para evitar isso, é essencial tratar o vetor linearizado com uma fosfatase para desfosforilar o 5 °2; Extremidades do vetor linearizado. Normalmente, o CIP é usado, embora outros estejam disponíveis. A otimização de ambas as unidades de fosfatase e o tempo de incubação podem ser necessárias para otimizar as ligaduras dos subseqüentes genes de interesse no vetor linearizado.
Por último, ao gerar um sistema policistrônico, o gene de interesse é inicialmente excisado pela digestão com XbaI e AvrII, gerando extremidades coesivas complementares nas extremidades 5 'e 3' do gene (Etapas 3.2 e 4.2). Esta inserção pode ser ligada no vetor linearizado na direção direta ou reversa. Como todas as extremidades coesivas são idênticas, nenhuma direccionalidade é aplicada através do recozimento complementar das extremidades coesivas. É, portanto, necessário exibir os clones resultantes para a direção de inserção correta do gene de interesse. Isto pode ser feito facilmente através da triagem de clones com um dos locais de restrição utilizados para clonar o gene original de interesse em pMGX (Passo 2). Tipicamente,EcoRI é usado, pois o site de restrição está na extremidade 3 'do gene e, portanto, é apropriado selecionar a direção, como mostrado na Figura 6 . Na maioria dos casos, 50% dos clones contendo o gene de interesse possuem o gene na orientação correta e 50% têm o gene na orientação oposta.
Infrequentemente (~ 10% das ligaduras), todos os clones de uma ligadura particular podem ter o gene de interesse inserido em apenas uma direção. Este resultado parece ser dependente da sequência, uma vez que é altamente reprodutível nestas instâncias específicas. Se isso ocorrer, e o gene de interesse está inserindo continuamente na orientação inversa, o vetor desejado geralmente pode ser acessado alternando a direção da clonagem. Por exemplo, em vez de clonar yfg2 no site AvrII de pMGX- yfg1 , yfg1 pode ser clonado no site XbaI de pMGX- yfg2 . Observe que isso dá o vetor final idênticosistema. Esta flexibilidade adicional que permite o acesso ao mesmo sistema a partir de diferentes estratégias de clonagem é altamente vantajosa.
Uma limitação importante desta metodologia é que as proteínas codificadas na extremidade 3 'do transcrito policórónico são tipicamente expressas em níveis inferiores às proteínas codificadas na extremidade 5' da transcrição, e esse efeito é mais pronunciado quanto mais a transcrição for . Isso significa que mudar a ordem dos genes de interesse no operão sintético pode afetar os níveis de expressão relativa das proteínas que codificam, proporcionando um mecanismo para ajustar os níveis de expressão relativa.
Em resumo, o sistema pMGX fornece um método confiável para a co-expressão de múltiplas proteínas a partir de um único plasmídeo em E. coli , que pode ser usado para uma variedade de aplicações de biologia sintética e para a caracterização da via bioquímica.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá.
Materiais
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Referências
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados