Method Article
Inducibile T7 RNA Polymerase-mediato sistema di espressione multigene, pMGX
In questo articolo
Riepilogo
Questo studio descrive metodi per la coespressione mediata da T7 di geni multipli da un singolo plasmide in Escherichia coli usando il sistema plasmid pMGX.
Abstract
La coespressione di molteplici proteine è sempre più essenziale per la biologia sintetica, studiando complessi proteico-proteici e caratterizzando e sfruttando percorsi biosintetici. In questo manoscritto è descritto l'uso di un sistema altamente efficace per la costruzione di operoni sintetici multigene sotto il controllo di una polimerasi RNA T7 indottabile. Questo sistema permette che molti geni siano espressi simultaneamente da un plasmide. In questo caso, un insieme di quattro vettori correlati, pMGX-A, pMGX-hisA, pMGX-K e pMGX-hisK, con il marker selettivo di resistenza a ampicillina o kanamycina (A e K) e posseggono o mancano di una esahistidina N-terminale (Il suo) sono divulgati. Sono forniti protocolli dettagliati per la costruzione di operoni sintetici che utilizzano questo sistema vettoriale, insieme ai dati corrispondenti, che dimostrano che un sistema basato su pMGX contenente cinque geni può essere facilmente costruito e utilizzato per produrre tutte e cinque le proteine codificate in Escherichia coli . Questo sistemaEm e il protocollo consentono ai ricercatori di esprimere in modo frequente moduli multipli complessi e percorsi in E. coli .
Introduzione
La coespressione di molteplici proteine è sempre più essenziale, in particolare nelle applicazioni di biologia sintetica, in cui devono essere espressi più moduli funzionali 1 ; Nello studio dei complessi proteico-proteici, in cui espressione e funzione richiedono spesso co-espressione 2 , 3 ; E nel caratterizzare e sfruttare percorsi biosintetici, in cui ogni gene nel percorso deve essere espresso 4 , 5 , 6 , 7 , 8 . Sono stati sviluppati diversi sistemi per la coespressione, in particolare nell'organismo ospitante Escherichia coli , il cavallo da lavoro per l'espressione di proteine ricombinanti di laboratorio 9 . Ad esempio, plasmidi multipli con differenti marcatori selectable possono essere utilizzati per esprimere singole proteine usando una ricchezza di diversi es Vettori di pressione 10 , 11 . I singoli sistemi plasmidi per l'espressione multipla di proteine hanno utilizzato sia promotori multipli per controllare l'espressione di ciascun gene 10 , 12 ; Operoni sintetici, in cui i geni multipli sono codificati su una singola trascrizione 2 , 13 ; O, in alcuni casi, un singolo gene che codifica un polipeptide che viene ultimamente elaborato in modo proteolitico, fornendo le proteine desiderate di interesse 14 .

Figura 1: flusso di lavoro pMGX che mostra la costruzione di un vettore policistronico. Il sistema pMGX fornisce una strategia flessibile e facile da usare per la costruzione di operoni sintetici sotto il controllo di un promotore T7 induttivo.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Fare clic qui per visualizzare una versione più grande di questa figura.
In questo manoscritto è descritto l'uso di un sistema altamente efficace per la costruzione di operoni sintetici multigene sotto il controllo di una polimerasi RNA T7 indottabile ( figura 1 ). Questo sistema permette che molti geni siano espressi simultaneamente da un plasmide. È basato su un sistema plasmidico, originariamente chiamato pKH22, che è stato utilizzato con successo per una serie di applicazioni diverse 6 , 7 , 8 . Qui, questo set di plasmidi viene ampliato per includere quattro vettori correlati: pMGX-A, un vettore di espressione privo di qualsiasi etichetta C- o N-terminale e con il marker di resistenza dell'ampicillina; PMGX-hisA, un vettore di espressione che codifica un tag di esahistidina N-terminale e con il marker di resistenza dell'ampicillina; PMGX-K, un vettore di espressione lAcking qualsiasi etichetta C- o N-terminale e con il marker di resistenza alla kanamycina; E pMGX-hisK, un vettore di espressione che codifica un tag di hexahistidine N-terminale e con il marker di resistenza alla kanamycina. In questo studio è stato dimostrato il metodo per la generazione di un vettore polistronicico contenente cinque geni usando il sistema pMGX, in particolare pMGX-A, insieme alla produzione di ogni singola proteina in Escherichia coli .
Protocollo
1. Ottenere Geni di Interesse
- Generi sintetici di progettazione.
- Ottimizzare una sequenza genica per l'espressione di E. coli .
- Rimuovere tutti i siti di restrizione problematica dalla sequenza (AvrII, NdeI, EcoRI e XbaI).
- Incorporare siti di restrizione per la clonazione; Si raccomanda un sito 5'-NdeI e un sito 3'-EcoRI. Altri siti possono essere usati, se necessario; Si riferisce alla regione multicloning del plasmide selezionato ( Figura S1- S4 ). Se si desidera, includere un tag di codifica 5 'o 3' per la rilevazione Western blot.
- Ordina commercialmente il gene progettato.
- O il clone blunt il gene in un vortice vettore utilizzando un kit di clonazione blunt, come per le istruzioni del produttore; Primer di progettazione per amplificare il gene (quindi procedere al punto 1.2.2); O aggiungere altri 5 'e 3' estremità per la clonazione diretta in un plasmide pMGX e procedere al punto 2. Qui,I risultati rappresentativi sono dalla clonazione sbilanciata dei geni di interesse.
- Amplificare il gene desiderato (da un gene sintetico progettato e ottimizzato o da un template DNA contenente il gene desiderato) 15 .
- Primer di progettazione con siti di restrizione per la clonazione; Si raccomanda un sito 5'-NdeI e un sito 3'-EcoRI. Altri siti possono essere usati, se necessario; Si riferisce alla regione multicloning del plasmide selezionato ( Figura S1- S4 ). Se si desidera, includere un tag di codifica 5 'o 3' per il rilevamento Western blot.
- PCR amplifica il gene desiderato 15 .
- Analizzare la PCR mediante elettroforesi 16 agarosica.
- Se esiste un'amplificazione non specificata, ripulire il gene amplificato mediante l'estrazione del gel. In caso contrario, utilizzare un kit di pulizia enzimatico.
- Quantificare il DNA utilizzando uno spettrofotometro controllando l'assorbimentoE a 260 nm; Vuoto con buffer di eluizione 17 .
2. Clonazione dei geni di interesse in un vettore del sistema di espressione multigene, pMGX 18
- La restrizione digerisce il gene di interesse ottenuto e il vettore desiderato, pMGX, con NdeI e EcoRI.
NOTA: Se si ottiene una grande quantità di plasmidi ricircolati, lasciare che la reazione NdeI proceda per 1 ora prima di aggiungere EcoRI.- Utilizzare una reazione digestata da 40 μL contenente 0,5-1,5 μg di DNA e 1 μl di ciascun enzima con 4 μl del appropriato tampone 10x.
- Per un digerimento di NdeI e EcoRI, utilizzare il tampone EcoRI e aggiungere in primo luogo l'endonucleasi di NdeI. Digestare per 1 h a 37 ° C. Quindi aggiungere l'endonucleasi EcoRI e consentire al digest di procedere per un'ora aggiuntiva. NdeI è sensibile alle fenditure vicine alla fine del DNA, per cui la digestione iniziale da parte di EcoRI può impedire una digestione efficace da NdeI.
- elettrOforesi la digestione di restrizione sul gel agarosico (per un gene da 1 kb, utilizzare un agarosio di 0,7% a 110 V per 55 minuti; per selezionare il percentuale di gel agarosio per diverse dimensioni del gene, fare riferimento al riferimento 16 .
- Inserto per accise e bande vettoriali usando uno scalpello pulito o una lama di rasoio e posizionare il segmento gel gelato in un tubo da 1,5 ml.
- Estrarre il DNA dal gel agarosico usando un kit di estrazione del gel secondo il protocollo del produttore.
- Ligare il gene di interesse nel vettore pMGX utilizzando un rapporto 3: 1 inserto-vettore; Impostare una legatura negativa di pMGX digerito senza l'inserto.
- Impostare una reazione di ligando di 20 μL contenente 1 μL di ligasi del DNA di T4, 0,15-0,5 μg di DNA vettoriale (~ 5 μL) e una quantità appropriata di inserto basata su un rapporto tra inserto-vettore 3: 1 e dimensione di Il gene che viene inserito; Le spine pMGX sono tra i 5.312 ei 5.504 bp di dimensioni. Includere una reazione di controllo negativo che contiene tutto buIl gene che viene inserito. Assicurarsi che la quantità di DNA vettoriale sia equivalente nella legatura del controllo negativo e nel vettore più le reazioni di legatura dell'inserto.
- Trasformare le reazioni di legame in cellule E. coli chimicamente competenti XL1-Blue e plasmidi ligati pMGX- yfg1 (contenenti gene di interesse 1), pMGX- yfg2 (contenente gene di interesse 2) ... pMGX- yfgn (contenente gene di interesse n). Utilizzare la tecnica asettica (all'interno di un armadio di biosicurezza o sotto una fiamma).
- Mescolare 100 microlitri aliquote di E. coli XL1-Blue E. coli chimicamente competenti per ghiaccio per 5 min e quindi aggiungere 5 μL delle reazioni di legatura. Incubare per 30 minuti in ghiaccio.
- Scuotere le cellule per 45 s in un bagno d'acqua tenuto a 42 ° C e quindi aggiungere 200 μL di LB freddo. Incubarli in ghiaccio per 2 min.
- Agitare le cellule a 37 ° C a 220 giri / min per 1 h e quindi distribuire la piastra 100 μL su una piastra LB-agar contenente un approfondimentoMarcatore selettivo selezionabile (sia ampicillina che kanamicina).
- Schermate i cloni per i trasformanti positivi.
- Confronti i conti delle colonie sulle piastre di controllo e legatura negative. È desiderato un rapporto di colonia maggiore di 1: 2. Se sul piatto di controllo negativo vi è un gran numero di colonie, tornare al punto 2.1 e rivedere la nota.
- Selezionare 4-8 singole colonie (a seconda del controllo negativo: rapporto di legatura) da ciascuna reazione di legatura dei diversi geni inseriti per la screening (1 n) e inoculare 4 ml di LB e la coltura di overnight / colonia individuale adeguata antibiotica. Coltivare le culture durante la notte con agitazione a 37 ° C e 220 giri / min.
- Isolare il plasmide DNA usando un kit di isolamento del DNA plasmidico secondo il protocollo del produttore.
- Impostare una reazione di 20 μl di digestione NdeI + EcoRI contenente 150-500 ng di DNA e 1 μl di ciascun enzima con 2 μl del appropriato tampone 10x. In thÈ il caso, aggiungere NdeI e EcoRI allo stesso tempo in cui il gene di interesse sarà tra i siti di restrizione. Si consiglia un controllo negativo con il vettore vuoto pMGX. Digestare per 2 ore a 37 ° C in un bagno d'acqua.
3. Inserimento del gene 2 nel vettore pMGX contenente il gene 1, pMGX- yfg1
- La restrizione digerisce pMGX- yfg1 con AvrII e tratta con fosfatasi intestinale del vitello (CIP).
- Utilizzare una reazione di digest di 40 μl contenente 0,5-1,5 μg di DNA vettoriale (~ 5-10 μL di DNA isolato) e 1 μL di AvrII con 4 μl del opportuno tampone da 10x. Digest per 1,5 h a 37 ° C e quindi aggiungere 1,5 μL di CIP. Lasciare a 37 ° C per altri 30 minuti.
- La restrizione digerisce pMGX- yfg2 con AvrII e XbaI per liberare il gene di interesse.
- Utilizzare una reazione digestata da 40 μL contenente 0,5-1,5 μg di DNA e 1 μ; L di ciascun enzima con 4 μl del buffer appropriato10x. Digestare per 2 ore a 37 ° C.
- Le restrizioni elettroforesi digeriscono su un opportuno gel percentuale (0,7%) di agarosio e esentano le bande di inserti e vettori usando una lama bruciata / rasoio pulita (vedere Punti 2.2-2.3).
- Estrarre il DNA utilizzando un kit di estrazione del gel secondo il protocollo del produttore e quantificare il DNA 17 .
- Ligate il gene 2 in pMGX- yfg1 utilizzando un rapporto 3: 1 inserto-vettore. Impostare una legatura negativa di pMGX- yfg1 digerito senza l'ulteriore inserto. Impostare come sopra nella fase 2.5.
- Trasformare 5 μL delle reazioni di legame in cellule E. coli chimicamente competenti XL1-Blue, plasmidi ligati pMGX- yfg1,2 (contenenti gene di interesse 1 e 2) e controllo negativo pMGX- yfg1 , come si vede nel passaggio 2.6.
- Paragonare il conteggio coloniale sulle piastre di controllo e di legatura negative. Un rapporto di colonia count gSi desidera un reater di 1: 2. Se ci sono un numero elevato di colonie sulla piastra di controllo negativa, tornare al punto 3.1 e rivedere il trattamento CIP.
- Selezionare 4-8 singole colonie (a seconda del controllo negativo: rapporto di legamento) dalla reazione di legatura e inoculare 4 mL di LB e l'antibiotico appropriato per singola colonia e crescere per una notte a 37 ° C e 220 rpm.
- Isolare il plasmide DNA usando un kit di isolamento del DNA plasmidico secondo il protocollo del produttore e quantificare il DNA 17 .
- Schermate l'efficace inserzione del secondo gene eseguendo una digestione di restrizione di pMGX- yfg1,2 con EcoRI.
- Utilizzare una reazione digestata da 20 μL contenente 150-500 ng di DNA e 1 μl di enzima EcoRI con 2 μl di EcoRI 10x buffer. Digestare per 2 ore a 37 ° C.
- Elettroforesi digerire la restrizione su un opportuno gel percentuale di agarosio; Cercare una banda che corrispondaS alla dimensione del gene 2 (vedere il punto 2.2). Un gene può inserire nel vettore nell'orientamento indesiderato e indesiderato.
4. Aggiunta di un terzo gene nel vettore pMGX contenente i geni 1 e 2, pMGX- yfg1,2
- La restrizione digerisce pMGX- yfg1,2 con AvrII e trattare con CIP, come si vede al punto 3.1.
- La restrizione digerisce pMGX- yfg3 con AvrII e XbaI, come si vede al punto 3.2.
- Gli elettroforesi digestano la restrizione su un opportuno gel percentuale di agarosio e nell'inserto di accisa e nelle bande vettoriali utilizzando una lama bruciata / rasoio pulita; Fare riferimento a Fasi 2.2-2.3.
- Estrarre il DNA dal gel agarosico usando un kit di estrazione del gel e quantificare il DNA 17 .
- Lignare il gene 3 in pMGX- yfg1,2 utilizzando un rapporto 3: 1 di inserto-vettore e impostare una legatura negativa di pMGX- yfg1,2 digerito senza un ulteriore inserto, come si vede al punto 3.5.
- Trasformare 5 μL delle reazioni di legame in XL1 cellule E. coli chimicamente competenti, plasmide ligati pMGX- yfg1,2,3 (contenenti geni di interesse 1, 2 e 3) e controllo negativo pMGX- yfg1,2 , come si vede al punto 2.6.
- Paragonare il conteggio coloniale sulle piastre di controllo e di legatura negative. Se sul piatto di controllo negativo vi è un gran numero di colonie, tornare al punto 4.1 e rivedere il trattamento CIP.
- Selezionare 4-8 singole colonie (a seconda del controllo negativo: rapporto di legatura) dalla reazione di ligation e inoculare 4 mL di LB e l'appropriato antibiotico per singola colonia; Crescono la notte a 37 ° C e 220 giri / min.
- Isolare il DNA del plasmide usando un kit di isolamento del DNA plasmidico secondo il protocollo del produttore.
- Schermate l'inserimento effettivo del terzo gene eseguendo una digestione di restrizione di pMGX- yfg1,2,3 con EcoRI, come si vede nel passaggio 3.10.
- Elettroforesi digerire la restrizione su un opportuno gel percentuale di agarosio; GuardaPer le bande che corrispondono alle dimensioni del gene 2 e del gene 3 (vedi punto 2.2). Nota: il gene 3 può inserire nel vettore nell'orientamento indesiderato e indesiderato. Se i geni 2 e 3 sono le stesse dimensioni, è necessario selezionare un altro sito di digestione appropriato per la screening.
NOTA: ripetere come necessario per ogni nuovo gene.
5. Produzione di proteine di interesse utilizzando un sistema di espressione multigene e valutazione della produzione da Western Blotting
- Trasformare il clone positivo contenente tutti i geni di interesse in E. coli , chimicamente competenti, produttrici di proteine, come BL21- (λDE3).
- Mescolare 100 μL aliquote di E. coli chimicamente competenti BL21- (λDE3) in ghiaccio per 5 min e quindi aggiungere 1 μL del DNA plasmidico positivo clonato; Incubare per 30 minuti in ghiaccio.
- Scongelamento caldo le cellule per 45 s in un bagno d'acqua tenuto a 42 ° C e poi aggiungere 200 μL di LB freddo. Incubare in ghiaccio per 2 min.
- Agitare le cellule a 37 ° C e 220 giri / min per 1 h e quindi distribuire la piastra 100 μL su una piastra LB-agar contenente il marcatore selettivo adatto (ampicillina o kanamicina).
- Esprimere la proteina mediante l'induzione di isopropil-β-D-1-tiogalattopiranoside (IPTG).
- Selezionare una colonia isolata dalla piastra di trasformazione B21- (λDE3) e inoculare 4 ml di LB e l'antibiotico appropriato; Crescono durante la notte, scuotendo a 37 ° C e 220 giri / min.
- Inoculare 100 ml di LB e la coltura antibiotica appropriata usando 1 ml di coltura overnight.
- Crescere a 37 ° C con agitazione a 220 giri / min ad un OD 600 di 0,6.
- Indurre la cultura con 100 μM IPTG e crescere per 15 h a 25 ° C e 220 giri / min.
- Rimuovere 1 ml della coltura e centrifugare a 13.000 xg per 1 min; Scartare il surnatante.
- Lyse le cellule utilizzando la soluzione di lisi secondo le istruzioni del produttoreE preformare una macchia Western della lisi cellulare solubile per determinare se tutte le proteine sono state prodotte con successo 19 .
Risultati
In questo studio, l'obiettivo era quello di co-esprimere cinque proteine da un unico plasmide. I frammenti di gene sintetici ottimizzati a cinque codoni che codificano i tag di esahistidine di N- o C-terminali sono stati acquistati in commercio. I geni sintetici sono stati amplificati mediante PCR e clonati individualmente in un vettore PCR-blunt e sequenziati. Per generare il plasmide policistronico, i cinque geni di interesse sono stati prima clonati in un idoneo plasmide pMGX, pMGX-A. La figura 2 mostra PCRBlunt- yfg1 e PCRBlunt- yfg2 in aggiunta a pMGX-A digerita con NdeI + EcoRI (vedi fase 2). Per clonare i primi due geni in pMGX-A, il plasmide è stato digerito con NdeI + EcoRI ( Figura 2 ). Dopo la clonazione dei geni di interesse in pMGX-A, i nuovi plasmidi, designati come pMGX- yfg1 e pMGX- yfg2 , sono stati digeriti con AvrII + XbaI per confermare che i cloni di successo sono stati ottenuti,Mostrato nelle bande da 1,1 a kb e 1,3 kb in Figura 3 .
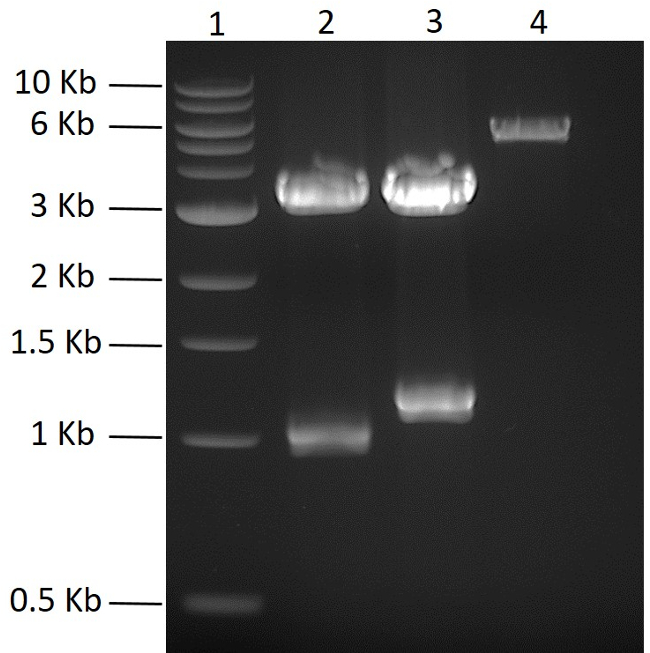
Figura 2: Clonazione di plasmidi pMGX. I risultati attesi di plasmidi PCR-blunt contenenti yfg1 e yfg2 dopo la digestione con NdeI + EcoRI sono mostrati su un agarosio di 0,7% (110V, 55 min). Lane 1, scala del DNA di 1 kb; Corsia 2, PCRBlunt- yfg1 ; Corsia 3, PCRBlunt- yfg2 ; Corsia 4, controllo pMGX-A. Clicca qui per visualizzare una versione più grande di questa figura.
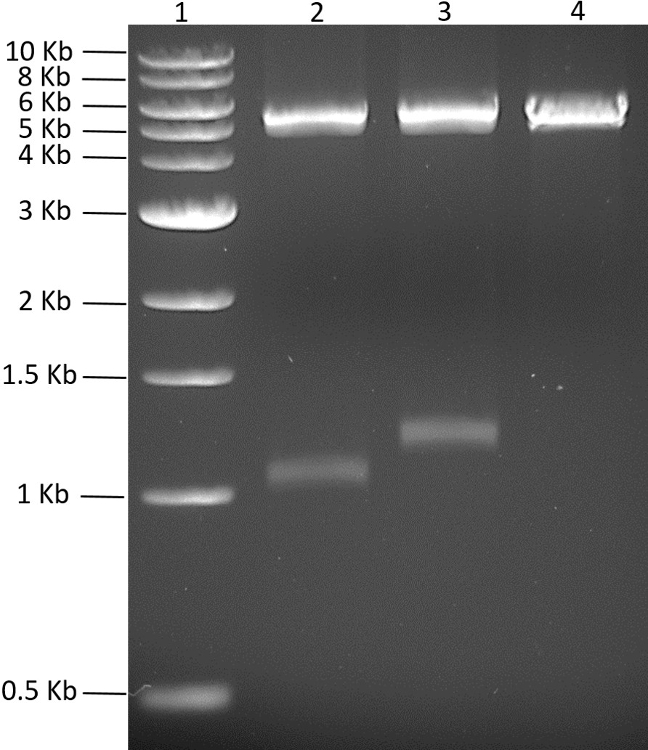
Figura 3: Screening di pMGX- yfg1 e pMGX- yfg2. Il 0,7% agarosio gel (110V, 55 min) mostra Le due bande generate da entrambi i pMGX- yfg1 e pMGX- yfg2 dopo la digestione con AvrII + XbaI. Lane 1, 1-kb scala del DNA; Corsia 2, pMGX- yfg1 ; La corsia 3, pMGX- yfg2 ; Corsia 4, controllo pMGX-A. Clicca qui per visualizzare una versione più grande di questa figura.
Con entrambi i geni nel corrispondente vettore pMGX, è stato costruito il plasmide policistronico. Per generare il plasmide pMGX- yfg1,2 , pMGX- yfg1 è stato digerito con AvrII, come mostrato in Figura 4 . PMGX- yfg2 è stato digerito con AvrII + XbaI e il gene da 1,3 kb contenente il frammento è stato legato nel sito AvrII di pMGX- yfg1 linearizzato, generando pMGX- yfg1,2 . La digestione di pMGX- yfg1,2 con EcoRI ha confermato la clonazione di successo del plasmide desiderato ed è mostrato in> Figura 5.

Figura 4: Generazione del plasmide bicistronic pMGX- yfg1,2. L'elettroforesi del gel agarosico del 0.7% (110V, 55 min) di pMGX- yfg1 digerito con AvrII e pMGX- yfg2 digeriti con AvrII + XbaI. Lane 1, 1-kb scala del DNA; La corsia 2, pMGX- yfg1 digerita con AvrII; La corsia 3, pMGX- yfg2 digerita con AvrII + XbaI; Corsia 4, controllo pMGX-A. Clicca qui per visualizzare una versione più grande di questa figura.
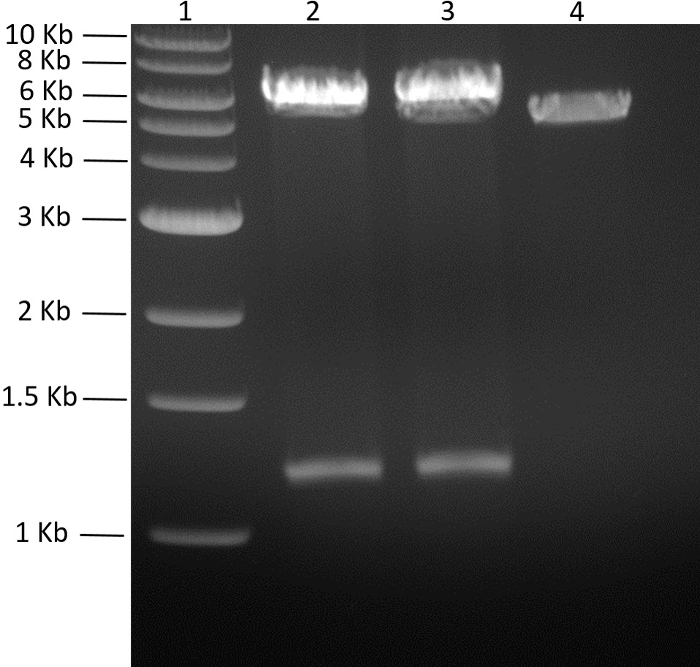
Figura 5: Screening dei cloni pMGX- yfg1,2 . Il risultante 0,7% agarosio gel (110 V, 55 min) di pMGX- yfSono mostrati g1,2 cloni digeriti con EcoRI. I cloni riusciti genereranno due bande, uno per il gene inserito yfg2 e l'altro corrispondente alla spina dorsale + yfg1 ( pMGX-yfg1 ). Lane 1, scala del DNA di 1 kb; Corsia 2, pMGX- yfg1,2 digerito con EcoRI, clone 1; Corsia 3, pMGX- yfg1,2 digerito con EcoRI, clone 2; Corsia 4, controllo pMGX-A. Clicca qui per visualizzare una versione più grande di questa figura.
Quando si genera un plasmide policistronico contenente due o più geni, si può incontrare un'inversione inversa indesiderata del gene clonato nel sito AvrII. La figura 6 è un gel che evidenzia la differenza tra i risultati di un plasmide digerito con un gene inserito nell'orientamento corretto, a differenza di un gene inserito nell'orientamento inverso. Se il gene èLegato e clonato nell'orientamento indesiderato, il sito EcoRI alla fine del gene sarà inserito proprio accanto al sito EcoRI del gene precedente. Ciò fornirà una dimensione di frammento che è praticamente indetectable da elettroforesi del DNA gel (meno di 50 bp). Inoltre, l'ultimo gene inserito apparirà nella spina dorsale, che è facilmente identificabile a causa della dimensione della spina dorsale più grande del previsto. La differenza nella dimensione della spina dorsale rispetto alla dimensione prevista indicherà la dimensione dell'ultimo gene inserito.

Figura 6: Screening dei cloni pMGX- yfg1-5 . Viene mostrato un 1,3% agarosio gel (110V, 65 min) che mostra i risultati di pMGX- yfg1-5 digeriti con EcoRI. I cloni di successo (corsia 2) genereranno una banda corrispondente all'ultimo gene inserito ( yfg5 in questo caso, 1,1 kb). lanE 1, 1-kb scala del DNA; Corsia 2, clone positivo di pMGX- yfg1-5, digerito con EcoRI; Corsia 3, clone negativo di pMGX- yfg1-5, digerito con EcoRI; Corsia 4, controllo pMGX- yfg1-4 . Clicca qui per visualizzare una versione più grande di questa figura.
Per completare il vettore di espressione polistronica a cinque gene, i restanti tre geni sono stati clonati uno dopo l'altro in pMGX- yfg1,2 per generare pMGX- yfg1-5 (vedi fase 5). Questo plasmide è stato quindi trasformato in BL21- (λDE3), e l'espressione delle proteine è stata indotta in una coltura di fase di mid-log con l'aggiunta di IPTG. La figura 7 è una macchia occidentale di lisati cellulari, confermando che tutte e cinque le proteine da un singolo plasmide contenente geni multipli in un singolo operone possono essere espressi. L'espressione di pMGX- yfg1 (contenente un gene),Sono mostrati pMGX- yfg1,2 (contenenti 2 geni) e pMGX- yfg1-5 (contenenti 5 geni).
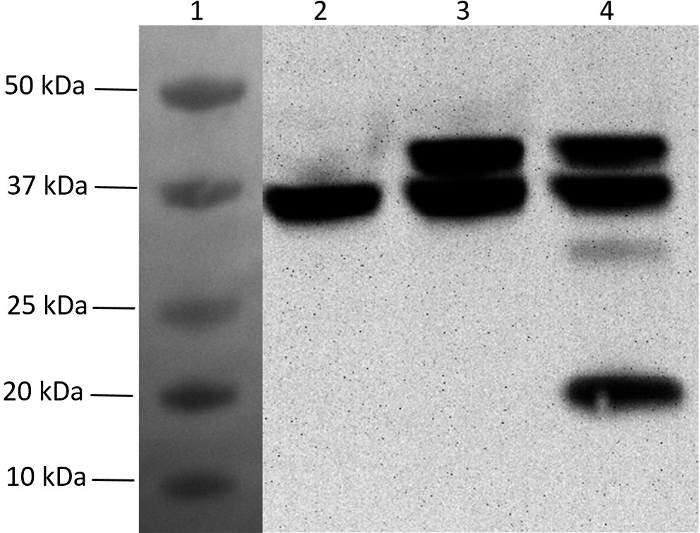
Figura 7: Analisi Western blot di espressione multi-gene usando il sistema pMGX. L'incorporazione di etichette esahistidiche di N- o C-terminali ha consentito l'individuazione Western blot di espressione proteica. I campioni sono stati separati su gel a base di acrilammide (200 V, 35 min, 10 cm x 8,5 cm) senza gradi di gradiente a gradiente di 4-20% e successivamente un trasferimento (40 V, 90 min) su una membrana nitrocellulosa (0,45 μm, 10 cm x 7 cm). L'anticorpo monoclonale anti-His coniugato con HRP, che non richiede un anticorpo secondario, è stato utilizzato. Tamponi di bloccaggio, trasferimento e tamponi di diluizione anticorpali sono stati preparati come raccomandato dal produttore di anticorpi. La rilevazione è stata eseguita con il substrato HRP Chemiluminescent occidentale seguendo le istruzioni del produttoretructions. La membrana risultante è stata visualizzata usando un imager senza filtro. Lane 1, standard a doppio colore trasferito sulla membrana (non chemiluminescente); La corsia 2, His6pMGX- yfg1 ; La corsia 3, His6pMGX- yfg1,2 ; La corsia 4, His6pMGX- yfg1-5. Nota: La proteina prodotta da yfg5 è la stessa dimensione della proteina prodotta da yfg1 (entrambi sono 36 kDa) e non possono essere separati sul gel. Clicca qui per visualizzare una versione più grande di questa figura.
Figure aggiuntive: Fare clic qui per scaricare questo file.
Discussione
La coespressione di geni multipli è sempre più essenziale, in particolare per caratterizzare e ricostituire complessi percorsi metabolici multigene 3 , 4 , 5 . Il sistema pMGX rende coespressione multigene in routine E. coli 6 , 7 , 8 e accessibile a diversi ricercatori. In questo studio sono state dimostrate che cinque proteine di interesse sono state prodotte contemporaneamente da un unico sistema plasmidico usando pMGX-A. Molti sistemi di coespressione attualmente disponibili consentono solo l'inserimento di due geni, generando plasmidi bicistronic come i vettori pET Duet 12 . Se si richiede l'aggiunta di più geni, sono necessari vettori bicistronic multipli con differenti marcatori selectable. Al contrario di altri sistemi di espressione multigene, il sistema pMGX consente l'facile clonazioneDi geni multipli in un plasmide, con la capacità di riutilizzare i siti di restrizione senza la necessità di diversi plasmidi donatori o di rimuovere siti multipli di restrizione dai geni 13 , 14 . I passi critici di questo protocollo includono la progettazione del gene, la prevenzione della ricircolazione di sfondo dei vettori pMGX singolarmente digeriti e schermi dei vettori policistronici per l'orientamento corretto del gene inserito.
Nel pianificare la strategia di clonazione per costruire un sistema policistronic, è essenziale garantire che i geni di interesse non contengano siti di restrizione necessari per la clonazione a valle. In particolare, questi includono XbaI e AvrII, che sono necessari per l'inserimento del secondo e dei successivi geni nel vettore pMGX contenente il primo gene di interesse (passaggi 3 e 4). Inoltre, è essenziale eliminare i siti di restrizione che saranno utilizzati per la clonazione iniziale del gDi interesse in pMGX (fase 2). Tipicamente, questi includono NdeI o NheI all'estremità 5 'del gene e BamHI o EcoRI all'estremità 3 del gene, sebbene altri siti di restrizione possono essere utilizzati anche per questo passaggio, se necessario. L'eliminazione di siti di restrizione extra-indesiderati può essere facilmente eseguita allo stadio di sintesi del gene o, nel caso di geni clonati da altre fonti, mediante sostituzioni nucleotidiche tramite la mutagenesi diretta sul sito 20 .
L'aggiunta di geni successivi di interesse in pMGX contenente il primo gene di interesse richiede linearizzazione di pMGX- yfg1 con AvrII (fase 3.1, nota che può anche essere linearizzata con XbaI). Questo vettore linearizzato verterà rapidamente durante l'inserimento del secondo gene di interesse (punto 3.5), portando ad un numero estremamente elevato di cloni di fondo. Per evitare questo, è essenziale trattare il vettore linearizzato con una fosfatasi per defosforilare il 5 °2; Estremità del vettore linearizzato. Tipicamente, viene utilizzato il CIP, anche se altri sono disponibili. L'ottimizzazione di entrambe le unità di fosfatasi e del tempo di incubazione può essere necessaria per ottimizzare le legazioni dei geni successivi di interesse nel vettore linearizzato.
Infine, quando si genera un sistema policistronico, il gene di interesse viene inizialmente eliminato dalla digestione con XbaI e AvrII, generando estremità coesive complementari alle estremità 5 'e 3' del gene (Fasi 3.2 e 4.2). Questo inserto può essere legato nel vettore linearizzato in direzione avanti o inversa. Poiché tutte le estremità coesive sono identiche, nessuna direzionalità viene applicata mediante l'addensamento complementare delle estremità coesi. È quindi necessario schermare i cloni risultanti per la corretta direzione di inserimento del gene di interesse. Ciò può essere fatto facilmente mediante la selezione di cloni con uno dei siti di restrizione utilizzati per clonare il gene originale di interesse in pMGX (punto 2). Tipicamente,Si usa EcoRI, in quanto il sito di restrizione si trova all'estremità 3 'del gene e quindi è opportuno controllare la direzionalità, come mostrato in Figura 6 . Nella maggior parte dei casi, il 50% dei cloni contenenti il gene di interesse ha il gene nell'orientamento corretto e il 50% ha il gene nell'orientamento opposto.
Infrequentemente (~ 10% delle legazioni), tutti i cloni di una particolare legatura possono avere il gene di interesse inserito in una sola direzione. Questo risultato sembra essere dipendente dalla sequenza, in quanto è altamente riproducibile in questi casi specifici. Se ciò si verifica e il gene di interesse viene continuamente inserito nell'orientamento inverso, è possibile accedere normalmente al vettore desiderato commutando la direzione della clonazione. Ad esempio, invece di clonare yfg2 nel sito AvrII di pMGX- yfg1 , yfg1 può essere clonata nel sito XbaI di pMGX- yfg2 . Si noti che questo fornisce il identico vettore finalesistema. Questa flessibilità aggiunta che consente l'accesso allo stesso sistema da diverse strategie di clonazione è estremamente vantaggioso.
Una limitazione importante di questa metodologia è che le proteine codificate all'estremità 3 'del trascritto polistirico sono tipicamente espresse a livelli inferiori rispetto alle proteine codificate all'estremità 5 della trascrizione e questo effetto è più pronunciato quanto più lungo è la trascrizione . Ciò significa che cambiare l'ordine dei geni di interesse nell'operone sintetico può influenzare i livelli di espressione relativa delle proteine che codificano, fornendo un meccanismo per il raffronto dei livelli di espressione relativa.
In sintesi, il sistema pMGX fornisce un metodo affidabile per la coespressione di più proteine da un singolo plasmide in E. coli , che può essere utilizzato per una varietà di applicazioni di biologia sintetica e per la caratterizzazione del percorso biochimico.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal Consiglio di Ricerca delle Scienze Naturali e Ingegneria del Canada.
Materiali
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Riferimenti
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon