Method Article
Induzierbares T7-RNA-Polymerase-vermitteltes Multigen-Expressionssystem, pMGX
In diesem Artikel
Zusammenfassung
Diese Studie beschreibt Methoden für die T7-vermittelte Co-Expression von mehreren Genen aus einem einzigen Plasmid in Escherichia coli unter Verwendung des pMGX-Plasmidsystems.
Zusammenfassung
Die Co-Expression mehrerer Proteine ist für die synthetische Biologie immer wichtiger, studiert Protein-Protein-Komplexe und charakterisiert und nutzt Biosynthesewege. In diesem Manuskript wird die Verwendung eines hochwirksamen Systems für die Konstruktion von multigenen synthetischen Operons unter der Kontrolle einer induzierbaren T7-RNA-Polymerase beschrieben. Dieses System erlaubt es, dass viele Gene gleichzeitig aus einem Plasmid exprimiert werden. Hier ist ein Satz von vier verwandten Vektoren, pMGX-A, pMGX-hisA, pMGX-K und pMGX-hisK, entweder mit dem Ampicillin- oder Kanamycin-resistente Marker (A und K) und entweder mit einem N-terminalen Hexahistidin oder einem N-terminalen Hexahistidin Tag (seine) werden offenbart. Detaillierte Protokolle für die Konstruktion von synthetischen Operen, die dieses Vektorsystem verwenden, werden zusammen mit den entsprechenden Daten bereitgestellt, was zeigt, dass ein pMGX-basiertes System, das fünf Gene enthält, leicht konstruiert und verwendet werden kann, um alle fünf codierten Proteine in Escherichia coli zu produzieren . Dieses systEm und Protokoll ermöglicht es Forschern, routinemäßig komplexe Mehrkomponentenmodule und Wege in E. coli auszudrücken.
Einleitung
Die Koexpression mehrerer Proteine ist zunehmend von wesentlicher Bedeutung, vor allem in synthetischen Biologieanwendungen, bei denen mehrere Funktionsmodule ausgedrückt werden müssen 1 ; Bei der Untersuchung von Protein-Protein-Komplexen, wo Expression und Funktion oft Co-Expression 2 , 3 erfordern; Und bei der Charakterisierung und Nutzung von Biosynthesewegen, wo jedes Gen im Weg ausgedrückt werden muss 4 , 5 , 6 , 7 , 8 . Eine Reihe von Systemen wurde für die Co-Expression entwickelt, insbesondere im Wirtsorganismus Escherichia coli , dem Arbeitspferd für die rekombinante Protein-Expression des Labors 9 . Zum Beispiel können mehrere Plasmide mit unterschiedlichen selektierbaren Markern verwendet werden, um einzelne Proteine unter Verwendung einer Fülle von verschiedenen ex zu exprimieren Druckvektoren 10 , 11 . Einzelne Plasmidsysteme für die multiple Proteinexpression haben entweder mehrere Promotoren verwendet, um die Expression jedes Gens 10 , 12 zu kontrollieren; Synthetische Operons, wo mehrere Gene auf einem einzigen Transkript 2 , 13 codiert sind; Oder in einigen Fällen ein einzelnes Gen, das für ein Polypeptid kodiert, das letztlich proteolytisch verarbeitet wird, wobei die gewünschten interessierenden Proteine 14 erhalten werden .

Abbildung 1: pMGX-Workflow, der den Aufbau eines polycistronischen Vektors zeigt. Das pMGX-System bietet eine flexible, einfach zu bedienende Strategie für den Bau von synthetischen Operons unter der Kontrolle eines induzierbaren T7-Promotors.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
In diesem Manuskript wird die Verwendung eines hochwirksamen Systems für die Konstruktion von multigenen synthetischen Operons unter der Kontrolle einer induzierbaren T7-RNA-Polymerase ( Fig. 1 ) beschrieben. Dieses System erlaubt es, dass viele Gene gleichzeitig aus einem Plasmid exprimiert werden. Es basiert auf einem Plasmidsystem, das ursprünglich pKH22 genannt wurde und das für eine Reihe von verschiedenen Anwendungen 6 , 7 , 8 erfolgreich eingesetzt wurde . Hier wird dieser Plasmidsatz um vier verwandte Vektoren erweitert: pMGX-A, ein Expressionsvektor ohne C- oder N-terminale Markierungen und mit dem Ampicillin-Resistenzmarker; PMGX-hisA, ein Expressionsvektor, der für ein N-terminales Hexahistidin-Tag und mit dem Ampicillin-Resistenzmarker kodiert; PMGX-K, ein Expressionsvektor lAcking alle C- oder N-terminalen Markierungen und mit dem Kanamycin-Resistenz-Marker; Und pMGX-hisK, ein Expressionsvektor, der für ein N-terminales Hexahistidin-Tag und mit dem Kanamycin-Resistenzmarker kodiert. In dieser Studie wird das Verfahren zur Erzeugung eines polycistronischen Vektors, der fünf Gene enthält, unter Verwendung des pMGX-Systems, insbesondere pMGX-A, zusammen mit der erfolgreichen Produktion jedes einzelnen Proteins in Escherichia coli gezeigt .
Protokoll
1. Erhalten von Genen von Interesse
- Design synthetische Gene.
- Optimieren Sie eine Gensequenz für die E. coli- Expression.
- Entfernen Sie alle problematischen Restriktionsstellen aus der Sequenz (AvrII, NdeI, EcoRI und XbaI).
- Einbindung von Restriktionsstellen für das Klonen; Eine 5'-NdeI-Stelle und eine 3'-EcoRI-Stelle werden empfohlen. Andere Standorte können bei Bedarf verwendet werden; Beziehen sich auf den Multicloning-Bereich des ausgewählten Plasmids ( Abbildung S1- S4 ). Falls gewünscht, bitte ein 5 'oder 3' Codierungs-Tag für die Western-Blot-Erkennung.
- Kommerziell das entworfene Gen bestellen
- Entweder stumpf klon das Gen in einen stumpfen Vektor unter Verwendung eines stumpfen Klonierungskits, wie nach den Anweisungen des Herstellers; Design-Primer zur Amplifikation des Gens (dann weiter mit Schritt 1.2.2); Oder zusätzliche 5'- und 3'-Enden zur direkten Klonierung in ein pMGX-Plasmid hinzufügen und mit Schritt 2 fortfahrenDie repräsentativen Ergebnisse stammen aus dem stumpfen Klonen der Gene von Interesse.
- Amplifizieren des gewünschten Gens (aus einem entworfenen und optimierten synthetischen Gen oder aus Templat-DNA, die das gewünschte Gen enthält) 15 .
- Design-Primer mit Restriktionsstellen für das Klonen; Eine 5'-NdeI-Stelle und eine 3'-EcoRI-Stelle werden empfohlen. Andere Standorte können bei Bedarf verwendet werden; Beziehen sich auf den Multicloning-Bereich des ausgewählten Plasmids ( Abbildung S1- S4 ). Wenn gewünscht, bitte entweder ein 5 'oder 3' Codierungs-Tag für Western Blot-Erkennung.
- PCR amplifiziert das gewünschte Gen 15 .
- Analysieren Sie die PCR durch Agarosegelelektrophorese 16 .
- Wenn es unspezifische Amplifikation gibt, reinigen Sie das amplifizierte Gen durch Gel-Extraktion. Wenn nicht, verwenden Sie ein Enzym-Clean-up-Kit.
- Quantifizierung der DNA unter Verwendung eines Spektrophotometers durch Überprüfung des AbsorptionsmittelsE bei 260 nm; Leer mit Elutionspuffer 17 .
2. Klonen von Genen von Interesse in ein Multigene Expression System Vector, pMGX 18
- Die Beschränkung verdrängt das erhaltene Gen von Interesse und den gewünschten Vektor, pMGX, mit NdeI und EcoRI.
HINWEIS: Wenn eine große Menge an rezirkulierten Plasmiden erhalten wird, lassen Sie die NdeI-Reaktion 1 h vor der Zugabe von EcoRI ablaufen.- Verwenden Sie eine 40 μl Digest-Reaktion mit 0,5-1,5 μg DNA und 1 μl jedes Enzyms mit 4 μl des entsprechenden 10x Puffers.
- Für einen NdeI- und EcoRI-Digest verwenden Sie EcoRI-Puffer und fügen Sie zuerst NdeI-Endonuklease hinzu. Digest für 1 h bei 37 ° C. Dann füge EcoRI-Endonuklease hinzu und lasse den Digest für eine weitere Stunde ablaufen. NdeI ist empfindlich gegenüber Spaltungen nahe am Ende der DNA, so dass die anfängliche Verdauung durch EcoRI eine wirksame Verdauung durch NdeI verhindern kann.
- ElektrOphorese die Restriktionsverdauung auf Agarosegel (für ein 1 kb-Gen, verwenden Sie ein 0,7% Agarosegel bei 110 V für 55 min, um den Prozentsatz des Agarosegels für unterschiedliche Gengrößen auszuwählen, siehe Referenz 16 .
- Exzisionseinsatz und Vektorbänder mit einem sauberen Skalpell oder Rasierklinge und legen das ausgeschnittene Gelsegment in ein 1,5 mL Röhrchen.
- Extrahieren Sie die DNA aus dem Agarosegel mit einem Gel-Extraktionskit gemäß dem Protokoll des Herstellers.
- Ligieren Sie das Gen von Interesse in den pMGX-Vektor unter Verwendung eines 3: 1-Insert-zu-Vektor-Verhältnisses; Eine negative Ligation von verdautem pMGX ohne den Einsatz einrichten.
- Stellen Sie eine 20 & mgr; l Ligationsreaktion ein, die 1 & mgr; l T4-DNA-Ligase, 0,15-0,5 & mgr; g Vektor-DNA (& ndash; 5 & mgr; l) und eine geeignete Menge an Insert auf der Basis eines 3: 1-Insert-zu-Vektor-Verhältnisses und der Grße von & lgr; Das Gen wird eingefügt; Die pMGX-Backbones sind zwischen 5.312 und 5.504 bp groß. Füge eine Negativkontrolle ein, die alles enthältWenn das Gen eingefügt wird. Sicherstellen, dass die Menge an Vektor-DNA in der Negativkontrollligatur äquivalent ist und der Vektor plus Insert-Ligationsreaktionen.
- Transformieren von Ligationsreaktionen in XL1-Blue-chemisch kompetente E. coli- Zellen und ligierte Plasmide pMGX- yfg1 (enthaltendes Gen von Interesse 1), pMGX- yfg2 (enthaltendes Gen von Interesse 2) ... pMGX- yfgn (mit Gen von Interesse n). Verwenden Sie aseptische Technik (innerhalb eines Biosicherheitskabinetts oder unter einer Flamme).
- 100 μl Aliquots von chemisch kompetenten XL1-Blue E. coli auf Eis für 5 min auftauen und dann 5 μl der Ligationsreaktionen hinzufügen. Inkubieren für 30 min auf Eis.
- Schütteln Sie die Zellen für 45 s in einem Wasserbad, das bei 42 ° C gehalten wird, und fügen Sie dann 200 & mgr; l kaltes LB hinzu. Inkubieren sie auf Eis für 2 min.
- Schütteln Sie die Zellen bei 37 ° C, 220 U / min für 1 h und dann die Platte 100 μl auf eine LB-Agar-Platte, die eine Zulassung enthält,Eine vorwählbare Markierung (entweder Ampicillin oder Kanamycin).
- Screen die Klone für positive Transformanten.
- Vergleichen Sie die Kolonie zählt auf der negativen Kontrolle und Ligation Platten. Ein Kolonenzählungsverhältnis größer als 1: 2 ist erwünscht. Wenn es eine große Anzahl von Kolonien auf der negativen Kontrollplatte gibt, gehen Sie zurück zu Schritt 2.1 und überprüfen Sie die Notiz.
- Wählen Sie aus jeder Ligationsreaktion der verschiedenen Gene, die zum Screening (1 n) eingefügt wurden, 4-8 einzelne Kolonien (abhängig von der Negativkontrolle: Ligationsverhältnis) und inokulieren Sie eine 4 ml LB und die entsprechende Antibiotika-Übernachtkultur / Einzelkolonie. Wachsen Sie Kulturen über Nacht mit Schütteln bei 37 ° C und 220 U / min.
- Isolieren Sie Plasmid-DNA unter Verwendung eines Plasmid-DNA-Isolationskits gemäß dem Protokoll des Herstellers.
- Richten Sie eine 20 μl NdeI + EcoRI-Digest-Reaktion ein, die 150-500 ng DNA und 1 μl jedes Enzyms mit 2 μl des entsprechenden 10x Puffers enthält. In thIst der Fall, füge NdeI und EcoRI zur gleichen Zeit hinzu, dass das Gen von Interesse zwischen den Restriktionsstellen liegt. Eine negative Kontrolle mit dem leeren pMGX-Vektor wird empfohlen. Digest für 2 h bei 37 ° C im Wasserbad.
3. Einfügen von Gen 2 in den pMGX-Vektor, der das Gen 1 enthält, pMGX- yfg1
- Restriktions-Digest pMGX- yfg1 mit AvrII und behandeln mit Kalb-Darm-Phosphatase (CIP).
- Verwenden Sie eine 40 μl Digest-Reaktion, die 0,5-1,5 μg Vektor-DNA (~ 5-10 μl isolierte DNA) und 1 μl AvrII mit 4 μl des entsprechenden 10x Puffers enthält. Digest für 1,5 h bei 37 ° C und füge dann 1,5 μl CIP hinzu. Lassen Sie es bei 37 ° C für weitere 30 min.
- Restriktionsverdauung pMGX- yfg2 mit AvrII und XbaI, um das Gen von Interesse zu befreien.
- Verwenden Sie eine 40 μl Digest-Reaktion, die 0,5-1,5 μg DNA und 1 μ enthält, L jedes Enzyms mit 4 & mgr; l des geeigneten 10 × Puffers. Digest für 2 h bei 37 ° C.
- Elektrophorese Restriktion verdaut auf einem entsprechenden Prozent (0,7%) Agarosegel und verbraucht die Insert- und Vektorbänder mit einem sauberen Skalpell / Rasierklinge (siehe Schritte 2.2-2.3).
- Extrahiere die DNA mit einem Gel-Extraktionskit nach dem Protokoll des Herstellers und quantifiziere die DNA 17 .
- Ligat-Gen 2 in pMGX- yfg1 unter Verwendung eines 3: 1-Insert-to-Vektor-Verhältnisses. Richten Sie eine negative Ligation von verdautem pMGX- yfg1 ohne den zusätzlichen Einsatz ein. Einrichten wie oben in Schritt 2.5.
- Verwandeln Sie 5 μl der Ligationsreaktionen in XL1-Blue-chemisch kompetente E. coli- Zellen, ligierte Plasmide pMGX- yfg1,2 (enthaltendes Gen von Interesse 1 und 2) und eine negative pMGX- yfg1- Kontrolle, wie in Schritt 2.6 zu sehen ist.
- Vergleichen Sie die Koloniezahl auf die Negativkontrolle und Ligationsplatten. Ein Kolonienzahlverhältnis gReater als 1: 2 ist erwünscht. Wenn es eine große Anzahl von Kolonien auf der negativen Kontrollplatte gibt, gehen Sie zurück zu Schritt 3.1 und überprüfen Sie die CIP-Behandlung.
- Wählen Sie aus der Ligationsreaktion 4-8 Einzelkolonien (abhängig von der Negativkontrolle: Ligationsverhältnis) und inokulieren Sie 4 ml LB plus das entsprechende Antibiotikum pro Einzelkolonie und wachsen über Nacht bei 37 ° C und 220 U / min.
- Isolieren Sie Plasmid-DNA unter Verwendung eines Plasmid-DNA-Isolationskits gemäß dem Protokoll des Herstellers und quantifizieren Sie die DNA 17 .
- Screen die effektive Insertion des zweiten Gens durch Durchführen einer Restriktionsverdauung von pMGX- yfg1,2 mit EcoRI.
- Verwenden Sie eine 20 μl Digest-Reaktion, die 150-500 ng DNA und 1 μl EcoRI-Enzym enthält, mit 2 μl EcoRI 10x Puffer. Digest für 2 h bei 37 ° C.
- Elektrophorese die Restriktionsverdauung auf einem geeigneten prozentualen Agarosegel; Suche nach einer Band, die korrespondiertS auf die Größe des Gens 2 (siehe Schritt 2.2). Ein Gen kann in den Vektor in die unerwünschte, umgekehrte Orientierung einfügen.
4. Hinzufügen eines dritten Gens in den pMGX-Vektor, der die Gene 1 und 2 enthält, pMGX- yfg1,2
- Beschränkung digest pMGX- yfg1,2 mit AvrII und behandeln mit CIP, wie in Schritt 3.1 gesehen.
- Einschränkung digest pMGX- yfg3 mit AvrII und XbaI, wie in Schritt 3.2 gesehen.
- Elektrophorese die Restriktion verdaut auf einem geeigneten Prozent Agarosegel und Exzisionseinsatz und Vektorbänder unter Verwendung einer sauberen Skalpell / Rasierklinge; Siehe Schritte 2.2-2.3.
- Extrahieren Sie die DNA aus dem Agarosegel mit einem Gel-Extraktionskit und quantifizieren Sie die DNA 17 .
- Ligat-Gen 3 in pMGX- yfg1,2 unter Verwendung eines 3: 1-Insert-to-Vector-Verhältnisses und stellte eine negative Ligation von verdautem pMGX- yfg1,2 ohne ein zusätzliches Insert auf, wie in Schritt 3.5 zu sehen ist.
- Umwandlung von 5 μl der Ligationsreaktionen in XL1-blaue chemisch kompetente E. coli- Zellen, ligiertes Plasmid pMGX- yfg1,2,3 (enthaltende Gene von Interesse 1, 2 und 3) und negative pMGX- yfg1,2- Kontrolle, wie in Schritt 2.6 gesehen.
- Vergleichen Sie die Koloniezahl auf die Negativkontrolle und Ligationsplatten. Wenn es eine große Anzahl von Kolonien auf der negativen Kontrollplatte gibt, gehen Sie zurück zu Schritt 4.1 und überprüfen Sie die CIP-Behandlung.
- Wählen Sie aus der Ligationsreaktion 4-8 einzelne Kolonien (abhängig von der Negativkontrolle: Ligationsverhältnis) und 4xL LB plus das entsprechende Antibiotikum pro Einzelkolonie inokulieren; Über Nacht bei 37 ° C und 220 U / min wachsen.
- Isolieren Sie die Plasmid-DNA unter Verwendung eines Plasmid-DNA-Isolationskits gemäß dem Protokoll des Herstellers.
- Screen die effektive Insertion des dritten Gens durch Durchführen einer Restriktionsverdauung von pMGX- yfg1,2,3 mit EcoRI, wie in Schritt 3.10 gesehen.
- Elektrophorese die Restriktionsverdauung auf einem geeigneten prozentualen Agarosegel; SchauFür Banden, die den Größen von Gen 2 und Gen 3 entsprechen (siehe Schritt 2.2). Anmerkung: Gen 3 kann in den Vektor in der unerwünschten, umgekehrten Orientierung einfügen. Wenn die Gene 2 und 3 die gleiche Größe haben, muss eine andere geeignete Beschränkungs-Digest-Site für das Screening ausgewählt werden.
HINWEIS: Wiederholen Sie, wie es für jedes neue Gen benötigt wird.
5. Herstellung von Proteinen von Interesse mit einem Multigene Expression System und Bewertung der Produktion durch Western Blotting
- Verwandeln Sie den positiven Klon, der alle Gene von Interesse enthält, in chemisch kompetente Proteinproduktion E. coli , wie BL21- (λDE3).
- 100 μl Aliquote von chemisch kompetenten BL21- (λDE3) E. coli auf Eis für 5 min auftauen und dann 1 μl der positiv klonierten Plasmid-DNA zugeben; 30 min auf Eis inkubieren
- Schütteln Sie die Zellen für 45 s in einem Wasserbad, das bei 42 ° C gehalten wird, und fügen Sie dann 200 & mgr; l kaltes LB hinzu. Inkubieren auf Eis für 2 min.
- Schütteln Sie die Zellen bei 37 ° C und 220 U / min für 1 h und dann die Platte 100 μl auf eine LB-Agar-Platte, die den geeigneten selektierbaren Marker (entweder Ampicillin oder Kanamycin) enthält.
- Expression des Proteins durch Isopropyl-β-D-1-thiogalactopyranosid (IPTG) Induktion.
- Wählen Sie eine isolierte Kolonie aus der B21- (λDE3) Transformationsplatte und inokulieren Sie 4 ml LB plus das entsprechende Antibiotikum; Wachsen über Nacht und schütteln bei 37 ° C und 220 U / min.
- Inokulieren Sie eine 100 ml LB plus die entsprechende antibiotische Kultur mit 1 ml über Nacht Kultur.
- Bei 37 ° C unter Schütteln bei 220 U / min auf eine OD 600 von 0,6 wachsen.
- Die Kultur mit 100 μM IPTG induzieren und 15 h bei 25 ° C und 220 U / min wachsen lassen.
- 1 ml der Kultur entfernen und bei 13000 xg für 1 min zentrifugieren; Den Überstand verwerfen.
- Lyse die Zellen mit Lyse-Lösung gemäß den Anweisungen des HerstellersUnd präformieren einen Western-Blot des löslichen Zelllysats, um festzustellen, ob alle Proteine erfolgreich produziert wurden 19 .
Ergebnisse
In dieser Studie war es das Ziel, fünf Proteine aus einem einzigen Plasmid zu exprimieren. Die Fünf-Codon-optimierten synthetischen Genfragmente, die entweder N- oder C-terminale Hexahistidin-Markierungen codieren, wurden kommerziell gekauft. Die synthetischen Gene wurden durch PCR amplifiziert und einzeln in einen PCR-stumpfen Vektor kloniert und sequenziert. Um das polycistronische Plasmid zu erzeugen, wurden die fünf interessierenden Gene zuerst in ein geeignetes pMGX-Plasmid pMGX-A kloniert. Abbildung 2 zeigt PCRBlunt- yfg1 und PCRBlunt- yfg2 zusätzlich zu pMGX-A, verdaut mit NdeI + EcoRI (siehe Schritt 2). Um die ersten beiden Gene in pMGX-A zu klonieren, wurde das Plasmid mit NdeI + EcoRI verdaut ( Abbildung 2 ). Bei der Klonierung der interessierenden Gene in pMGX-A wurden die neu konstruierten Plasmide, die als pMGX- yfg1 und pMGX- yfg2 bezeichnet wurden, mit AvrII + XbaI verdaut, um zu bestätigen, dass erfolgreiche Klone erhalten wurdenGezeigt in den in Fig. 3 erhaltenen 1,1-kb- und 1,3-kb-Bändern.
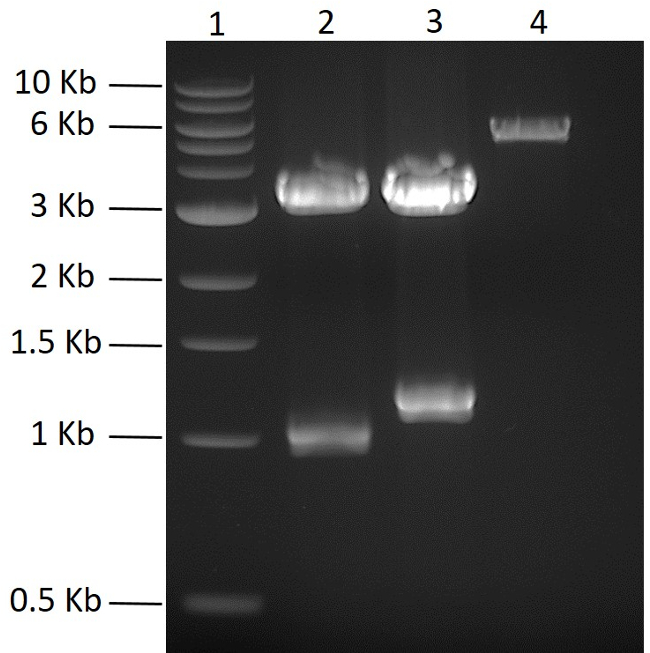
Abbildung 2: Klonierung von pMGX-Plasmiden. Die erwarteten Ergebnisse von PCR-stumpfen Plasmiden, die yfg1 und yfg2 nach dem Verdau mit NdeI + EcoRI enthalten, sind auf einem 0,7% Agarosegel (110 V, 55 min) gezeigt. Spur 1, 1 kb DNA Leiter; Spur 2, PCRBlunt- yfg1 ; Spur 3, PCRBlunt- yfg2 ; Spur 4, pMGX-A Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
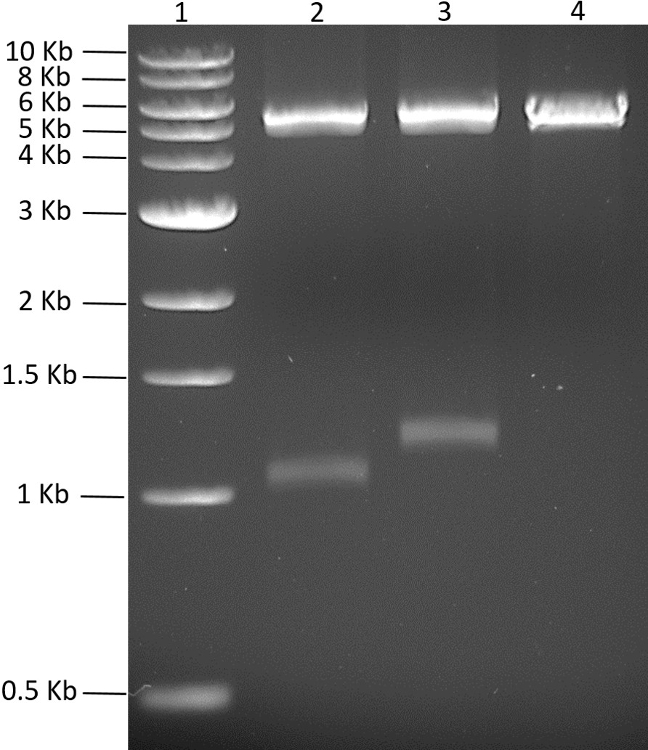
Abbildung 3: Screening von pMGX- yfg1 und pMGX- yfg2. Das 0,7% Agarosegel (110 V, 55 min) zeigt Die beiden Banden, die nach dem Verdau mit AvrII + XbaI von pMGX- yfg1 und pMGX- yfg2 erzeugt werden. Spur 1, 1-kb DNA Leiter; Spur 2, pMGX- yfg1 ; Spur 3, pMGX- yfg2 ; Spur 4, pMGX-A Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Bei beiden Genen in dem entsprechenden pMGX-Vektor wurde das polycistronische Plasmid konstruiert. Um das Plasmid pMGX- yfg1,2 zu erzeugen, wurde pMGX- yfg1 mit AvrII verdaut, wie in Abbildung 4 gezeigt . PMGX- yfg2 wurde mit AvrII + XbaI verdaut und das 1,3-kb-Gen, das das Fragment enthielt, wurde in die AvrII-Stelle von linearisiertem pMGX- yfg1 ligiert , wodurch pMGX- yfg1,2 erzeugt wurde . Die Verdauung von pMGX- yfg1,2 mit EcoRI bestätigte die erfolgreiche Klonierung des gewünschten Plasmids und ist in gezeigt> Abbildung 5

Abbildung 4: Erzeugung des bicistronischen Plasmids pMGX- yfg1,2. Die 0,7% Agarose-Gelelektrophorese resultiert (110 V, 55 min) von pMGX- yfg1, verdaut mit AvrII und pMGX- yfg2, verdaut mit AvrII + XbaI. Spur 1, 1-kb DNA Leiter; Spur 2, pMGX- yfg1 verdaut mit AvrII; Spur 3, pMGX- yfg2 verdaut mit AvrII + XbaI; Spur 4, pMGX-A Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
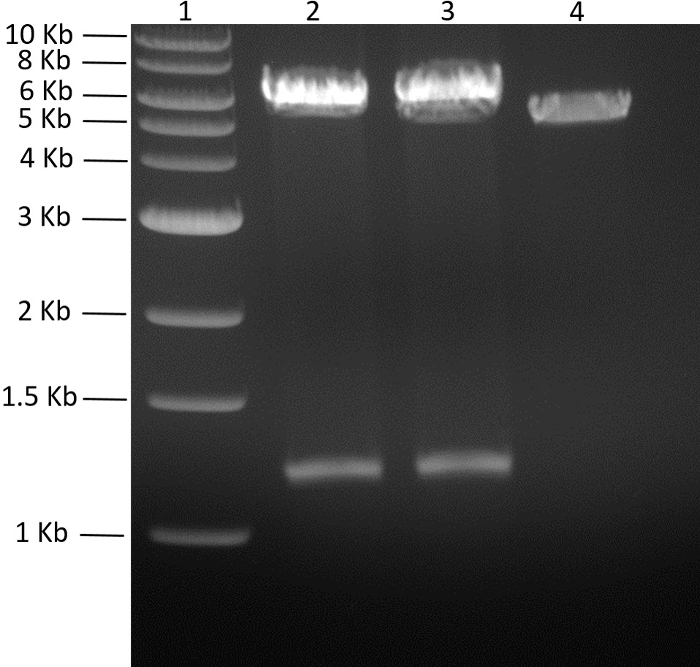
Abbildung 5: Screening von pMGX- yfg1,2 Klonen . Das resultierende 0,7% Agarosegel (110 V, 55 min) von pMGX- yfG1,2 Klone, die mit EcoRI verdaut wurden, wird gezeigt. Erfolgreiche Klone erzeugen zwei Banden, eine für das eingefügte Gen yfg2 und die andere dem Backbone + yfg1 ( pMGX-yfg1 ). Spur 1, 1 kb DNA Leiter; Spur 2, pMGX- yfg1,2 verdaut mit EcoRI, Klon 1; Spur 3, pMGX- yfg1,2 verdaut mit EcoRI, Klon 2; Spur 4, pMGX-A Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Bei der Erzeugung eines polycistronischen Plasmids, das zwei oder mehr Gene enthält, kann man eine unerwünschte umgekehrte Insertion des in die AvrII-Stelle klonierten Gens treffen. Fig. 6 ist ein Gel, das den Unterschied zwischen den Ergebnissen eines verdauten Plasmids mit einem in die korrekte Orientierung eingefügten Gen im Gegensatz zu einem Gen, das in die umgekehrte Orientierung eingefügt ist, hervorhebt. Wenn das Gen istLigiert und in der unerwünschten Orientierung kloniert, wird die EcoRI-Stelle am Ende des Gens direkt neben der EcoRI-Stelle des vorherigen Gens inseriert. Dies würde eine Fragmentgröße ergeben, die durch DNA-Gelelektrophorese (weniger als 50 bp) praktisch nicht nachweisbar ist. Zusätzlich würde das zuletzt eingefügte Gen im Rückgrat erscheinen, was leicht identifizierbar ist, da die Backbone-Größe größer als erwartet ist. Der Unterschied in der Größe des Rückgrats aus der erwarteten Größe gibt die Größe des zuletzt eingefügten Gens an.

Abbildung 6: Screening von pMGX- yfg1-5 Klonen . Es wurde ein 1,3% Agarosegel (110 V, 65 min) gezeigt, das die mit EcoRI verdauten Ergebnisse von pMGX- yfg1-5 darstellt . Erfolgreiche Klone (Spur 2) erzeugen eine Bande, die dem zuletzt eingefügten Gen entsprechen ( yfg5 in diesem Fall 1,1 kb). LanE 1, 1-kb DNA Leiter; Spur 2, positiver Klon von pMGX- yfg1-5, verdaut mit EcoRI; Spur 3, negativer Klon von pMGX- yfg1-5, verdaut mit EcoRI; Spur 4, pMGX- yfg1-4 Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Um den fünf-Gen-polycistronischen Expressionsvektor zu vervollständigen, wurden die verbleibenden drei Gene nacheinander in pMGX- yfg1,2 kloniert, um pMGX- yfg1-5 zu erzeugen (siehe Schritt 5). Dieses Plasmid wurde dann in BL21- (λDE3) transformiert, und die Expression der Proteine wurde in einer Mittel-Log-Phase-Kultur durch Zugabe von IPTG induziert. Fig. 7 ist ein Western-Blot von Zelllysaten und bestätigt, daß alle fünf Proteine aus einem einzigen Plasmid, das mehrere Gene in einem einzigen Operon enthält, exprimiert werden können. Die Expression von pMGX- yfg1 (mit 1 Gen),PMGX- yfg1,2 (mit 2 Genen) und pMGX- yfg1-5 (mit 5 Genen) werden gezeigt.
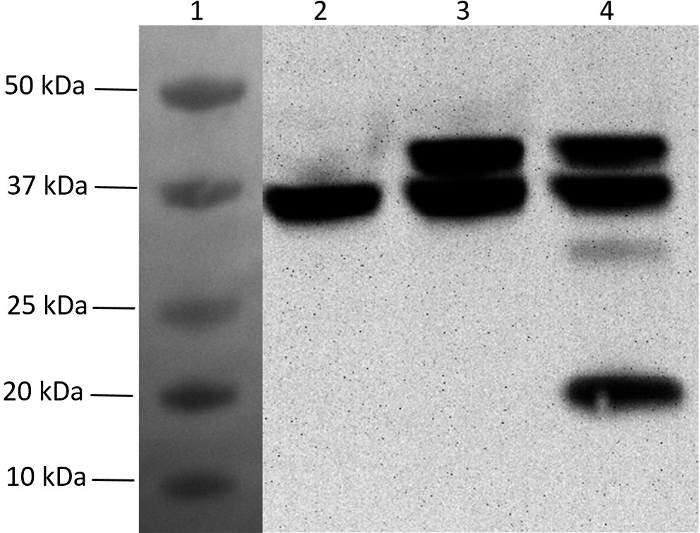
Abbildung 7: Western-Blot-Analyse der Multi-Gen-Expression mit dem pMGX-System. Die Einarbeitung von entweder N- oder C-terminalen Hexahistidin-Markierungen ermöglichte die Western-Blot-Detektion der Proteinexpression. Die Proben wurden auf vorgegossenen 4-20% Gradienten-stammfreien Acrylamidgelen (200 V, 35 min, 10 cm x 8,5 cm) und anschließend einem Transfer (40 V, 90 min) auf eine Nitrozellulosemembran (0,45 μm, 10 cm x 7 cm) durchgeführt. HRP-konjugierter Anti-His-monoklonaler Antikörper, der keinen sekundären Antikörper erfordert, wurde verwendet. Blockierungs-, Transfer- und Antikörper-Verdünnungspuffer wurden, wie vom Antikörperhersteller empfohlen, hergestellt. Die Detektion wurde mit dem Western Chemiluminescent HRP-Substrat nach dem Hersteller durchgeführtTruktionen Die resultierende Membran wurde mit einem Imager ohne Filter sichtbar gemacht. Spur 1, zweifarbiger Standard auf die Membran übertragen (nicht chemilumineszierend); Spur 2, His6pMGX- yfg1 ; Spur 3, His6pMGX- yfg1,2 ; Spur 4, His6pMGX- yfg1-5. Anmerkung: Das von Yfg5 hergestellte Protein ist gleich groß wie das von yfg1 produzierte Protein (beide sind 36 kDa) und können nicht auf dem Gel abgetrennt werden. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Ergänzende Zahlen: Klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Koexpression mehrerer Gene ist immer wichtiger, insbesondere bei der Charakterisierung und Rekonstitution komplexer, multigener Stoffwechselwege 3 , 4 , 5 . Das pMGX-System macht die Multigene-Koexpression in der E. coli- Routine 6 , 7 , 8 und ist für verschiedene Forscher zugänglich. In dieser Studie wurden fünf Proteine von Interesse gezeigt, die gleichzeitig aus einem einzigen Plasmidsystem unter Verwendung von pMGX-A hergestellt wurden. Viele Co-Expression-Systeme, die derzeit verfügbar sind, erlauben nur die Insertion von zwei Genen, die Erzeugung von bicistronischen Plasmiden wie pET-Duet-Vektoren 12 . Wenn man die Zugabe von mehr Genen erfordert, sind mehrere bikistronische Vektoren mit unterschiedlichen selektierbaren Markern erforderlich. Im Gegensatz zu anderen multigenen Expressionssystemen ermöglicht das pMGX-System das einfache KlonenVon mehreren Genen in ein Plasmid mit der Fähigkeit, Restriktionsstellen ohne die Notwendigkeit verschiedener Donorplasmide oder zur Entfernung mehrerer Restriktionsenzymstellen aus den Genen 13 , 14 wiederzuverwenden. Kritische Schritte in diesem Protokoll umfassen Gendesign, Verhinderung der Hintergrundrezirkularisierung von einzeln verdauten pMGX-Vektoren und Bildschirme der polycistronischen Vektoren für die korrekte Orientierung des insertierten Gens.
Bei der Planung der Klonierungsstrategie zur Konstruktion eines polycistronischen Systems ist es wichtig, sicherzustellen, dass die interessierenden Gene keine Restriktionsstellen enthalten, die für die nachgeschaltete Klonierung erforderlich sind. Insbesondere umfassen diese XbaI und AvrII, die für die Insertion der zweiten und nachfolgenden Gene in den pMGX-Vektor mit dem ersten Gen von Interesse (Schritte 3 und 4) erforderlich sind. Darüber hinaus ist es wichtig, Restriktionsstellen zu eliminieren, die für die anfängliche Klonierung der g verwendet werdenInteresse an pMGX (Schritt 2). Typischerweise schließen diese NdeI oder NheI am 5'-Ende des Gens und BamHI oder EcoRI am 3'-Ende des Gens ein, obwohl andere Restriktionsstellen auch für diesen Schritt verwendet werden können, falls erforderlich. Die Eliminierung von extra-, unerwünschten Restriktionsstellen kann leicht bei der Gensynthesestufe durchgeführt werden, oder im Falle von aus anderen Quellen klonierten Genen auch durch Nukleotidsubstitutionen über ortsgerichtete Mutagenese 20 .
Die Addition von nachfolgenden interessierenden Genen in pMGX, die das erste Gen von Interesse enthält, erfordert eine Linearisierung von pMGX- yfg1 mit AvrII (Schritt 3.1, man beachte, dass es auch mit XbaI linearisiert werden kann). Dieser linearisierte Vektor wird während der Einfügung des zweiten Gens von Interesse (Schritt 3.5) schnell religieren, was zu einer extrem hohen Anzahl von Hintergrundklonen führt. Um dies zu vermeiden, ist es wesentlich, den linearisierten Vektor mit einer Phosphatase zu behandeln, um die 5 to zu dephosphorylieren2; Endungen des linearisierten vektors Typischerweise wird CIP verwendet, obwohl andere verfügbar sind. Eine Optimierung sowohl der Einheiten der Phosphatase als auch der Inkubationszeit kann notwendig sein, um die Ligationen der nachfolgenden Gene von Interesse in den linearisierten Vektor zu optimieren.
Schließlich wird bei der Erzeugung eines polycistronischen Systems das Gen von Interesse zunächst durch Verdau mit XbaI und AvrII ausgeschnitten, wodurch komplementäre zusammenhängende Enden an den 5'- und 3'-Enden des Gens entstehen (Schritte 3.2 und 4.2). Dieser Einsatz kann in den linearisierten Vektor in Vorwärts- oder Rückwärtsrichtung ligiert werden. Da alle zusammenhängenden Enden identisch sind, wird durch das komplementäre Glühen der zusammenhängenden Enden keine Richtcharakteristik erzwungen. Es ist daher notwendig, die resultierenden Klone für die korrekte Insertionsrichtung des Gens von Interesse zu screenen. Dies kann leicht durch Screening von Klonen mit einer der Restriktionsstellen erfolgen, die verwendet werden, um das ursprüngliche Gen von Interesse in pMGX zu klonieren (Schritt 2). TypischerweiseEcoRI wird verwendet, da die Restriktionsstelle am 3'-Ende des Gens liegt, und es ist daher angebracht, für die Direktionalität zu screenen, wie in Fig. 6 gezeigt . In den meisten Fällen haben 50% der Klone, die das Gen von Interesse enthalten, das Gen in der korrekten Orientierung und 50% haben das Gen in der entgegengesetzten Orientierung.
In seltenen (~ 10% der Ligationen) können alle Klone aus einer bestimmten Ligation das Gen von Interesse in nur eine Richtung eingefügt haben. Dieses Ergebnis scheint sequenzabhängig zu sein, da es in diesen spezifischen Fällen sehr reproduzierbar ist. Wenn dies geschieht, und das Gen von Interesse kontinuierlich in die umgekehrte Orientierung einfügt, kann der gewünschte Vektor gewöhnlich durch Umschalten der Richtung der Klonierung zugegriffen werden. Beispielsweise kann yfg1 anstelle der Klonierung von yfg2 in die AvrII-Stelle von pMGX- yfg1 in die XbaI-Stelle von pMGX- yfg2 kloniert werden . Beachten Sie, dass dies den identischen endgültigen Vektor gibtSystem. Diese Flexibilität, die den Zugriff auf das gleiche System aus verschiedenen Klonierungsstrategien ermöglicht, ist sehr vorteilhaft.
Eine wichtige Einschränkung dieser Methodik ist, dass die am 3'-Ende des polycistronischen Transkripts codierten Proteine typischerweise auf niedrigeren Ebenen exprimiert werden als die am 5'-Ende des Transkripts codierten Proteine, und dieser Effekt ist stärker ausgeprägt, je länger das Transkript ist . Dies bedeutet, dass das Ändern der Reihenfolge der Gene, die für das synthetische Operon interessant sind, die relativen Expressionsniveaus der Proteine beeinflussen kann, die sie codieren, was einen Mechanismus zur Feinabstimmung der relativen Expressionsniveaus bereitstellt.
Zusammenfassend stellt das pMGX-System eine zuverlässige Methode für die Co-Expression von mehreren Proteinen aus einem einzigen Plasmid in E. coli zur Verfügung , die für eine Vielzahl von synthetischen biologischen Anwendungen und für die biochemische Wegcharakterisierung verwendet werden können.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde vom Naturwissenschaften und Ingenieurforschungsrat von Kanada unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Referenzen
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten