Method Article
Système d'expression multigène médié par l'ARN T7 à ADN polymère induible, pMGX
Dans cet article
Résumé
Cette étude décrit des méthodes pour la co-expression médiée par T7 de plusieurs gènes à partir d'un seul plasmide dans Escherichia coli en utilisant le système de plasmide pMGX.
Résumé
La co-expression de protéines multiples est de plus en plus essentielle pour la biologie synthétique, l'étude des complexes protéines-protéines, et la caractérisation et l'atténuation des voies de biosynthèse. Dans ce manuscrit, on décrit l'utilisation d'un système hautement efficace pour la construction d'opérons synthétiques multigènes sous le contrôle d'une ARN polymérase T7 inducible. Ce système permet d'exprimer plusieurs gènes simultanément à partir d'un plasmide. Ici, un ensemble de quatre vecteurs apparentés, pMGX-A, pMGX-hisA, pMGX-K et pMGX-hisK, avec le marqueur sélectionnable par résistance à l'ampicilline ou à la kanamycine (A et K) et possédant ou n'ayant pas d'hexahistidine N-terminale Tag (son) est divulgué. Des protocoles détaillés pour la construction d'opérons synthétiques utilisant ce système vectoriel sont fournis avec les données correspondantes, montrant qu'un système à base de pMGX contenant cinq gènes peut être facilement construit et utilisé pour produire les cinq protéines codées dans Escherichia coli . Ce systèmeEm et le protocole permet aux chercheurs d'exprimer systématiquement des modules et des chemins multi-composants complexes dans E. coli .
Introduction
La co-expression de multiples protéines est de plus en plus essentielle, en particulier dans les applications de biologie synthétique, où plusieurs modules fonctionnels doivent être exprimés 1 ; Dans l'étude des complexes protéine-protéine, où l'expression et la fonction nécessitent souvent une co-expression 2 , 3 ; Et dans la caractérisation et l'exploitation des voies de biosynthèse, où chaque gène dans la voie doit être exprimé 4 , 5 , 6 , 7 , 8 . Un certain nombre de systèmes ont été développés pour la co-expression, en particulier dans l'organisme hôte Escherichia coli , le cheval de travail pour l'expression de protéines recombinantes en laboratoire 9 . Par exemple, de multiples plasmides avec différents marqueurs sélectionnables peuvent être utilisés pour exprimer des protéines individuelles en utilisant une multitude d'ex différents Vecteurs de pression 10 , 11 . Des systèmes plasmidiques simples pour une expression de protéines multiples ont utilisé soit des promoteurs multiples pour contrôler l'expression de chaque gène 10 , 12 ; Opérons synthétiques, où plusieurs gènes sont codés sur une seule transcription 2 , 13 ; Ou, dans certains cas, un seul gène codant pour un polypeptide qui est finalement transformé de façon protéolytique, donnant les protéines désirées d'intérêt 14 .

Figure 1: flux de travail pMGX montrant la construction d'un vecteur polycistronique. Le système pMGX offre une stratégie flexible et facile à utiliser pour la construction d'opérons synthétiques sous le contrôle d'un promoteur T7 inducible.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Cliquez ici pour voir une version plus grande de cette figure.
Dans ce manuscrit, on décrit l'utilisation d'un système très efficace pour la construction d'opérons synthétiques multigène sous le contrôle d'une ARN polymérase T7 induite ( figure 1 ). Ce système permet d'exprimer plusieurs gènes simultanément à partir d'un plasmide. Il est basé sur un système plasmide, initialement appelé pKH22, qui a été utilisé avec succès pour un certain nombre d'applications différentes 6 , 7 , 8 . Ici, ce jeu de plasmide est développé pour inclure quatre vecteurs apparentés: pMGX-A, un vecteur d'expression dépourvu de marqueurs C ou N-terminaux et avec le marqueur de résistance à l'ampicilline; PMGX-hisA, un vecteur d'expression codant une étiquette d'hexahistidine N-terminal et avec le marqueur de résistance à l'ampicilline; PMGX-K, un vecteur d'expression lAvoir des marqueurs C ou N-terminaux et avec le marqueur de résistance à la kanamycine; Et pMGX-hisK, un vecteur d'expression codant une étiquette d'hexahistidine N-terminal et avec le marqueur de résistance à la kanamycine. Dans cette étude, le procédé de génération d'un vecteur polycistronique contenant cinq gènes utilisant le système pMGX, en particulier pMGX-A, est démontré avec la production réussie de chaque protéine individuelle dans Escherichia coli .
Protocole
1. Obtention de gènes d'intérêt
- Concevoir des gènes synthétiques.
- Optimiser une séquence de gènes pour l'expression de E. coli .
- Supprimez tous les sites de restriction problématiques de la séquence (AvrII, NdeI, EcoRI et XbaI).
- Incorporer des sites de restriction pour le clonage; Un site 5'-NdeI et un site 3'-EcoRI sont recommandés. D'autres sites peuvent être utilisés, si nécessaire; Se référer à la région de multiclonation du plasmide sélectionné ( Figure S1 -S4 ). Si vous le souhaitez, incluez une étiquette de codage 5 'ou 3' pour la détection Western Blot.
- Commandez commercialement le gène conçu.
- L'un ou l'autre clone clone le gène en un vecteur émoussé en utilisant un kit de clonage brutal, conformément aux instructions du fabricant; Concevoir des amorces pour amplifier le gène (puis passer à l'étape 1.2.2); Ou ajouter des extrémités 5 'et 3' supplémentaires pour le clonage direct dans un plasmide pMGX et passer à l'étape 2. Ici, leLes résultats représentatifs proviennent du clonage brutal des gènes d'intérêt.
- Amplifier le gène souhaité (à partir d'un gène synthétique conçu et optimisé ou d'un ADN modèle contenant le gène désiré) 15 .
- Concevoir des amorces avec des sites de restriction pour le clonage; Un site 5'-NdeI et un site 3'-EcoRI sont recommandés. D'autres sites peuvent être utilisés, si nécessaire; Se référer à la région de multiclonation du plasmide sélectionné ( Figure S1 -S4 ). Si vous le souhaitez, incluez une étiquette de codage 5 'ou 3' pour la détection Western Blot.
- La PCR amplifie le gène désiré 15 .
- Analyser la PCR par électrophorèse sur gel d'agarose 16 .
- S'il existe une amplification non spécifique, nettoyer le gène amplifié par extraction au gel. Sinon, utilisez un kit de nettoyage enzymatique.
- Quantifier l'ADN en utilisant un spectrophotomètre en vérifiant l'absorbantE à 260 nm; Blanc avec tampon d'élution 17 .
2. Clonage de gènes d'intérêt dans un vecteur de système d'expression multigène, pMGX 18
- La restriction digère le gène obtenu d'intérêt et le vecteur désiré, pMGX, avec NdeI et EcoRI.
REMARQUE: Si une grande quantité de plasmides recirculés est obtenue, la réaction NdeI doit se poursuivre pendant 1 h avant d'ajouter EcoRI.- Utilisez une réaction de digestion de 40 μl contenant 0,5-1,5 μg d'ADN et 1 μL de chaque enzyme avec 4 μL du tampon 10x approprié.
- Pour un résumé NdeI et EcoRI, utilisez un tampon EcoRI et commencez par ajouter de l'endonucléase NdeI. Digest pendant 1 heure à 37 ° C. Ensuite, ajoutez l'endonucléase EcoRI, et permettez au produit digeste de continuer une heure supplémentaire. NdeI est sensible aux clivages proches de la fin de l'ADN, de sorte que la digestion initiale par EcoRI peut empêcher une digestion efficace par NdeI.
- ElectrOmettre la digestion de restriction sur gel d'agarose (pour un gène de 1 kb, utiliser un gel d'agarose à 0,7% à 110 V pendant 55 minutes, pour sélectionner le pourcentage de gel d'agarose pour différentes tailles de gènes, se référer à la référence 16 .
- Insertion d'accise et bandes de vecteurs en utilisant un scalpel propre ou une lame de rasoir et placez le segment de gel excisé dans un tube de 1,5 ml.
- Extraire l'ADN du gel d'agarose en utilisant un kit d'extraction de gel selon le protocole du fabricant.
- Relier le gène d'intérêt dans le vecteur pMGX en utilisant un rapport insert-vecteur 3: 1; Mettre en place une ligature négative de pMGX digérée sans l'insert.
- Mettre en place une réaction de ligature de 20 μl contenant 1 μl d'ADN ligase T4, 0,15-0,5 μg d'ADN vecteur (~ 5 μL) et une quantité appropriée d'insert basée sur un rapport insert-vecteur 3: 1 et la taille de Le gène étant inséré; Les backbones pMGX sont entre 5 311 et 5 504 pb. Inclure une réaction de contrôle négatif qui contient tout buT le gène étant inséré. Assurez-vous que la quantité d'ADN vectoriel est équivalente dans la ligature de contrôle négative et les réactions de ligature vectorielle plus de ligature.
- Les réactions de ligature de transformation dans des cellules de E. coli chimiquement compétentes XL1-Blue et des plasmides ligaturés pMGX- yfg1 (contenant le gène d'intérêt 1), pMGX- yfg2 (contenant le gène d'intérêt 2) ... pMGX- yfgn (contenant le gène d'intérêt n). Utiliser une technique aseptique (à l'intérieur d'une armoire à biosécurité ou sous une flamme).
- Décongeler 100 μl d'aliquotes de XL1-Blue E. coli chimiquement compétent sur de la glace pendant 5 min, puis ajouter 5 μL des réactions de ligature. Incuber pendant 30 minutes sur de la glace.
- Choc de chaleur les cellules pendant 45 s dans un bain d'eau maintenu à 42 ° C, puis ajouter 200 μL de LB froide. Incuber les sur glace pendant 2 min.
- Agiter les cellules à 37 ° C, 220 tr / min pendant 1 h, puis étaler la plaque 100 μL sur une plaque de LB-agar contenant un approMarqueur sélectionnable approprié (ampicilline ou kanamycine).
- Écran des clones pour les transformants positifs.
- Comparez le nombre de colonies sur les plaques de contrôle négatif et de ligature. Un rapport de comptage de colonie supérieur à 1: 2 est désiré. S'il y a un grand nombre de colonies sur la plaque témoin négative, revenez à l'étape 2.1 et passez en revue la note.
- Sélectionnez 4-8 colonies individuelles (selon le témoin négatif: rapport de ligature) de chaque réaction de ligature des différents gènes insérés pour le dépistage (1 n) et inoculez un LB de 4 mL et une culture de nuit appropriée d'antibiotiques / colonie individuelle. Cultiver des cultures pendant la nuit avec agitation à 37 ° C et 220 tr / min.
- Isoler l'ADN plasmidique en utilisant un kit d'isolation d'ADN plasmidique selon le protocole du fabricant.
- Mettre en place une réaction de digestion de 20 μL NdeI + EcoRI contenant 150-500 ng d'ADN et 1 μL de chaque enzyme avec 2 μL du tampon 10x approprié. Dans leEst un cas, ajoute NdeI et EcoRI en même temps que le gène d'intérêt sera entre les sites de restriction. Un contrôle négatif avec le vecteur pMGX vide est recommandé. Digérer pendant 2 h à 37 ° C dans un bain-marie.
3. Insertion du gène 2 dans le vecteur pMGX contenant le gène 1, pMGX- yfg1
- Restriction digest pMGX- yfg1 avec AvrII et traiter avec la phosphatase intestinale du veau (CIP).
- Utilisez une réaction de digestion de 40 μl contenant 0,5-1,5 μg d'ADN vectoriel (~ 5-10 μl d'ADN isolé) et 1 μL d'AvrII avec 4 μL du tampon 10x approprié. Digérer pendant 1,5 h à 37 ° C puis ajouter 1,5 μL de CIP. Laissez-le à 37 ° C pendant 30 minutes supplémentaires.
- Restriction digest pMGX- yfg2 avec AvrII et XbaI pour libérer le gène d'intérêt.
- Utilisez une réaction de digestion de 40 μl contenant 0,5-1,5 μg d'ADN et 1 μ; L de chaque enzyme avec 4 μL du tampon 10x approprié. Digest pendant 2 h à 37 ° C.
- La restriction d'électrophorèse digère un gel d'agarose (0,7%) approprié et accise les bandes d'insertion et de vecteur à l'aide d'un scalpel propre / lame de rasoir (voir les étapes 2.2-2.3).
- Extraire l'ADN en utilisant un kit d'extraction de gel selon le protocole du fabricant et quantifier l'ADN 17 .
- Le gène de ligature 2 en pMGX- yfg1 en utilisant un rapport insert-vecteur 3: 1. Configurez une ligature négative de pMGX- yfg1 digérée sans l'insert supplémentaire. Configurez comme ci-dessus à l'étape 2.5.
- Transforme 5 μL des réactions de ligature en cellules E. coli chimiquement compétentes XL1-Blue, plasmides ligaturés pMGX- yfg1,2 (contenant le gène d'intérêt 1 et 2) et le contrôle négatif de pMGX- yfg1 , comme on le voit à l'étape 2.6.
- Comparez le nombre de colonies sur les plaques de contrôle négatif et de ligature. Un rapport de comptage de colonie gUn compteur de 1: 2 est désiré. S'il y a un grand nombre de colonies sur la plaque témoin négative, revenez à l'étape 3.1 et révisez le traitement CIP.
- Sélectionnez 4-8 colonies individuelles (selon le témoin négatif: rapport de ligature) de la réaction de ligature et inocule 4 mL de LB plus l'antibiotique approprié par colonie individuelle et passez une nuit à 37 ° C et 220 tr / min.
- Isoler l'ADN plasmidique en utilisant un kit d'isolation d'ADN plasmidique selon le protocole du fabricant et quantifier l'ADN 17 .
- Taper l'insertion effective du second gène en effectuant un résumé de restriction de pMGX- yfg1,2 avec EcoRI.
- Utilisez une réaction de digestion de 20 μl contenant 150 à 500 ng d'ADN et 1 μl d'enzyme EcoRI avec 2 μL de tampon EcoRI 10x. Digest pendant 2 h à 37 ° C.
- Électrophorèse la digestion de restriction sur un pourcentage approprié de gel d'agarose; Recherchez un groupe qui correspondS à la taille du gène 2 (voir l'étape 2.2). Un gène peut s'insérer dans le vecteur dans l'orientation inverse non souhaitée.
4. Ajout d'un troisième gène dans le vecteur pMGX contenant les gènes 1 et 2, pMGX- yfg1,2
- Restriction digest pMGX- yfg1,2 avec AvrII et traiter avec CIP, comme indiqué à l'étape 3.1.
- Restriction digest pMGX- yfg3 avec AvrII et XbaI, comme on le voit à l'étape 3.2.
- Électrophorèse les digestion de restriction sur un pourcentage approprié de gel d'agarose et d'insertion et de bandes vectorielles à l'aide d'un scalpel propre / lame de rasoir; Se reporter aux étapes 2.2-2.3.
- Extraire l'ADN du gel d'agarose en utilisant un kit d'extraction de gel et quantifier l'ADN 17 .
- Le gène de ligature 3 en pMGX- yfg1,2 en utilisant un rapport d'insert-vecteur 3: 1 et a mis en place une ligation négative de pMGX- yfg1,2 digérée sans insertion supplémentaire, comme on le voit à l'étape 3.5.
- Transformer 5 μL des réactions de ligature en XLDes cellules de E. coli chimiquement compétentes de 1-Blue, le plasmide ligaturé pMGX- yfg1,2,3 (contenant les gènes d'intérêt 1, 2 et 3) et le contrôle négatif de pMGX- yfg1,2 , comme on le voit à l'étape 2.6.
- Comparez le nombre de colonies sur les plaques de contrôle négatif et de ligature. S'il y a un grand nombre de colonies sur la plaque témoin négative, revenez à l'étape 4.1 et révisez le traitement CIP.
- Sélectionnez 4-8 colonies individuelles (selon le témoin négatif: rapport de ligature) de la réaction de ligature et inocule 4 mL de LB plus l'antibiotique approprié par colonie individuelle; Croître de nuit à 37 ° C et 220 tr / min.
- Isoler l'ADN plasmidique en utilisant un kit d'isolation d'ADN plasmidique selon le protocole du fabricant.
- Taper l'insertion efficace du troisième gène en effectuant un résumé de restriction de pMGX- yfg1,2,3 avec EcoRI, comme on le voit à l'étape 3.10.
- Électrophorèse la digestion de restriction sur un pourcentage approprié de gel d'agarose; RegardezPour les bandes qui correspondent aux tailles du gène 2 et du gène 3 (voir l'étape 2.2). Remarque: le gène 3 peut être inséré dans le vecteur dans l'orientation inverse non souhaitée. Si les gènes 2 et 3 ont la même taille, un autre site de restriction de restriction approprié doit être sélectionné pour le dépistage.
REMARQUE: Répétez le cas échéant pour chaque nouveau gène.
5. Produire des protéines d'intérêt à l'aide d'un système d'expression multigène et évaluer la production par Western Blot
- Transformer le clone positif contenant tous les gènes d'intérêt dans la production de protéines chimiquement compétente E. coli , tel que BL21- (λDE3).
- Décongeler des aliquotes de 100 μL de E. coli BL21- (λDE3) chimiquement compétent sur de la glace pendant 5 min, puis ajouter 1 μl de l'ADN plasmidique clone positif; Incuber pendant 30 minutes sur de la glace.
- Choc thermique des cellules pendant 45 s dans un bain d'eau maintenu à 42 ° C, puis ajouter 200 μL de LB froide. Incuber sur de la glace pendant 2 min.
- Agiter les cellules à 37 ° C et 220 tr / min pendant 1 h, puis étaler la plaque de 100 μL sur une plaque de LB-agar contenant le marqueur sélectionnable approprié (soit l'ampicilline soit la kanamycine).
- Exprimer la protéine par l'induction d'isopropyl-β-D-1-thiogalactopyranoside (IPTG).
- Sélectionnez une colonie isolée de la plaque de transformation B21- (λDE3) et inoculez 4 mL de LB plus l'antibiotique approprié; Croître toute la nuit, tremper à 37 ° C et 220 tr / min.
- Inoculer une LB de 100 ml plus la culture d'antibiotiques appropriée en utilisant 1 mL de culture pendant la nuit.
- Cultiver à 37 ° C avec agitation à 220 tr / min jusqu'à une DO 600 de 0,6.
- Induire la culture avec 100 μM d'IPTG et pousser pendant 15 h à 25 ° C et 220 tr / min.
- Retirer 1 mL de la culture et centrifuger à 13 000 xg pendant 1 min; Jeter le surnageant.
- Lyse les cellules en utilisant une solution de lyse selon les instructions du fabricantEt préformer une Western blot du lysat cellulaire soluble pour déterminer si toutes les protéines ont été produites avec succès 19 .
Résultats
Dans cette étude, l'objectif était de coexprimer cinq protéines à partir d'un seul plasmide. Les fragments de gènes synthétiques optimisés à cinq codons codant pour les marqueurs hexahistidine N- ou C-terminaux ont été achetés dans le commerce. Les gènes synthétiques ont été amplifiés par PCR et clonés individuellement dans un vecteur brut de PCR et séquencés. Pour générer le plasmide polycistronique, les cinq gènes d'intérêt ont d'abord été clonés dans un plasmide pMGX approprié, pMGX-A. La figure 2 montre PCRBlunt- yfg1 et PCRBlunt- yfg2 en plus du pMGX-A digéré avec NdeI + EcoRI (voir étape 2). Pour cloner les deux premiers gènes dans pMGX-A, le plasmide a été digéré avec NdeI + EcoRI ( Figure 2 ). Lors du clonage des gènes d'intérêt dans pMGX-A, les plasmides nouvellement construits, désignés par pMGX- yfg1 et pMGX- yfg2 , ont été digérés avec AvrII + XbaI pour confirmer que des clones réussis ont été obtenus,Montré dans les bandes de 1,1 kb et 1,3 kb obtenues à la figure 3 .
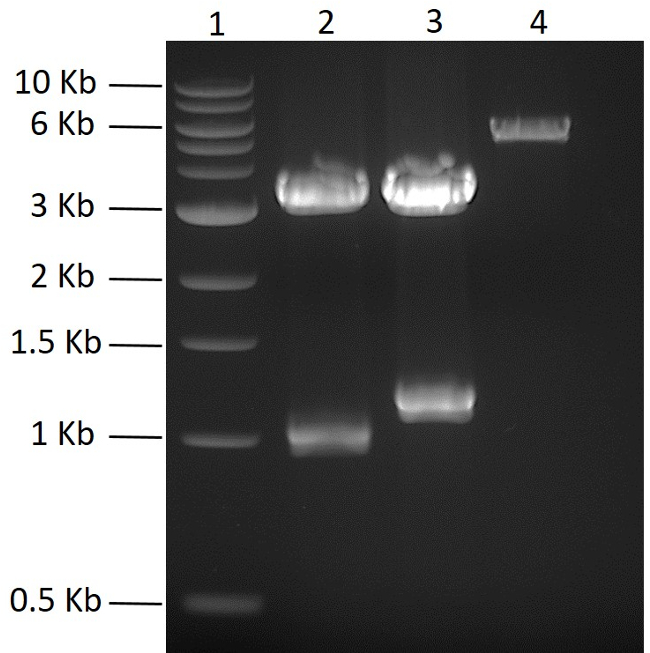
Figure 2: Clonage de plasmides pMGX. Les résultats attendus des plasmides concomitants par PCR contenant yfg1 et yfg2 après digestion avec NdeI + EcoRI sont indiqués sur un gel d'agarose à 0,7% (110 V, 55 min). Voie 1, échelle d'ADN de 1 kb; Voie 2, PCRBlunt- yfg1 ; Voie 3, PCRBlunt- yfg2 ; Lane 4, contrôle pMGX-A. Cliquez ici pour voir une version plus grande de ce chiffre.
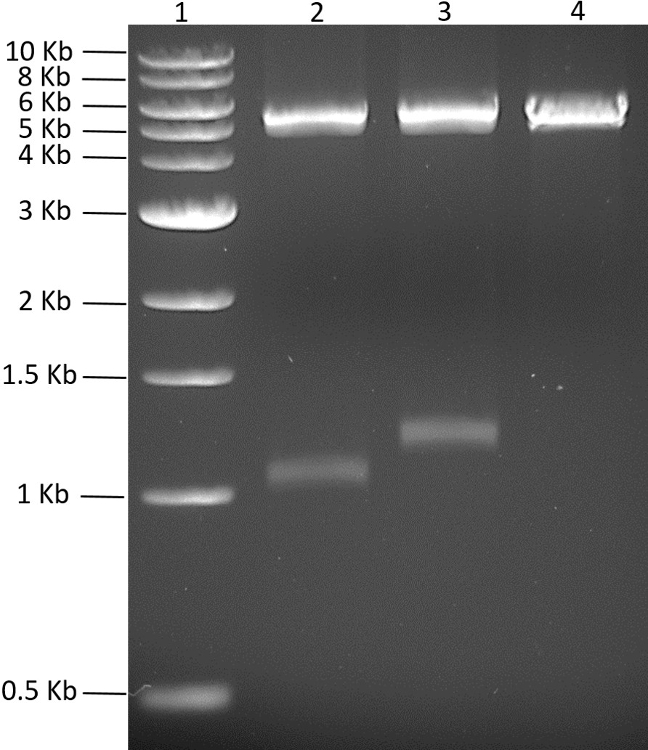
Figure 3: Dépistage de pMGX- yfg1 et pMGX- yfg2. Le gel d'agarose à 0,7% (110 V, 55 min) montre Les deux bandes générées par pMGX- yfg1 et pMGX- yfg2 après digestion avec AvrII + XbaI. Voie 1, échelle d'ADN de 1 kb; Voie 2, pMGX- yfg1 ; Voie 3, pMGX- yfg2 ; Lane 4, contrôle pMGX-A. Cliquez ici pour voir une version plus grande de ce chiffre.
Avec les deux gènes dans le vecteur pMGX approprié, le plasmide polycistronique a été construit. Pour générer le plasmide pMGX- yfg1,2 , pMGX- yfg1 a été digéré avec AvrII, comme le montre la figure 4 . PMGX- yfg2 a été digéré avec AvrII + XbaI et le gène de 1,3 kb contenant le fragment a été ligaturé dans le site AvrII de pMGX- yfg1 linéarisé, générant pMGX- yfg1,2 . La digestion de pMGX- yfg1,2 avec EcoRI a confirmé le clonage réussi du plasmide souhaité et est représentée en> Figure 5.

Figure 4: Génération du plasmide bicistronique pMGX-yfg1,2. Les résultats d'électrophorèse sur gel d'agarose à 0,7% (110 V, 55 minutes) de pMGX- yfg1 digérés avec AvrII et pMGX- yfg2 digérés avec AvrII + XbaI. Voie 1, échelle d'ADN de 1 kb; Voie 2, pMGX- yfg1 digérée avec AvrII; Voie 3, pMGX- yfg2 digéré avec AvrII + XbaI; Lane 4, contrôle pMGX-A. Cliquez ici pour voir une version plus grande de ce chiffre.
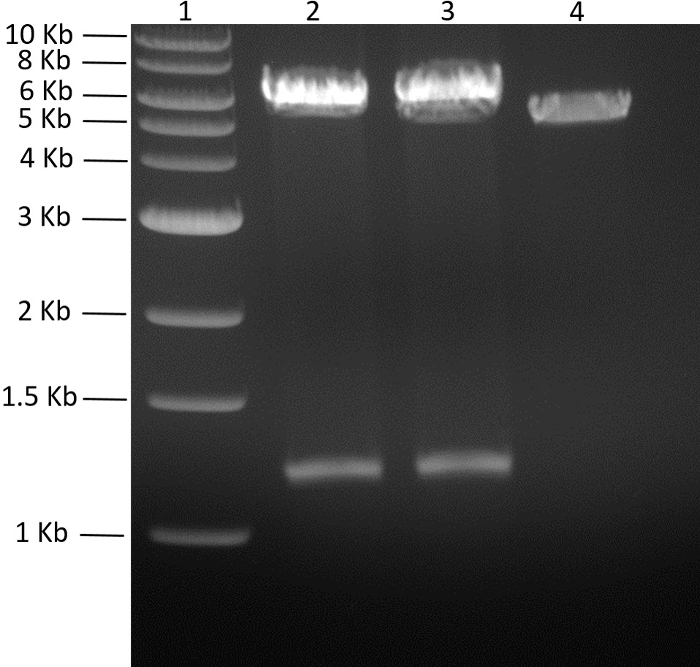
Figure 5: Dépistage des clones pMGX- yfg1,2 . Le gel d'agarose à 0,7% résultant (110 V, 55 min) de pMGX- yfDes clones de g1,2 digérés avec EcoRI sont présentés. Les clones réussis généreront deux bandes, une pour le gène inséré yfg2 et l'autre correspondant au squelette + yfg1 ( pMGX-yfg1 ). Voie 1, échelle d'ADN de 1 kb; Voie 2, pMGX- yfg1,2 digérée avec EcoRI, clone 1; Piste 3, pMGX- yfg1,2 digérée avec EcoRI, clone 2; Lane 4, contrôle pMGX-A. Cliquez ici pour voir une version plus grande de ce chiffre.
Lors de la génération d'un plasmide polycistronique contenant deux gènes ou plus, on peut rencontrer une insertion inversée indésirable du gène clone dans le site AvrII. La figure 6 est un gel mettant en évidence la différence entre les résultats d'un plasmide digéré avec un gène inséré dans l'orientation correcte par opposition à un gène qui est inséré dans l'orientation inversée. Si le gène estLigaturé et clone dans l'orientation indésirable, le site EcoRI à la fin du gène sera inséré juste à côté du site EcoRI du gène précédent. Cela donnerait une taille de fragment qui est pratiquement indétectable par électrophorèse sur gel d'ADN (moins de 50 pb). De plus, le dernier gène inséré apparaîtra dans l'ossature, ce qui est facilement identifiable en raison de la taille du squelette plus grande que prévu. La différence dans la taille de l'os à partir de la taille attendue indiquera la taille du dernier gène inséré.

Figure 6: Dépistage des clones pMGX- yfg1-5 . On montre un gel d'agarose à 1,3% (110 V, 65 min) représentant les résultats de pMGX- yfg1-5 digérés avec EcoRI. Les clones réussis (ligne 2) généreront une bande correspondant au dernier gène inséré ( yfg5 dans ce cas, 1,1 kb). LanE 1, 1-kb échelle d'ADN; Voie 2, clone positif de pMGX- yfg1-5, digéré avec EcoRI; Voie 3, clone négatif de pMGX- yfg1-5, digéré avec EcoRI; Lane 4, pMGX- yfg1-4 de contrôle. Cliquez ici pour voir une version plus grande de ce chiffre.
Pour compléter le vecteur d'expression polycistronique à cinq gènes, les trois gènes restants ont été clones l'un après l'autre dans pMGX- yfg1,2 pour générer pMGX- yfg1-5 (voir l'étape 5). Ce plasmide a ensuite été transformé en BL21- (λDE3), et l'expression des protéines a été induite dans une culture en phase intermédiaire de phase par addition d'IPTG. La figure 7 est un transfert Western de lysats cellulaires, confirmant que les cinq protéines provenant d'un seul plasmide contenant plusieurs gènes dans un seul opéron peuvent être exprimées. L'expression de pMGX- yfg1 (contenant 1 gène),PMGX- yfg1,2 (contenant 2 gènes), et pMGX- yfg1-5 (contenant 5 gènes) sont présentés.
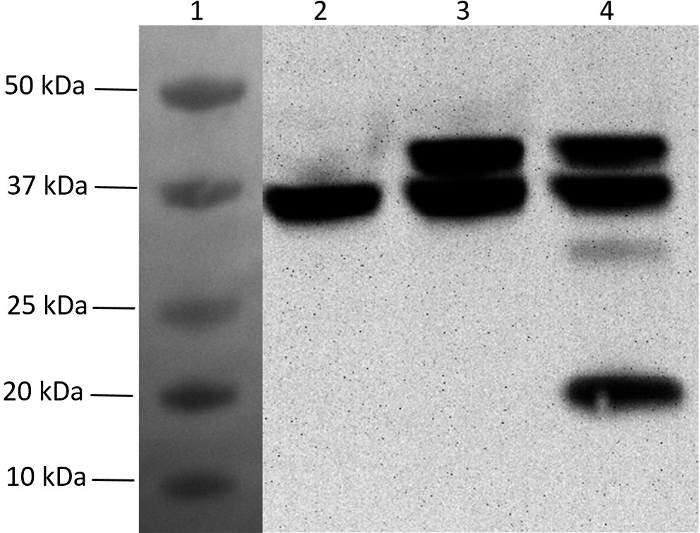
Figure 7: analyse Western Blot de l'expression multigène à l'aide du système pMGX. L'incorporation d'étiquettes d'hexahistidine N- ou C-terminal a permis la détection de Western blot de l'expression de protéines. Les échantillons ont été séparés sur des gels d'acrylamide exempts de déformation à gradient de 4-20% pré-coulé (200 V, 35 min, 10 cm x 8,5 cm), puis un transfert (40 V, 90 min) sur une membrane de nitrocellulose (0,45 μm, 10 cm x 7 cm). L'anticorps monoclonal anti-His conjugué au HRP, qui ne nécessite pas d'anticorps secondaire, a été utilisé. Les tampons de blocage, de transfert et de dilution d'anticorps ont été préparés comme recommandé par le fabricant de l'anticorps. La détection a été réalisée avec un substrat chimique à base de HRP chimiluminescents suite aux inscriptions du fabricantTructions. La membrane résultante a été visualisée à l'aide d'un imageur sans filtre. Voie 1, standard double couleur transféré sur la membrane (non chimioluminescente); Voie 2, His6pMGX- yfg1 ; Voie 3, His6pMGX- yfg1,2 ; Lane 4, His6pMGX- yfg1-5. Note: Les protéines produites par yfg5 ont la même taille que la protéine produite par yfg1 (les deux sont 36 kDa) et ne peuvent être séparées sur le gel. Cliquez ici pour voir une version plus grande de ce chiffre.
Chiffres supplémentaires: cliquez ici pour télécharger ce fichier.
Discussion
La co-expression de multiples gènes est de plus en plus essentielle, en particulier dans la caractérisation et la reconstitution des voies métaboliques multigène complexes 3 , 4 , 5 . Le système pMGX fait une co-expression multigène dans la routine E. coli 6 , 7 , 8 et accessible à divers chercheurs. Dans cette étude, on a montré que cinq protéines d'intérêt étaient produites simultanément à partir d'un seul système de plasmide utilisant pMGX-A. De nombreux systèmes de co-expression actuellement disponibles ne permettent que l'insertion de deux gènes, générant des plasmides bicistroniques tels que les vecteurs pET Duet 12 . Si l'on exige l'addition de plus de gènes, de multiples vecteurs bicistroniques avec différents marqueurs sélectionnables sont nécessaires. Contrairement à d'autres systèmes d'expression multigène, le système pMGX permet le clonage facileDe multiples gènes dans un plasmide, avec la possibilité de réutiliser les sites de restriction sans avoir besoin de différents plasmides donneurs ou pour éliminer les sites d'enzymes de restriction multiples des gènes 13,14 . Les étapes critiques de ce protocole comprennent la conception de gènes, la prévention de la recirculation d'arrière-plan de vecteurs pMGX digérés individuellement et des écrans des vecteurs polycistroniques pour l'orientation correcte du gène inséré.
En planifiant la stratégie de clonage pour construire un système polycistronique, il est essentiel de s'assurer que les gènes d'intérêt ne contiennent pas de sites de restriction requis pour le clonage en aval. En particulier, ceux-ci incluent XbaI et AvrII, qui sont nécessaires pour l'insertion du second et des gènes subséquents dans le vecteur pMGX contenant le premier gène d'intérêt (étapes 3 et 4). En outre, il est essentiel d'éliminer les sites de restriction qui seront utilisés pour le clonage initial du gD'intérêt dans pMGX (étape 2). Typiquement, ceux-ci comprennent NdeI ou NheI à l'extrémité 5 'du gène et BamHI ou EcoRI à l'extrémité 3' du gène, bien que d'autres sites de restriction puissent également être utilisés pour cette étape, si nécessaire. L'élimination des sites de restriction extra indésirables peut être effectuée facilement au stade de la synthèse des gènes ou, dans le cas des gènes clonés provenant d'autres sources, par des substitutions synonymes de nucleotides par mutagenèse dirigée 20 .
L'ajout de gènes d'intérêt ultérieurs dans pMGX contenant le premier gène d'intérêt nécessite une linéarisation de pMGX- yfg1 avec AvrII (étape 3.1, notez qu'il peut également être linéarisé avec XbaI). Ce vecteur linéarisé se religatera rapidement lors de l'insertion du second gène d'intérêt (Étape 3.5), ce qui entraîne un nombre extrêmement élevé de clones d'arrière-plan. Pour éviter cela, il est essentiel de traiter le vecteur linéarisé avec une phosphatase pour déphosphoryler le 5̸2; Extrémités du vecteur linéarisé. Généralement, le CIP est utilisé, bien que d'autres soient disponibles. L'optimisation des deux unités de phosphatase et le temps d'incubation peuvent être nécessaires pour optimiser les ligatures des gènes d'intérêt ultérieurs dans le vecteur linéarisé.
Enfin, lors de la génération d'un système polycistronique, le gène d'intérêt est initialement excisé par la digestion avec XbaI et AvrII, générant des extrémités cohésives complémentaires aux extrémités 5 'et 3' du gène (Etapes 3.2 et 4.2). Cet insert peut être ligaturé dans le vecteur linéarisé dans le sens inverse ou inversé. Comme toutes les extrémités cohésives sont identiques, aucune directionnalité n'est appliquée par un recuit complémentaire des extrémités cohésives. Il est donc nécessaire de filtrer les clones résultants pour la direction d'insertion correcte du gène d'intérêt. Ceci peut être effectué facilement en sélectionnant des clones avec l'un des sites de restriction utilisés pour cloner le gène original d'intérêt dans pMGX (étape 2). Typiquement,EcoRI est utilisé, car le site de restriction est à la fin 3 'du gène, et il est donc approprié d'examiner la directionnalité, comme le montre la figure 6 . Dans la plupart des cas, 50% des clones contenant le gène d'intérêt ont le gène dans l'orientation correcte et 50% ont le gène dans l'orientation opposée.
Peu fréquemment (~ 10% des ligatures), tous les clones provenant d'une ligature particulière peuvent avoir le gène d'intérêt inséré dans une seule direction. Ce résultat semble être dépendant de la séquence, car il est hautement reproductible dans ces cas spécifiques. Si cela se produit, et le gène d'intérêt s'insère continuellement dans l'orientation inverse, on peut habituellement accéder au vecteur souhaité en commutant la direction du clonage. Par exemple, au lieu de cloner yfg2 dans le site AvrII de pMGX- yfg1 , yfg1 peut être clone dans le site XbaI de pMGX- yfg2 . Notez que cela donne le vecteur final identiquesystème. Cette flexibilité supplémentaire permettant l'accès au même système à partir de différentes stratégies de clonage est très avantageuse.
Une limitation importante de cette méthodologie est que les protéines codées à l'extrémité 3 'du transcrit polycistronique sont typiquement exprimées à des niveaux inférieurs à ceux des protéines codées à l'extrémité 5' de la transcription, et cet effet est plus prononcé plus la transcription est longue . Cela signifie que la modification de l'ordre des gènes d'intérêt dans l'opéron synthétique peut avoir une incidence sur les niveaux d'expression relatifs des protéines qu'ils encodent, fournissant un mécanisme pour affiner les niveaux d'expression relative.
En résumé, le système pMGX fournit une méthode fiable pour la co-expression de protéines multiples à partir d'un seul plasmide dans E. coli , qui peut être utilisé pour une variété d'applications de biologie synthétique et pour la caractérisation des voies biochimiques.
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Ce travail a été appuyé par le Conseil de recherches en sciences naturelles et en génie du Canada.
matériels
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Références
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon