Method Article
Identificação de pares Kinase-substrato utilizando High Throughput Screening
Neste Artigo
Resumo
Protein phosphorylation is a central feature of how cells interpret and respond to information in their extracellular milieu. Here, we present a high throughput screening protocol using kinases purified from mammalian cells to rapidly identify kinases that phosphorylate a substrate(s) of interest.
Resumo
Nós desenvolvemos uma plataforma de rastreio para identificar proteínas quinases específicas de substratos fosforilados humanos que podem ser utilizados para elucidar novas vias de transdução de sinal. A nossa abordagem apresenta a utilização de uma biblioteca de proteínas quinases humano marcado com GST purificada e um substrato da proteína recombinante de interesse. Usámos esta tecnologia para identificar MAP / microtúbulo-regulação afinidade quinase 2 (MARK2) como a quinase para um local regulada por glucose, em reguladas pela CREB transcricional coativador 2 (CRTC2), uma proteína necessária para a proliferação das células beta, assim como a família de tirosina-quinases Axl como reguladores de metástases de células por fosforilação da proteína adaptadora ELMO. Descrevemos essa tecnologia e discutir como ela pode ajudar a estabelecer um mapa abrangente de como as células respondem a estímulos ambientais.
Introdução
Proteína modificações pós-traducionais (PTMs) são essenciais para a comunicação intracelular. Talvez o melhor estudada de todas as PTMs fosforilação é catalisada por proteínas quinases, que regulam uma miríade de funções de proteínas, incluindo a sua actividade bioquímica, localização subcelular, conformação, e a estabilidade. A identificação de sítios de fosforilação de proteínas alvo pode ser realizada através de mapeamento triptico fosfopéptido ou por técnicas-padrão de proteómica agora utilizando amostras enriquecidas para péptidos fosforilados 1,2. Enquanto três quartos do proteoma expressas são esperados para ser fosforilada 3 e um identificado 200.000 locais de fosforilação 5, com estimativas de até 1 milhão de 6, muitos deles não têm atribuído biologia, via de sinalização, ou proteína quinase.
Enquanto identificação de locais fosforilados é relativamente simples, uma comparativamente maior desafio éidentificar a cinase cognato (s) que tem como alvo estes locais, um processo que nos referimos como mapeamento de quinase: substrato pares. Várias abordagens para a identificação de quinase: substrato pares foram descritas, começando com uma quinase de interesse e procurando os seus substratos, ou a partir de um substrato de interesse e a tentativa de encontrar um modificador quinase 7-11 experimentalmente ou computacionalmente 12. Para identificar quinases para um substrato fosforilado conhecido, bioinformática pode ser usado para identificar proteínas que contêm uma sequência conservada curto de aminoácidos que flanqueiam o resíduo fosforilado (o local de consenso), bem como a identificação de quinases que formam um complexo com o substrato precipitável. No entanto, estas abordagens são demorados e muitas vezes não cumprem com sucesso.
Nós desenvolvemos uma abordagem funcional sistemático para identificar rapidamente quinases que fosforilam pode um determinado substrato 13. O ensaio tela produz excelente específicodade, com a seleção muito clara para os potenciais quinases cognatas. Dada a centralidade da fosforilação de sinalização biológica, a tela é útil para a descoberta em praticamente todos os celulares vias de sinalização 14-16. A tela envolve a realização de um ensaio de quinase em larga escala com uma biblioteca de proteínas quinases humanos. As cinases foram marcados com glutationa bacteriano S-transferase (GST) de proteínas e são purificados a partir de extractos de células de mamíferos, o que significa que as enzimas recombinantes - ao contrário do que as preparadas a partir de bactérias - são gerados na presença das proteínas quinases a montante muitas vezes necessários para a enzimas recombinantes para ter actividade in vitro. Com efeito, enquanto que a actividade da cinase de serina, treonina e tirosina necessária para a activação a jusante de quinase estão presentes na levedura 10, o genoma da levedura codifica 122 cinases de proteína, indicando que o kinome de mamífero, com mais de 500 genes 17, tornou-se significativamente mais complexa, a fim de regulate os processos originais para os organismos de ordem mais elevada. Além disso, o efeito de diferentes estímulos relevantes para a biologia celular e doença humana (tal como pequenas moléculas, factores de crescimento, hormonas, etc.) podem ser usados para modular a actividade de quinase 14,15 num contexto apropriado.
Protocolo
1. Preparação dos reagentes, placas, e Células
- Adicione 500 ml de tampão de lise: Tris 25 mM, pH 7,5, NaCl 150 mM, NaF 50 mM, 0,5 mM EDTA pH 8,0, 0,5% Triton X-100, 5 mM beta-glicerofosfato, 5% de glicerol. Armazenar a 4 ° C. Imediatamente antes da utilização, adicionar 1 mM de ditiotreitol (DTT), 1 mM de fluoreto de fenilmetil sulfonil (PMSF), e 1 mM de vanadato de sódio. Após este passo, PMSF não é necessária em qualquer tampão de lavagem.
- Adicione 20 ml de cinase 10X: Tris Buffer de 200 mM de pH 7,5, 50 mM beta-glicerofosfato (FW 216), vanadato de sódio a 2 mM. Da alíquota em tubos de 1 ml e armazenar a -20 ° C. Imediatamente antes de usar, adicionar DTT 5 mM.
- Adicione 20 mL de tampão H-ATP 10x: 300 uM de adenosina trifosfato (ATP), 66 mM de MgCl2, 33 mM de MnCl 2. alíquota para tubos de 1 ml e armazenar a -20 ° C.
- Adicione 100 ml de 2x dodecil sulfato de sódio (SDS) de tampão de lise: 1,5 g de base Tris, 20 ml de glicerol, 30 ml de H 2 O. Dissolver e ajustar o pH para 6,8 com HCl. Adicionar 40ml de SDS a 10%, ajustar o volume para 100 ml. Adicionar 25 mg de azul de bromofenol.
- Spot 4 ul de 25 ng / uL do plasmídeo de expressão de mamífero que codificam uma cinase de GST-14 por poço em placas de conjuntos de 96 poços e as placas de etiqueta em conformidade. Seal e congelar placas a -20 ° C até à sua utilização.
- 1-2 dias antes da transfecção, as células HEK293T cultura em meio de Eagle modificado por Dulbecco completo (DMEM) + 10% de soro fetal de bovino (FBS) e antibióticos numa incubadora a 37 ° C com CO 2 suplementado (final de 5%). Passage com tripsina e não permitir que o estoque para se tornar> 80% confluentes durante a expansão. Um mínimo de 6 x 10 7 células são necessárias para a tela (aproximadamente 3 x 15 cm pratos de células HEK293T em 80% de confluência).
2. Transfecção
Nota: Consulte a Figura 1 para um fluxograma de protocolo inteiro.
- Se placas contendo plasmídeos quinase foram congelados, descongele em temperatura ambiente umaND, centrifugar a 1900 xg durante 3 minutos para recolher a humidade no fundo dos poços.
- Misturar 8,6 ml de meio de soro reduzido (por exemplo, meio OPTI-MEM), com 312,7 ul de reagente de transfecção à base de lípidos. Deixe descansar por 5 min.
- Adicionar 10 ul de meio de soro reduzido para cada cavidade utilizando um dispensador de líquido automático equipado com um pequeno volume de cassete.
- Adiciona-se 10 uL por poço de reagente de forma a mistura de soro / transfecção reduzida a partir do passo 2.2 utilizando um dispensador de líquido automático equipado com um pequeno volume de cassete. Deixe descansar por 20 a 45 min.
- Ressuspender as células 293T em 7,5 x 10 5 células / ml em 80 ml de DMEM completo. Adicionar 100 ul de suspensão de células (isto é, 7,5 x 10 4 células) por poço usando um distribuidor de líquido automático equipado com uma cassete de volume padrão.
- Verifique poços sob o microscópio para distribuição uniforme de células e retornar à incubadora durante 24 h.
3. pulldown de GST-cinase
- Faça fresh: 4 mM de solução pervanadato por mistura de 60 ul de 0,2 M de vanadato de sódio, com 540 mL de H 2 O. Numa segunda mistura tubo de 2,7 mL de peróxido de 30% e 1,4 ml de PBS. Adicionar as duas soluções em conjunto e deixe descansar por 15 min antes do uso.
- Usando uma pipeta de canais múltiplos (não utilizar um distribuidor de líquido automatizado uma vez que cria demasiada turbulência no poço) dispensar 2 ul de 0,25 M de CaCl2 em cada poço, seguido de 2,5 mL do pervanadato preparado no passo 3.1. Incubar cada placa a 37 ° C durante 10 min e, em seguida, colocar em gelo.
- Prepare 35 ml de tampão de lise adicionando DTT, PMSF, e vanadato de sódio, tal como indicado na Seção 1 (Preparação de Reagentes).
- Mantendo as placas em gelo, remover o meio de cada poço utilizando um aspirador de vácuo. Imediatamente adicionar 50 ul / poço de tampão de lise arrefecido em gelo utilizando um dispensador de líquido automático equipado com uma cassete de padrão. Deixe descansar 30 min em gelo para lise (opcional: pode parar por aqui, se necessário, por meio de selagem platE e armazenar a -80 ° C. Para continuar, placas de descongelamento no gelo).
- Chapas giram a 1900 xg durante 3 min a 4 ° C.
- Raspe células de cada poço utilizando uma pipeta de canais múltiplos e para transferir todos os conteúdos apropriadamente marcada placas de 96 poços de fundo em V. Chapas giram a 1900 xg durante 10 min a 4 ° C.
- Durante a rotação, encher placas revestidas com glutationa (um para cada placa de 96 poços) com 100 ul / poço de tampão de lise arrefecido em gelo (sem adição de PMSF) como uma lavagem. Mantenha as placas no gelo.
- Remover placas de fundo em V da centrífuga. Um de cada vez, inverter as placas de glutationa sobre uma pia para esvaziar a solução de lise e blot em uma toalha de papel. Transferir tampão de lise a partir das placas de fundo em V para as placas de glutationa por inclinação da placa e utilizando uma pipeta de canais múltiplos, tendo o cuidado de não perturbar o sedimento no fundo. Cubra as placas e deixar em gelo durante mínimo 2 horas de se ligar.
- Perto do fim do passo de ligação 2 h, preparar uma estação de trabalho radioactividade, assegurando tele precauções de segurança necessárias estão no lugar para o trabalho radioativo. Definir forno de hibridação a 30 ° C.
- Preparar 200 ml de tampão de lise por adição de DTT e vanadato de sódio tal como indicado no ponto 1. PMSF não é necessária nesta fase.
- Inverter as placas de glutationa sobre uma pia para sacudir a tampão de lise e blot em uma toalha de papel. Lavar os poços 3x com 100 ul de tampão de lise (sem adição de PMSF). Não deixe que os poços sentar seco - mantê-los na lavagem até que esteja pronto para prosseguir.
- Preparar 55 ml de tampão de quinase 1x (KB) diluindo o estoque de 10x e adição de DTT, tal como indicado no ponto 1. Lavar as placas uma vez com 50 ul de 1x KB utilizando um dispensador de líquido automático equipado com uma cassete de volume padrão. Deixar 1x KB em poços até uma solução pronta é:
- Prepare a Solução A pela adição de 500 a 530 ug de o substrato de interesse, 500 ug de proteína básica de mielina (MBP), 2,65 ml de 10x KB, 13,25 ul de DTT 1 M, e H2O até 15,9 ml.
- Um de cada vez, inverter as placas sobre uma pia para remover 1x KB enxaguamento, seque sobre toalha de papel, e imediatamente adicionar 30 ul de uma solução utilizando um dispensador de líquido automático equipado com um pequeno volume de cassete. Mantenha as placas no gelo.
- Preparar a solução B na área de trabalho a radioactividade por adição de 2,5 ml de 10x H-ATP, 500 uCi de 32 P ATP gama-, e H 2 O a 10 ml.
- Adicionar 20 ul de solução B por poço utilizando uma pipeta de repetidor que auxilia na mistura, devido à força de ejecção. Cobrir e incubar em 30 ° C forno de hibridação durante 30 min.
- Após 30 minutos, a transferência de volta para as placas de gelo. Adicionar 50 ul de tampão de lise 2x SDS para cada poço utilizando uma pipeta de canais múltiplos. Pode prosseguir neste ponto para a próxima etapa, ou selar as placas com folha de alumínio e armazenar a -20 ° C até conveniente.
4. corrida, coloração e géis de secagem
Nota: Todo o trabalho deve ser realizado em uma área DESIGNAted para a radioactividade.
- Ligue forno de hibridação e ajustado para 85 ° C. Placas de descongelamento em temperatura ambiente. Uma vez forno atingiu a temperatura, placas de transferência para forno e incubar por 10 min para desnaturar amostras.
- Carga 26 poços géis pré-moldados com 15 ul de cada reacção utilizando uma pipeta de canais múltiplos para preencher vários poços de uma só vez. Cuidados devem ser tomados para que todas as dicas alinhar com as correspondentes bem antes de adicionar amostras. Execute gel em 150 V. Não deixe que o corante de rastreio (linha azul) funcionar fora do fundo do gel como esta contém a ATP não incorporado.
- Desmontar os géis e cortou a ATP não incorporado (linha azul), utilizando um bisturi ou borda reta como ele vai expor demais os filmes. Coloque os gels em recipientes rotulados e cubra com Coomassie mancha para 15 min.
- Remover a mancha de Coomassie, brevemente enxaguar os géis com água, e adicionar destain solução. Destain os géis até que as proteínas são claramente visíveis. Uma banda para MBP e uma banda para o substrato deveser visível para cada amostra.
- Para secar o gel:
- Corte uma folha grande de papel de filtro e coloque-o no secador.
- Molhe uma folha de celofane em água destilada até que esteja lisa e sem rugas, e colocá-lo no topo do papel.
- Coloque os géis em cima da folha de celofane, fazendo uma nota da ordem de géis. Molhar uma segunda folha de celofane e colocar na parte superior dos géis.
- Rolar para fora todas as bolhas (um rolo de vedação placa funciona bem para isso) para uma superfície uniforme agradável. Fechar a aba, ligar o vácuo, e secar o gel durante 3 horas a 80 ° C.
- Uma vez géis estão secos, expô-los a filme XAR com um ecrã de intensificar o sinal. Enrole a fita com saran wrap ou um saco plástico e feche com fita adesiva para impedir a entrada de geada. Armazenar a cassete a -80 ° C durante a noite.
5. Desenvolver XAR Films
- No dia seguinte, remove cassete do congelador e deixar descongelar à temperatura ambiente. Desenvolver o filme emuma câmara escura usando um processador de filme de acordo com as instruções do fabricante.
Nota: Examinar os filmes para provas de pares quinase-substrato. Uma segunda exposição mais longa pode também ser útil para detectar eventos de fosforilação fracas.
Resultados
Os resultados representativos de uma tela são mostrados na Figura 2. 180 quinases foram rastreadas utilizando um substrato de péptido marcado com GST correspondente aos aa 268-283 da CRTC2, bem como a proteína básica clássico ensaio de quinase substrato de mielina (MBP). Apenas duas cinases, MARK2 e o MARK3-quinase altamente relacionados com o peptídeo fosforilado CRTC2. MBP é incluído como um controlo interno em todos os ensaios, uma vez que contém muitos resíduos fosforilável e roda a 18 kDa, em direcção ao fundo do gel. Isto permite uma interpretação da especificidade: alguns quinase vai robustamente fosforilar um substrato e MBP. De notar aqui é que os poços que contêm GST isolada (ou seja, os controlos sem quinase) sempre purificar alguma actividade de quinase endógena, portanto, existe sempre a fosforilação de fundo no ensaio. Enquanto isto não exclui que a fosforilação do substrato é real, ela sugere que in vitro no ajuste da quinase pode ser menos selectiva.É particularmente informativo para incluir vários substratos de diferentes MW para tirar conclusões acerca da especificidade quinase-substrato.
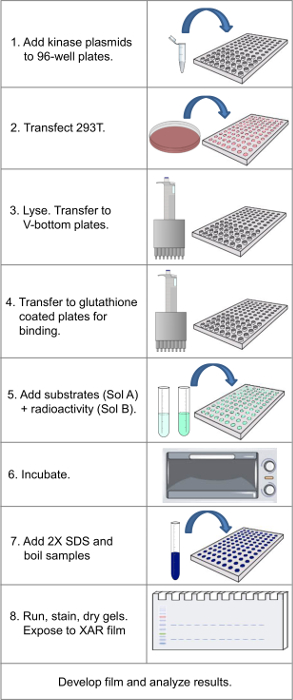
Figura 1. Fluxograma mostrando etapas fundamentais no protocolo. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2. Exemplo da tela resultado. Autorradiografias de uma tela de uma versão da biblioteca (180 proteínas cinases humanos) realizada utilizando substrato GST-péptido correspondente aos aa 268-283 de rato CRTC2 (reproduzido de Jansson et ai., 2008). MBP, proteína básica de mielina é indicado. O elevado grau de sseleção ubstrate por quinases relacionadas é uma característica da tela. Cada uma das pistas representa os produtos de reacção de um ensaio de cinase distinta. MARK3 (Gel 1) e MARK2 (Gel 16), os únicos quinases para fosforilar TORC2 268-283 peptídeo, são indicadas. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
Uma vez que as publicações originais que descrevem a abordagem 14,15, a biblioteca original de 180 GST-quinases foi ampliada para 420 membros, ou ~ 80% do kinome proteína humana. Com a biblioteca ampliada, o protocolo conforme descrito leva 4-5 dias e, em seguida, 1-4 dias para desenvolver filmes (como necessário), o que pode ser encurtado através da utilização de imagiologia com fósforo e o aumento do sinal digital. Há várias etapas-chave em que os cuidados devem ser tomados (veja a Figura 1 para uma descrição de protocolo). Primeiro, é a saúde da população de células (por micoplasma livre, nunca permitiu atingir a confluência, durante a expansão, e tratou-se com tripsina a cada passagem) durante a fase de expansão do lote e nas placas de 96 poços no momento da transfecção. As células devem ser uniformemente semeado, e 80% confluente quando transfecção tem lugar (Passo 1.5).
Em segundo lugar, é ideal durante a transfecção para utilizar um distribuidor de líquido automatizado para minimizar o volume transferros de er, limitando assim o coeficiente de variação ("cvs") entre os poços. Para pequenos volumes, ajustando o dispensador com um volume menor perda de limites cassete de reagente, devido à redução dos volumes mortos e aumentos de dispensação de precisão. O passo de tratamento com pervanadato de sódio, um inibidor de tirosina-fosfatase panela, é usado como um estímulo para aumentar geral estado de fosforilação de cinases alvo. Inclusão de cálcio nesta etapa aumenta a adesão de células-matriz, evitando a perda de células devido a arredondamentos provocada por tratamento pervanadato prolongado. Esta etapa deve ser feito com a estrita observância do tempo de incubação de 10 minutos.
Em terceiro lugar, para o ensaio de quinase em si, há várias considerações ao seleccionar o substrato recombinante: péptidos, domínios de proteína, ou proteínas intactas tão grandes como 120 kDa podem ser utilizados. Se um local de fosforilação específica é conhecida, um substrato péptido é a abordagem mais direta, mas precisa ser suficientemente grande para detectarnum gel de SDS-PAGE. Assim, os substratos de péptidos podem ser fundidos com GST e purificado a partir de bactérias para dar um peso molecular de> 25. Proteínas maiores têm a vantagem de que eles são provavelmente dobrada e os seus resíduos fosforilável exposta como estariam na célula, mas possuem a desvantagem de que eles vão incluir resíduos adicionais, que podem ser fosforilados e dão sinal no ensaio. A nossa preferência é para executar uma tela utilizando um substrato péptido constituído por 15-20 aminoácidos que rodeiam um resíduo que é conhecido por ser fosforilada in vivo e é conhecido por regular um evento biológico, já que isto torna a validação das cinases candidatos a partir da tela, tanto in vitro e in vivo muito mais rápido.
Por último, de preferência a quantidade de abordagens de proteína ou superior a 1 ug por poço; Isto, obviamente, varia de acordo com o PM do substrato. Menos de proteína por poço pode produzir sucessos significativos, mas a "mais é melhor" regra aplica-se à medida que aumenta sinal: noiSE. Padrão, géis de gradiente não são recomendados como géis de gradiente de rachar muito frequentemente durante a secagem. Após a secagem dos geles, a detecção de um sinal radioactivo sobre o fundo com um contador Geiger dá uma excelente indicação de sucesso do ensaio.
À medida que a tela é in vitro e níveis adicionais de complexidade existir in vivo, um candidato quinase (s) para um dado substrato deve ser validado em células. Especificamente, o ecrã pode identificar um membro da família que tem a capacidade para fosforilar a bioquímica substrato in vitro, mas não é expresso no mesmo tipo de célula ou no mesmo compartimento subcelular como substrato. Por exemplo, enquanto MARK2 e MARK3 eram ambos os acessos em uma tela para cinase que podem fosforilar CRTC2 em Ser275, única MARK2 formado um complexo com o substrato 14 em células. Segue-se uma série de experiências adicionais que podem ser utilizados para confirmar a relevância fisiológica de um candidato quinase. Abetor, os desvios de mobilidade do substrato em SDS-PAGE a seguir a co-expressão da quinase candidato pode ser utilizado como um controlo de confirmação. Controles apropriados, tais cotransfecção de uma quinase cataliticamente inativa pode confirmar especificidade. Em segundo lugar, o substrato pode ser imunoprecipitada a partir de extractos celulares que foram incubadas com 32 P-ortofosfato para confirmar a incorporação de fosfato no substrato na presença do tipo selvagem e não uma quinase cataliticamente inactivo. Em terceiro lugar, um anticorpo pode ser gerado fosfoespec�ico contra a sequência phosphoacceptor (se conhecido) e utilizado para confirmar um aumento na fosforilação do substrato quando a cinase é sobre-expresso. Um ecrã secundário utilizando um substrato mutantes portadoras de um resíduo não-fosforilável no local alvo pode confirmar a especificidade antes da geração de um phosphoantibody, um controlo particularmente importante com substratos e maior domínio de proteína de comprimento completo. A inibição farmacológica abordagens para inibir um candidata quinase também pode ser usado, mas o cuidado deve ser exercido como inibição das cinases relacionadas é uma realidade subvalorizado. Por último, mediada por ARNi silenciamento do candidato quinase (s) nas células seguidos e western blotting para monitorizar a perda de local alvo fosforilação com um anticorpo fosfoespec�ico pode ser realizada.
Divulgações
The authors have nothing to disclose.
Agradecimentos
Este trabalho foi apoiado pela concessão NSERC 386634. Gostaríamos de agradecer aos membros do Laboratório Screaton para discussões úteis.
Materiais
| Name | Company | Catalog Number | Comments |
| Lysis buffer | Made in house | See Protocol step 1.1 | |
| 10x kinase buffer | Made in house | See Protocol step 1.2 | |
| 10x M-ATP | Made in house | See Protocol step 1.3 | |
| Human kinase plasmids | Orfeome, Invitrogen, Origene | GST-tagged in house | |
| 96 well plates | Fisher Scientific | CS003595 | |
| 293T cells | ATCC | CRL-11268 | |
| DMEM | Fisher Scientific | SH3002201 | supplement with 100 U/ml penicillin, 100 μg/ml streptomycin, 10% fetal calf serum. |
| CO2 incubator | Sanyo | MCO-17AIC | |
| 15 cm cell culture dishes | Fisher Scientific | 877224 | |
| Reduced serum medium | Invitrogen | 22600-050 | |
| Lipid-based transfection reagent | Invitrogen | 11668-019 | |
| Automated liquid dispenser | Thermo Scientific | 5840300 | |
| Small cassette attachment | Thermo Scientific | 24073295 | |
| Standard cassette attachment | Thermo Scientific | 14072670 | |
| 4 mM pervanadate | Made in house | See Protocol step 3.1 | |
| 0.25 M CaCl2 | Made in house | ||
| Multichannel pipette (20-200 μl) | Labnet | p4812-200 | |
| Multichannel pipette (1-10 μl) | Thermo Scientific | 4661040 | |
| V-bottom 6-well plates | Evergreen Scientific | 290-8116-01V | |
| Glutathione coated 96-well plates | Fisher Scientific | PI-15240 | |
| Hybridization oven | Biostad | 350355 | |
| GST tagged substrate | Made in house | ||
| Myelin Basic Protein (MBP) | Sigma | M1891 | |
| Repeater pipette (1 ml) | Eppendorf | 22266209 | |
| 32P gamma-ATP | Perkin Elmer | BLU502Z500UC | |
| 2x SDS lysis buffer (100 ml) | Made in house | See Protocol step 1.4 | |
| 26-well precast TGX gels | BioRad | 567-1045 | gel percentage required is dependent on the molecular weight of the substrate of interest |
| Coomassie stain | Made in house | 0.1% Coomassie R250, 10% acetic acid, 40% methanol | |
| Coomassie destain | Made in house | 10% acetic acid, 20% methanol | |
| Labeled gel containers | Made in house | Used plastic lids from empty tip boxes, just big enough to contain one gel | |
| Whatman filter paper | Fisher Scientific | 57144 | |
| Cellophane sheets (2) | BioRad | 165-0963 | |
| Gel dryer | Labconco | 4330150 | |
| Double emulsion autoradiography film | VWR | IB1651454 | |
| Film cassette | Fisher Scientific | FBAC-1417 | |
| Intensifying screen | Fisher Scientific | FBIS-1417 | |
| Plate sealing rubber roller | Sigma | R1275 |
Referências
- Meisenhelder, J., Hunter, T., van der Geer, P. Phosphopeptide mapping and identification of phosphorylation sites. Curr Protoc Mol Biol. 18, Unit 18 19 (2001).
- Doll, S., Burlingame, A. L. Mass spectrometry-based detection and assignment of protein posttranslational modifications. ACS chem. 10, 63-71 (2015).
- Sharma, K., et al. Ultradeep human phosphoproteome reveals a distinct regulatory nature of Tyr and Ser/Thr-based signaling. Cell rep. 8, 1583-1594 (2014).
- Cohen, P. The regulation of protein function by multisite phosphorylation--a 25 year update. Trends Biochem Sci. 25, 596-601 (2000).
- Walsh, C. T. . Posttranslation Modification of Proteins: Expanding Nature's Inventory. , (2006).
- Boersema, P. J., et al. In-depth qualitative and quantitative profiling of tyrosine phosphorylation using a combination of phosphopeptide immunoaffinity purification and stable isotope dimethyl labeling. Mol Cell Proteomics. 9, 84-99 (2010).
- Hutti, J. E., et al. A rapid method for determining protein kinase phosphorylation specificity. Nat Methods. 1, 27-29 (2004).
- Johnson, S. A., Hunter, T. Kinomics: methods for deciphering the kinome. Nat Methods. 2, 17-25 (2005).
- Pawson, T., Nash, P. Assembly of cell regulatory systems through protein interaction domains. Science. 300, 445-452 (2003).
- Zhu, H., et al. Analysis of yeast protein kinases using protein chips. Nat Genet. 26, 283-289 (2000).
- Shah, K., Shokat, K. M. A chemical genetic approach for the identification of direct substrates of protein kinases. Methods Mol Biol. 233, 253-271 (2003).
- Zou, L., et al. PKIS: computational identification of protein kinases for experimentally discovered protein phosphorylation sites. BMC bioinform. 14, 247 (2013).
- Varjosalo, M., et al. Application of active and kinase-deficient kinome collection for identification of kinases regulating hedgehog signaling. Cell. 133, 537-548 (2008).
- Jansson, D., et al. Glucose controls CREB activity in islet cells via regulated phosphorylation of TORC2. Proc Natl Acad Sci U S A. 105, 10161-10166 (2008).
- Fu, A., Screaton, R. A. Using kinomics to delineate signaling pathways: control of CRTC2/TORC2 by the AMPK family. Cell Cycle. 7, 3823-3828 (2008).
- Abu-Thuraia, A., et al. Axl phosphorylates elmo scaffold proteins to promote rac activation and cell invasion. Mol Cell Biol. 35, 76-87 (2015).
- Manning, G., Whyte, D. B., Martinez, R., Hunter, T., Sudarsanam, S. The protein kinase complement of the human genome. Science. 298, 1912-1934 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados