Method Article
Identifikation von Kinase-Substrat-Pairs Mit High Throughput Screening
In diesem Artikel
Zusammenfassung
Protein phosphorylation is a central feature of how cells interpret and respond to information in their extracellular milieu. Here, we present a high throughput screening protocol using kinases purified from mammalian cells to rapidly identify kinases that phosphorylate a substrate(s) of interest.
Zusammenfassung
Wir haben ein Screening-Plattform zu dedizierten menschliche Proteinkinasen phosphoryliert für Substrate, die verwendet werden können, um neue Signaltransduktionswege aufzuklären identifizieren. Unsere Vorgehensweise bietet die Verwendung einer Bibliothek von gereinigtem GST-markiertes humanes Protein-Kinasen und ein rekombinantes Protein Substrat von Interesse. Wir haben diese Technologie ein Protein für die Beta-Zellproliferation erforderlich ist, sowie die verwendet werden, um MAP / Mikrotubuli-Affinität regulierenden Kinase 2 (Modell 2) als Kinase für eine Glucose-regulierten Website auf CREB-Regulated Transcriptional Koaktivator 2 (CRTC2) zu identifizieren, Axl Familie von Tyrosinkinasen als Regulatoren der Zellmetastasen durch Phosphorylierung des Adapterprotein ELMO. Wir beschreiben diese Technologie und diskutieren, wie sie helfen können, eine umfassende Karte, wie Zellen auf Reize aus der Umwelt zu etablieren.
Einleitung
Protein posttranslationale Modifikationen (PTMs) essentiell für die intrazelluläre Kommunikation. Vielleicht ist die am besten untersuchten aller PTMs ist Phosphorylierung durch Proteinkinasen, die eine Vielzahl von Proteinfunktionen, einschließlich ihrer biochemischen Aktivität, subzelluläre Lokalisierung, Konformation und Stabilität regulieren katalysiert. Die Identifizierung von Phosphorylierungsstellen auf Zielproteine können durch tryptische Phosphopeptid Kartierung oder jetzt Standard Proteomics Techniken unter Verwendung von Proben für die phosphorylierten Peptide 1,2 angereichert erreicht werden. Während drei Viertel der zum Ausdruck Proteom sollen phosphoryliert 3 und eine identifizierte 200.000 Phosphorylierungsstellen 5, wobei die Schätzungen bis zu 1 Mio. 6, viele von ihnen haben keine zugewiesenen Biologie, Signalwegs oder Proteinkinase.
Während Identifizierung der phosphorylierten Websites ist relativ einfach, eine vergleichsweise größere Herausforderung ist es,identifizieren die verwandten Kinase (n), dass diese Seiten abzielt, ein Prozess, wir als Mapping-Kinase: Substrat-Paare. Mehrere Ansätze zum Identifizieren von Kinase: Substrat-Paare beschrieben wurden, entweder ausgehend von einer Kinase von Interesse und der nach seiner Substrate oder beginnend mit einem Substrat von Interesse und zu versuchen, eine modifizierende Kinase experimentell 7-11 oder rechnerisch 12 finden. Kinasen für eine bekannte phosphorylierte Substrat identifizieren kann Bioinformatik verwendet, um Proteine, die eine kurze konservierte Aminosäuresequenz flankieren den phosphorylierten Rest (der Konsensusstelle), sowie die Identifizierung Kinasen, die einen präzipitierbaren Komplex mit dem Substrat enthalten identifizieren. Allerdings sind diese Ansätze zeitaufwendig und oft nicht von Erfolg gekrönt.
Wir entwickelten eine systematische funktionalen Ansatz schnell zu identifizieren Kinasen, die eine gegebene Substrat 13 phosphorylieren kann. Der Bildschirm Assay produziert hervorragende spezifischekeit, mit sehr klaren Auswahl für eine mögliche verwandte Kinasen. Angesichts der Zentralität der Phosphorylierung an biologischen Signal, der Bildschirm ist nützlich für die Entdeckung in nahezu allen Zellsignalwege 14-16. Der Bildschirm beinhaltet die Durchführung einer großen Kinase-Assay mit einer Bibliothek von menschlichen Proteinkinasen. Die Kinasen wurden mit bakteriellen Glutathion-S-Transferase (GST) -Protein, markiert worden und werden aus Säugetierzellextrakten gereinigt ist, was bedeutet, daß die rekombinanten Enzyme - im Gegensatz zu den aus Bakterien hergestellt, - in Gegenwart der vorgeschalteten Proteinkinasen oft die erforderliche erzeugten rekombinante Enzyme Aktivität haben in vitro. Tatsächlich, während Serin, Threonin und Tyrosin Kinaseaktivität für nachgeschaltete Kinaseaktivierung in Hefe 10 vorhanden sind, codiert das Hefegenom 122 Proteinkinasen, die anzeigt, dass die Säuger Kinoms, mit mehr als 500 Gene 17 ist wesentlich komplexer zu werden, um Regulate die Prozesse eindeutig höherer Ordnung Organismen. Darüber hinaus kann die Wirkung der relevanten Zellbiologie und menschlichen Erkrankungen (wie beispielsweise kleinen Molekülen, Wachstumsfaktoren, Hormone, etc.) verschiedene Reize zu 14,15 modulate Kinase-Aktivität in einem geeigneten Kontext eingesetzt werden.
Protokoll
1. Vorbereitung der Reagenzien, Platten und Zellen
- Bilden 500 ml Lyse-Puffer: 25 mM Tris pH 7,5, 150 mM NaCl, 50 mM NaF, 0,5 mM EDTA pH 8,0, 0,5% Triton X-100, 5 mM beta-Glycerophosphat, 5% Glycerin. Lagerung bei 4 ° C. Unmittelbar vor der Verwendung, fügen Sie 1 mM Dithiothreitol (DTT), 1 mM Phenylmethylsulfonylfluorid (PMSF) und 1 mM Natriumvanadat. Nach diesem Schritt wird PMSF in keiner Spülpuffer erforderlich.
- Machen 20 ml 10x Kinase-Puffer: 200 mM Tris pH 7,5, 50 mM beta-Glycerophosphat (FW 216), 2 mM Natriumvanadat. Aliquot in 1-ml-Röhrchen und bei -20 ° C. Unmittelbar vor der Verwendung, fügen Sie 5 mM DTT.
- Machen Sie 20 ml 10x M-ATP-Puffer: 300 um Adenosintriphosphat (ATP), 66 mM MgCl 2, 33 mM MnCl 2 Aliquot in 1-ml-Röhrchen und bei -20 ° C.
- Auf 100 ml 2x Natriumdodecylsulfat (SDS) Lysepuffer: 1,5 g Tris-Base, 20 ml Glycerin, 30 ml H 2 O. Auflösen und pH-Wert auf 6,8 mit HCl. In 40ml 10% SDS, die Lautstärke auf 100 ml. In 25 mg Bromphenolblau.
- Spot 4 ul 25 ng / ul Säuger-Expressionsplasmid ein GST-Kinase 14 pro in Sätze von 96-Well-Platten und Kennzeichenschilder entsprechend kodiert gut. Dichtung und gefrier Platten bei -20 ° C bis zur Verwendung.
- 1-2 Tage vor der Transfektion, Kultur HEK293T-Zellen in Komplett Dulbeccos modifiziertem Eagle-Medium (DMEM) + 10% fötalem Rinderserum (FBS) und Antibiotika in einem 37 ° C-Inkubator mit CO 2 (5% final) ergänzt. Passage mit Trypsin und erlauben nicht die Aktie um> 80% konfluent während der Expansion zu werden. Für den Bildschirm (ca. 3 x 15 cm-Schalen aus HEK293T-Zellen bei 80% Konfluenz) sind mindestens 6 x 10 7 Zellen erforderlich.
2. Transfektion
Hinweis: Siehe Abbildung 1 für ein Flussdiagramm des gesamten Protokoll.
- Wenn Platten, die Kinase-Plasmide wurden eingefroren, bei Raumtemperatur auftauen einnd Zentrifuge bei 1.900 × g für 3 min, vorhandene Feuchtigkeit an der Unterseite der Vertiefungen sammeln.
- Mischungs 8,6 ml reduzierte Serummedium (zB OPTI-MEM) mit 312,7 & mgr; l Lipid basierende Transfektionsreagenz. Lassen Sie sitzen für 5 min.
- Zugabe von 10 & mgr; l der reduzierten Serummedium zu jeder Vertiefung unter Verwendung eines automatisierten Flüssigkeitsabgabevorrichtung mit einem kleinen Volumen Kassette angebracht.
- In 10 ul pro Vertiefung der reduzierten Serum-Medium / Transfektionsreagenz Mischung aus Schritt 2.2 unter Verwendung eines automatisierten Flüssigkeitsspender mit einem kleinen Volumen Kassette ausgestattet. Lassen Sie sich für 20 bis 45 min.
- Resuspendieren 293T-Zellen mit 7,5 × 10 5 Zellen / ml in 80 ml vollständigem DMEM. 100 ul Zellsuspension (dh 7,5 × 10 4 Zellen) pro Vertiefung unter Verwendung eines automatisierten Flüssigkeitsabgabevorrichtung mit einem Standardvolumen Kassette angebracht.
- Überprüfen Brunnen unter dem Mikroskop für eine gleichmäßige Zellverteilung und zum Inkubator für 24 Stunden.
3. GST-Pulldown-Kinase
- Stellen Sie fresh: 4 mM Pervanadat-Lösung durch Mischen von 60 ul 0,2 M Natriumvanadat mit 540 & mgr; l H 2 O In einem zweiten Rohr Mischung 2,7 ul 30% Wasserstoffperoxid und 1,4 ml PBS. Fügen Sie die beiden Lösungen zusammen und lassen Sie sich für 15 Minuten vor dem Gebrauch.
- Mit Hilfe einer Mehrkanalpipette (verwenden Sie nicht eine automatische Flüssigkeitsspender, wie es erzeugt zu viel Turbulenz in der gut) verzichtet werden 2 ul von 0,25 M CaCl 2 in jede Vertiefung, gefolgt von 2,5 ul des Pervanadat in Schritt 3.1 vorbereitet. Inkubieren jede Platte bei 37 ° C für 10 min und dann auf Eis zu stellen.
- Vorbereitung 35 ml Lyse-Puffer durch Zugabe von DTT, PMSF und Natriumvanadat, wie in Abschnitt 1 (Herstellung der Reagentien) angegeben.
- Keeping Platten auf Eis zu entfernen Medium aus jeder Vertiefung unter Verwendung einer Vakuumstrahlpumpe. Sofort im 50 ul / Vertiefung von eiskaltem Lyse-Puffer unter Verwendung eines automatisierten Flüssigkeitsspender mit einer Standardkassette ausgestattet. Lassen Sie sitzen 30 Minuten lang auf Eis zu lysieren (optional: Hier können gegebenenfalls durch Versiegelung plat stoppene und Lagerung bei -80 ° C. Um auftauen Platten auf Eis) fortsetzen.
- Spin-Platten bei 1.900 × g für 3 min bei 4 ° C.
- Kratzen Zellen aus jeder Vertiefung mit einer Mehrkanalpipette und übertragen Sie alle Inhalte in geeigneter Weise V-Boden Platten mit 96 Vertiefungen gekennzeichnet. Spin-Platten bei 1.900 × g für 10 min bei 4 ° C.
- Während Spin, füllen Glutathion-beschichteten Platten (eine für jede 96-Well-Platte) mit 100 ul / Vertiefung von eiskaltem Lyse-Puffer (ohne PMSF) als Spülung. Halten Platten auf Eis.
- Entfernen V-Bodenplatten aus der Zentrifuge. Ein zu einer Zeit, zu invertieren die Glutathion-Platten über ein Waschbecken zu schütteln den Lysepuffer und Blot auf einem Papiertuch. Bringen Lysepuffer aus den V-Bodenplatten auf die Glutathion-Platten durch Kippen der Platte und mit einer Mehrkanalpipette, man aufpassen, nicht das Pellet am Boden stören. Abdeckplatten und lassen auf Eis für mindestens 2 Stunden zu binden.
- In der Nähe des Ende der 2 Stunden Bindungsschritt, bereiten Sie einen Radiostation, die Gewährleistung tEr erforderlichen Sicherheitsvorkehrungen bestehen für radioaktive Arbeit. Stellen Hybridisierungsofen bis 30 ° C.
- Vorbereitung 200 ml Lysispuffer durch Zugabe von DTT und Natriumvanadat wie in Abschnitt 1. PMSF deutet ist in diesem Stadium nicht erforderlich.
- Kehren Sie die Glutathion-Platten über ein Waschbecken zu schütteln den Lysepuffer und tupfen Sie auf einem Papiertuch. Spülen Sie die Taschen 3x mit 100 ul Lysepuffer (ohne PMSF). Lassen Sie sich nicht Brunnen sitzen trocken - halten sie in Spülung bis bereit zu gehen.
- Bereiten Sie 55 ml 1x Kinasepuffer (KB) durch Verdünnen des 10fach und das Hinzufügen von DTT, wie in Abschnitt 1. Spülen Platten einmal mit 50 ul 1x KB mit Hilfe eines automatisierten Flüssigkeitsspender mit einem Standardvolumen Kassette ausgerüstet angezeigt. Lassen 1x KB in Brunnen, bis die Lösung A bereit ist:
- Vorbereitung der Lösung A durch Zugabe von 500 bis 530 ug des Substrats von Interesse ist, 500 & mgr; g des basischen Myelinproteins (MBP), 2,65 ml 10 × KB, 13,25 & mgr; l 1 M DTT, und H 2 O bis auf 15,9 ml.
- Ein zu einer Zeit, zu invertieren die Platten über ein Waschbecken, um 1x KB Spülung entfernen, Fleck auf Papiertuch, und sofort fügen 30 ul Lösung A unter Verwendung eines automatisierten Flüssigkeitsspender mit einem kleinen Volumen Kassette ausgestattet. Halten Platten auf Eis.
- Vorbereitung der Lösung B in den Arbeitsbereich der Radioaktivität durch Zugabe von 2,5 ml 10x M-ATP, 500 & mgr; Ci 32 P ATP gamma- und H 2 O auf 10 ml.
- In 20 ul Lösung B pro Vertiefung mit einem Repeater-Pipette, die aufgrund von Ausstoßkraft Mischhilfen. Decken und Inkubation in 30 ° C Hybridisierungsofen für 30 min.
- Nach 30 min Übertragungsplatten wieder auf Eis. Zugabe von 50 ul 2 x SDS-Lysepuffer zu jedem Well mit einer Mehrkanalpipette. Kann an dieser Stelle, um zum nächsten Schritt, oder dichten die Platten mit Aluminiumfolie und bei -20 ° C, bis praktisch.
4. Laufsport, Färbung und Trocknung Gele
Hinweis: Alle Arbeiten sind in einem Gebiet durchgeführt werden DESIGNAted auf Radioaktivität.
- Schalten Sie Hybridisierungsofen und auf 85 ° C eingestellt. Tau-Platten bei Raumtemperatur. Sobald Backofen Temperatur, Übertragungsplatten zum Ofen und Inkubation für 10 min, um Proben zu denaturieren erreicht.
- Last 26-Well-Fertig Gele mit 15 ul jeder Reaktion unter Verwendung einer Mehrkanalpipette um mehrere Brunnen auf einmal zu füllen. Ist darauf zu achten, dass alle Spitzen ausgerichtet entsprechend gut, bevor Proben werden. Führen Gel bei 150 V Lassen Sie die Markierungsfarbstoff (blaue Linie) ablaufen der Unterseite des Gels, da dies enthält den unincorporated ATP.
- Demontieren Sie die Gele und schneiden Sie die nicht rechtsfähige ATP (blaue Linie) mit einem Skalpell oder gerade Kante, wie es die Filme überbelichtet. Legen Sie die Gele in beschriftete Behälter geben und mit Coomassie-Färbung für 15 min.
- Entfernen Sie die Coomassie-Färbung, kurz spülen Sie die Gele mit Wasser, und fügen Sie destain Lösung. Entfärben der Gele bis die Proteine sind deutlich sichtbar. Ein Band für MBP und ein Band für das Substrat solltewerden für jede Probe sichtbar.
- Um die Gele trocken:
- Schneiden Sie ein großes Blatt Filterpapier und legen Sie es auf dem Trockner.
- Befeuchten Sie ein Cellophanfolie in destilliertem Wasser, bis es glatt ist und knitterfrei, und legen Sie sie auf dem Papier.
- Verlegen Sie die Gele auf dem Zellophan Blech, die eine Anmerkung in der Größenordnung von Gelen. Befeuchten Sie ein zweites Blatt Cellophan und legen Sie auf der Gele.
- Rollen Sie alle Luftblasen (eine Platte Siegelwalze funktioniert gut für diese) für eine schöne gleichmäßige Oberfläche. Schließen Sie die Klappe, schalten Sie das Vakuum und trocknen Sie die Gele für 3 Stunden bei 80 ° C.
- Sobald Gele trocken sind, setzen Sie sie XAR-Film mit einem Bildschirm, um das Signal zu verstärken. Wickeln Sie die Kassette mit Frischhaltefolie oder eine Plastiktüte und verschließen Sie mit Klebeband gegen Frost draußen zu halten. Bewahren Sie die Kassette bei -80 ° C über Nacht.
5. Entwicklung XAR Films
- Am folgenden Tag, entfernen Kassette aus dem Gefrierschrank und lassen bei Raumtemperatur auftauen. Entwickeln Sie den Film inDunkelkammer unter Verwendung eines Filmprozessors gemß den Anweisungen des Herstellers.
Hinweis: Überprüfen Sie die Folien nach Beweisen für Kinase-Substrat-Paare. Ein zweiter längerer Exposition kann auch nützlich sein schwächer Phosphorylierungsereignisse detektieren.
Ergebnisse
Repräsentative Ergebnisse von einem Bildschirm werden in Abbildung 2 dargestellt. 180 Kinasen wurden unter Verwendung eines GST-tagged Peptidsubstrat entsprechend zu AS 268-283 von CRTC2 sowie klassischen Kinaseassays Substrat das basische Myelinprotein (MBP) gescreent. Nur zwei Kinasen, MARK2 und die eng verwandte Kinase phosphoryliert das MARK3 CRTC2 Peptids. MBP ist als eine interne Kontrolle in allen Assays eingeschlossen, da es enthält viele phosphorylierbare Reste und läuft bei 18 kDa, in Richtung der Unterseite des Gels. Dies ermöglicht eine Interpretation der Spezifität: einige Kinase phosphoryliert wird kräftig ein Substrat und MBP. Der Hinweis ist, dass die Wells GST alleine (dh ohne die Kinase Kontrollen) enthalten reinigen immer eine endogene Kinaseaktivität, wodurch es immer Hintergrund Phosphorylierung in dem Assay. Während dies nicht ausschließen, dass die Phosphorylierung des Substrats ist real, legt es nahe, daß bei der in vitro-Setzen der Kinase kann weniger selektiv.Es ist besonders informativ, um mehrere Substrate unterschiedlicher MW Rückschlüsse auf Kinase-Substratspezifität zu ziehen sind.
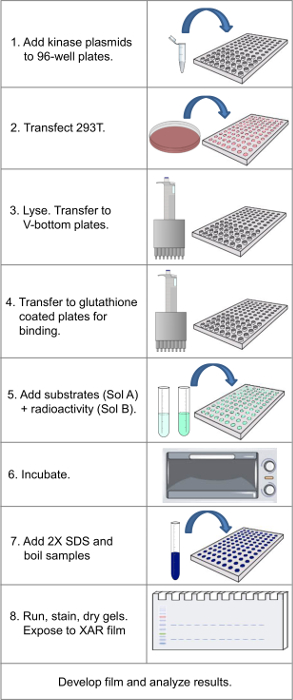
Abbildung 1. Flussdiagramm, das wesentliche Schritte in Protokoll. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 2. Beispiel Bildschirm Ergebnis. Autoradiographien eines Bildschirms der Version 1 der Bibliothek (180 menschliche Proteinkinasen) durchgeführt unter Verwendung von GST-Peptid-Substrat entsprechend zu AS 268-283 des Maus CRTC2 (von Jansson reproduziert et al., 2008). MBP, das basische Myelinprotein ist angegeben. Der hohe Grad der substrate Auswahl von verwandten Kinasen ist eine Funktion des Bildschirms. Jede Spur stellt die Reaktionsprodukte von einer verschiedenen Kinase-Assay. MARK3 (Gel 1) und MARK2 (Gel 16), die einzigen Kinasen zu TORC2 268-283 Peptid phosphorylieren, sind angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Seit den ursprünglichen Veröffentlichungen über den Ansatz 14,15 ist die ursprüngliche Bibliothek 180 GST-Kinasen wurden in 420 Mitglieder oder ~ 80% des menschlichen Protein Kinoms erweitert. Der erweiterte Bibliothek, die wie beschrieben Protokoll dauert 4-5 Tage und dann 1-4 Tage, um Filme zu entwickeln (wie dies notwendig erscheint), die durch die Verwendung von Phosphorimaging und digitale Signalverstärkung verkürzt werden konnte. Es gibt mehrere wichtige Schritte, bei denen darauf geachtet werden muss (siehe Abbildung 1 für die Übersicht über Protokoll). Erstens ist der gesunde Bestand von Zellen (Mykoplasmen freie, niemals zur Konfluenz während der Expansion zu erreichen, und mit Trypsin behandelt bei jedem Durchgang) während des Chargenexpansionsphase und in den 96-Well-Platten bei der Transfektion. Zellen sollten gleichmäßig ausgesät werden, und 80% konfluent, wenn die Transfektion erfolgt (Schritt 1.5).
Zweitens ist es ideal bei der Transfektion ein automatisiertes Flüssigkeitsspender zu verwenden, um die Lautstärke zu minimieren transfer Fehler, so Begrenzungskoeffizienten von Variationen ("cvs") zwischen den Wells. Für kleine Volumina, Einpassen der Spender mit einem kleineren Volumen Kassettengrenzen Reagenzienverlust aufgrund der reduzierten Totvolumina und erhöht die Dosiergenauigkeit. Der Behandlungsschritt mit Natrium Pervanadat, einer Pfanne Tyrosinphosphataseinhibitor wird als allgemeiner Anreiz zur Phosphorylierung von Zielkinasen erhöhen. Die Aufnahme von Calcium in diesem Schritt erhöht die Zell-Matrix-Adhäsion, verhindert Zellverlust durch Runden der Zahlen bei längerer Pervanadat Behandlung ausgelöst. Dieser Schritt sollte nur unter Einhaltung der 10-minütigen Inkubationszeit durchgeführt werden.
Drittens, für den Kinase-Assay selbst, gibt es verschiedene Überlegungen bei der Auswahl des rekombinanten Substrats: Peptide, Proteindomänen, oder intakte Proteine so groß wie 120 kDa können alle verwendet werden. Wenn eine spezifische Phosphorylierungsstelle ist bekanntlich ein Peptidsubstrat die geradlinige Ansatz, aber groß genug sein muss, um zu erkennen seinauf einem SDS-PAGE Gel. Somit können Peptidsubstrate mit GST fusioniert und gereinigt werden von den Bakterien bis ein MG von> 25 ergeben. Größere Proteine haben den Vorteil, daß sie wahrscheinlich gefaltet und ihre phosphorylierten Resten ausgesetzt, wie sie in der Zelle sein, doch sind mit dem Nachteil, dass sie zusätzliche Reste, die phosphoryliert werden kann und gibt Signal in dem Assay umfassen. Wir bevorzugen, um einen Bildschirm unter Benutzung einer Peptidsubstrat von 15-20 Aminosäuren einen Rückstand, der bekanntlich in vivo phosphoryliert werden und ist dafür bekannt, eine biologische Ereignis regulieren Umgebung besteht, wie dies macht Validierung der Kandidaten Kinasen vom Bildschirm sowohl in vitro und in vivo viel schneller.
Letzten, im Idealfall der Menge an Protein, Ansätze oder mehr als 1 & mgr; g pro Napf; dies natürlich variiert mit dem Molekulargewicht von dem Substrat. Weniger Protein pro Vertiefung kann sinn Treffer ergeben, aber die "mehr ist besser" gilt, wie es erhöht Signal: noise. Norm wird, sind nicht zu empfehlen, da Gradientengelen Gradientengelen knacken zu oft beim Trocknen. Nach Trocknung der Gele, Feststellung eines radioaktiven Signals gegenüber dem Hintergrund mit einem Geigerzähler gibt einen ausgezeichneten Erfolgsmeldung des Assays.
Der Bildschirm ist in vitro und weitere Komplexitätsebenen in vivo vorhanden ist, muss ein Kandidat-Kinase (n) für ein gegebenes Substrat in Zellen bestätigt werden. Insbesondere kann der Bildschirm ein Mitglied der Familie, die die biochemische Kapazität, um das Substrat in vitro phosphorylieren hat, aber doch nicht im gleichen Zelltyp oder in demselben subzellulären Kompartiment als Substrat exprimiert werden. Zum Beispiel, während MARK2 und MARK3 waren beide Treffer in einem Bildschirm für Kinase, CRTC2 auf Ser275 phosphorylieren konnten nur MARK2 gebildete Komplex mit dem Substrat in den Zellen 14. Im folgenden sind eine Reihe weiterer Experimente, die verwendet werden, um die physiologische Relevanz der Kandidat-Kinase zu bestätigen. First, Mobilitätsverschiebungen des Substrats, auf SDS-PAGE folgenden Coexpression des Kandidaten Kinase kann als Bestätigungssteuerung verwendet werden. Geeignete Kontrollen wie Co-Transfektion eines katalytisch inaktiven Kinase kann Spezifität zu bestätigen. Zweitens kann das Substrat aus Zellextrakten, die mit 32 P-Orthophosphat eine katalytisch inaktive Kinase inkubiert wurden, um den Einbau von Phosphat in das Substrat in Gegenwart des Wildtyp zu bestätigen und nicht immungefällt werden. Drittens kann ein phosphospecific Antikörper gegen das phosphoacceptor Sequenz (falls bekannt) erzeugt und verwendet, um einen Anstieg der Substratphosphorylierung zu bestätigen, wenn die Kinase überexprimiert werden. Ein zweites Screening unter Verwendung eines Mutanten-Substrat, das eine nicht-phosphorylierten Rest an der Zielstelle kann Spezifität vor der Erzeugung eines phosphoantibody eine besonders wichtige Steuer mit größeren Domäne und Vollängenprotein Materialien zu prüfen. Pharmakologische Hemmung nähert, um einen Kandidaten zu hemmenDatum Kinase kann auch verwendet werden, jedoch ist Vorsicht geboten, wie die Hemmung der verwandten Kinasen ausgeübt wird, ist eine underappreciated Realität. Schließlich RNAi-vermittelte Silencing des Kandidaten-Kinase (n) in Zellen verfolgt und Western-Blotting zum Verlust der Zielstelle mit einer Phosphorylierung phosphospecific Antikörper Überwachung durchgeführt werden kann.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Diese Arbeit wurde vom NSERC Zuschuss unterstützt 386634. Wir möchten die Mitglieder des Screaton Lab für hilfreiche Diskussionen danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Lysis buffer | Made in house | See Protocol step 1.1 | |
| 10x kinase buffer | Made in house | See Protocol step 1.2 | |
| 10x M-ATP | Made in house | See Protocol step 1.3 | |
| Human kinase plasmids | Orfeome, Invitrogen, Origene | GST-tagged in house | |
| 96 well plates | Fisher Scientific | CS003595 | |
| 293T cells | ATCC | CRL-11268 | |
| DMEM | Fisher Scientific | SH3002201 | supplement with 100 U/ml penicillin, 100 μg/ml streptomycin, 10% fetal calf serum. |
| CO2 incubator | Sanyo | MCO-17AIC | |
| 15 cm cell culture dishes | Fisher Scientific | 877224 | |
| Reduced serum medium | Invitrogen | 22600-050 | |
| Lipid-based transfection reagent | Invitrogen | 11668-019 | |
| Automated liquid dispenser | Thermo Scientific | 5840300 | |
| Small cassette attachment | Thermo Scientific | 24073295 | |
| Standard cassette attachment | Thermo Scientific | 14072670 | |
| 4 mM pervanadate | Made in house | See Protocol step 3.1 | |
| 0.25 M CaCl2 | Made in house | ||
| Multichannel pipette (20-200 μl) | Labnet | p4812-200 | |
| Multichannel pipette (1-10 μl) | Thermo Scientific | 4661040 | |
| V-bottom 6-well plates | Evergreen Scientific | 290-8116-01V | |
| Glutathione coated 96-well plates | Fisher Scientific | PI-15240 | |
| Hybridization oven | Biostad | 350355 | |
| GST tagged substrate | Made in house | ||
| Myelin Basic Protein (MBP) | Sigma | M1891 | |
| Repeater pipette (1 ml) | Eppendorf | 22266209 | |
| 32P gamma-ATP | Perkin Elmer | BLU502Z500UC | |
| 2x SDS lysis buffer (100 ml) | Made in house | See Protocol step 1.4 | |
| 26-well precast TGX gels | BioRad | 567-1045 | gel percentage required is dependent on the molecular weight of the substrate of interest |
| Coomassie stain | Made in house | 0.1% Coomassie R250, 10% acetic acid, 40% methanol | |
| Coomassie destain | Made in house | 10% acetic acid, 20% methanol | |
| Labeled gel containers | Made in house | Used plastic lids from empty tip boxes, just big enough to contain one gel | |
| Whatman filter paper | Fisher Scientific | 57144 | |
| Cellophane sheets (2) | BioRad | 165-0963 | |
| Gel dryer | Labconco | 4330150 | |
| Double emulsion autoradiography film | VWR | IB1651454 | |
| Film cassette | Fisher Scientific | FBAC-1417 | |
| Intensifying screen | Fisher Scientific | FBIS-1417 | |
| Plate sealing rubber roller | Sigma | R1275 |
Referenzen
- Meisenhelder, J., Hunter, T., van der Geer, P. Phosphopeptide mapping and identification of phosphorylation sites. Curr Protoc Mol Biol. 18, Unit 18 19 (2001).
- Doll, S., Burlingame, A. L. Mass spectrometry-based detection and assignment of protein posttranslational modifications. ACS chem. 10, 63-71 (2015).
- Sharma, K., et al. Ultradeep human phosphoproteome reveals a distinct regulatory nature of Tyr and Ser/Thr-based signaling. Cell rep. 8, 1583-1594 (2014).
- Cohen, P. The regulation of protein function by multisite phosphorylation--a 25 year update. Trends Biochem Sci. 25, 596-601 (2000).
- Walsh, C. T. . Posttranslation Modification of Proteins: Expanding Nature's Inventory. , (2006).
- Boersema, P. J., et al. In-depth qualitative and quantitative profiling of tyrosine phosphorylation using a combination of phosphopeptide immunoaffinity purification and stable isotope dimethyl labeling. Mol Cell Proteomics. 9, 84-99 (2010).
- Hutti, J. E., et al. A rapid method for determining protein kinase phosphorylation specificity. Nat Methods. 1, 27-29 (2004).
- Johnson, S. A., Hunter, T. Kinomics: methods for deciphering the kinome. Nat Methods. 2, 17-25 (2005).
- Pawson, T., Nash, P. Assembly of cell regulatory systems through protein interaction domains. Science. 300, 445-452 (2003).
- Zhu, H., et al. Analysis of yeast protein kinases using protein chips. Nat Genet. 26, 283-289 (2000).
- Shah, K., Shokat, K. M. A chemical genetic approach for the identification of direct substrates of protein kinases. Methods Mol Biol. 233, 253-271 (2003).
- Zou, L., et al. PKIS: computational identification of protein kinases for experimentally discovered protein phosphorylation sites. BMC bioinform. 14, 247 (2013).
- Varjosalo, M., et al. Application of active and kinase-deficient kinome collection for identification of kinases regulating hedgehog signaling. Cell. 133, 537-548 (2008).
- Jansson, D., et al. Glucose controls CREB activity in islet cells via regulated phosphorylation of TORC2. Proc Natl Acad Sci U S A. 105, 10161-10166 (2008).
- Fu, A., Screaton, R. A. Using kinomics to delineate signaling pathways: control of CRTC2/TORC2 by the AMPK family. Cell Cycle. 7, 3823-3828 (2008).
- Abu-Thuraia, A., et al. Axl phosphorylates elmo scaffold proteins to promote rac activation and cell invasion. Mol Cell Biol. 35, 76-87 (2015).
- Manning, G., Whyte, D. B., Martinez, R., Hunter, T., Sudarsanam, S. The protein kinase complement of the human genome. Science. 298, 1912-1934 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten