Method Article
ハイスループットスクリーニングを使用したキナーゼ基質ペアの同定
要約
Protein phosphorylation is a central feature of how cells interpret and respond to information in their extracellular milieu. Here, we present a high throughput screening protocol using kinases purified from mammalian cells to rapidly identify kinases that phosphorylate a substrate(s) of interest.
要約
我々は、新規のシグナル伝達経路を解明するために使用することができ、リン酸化基質のための専用のヒトプロテインキナーゼを同定するためのスクリーニングプラットフォームを開発しました。我々のアプローチは、精製GSTタグ付きヒトプロテインキナーゼのライブラリーを使用すると、目的の組換えタンパク質基質を備えています。私たちは、CREB-規制転写コアクチベーター2(CRTC2)のグルコース調節サイトのキナーゼとしてのMAP /微小管親和性調節キナーゼ2(MARK2)を識別するために、β細胞の増殖に必要なタンパク質、ならびにこの技術を使用していますアダプタータンパク質ELMOのリン酸化による細胞の転移の調節因子としてのチロシンキナーゼのAXLファミリー。私たちはこの技術を記述し、それは、細胞が環境刺激に応答する方法の包括的なマップを確立するのを助けることができる方法について説明します。
概要
タンパク質の翻訳後修飾(PTMを)は、細胞内の通信のために不可欠です。おそらく全てのPTMの検討最高の細胞内局在性、コンフォメーション、安定性、それらの生化学的活性を含むタンパク質の機能の無数を調節するタンパク質キナーゼによって触媒されるリン酸化は、です。標的タンパク質上のリン酸化部位の同定は、トリプシンリンペプチドマッピングにより、またはリン酸化ペプチド1,2-について富化サンプルを使用して、今、標準プロテオミクス技術によって達成することができます。表現プロテオームの四分の三が3リン酸化されることが予想され、100万6までの推計で20万リン酸化部位5を特定しているが、これらの多くは、経路、またはプロテインキナーゼシグナル伝達、全く割り当てられた生物学を持っていません。
リン酸化部位の同定は比較的簡単ですが、比較的大きな挑戦はにあります基板のペア:これらのサイトを対象と同族キナーゼ(複数可)、我々はマッピングキナーゼと呼ぶプロセスが識別されます。いくつかのキナーゼを同定するための方法:基板対は、目的のキナーゼで開始し、その基質を探しまたは対象の基板から始まると修飾キナーゼ実験7-11または計算12を検索しようとするいずれかの記載されています。既知のリン酸化基質のためのキナーゼを同定するために、バイオインフォマティクスは、基板と沈殿複合体を形成するキナーゼをリン酸化残基(コンセンサス部位)に隣接する、ならびに特定のアミノ酸の短い保存配列を含むタンパク質を同定するために使用することができます。しかしながら、これらの方法は時間がかかり、多くの場合、成功して満たしていません。
私たちは、急速に所定の基板13をリン酸化することができるキナーゼを同定するための体系的な機能的なアプローチを開発しました。スクリーンアッセイは、優れた特定の生成します潜在的な同族のキナーゼのために非常に明確な選択と性、。生物学的シグナル伝達にリン酸化の中心性を考えると、画面は、事実上すべての細胞シグナル伝達経路14-16で発見するのに便利です。画面には、ヒトタンパク質キナーゼのライブラリーと、大規模なキナーゼアッセイを実施することを含みます。キナーゼは、細菌のグルタチオンS-トランスフェラーゼ(GST)タンパク質でタグ付けされており、組換え酵素があることを意味する、哺乳類細胞抽出物から精製される - 細菌から調製されたものとは異なり、 - 多くの場合に必要な上流のプロテインキナーゼの存在下で生成されます組換え酵素は、インビトロで活性を有すること。セリン、スレオニン、およびチロシンキナーゼ活性は、下流のキナーゼの活性化のために必要としながら実際には、酵母10内に存在する、酵母ゲノムは、500を超える遺伝子17と哺乳類キノームは、するために、かなり複雑になっていることを示す、122プロテインキナーゼをコードしレギュレーションより高次の生物に固有のプロセスをTE。さらに、(例えば、 その他の小分子、増殖因子、ホルモンなど)、細胞生物学およびヒトの疾患に関連した様々な刺激の効果は、適切なコンテキストに14,15モジュレートキナーゼ活性のために使用することができます。
プロトコル
試薬、プレート、および細胞の作製
- 25mMトリスpH7.5で、150mMのNaCl、50mMのNaFを、0.5mMのEDTA pH8.0の、0.5%トリトンX-100、5mMのβグリセロリン酸、5%グリセロール:500 mlの溶解バッファー行います。 4℃で保存します。使用1mMジチオスレイトール(DTT)、1mMのフェニルメチルスルホニルフルオリド(PMSF)及び1mMのバナジン酸ナトリウムを添加する直前。このステップの後、PMSFは、任意のすすぎバッファに必要とされません。
- 200 mMトリスpH7.5の、50mMのベータ - グリセロホスフェート(FW 216)、2mMのバナジン酸ナトリウム:10倍キナーゼ緩衝液20mlのを行います。 -20℃で1 mlチューブで分注し、店舗。使用直前に、5mMのDTTを追加します。
- 300μMのアデノシン三リン酸(ATP)、66mMのMgCl 2を、-20℃で33 mMでのMnCl 2.1 mlチューブで小分けして保存:10倍、M-ATPバッファの20ミリリットルを行います。
- 1.5グラムのトリス塩基、20 mlのグリセロール、30mLのH 2 O:2Xドデシル硫酸ナトリウム(SDS)の溶解緩衝液100mlを作ります溶解し、HClでpHを6.8に調整します。 40を追加mlの10%SDS、100ミリリットルにボリュームを調整します。 25 mgのブロモフェノールブルーを追加します。
- スポット応じて96ウェルプレートとラベルプレートのセットで、ウェルあたりのGST-キナーゼ14をコードする 25 ngの/μlの哺乳動物発現プラスミドの4μlの。使用するまで-20℃でプレートを密封し、凍結。
- トランスフェクションの2日前に、CO 2(最終5%)を補充した37℃のインキュベーター内で培養HEK293Tの完全ダルベッコ改変イーグル培地(DMEM)+ 10%ウシ胎児血清(FBS)中の細胞及び抗生物質。そしてトリプシンで通過は在庫が拡張中に、> 80%コンフルエントになることはできません。 6×10 7細胞の最小画面(80%コンフルエンスでHEK293T細胞の約3×15cmの皿)に必要とされます。
2.トランスフェクション
注意:プロトコル全体のフローチャートについては、図1を参照してください 。
- キナーゼプラスミドを含むプレートは、凍結されている場合は、室温aの解凍NDウェルの底に任意の水分を収集するために、3分間1900×gで遠心します。
- 脂質ベースのトランスフェクション試薬の312.7μlの低血清培地(例えば、OPTI-MEM)の8.6ミリリットルを混ぜます。 5分間座ってみましょう。
- 小容量のカセットを装着した自動液体ディスペンサーを用いて各ウェルに減少血清培地10μlのを追加します。
- 小容量のカセットを装着した自動液体ディスペンサーを用いて、ステップ2.2から減少血清培地/トランスフェクション試薬混合物のウェルあたり10μLを追加します。 20〜45分間座ってみましょう。
- 完全DMEM 80mlに7.5×10 5細胞/ mlで再懸濁した293T細胞。よく標準ボリュームカセットを装着した自動液体ディスペンサーを用いあたり100μlの細胞懸濁液( すなわち 7.5×10 4細胞)を追加します。
- 均一な細胞分布のために顕微鏡下で井戸を確認し、24時間インキュベーターに戻します。
3. GST-キナーゼプルダウン
- Fを作りますレッシュ:H 2 Oの540μlの0.2 Mバナジン酸ナトリウムの60μLを混合することにより、4 mMのペルバナデートソリューション第2のチューブミックス30%の過酸化物の2.7マイクロリットルのPBS 1.4ミリリットルで。 2つのソリューションを追加し、使用前に15分間放置します。
- マルチチャンネルピ ペットを使用して(それもであまりにも多くの乱流を生成するように自動液体ディスペンサーを使用しないでください)ステップ3.1で調製した過バナジン酸の2.5μlのに続いて、各ウェルに0.25 M CaCl 2を2μLを分注します。 37℃で10分間各プレートをインキュベートした後、氷の上に置きます。
- 第1(試薬の調製)に示すようにDTT、PMSFおよびバナジン酸ナトリウムを添加することによって溶解緩衝液の35ミリリットルを準備します。
- 氷の上でプレートを維持、真空吸引器を使用して、各ウェルから培地を除去します。すぐに標準カセットを装着した自動液体ディスペンサーを用いて氷冷溶解緩衝液50μl/ウェルを追加します。オプション(溶解し、氷上で30分間座ってみましょう:必要に応じてPLATをシールすることによってここに停止することができますE、-80℃で保存します。氷の上で、解凍プレート)を続行します。
- 4℃で3分間、1,900×gでスピンプレート。
- 各ウェルマルチチャンネルピペットを使用してから細胞をこすり落とし、すべての内容を転送は、適切にV底96ウェルプレートをラベル付けします。 4℃で10分間、1900×gでスピンプレート。
- スピン中、リンスとして(PMSFなし)の氷冷溶解緩衝液100μl/ウェルでグルタチオン被覆したプレート(各96ウェルプレートに1つずつ)を入力します。氷の上でプレートを保管してください。
- 遠心機からのV底プレートを取り外します。当時の一つは、ペーパータオルの溶解バッファーとブロットを振るために、シンクの上にグルタチオンプレートを反転。底にペレットを乱さないように注意しながら、プレートを傾けると、マルチチャンネルピペットを用いて、グルタチオンプレートにV底プレートから溶解バッファーを転送します。プレートをカバーして結合するための最小2時間、氷上に残します。
- 2時間結合ステップの終わりに近く、トンを確保し、放射能のワークステーションを準備します彼に必要な安全上の注意事項は、放射性の仕事のための場所です。 30℃のハイブリダイゼーションオーブンを設定します。
- 1. PMSFこの段階で必要とされていないセクションに示すようにDTTおよびナトリウムバナジン酸を加えることによって、溶解緩衝液200ミリリットルを準備します。
- 溶解バッファーを振る、ペーパータオルにブロットするシンクの上にグルタチオンプレートを反転します。ウェルは(PMSFなし)、100μlの溶解緩衝液で3倍すすいでください。井戸が乾燥座らせてはいけない - 続行する準備ができるまで、すすぎに保管してください。
- 10倍の株式を希釈し、標準ボリュームのカセットを装着した自動液体ディスペンサーを用いて1×KB50μlの1リンスプレート一度節に示すようにDTTを追加することによって、1×キナーゼ緩衝液(KB)の55ミリリットルを準備します。溶液Aの準備ができるまでのウェルで1倍KBを残します:
- 15.9ミリリットルまで530関心の基板μgの、ミエリン塩基性タンパク質の500μgの(MBP)、10倍の2.65ミリリットルKB、1 M DTTの13.25μL、 および H 2 Oに500を追加することによって、溶液Aを準備します。
- 一度に1つは、1×KBリンスを除去ペーパータオルにブロットし、すぐに小容量のカセットを装着した自動液体ディスペンサーを用いて溶液Aの30μlを添加するシンクの上にプレートを反転。氷の上でプレートを保管してください。
- 2.5ミリリットルの10倍のM-ATP、500μCiのガンマ- 32 PのATP、 および H 2 Oに10ミリリットルを追加することによって、放射能作業領域に溶液Bを準備します。
- よく吐出力による混合に役立つリピーターピペットを用いあたり溶液Bの20μLを加えます。覆い、30分間30℃のハイブリダイゼーションオーブンでインキュベート。
- 30分後、氷にバックプレートを転送します。各ウェルマルチチャンネルピペットを使用して2倍のSDS溶解緩衝液50μlを追加します。次のステップにこの時点で進行、または便利になるまで-20℃でアルミホイルや店舗でプレートをシールすることができます。
4.ランニング、染色、乾燥ゲル
注:すべての作業は、エリアdesignaで実行する必要があります放射能のためのテッド。
- ハイブリダイゼーションオーブンをオンにして、85℃に設定してください。室温で解凍プレート。一度オーブンオーブンで10分間サンプルを変性するためにインキュベートした温度、転写板に到達しました。
- ロード一度に複数の井戸を埋めるためにマルチチャンネルピペットを用いて、各反応15μlの26ウェルのプレキャストゲル。ケアは、すべてのヒントは、サンプルを追加する前に十分に対応して整列するように注意しなければなりません。これが組み込まれていないATPが含まれているとして、追跡用色素(青線)は、ゲルの底部から実行させてはいけない150 Vでゲルを実行します。
- ゲルを解体して、フィルムを露出オーバーになりますようにメスまたはストレートエッジを使用して、取り込まれていないATP(青線)を遮断します。ラベル付き容器にゲルを置き、15分間のクマシー染色でカバーしています。
- 簡単に、クマシー染色を取り除くには、水でゲルを洗浄し、溶液を脱色追加。タンパク質がはっきりと見えるようになるまで、ゲルを脱色。 MBPのためのバンドと基板用バンドべき各サンプルの見えること。
- ゲルを乾燥させるには:
- ろ紙の大きなシートをカットし、乾燥機の上に置きます。
- それは滑らかで、しわがなくなるまで蒸留水にセロファンシートを湿らせて、紙の上に置きます。
- ゲルのためのノートを作り、セロハンシートの上にゲルを置きます。二セロファンシートを湿らせ、ゲルの上に置きます。
- 素敵な均一な表面のためのすべてのバブルを(プレートシールローラは、この適しています)ロールアウト。 、フラップを閉じ、真空をオンにし、80℃で3時間、ゲルを乾燥させます。
- ゲルが乾燥したら、信号を強化するには、画面を使用してXARフィルムにそれらを公開します。サランラップやビニール袋でカセットをラップし、霜を保つためにテープで密封します。 -80℃で一晩カセットを保管してください。
5. XARフィルムを開発
- 次の日、冷凍庫からカセットを除去し、室温で解凍してみましょう。フィルムを開発製造業者の指示に従ってフィルムプロセッサを用いて暗室。
注:キナーゼ基質のペアの証拠のためのフィルムを調べます。より長い第2の露光はまた、より弱いリン酸化事象を検出するのに有用であり得ます。
結果
画面の代表的な結果を図2に示す 。180は、CRTC2キナーゼならびに古典的なキナーゼアッセイ基質ミエリン塩基性タンパク質(MBP)のアミノ酸268から283に対応するGSTタグ化ペプチド基質を用いてスクリーニングしました。唯一の2つのキナーゼ、MARK2および高度に関連するキナーゼMARK3は、CRTC2ペプチドをリン酸化しました。それは多くのリン酸化残基を含有し、ゲルの底に向かっ、18 kDaので動作するようMBPは、全てのアッセイで内部コントロールとして含まれています。これは特異性の解釈を可能にする:いくつかのキナーゼは、堅牢基板およびMBPをリン酸化します。ここで注目すべきは、このように常にアッセイにおけるバックグラウンドのリン酸化があり、(無キナーゼコントロールすなわち )GSTのみを含むウェルは、常にいくつかの内因性キナーゼ活性を浄化することです。これは基質のリン酸化が本物であることを排除していませんが、それは 、インビトロでのキナーゼの設定は以下の選択であることを示唆しているん。これは、キナーゼ基質特異性に関する結論を引き出すためにMWの異なる複数の基板を含むことが特に有益です。
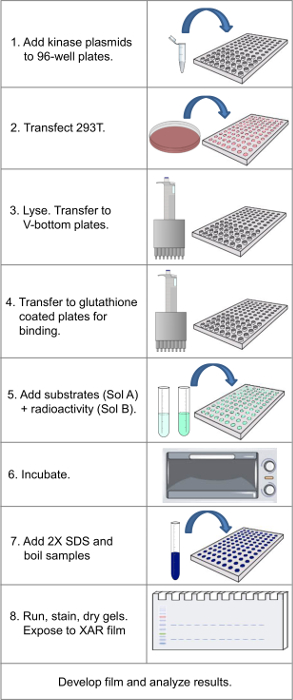
プロトコルにおける重要なステップを示す図1のフローチャート 。 この図の拡大版をご覧になるにはこちらをクリックしてください。

ライブラリー(180ヒトプロテインキナーゼ)のバージョン1の画面の図の画面結果の2例。オートラジオグラフ(ヤンソンから再生ら 、2008)、マウスCRTC2のAA 268から283に対応するGST-ペプチド基質を使用して行います。 MBP、ミエリン塩基性タンパク質を示しています。複数の高度関連キナーゼによるubstrate選択は画面の機能です。各レーンは、異なるキナーゼアッセイからの反応生成物を表します。 MARK3(ゲル1)とMARK2(ゲル16)が、唯一のキナーゼはTORC2 268から283ペプチドをリン酸化するために、示されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
アプローチ14,15を記述する元の出版以来、180 GST-キナーゼの元のライブラリは、ヒトタンパク質キノームの420のメンバー、または〜80% にまで拡大されました。拡大ライブラリを使用すると、説明したようにプロトコルは、ホスホおよびデジタル信号増強を使用することによって短縮することができた、(必要と認めた場合)フィルムを開発するために4〜5日、その後1-4日かかります。注意しなければならないいくつかの重要なステップは、(プロトコルの概要について、図1を参照してください )があります。まず、トランスフェクションの時点でバッチ拡張段階、96ウェルプレート中で(マイコプラズマフリー、拡張中にコンフルエントに到達させず、各継代でのトリプシン処理決して)細胞のストックの健康です。トランスフェクションが行われ(ステップ1.5)を取るとき、細胞を均一に播種すると、80%のコンフルエントにする必要があります。
第二に、ボリュームTRANSFを最小にするために、自動化液体ディスペンサを使用するトランスフェクション時に理想的ですERエラーは、このように、ウェル間の変動係数(「CVを ")制限します。小さなボリュームの場合、精度を分配減少デッドボリュームと増加に起因する小容量カセット制限試薬損失でディスペンサーを当てはめます。過バナジン酸ナトリウム、パンチロシンホスファターゼ阻害剤を用いた治療工程は、標的キナーゼのリン酸化状態を増加させるために、一般的な刺激として使用されます。この段階でのカルシウムの含有は、長引くペルバナデート治療によって誘発される丸めに細胞の損失を防ぐ、細胞 - マトリックス接着を向上させます。このステップは、10分間のインキュベーション時間を厳守して行われるべきです。
すべてを使用することができる120 kDaのと同じ大きさのペプチド、タンパク質ドメイン、またはインタクトなタンパク質:第三に、キナーゼアッセイ自身のために、いくつかの組換え基質を選択する考慮事項があります。特定のリン酸化部位が知られている場合、ペプチド基質は、最も直接的なアプローチであるが、検出するのに十分大きいことが必要ですSDS-PAGEゲル上で。したがって、ペプチド基質は> 25のMWを与える細菌からGSTに融合させ、精製することができます。より大きなタンパク質は、それらがおそらく折り畳まれ、それらが細胞にあるであろうとしてのリン酸化残基を露出、まだ彼らはリン酸化されることができ、追加の残基を含む、アッセイの信号を与えるという欠点が付属しているという利点があります。これは、両方の画面から候補キナーゼの検証をレンダリングするように私達の好みは、 インビボでリン酸化されることが知られており、生物学的事象を調節することが知られている残基を囲む15-20個のアミノ酸からなるペプチド基質を使用して画面を実行することですin vitroおよびin vivoではるかに高速です。
最後に、理想的には、タンパク質のアプローチの量や、ウェル当たり1μgのを超えています。もちろん、これは、基板のMWによって変化します。ウェル当たり少ないタンパク質は、意味のあるヒットを得ることができるが、それは信号を増大させるよう、ルールが適用される」よりは優れている ':NOISE。勾配ゲルが乾燥時にあまりにも頻繁に割れている標準は、非勾配ゲルを推奨します。ゲルを乾燥させた後、ガイガーカウンターとバックグラウンドを超える放射性シグナルの検出は、アッセイの成功の優れた指標を与えます。
画面がインビトロであると複雑さの追加レベルが in vivoで存在するように、所定の基板の候補キナーゼ(複数可)は、細胞内で検証する必要があります。具体的には、画面はまだ、同じ細胞型において、または基板と同じ細胞内区画において発現されない、 インビトロで基質をリン酸化する生化学的能力を有している家族を識別することができます。 MARK2とMARK3はSer275にCRTC2をリン酸化することができキナーゼの画面の両方でヒットした一方で、例えば、唯一のMARK2はセル 14内の基板との複合体を形成しました。候補キナーゼの生理学的な関連性を確認するために使用することができる追加の一連の実験は以下のとおり。ファーSTは、候補キナーゼの同時発現以下のSDS-PAGE上の基板の移動度シフトは、確認コントロールとして使用することができます。適切なコントロールは、触媒的に不活性なキナーゼのような同時トランスフェクションは、特異性を確認することができます。第二に、基板は、野生型ではなく、触媒的に不活性キナーゼの存在下での基質へのリン酸の取り込みを確認するために、32 P-オルトリン酸と共にインキュベートされた細胞抽出物から免疫沈降させることができます。第三に、リン酸特異的抗体は、(既知の場合)ホスホ配列に対して生成され、キナーゼが過剰発現されたときに基質リン酸化の増加を確認することができます。標的部位で非リン酸化残基を担持する変異体基板を用いた二次スクリーニングは、事前phosphoantibodyの発生に大きなドメインおよび完全長タンパク質基質に特に重要な制御の特異性を確認することができます。キャンディを阻害する薬理学的阻害アプローチ日付キナーゼを用いることも可能で、まだ関連するキナーゼの阻害が過小評価現実であるよう注意が必要。最後に、細胞中の候補キナーゼ(単数または複数)のRNAi媒介性サイレンシングが続き、リン酸特異的抗体を用いて標的部位のリン酸化の減少をモニターするウェスタンブロッティングを行うことができます。
開示事項
The authors have nothing to disclose.
謝辞
この作品は、NSERCの助成金によってサポートされていました 386634.私たちは有用な議論のためにScreatonラボのメンバーに感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| Lysis buffer | Made in house | See Protocol step 1.1 | |
| 10x kinase buffer | Made in house | See Protocol step 1.2 | |
| 10x M-ATP | Made in house | See Protocol step 1.3 | |
| Human kinase plasmids | Orfeome, Invitrogen, Origene | GST-tagged in house | |
| 96 well plates | Fisher Scientific | CS003595 | |
| 293T cells | ATCC | CRL-11268 | |
| DMEM | Fisher Scientific | SH3002201 | supplement with 100 U/ml penicillin, 100 μg/ml streptomycin, 10% fetal calf serum. |
| CO2 incubator | Sanyo | MCO-17AIC | |
| 15 cm cell culture dishes | Fisher Scientific | 877224 | |
| Reduced serum medium | Invitrogen | 22600-050 | |
| Lipid-based transfection reagent | Invitrogen | 11668-019 | |
| Automated liquid dispenser | Thermo Scientific | 5840300 | |
| Small cassette attachment | Thermo Scientific | 24073295 | |
| Standard cassette attachment | Thermo Scientific | 14072670 | |
| 4 mM pervanadate | Made in house | See Protocol step 3.1 | |
| 0.25 M CaCl2 | Made in house | ||
| Multichannel pipette (20-200 μl) | Labnet | p4812-200 | |
| Multichannel pipette (1-10 μl) | Thermo Scientific | 4661040 | |
| V-bottom 6-well plates | Evergreen Scientific | 290-8116-01V | |
| Glutathione coated 96-well plates | Fisher Scientific | PI-15240 | |
| Hybridization oven | Biostad | 350355 | |
| GST tagged substrate | Made in house | ||
| Myelin Basic Protein (MBP) | Sigma | M1891 | |
| Repeater pipette (1 ml) | Eppendorf | 22266209 | |
| 32P gamma-ATP | Perkin Elmer | BLU502Z500UC | |
| 2x SDS lysis buffer (100 ml) | Made in house | See Protocol step 1.4 | |
| 26-well precast TGX gels | BioRad | 567-1045 | gel percentage required is dependent on the molecular weight of the substrate of interest |
| Coomassie stain | Made in house | 0.1% Coomassie R250, 10% acetic acid, 40% methanol | |
| Coomassie destain | Made in house | 10% acetic acid, 20% methanol | |
| Labeled gel containers | Made in house | Used plastic lids from empty tip boxes, just big enough to contain one gel | |
| Whatman filter paper | Fisher Scientific | 57144 | |
| Cellophane sheets (2) | BioRad | 165-0963 | |
| Gel dryer | Labconco | 4330150 | |
| Double emulsion autoradiography film | VWR | IB1651454 | |
| Film cassette | Fisher Scientific | FBAC-1417 | |
| Intensifying screen | Fisher Scientific | FBIS-1417 | |
| Plate sealing rubber roller | Sigma | R1275 |
参考文献
- Meisenhelder, J., Hunter, T., van der Geer, P. Phosphopeptide mapping and identification of phosphorylation sites. Curr Protoc Mol Biol. 18, Unit 18 19 (2001).
- Doll, S., Burlingame, A. L. Mass spectrometry-based detection and assignment of protein posttranslational modifications. ACS chem. 10, 63-71 (2015).
- Sharma, K., et al. Ultradeep human phosphoproteome reveals a distinct regulatory nature of Tyr and Ser/Thr-based signaling. Cell rep. 8, 1583-1594 (2014).
- Cohen, P. The regulation of protein function by multisite phosphorylation--a 25 year update. Trends Biochem Sci. 25, 596-601 (2000).
- Walsh, C. T. . Posttranslation Modification of Proteins: Expanding Nature's Inventory. , (2006).
- Boersema, P. J., et al. In-depth qualitative and quantitative profiling of tyrosine phosphorylation using a combination of phosphopeptide immunoaffinity purification and stable isotope dimethyl labeling. Mol Cell Proteomics. 9, 84-99 (2010).
- Hutti, J. E., et al. A rapid method for determining protein kinase phosphorylation specificity. Nat Methods. 1, 27-29 (2004).
- Johnson, S. A., Hunter, T. Kinomics: methods for deciphering the kinome. Nat Methods. 2, 17-25 (2005).
- Pawson, T., Nash, P. Assembly of cell regulatory systems through protein interaction domains. Science. 300, 445-452 (2003).
- Zhu, H., et al. Analysis of yeast protein kinases using protein chips. Nat Genet. 26, 283-289 (2000).
- Shah, K., Shokat, K. M. A chemical genetic approach for the identification of direct substrates of protein kinases. Methods Mol Biol. 233, 253-271 (2003).
- Zou, L., et al. PKIS: computational identification of protein kinases for experimentally discovered protein phosphorylation sites. BMC bioinform. 14, 247 (2013).
- Varjosalo, M., et al. Application of active and kinase-deficient kinome collection for identification of kinases regulating hedgehog signaling. Cell. 133, 537-548 (2008).
- Jansson, D., et al. Glucose controls CREB activity in islet cells via regulated phosphorylation of TORC2. Proc Natl Acad Sci U S A. 105, 10161-10166 (2008).
- Fu, A., Screaton, R. A. Using kinomics to delineate signaling pathways: control of CRTC2/TORC2 by the AMPK family. Cell Cycle. 7, 3823-3828 (2008).
- Abu-Thuraia, A., et al. Axl phosphorylates elmo scaffold proteins to promote rac activation and cell invasion. Mol Cell Biol. 35, 76-87 (2015).
- Manning, G., Whyte, D. B., Martinez, R., Hunter, T., Sudarsanam, S. The protein kinase complement of the human genome. Science. 298, 1912-1934 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。
