Method Article
Reação selecionada Monitoramento Espectrometria de Massa para Absolute Quantificação de Proteína
Neste Artigo
Resumo
This protocol describes how to perform absolute quantification assays of target proteins within complex biological samples using selected reaction monitoring. It was used to accurately quantify proteins of the mouse macrophage chemotaxis signaling pathway. Target peptide selection, assay development, and qualitative and quantitative assays are described in detail.
Resumo
Absolute quantification of target proteins within complex biological samples is critical to a wide range of research and clinical applications. This protocol provides step-by-step instructions for the development and application of quantitative assays using selected reaction monitoring (SRM) mass spectrometry (MS). First, likely quantotypic target peptides are identified based on numerous criteria. This includes identifying proteotypic peptides, avoiding sites of posttranslational modification, and analyzing the uniqueness of the target peptide to the target protein. Next, crude external peptide standards are synthesized and used to develop SRM assays, and the resulting assays are used to perform qualitative analyses of the biological samples. Finally, purified, quantified, heavy isotope labeled internal peptide standards are prepared and used to perform isotope dilution series SRM assays. Analysis of all of the resulting MS data is presented. This protocol was used to accurately assay the absolute abundance of proteins of the chemotaxis signaling pathway within RAW 264.7 cells (a mouse monocyte/macrophage cell line). The quantification of Gi2 (a heterotrimeric G-protein α-subunit) is described in detail.
Introdução
Experimentos de proteômica que usam a espectrometria de massa (MS) pode ser projetado para usar um não-alvo (espingarda) ou métodos direcionados. Proteómica descoberta baseia-se na espingarda geralmente de baixo para cima MS, usando um modo de aquisição dependente dos dados tradicional, ou utilizando uma das técnicas independentes de dados desenvolvidas recentemente (por exemplo, MS E, SWATH) 1,2. Proteómica espingarda é uma ferramenta poderosa para a identificação de alto rendimento péptido e quantificação relativa, mas é geralmente inadequada para a quantificação absoluta ou para o direccionamento pequenos conjuntos, definidos ~ (RTE) de proteínas. O método MS mais frequentemente usado para proteômica alvejados é selecionado monitoramento de reação (SRM) por causa de sua alta sensibilidade, velocidade e alcance dinâmico 3-5. Alternativas a SRM incluem monitoramento de reação paralela, que tira proveito de alta resolução, completa varredura MS 6.
SRM é geralmente realizada utilizando um fluxo de rever nano-sed cromatografia em fase líquida de alta eficiência (nano-RP-LC) acoplado a um instrumento de ionização por electropulverização de nano-(nano-ESI) ligado a fonte de iões de um espectrómetro de massa triplo quadrupolo (QQQ-MS). Numa experiência típica, as proteínas da amostra são proteoliticamente digeridos, e os peptídeos resultantes foram separados cromatograficamente, dessorvido, e ionizado. Os iões resultantes são precursoras m / z filtrada por o primeiro quadrupolo (Q1) e fragmentada no segundo quadrupolo (Q2) por colidir com um gás de colisão. Os iões resultantes são fragmento m / no terceiro quadrupolo (Q3) e quantificada por um dínodo filtrou-z. Cada par de iões fragmento precursor e é referido como uma transição, e cada transição é monitorizado por um período determinado de tempo (tempo de espera, tipicamente 2-50 ms). Durante LC-SRM, os ciclos QQQ-MS por meio de uma lista pré-definida de transição (o ciclo de serviço é tipicamente ≤3 seg), e um cromatograma de cada transição é produzido.
Strateg alternativas para a quantificação de proteínas normalmente utilizam imunoensaios tais como borrões ponto, manchas de Western, ELISAs, microarrays de anticorpos, microarrays de proteínas de fase reversa, imunoensaios microfluídicos, ELISAs digitais e imunoensaios à base de microesferas 7. Os melhores imunoensaios podem ser significativamente mais sensíveis do que os LC-SRM, e o manuseamento das amostras de imunoensaios podem ser significativamente mais elevada do que a da LC-5 SRM. No entanto, imunoensaios em desenvolvimento pode ser caro e / ou demorado, e os ensaios resultantes podem estar vulneráveis a reactividade cruzada e / ou interferências, incompatível com células / tecido de lise / métodos de homogeneização, e / ou não passíveis de multiplexagem 5,8. Algumas dessas questões podem ser abordadas por acoplamento técnicas anticorpo-e baseado no MS. Por exemplo, as proteínas alvo podem ser enriquecidas utilizando imunoprecipitação prévia à proteólise e LC-SRM 9-12. Alternativamente, a técnica emprega SISCAPA imunoprecipitação subsequente à proteólise no leve peptídeol 13,14. Além immunoenrichment estratégias, imunodepleção de proteínas de alta abundância pode ser empregue para aumentar a sensibilidade de LC-SRM, reduzindo a interferência por coeluting analitos 15,16.
Quantificação de proteína baseada em MS pode ser dividido em quantificação relativa e absoluta, e também para rotulagem isótopo estável e livre de marcador (por exemplo, a etiquetagem metabólica, marcação química, e pesada da proteína marcada com padrões internos e peptídicos). Técnicas livre de marcador pode ser útil para a quantificação relativa de proteína, mas não são adequados para a quantificação exacta absoluta. Por comparação, técnicas de marcação já reduzida de erro associado com a preparação da amostra e da variância MS, e são muitas vezes utilizados para a quantificação relativa de proteína 17. Por exemplo, os padrões de isótopos estáveis proteoma marcado (SILAP) preparado utilizando uma linha de células humanas de cultura permitiu a quantificação relativa de biomarcadores potenciais através de LC-SRM de soro humano 18. Accurate quantificação absoluta de proteína por MS requer que as normas internas purificada, quantificado, proteínas isotopicamente marcada ou peptídeo ser cravado-em amostras biológicas antes de MS. A incorporação de padrões internos marcados com isótopos pesados em um fluxo de trabalho LC-SRM permite a quantificação absoluta de que foi mostrado ser altamente reprodutível e transferível entre laboratórios 16,19.
Rotulados padrões internos de isótopos estáveis para a quantificação absoluta de proteína por MS incluem padrões de peptídeos preparados usando a síntese em fase sólida 20, proteínas compostas de padrões de peptídeos protease clivável concatenadas 21, e padrões de proteína de comprimento total 22. Alvo modificação covalente de proteínas e preparação de amostras incompletas (por exemplo, a lise da amostra incompleta e homogeneização, e solubilização de proteínas incompletas, a desnaturação, a alquilação, e a proteólise) pode prejudicar a quantificação precisa. P Internonormas rotein são os menos susceptíveis de serem afectadas pela maioria desses problemas potenciais, mas eles geralmente são os mais difíceis de preparar. Uma alternativa é a de analisar cada proteína alvo utilizando vários padrões peptídicos internos que são concebidos para incluir os resíduos de flanqueamento nativos amino e carboxi-terminais. Independentemente de qual o tipo de padrão interno é utilizado, ele deve ser cravado-os em amostras biológicas, o mais a altura durante a preparação da amostra quanto possível. Além disso, técnicas de preparação de amostras múltiplas (por exemplo, as diferentes condições de desnaturação) deve ser testado. O uso de várias técnicas experimentais ortogonais (de validação cruzada experimental) é uma estratégia viável para a superação de mais quantificação potencial desafia 23-25.
LC-SRM quantificação de proteínas é uma técnica altamente flexível, que foi utilizado numa ampla variedade de aplicações. Notavelmente, tem sido utilizada para estudar peptídicos e proteicos biomarcadores dentroamostras clínicas, tais como soro, biópsias, e punção aspirativa por agulha fina 5. LC-SRM também tem sido utilizado para medir a estequiometria de complexos de proteínas 5,26, para detectar neurotoxinas botulínicas 27, para quantificar dinâmica de fosforilação de proteína dentro de vias de sinalização 5, e para quantificar as alterações na conformação da proteína 28.
Nosso laboratório está usando LC-SRM para quantificar as proteínas sinalizadoras que medeiam a quimiotaxia de macrófagos para apoiar o desenvolvimento de simulações via quimiotaxia. O esquema geral do protocolo (Figura 1) começa com o ranking dos peptídeos alvo provisórias. Subsequentemente, padrões externos de péptidos em bruto são sintetizados e utilizados para desenvolver testes de LC-SRM para análises qualitativas de amostras biológicas. Se o péptido alvo biológico derivado de amostra é detectada, padrões peptídicos internos pesados marcado purificados são preparados por LC-quantitativa SRM. Este protocolo pode ser utilizado para accurately quantificar proteínas a partir de uma grande variedade de amostras biológicas, e para apoiar as investigações de uma grande variedade de alvos de proteína.
Protocolo
NOTA: Este método foi descrito anteriormente 56.
1. Peptide seleção de alvos
- Compilar uma lista das proteínas alvo, e incluem um pequeno número de proteínas de limpeza para a normalização entre amostras biológicas, e também incluir um padrão interno de proteínas (por exemplo, luciferase de pirilampo). Digerir as proteínas-alvo em peptídeos trípticos in silico usando uma ferramenta de software, como a proteína digestão Simulator 29,30.
- Exigir que os peptídeos são totalmente tríptico e não contêm locais de clivagem de tripsina em falta. Evitar péptidos com locais de clivagem de tripsina vizinhos para evitar a digestão de tripsina potencialmente incompleto 31.
- Exigem que o comprimento de cada péptido é 5-20 resíduos. Considere péptidos mais longos, mas note que eles são geralmente mais caros de sintetizar.
- Evitar um péptido se ele corresponde a uma variante genética natural (por exemplo, um único nucleotidpolimorfismo e). Também confirmam que os sites de tripsina não são afetados (Passo 1.2).
- Evite um peptídeo se corresponder a um local de modificação pós-tradução (PTM). Da mesma forma, evitar um péptido se que seria propenso a modificação química durante o LC-MS a preparação da amostra. Especificamente, evitar péptidos propensas à cisteína e à oxidação de metionina, asparagina e glutamina desamidação, e a formação de glutamina no terminal amino de piroglutamato. Confirme que os locais de tripsina não são afetados (Passo 1.2).
- Além disso, evitar a péptidos que se originam a partir de proteínas e amino terminais carboxi terminais da proteína porque são propensos a modificação pós-tradução (perda de metionina amino-terminal e acetilação, e amidação do terminal carboxi).
- Exigir que cada péptido alvo é única para cada proteína alvo. Se isso não for possível, requerem que o péptido é único para um conjunto de isoformas / homólogos. Trate péptidos diferindo apenas pelas leucina / substituição isoleucinacomo se fossem idênticos ao determinar peptídeo singularidade porque estes executam quase idêntica durante LC-SRM.
- Se uma análise do transcriptoma abrangente de todas as amostras biológicas tem sido realizada (por exemplo, ARN-SEQ), determinar péptido usando singularidade em traduções in silico da transcrição sequências 32 em vez de usar todo o proteoma da espécie.
- Exigir que os péptidos alvo são proteotípicos. Identificar os péptidos utilizando espectrometria de massa proteotípicos espingarda 1,2 das amostras biológicas a ser estudada. Alternativamente, recuperar identificações peptídicas de péptidos proteotípicos de bancos de dados de proteômica online, como a espectrometria de massa NIST bibliotecas de 33 (http://peptide.nist.gov/), A Máquina Proteome global 34 (X CAÇADOR bibliotecas espectrais de http: // www .thegpm.org / HUNTER / index.html, e GPMDB identificações peptídicos de http://gpmdb.thegpm.org/), PeptideAtlas 35 (http://www.peptideatlas.org/), eo banco de dados PRIDE 36 (http://www.ebi.ac.uk/pride/archive/).
- Se análise quantitativa do nível de transcrição as amostras biológicas foram realizadas (por exemplo, ARN-SEQ, qPCR), em seguida, evitar os péptidos alvo que correspondem aos transcritos relativamente baixa abundância.
- Se necessário, seleccione péptidos alvo que possam ser utilizados para ensaios de LC-SRM de ortólogos proteína alvo (por exemplo, para ensaiar a ambos as formas de ratinho de uma proteína alvo e humano).
- Seleccionar pelo menos dois péptidos alvo por proteína alvo (se possível).
2. Preparação do Péptido Padrões
NOTA: Esta seção do protocolo descreve a preparação de um conjunto de vinte padrões de peptídeos liofilizados (cada um sendo 1 nmol em quantidade) para análises a jusante. Para um número diferente de péptidos, ou em quantidades diferentes de péptidos, que terá de ser ajustado em conformidade.
- Preparar padrões liofilizados peptídicas (≥1 nmol), utilizando uma tecnologia de síntese de peptídeos, tais como SPOT síntese 20,37.
- Assegurar que os resíduos de cisteína de padrões de peptídeos externos são carbamidomethylated (a estrutura química formada por alquilação iodoacetamida). Em contraste, a garantir que os resíduos de cisteína de padrões peptídicos internos são não modificada (estes irão ser alquilado durante a preparação da amostra, descrito abaixo).
- Conceber padrões peptídicos internos de modo a que eles contêm nativa de amino e carboxi-terminal de resíduos que flanqueiam (para controlar a eficiência da digestão de tripsina; cada um é normalmente de três a seis resíduos de comprimento).
- Projetar padrões de peptídeos internos para que eles sejam rotulados isótopo estável. Considere o perfil isotópica natural do péptido alvo e a precisão da medição da massa MS quando se selecciona uma estratégia de rotulagem péptido. Não use padrões de peptídeos deuterados porque causas peptídeo deuterização de fase reversa tempo de retenção LCs para deslocar 38.
Nota: Normalmente, os padrões de péptidos trípticos internos são sintetizados usando ~ 98% puro [13 C 6, 15 N 4] Arg e [13 C 6, 15 N 2] Lys incorporada no carboxi-terminais. - Purificar padrões de peptídeos internos utilizando HPLC 39, e quantificá-los com precisão 40.
- Adicionar 100 ul de 0,1% v / v de ácido fórmico, 20% v / v de acetonitrilo (ACN) a cada 1 nmol padrão péptido liofilizado para produzir uma concentração de péptido de 10 uM (utilização de cuidados de que o péptido liofilizado pode ter uma muito baixa densidade e pode ser facilmente perdido). Agitar as amostras durante 2 min e sonicar banho-los, durante 5 minutos para assegurar que a dissolução está completa péptido.
- Reúnem-se os péptidos dissolvidos, e concentra-se a mistura resultante num concentrador de vácuo a um volume final de 80 ul. Adicionar 20 ul de ACN para dissolver qualquer precipitado de péptido.
Nota: A amostra agora contains 10 uM de cada péptido e 20% v / v de ACN. - Para os padrões de peptídeos internos, preparar uma série de diluição para quantitativa LC-SRM:
- Prepare a 100 ul de uma diluição de 1 uM: combinar 90 ul de 20% v / v de ACN e 10 ul da mistura de péptidos de 10 uM.
- Prepare a 100 ul de uma diluição de 100 nM: combinar 90 ul de 20% v / v de ACN e 10 ul da mistura de péptidos 1 uM.
- Para padrões de péptidos externos, preparar a 100 ul de uma diluição de 1 uM para LC-MS através da combinação de 90 ul de 0,1% v / v de ácido fórmico e 10 ul da mistura de péptidos de 10 uM.
Nota: A amostra agora contém 1 uM de cada péptido, v / v de ácido fórmico a 0,1% e 2% v / v de ACN, e está pronto a ser utilizado para a LC-SRM desenvolvimento do ensaio.
3. LC-SRM Ensaio Desenvolvimento
- Analisar as misturas de péptidos de os padrões externos (cerca de 1-10 pmol de cada péptido por injecção) por shotgun MS usando um HPL nano-flowC sistema acoplado a um espectrômetro de massas triplo quadrupolo (LC-qq-MS (/ MS)).
- Use um sistema LC-MS equipado com uma coluna capilar embalado com C-18 mídia (≤5 mm de diâmetro, ~ 200 poros A, de comprimento ≥10 cm, id = 50-100 mm) e uma ponta nano-ESI (tipicamente produzida usando um laser-extrator). Certifique-se de que cada série de LC-MS inclui um gradiente linear de ~ 60 min (tipicamente, 0-40% de solvente B), um passo de regeneração de coluna (~ 80% de Solvente B), e uma etapa de re-equilibração da coluna (~ 0% de Solvente B ) (solvente A = 0,1% v / v de ácido fórmico em H2O, Solvente B = 0,1% v taxa / v de ácido fórmico em ACN, fluxo = 200-800 nl / min, tensão ESI = 1800 V, largura isolamento Q3 = 0,7 m / z, a pressão de árgon q2 = 1,5 mTorr).
- Para cada verificação de iões precursor, selecione dinamicamente o top ~ 10 íons precursores mais intensos para a espectrometria de massa em tandem (MS / MS). Executar cada amostra em duplicado, de modo que duas rampas de energia de colisão são usados diferentes: um concebido para fragmentar optimamente dois iões precursores, e os outrospara 3 precursor íons 41.
- Realize o LC-qq-MS (/ MS) utilizando análises de varredura ion Q3 precursor (digitalização de iões precursor Q1 pode causar tailing dos picos de iões precursor resultantes de colisões de argônio de baixa energia em Q2).
- Certifique-se de que o sistema LC-MS está executando adequadamente, analisando amostras de CQ e repetições técnicas. Prevenir a mistura de amostras utilizando colunas para LC feitas na hora e executando vários espaços em branco entre as amostras.
- Analisar os espingarda LC-qq-MS (/ MS) os dados resultantes usando banco de dados busca contra as sequências dos padrões de peptídeos externos 42. Se for o caso, descartar identificações peptídicas ambíguas usando os escores de identificação de péptidos de confiança (por exemplo, valores esperados) ou utilizando modelagem estatística 42. Independentemente disso, rever manualmente todas as identificações de péptidos 43 para garantir que todos eles são inequívocas.
- Use a espingarda MS identificações peptídicas para construct uma biblioteca espectro usando um programa de software como o Skyline.
- Prepare uma lista de transição LC-SRM utilizando os 3-10 transições mais intensos por íon precursor (2 e 3 íons precursores; 1 e 2 de fragmentos de íons; Y-, B-, e A-iões que são ≥2 resíduos grandes).
- Descartar uma transição se os valores de precursor e o fragmento de iões m / z sobrepõem dentro da precisão de medição de massa do QQQ-EM (ião precursor de fragmentação é por vezes incompleto; lembrar de considerar o monoisotópico e os isótopos pesados formas naturais, bem como a não marcado e formas heavy-identificadas).
- Da mesma forma, se uma transição descartar o fragmento ião m / z que se sobrepõe de um fragmento ião diferente a partir do mesmo precursor de ião.
- Utilize as listas de transição resultante para efectuar análises de LC-SRM das misturas dos padrões de péptidos externos (executar espectrometria de massa, conforme descrito no Passo 3.1; Q1 e Q3 isolamento largura = 0,7 m / z, tempo de retenção = 2-50 ms).
- Mamãenually rever os dados resultantes LC-SRM e excluir quaisquer ensaios de desempenho insatisfatório (a análise dos dados LC-SRM é descrito na Seção 5).
4. LC-SRM Os ensaios de amostras biológicas
- Preparar os pratos de células cultivadas 44. Se várias condições experimentais estão sendo comparados, bloco e embaralhar as amostras para reduzir eventuais desvios sistemáticos 45.
- Para cada placa de células, aspirar o meio de cultura de células para o lixo e suspender as células num tampão isento de soro 44. Se necessário, lavar as células num tampão isento de soro para remover qualquer proteína extracelular restante.
- Para cada amostra, contar o número de células viáveis e totais utilizando uma mancha de viabilidade (por exemplo, azul de tripano) e um hemocitómetro ou um contador de células automatizado 44.
- Agregar as células por centrifugação, e aspirar os sobrenadantes para o lixo 44 (um volume de sedimento celular típica é de ~ 30 ul).
- Adicionar 400 ul de tampão de lise de ureia ou Tensioactivo tampão de lise a cada sedimento de células (100 mM de HEPES ∙ NaOH a pH 8, 10 uM de bestatina, 10 ^ M de cloridrato de pepstatina A, e ou 8 M de ureia ou 0,1% w / v de agente tensioactivo (respectivamente ); preparados na hora, use um surfactante compatível MS-ácido-lábil, como RapiGest SF ou PPS silenciosa surfactante). Se utilizar outros inibidores da protease, garantir que eles não inibem a tripsina.
- Misture cada amostra utilizando pipetagem suave, e transferir para um tubo de cada 2 ml (com uma tampa de rosca com um anel-O) contendo ~ 100 l de 0,1 milímetros de zircônia / contas de sílica (note que as contas podem causar tubos snap-cap a vazar ). Lisar as células por vórtice as amostras durante 5 minutos à velocidade máxima (isto lisa as células por talão de batimento).
- Alternativamente, lisar as células com um processo mecânico diferente (por exemplo, usando um francês de imprensa ou de homogeneização dicas).
Nota: Evitar centrifugação lisados de células porque este pode sedimentar a proteína precipitada que poderia otherwise tryptically ser digerido e detectado por LC-MS.
- Alternativamente, lisar as células com um processo mecânico diferente (por exemplo, usando um francês de imprensa ou de homogeneização dicas).
- Para as amostras desnaturadas surfactante, incubá-las a 90 ° C durante 10 minutos para auxiliar a homogeneização da amostra e a desnaturação de proteínas.
- Banho sonicar as amostras durante 10 min a temperatura ambiente para ajudar a homogeneização e a desnaturação de proteínas.
Nota: O tampão de lise e os lisados podem ser armazenados a -80 ° C (o tampão de lise será necessário para obter as etapas 4.9 e 4.13). - Realizar um ensaio de concentração de proteína de 46 (por exemplo, um ensaio de ácido bicinconínico) dos lisados (e do tampão de lise como uma experiência de controlo).
- Para as análises qualitativas (ou seja, não há padrões de peptídeos internos serão incrementadas-nas amostras), pipetas 200 mg (massa de proteína) de ligado celular em um novo tubo de microcentrífuga de 1,5 ml. Para análises quantitativas, prepare quatro amostras, para uma série de diluição de isótopos estáveis.
- Para cada amostra, adicionar um padrão de proteína interna (por exemplo </ Em>, adicione ~ 5 pmol de ~ 98% de vaga-lume puro luciferase).
- Para análise quantitativa, adicionar a série de diluição com isótopos estáveis da mistura equimolar dos padrões peptídicos internos (por exemplo, 0, 0,2, 2, 20 pmol de cada peptídeo) com as amostras.
- Em paralelo, preparar amostras de controlo, usando os padrões peptídicos interno sozinho (isto é, sem lisado celular) (por exemplo, 0, 0,2, 2, 20 pmol de cada péptido).
- Também preparar amostras de controlo que utilizam os padrões de péptidos externos e internos (por exemplo: 2 pmoles de cada péptido padrão externo, e 0, 0,2, 2, e 20 pmol de cada péptido padrão interno).
Nota: Certifique-se de que os padrões de peptídeos são livres de ácido fórmico, pois pode interferir com a digestão de tripsina.
- Adicionar tampão de lise, de modo que todas as amostras são volumes idênticos. Nota: Um volume de amostra típico é 70 ul, de modo que este volume irá ser utilizada para este protocolo.
- Reduzir os resíduos de cisteína da proteína por adição de 0.7 ul de DTT 1 M (preparada de fresco) a cada amostra (a concentração final é de DTT 10 mM) e incubou-se as amostras a 60 ° C durante 30 min.
- Cistinas alquilato de proteínas por adição de 7 uL de iodoacetamida tamponada (500 mM de iodoacetamida, 1 M de HEPES ∙ NaOH a pH 8;) preparadas de fresco para cada amostra (a concentração final é de iodoacetamida 50 mM) e a incubação das amostras à temperatura ambiente durante 20 minutos na escuridão (iodoacetamida é sensível à luz). Se necessário, extinguir a iodoacetamida remanescente por adição de DTT até uma concentração final de 50 mM.
- Para cada uma das amostras desnaturadas usando ureia, adicionar 482 ul de 100 mM de HEPES ∙ NaOH a pH 8, de modo que a concentração final é de ureia a 1 M (tripsina é significativamente inibida a> 1 47 M de ureia).
- Tryptically digerir as proteínas em péptidos.
- Para cada amostra, que contém lisado celular, adicionar 8 mL de 0,5 ug de grau de sequenciação / uL de tripsina modificada de modo que a última concentrat tripsinaião é de 1:50 (w proteína: w) de tripsina: amostra, e incuba-se a amostra a 37 ° C durante 18 h.
- Para cada amostra que não contenha lisado celular, adicionar 0,5 mL de 0,5 ug / sequenciação de grau modificado ul de tripsina, e incuba-se a amostra a 37 ° C durante 2 horas (cada uma destas amostras são para experiências de controlo, e contêm tipicamente apenas 10 ng -10 ug de péptido + massa de proteína).
- Para cada uma das amostras desnaturadas ureia, adicionar 440 ul de 2% v / v de ácido fórmico (a concentração final de ácido fórmico é de 1% v / v). Confirme que estas amostras são pH ~ 3.
- Para cada uma das amostras desnaturadas surfactante, adicionar 914 ul de 0,5% v / v de TFA (a concentração final é de TFA a 0,5% v / v). Confirme que estas amostras são pH ~ 1,5. Incubar as amostras a 37 ° C durante 60 minutos para hidrolisar o surfactante.
- Microcentrífuga todas as amostras a 21.000 × g durante 20 min à temperatura ambiente para sedimentar o grupo de cauda e qualquer outro agente tensioactivo precipitates que iria entupir um cartucho de SPE C-18.
- Fase sólida extrair cada um dos sobrenadantes utilizando um C-18 descartável cartucho de SPE (Tampão A = 0,1% v / v de ácido fórmico; tampão B = 0,1% v / v de ácido fórmico, 80% v / v de ACN; ~ 100 mg de C- 18 resina por cartucho). Forçar as fases móveis, através do cartucho de SPE C-18 utilizando uma lâmpada de borracha de superfície plana, usando uma conduta distribuidora de extracção, ou por centrifugação do cartucho num tubo de centrífuga de 15 ml em 10 x g durante 5 min.
- Molhar da coluna por aplicação de 1 ml de Tampão B, equilibrar-lo através da aplicação de 1 ml de Tampão A, duas vezes, aplicar a amostra, e lava-se o cartucho através da aplicação de 1 ml de Tampão A, duas vezes. Elui-se os péptidos através da aplicação de 1 ml de Tampão B lentamente (~ 2 min).
- Concentra-se cada eluído num concentrador de vácuo, para um volume final de 100 ul para evaporar ACN. Adicionar 200 ul de H2O a cada amostra, e concentra-se cada uma em um concentrador de vácuo, para um volume final de 98 ul para evaporar qualquer re possívelmaining ACN.
- Adicionar 2 mL de 5% v / v de ácido fórmico em ACN a cada amostra (as concentrações finais de amostra são de 0,1% v / v de ácido fórmico, a 2% v / v ACN).
Nota: As amostras podem ser armazenadas a -80 ° C. - Analisar as amostras utilizando LC-SRM (como no Passo 3.4). Para as análises qualitativa LC-SRM, execute as amostras biológicas e os padrões de peptídeos externos, e analisar todos os dados resultantes juntos. Para as análises quantitativa LC-SRM, executar a série de diluição isotópica de cada amostra biológica, e também executar os exemplos que consistem nos padrões de peptídeos sozinho, e analisar todos os dados resultantes juntos.
5. Análise de Dados LC-SRM
NOTA: identificação e quantificação Peptide pode ser altamente simplificada e parcialmente automatizados usando softwares como o Skyline, mas ainda é altamente recomendável que todos os dados da anotação ser revistos manualmente. Além disso, é melhor para excluir informações sobre o nível de proteínas durante annot Manualção de dados de LC-SRM para evitar viés.
- Para cada péptido de identificação, confirmar que a forma de cada perfil de eluição de transição é de aproximadamente Gaussiana. Para cada transição, confirmar que o perfil de eluição é o produto dos múltiplos sinais medidos pelo detector MS (isto é, não é o produto de um ou dois pontos aleatórios de ruído MS).
- Para cada péptido de identificação, assegurar que todo o perfil de eluição de péptidos é seleccionada. Evitar ajustar manualmente os limites do perfil de eluição de subcomponentes do péptido de identificação (o péptido pesado forma marcada, a forma não marcada, os iões individuais de precursor, e transições individuais).
- Confirmar que cada perfil de eluição de transição tem uma relação sinal-para-ruído (S / N) ≥3. Nota: "Ruído" refere-se ao ruído aleatório MS, não reprodutíveis sinal de analitos coeluting. Cromatograma funções de suavização pode ajudar muito a análise dos dados ruidosos.
- Para cada péptido de identificação,confirmar que todas as transições têm perfis de eluição quase idênticas (e não apenas vezes vértice do pico iguais; os perfis de eluição podem ter amplitudes diferentes).
- Para as análises de LC-SRM de uma grande quantidade de padrões de péptidos (tipicamente ≥100 fmol de cada péptido), confirmar que os perfis de eluição de péptidos correspondentes são muito intensos (normalmente s / n ≥100), e que as identificações de péptidos não são ambíguas. Nota: Estas identificações altamente confiável serão críticos para a identificação dos péptidos correspondentes derivados em amostras biológicas.
- Para cada identificação de péptidos, confirmar que as intensidades de transição relativos coincidir com os do padrão de peptídeo com confiança identificados. Se necessário, descarte, relativamente baixa intensidade perfis de eluição de transição ruidosos, mas confirmam que nenhuma das transições relativamente intensos estão faltando. Desconsidere perfis de eluição de transição relativamente intensos que são significativamente e de forma inequívoca afetados por uma coeluindo contaminante.
- Para cada identificação de péptidos, estimar a probabilidade de que ele foi produzido aleatoriamente pelo sinal de fundo (ruído aleatório MS acrescido de sinal reprodutível de analitos coeluting).
- Estimar essa probabilidade manualmente, examinando todo o cromatograma LC-SRM e estimar a singularidade de cada identificação de péptidos. Produzir um conjunto de identificações peptídicas confiantes que tem uma taxa estimada falsa descoberta (FDR) de 5%. Use critérios mais rigorosos (por exemplo, FDR ≤1%) se for necessária uma maior confiança conjunto de identificações (não é recomendado o uso de critérios mais flexíveis).
- Em alternativa, usar o algoritmo mProphet 48 ou o algoritmo de Auditoria 49, mas reveja manualmente os resultados.
- Para cada péptido de identificação, confirmam que o tempo de eluição observado é consistente com a hidrofobicidade do péptido (Skyline é concebido para desempenhar este cálculo).
- Para cada péptido, que confirmamseu tempo de eluição é consistente entre os LC é executado.
NOTA: LC-SRM análises, por vezes, produzir perfis de eluição que são sistematicamente enviesado entre as execuções. Desvios sistemáticos são de esperar depois alterações do instrumento, tais como a substituição de uma coluna. Além disso, os peptídeos que eluem cedo pode ser propenso a deslocar (em especial se a coluna é carregada perto de capacidade), e péptidos que eluem final também pode ser propenso a deslocar. Identificação Peptide agora está completa. - Para melhorar a quantificação péptido, inspecionar novamente cada péptido de identificação e descartar um perfil de eluição de transição se é especialmente barulhento em comparação com os outros perfis de eluição de transição do péptido de identificação. Descarte um perfil de eluição de transição, se a sua intensidade de transição relativo é errado (em comparação com o padrão de péptido).
- Inspeccionar os limites de cada um dos perfis de eluição e ião precursor de transição. Se necessário, ajuste com cuidado as fronteiras perfil de eluição para cortar afastado sinal de fundo (ou ifundo ELHORAR estimativa do sinal).
- Para cada padrão de péptido interno, verificar se está contaminado com uma quantidade detectável de péptido luz (determinado por LC-SRM análises dos padrões peptídicos internos sozinho, descrito acima). Posteriormente, descartar quaisquer perfis leves de eluição de péptidos que foram comprometidos de forma significativa pela contaminação de luz peptídeo dentro dos padrões de peptídeos internos.
- Quantificar cada perfil de eluição de transição calculando a sua área de pico LC. Se for caso disso, deduzir o sinal de fundo estimado. Quantificar cada perfil de eluição de péptidos pela soma dos valores de quantificação transição correspondentes (a seguir, isto é referido como o valor "Sum_Peak_Area"). Para cada valor Sum_Peak_Area péptido amostra biológica, calcular a abundância molar de péptido amostra biológica usando dados de LC-SRM de padrões peptídicos internos ou externos (uso apenas as transições que foram quantificados com sucesso tanto pelo péptido amostra biológicaLC-SRM e que o padrão de péptido).
- Quantificação utilizando padrões de peptídeos externos (não recomendado):
- Traçar os valores Sum_Peak_Area padrão peptídicas externo versus os valores de abundância padrão molar peptídeo externo. Produzir uma curva padrão por ajuste de uma regressão linear aos dados (tipicamente, apenas o componente linear da faixa dinâmica devem ser utilizados, e qualquer componente não linear deve ser usado com cuidado extremo). Para cada valor Sum_Peak_Area péptido amostra biológica, usar a curva padrão para calcular a abundância molar de péptido.
Nota: Este método de quantificação não é recomendado porque ele requer que a preparação da amostra e LC-SRM ser demonstrabilidade robusta porque as amostras biológicas e padrões de peptídeos externos são preparadas e analisadas por LC-SRM separadamente. Além disso, ele não tem em conta os efeitos da matriz (efeitos devido a componentes de uma amostra que não seja a substância a analisar).
- Traçar os valores Sum_Peak_Area padrão peptídicas externo versus os valores de abundância padrão molar peptídeo externo. Produzir uma curva padrão por ajuste de uma regressão linear aos dados (tipicamente, apenas o componente linear da faixa dinâmica devem ser utilizados, e qualquer componente não linear deve ser usado com cuidado extremo). Para cada valor Sum_Peak_Area péptido amostra biológica, usar a curva padrão para calcular a abundância molar de péptido.
- Quantificação usando internopadrões de peptídeos (recomendado):
- Para cada valor Sum_Peak_Area peptídeo amostra biológica e correspondente peptídeo interno valor Sum_Peak_Area padrão heavy-rotulados, calcular a razão leve / pesado. Use este rácio como uma medida da / péptido pesado proporção molar abundância luz correspondente para calcular a abundância molar do péptido amostra biológica. Especificamente, a abundância molar peptídeo amostra biológica é igual ao peptídeo interno abundância molar padrão multiplicado pela relação Sum_Peak_Area leve / pesado.
- Para cada replicado e péptido alvo biológico, identificar a gama linear da quantificação LC-SRM, criando uma curva padrão, utilizando uma regressão linear. Especificamente, plotar os valores de abundância molar padrão peptídeo interno versus os valores da relação pesados de luz / Sum_Peak_Area (note que a massa da amostra biológica é constante em todo o conjunto de dados). Exigir que cada amostra valor peptídeo Sum_Peak_Area biológica está dentro da faixa linear (typicaliado, apenas o componente linear da faixa dinâmica deve ser utilizado; qualquer componente não linear deve ser usado com cuidado extremo). Da mesma forma, executar esta etapa para cada transição peptídeo alvo.
Nota: Analyte quantificação por LC-qq-SRM deve ser linear ao longo de uma ampla faixa dinâmica (~ 10000). Na extremidade baixa, sinal de fundo (ruído aleatório MS acrescido de sinal reprodutível de analitos coeluting) irá reduzir a precisão quantificação e precisão. Na parte alta, saturação detector causará linearidade (em última análise, isso pode causar perfis de eluição de partida para ser plana em uma intensidade de sinal específico).
- Quantificação utilizando padrões de peptídeos externos (não recomendado):
- Se apropriado, a uma normalização global em amostras, tais como uma normalização tendência central 50, para corrigir pequenas diferenças na quantidade de amostra, antes da cravação-no dos padrões peptídicos internos. Se necessário, use apenas as proteínas de limpeza do Passo 1.1.
- Se necessário, imputar falta quantification valoriza 51,52.
Nota: É impróprio para simplesmente substituir os valores em falta com a metade do limite inferior de quantificação (LIQ) ou a metade do limite de detecção (LOD), pois isso reduziria artificialmente variância quantificação, e pode resultar em um teste estatístico (por exemplo, uma análise de variância ), produzindo um resultado falso positivo (um erro de tipo I). No entanto, ignorando os valores em falta também pode ser problemática. Por exemplo, se metade dos valores de abundância verdadeiros estão abaixo do LOD, ea outra metade estão acima do LLOQ, em seguida, a média observada abundância seria superestimar a verdadeira abundância dizer. - Certifique-se de que os ensaios satisfazer as orientações amplamente aceitas para LC-SRM 23-25. Notavelmente, exigem que o coeficiente de variação dos valores de quantificação é tipicamente ≤25% para ensaios clínicos, e ≤35% para os ensaios clínicos não-25. Para os ensaios de LC-SRM relacionados à saúde e veterinária produtos ou serviços que estão a ser consideradas para a aplicação de regulamentaçãoRoval, exigem que os ensaios de satisfazer os mais estritos requisitos de precisão para estes ensaios: "A precisão determinado em cada nível de concentração não deve exceder 15% do coeficiente de variação (CV), excepto para o LIQ, em que não deve exceder 20% do CV "23,24.
Resultados
O desenvolvimento de modelos computacionais preditivos de vias de transdução de sinal é um dos objectivos fundamentais da Biologia de Sistemas 53. Infelizmente, mesmo para as vias de sinalização que têm sido estudados extensivamente e têm um elevado significado clínico, que ainda não é geralmente possível prever o comportamento quantitativamente via em resposta a perturbações (por exemplo, isto é verdadeiro para a MAPK / ERK 54). Recentemente, uma investigação empregada proteômica direcionados, transcriptomics e modelagem e simulação computacional para estudar a quimiotaxia rato macrófagos via de sinalização 56. O foco da investigação foi esfingosina-1-fosfato quimiotaxia mediada de células RAW 264.7 (uma linha celular do mouse monócitos / macrófagos). Para facilitar a modelagem via, ensaios de LC-SRM foram desenvolvidos e realizados para medir a abundância absoluta das proteínas da via de quimiotaxia dentro de células RAW 264.7. Os valores de abundância resultantes eram usod como parâmetros do modelo de via.
O esquema experimental global (Figura 1) começou com uma lista de proteínas alvo, que incluíam L i2, uma proteína G heterotrimérica α-subunidade. O sucesso do protocolo geral é altamente dependente da selecção de alvos de péptidos que são proteotípicos e quantotypic, tais como YDEAASYIQSK. Misturas de padrões de péptidos externos foram preparadas e analisadas por LC-espingarda QQQ-MS (/ MS). O espectro de massa em tandem YDEAASYIQSK resultante era composto por iões de fragmento, e que tinha um fundo baixo (Figura 2). Os espectros foram usadas para compor listas de alvos LC-SRM contendo os dez íons fragmento mais intensos por íon precursor.
Uma mistura de 409 padrões de péptidos externos ("JPT409") foi analisada em triplicado por qualitativa LC-SRM (dados não mostrados). Esta amostra incluídos três péptidos i2 G, e a identificação de todos os três erajulgados para estar confiante. Subsequentemente, RAW 264.7 (amostras réplicas biológicas "raw1" e "raw2") e a amostra foram JPT409 cada analisadas em duplicado (duas repetições técnicas LC-SRM) qualitativa por LC-SRM (estes seis LC-SRM análises foram realizadas de forma semelhante como a possível). O péptido foi YDEAASYIQSK confiança identificada em todas as seis análises (Figuras 3A - C). Os padrões de intensidade de transição YDEAASYIQSK foram consistentes ao longo analisa a seis LC-SRM, e estes foram mais ou menos semelhante à da espingarda LC-MS (/ MS) padrão (a "biblioteca" de replicar Figura 3C).
O padrão de intensidades de transição por si só não é sempre suficiente para a identificação de um péptido confiante. A hidrofobicidade péptido e medido o tempo de retenção LC deve ser consistente. Além disso, as normas de péptidos externos e os péptidos correspondentes de amostras biológicas devem ter, aproximadamente,tempos de retenção iguais. A hidrofobicidade (estimada utilizando a versão 3.0 SSRCalc 100 algoritmo A 55) e tempo de retenção observado de YDEAASYIQSK foram encontrados para ser consistente (Figura 4A). Além disso, o tempo de retenção YDEAASYIQSK foi medida utilizando ambos os RAW 264.7 análises e as análises dos padrões de péptidos externos, e todos os valores de tempo de retenção foram aproximadamente iguais (Figura 4B).
A maior parte do qualitativa LC-SRM análises das amostras biológicas foram bem sucedidas, de modo correspondente pesada marcado, purificado, quantificado padrões peptídicos internos foram preparadas e adicionadas em RAW-264.7 lisados celulares. Isótopos série de diluições LC-SRM foi utilizado para analisar amostras que consistem em os padrões peptídicos interno sozinho (amostra "R0"), o interno e os padrões de péptidos externos (amostra "R1"), e seis réplicas biológicas RAW 264.7 (amostras "R2" - "R7"). Dois different desnaturantes de proteínas foram utilizadas para testar possíveis problemas de solubilização da proteína, desnaturação, alquilação e / ou de digestão que possam comprometer a quantificação exacta proteína alvo (amostras R2-R4 utilizada ureia, e R5-R7 amostras usadas RapiGest SF). Os leves e pesados formas de YDEAASYIQSK continha padrões de intensidade de transição quase idêntico e perfis de eluição (Figuras 5A - C). LC-SRM resumida das proporções de áreas de pico foram usadas como uma medida da abundância relativa de péptido, e os rácios YDEAASYIQSK foram usadas para calcular valores de abundância G i2 em unidades de cópias por células RAW 264.7. Em paralelo, uma segunda G i2 padrão peptídeo interno (IAQSDYIPTQQDVLR) foi utilizado para realizar ensaios G i2 LC-SRM quantitativas dos mesmos RAW 264.7 amostras, e os dois ensaios produzidos medições abundância G i2 altamente semelhantes em todas as réplicas biológicas (Figura 5D). O acordo dos dois G ensaios i2 é uma forte evidência de que ambos os péptidos são quantotypic, e que todos os L i2 ensaios quantitativos LC-SRM foram exactos e precisos.
Em geral, 35 proteínas foram quantificadas usando 58 padrões peptídicos internos (Tabela 1). Nomeadamente, os ensaios de LC-SRM do padrão interno de proteína (a luciferase do pirilampo; 4.11) Etapa foram exactos e precisos. O conjunto abrangente de dados Skyline do presente inquérito está disponível no banco de dados LC-SRM on-line Panorama (na pasta Manes_RAW_Chemotaxis em https://panoramaweb.org/labkey/project/NIH_NitaLazar/begin.view).
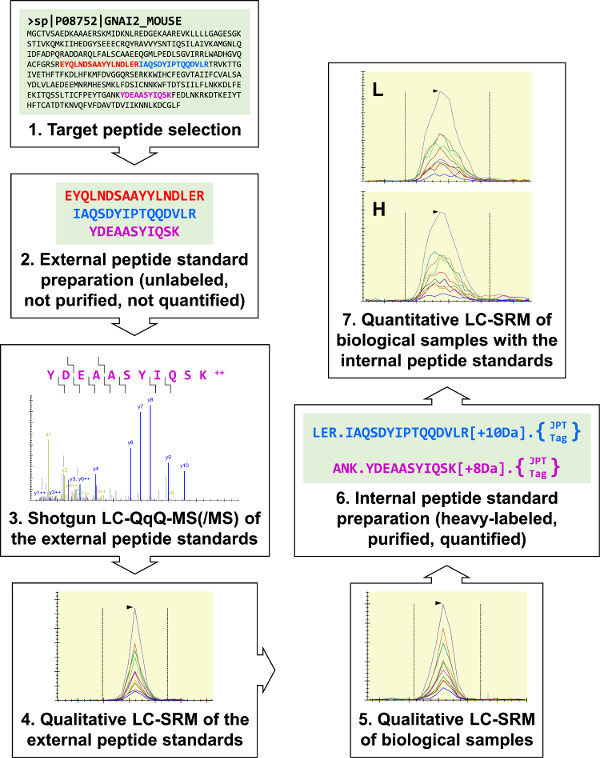
Figura 1:. Descrições dos protocolo Três péptidos alvo tentativas para G I2 (uma proteína G heterotrimérica α-subunidade) foram seleccionados para a síntese de péptido padrão externo. Estes foram Analyzado por espingarda LC-MS (/ MS) para desenvolver três L i2 ensaios LC-SRM, e estes ensaios foram usados para realizar análises qualitativas de amostras biológicas. Foram identificados os três péptidos G-alvo i2, e dois foram selecionados para a preparação padrão peptídeo interno. Foram usadas tripsina cliv�el "JPT-Tag" para quantificar os padrões de peptídeos internos utilizando espectrofotometria UV. Os padrões de peptídeos internos foram usadas para executar G i2 ensaios LC-SRM quantitativos de RAW 264.7 amostras. Por favor clique aqui para ver uma versão maior desta figura.
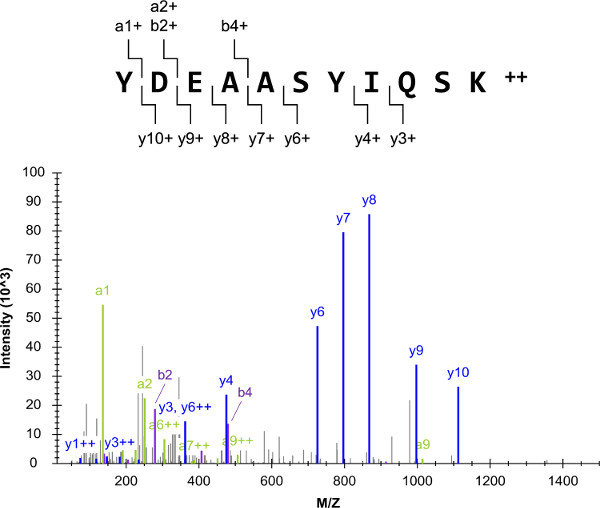
Figura 2:. Espingarda LC-MS (/ MS) O espectro de massa representada originado a partir das análises de uma das i2 padrões de péptidos externos G (YDEAASYIQSK). Para este precursorion, os dez melhores transições mais intensos foram selecionados para LC-SRM (excluindo o íon a1 1+ fragmento devido à sua curta duração). Por favor clique aqui para ver uma versão maior desta figura.
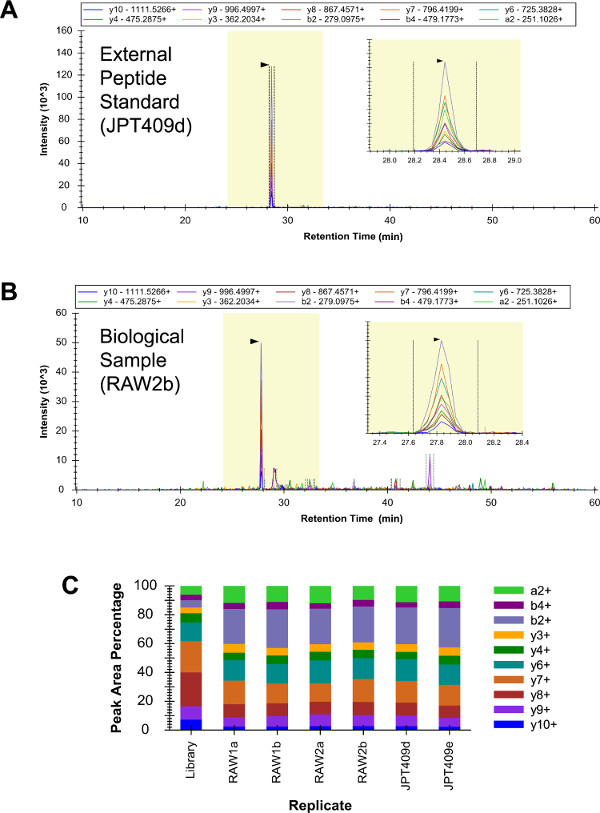
Figura 3:. Qualitativa LC-SRM de L i2 O cromatograma YDEAASYIQSK do JPT409 análises continha um fundo muito baixo e o péptido foi identificado com confiança (A). As análises RAW 264.7 correspondentes resultou em mais sinal de fundo, mas a identificação do péptido foi ainda não ambígua (B). Os padrões de intensidade de transição relativos foram consistentes em todos os seis LC-SRM análises (duas repetições técnicos de três amostras independentes), e estes foram aproximadamente simiLar para a espingarda correspondente LC-MS (/ MS) padrão (a "Library" repetição) (C). Por favor clique aqui para ver uma versão maior desta figura.

Figura 4: previsão de tempo de retenção e o Péptido variação entre corridas Uma regressão linear foi calculada usando a hidrofobicidade estimado e medido LC-SRM tempo de eluição qualitativa de todos os péptidos alvo, e os tempos de eluição previstos e medidos de YDEAASYIQSK eram consistentes (A). . Além disso, a consistência do tempo de eluição observado de cada péptido em toda a LC-SRM análises foi determinada. Os tempos de eluição de YDEAASYIQSK abrangeram uma gama de ~ 40 segundos, o que foi consistente com a precisão do instrumento LC-SRM que foi usada (valoressão o horário de pico ápice +/- a largura total a meia-max, e também a toda a largura na base do pico) (B). Por favor clique aqui para ver uma versão maior desta figura.
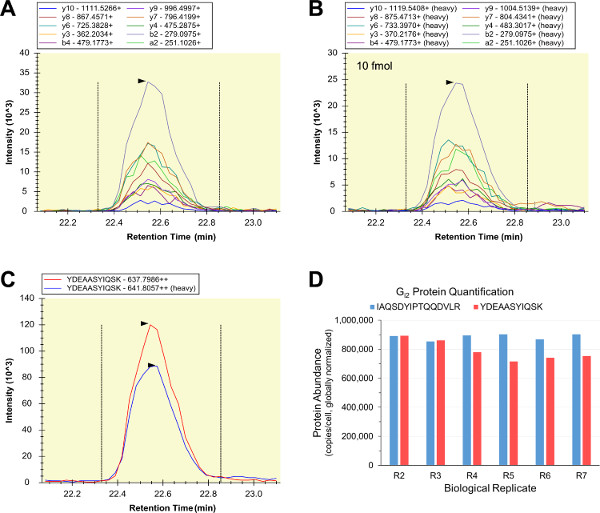
Figura 5:. Quantitativa LC-SRM de L i2 Os cromatogramas de transição do péptido amostra biológica (A) e o péptido padrão interno (B) têm intensidades relativas consistentes de transição, e foram somados (C) (representada é a análise de "R2" para YDEAASYIQSK usando 10 fmol padrão peptídeo interno). Os perfis de eluição leves e pesadas eram consistentes (C), e a razão entre a área sob estas curvas foi usado como uma medida da light / abundância péptido pesado. Um segundo péptido L i2 padrão interno (IAQSDYIPTQQDVLR) foi utilizado para quantitativa LC-SRM em paralelo, e os valores de abundância L i2 resultantes foram consistentes em ambas as seis réplicas biológicas e os dois péptidos alvo (totais, n = 12 e CV = 8,38 %) (D). Por favor clique aqui para ver uma versão maior desta figura.
| UniProt Adesão | Proteína alvo | Alvo Peptide | Abundância (fmol / mg) | Abundância (cópias / célula, normalizado) | CV |
| P08659 | Luciferase | (UV a 280 nm) | 23,824 | n / D | n / D |
| P08659 | Luciferase | VVDLDTGK | 24,103 | n / D | 7% |
| P08659 | Luciferase | VVPFFEAK | 24,717 | n / D | 4% |
| P60710 | Actina, 1 citoplasmática | IWHHTFYNELR | 760,448 | 61762598 | 3% |
| P16858 | GAPDH | LISWYDNEYGYSNR | 357,803 | 28906524 | 14% |
| P06151 | Lactato desidrogenase | LLIVSNPVDILTYVAWK | 129,623 | 10633145 | 30% |
| P99024 | Tubulin β 5 | ALTVPELTQQVFDAK | 78,765 | 6398971 | 3% |
| P20152 | Vimentin | SLYSSSPGGAYVTR | 121,100 | 9807488 | 10% |
| P60766 | CDC42 | DDPSTIEK | 86,647 | 6957317 | 27% |
| P60766 | CDC42 | QKPITPETAEK | 26,475 | 2153669 | 6% |
| Q8C3J5 | DOCK2 | ETLYETIIGYFDK | 1.459 | 118539 | 15% |
| Q8C3J5 | DOCK2 | ISSSPTHSLYVFVR | 1.795 | 143440 | 33% |
| Q8BPU7 | ELMO1 | ALTTKPSSLDQFK | 2.302 | 186754 | 7% |
| Q8BPU7 | ELMO1 | SAIDISILQR | 1.244 | 99.728 | 25% |
| Q8BGM0 | FGR | GAYSLSIR | 1.099 | 89.473 | 17% |
| Q8BGM0 | FGR | WTAPEAALFGR | 0,319 | 24.766 | 6% |
| P27601 | Gu 13 | GIHEYDFEIK | 0,843 | 68467 | 15% |
| P27601 | Gu 13 | VFLQYLPAIR | 1.295 | 106176 | 23% |
| P08752 | Ga (i) 2 | IAQSDYIPTQQDVLR | 10.900 | 885701 | 2% |
| P08752 | Ga (i) 2 | YDEAASYIQSK | 9,774 | 790616 | 9% |
| Q9DC51 | Ga (k) | EYQLNDSASYYLNDLDR | 6,574 | 537862 | 15% |
| Q9DC51 | Ga (k) | ISQTNYIPTQQDVLR | 3.504 | 285784 | 10% |
| P62874 | Gβ 1 | AGVLAGHDNR | 17,078 | 1385578 | 4% |
| Q9CXP8 | Gγ 10 | DALLLGVPAGSNPFR | 2.167 | 179178 | 36% |
| P63213 | Gγ 2 | EDPLLTPVPASENPFR | 3,284 | 266360 | 8% |
| Q80SZ7 | Gγ 5 | VSQAAADLK | 6,087 | 493512 | 6% |
| P08103 | HCK | GPVYVPDPTSSSK | 1.143 | 93.044 | 12% |
| P08103 | HCK | IIEDNEYTAR | 0,944 | 77.147 | 19% |
| P43406 | Integrina α V | AGTQLLAGLR | 0,276 | 22.264 | 32% |
| P43406 | Integrina α V | SHQWFGASVR | 0,443 | 34.235 | 5% |
| P25911 | LYN | VIEDNEYTAR | 1.461 | 119377 | 13% |
| Q5SW28 | PI3K 5 regulamentar | AGFPGILDTASPGK | 0,301 | 24.331 | 11% |
| Q8K3B3 | PI3K α regulamentar | LYEEYTR | 0,472 | 38.592 | 16% |
| Q8K3B3 | PI3K α regulamentar | TWNVGSSNR | 0,524 | 42.697 | 12% |
| Q8K3B3 | PI3K α regulamentar | VLSEIFSPVLFR | 0.440 | 35.902 | 20% |
| Q5U3K7 | PI3K β regulamentar | DTPDGTFLVR | 0,188 | 15.262 | 30% |
| Q5U3K7 | PI3K β regulamentar | IAEIHESR | 0,282 | 22.561 | 13% |
| Q0VGQ5 | Α PI3K | LINLTDILK | 0,102 | 8494 | 38% |
| Q8CI98 | PI3K δ | HEVQEHFPEALAR | 0,178 | 14.566 | 22% |
| Q8CI98 | PI3K δ | ITEEEQLQLR | 0,481 | 38.709 | 24% |
| Q9ES52 | PIP3 5-fosfatase 1 | IVVLAKPEHENR | 0,486 | 39.194 | 19% |
| Q9ES52 | PIP3 5-fosfatase 1 | LSQLTSLLSSIEDK | 2.056 | 167123 | 7% |
| Q69ZK0 | Rac 1 GEF-dependente PIP3 | DSVLSYTSVR | 0,647 | 52.712 | 32% |
| Q69ZK0 | Rac 1 GEF-dependente PIP3 | NQLLLALLK | 0,354 | 27.425 | 5% |
| Q9CQE5 | RGS 10 | ASSQVNVEGQSR | 2.460 | 199782 | 4% |
| Q9CQE5 | RGS 10 | WASSLENLLEDPEGVQR | 20,647 | 206005 | 11% |
| Q9CX84 | RGS 19 | AEANQHVVDEK | 0,495 | 39.753 | 21% |
| Q9CX84 | RGS 19 | LIYEDYVSILSPK | 0,846 | 68.481 | 22% |
| B9EKC3 | Rho GAP 5 | DGLAQELANEIR | 0,448 | 34.653 | 10% |
| Q99PT1 | Rho GDI 1 | SIQEIQELDK | 3.156 | 267063 | 16% |
| Q99PT1 | Rho GDI 1 | VAVSADPNVPNVIVTR | 37,077 | 3006168 | 5% |
| Q61599 | Rho GDI 2 | LNYKPPPQK | 37,975 | 3086596 | 3% |
| Q61599 | Rho GDI 2 | YVQHTYR | 21,436 | 1711908 | 47% |
| Q61210 | Rho GEF 1 | FDGAEGSWFQK | 2.149 | 176139 | 47% |
| Q61210 | Rho GEF 1 | SGLELEPEEPPGWR | 2.911 | 236483 | 8% |
| Q9QUI0 | RhoA | QVELALWDTAGQEDYDR | 43.500 | 3532438 | 8% |
| P70336 | Rock2 | GAFGEVQLVR | 0,527 | 42.800 | 23% |
| P70336 | Rock2 | IYESIEEAK | 1.011 | 83.794 | 33% |
| P70336 | Rock2 | LEGWLSLPVR | 0,573 | 48.259 | 22% |
| Q8R0X7 | S1P lyase 1 | AGYPLEKPFDFR | 1.787 | 147043 | 27% |
| Q8R0X7 | S1P lyase 1 | TPEIVAPESAHAAFDK | 3,562 | 291791 | 18% |
Tabela 1: LC-SRM de RAW 264,7 proteínas celulares quantitativos Trinta e cinco proteínas de células RAW 264.7 foram quantificados utilizando Cinqüenta e oito padrões de peptídeos internos e seis repetições biológicas.. Cinco das proteínas alvo foram arrumação proteínas (actina, GAPDH, lactato desidrogenase, tubulina, e vimentina), e foram quantificados para permitir a normalização através de amostras biológicas (Passo 1.1). Além disso, uma proteína padrão interno foi cravado-em cada ligado de células e quantificado por LC-SRM (4,765 pmol de luciferase do pirilampo por 200 g de amostra; 98% pura por SDS-PAGE; quantificado espectrofotometricamente a 280 nm; Passo 4,11). Os valores de CV foram calculadas entre os seis repetições biológicos utilizando os valores de abundância globalmente normalizados (exceto para luciferase; Passo 5,15).
Discussão
Quantificação absoluta de proteína é essencial para uma gama muito diversificada de aplicações biomédicas tais como validação de biomarcadores e modelagem via de transdução de sinal. Recentemente, proteômica direcionados utilizando LC-SRM beneficiou de melhorias para várias tecnologias, incluindo a preparação de péptidos padrão, HPLC, qq-MS, e análise de dados LC-SRM. Por conseguinte, tornou-se uma alternativa poderosa para imunoensaios. Imunoensaios podem ser extremamente sensíveis e de alto rendimento, mas o desenvolvimento de um imunoensaio robusta pode ser extremamente desafiador porque imunoensaios podem ser vulneráveis a reactividade cruzada e / ou interferência, incompatível com células / tecidos métodos de lise / homogeneização, e / ou não passíveis de multiplexação 5,8. Por exemplo, o teste mais abrangente para reatividade cruzada é realizar o imunoensaio utilizando amostras que originaram de nocautes gene, que pode ser um desafio para se preparar.
Este protocolo descreve Pept alvoseleção ide, LC-SRM desenvolvimento do ensaio, qualitativa e quantitativa LC-SRM, e análise de dados LC-SRM. Ele foi usado para medir a abundância absoluta das proteínas da via 36 de quimiotaxia dentro RAW 264.7 56, mas sua aplicabilidade se estende muito para além desta aplicação específica. Apesar de ter sido concebido para quantificar proteínas dentro de peletes de células, pode ser ajustada para a análise de outras amostras biológicas (por exemplo, Biofluids) e outros alvos SRM (por exemplo, fosfopéptidos). Por exemplo, alterando o protocolo de homogeneização e / ou proteína de digestão pode melhorar significativamente a solubilização, a desnaturação, a alquilação, a digestão, e a quantificação de proteínas alvo especialmente difíceis (por exemplo, proteínas de membrana), ou pode permitir a análise de amostras especialmente exigentes (por exemplo, amostras contendo <100 mg de massa de proteína).
A selecção inicial de péptidos alvo é crítico mas pode ser demorado. Para hundreds de proteínas alvo, uma pontuação pontual pode ser utilizada para cada critério, e os péptidos alvo experimentais podem ser classificados com base na soma das pontuações, tal como foi feito anteriormente 56. Em alternativa, esta análise pode ser automatizado usando PeptidePicker, uma interface web que simplifica muito a seleção peptídeo meta de 30 (http://mrmpeptidepicker.proteincentre.com/).
Depois de os péptidos alvo foram seleccionados e os ensaios de LC-SRM têm sido desenvolvidos, é fundamental que os ensaios qualitativos LC-SRM das amostras biológicas ser executada porque mesmo um péptido altamente proteotípicos quantotypic e não será detectável se a proteína é expressa em níveis abaixo do limiar de sensibilidade do instrumento, ou se a interferência de fundo é especialmente problemática. Uma segunda dimensão de separação (por exemplo, HPLC de permuta catiónica forte, de alto pH HPLC de fase inversa, electroforese e isento de gel) pode aumentar a profundidade proteómica, mas irá exigirsignificativamente mais tempo do instrumento e análise de dados. Alternativamente, uma estratégia de enriquecimento (por exemplo, immunoenrichment p�tido e de nível de proteína e o fraccionamento de células) podem melhorar a profundidade proteómica, e imunodepleção de proteínas altamente abundantes pode ser usado para reduzir a interferência por coeluting analitos.
LC-SRM do ~ dezenas de amostras para ~ centenas ou milhares de transições ~ normalmente requer tempo extenso instrumento. Embora não tenha sido utilizado para este estudo, LC-SRM agendamento (medindo transições durante a eluição janelas de tempo pré-especificado) permite a análise de mais transições por corrida. Além disso, reduz o ciclo de programação dever SRM durante períodos relativamente vagos do gradiente de LC, e isto pode resultar na melhoria da identificação e quantificação péptido. No entanto, a programação requer que o tempo de eluição de péptidos com confiança ser determinada a partir de uma análise por LC-SRM qualitativa. Mudanças sutis para a preparação da amostra, o instrumento LC-MS,ou o método LC-MS pode causar agendamento para cortar ou até inteiramente perca peptídeos alvo. Por exemplo, os perfis de eluição da amostra YDEAASYIQSK biológicos foram ligeiramente deslocados em relação às do péptido padrão externo (Figura 4B), possivelmente devido a efeitos de matriz.
Em resumo, um protocolo passo-a-passo é apresentado para o desenvolvimento e aplicação de LC-SRM para a quantificação absoluta de proteína. Quantificação absoluta de proteínas por LC-SRM já foi demonstrado ser reprodutível entre laboratórios 16,19. Proteómica tecnologias, incluindo a preparação da amostra (por exemplo, automação), cromatografia líquida, espectrometria de massa e análise de dados, estão a melhorar rapidamente, e está permitindo LC-SRM para se tornar prático para investigação em grande escala e aplicações clínicas. A especificidade, sensibilidade, precisão, reprodutível e de alta taxa de transferência de quantitativo LC-SRM torna uma ferramenta poderosa para a pesquisa básica e biomedicina.
Divulgações
The authors have nothing to disclose.
Agradecimentos
This research was supported by the Intramural Research Program of the NIH, National Institute of Allergy and Infectious Diseases.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| BCA (bicinchoninic acid) protein assay kit | Fisher | 23235 | |
| Beads for bead beating, zirconia-silica, 0.1 mm | BioSpec Products | 11079101z | |
| Bestatin hydrochloride | Sigma | B8385-10MG | |
| Cell culture DMEM (with glucose, without L-glutamine) | Lonza | 12-614F | |
| Cell culture EDTA, 500 mM, pH8 | Gibco | 15575 | |
| Cell culture fetal bovine serum (FBS) | Atlanta Biologicals | S11550 | |
| Cell culture L-glutamine | Sigma | G8540-25G | |
| Cell culture phosphate buffered saline (PBS) pH 7.4 | Gibco | 10010-049 | |
| Cell culture Trypan Blue viability stain, 0.4% w/v | Lonza | 17-942E | |
| Cellometer Auto T4 cell counter | Nexcelom Bioscience | Cellometer Auto T4 | |
| Cellometer Auto T4 disposable counting chambers | Nexcelom Bioscience | CHT4-SD100-014 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Formic acid, LC-MS grade, ampules | Fisher | A117-10X1AMP | |
| Hemocytometer, Neubauer-improved, 0.1 mm deep | Marienfeld-Superior | 0640030 | |
| HEPES, 1 M, pH 7.2 | Mediatech | 25-060-CI | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Laser Based Micropipette Puller | Sutter Instrument Co. | P-2000 | |
| LC coated silica capillary, 50 µm id | Polymicro Technologies | 1068150017 | |
| LC vial, autosampler, 12 mm x 32 mm polypropylene | SUN SRI | 200-268 | |
| LC vial screw cap, autosampler, pre-slit PTFE/silicone | SUN SRI | 500-061 | |
| Luciferase, from Photinus pyralis | Sigma | L9506-1MG | |
| Pepstatin A | EMD Millipore | 516481-25MG | |
| pH strips colorpHast (pH 0.0-6.0) | EMD Chemicals | 9586-1 | |
| PhosStop phosphatase inhibitor cocktail | Roche | 04906837001 | |
| RapiGest SF | Waters | 186001861 | |
| Sep-Pak SPE, C18 1 ml 100 mg cartridge | Waters | WAT023590 | |
| Sep-Pak SPE, extraction manifold, 20 position | Waters | WAT200609 | |
| Sep-Pak SPE, flat-surfaced rubber bulb | Fisher | 03-448-25 | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| SpeedVac vacuum concentrator | Fisher | SPD111V | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Tube decapper for Micronic tubes | USA Scientific | 1765-4000 | |
| Tubes, 2 ml microcentrifuge, o-ring screw-cap, sterile | Sarstedt | 72.694.006 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS grade | Fisher | W6-1 |
Referências
- Cox, J., Mann, M. Quantitative high-resolution proteomics for data-driven systems biology. Annu Rev Biochem. 80, 273-299 (2011).
- Zhang, Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. 3rd Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 113, 2343-2394 (2013).
- Boja, E. S., Rodriguez, H. Mass spectrometry-based targeted quantitative proteomics: achieving sensitive and reproducible detection of proteins. Proteomics. 12, 1093-1110 (2012).
- Gillette, M. A., Carr, S. A. Quantitative analysis of peptides and proteins in biomedicine by targeted mass spectrometry. Nat Methods. 10, 28-34 (2013).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nat Methods. 9, 555-566 (2012).
- Lesur, A., Domon, B. Advances in high-resolution accurate mass spectrometry application to targeted proteomics. Proteomics. , (2015).
- Wild, D. The immunoassay handbook : theory and applications of ligand binding ELISA., and related techniques. , 4th edn, Elsevier. (2013).
- Sturgeon, C. M., Viljoen, A. Analytical error and interference in immunoassay: minimizing risk. Ann Clin Biochem. 48, 418-432 (2011).
- Adrait, A., et al. Development of a Protein Standard Absolute Quantification (PSAQ) assay for the quantification of Staphylococcus aureus enterotoxin A in serum. J Proteomics. 75, 3041-3049 (2012).
- Lin, D., Alborn, W. E., Slebos, R. J., Liebler, D. C. Comparison of protein immunoprecipitation-multiple reaction monitoring with ELISA for assay of biomarker candidates in plasma. J Proteome Res. 12, 5996-6003 (2013).
- Weiss, F., et al. Catch and measure-mass spectrometry-based immunoassays in biomarker research. Biochim Biophys Acta. 1844, 927-932 (2014).
- Yassine, H., et al. Mass spectrometric immunoassay and MRM as targeted MS-based quantitative approaches in biomarker development: potential applications to cardiovascular disease and diabetes. Proteomics Clin Appl. 7, 528-540 (2013).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. , (2011).
- Becker, J. O., Hoofnagle, A. N. Replacing immunoassays with tryptic digestion-peptide immunoaffinity enrichment and LC-MS/MS. 4, 281-290 (2012).
- Wasinger, V. C., Zeng, M., Yau, Y. Current status and advances in quantitative proteomic mass spectrometry. Int J Proteomics. 2013, 180605(2013).
- Abbatiello, S. E., et al. Large-scale inter-laboratory study to develop, analytically validate and apply highly multiplexed, quantitative peptide assays to measure cancer-relevant proteins in plasma. Mol Cell Proteomics. , (2015).
- Rodriguez-Suarez, E., Whetton, A. D. The application of quantification techniques in proteomics for biomedical research. Mass Spectrom Rev. 32, 1-26 (2013).
- Wehr, A. Y., Hwang, W. T., Blair, I. A., Yu, K. H. Relative quantification of serum proteins from pancreatic ductal adenocarcinoma patients by stable isotope dilution liquid chromatography-mass spectrometry. J Proteome Res. 11, 1749-1758 (2012).
- Kennedy, J. J., et al. Demonstrating the feasibility of large-scale development of standardized assays to quantify human proteins. Nat Methods. 11, 149-155 (2014).
- Jensen, K. J., Shelton, P. T., Pedersen, S. L. Peptide synthesis and applications. , 2nd edn, Humana Press Springer. (2013).
- Pratt, J. M., et al. Multiplexed absolute quantification for proteomics using concatenated signature peptides encoded by QconCAT genes. Nat Protoc. 1, 1029-1043 (2006).
- Brun, V., et al. Isotope-labeled protein standards: toward absolute quantitative proteomics. Mol Cell Proteomics. 6, 2139-2149 (2007).
- Guidance for Industry: Bioanalytical Method Validation. , U.S. Food and Drug Administration. Available from: http://www.fda.gov/ucm/groups/fdagov-public/@fdagov-drugs-gen/documents/document/ucm070107.pdf (2001).
- Guidance for Industry: Bioanalytical Method Validation. , U.S. Food and Drug Administration; Revision 1 (draft). Available from: http://www.fda.gov/ucm/groups/fdagov-public/@fdagov-drugs-gen/documents/document/ucm368107.pdf (2013).
- Carr, S. A., et al. Targeted peptide measurements in biology and medicine: best practices for mass spectrometry-based assay development using a fit-for-purpose approach. Mol Cell Proteomics. 13, 907-917 (2014).
- Ori, A., Andres-Pons, A., Beck, M. The use of targeted proteomics to determine the stoichiometry of large macromolecular assemblies. Methods Cell Biol. 122, 117-146 (2014).
- Rosen, O., Feldberg, L., Gura, S., Zichel, R. A new peptide substrate for enhanced botulinum neurotoxin type B detection by endopeptidase-liquid chromatography-tandem mass spectrometry/multiple reaction monitoring assay. Anal Biochem. , (2015).
- Feng, Y., et al. Global analysis of protein structural changes in complex proteomes. Nat Biotechnol. 32, 1036-1044 (2014).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Mohammed, Y., et al. PeptidePicker: a scientific workflow with web interface for selecting appropriate peptides for targeted proteomics experiments. J Proteomics. 106, 151-161 (2014).
- Rodriguez, J., Gupta, N., Smith, R. D., Pevzner, P. A. Does trypsin cut before proline. J Proteome Res. 7, 300-305 (2008).
- Min, X. J., Butler, G., Storms, R., Tsang, A. OrfPredictor: predicting protein-coding regions in EST-derived sequences. Nucleic Acids Res. 33, W677-W680 (2005).
- Lam, H., et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods. 5, 873-875 (2008).
- Craig, R., Cortens, J. P., Beavis, R. C. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res. 3, 1234-1242 (2004).
- Desiere, F., et al. The PeptideAtlas project. Nucleic Acids Res. 34, D655-D658 (2006).
- Vizcaino, J. A., et al. The PRoteomics IDEntifications (PRIDE) database and associated tools: status in 2013. Nucleic Acids Res. 41, D1063-D1069 (2013).
- Frank, R. The SPOT-synthesis technique. Synthetic peptide arrays on membrane supports--principles and applications. J Immunol Methods. 267, 13-26 (2002).
- Ong, S. E., Kratchmarova, I., Mann, M. Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). J Proteome Res. 2, 173-181 (2003).
- Mant, C. T., et al. HPLC analysis and purification of peptides. Methods Mol Biol. 386, 3-55 (2007).
- Alterman, M. A., Hunziker, P. Amino acid analysis : methods and protocols. , Humana Press. (2012).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Nesvizhskii, A. I. A survey of computational methods and error rate estimation procedures for peptide and protein identification in shotgun proteomics. J Proteomics. 73, 2092-2123 (2010).
- Tabb, D. L., Friedman, D. B., Ham, A. J. Verification of automated peptide identifications from proteomic tandem mass spectra. Nat Protoc. 1, 2213-2222 (2006).
- Freshney, R. I. Culture of animal cells : a manual of basic technique and specialized applications. , 6th edn, Wiley-Blackwell. (2010).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. J Proteome Res. 8, 2144-2156 (2009).
- Noble, J. E., Bailey, M. J. Quantitation of protein. Methods Enzymol. 463, 73-95 (2009).
- Kiser, J. Z., Post, M., Wang, B., Miyagi, M. Streptomyces erythraeus trypsin for proteomics applications. J Proteome Res. 8, 1810-1817 (2009).
- Reiter, L., et al. mProphet: automated data processing and statistical validation for large-scale SRM experiments. Nat Methods. 8, 430-435 (2011).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Callister, S. J., et al. Normalization approaches for removing systematic biases associated with mass spectrometry and label-free proteomics. J Proteome Res. 5, 277-286 (2006).
- Karpievitch, Y. V., Dabney, A. R., Smith, R. D. Normalization and missing value imputation for label-free LC-MS analysis. BMC Bioinformatics. 13, Suppl 16. S5(2012).
- Oh, S., Kang, D. D., Brock, G. N., Tseng, G. C. Biological impact of missing-value imputation on downstream analyses of gene expression profiles. Bioinformatics. 27, 78-86 (2011).
- Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A., Fraser, I. D. Systems biology in immunology: a computational modeling perspective. Annu Rev Immunol. 29, 527-585 (2011).
- Futran, A. S., Link, A. J., Seger, R., Shvartsman, S. Y. ERK as a model for systems biology of enzyme kinetics in cells. Curr Biol. 23, R972-R979 (2013).
- Krokhin, O. V. Sequence-specific retention calculator. Algorithm for peptide retention prediction in ion-pair RP-HPLC: application to 300- and 100-A pore size C18 sorbents. Anal Chem. 78, 7785-7795 (2006).
- Manes, N. P., Angermann, B. R., Koppenol-Raab, M., An, E., Sjoelund, V. H., Sun, J., Ishii, M., Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A. Targeted Proteomics-Driven Computational Modeling of Macrophage S1P Chemosensing. Mol Cell Proteomics. , Forthcoming.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados