Method Article
Reazione selezionato Monitoraggio spettrometria di massa per Absolute Protein Quantificazione
In questo articolo
Riepilogo
This protocol describes how to perform absolute quantification assays of target proteins within complex biological samples using selected reaction monitoring. It was used to accurately quantify proteins of the mouse macrophage chemotaxis signaling pathway. Target peptide selection, assay development, and qualitative and quantitative assays are described in detail.
Abstract
Absolute quantification of target proteins within complex biological samples is critical to a wide range of research and clinical applications. This protocol provides step-by-step instructions for the development and application of quantitative assays using selected reaction monitoring (SRM) mass spectrometry (MS). First, likely quantotypic target peptides are identified based on numerous criteria. This includes identifying proteotypic peptides, avoiding sites of posttranslational modification, and analyzing the uniqueness of the target peptide to the target protein. Next, crude external peptide standards are synthesized and used to develop SRM assays, and the resulting assays are used to perform qualitative analyses of the biological samples. Finally, purified, quantified, heavy isotope labeled internal peptide standards are prepared and used to perform isotope dilution series SRM assays. Analysis of all of the resulting MS data is presented. This protocol was used to accurately assay the absolute abundance of proteins of the chemotaxis signaling pathway within RAW 264.7 cells (a mouse monocyte/macrophage cell line). The quantification of Gi2 (a heterotrimeric G-protein α-subunit) is described in detail.
Introduzione
Esperimenti di proteomica che utilizzano la spettrometria di massa (MS) possono essere progettati per utilizzare non mirati (shotgun) o metodi mirati. Proteomica Discovery genere si basa su fucile bottom-up MS, sia utilizzando una tradizionale modalità di acquisizione dati dipendente, oppure utilizzando una delle tecniche di dati indipendenti recentemente sviluppati (ad esempio, MS E, SWATH) 1,2. Proteomica Shotgun è un potente strumento per l'identificazione peptide high-throughput e relativa quantificazione, ma è generalmente adatto per la quantificazione assoluta o per il targeting piccoli insiemi definiti (~ decine) di proteine. Il metodo MS più spesso utilizzato per proteomica mirati è selezionato monitoraggio reazione (SRM) a causa della sua elevata sensibilità, velocità e gamma dinamica 3-5. Alternative al SRM comprendono il monitoraggio reazione parallela, che sfrutta ad alta risoluzione, scansione MS pieno 6.
SRM è di solito eseguita con un rever nano-flowsed fase cromatografia liquida ad alte prestazioni (nano-RP-LC) strumento accoppiato ad un ionizzazione nano-elettrospray (nano-ESI) sorgente di ioni collegato a uno spettrometro di massa a triplo quadrupolo (QQQ-MS). In un esperimento tipico, proteine del campione sono proteolytically digeriti, e peptidi risultanti vengono cromatograficamente separati, desorbiti e ionizzati. Gli ioni precursori risultanti sono m / z filtrato dal primo quadrupolo (Q1) e frammentata nel secondo quadrupolo (q2) da loro collisione con un gas di collisione. Gli ioni frammento risultante sono m / z filtrata nel terzo quadrupolo (Q3) e quantificata da un dinodi. Ogni coppia precursore e frammento di ioni viene indicato come una transizione, e ogni transizione viene monitorato per un determinato periodo di tempo (il tempo di sosta, in genere 2-50 msec). Durante LC-SRM, i cicli QQQ-MS tramite un elenco predefinito di transizioni (duty cycle è tipicamente ≤3 sec), e un cromatogramma di ogni transizione è prodotto.
Strateg Alternativei per le proteine quantificazione utilizzano in genere saggi immunologici, come macchie di punti, macchie occidentali, ELISA, microarrays dell'anticorpo, di microarrays della proteina di fase inversa, immunodosaggi microfluidica, ELISA digitali e immunologici basati microsfere-7. I migliori immunodosaggi possono essere significativamente più sensibile LC-SRM, e la produttività del campione di immunodosaggi possono essere significativamente superiore a quello di LC-SRM 5. Tuttavia, immunodosaggi sviluppo possono essere costosi e / o di tempo, e le analisi risultanti possono essere vulnerabile a cross-reattività e / o interferenze, compatibile con la cellula / tessuto lisi / metodi di omogeneizzazione, e / o non suscettibili di multiplexing 5,8. Alcuni di questi problemi possono essere affrontati con l'accoppiamento di tecniche anticorpo-e basate su MS. Per esempio, le proteine bersaglio possono essere arricchiti con immunoprecipitazione prima proteolisi e LC-SRM 9-12. In alternativa, la tecnica SISCAPA impiega immunoprecipitazione successiva alla proteolisi al leve peptidel 13,14. Oltre a immunoenrichment strategie, immunodeplezione di proteine elevate abbondanza può essere impiegato per aumentare la sensibilità LC-SRM riducendo interferenze da coeluiscono analiti 15,16.
Proteina quantificazione basata su MS può essere diviso in quantificazione relativa e assoluta, e anche in etichettatura isotopo stabile e priva di etichetta (ad esempio, in materia di etichettatura del metabolismo, l'etichettatura chimica, e proteine pesante marcati e peptide standard interni). Tecniche senza etichetta può essere utile per la proteina relativa quantificazione, ma non sono adatti per un accurato quantificazione assoluta. In confronto, le tecniche di etichettatura hanno ridotto errore associato con la preparazione del campione e MS varianza, e sono spesso utilizzati per la proteina relativa quantificazione 17. Ad esempio, gli standard degli isotopi stabili proteoma marcati (SILAP) preparati con una linea di cellule umane in coltura permesso quantificazione relativa dei potenziali biomarcatori tramite LC-SRM di siero umano 18. Accurate proteina quantificazione assoluta da MS è necessario che le norme interne purificato, quantificato, proteine o peptidi isotopi marcati venire addizionate-in campioni biologici prima della SM. L'incorporazione di isotopo pesante standard interni marcati in un flusso di lavoro LC-SRM consente una quantificazione assoluta che ha dimostrato di essere altamente riproducibile e trasferibili tra i laboratori 16,19.
Isotopo stabile etichettati standard interni per le proteine quantificazione assoluta da MS sono gli standard di peptide preparati con solida sintesi in fase 20, proteine composte da concatenati standard peptide proteasi cleavable 21, e full-length standard proteina 22. Obiettivo modifica proteine covalente e preparazione dei campioni incompleti (vale a dire, la lisi incompleta del campione e l'omogeneizzazione, e incompleta delle proteine solubilizzazione, denaturazione, alchilazione e proteolisi) può minare la quantificazione accurata. P internonorme rotein sono i meno probabilità di essere colpiti dalla maggior parte di questi potenziali problemi, ma di solito sono le più difficili da preparare. Un'alternativa è quella di analizzare ogni proteina bersaglio utilizzando più standard peptide interni che sono progettati per includere i residui di nativi di accompagnamento amino e carbossi-terminale. Indipendentemente da quale è impiegato il tipo di standard interno, occorre spillo-nei campioni biologici fin dalle prime punto durante la preparazione del campione possibile. Inoltre, le tecniche di preparazione dei campioni multipli (ad esempio, diverse condizioni di denaturazione) devono essere testati. L'uso di molteplici tecniche sperimentali ortogonali (cross-validazione sperimentale) è una strategia praticabile per superare più potenziale quantificazione sfide 23-25.
LC-SRM quantificazione delle proteine è una tecnica estremamente flessibile che è stato utilizzato in un'ampia varietà di applicazioni. In particolare, è stato utilizzato per studiare peptidi e proteine biomarker all'internocampioni clinici quali siero, biopsie, e aspirati con ago sottile 5. LC-SRM è stato utilizzato anche per misurare la stechiometria di complessi proteici 5,26, per rilevare neurotossine botuliniche 27, per quantificare la dinamica delle proteine di fosforilazione in vie di segnalazione 5, e di quantificare i cambiamenti di conformazione proteica 28.
Il nostro laboratorio sta usando LC-SRM per quantificare le proteine di segnalazione che mediano la chemiotassi macrofagi per sostenere lo sviluppo di simulazioni chemiotassi pathway. Lo schema generale del protocollo (Figura 1) inizia con classifica i timidi peptidi di destinazione. Successivamente, gli standard di peptide esterni grezzi vengono sintetizzati e utilizzati per sviluppare test LC-SRM per le analisi qualitative dei campioni biologici. Se viene rilevato il biologico campione di derivazione bersaglio peptide, peptide norme interne pesanti marcato purificate sono preparati per quantitativa LC-SRM. Questo protocollo può essere utilizzato per acaccurata- quantificare proteine da un'ampia varietà di campioni biologici, e di agevolare le indagini di un'ampia varietà di proteine target.
Protocollo
NOTA: Questo metodo è stato descritto in precedenza 56.
1. Peptide Target Selection
- Compilare un elenco delle proteine bersaglio, e comprendono un piccolo numero di proteine di pulizia per la normalizzazione di tutti i campioni biologici, e comprendono anche uno standard di proteine interna (ad esempio, la lucciola luciferasi). Digeriscono le proteine bersaglio in peptidi triptici in silico utilizzando uno strumento software come la digestione delle proteine Simulator 29,30.
- Richiedere che i peptidi sono completamente triptica e non contengono siti tripsina scissione mancanti. Evitare di peptidi con vicini siti di rottura della tripsina per evitare potenzialmente incompleta digestione tripsina 31.
- Richiede che la lunghezza di ciascun peptide è 5-20 residui. Considerate peptidi più lunghi, ma è da notare che essi sono generalmente più costosi di sintetizzare.
- Evitare un peptide se corrisponde una variante genetica naturale (ad esempio, un singolo nucleotide polimorfismo). Inoltre confermano che i siti tripsina sono influenzati (punto 1.2).
- Evitare un peptide se corrisponde a un sito di modificazione post-traslazionale (PTM). Analogamente, evitare un peptide se sarebbe soggetta a modificazione chimica durante la loro preparazione LC-MS. In particolare, evitare di peptidi inclini a cisteina e metionina ossidazione, asparagina e glutammina deamidazione, e glutammina formazione amino-terminale di piroglutammato. Verificare che i siti tripsina sono influenzati (Passo 1.2).
- Inoltre, evitare di peptidi che provengono da proteine ammino- e carbossi-termini, perché termini di proteine sono soggetti a modificazione post-traslazionale (perdita metionina amino-terminale e acetilazione, e carbossi-terminale amidazione).
- Richiede che ogni peptide bersaglio è unico per ogni proteina bersaglio. Se questo non è possibile, richiedere che il peptide è unico per un insieme di isoforme / omologhi. Trattare peptidi differiscono solo da leucina / sostituzione isoleucinacome se fossero identici per determinare peptide unicità perché questi eseguono quasi identico durante LC-SRM.
- Se una completa analisi trascrittomica di tutti i campioni biologici è stata eseguita (per esempio, RNA-seq), determinare peptide unicità utilizzando in traduzioni silico della trascrizione sequenze 32 invece di utilizzare l'intero proteoma delle specie.
- Richiedere che i peptidi target sono proteotypic. Identificare peptidi proteotypic utilizzando fucile spettrometria di massa 1,2 dei campioni biologici in fase di studio. In alternativa, recuperare identificazioni peptide di peptidi proteotypic da database proteomica online come la spettrometria di massa NIST librerie 33 (http://peptide.nist.gov/), proteoma macchina Globale 34 (X librerie spettrali HUNTER da http: // www .thegpm.org / HUNTER / index.html e GPMDB peptide identificazioni da http://gpmdb.thegpm.org/), PeptideAtlas 35 (http://www.peptideatlas.org/), e il database PRIDE 36 (http://www.ebi.ac.uk/pride/archive/).
- Se a livello di trascrizione analisi quantitative dei campioni biologici sono state eseguite (per esempio, RNA-seq, qPCR), quindi evitare il targeting peptidi corrispondenti ai trascritti relativamente bassa abbondanza.
- Se necessario, selezionate peptidi bersaglio che potrebbero essere utilizzati per test LC-SRM di bersaglio ortologhi di proteine (ad esempio, per saggiare sia la le forme del mouse di una proteina bersaglio umano e).
- Seleziona almeno due peptidi bersaglio per proteina bersaglio (se possibile).
2. Preparazione del Peptide Standards
NOTA: Questa sezione del protocollo descrive la preparazione di una serie di venti norme peptidici liofilizzati (ciascuna essendo 1 nmol in quantità) per analisi a valle. Per un numero diverso di peptidi, o per le diverse quantità di peptidi, dovrà essere adeguato di conseguenza.
- Preparare gli standard liofilizzati peptide (≥1 nmol) utilizzando una tecnologia di sintesi peptidica come la sintesi SPOT 20,37.
- Assicurarsi che residui di cisteina di standard peptide esterni sono carbamidomethylated (la struttura chimica formata da alchilazione iodoacetamide). Al contrario, garantire che i residui di cisteina di standard peptidici interne non sono modificati (questi saranno alchilata durante la preparazione del campione, descritto di seguito).
- Progettare standard peptide interni in modo che essi contengono nativo ammino- e carbossi-terminale di accompagnamento residui (per controllare l'efficienza tripsina digestione; ciascuno è in genere a lungo 3-6 residui).
- Progettare standard peptidici interne in modo che siano isotopo stabile etichettati. Si consideri il profilo isotopico naturale del peptide bersaglio e la precisione di misura di massa del MS quando si seleziona una strategia di etichettatura peptide. Non utilizzare gli standard del peptide deuterati perché cause peptide deuterazione fase inversa tempo di ritenzione LCs di spostare 38.
Nota: in genere, le norme interne peptide triptici sono sintetizzati con ~ 98% [13 C 6, 15 N 4] Arg e [13 C 6, 15 N 2] Lys costituita a carbossi-terminale. - Purificare standard peptide interni utilizzando HPLC 39, e con precisione le 40 quantificare.
- Aggiungere 100 ml di 0,1% v / v di acido formico, 20% v / v acetonitrile (ACN) per ogni 1 nmol norma peptide liofilizzata per ottenere una concentrazione di 10 mM peptide (uso diligenza peptide liofilizzato può avere una bassa densità e può essere perso facilmente). Vortex i campioni per 2 min e bagno li sonicazione per 5 minuti per assicurare che peptide completa dissoluzione.
- Si riuniscono gli peptidi disciolti, e concentrare la miscela risultante in un concentratore a vuoto ad un volume finale di 80 microlitri. Aggiungere 20 ml di ACN per sciogliere qualsiasi peptide precipitato.
Nota: Il campione ora contains 10 pM di ciascun peptide e il 20% v / v ACN. - Per gli standard peptide interne, preparare una serie di diluizioni per quantitativa LC-SRM:
- Preparare 100 ml di una diluizione 1 mM: unire 90 ml di 20% v / v ACN e 10 microlitri della miscela peptidica 10 pM.
- Preparare 100 ml di una diluizione di 100 nM: unire 90 ml di 20% v / v ACN e 10 microlitri della miscela peptidica 1 pM.
- Per peptide standard esterni, preparare 100 ml di una diluizione 1 pM per LC-MS combinando 90 ml di 0,1% v / v di acido formico e 10 microlitri della miscela peptidica 10 pM.
Nota: Il campione contiene ora 1 pM di ciascun peptide, 0,1% v / v di acido formico, e 2% v / v ACN, ed è pronto per essere utilizzato per lo sviluppo test LC-SRM.
3. LC-SRM Assay Development
- Analizzare le miscele delle norme peptidici esterni (circa 1-10 pmol di ogni peptide per iniezione) per fucile MS utilizzando un HPL nano-flowSistema C accoppiato ad uno spettrometro di massa a triplo quadrupolo (LC-MS-QQQ (/ MS)).
- Utilizzare un sistema di LC-MS dotato di una colonna capillare ricco di C-18 supporti (≤5 diametro micron, ~ 200 pori Å, lunghezza ≥10 cm, id = 50-100 micron) e una punta nano-ESI (in genere prodotto con un laser-estrattore). Assicurarsi che ogni run LC-MS include un gradiente ~ 60 min lineare (tipicamente, 0-40% Solvent B), una fase di rigenerazione della colonna (~ 80% Solvent B), e un passo di colonna riequilibrio (~ 0% solvente B ) (Solvente A = 0,1% v / v di acido formico in H 2 O, Solvente B = 0,1% v / v tasso acido formico in ACN, flusso = 200-800 nl / min, tensione ESI = 1.800 V, larghezza isolamento Q3 = 0,7 m / z, q2 pressione argon = 1,5 mTorr).
- Per ogni scansione precursore ione, selezionare dinamicamente il top ~ 10 ioni precursori più intensi per la spettrometria di massa tandem (MS / MS). Eseguire ciascun campione in duplicato in modo da utilizzare due diverse rampe collisione energetiche: una destinata a frammentare ottimale +2 ioni precursori, e l'altraper 3 precursore ioni 41.
- Eseguire la LC-QQQ-MS (/ MS) analisi utilizzando la scansione ioni Q3 precursore (Q1 scansione precursore ione può causare pedinamenti dei picchi precursore di ioni derivanti dalle collisioni argon a bassa energia in q2).
- Assicurarsi che il sistema LC-MS sta eseguendo adeguatamente analizzando campioni QC e replicati tecnici. Evitare diffusione del campione utilizzando colonne LC appena fatte e l'esecuzione di più spazi vuoti tra i campioni.
- Analizzare le fucile LC-QQQ-MS (/ MS) dei dati risultanti utilizzando database di ricerca contro le sequenze delle norme peptide esterni 42. Se del caso, eliminare identificazioni peptide ambigui con i punteggi di identificazione peptide di fiducia (ad esempio, valori di aspettativa) oppure utilizzando modelli statistici 42. Indipendentemente da ciò, rivedere manualmente tutti i identificazioni peptide 43 per garantire che essi sono tutti inequivocabili.
- Utilizzare i fucile MS identificazioni peptide di construct una biblioteca spettro utilizzando un programma software come Skyline.
- Preparare una lista di transizione LC-SRM utilizzando i 3-10 più intense transizioni per ioni precursore (+2 e +3 ioni precursori, +1 e +2 frammento ioni, y-, B, e A-ioni che sono ≥2 residui lungo).
- Scartare una transizione se i valori m / z precursore e frammento ioni sovrappongono entro la precisione di misura massa del QQQ-MS (frammentazione precursore ione è talvolta incompleta; ricordarsi di considerare la monoisotopico e le pesanti forme isotopi naturali, così come la non marcato e forme pesante marcato).
- Analogamente, scartare una transizione se il frammento ioni m / z sovrappone a quella di un altro ione frammento stesso ione precursore.
- Utilizzare gli elenchi di transizione derivanti effettuare LC-SRM analisi delle miscele delle norme peptidici esterni (eseguire la spettrometria di massa, come descritto al punto 3.1, Q1 e la larghezza isolamento Q3 = 0,7 m / z; tempo di sosta = 2-50 msec).
- Mammanualmente rivedere i risultanti dati LC-SRM ed eliminare tutti i test a basso rendimento (l'analisi dei dati LC-SRM è descritta nella sezione 5).
4. LC-SRM Saggi di campioni biologici
- Preparare i piatti di cellule in coltura 44. Se più condizioni sperimentali sono stati confrontati, blocco e casuale l'campioni per ridurre eventuali distorsioni sistematiche 45.
- Per ogni piatto di cellule, aspirare il terreno di coltura cellulare per i rifiuti e di sospendere le cellule in un siero buffer libero 44. Se necessario, lavare le cellule in un buffer siero libero per rimuovere qualsiasi proteina extracellulare rimanente.
- Per ogni campione, contare il numero di cellule vitali e totali utilizzando un colorante vitalità (es trypan blu) e un emocitometro o un contatore di cellule automatizzato 44.
- Agglomerare le cellule per centrifugazione, e aspirare il surnatante sprecare 44 (un normale carico pellet di cellule è ~ 30 microlitri).
- aggiungere 400 ml di urea Lysis Buffer o Surfactant Lysis Buffer per ogni pellet (100 mM HEPES ∙ NaOH pH 8, 10 micron Bestatin cloridrato, 10 micron pepstatina A, e sia 8 M urea o 0.1% w / v tensioattivo (rispettivamente ), preparati al momento, utilizzare un tensioattivo compatibile con MS acido labile come RapiGest SF o PPS Silenzioso Tensioattivo). Se si utilizzano altri inibitori della proteasi, in modo che essi non inibiscono tripsina.
- Mescolare ogni campione utilizzando dolce pipettaggio, e trasferire ogni in una provetta da 2 ml (con tappo a vite con un O-ring) contenente circa 100 ml di 0,1 mm di zirconia / perle di silice (si noti che le perle possono causare tubi con chiusura a scatto a perdere ). Lisare le cellule vortexando campioni per 5 min a piena velocità (questa lisa le cellule tallone battitura).
- In alternativa, lisare le cellule utilizzando un metodo meccanico diverso (ad esempio, mediante una funzione suggerimenti comunicati o omogeneizzazione francesi).
Nota: evitare di centrifugazione lisati cellulari perché ciò potrebbe pellet proteina precipitata che potrebbero otherwise essere tryptically digeriti e rilevato da LC-MS.
- In alternativa, lisare le cellule utilizzando un metodo meccanico diverso (ad esempio, mediante una funzione suggerimenti comunicati o omogeneizzazione francesi).
- Per i tensioattivi campioni denaturati, incubare a 90 ° C per 10 min per assistere omogeneizzazione del campione e denaturazione proteica.
- Bath Sonicare i campioni per 10 min a temperatura ambiente per assistere omogeneizzazione e denaturazione proteica.
Nota: Il tampone lisati e lisi può essere conservato a -80 ° C (sarà necessario il buffer di lisi per Steps 4.9 e 4.13). - Effettuare un saggio concentrazione proteica 46 (ad esempio, un test di acido bicinconinico) dei lisati (e del tampone di lisi come un esperimento di controllo).
- Per le analisi qualitative (ad esempio, non esistono norme peptidi interne saranno spillo-in dei campioni), pipetta 200 mcg (massa proteine) di lisato cellulare in un fresco 1,5 ml provetta. Per le analisi quantitative, preparare quattro questi campioni per una serie stabile diluizione isotopica.
- Per ogni campione, aggiungere un livello di proteine interna (ad esempio </ Em>, aggiungere ~ 5 pmol del ~ 98% lucciola puro luciferasi).
- Per le analisi quantitative, aggiungere una serie di diluizioni isotopo stabile della miscela equimolare di norme peptidici interni (ad esempio, 0, 0,2, 2, 20 pmol di ciascun peptide) ai campioni.
- Parallelamente, preparare campioni di controllo utilizzando solo le norme peptide interna (cioè, senza lisato cellulare) (ad esempio, 0, 0,2, 2, 20 pmol di ciascun peptide).
- Anche preparare campioni di controllo di standard di peptide esterni e interni (per esempio: 2 pmol di ciascuno standard peptide esterna, e 0, 0,2, 2, e 20 pmol di ciascuno standard peptide interno).
Nota: assicurarsi che gli standard di peptide sono privi di acido formico in quanto potrebbe interferire con la digestione tripsina.
- Aggiungere tampone di lisi in modo che tutti i campioni sono volumi identici. Nota: Un volume tipico del campione è 70 microlitri, quindi questo volume verrà utilizzato per questo protocollo.
- Ridurre i residui di proteine cisteina con l'aggiunta di 0.7 ml di 1 M DTT (preparato fresco) di ciascun campione (la concentrazione finale DTT è 10 mM) e incubando i campioni a 60 ° C per 30 min.
- Cystines alchilata proteina mediante aggiunta di 7 ml di iodoacetamide tamponata (500 mM iodoacetamide, 1 M HEPES ∙ NaOH pH 8; preparate) per ogni campione (la concentrazione finale iodoacetamide 50 mM) e incubando i campioni a temperatura ambiente per 20 min al buio (iodoacetamide è sensibile alla luce). Se necessario, spegnere la iodoacetamide restante aggiungendo DTT ad una concentrazione finale di 50 mM.
- Per ciascuno dei campioni denaturato con urea, aggiungere 482 ml di HEPES 100 mM ∙ NaOH pH 8 in modo che la concentrazione finale di urea è 1 M (tripsina è significativamente inibita a> 1 M urea 47).
- Tryptically digerire le proteine in peptidi.
- Per ogni campione contenente lisato cellulare, aggiungere 8 ml di grado sequenziamento / microlitri tripsina modificata 0,5 mg in modo che la tripsina concentrat finaleione è 01:50 (proteina w: w) tripsina: campione, e incubare il campione a 37 ° C per 18 ore.
- Per ogni campione che non contiene lisato cellulare, aggiungere 0,5 ml di 0,5 mg / mL di grado modificato tripsina sequenziamento, e incubare il campione a 37 ° C per 2 ore (ciascuno di questi campioni sono per esperimenti di controllo, e in genere contengono solo 10 ng -10 mg di peptide + massa proteica).
- Per ciascuno dei urea campioni denaturati, aggiungere 440 ml di 2% v / v di acido formico (la concentrazione finale di acido formico è 1% v / v). Verificare che questi campioni sono pH ~ 3.
- Per ciascuno dei tensioattivi campioni denaturati, aggiungere 914 ml di 0,5% v / v TFA (la concentrazione finale è TFA 0,5% v / v). Verificare che questi campioni sono pH ~ 1.5. Incubare questi campioni a 37 ° C per 60 min per idrolizzare il tensioattivo.
- Microcentrifuga tutti i campioni a 21.000 xg per 20 min a temperatura ambiente per far sedimentare gruppo tensioattivo coda e qualsiasi altro precipitates che intasare una cartuccia SPE C-18.
- Estrarre fase solida ciascuno dei supernatanti utilizzando un monouso C-18 cartuccia SPE (tampone A = 0,1% v / v di acido formico; Tampone B = 0,1% v / v di acido formico, 80% v / v ACN; ~ 100 mg C- 18 resina per cartuccia). Forzare le fasi mobili attraverso la cartuccia SPE C-18 utilizzando un bulbo di gomma piatta a superficie, utilizzando un collettore di estrazione, o mediante centrifugazione la cartuccia in una provetta da 15 ml centrifugare a 10 g per 5 min.
- Bagnare colonna applicando 1 ml di tampone B, equilibrare esso applicando 1 ml di tampone A due volte, applicare il campione, e lavare la cartuccia applicando 1 ml di tampone A due volte. Eluire i peptidi applicando 1 ml di tampone B lentamente (~ 2 min).
- Concentrato ogni eluato in un concentratore a vuoto ad un volume finale di 100 microlitri evaporare via ACN. Aggiungere 200 ml di H 2 O a ciascun campione, e concentrare ciascuno in un concentratore a vuoto ad un volume finale di 98 microlitri evaporare via ogni possibile rirestante ACN.
- Aggiungere 2 ml di 5% v / v di acido formico in ACN per ogni campione (le concentrazioni dei campioni finali sono 0,1% v / v di acido formico, 2% v / v ACN).
Nota: I campioni possono essere conservati a -80 ° C. - Analizzare i campioni utilizzando LC-SRM (come al punto 3.4). Per qualitativo LC-SRM analizza, eseguire i campioni biologici e gli standard di peptide esterni, e analizzare tutti i dati risultanti insieme. Per quantitativa LC-SRM analizza, eseguire la serie di diluizioni isotopo di ogni campione biologico, e anche eseguire gli esempi costituiti dalle norme peptide da solo, e analizzare tutti i dati risultanti insieme.
Analisi 5. LC-SRM dati
NOTA: l'identificazione e quantificazione Peptide possono essere altamente semplificate e parzialmente automatizzate utilizzando software come urbano, ma è ancora fortemente raccomandato che tutti i dati di annotazione riesaminata manualmente. Inoltre, è meglio escludere informazioni a livello di proteine durante annot manualezione dei dati LC-SRM per evitare pregiudizi.
- Per ciascun peptide identificazione, confermano che la forma di ogni profilo transizione eluizione è all'incirca gaussiano. Per ogni transizione, confermano che il profilo di eluizione è il prodotto di molteplici segnali misurati dal rivelatore MS (cioè, non è il prodotto di una o due picchi casuali di rumore MS).
- Per ogni identificazione peptide, assicurarsi che sia selezionata l'intero profilo peptide eluizione. Evitare manualmente che modifica i limiti profilo eluizione di sottocomponenti dell'identificazione peptide (la forma peptide pesante marcata, la forma non marcata, singoli ioni precursori, e singole transizioni).
- Confermare che ogni profilo transizione eluizione ha un rapporto segnale-rumore (s / n) ≥3. Nota: "Noise" si riferisce al rumore MS casuali, non riproducibile segnale da analiti coeluiscono. Cromatogramma funzioni smoothing possono notevolmente aiutare l'analisi dei dati rumorosi.
- Per ogni identificazione peptide,verificare che tutte le transizioni hanno profili di eluizione quasi identici (non solo volte all'apice del picco pari, i profili di eluizione possono avere differenti ampiezze).
- Per LC-SRM analisi di una grande quantità di norme peptidici (tipicamente ≥100 fmol di ciascun peptide), conferma che i corrispondenti profili di eluizione peptide sono molto intense (tipicamente s / n ≥100), e che le identificazioni peptide sono inequivocabili. Nota: Queste identificazioni altamente sicuri saranno fondamentali per l'identificazione dei corrispondenti peptidi derivati dal campione biologico.
- Per ogni identificazione peptide, confermano che le intensità di transizione relativi corrispondono a quelle dello standard peptide fiducia identificato. Se necessario, scartare rumorosi, relativamente bassa intensità profili di eluizione transizione, ma confermare che nessuna delle transizioni relativamente intensi mancano. Ignorare relativamente intense profili transizione eluizione che sono significativamente e inequivocabilmente colpiti da un coeluting contaminante.
- Per ogni identificazione peptide, stimare la probabilità che è stato prodotto in modo casuale dal segnale di fondo (rumore MS casuale più segnale riproducibile da analiti coeluiscono).
- Stimare questa probabilità manualmente esaminando l'intero LC-SRM cromatogramma e stimare l'unicità di ogni identificazione peptide. Produrre una serie di identificazioni peptide fiduciosi che ha un tasso stimato falsi scoperta (FDR) del 5%. Utilizzare criteri più rigorosi (per esempio, FDR ≤1%) se è necessaria una maggior fiducia insieme di identificazioni (uso di criteri più flessibile è sconsigliato).
- In alternativa, utilizzare l'algoritmo mProphet 48 o l'algoritmo AUDIT 49, ma rivedere manualmente i risultati.
- Per ciascun peptide identificazione, verificare che il tempo di eluizione osservato è coerente con l'idrofobicità del peptide (Skyline è progettato per eseguire questo calcolo).
- Per ogni peptide, confermare cheil suo tempo di eluizione è coerente tra le piste LC.
NOTA: LC-SRM analisi a volte producono profili di eluizione che vengono sistematicamente distorte tra le esecuzioni. Spostamenti sistematici sono attesi dopo le alterazioni dello strumento, come la sostituzione di una colonna. Inoltre, i peptidi presto stent possono essere soggetti a spostamento (soprattutto se la colonna viene caricata vicino alla capacità), e peptidi eluizione fine possono anche essere soggetti a spostamento. Identificazione Peptide è ora completa. - Per migliorare peptide quantificazione, reinspect ogni identificazione peptide e scartare un profilo di eluizione di transizione se è particolarmente rumoroso rispetto agli altri profili di transizione eluizione dell'identificazione peptide. Eliminare un profilo di transizione di eluizione se la sua intensità di transizione relativa è sbagliato (rispetto allo standard peptide).
- Ispezionare i confini di ciascun profilo di ioni precursore e di transizione eluizione. Se necessario, regolare con cura i confini profilo eluizione di ritagliare via segnale di fondo (o per isfondo mprove stima del segnale).
- Per ogni standard peptide interna, verificare se è contaminato con una quantità rilevabile di peptide luce (determinato dal LC-SRM analisi delle norme di peptide interni da solo, descritto in precedenza). Successivamente, eliminare eventuali profili peptide eluizione di luce che sono state significativamente compromesse dalla contaminazione luce-peptide nel rispetto degli standard di peptidi interni.
- Quantificare ogni profilo di eluizione transizione calcolando la sua area di picco LC. Se del caso, dedurre il segnale di fondo stimata. Quantificare ciascun profilo di eluizione peptide sommando i valori di transizione di quantificazione corrispondenti (qui di seguito, questo viene indicato come il valore "Sum_Peak_Area"). Per ogni valore Sum_Peak_Area campione peptide biologico, calcolare il peptide campione biologico abbondanza molare utilizzando i dati di LC-SRM di norme peptidici interni o esterni (uso solo le transizioni che sono stati quantificati con successo sia dal peptide campione biologicoLC-SRM e quella dello standard peptide).
- Quantificazione utilizzando standard peptidici esterni (non consigliato):
- Tracciare i peptidi esterno valori Sum_Peak_Area standard di rispetto dei valori di abbondanza molari standard di peptide esterno. Produrre una curva standard montando una regressione lineare per i dati (tipicamente, deve essere usato solo la componente lineare del range dinamico, qualsiasi componente non lineare deve essere usato con estrema cautela). Per ogni valore Sum_Peak_Area campione peptide biologica, utilizzare la curva standard per calcolare il peptide abbondanza molare.
Nota: questo metodo di quantificazione non è raccomandato perché richiede che la preparazione del campione e LC-SRM essere dimostrabilità robusto perché i campioni biologici e gli standard di peptide esterni sono preparati e analizzati da LC-SRM separatamente. Inoltre, non tiene conto degli effetti matrice (effetti dovuti a componenti di un campione diverso l'analita).
- Tracciare i peptidi esterno valori Sum_Peak_Area standard di rispetto dei valori di abbondanza molari standard di peptide esterno. Produrre una curva standard montando una regressione lineare per i dati (tipicamente, deve essere usato solo la componente lineare del range dinamico, qualsiasi componente non lineare deve essere usato con estrema cautela). Per ogni valore Sum_Peak_Area campione peptide biologica, utilizzare la curva standard per calcolare il peptide abbondanza molare.
- Quantificazione impiegando modelli interninorme peptide (consigliato):
- Per ogni valore Sum_Peak_Area campione peptide biologico e corrispondente peptide interno valore Sum_Peak_Area serie pesante marcato, calcolare il / pesante rapporto chiaro. Utilizzare questo rapporto come misura del corrispondente / pesante peptide rapporto di abbondanza molare luce per calcolare l'abbondanza molare del peptide campione biologico. In particolare, il peptide campione biologico abbondanza molare è uguale al peptide interno molare standard di abbondanza moltiplicato per il / pesante rapporto Sum_Peak_Area luce.
- Per ogni biologica replica e destinazione peptide, individuare l'intervallo lineare del quantificazione LC-SRM creando una curva standard utilizzando una regressione lineare. In particolare, tracciare i normali peptide interno valori di abbondanza molare rispetto ai valori pesante / leggero Sum_Peak_Area rapporto (si noti che la massa campione biologico è costante in tutto il set di dati). Richiedere che ogni campione valore peptide Sum_Peak_Area biologico è all'interno della gamma lineare (typically, deve essere utilizzato solo la componente lineare del campo dinamico; qualsiasi componente non lineare deve essere usato con estrema cautela). Allo stesso modo, eseguire questo passaggio per ogni transizione bersaglio peptide.
Nota: Analita quantificazione da LC-QQQ-SRM dovrebbe essere lineare su un ampio range dinamico (~ 10.000). Alla fine basso, segnale di fondo (rumore MS casuale più segnale riproducibile da analiti coeluiscono) ridurrà la precisione la quantificazione e la precisione. Alla fascia alta, la saturazione del rivelatore causerà non linearità (in definitiva, questo può causare profili di eluizione di essere Flat A partire da un'intensità di segnale specifico).
- Quantificazione utilizzando standard peptidici esterni (non consigliato):
- Se del caso, effettuare una normalizzazione globale di tutti i campioni, come ad esempio una tendenza normalizzazione centrale 50, per correggere leggere differenze di quantità di campione prima della spiking-in degli standard peptide interne. Se necessario, utilizzare solo le proteine di pulizia a partire dal punto 1.1.
- Se necessario, imputare mancante quantificatValori ion 51,52.
Nota: E 'improprio semplicemente sostituire i valori mancanti con la metà del limite inferiore di quantificazione (LLOQ) o metà del limite di rilevazione (LOD), perché così facendo ridurre artificiosamente la quantificazione della varianza, e potrebbe tradursi in un test statistico (ad esempio, un ANOVA ) che produce un risultato falso positivo (un errore di I tipo). Tuttavia, ignorando i valori mancanti può essere problematico. Ad esempio, se la metà dei valori di abbondanza veri sono al di sotto del LOD, e l'altra metà sono sopra il LLOQ, allora la media osservata abbondanza sarebbe sopravvalutare la vera abbondanza significa. - Assicurarsi che i saggi soddisfano le linee guida largamente accettate per LC-SRM 23-25. In particolare, richiedono che il coefficiente di variazione dei valori di quantificazione è tipicamente ≤25% per test clinici, e ≤35% per i test non clinici 25. Per i saggi LC-SRM relativi alla assistenza sanitaria o veterinari prodotti o servizi che devono essere considerati per app normativoroval, richiedono che i saggi soddisfano i requisiti rigorosi di precisione per tali dosaggi: "La precisione determinata ad ogni livello di concentrazione non deve superare il 15% del coefficiente di variazione (CV) ad eccezione del LLOQ, dove non deve superare il 20% del CV "23,24.
Risultati
Lo sviluppo di modelli computazionali predittivi di trasduzione del segnale è uno degli obiettivi fondamentali della biologia dei sistemi 53. Purtroppo, anche per vie di segnalazione che sono stati studiati estesamente e hanno un alto significato clinico, non è ancora generalmente possibile predire quantitativamente il comportamento percorso in risposta a perturbazioni (ad esempio, questo è vero per la MAPK / ERK 54). Recentemente, una ricerca impiegato proteomica mirati, trascrittomica e la modellazione computazionale e di simulazione per studiare le chemiotassi topo macrofagi segnalazione via 56. L'obiettivo della ricerca era sfingosina-1-fosfato chemiotassi mediati di RAW 264.7 cellule (una linea cellulare di topo monociti / macrofagi). Per facilitare la modellazione percorso, test LC-SRM sono stati sviluppati ed eseguiti per misurare l'abbondanza assoluta delle proteine chemiotassi pathway all'interno RAW 264.7 cellule. I valori di abbondanza risultanti erano usod come parametri del modello pathway.
Lo schema sperimentale generale (figura 1) ha avuto inizio con una lista di proteine bersaglio, che comprendeva G i2, un heterotrimeric proteina G α-subunità. Il successo del protocollo generale è fortemente dipendente dalla selezione del target peptide che sono proteotypic e quantotypic, come YDEAASYIQSK. Miscele di norme peptidici esterni sono stati preparati e analizzati da fucile LC-QQQ-MS (/ MS). Il risultante spettro di massa tandem YDEAASYIQSK fu composta da diversi frammenti di ioni, e aveva un fondo basso (Figura 2). Gli spettri sono stati usati per comporre le liste obiettivo LC-SRM contenenti i primi dieci frammenti di ioni più intensi al precursore di ioni.
Una miscela di 409 norme peptide esterni ("JPT409") è stato analizzato in triplicato da qualitative LC-SRM (dati non mostrati). Questo campione includeva tre G peptidi i2, e l'identificazione di tutti e tre eragiudicato per essere fiduciosi. Successivamente, RAW 264.7 campioni (repliche biologiche "raw1" e "raw2") e il campione sono stati JPT409 ogni analizzati in doppio (due repliche tecniche LC-SRM) da indicatori qualitativi LC-SRM (questi sei LC-SRM analisi sono state eseguite come analogamente possibile). Il peptide YDEAASYIQSK è stato identificato con sicurezza in tutte e sei le analisi (Figure 3A - C). I YDEAASYIQSK transizione modelli di intensità sono stati coerenti in tutto il sei LC-SRM analisi, e questi erano più o meno simile al fucile LC-MS (/ MS) modello (la "Library" replica della Figura 3C).
Il modello di intensità di transizione da sola non sempre è sufficiente per l'identificazione sicura di un peptide. L'idrofobia peptide e misurato il tempo di ritenzione LC devono essere coerenti. Inoltre, le norme peptide esterne e le corrispondenti peptidi campioni biologici devono avere approssimativamentetempi di ritenzione pari. La idrofobicità (stimato utilizzando la versione 3.0 100 SSRCalc algoritmo A 55) e tempo di ritenzione osservato di YDEAASYIQSK sono stati trovati ad essere coerente (figura 4A). Inoltre, il tempo di ritenzione YDEAASYIQSK stata misurata utilizzando sia i RAW 264.7 analisi e le analisi dei peptidi standard esterni, e tutti i valori di tempo di ritenzione erano approssimativamente uguali (Figura 4B).
La maggior parte delle qualitativo LC-SRM analisi dei campioni biologici hanno avuto successo, così corrispondente pesante marcato, purificato, quantificato standard peptidi interni sono stati preparati e addizionati-in RAW 264.7 lisati cellulari. Serie di diluizioni Isotope LC-SRM è stato utilizzato per analizzare campioni costituiti dalle norme peptide interno da solo (campione "R0"), il interno e le norme peptide esterni (campione "R1"), e sei RAW 264.7 repliche biologiche (campioni "R2" - "R7"). Due different denaturanti di proteine sono stati utilizzati per verificare eventuali problemi di proteine solubilizzazione, denaturazione, alchilazione, e / o di digestione che possono minare precisa quantificazione proteina bersaglio (campioni R2-R4 utilizzato urea, e campioni R5-R7 utilizzato RapiGest SF). Le forme leggeri e pesanti di YDEAASYIQSK contenevano modelli di intensità di transizione quasi identici e profili di eluizione (Figure 5A - C). LC-SRM riassunta rapporti di area di picco sono usati come misura di relativa peptide abbondanza, ei coefficienti YDEAASYIQSK stati usati per calcolare i valori G i2 abbondanza in unità di copie per RAW 264.7 cellule. Parallelamente, una seconda G i2 standard di peptide interno (IAQSDYIPTQQDVLR) è stato utilizzato per eseguire analisi quantitative G i2 LC-SRM degli stessi RAW 264.7 campioni, e le due saggi prodotto altamente simili misure G i2 abbondanza attraverso tutte le repliche biologiche (Figura 5D). L'accordo delle due G test i2 è una forte evidenza che entrambi i peptidi sono quantotypic, e che tutte le G i2 test LC-SRM quantitativi erano accurate e precise.
Nel complesso, 35 proteine sono state quantificate utilizzando 58 standard peptide interni (Tabella 1). In particolare, i test LC-SRM dello standard di proteine interno (lucciola luciferasi; Passo 4.11) erano accurate e precise. La serie completa di dati Skyline da questa indagine è disponibile presso la banca dati on-line LC-SRM Panorama (nella cartella Manes_RAW_Chemotaxis a https://panoramaweb.org/labkey/project/NIH_NitaLazar/begin.view).
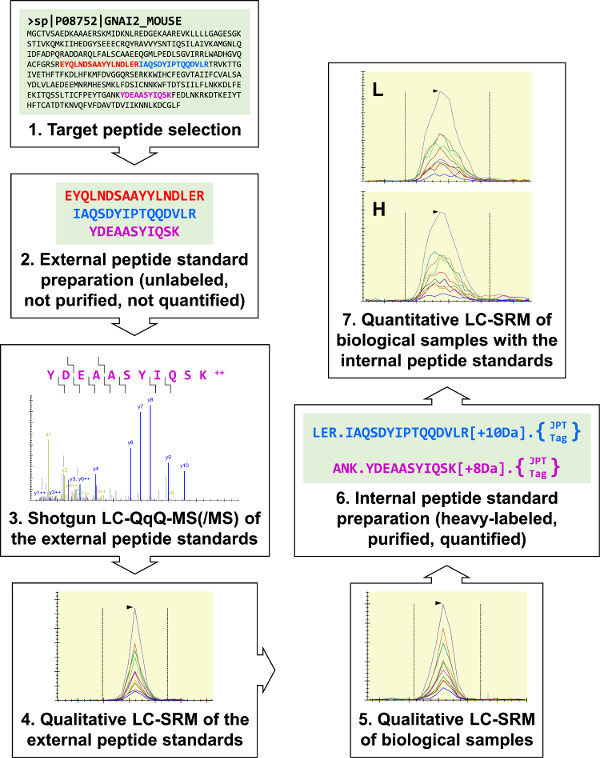
Figura 1:. Presentazione del protocollo Tre tentativi peptidi target per G i2 (un eterotrimeriche proteina G α-subunità) sono stati selezionati per il peptide esterno sintesi standard. Questi erano analyzed per fucile LC-MS (/ MS) per sviluppare tre G i2 test LC-SRM, e questi test sono stati usati per eseguire analisi qualitative dei campioni biologici. Tutti e tre i peptidi G bersaglio i2 sono stati identificati, e due sono stati selezionati per il peptide interno preparato standard. Tripsina-cleavable "JPT-Tag" sono stati utilizzati per quantificare gli standard peptide interne tramite spettrofotometria UV. Le norme peptide interni sono stati usati per eseguire quantitativi G i2 LC-SRM saggi di RAW 264.7 campioni. Cliccate qui per vedere una versione più grande di questa figura.
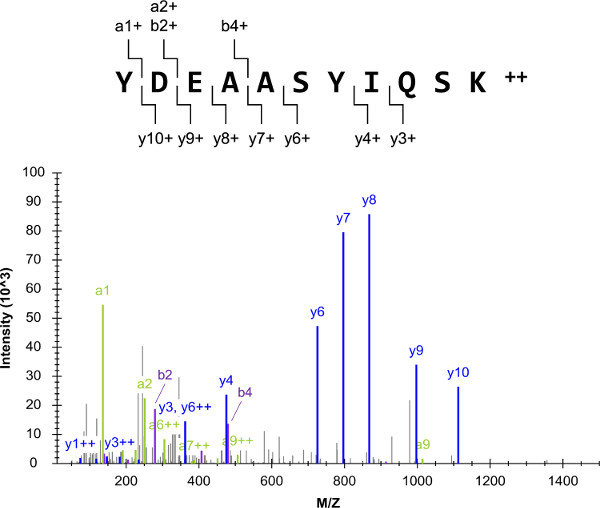
Figura 2:. Fucile LC-MS (/ MS) Lo spettro di massa raffigurata originato dalle analisi di uno dei i2 standard peptidici esterni G (YDEAASYIQSK). Per questo precursoreion, i primi dieci passaggi più intensi sono stati selezionati per LC-SRM (escludendo lo ione a1 frammento 1+ a causa della sua breve durata). Cliccate qui per vedere una versione più grande di questa figura.
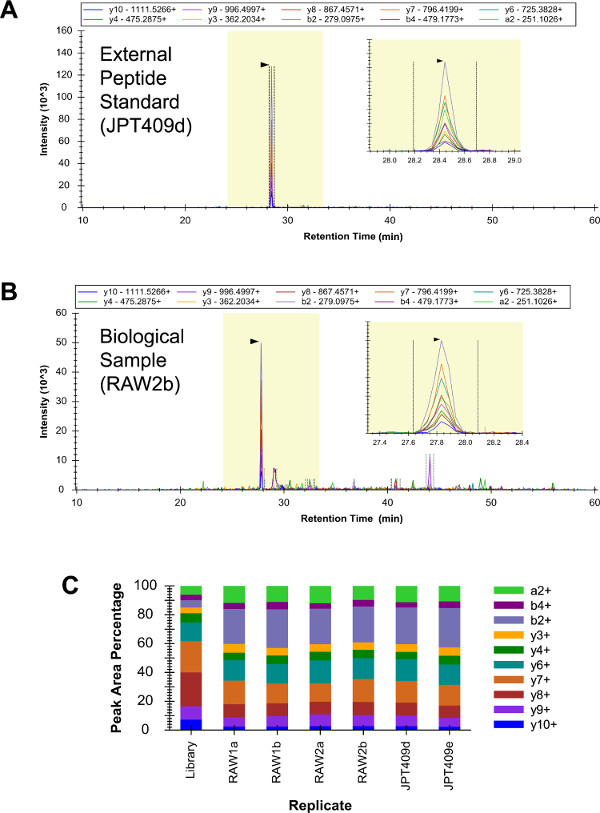
Figura 3:. Qualitative LC-SRM di G i2 Il cromatogramma YDEAASYIQSK dal JPT409 analisi contenute fondo molto basso e il peptide è stato identificato con sicurezza (A). Le corrispondenti RAW 264.7 analisi provocato più segnale di fondo, ma l'identificazione peptide era ancora priva di ambiguità (B). I relativi modelli di transizione di intensità sono stati coerenti in tutti sei LC-SRM analisi (due repliche tecniche dei tre campioni indipendenti), e questi erano circa similar al corrispondente fucile LC-MS (/ MS) modello (la "Library" replicare) (C). Cliccate qui per vedere una versione più grande di questa figura.

Figura 4: Peptide previsione tempo di ritenzione e di variazione attraverso i funzionamenti Una regressione lineare è stata calcolata usando il idrofobicità stimata e misurata LC-SRM tempo di eluizione qualitativa di tutte le peptidi di destinazione, ed i tempi di eluizione previsti e misurati YDEAASYIQSK erano coerenti (A). . Inoltre, la consistenza del tempo di eluizione osservata di ciascun peptide attraverso la LC-SRM analisi è stata determinata. I tempi di eluizione di YDEAASYIQSK riguardano una gamma di ~ 40 sec, che è coerente con la precisione dello strumento LC-SRM utilizzata (valorisono l'ora di punta apice +/- la larghezza a metà-max, e anche la larghezza alla base del picco) (B). Cliccate qui per vedere una versione più grande di questa figura.
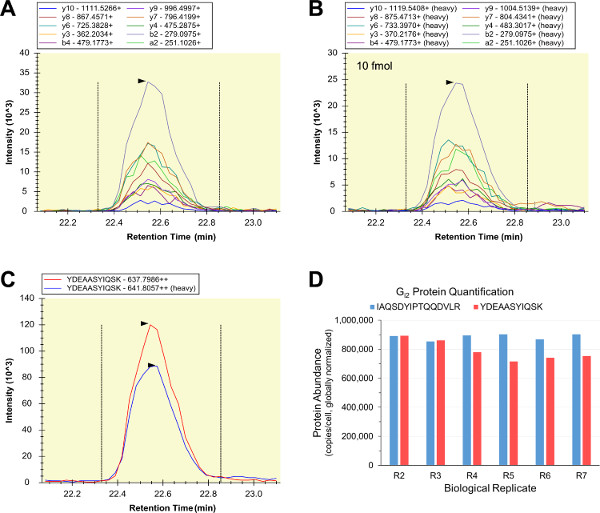
Figura 5:. Quantitative LC-SRM di G i2 I cromatogrammi di transizione del peptide campione biologico (A) e lo standard interno peptide (B) ha avuto consistenti intensità transizione relativi, e sono sommate (C) (rappresentata è l'analisi "R2" per YDEAASYIQSK utilizzando 10 fmol standard di peptide interno). I profili di eluizione leggeri e pesanti erano coerenti (C), e il rapporto tra l'area sotto queste curve è stato utilizzato come misura del ligHT / pesante abbondanza peptide. Una seconda G i2 standard di peptide interno (IAQSDYIPTQQDVLR) è stato utilizzato per quantitativa LC-SRM in parallelo, e le conseguenti valori di abbondanza G i2 fosse coerente in entrambe le sei repliche biologiche e le due peptidi bersaglio (complessivamente, n = 12 e CV = 8.38 %) (D). Cliccate qui per vedere una versione più grande di questa figura.
| UniProt adesione | Proteina bersaglio | Obiettivo Peptide | Abbondanza (fmol / mg) | Abbondanza (copie / cellula, normalizzato) | CV |
| P08659 | Luciferasi | (UV a 280 nm) | 23,824 | n / a | n / a |
| P08659 | Luciferasi | VVDLDTGK | 24,103 | n / a | 7% |
| P08659 | Luciferasi | VVPFFEAK | 24,717 | n / a | 4% |
| P60710 | Actina, citoplasmatica 1 | IWHHTFYNELR | 760,448 | 61.762.598 | 3% |
| P16858 | GAPDH | LISWYDNEYGYSNR | 357,803 | 28.906.524 | 14% |
| P06151 | Lattato deidrogenasi | LLIVSNPVDILTYVAWK | 129,623 | 10.633.145 | 30% |
| P99024 | Tubulin β 5 | ALTVPELTQQVFDAK | 78,765 | 6398971 | 3% |
| P20152 | Vimentin | SLYSSSPGGAYVTR | 121,100 | 9807488 | 10% |
| P60766 | CDC42 | DDPSTIEK | 86,647 | 6957317 | 27% |
| P60766 | CDC42 | QKPITPETAEK | 26,475 | 2153669 | 6% |
| Q8C3J5 | DOCK2 | ETLYETIIGYFDK | 1.459 | 118.539 | 15% |
| Q8C3J5 | DOCK2 | ISSSPTHSLYVFVR | 1.795 | 143.440 | 33% |
| Q8BPU7 | ELMO1 | ALTTKPSSLDQFK | 2.302 | 186.754 | 7% |
| Q8BPU7 | ELMO1 | SAIDISILQR | 1.244 | 99.728 | 25% |
| Q8BGM0 | FGR | GAYSLSIR | 1.099 | 89.473 | 17% |
| Q8BGM0 | FGR | WTAPEAALFGR | 0,319 | 24.766 | 6% |
| P27601 | Gα 13 | GIHEYDFEIK | 0,843 | 68.467 | 15% |
| P27601 | Gα 13 | VFLQYLPAIR | 1.295 | 106.176 | 23% |
| P08752 | Gα (i) 2 | IAQSDYIPTQQDVLR | 10.900 | 885.701 | 2% |
| P08752 | Gα (i) 2 | YDEAASYIQSK | 9,774 | 790.616 | 9% |
| Q9DC51 | Gα (k) | EYQLNDSASYYLNDLDR | 6,574 | 537.862 | 15% |
| Q9DC51 | Gα (k) | ISQTNYIPTQQDVLR | 3,504 | 285.784 | 10% |
| P62874 | Gβ 1 | AGVLAGHDNR | 17,078 | 1385578 | 4% |
| Q9CXP8 | Gγ 10 | DALLLGVPAGSNPFR | 2.167 | 179.178 | 36% |
| P63213 | Gγ 2 | EDPLLTPVPASENPFR | 3.284 | 266.360 | 8% |
| Q80SZ7 | Gγ 5 | VSQAAADLK | 6,087 | 493.512 | 6% |
| P08103 | HCK | GPVYVPDPTSSSK | 1.143 | 93.044 | 12% |
| P08103 | HCK | IIEDNEYTAR | 0,944 | 77.147 | 19% |
| P43406 | Integrina α V | AGTQLLAGLR | 0,276 | 22.264 | 32% |
| P43406 | Integrina α V | SHQWFGASVR | 0.443 | 34.235 | 5% |
| P25911 | LYN | VIEDNEYTAR | 1.461 | 119.377 | 13% |
| Q5SW28 | PI3K 5 normativo | AGFPGILDTASPGK | 0,301 | 24.331 | 11% |
| Q8K3B3 | PI3K α normativo | LYEEYTR | 0,472 | 38.592 | 16% |
| Q8K3B3 | PI3K α normativo | TWNVGSSNR | 0,524 | 42.697 | 12% |
| Q8K3B3 | PI3K α normativo | VLSEIFSPVLFR | 0,440 | 35.902 | 20% |
| Q5U3K7 | PI3K β normativo | DTPDGTFLVR | 0,188 | 15.262 | 30% |
| Q5U3K7 | PI3K β normativo | IAEIHESR | 0,282 | 22.561 | 13% |
| Q0VGQ5 | Α PI3K | LINLTDILK | 0,102 | 8494 | 38% |
| Q8CI98 | PI3K δ | HEVQEHFPEALAR | 0,178 | 14.566 | 22% |
| Q8CI98 | PI3K δ | ITEEEQLQLR | 0,481 | 38.709 | 24% |
| Q9ES52 | PIP3 5-fosfatasi 1 | IVVLAKPEHENR | 0,486 | 39.194 | 19% |
| Q9ES52 | PIP3 5-fosfatasi 1 | LSQLTSLLSSIEDK | 2.056 | 167.123 | 7% |
| Q69ZK0 | PIP3-dipendente Rac GEF 1 | DSVLSYTSVR | 0,647 | 52.712 | 32% |
| Q69ZK0 | PIP3-dipendente Rac GEF 1 | NQLLLALLK | 0,354 | 27.425 | 5% |
| Q9CQE5 | RGS 10 | ASSQVNVEGQSR | 2.460 | 199.782 | 4% |
| Q9CQE5 | RGS 10 | WASSLENLLEDPEGVQR | 20,647 | 206.005 | 11% |
| Q9CX84 | RGS 19 | AEANQHVVDEK | 0,495 | 39.753 | 21% |
| Q9CX84 | RGS 19 | LIYEDYVSILSPK | 0,846 | 68.481 | 22% |
| B9EKC3 | Rho GAP 5 | DGLAQELANEIR | 0.448 | 34.653 | 10% |
| Q99PT1 | Rho GDI 1 | SIQEIQELDK | 3,156 | 267.063 | 16% |
| Q99PT1 | Rho GDI 1 | VAVSADPNVPNVIVTR | 37,077 | 3006168 | 5% |
| Q61599 | Rho GDI 2 | LNYKPPPQK | 37,975 | 3086596 | 3% |
| Q61599 | Rho GDI 2 | YVQHTYR | 21,436 | 1711908 | 47% |
| Q61210 | Rho GEF 1 | FDGAEGSWFQK | 2.149 | 176.139 | 47% |
| Q61210 | Rho GEF 1 | SGLELEPEEPPGWR | 2.911 | 236.483 | 8% |
| Q9QUI0 | RhoA | QVELALWDTAGQEDYDR | 43.500 | 3532438 | 8% |
| P70336 | ROCK2 | GAFGEVQLVR | 0,527 | 42.800 | 23% |
| P70336 | ROCK2 | IYESIEEAK | 1.011 | 83.794 | 33% |
| P70336 | ROCK2 | LEGWLSLPVR | 0,573 | 48.259 | 22% |
| Q8R0X7 | S1P liasi 1 | AGYPLEKPFDFR | 1.787 | 147.043 | 27% |
| Q8R0X7 | S1P liasi 1 | TPEIVAPESAHAAFDK | 3,562 | 291.791 | 18% |
Tabella 1: LC-SRM di RAW proteine 264.7 cellulari quantitativi Trentacinque RAW 264.7 proteine cellulari sono stati quantificati con cinquantotto standard peptide interne e sei repliche biologiche.. Cinque delle proteine bersaglio sono stati PULIZIE proteine (actina, GAPDH, lattato deidrogenasi, tubulina, e vimentina), e sono stati quantificati per consentire la normalizzazione di tutti i campioni biologici (punto 1.1). Inoltre, uno standard di proteine interno è stata aggiunta, in ogni lisato cellulare e quantificata da LC-SRM (4,765 pmol della luciferasi di lucciola per 200 mg del campione; puro al 98% mediante SDS-PAGE, quantificato spettrofotometricamente a 280 nm; punto 4.11). I valori di CV sono stati calcolati attraverso le sei repliche biologiche utilizzando i valori di abbondanza globale normalizzati (ad eccezione di luciferasi; Passo 5.15).
Discussione
Proteina assoluta quantificazione è essenziale per una gamma molto diversificata di applicazioni biomediche, come la convalida biomarker e trasduzione del segnale modellazione via. Recentemente, la proteomica mirati utilizzando LC-SRM ha beneficiato di miglioramenti a numerose tecnologie, tra cui il peptide preparazione standard HPLC, QQQ-MS e LC-SRM analisi dei dati. Di conseguenza, è diventato un potente alternativa a immunodosaggi. Immunoassays possono essere estremamente sensibili e high-throughput, ma lo sviluppo di un test immunologico robusta può essere estremamente impegnativo perché immunologici possono essere vulnerabili a cross-reattività e / o interferenze, incompatibile con cellule / tessuti metodi lisi / omogeneizzazione, e / o non suscettibili di multiplexing 5,8. Ad esempio, il test più completo per il cross-reattività è quello di eseguire il immunodosaggio utilizzando campioni che provenivano da knockout genici, che può essere difficile da preparare.
Questo protocollo descrive bersaglio PEPTselezione ide, sviluppo di analisi LC-SRM, qualitativa e quantitativa LC-SRM, e LC-SRM analisi dei dati. E 'stato utilizzato per misurare l'abbondanza assoluta delle proteine pathway 36 chemiotassi entro RAW 264.7 cellule 56, ma la sua applicabilità estende molto oltre questa specifica applicazione. Anche se è stato progettato per quantificare proteine all'interno pellet cellulari, potrebbe essere regolata per l'analisi di altri campioni biologici (ad esempio, fluidi biologici) e altri obiettivi SRM (es fosfopeptidi). Ad esempio, cambiare il protocollo omogeneizzazione e / o proteine di digestione può migliorare significativamente solubilizzazione, denaturazione, alchilazione, la digestione, e la quantificazione di specie difficili proteine bersaglio (ad esempio, proteine di membrana), o può consentire l'analisi dei campioni particolarmente difficili (ad esempio, campioni contenente <100 mg massa proteica).
La selezione iniziale di peptidi bersaglio è critica, ma può richiedere molto tempo. Per hundreds di proteine bersaglio, un punteggio ad hoc potrebbero essere utilizzati per ogni criterio, e gli incerti peptidi di destinazione possono essere classificati in base alla somma dei punteggi, come è stato fatto in precedenza 56. In alternativa, questa analisi può essere automatizzato utilizzando PeptidePicker, un'interfaccia web che semplifica notevolmente la selezione del target peptide 30 (http://mrmpeptidepicker.proteincentre.com/).
Dopo che i peptidi bersaglio sono stati selezionati e sono stati sviluppati saggi LC-SRM, è fondamentale che qualitativi saggi LC-SRM dei campioni biologici essere eseguite perché anche un peptide altamente proteotypic e quantotypic non sarà rilevabile se la proteina è espressa in livelli di sotto della soglia di sensibilità dello strumento, o se l'interferenza di fondo è particolarmente problematico. Una seconda dimensione della separazione (ad esempio, forte scambio cationico HPLC, alto pH invertito fase HPLC, e senza gel elettroforesi) può aumentare la profondità di proteomica, ma richiederàmolto più tempo strumento e analisi dei dati. In alternativa, una strategia di arricchimento (ad esempio, immunoenrichment peptide- ea livello di proteine e frazionamento cellulare) può migliorare la profondità di proteomica, e immunodeplezione di proteine altamente abbondanti può essere utilizzato per ridurre le interferenze da coeluiscono analiti.
LC-SRM di ~ decine di campioni per ~ ~ centinaia o migliaia di transizioni richiedono tipicamente tempo esteso strumento. Anche se non è stato utilizzato per questo studio, la pianificazione di LC-SRM (che misura le transizioni durante le finestre pre-specificati tempo di eluizione) consente l'analisi di più transizioni per corsa. Inoltre, la programmazione riduce il SRM ciclo durante periodi relativamente vacanti del gradiente LC, e questo può provocare identificazione peptide migliorato e quantificazione. Tuttavia, la programmazione richiede che il tempo di eluizione peptide essere determinato con sicurezza da una qualitativa analisi LC-SRM. Sottili cambiamenti alla preparazione del campione, lo strumento LC-MS,o il metodo LC-MS può causare la pianificazione per ritagliare o addirittura del tutto perdere peptidi di destinazione. Ad esempio, i profili di eluizione campione YDEAASYIQSK biologici sono leggermente spostati rispetto a quelli standard peptide esterna (Figura 4B), probabilmente a causa dell'effetto matrice.
In sintesi, un protocollo passo-passo è presentato per lo sviluppo e l'applicazione di LC-SRM per le proteine quantificazione assoluta. Quantificazione assoluta di proteine da LC-SRM è stato già dimostrato di essere riproducibile tra laboratori 16,19. Tecnologie Proteomica, compresa la preparazione del campione (per esempio, l'automazione), cromatografia liquida, spettrometria di massa, e l'analisi dei dati, stanno migliorando rapidamente, e sta permettendo LC-SRM per diventare pratica per la ricerca su larga scala e applicazioni cliniche. La specificità, la sensibilità, l'accuratezza, riproducibile, e high-throughput di quantitative LC-SRM rende un potente strumento per la ricerca e la biomedicina di base.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This research was supported by the Intramural Research Program of the NIH, National Institute of Allergy and Infectious Diseases.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| BCA (bicinchoninic acid) protein assay kit | Fisher | 23235 | |
| Beads for bead beating, zirconia-silica, 0.1 mm | BioSpec Products | 11079101z | |
| Bestatin hydrochloride | Sigma | B8385-10MG | |
| Cell culture DMEM (with glucose, without L-glutamine) | Lonza | 12-614F | |
| Cell culture EDTA, 500 mM, pH8 | Gibco | 15575 | |
| Cell culture fetal bovine serum (FBS) | Atlanta Biologicals | S11550 | |
| Cell culture L-glutamine | Sigma | G8540-25G | |
| Cell culture phosphate buffered saline (PBS) pH 7.4 | Gibco | 10010-049 | |
| Cell culture Trypan Blue viability stain, 0.4% w/v | Lonza | 17-942E | |
| Cellometer Auto T4 cell counter | Nexcelom Bioscience | Cellometer Auto T4 | |
| Cellometer Auto T4 disposable counting chambers | Nexcelom Bioscience | CHT4-SD100-014 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Formic acid, LC-MS grade, ampules | Fisher | A117-10X1AMP | |
| Hemocytometer, Neubauer-improved, 0.1 mm deep | Marienfeld-Superior | 0640030 | |
| HEPES, 1 M, pH 7.2 | Mediatech | 25-060-CI | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Laser Based Micropipette Puller | Sutter Instrument Co. | P-2000 | |
| LC coated silica capillary, 50 µm id | Polymicro Technologies | 1068150017 | |
| LC vial, autosampler, 12 mm x 32 mm polypropylene | SUN SRI | 200-268 | |
| LC vial screw cap, autosampler, pre-slit PTFE/silicone | SUN SRI | 500-061 | |
| Luciferase, from Photinus pyralis | Sigma | L9506-1MG | |
| Pepstatin A | EMD Millipore | 516481-25MG | |
| pH strips colorpHast (pH 0.0-6.0) | EMD Chemicals | 9586-1 | |
| PhosStop phosphatase inhibitor cocktail | Roche | 04906837001 | |
| RapiGest SF | Waters | 186001861 | |
| Sep-Pak SPE, C18 1 ml 100 mg cartridge | Waters | WAT023590 | |
| Sep-Pak SPE, extraction manifold, 20 position | Waters | WAT200609 | |
| Sep-Pak SPE, flat-surfaced rubber bulb | Fisher | 03-448-25 | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| SpeedVac vacuum concentrator | Fisher | SPD111V | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Tube decapper for Micronic tubes | USA Scientific | 1765-4000 | |
| Tubes, 2 ml microcentrifuge, o-ring screw-cap, sterile | Sarstedt | 72.694.006 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS grade | Fisher | W6-1 |
Riferimenti
- Cox, J., Mann, M. Quantitative high-resolution proteomics for data-driven systems biology. Annu Rev Biochem. 80, 273-299 (2011).
- Zhang, Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 113, 2343-2394 (2013).
- Boja, E. S., Rodriguez, H. Mass spectrometry-based targeted quantitative proteomics: achieving sensitive and reproducible detection of proteins. Proteomics. 12, 1093-1110 (2012).
- Gillette, M. A., Carr, S. A. Quantitative analysis of peptides and proteins in biomedicine by targeted mass spectrometry. Nat Methods. 10, 28-34 (2013).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nat Methods. 9, 555-566 (2012).
- Lesur, A., Domon, B. Advances in high-resolution accurate mass spectrometry application to targeted proteomics. Proteomics. , (2015).
- Wild, D. . The immunoassay handbook : theory and applications of ligand binding ELISA., and related techniques. , (2013).
- Sturgeon, C. M., Viljoen, A. Analytical error and interference in immunoassay: minimizing risk. Ann Clin Biochem. 48, 418-432 (2011).
- Adrait, A., et al. Development of a Protein Standard Absolute Quantification (PSAQ) assay for the quantification of Staphylococcus aureus enterotoxin A in serum. J Proteomics. 75, 3041-3049 (2012).
- Lin, D., Alborn, W. E., Slebos, R. J., Liebler, D. C. Comparison of protein immunoprecipitation-multiple reaction monitoring with ELISA for assay of biomarker candidates in plasma. J Proteome Res. 12, 5996-6003 (2013).
- Weiss, F., et al. Catch and measure-mass spectrometry-based immunoassays in biomarker research. Biochim Biophys Acta. 1844, 927-932 (2014).
- Yassine, H., et al. Mass spectrometric immunoassay and MRM as targeted MS-based quantitative approaches in biomarker development: potential applications to cardiovascular disease and diabetes. Proteomics Clin Appl. 7, 528-540 (2013).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. , (2011).
- Becker, J. O., Hoofnagle, A. N. Replacing immunoassays with tryptic digestion-peptide immunoaffinity enrichment and LC-MS/MS. 4, 281-290 (2012).
- Wasinger, V. C., Zeng, M., Yau, Y. Current status and advances in quantitative proteomic mass spectrometry. Int J Proteomics. 2013, 180605 (2013).
- Abbatiello, S. E., et al. Large-scale inter-laboratory study to develop, analytically validate and apply highly multiplexed, quantitative peptide assays to measure cancer-relevant proteins in plasma. Mol Cell Proteomics. , (2015).
- Rodriguez-Suarez, E., Whetton, A. D. The application of quantification techniques in proteomics for biomedical research. Mass Spectrom Rev. 32, 1-26 (2013).
- Wehr, A. Y., Hwang, W. T., Blair, I. A., Yu, K. H. Relative quantification of serum proteins from pancreatic ductal adenocarcinoma patients by stable isotope dilution liquid chromatography-mass spectrometry. J Proteome Res. 11, 1749-1758 (2012).
- Kennedy, J. J., et al. Demonstrating the feasibility of large-scale development of standardized assays to quantify human proteins. Nat Methods. 11, 149-155 (2014).
- Jensen, K. J., Shelton, P. T., Pedersen, S. L. . Peptide synthesis and applications. , (2013).
- Pratt, J. M., et al. Multiplexed absolute quantification for proteomics using concatenated signature peptides encoded by QconCAT genes. Nat Protoc. 1, 1029-1043 (2006).
- Brun, V., et al. Isotope-labeled protein standards: toward absolute quantitative proteomics. Mol Cell Proteomics. 6, 2139-2149 (2007).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2001).
- . . Guidance for Industry: Bioanalytical Method Validation. , (2013).
- Carr, S. A., et al. Targeted peptide measurements in biology and medicine: best practices for mass spectrometry-based assay development using a fit-for-purpose approach. Mol Cell Proteomics. 13, 907-917 (2014).
- Ori, A., Andres-Pons, A., Beck, M. The use of targeted proteomics to determine the stoichiometry of large macromolecular assemblies. Methods Cell Biol. 122, 117-146 (2014).
- Rosen, O., Feldberg, L., Gura, S., Zichel, R. A new peptide substrate for enhanced botulinum neurotoxin type B detection by endopeptidase-liquid chromatography-tandem mass spectrometry/multiple reaction monitoring assay. Anal Biochem. , (2015).
- Feng, Y., et al. Global analysis of protein structural changes in complex proteomes. Nat Biotechnol. 32, 1036-1044 (2014).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Mohammed, Y., et al. PeptidePicker: a scientific workflow with web interface for selecting appropriate peptides for targeted proteomics experiments. J Proteomics. 106, 151-161 (2014).
- Rodriguez, J., Gupta, N., Smith, R. D., Pevzner, P. A. Does trypsin cut before proline. J Proteome Res. 7, 300-305 (2008).
- Min, X. J., Butler, G., Storms, R., Tsang, A. OrfPredictor: predicting protein-coding regions in EST-derived sequences. Nucleic Acids Res. 33, W677-W680 (2005).
- Lam, H., et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods. 5, 873-875 (2008).
- Craig, R., Cortens, J. P., Beavis, R. C. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res. 3, 1234-1242 (2004).
- Desiere, F., et al. The PeptideAtlas project. Nucleic Acids Res. 34, D655-D658 (2006).
- Vizcaino, J. A., et al. The PRoteomics IDEntifications (PRIDE) database and associated tools: status in 2013. Nucleic Acids Res. 41, D1063-D1069 (2013).
- Frank, R. The SPOT-synthesis technique. Synthetic peptide arrays on membrane supports--principles and applications. J Immunol Methods. 267, 13-26 (2002).
- Ong, S. E., Kratchmarova, I., Mann, M. Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). J Proteome Res. 2, 173-181 (2003).
- Mant, C. T., et al. HPLC analysis and purification of peptides. Methods Mol Biol. 386, 3-55 (2007).
- Alterman, M. A., Hunziker, P. . Amino acid analysis : methods and protocols. , (2012).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Nesvizhskii, A. I. A survey of computational methods and error rate estimation procedures for peptide and protein identification in shotgun proteomics. J Proteomics. 73, 2092-2123 (2010).
- Tabb, D. L., Friedman, D. B., Ham, A. J. Verification of automated peptide identifications from proteomic tandem mass spectra. Nat Protoc. 1, 2213-2222 (2006).
- Freshney, R. I. . Culture of animal cells : a manual of basic technique and specialized applications. , (2010).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. J Proteome Res. 8, 2144-2156 (2009).
- Noble, J. E., Bailey, M. J. Quantitation of protein. Methods Enzymol. 463, 73-95 (2009).
- Kiser, J. Z., Post, M., Wang, B., Miyagi, M. Streptomyces erythraeus trypsin for proteomics applications. J Proteome Res. 8, 1810-1817 (2009).
- Reiter, L., et al. mProphet: automated data processing and statistical validation for large-scale SRM experiments. Nat Methods. 8, 430-435 (2011).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Callister, S. J., et al. Normalization approaches for removing systematic biases associated with mass spectrometry and label-free proteomics. J Proteome Res. 5, 277-286 (2006).
- Karpievitch, Y. V., Dabney, A. R., Smith, R. D. Normalization and missing value imputation for label-free LC-MS analysis. BMC Bioinformatics. 13, S5 (2012).
- Oh, S., Kang, D. D., Brock, G. N., Tseng, G. C. Biological impact of missing-value imputation on downstream analyses of gene expression profiles. Bioinformatics. 27, 78-86 (2011).
- Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A., Fraser, I. D. Systems biology in immunology: a computational modeling perspective. Annu Rev Immunol. 29, 527-585 (2011).
- Futran, A. S., Link, A. J., Seger, R., Shvartsman, S. Y. ERK as a model for systems biology of enzyme kinetics in cells. Curr Biol. 23, R972-R979 (2013).
- Krokhin, O. V. Sequence-specific retention calculator. Algorithm for peptide retention prediction in ion-pair RP-HPLC: application to 300- and 100-A pore size C18 sorbents. Anal Chem. 78, 7785-7795 (2006).
- Manes, N. P., Angermann, B. R., Koppenol-Raab, M., An, E., Sjoelund, V. H., Sun, J., Ishii, M., Germain, R. N., Meier-Schellersheim, M., Nita-Lazar, A. Targeted Proteomics-Driven Computational Modeling of Macrophage S1P Chemosensing. . Mol Cell Proteomics. , .
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon